扩展功能
文章信息
- 吕鹏翼, 罗金学, 韩振飞, 黄占斌
- LÜ Peng-Yi, LUO Jin-Xue, HAN Zhen-Fei, HUANG Zhan-Bin
- 生物膜法强化净化氨氮污染水体及其微生物群落解析
- Purification of ammonia-polluted water through biofilm augmentation:operating conditions and key microbial community
- 微生物学通报, 2017, 44(9): 2055-2066
- Microbiology China, 2017, 44(9): 2055-2066
- DOI: 10.13344/j.microbiol.china.170115
-
文章历史
- 收稿日期: 2017-02-14
- 接受日期: 2017-04-11
- 优先数字出版日期(www.cnki.net): 2017-04-20
2. 中国科学院生态环境研究中心 中国科学院环境生物技术重点实验室 北京 100085
2. CAS Key Laboratory of Environmental Biotechnology, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China
近年来,地表水体中氨氮浓度远超出受纳水体的自净能力,已成为我国水环境保护面临的一大难题。环境保护部公布的2016年上半年地表水环境质量状况显示,全国地表水环境质量监测网1 907个断面中,劣Ⅴ类断面所占比例为10.5%,其中,氨氮为主要污染指标之一。依据2015年《环境统计年报》,我国水环境中氨氮排放源主要为城镇生活污水(70.12%)、农业面源污染(24.93%)、集中式治理设施排放(3.09%)及工业废水点源污染(1.86%)。水体中氨氮污染不仅会引起富营养化等一系列环境问题[1],同时也是威胁人类健康的一大隐患[2]。
生物膜技术以其修复时间短、净化效果好等优势逐步成为原位修复的研究热点[3]。当前应用生物膜技术原位修复污染水体在我国已有一定的研究基础[4-6],但是关于预固定脱氮菌的生物膜技术在污染水体处理中的应用及生物膜技术修复过程中微生物群落多样性的研究鲜有报道。在原位修复过程中由于各环境因素的制约,在实际应用前进行小试研究,衡量生物修复技术的可行性不可或缺。末端限制性片段长度多态性(Terminal restriction fragment length polymorphism,T-RFLP)是通过对荧光标记的PCR扩增产物进行限制性酶切后,通过荧光检测得到限制性末端片段(T-RFs)[7]。理论上每一种菌对应一个T-RF,一个T-RF可认为是一个操作分类单元(OTU),每个T-RF的相对峰面积代表该T-RF的相对数量,通过与相应数据库分析可获得T-RF对应的菌种信息,因此可以根据T-RFs的多样性对细菌群落结构快速进行初步的定量、定性分析[8]。
本研究以自然成膜的生物膜反应器和预固定脱氮菌的生物膜反应器为研究对象,考察不同营养条件下两种挂膜方式的生物膜反应器的脱氮效果,研究附菌强化挂膜技术的可行性,并基于T-RFLP技术获得的大量T-RFs对不同挂膜方式的成膜过程及微生物群落组成进行考察,以期为原位生物膜修复技术的应用提供一种新的思路及理论依据。
1 材料与方法 1.1 主要试剂和仪器 1.1.1 主要试剂: T-RFLP所用引物27F-FAM (5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R (5′-G GTTACCTTGTTACGACTT-3′),北京睿博兴科生物技术有限公司;2×Taq PCR Master Mix,北京博迈德基因技术有限公司;琼脂糖凝胶回收试剂盒,OMEGA Bio-Tek公司;限制性内切酶Rsa Ⅰ,NEB公司。 1.1.2 主要仪器: PCR仪,德国Eppendorf公司;凝胶成像仪,美国Bio-Rad公司;NanoDrop分光光度计,美国Thermo Scientific公司。 1.2 生物膜反应器模拟装置设置3组对照实验,(1) 空白组(Blank):未安装空载填料,未添加菌剂;(2) 自然成膜组(Raw):安装填料,未附菌;(3) 预附脱氮菌强化挂膜组(PCC):安装填料并预附脱氮副球菌Paracoccus denitrificans PD1222。载体填料为生物膜接触氧化的核心构件,实验填料选用绳索状聚丙烯纤维,其比表面积为2 580 m2/m3。启动前,分别将空载的聚丙烯纤维填料和附菌的填料垂直于水体进行排布,填料纤维绳浸没并悬浮于水体中,同时在填料纤维绳悬浮端底布设置曝气点进行人工曝气。每个反应器的有效容积为50 L;3个反应器底部均铺设厚度约为1-2 cm的底泥,反应器初始水体及底泥取自于凉水河小红门污水处理厂出水口下游500 m处,实验前在实验室条件下驯化30 d。反应器进水为人工合成污水,氨氮含量为5 mg/L。污水在蠕动泵作用下进入3个反应器,Raw与PCC中污水经生物膜处理后排出,模拟装置如图 1所示。在不同C/N条件下反应器以连续流方式连续运行60 d,其中1-25 d的C/N=1:1 (5 mg/L TOC : 5 mg/L NH4+-N),26-60 d的C/N=2:1 (10 mg/L TOC : 5 mg/L NH4+-N);整个运行过程中反应器水力停留时间(HRT)均为21.6 h,溶解氧(DO)控制在3-5 mg/L,pH波动范围为7.0-8.0。实时监测反应器对氨氮、硝态氮、亚硝态氮的消减情况,比较生物膜反应器(Raw、PCC)与水体自净(Blank)对含氮污水的处理能力。
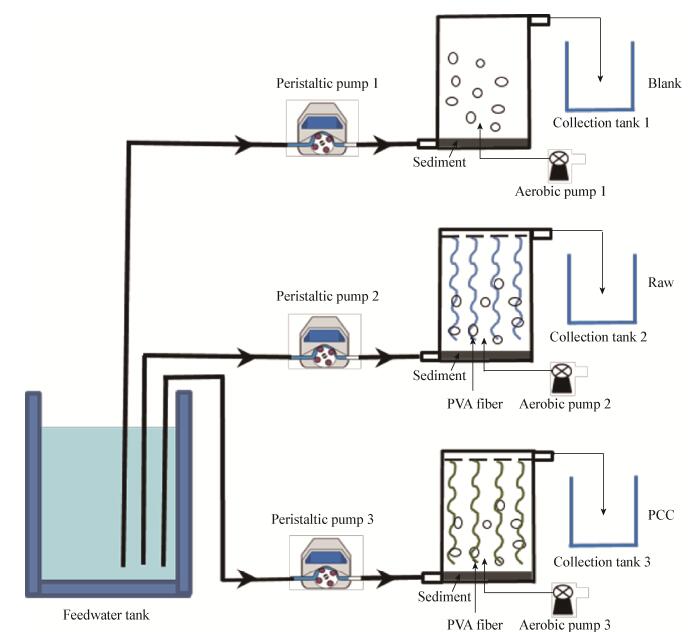
|
| 图 1 生物膜反应器模拟示意图 Figure 1 Simulation diagram of the biofilm reactors 注:Blank:空白组反应器;Raw:自然成膜反应器;PCC:预附脱氮菌强化挂膜反应器. Note: Blank: The control reactor; Raw: The biofilm reactor with the raw carrier; PCC: The biofilm reactor with the functional bacteria-immobilized carrier. |
|
|
在反应器整个运行过程中共取样5次,采样位点为反应器的水体和生物膜,每个位点均取3个平行样品。水样从反应器出水口取水1 L,用0.45 μm水系微孔滤膜过滤收集水体中菌体;生物膜样直接从成膜的载体填料上获取。分别在第1、8、16天(C/N=1:1) 及第30、60天(C/N=2:1) 时采集样品提取DNA。
1.4 样品总DNA提取样品中微生物总DNA用CTAB-PEG方法提取[9-10]。用NanoDrop分光光度计测定各样品DNA浓度及提取质量,DNA样品A260/A280比值均在1.8-2.0范围内,A260/A230比值均在1.9-2.1范围内,表明提取的DNA质量较高。DNA样品于-80 ℃保存。
1.5 T-RFLP分析 1.5.1 目的片段16S rRNA基因PCR: 选择具有荧光标记的引物27F-FAM和1492R对16S rRNA基因进行特异性扩增[11]。50 μL PCR反应体系:2×Taq PCR Master Mix (不含染料) 25 μL,27F-FAM (10 μmol/L) 1 μL,1492R (10 μmol/L) 1 μL,DNA模板(10 mg/L) 1 μL,ddH2O 22 μL。PCR反应条件:95 ℃ 5 min;94 ℃ 1 min,50 ℃ 45 s,72 ℃ 1.5 min,30个循环;72 ℃ 10 min。使用OMEGA琼脂糖凝胶回收试剂盒对PCR产物进行切胶回收。 1.5.2 PCR产物限制性酶切: 选用识别位点为4个碱基(5′-GTAC-3′)的限制性内切酶Rsa Ⅰ对纯化的PCR产物进行酶切。酶切消化反应体系(20 μL):10×NEB buffer 2 μL,Rsa Ⅰ (10 units/μL) 0.5 μL,PCR纯化产物15 μL,ddH2O 2.5 μL。37 ℃酶切4 h。利用凝胶电泳成像验证酶切片段质量,将酶切产物经过毛细管电泳检测分析得到T-RFLP图谱。酶切产物由北京睿博兴科生物技术有限公司进行基因扫描(Gene scan)。 1.6 数据分析T-RFLP图谱用Gene marker V 2.2.0软件(Soft Genetics,USA)进行处理,内标选择GS1200LIZ,分析片段长度范围设置为60–1 000 bp,去掉相对丰度小于1%的T-RFs[12]。将通过T-RFLP分析所得的T-RFs与微生物群落分析数据库MiCA进行在线比对(http://mica.ibest.uidaho.edu/),得出T-RFs可能代表的菌群[13]。
考察一个群落或生境中物种多样性一般从物种丰富度(species richness)和均匀度(Evenness)进行综合评估[14]。(1) S代表直观可统计的物种数(The observed species numbers)[14]。(2) 香农指数(Shannon index,H')[15]反映了一个群落物种的多样性程度,其数值越大代表该群落物种多样性越高,香农指数对物种的丰富度更为敏感。(3) 均匀度指数(Pielou’s evenness index,J')[16]用于评估一个群落中各物种数目分配的均匀程度。多样性指数采用Primer 6.0软件分析。
香农指数计算公式:

|
均匀度指数计算公式:
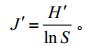
|
Pi为种i的个体数在群落总个体数中所占的比例,S为总物种数。
不同反应器不同生态位微生物群落动态变化过程主要用NMDS展示,由Primer 6.0完成。群落间的相似性用Bray-Curtis指数表征[17]。不同群落之间的差异性分析通过Primer 6.0中的“SIMPER”命令完成[17]。
2 结果与分析 2.1 生物膜反应器对氨氮的去除效果各反应器连续运行期间氨氮消减变化如图 2所示。反应器运行初期(1–10 d),Blank和Raw的氨氮去除率分别为0–5%和0–10%,PCC的NH4+-N去除率逐渐升高,在第5天时达到最高为70%,此时填料上所附的Paracoccus denitrificans PD1222 (异养硝化-好氧反硝化模式菌株)起主要的作用。11–25 d运行过程中,Blank、Raw和PCC三组反应器的氨氮去除率均在13%–18%范围内。第25天提高碳氮比后,3组反应器氨氮去除率不同程度提高,C/N由1:1改为2:1反应器运行24 h后Blank、Raw和PCC的NH4+-N去除率分别为37.81%、82.83%和66.63%。随着微生物生物量增大及生物膜的成熟,在第46天时Blank的氨氮去除率为90%,达到《地表水环境质量标准(GB 3838-2002)》Ⅱ类水水质(0.5 mg/L)标准,而两个挂膜反应器Raw与PCC仅在运行第29天时即达到相同处理效果,此时的硝化作用可能主要由异养微生物主导。由运行结果可知生物膜反应器具有较好的氨氮转化性能,且相比于水体自净(Blank)大大缩短了,达到同一处理效率的时间。反应器运行稳定后Raw与PCC的氨氮转化率均达到95%以上,生物膜的处理能力可能超过了污染负荷;同时应注意到运行后期Blank同样具有较高的氨氮转化率,推测原因可能是实验的HRT较长,导致Blank中功能微生物生物量的累积达到了可以处理进水中的氨态氮的浓度。在整个运行过程中随着NH4+-N的去除NO3--N有一定程度的积累,NO2--N无显著累积。
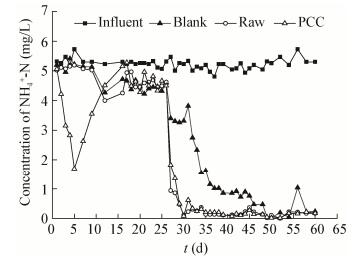
|
| 图 2 生物膜反应器运行过程中NH4+-N消减情况 Figure 2 The variation of NH4+-N concentration during the operation of the biofilm reactors |
|
|
基于T-RFLP结果对3个反应器中细菌群落结构进行分析。不同运行条件下3个反应器水体中共检测到优势片段17个,Raw和PCC的生物膜中共检测到优势片段19个(表 1)。不同碳氮比条件下不同生态位(水体与生物膜)细菌群落结构差异性较大。将T-RFLP分析所得的22个T-RFs与MiCA平台的细菌16S rRNA基因RDP数据库比对(表 1),结果表明在3个反应器中门水平上的细菌主要有变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、硝化螺旋菌门(Nitrospirae)、浮霉菌门(Planctomycetes)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)及芽单胞菌门(Gemmatimonadetes)。
| T-RFs长度 T-RFs length (bp) |
相似菌株 The similar strain |
NCBI登录号 NCBI accession No. |
门水平分类 Phylum level classification |
| 67a & b | Micromonosporaceae bacterium YIM 65646 | GU367156 | Actinobacteria |
| 114a & b | uncultured Planctomyces sp. A05-07C | FJ542858 | Planctomycetes |
| 116a & b | uncultured Xanthobacter sp. A03-02E | FJ542831 | Proteobacteria |
| 122a & b | uncultured Hydrogenophilaceae bacterium | EU266782 | Proteobacteria |
| 149a | uncultured Gemmatimonas sp. X-18 | HQ132388 | Gemmatimonadetes |
| 415a & b | uncultured Acidobacteria bacterium XA2A0 | FJ269265 | Acidobacteria |
| 420b | Paracoccus denitrificans PD1222 | CP000489 | Proteobacteria |
| 423a & b | Rhodobacter sp. TCRI | AB017798 | Proteobacteria |
| 426a & b | uncultured Comamonadaceae bacterium D2514 | EU266893 | Proteobacteria |
| 447b | Brevibacillus sp. AV-Pb | AJ457160 | Firmicutes |
| 463a & b | uncultured Nitrospiraceae bacterium D2514 | EU266889 | Nitrospirae |
| 467a & b | uncultured Rhodocyclaceae bacterium 48 | HQ184352 | Proteobacteria |
| 472a & b | Burkholderiaceae bacterium KVD-1982-1 | DQ490286 | Proteobacteria |
| 482b | Pseudoxanthomonas sp. M1-3 | AB039330 | Proteobacteria |
| 564a | Pseudoalteromonas sp. AS-43 | AJ391204 | Proteobacteria |
| 635a | Mycobacterium tuberculosis TB36 | AM283534 | Actinobacteria |
| 641a | Uncultured Pseudomonadaceae bacterium B02-07B | FJ542959 | Proteobacteria |
| 744a & b | Acinetobacter sp. BD189 | HM120259 | Proteobacteria |
| 821a & b | Rhizobium sp. TJ171 | AJ505298 | Proteobacteria |
| 860a | Corynebacterium sp. ATCC 43833 | AF262996 | Actinobacteria |
| 867a & b | Allochromatium vinosum DSM 180 | CP001896 | Proteobacteria |
| 874a & b | Pseudomonas sp. JT08 | AB554725 | Proteobacteria |
| 注:a:存在于水体中的限制性末端片段;b:存在于生物膜中的限制性末端片段;a & b:水体和生物膜中均存在的限制性末端片段. Note: a: T-RFs in water samples; b: T-RFs in biofilm samples; a & b: T-RFs both in water and biofilm samples. |
|||
微生物多样性指数从不同角度反映了微生物群落结构的多样性。改变C/N后,3组反应器水体和生物膜中微生物群落结构均发生了显著变化(表 2)。(1) 物种数S。高碳氮比条件(C/N=2:1) 下3个反应器水体中物种数目均显著降低。反应器水体中的微生物主要来自于凉水河的水体和底泥,反应器水体中物种丰度减少可能是由于某些土著微生物不适应反应器中的生态环境从而被适应性更强的微生物所演替。此外,相比于浮游状态微生物更易在载体上富集形成生物膜,因此Raw与PCC水体中微生物种类减少也可能是部分易成膜微生物倾向于定殖于载体填料,该结果与香农指数H′相一致。(2) 香农指数H′。富营养条件(C/N=2:1) 下RB (Raw biofilm)和PB (PCC biofilm)的香农指数显著上升,表明生物膜中微生物种类更为丰富,更多的微生物吸附定殖于载体填料。(3) 均匀度J′。PB在寡营养条件(C/N=1:1) 下物种均匀度指数范围为0.5–0.6,富营养条件下物种均匀度指数范围为0.8–0.9,PB中优势微生物由单一的Paracoccus denitrificans PD1222演变成多种优势物种共存,表明填料表面预固定的外源微生物的生长受到了土著微生物物种的竞争性抑制。
| 样品 Samples |
S | H′ | J′ |
| BW-1 | 13 | 2.836 | 0.766 4 |
| BW-8 | 15 | 3.225 | 0.825 5 |
| BW-16 | 11 | 2.557 | 0.739 1 |
| BW-30 | 10 | 2.935 | 0.883 6 |
| BW-60 | 7 | 2.550 | 0.908 3 |
| RW-1 | 13 | 2.769 | 0.748 2 |
| RW-8 | 14 | 2.758 | 0.724 5 |
| RW-16 | 10 | 2.708 | 0.815 1 |
| RW-30 | 12 | 2.818 | 0.786 1 |
| RW-60 | 8 | 2.442 | 0.814 0 |
| PW-1 | 12 | 2.402 | 0.670 0 |
| PW-8 | 12 | 2.855 | 0.796 3 |
| PW-16 | 11 | 2.957 | 0.854 9 |
| PW-30 | 8 | 2.511 | 0.837 2 |
| PW-60 | 8 | 2.245 | 0.748 4 |
| RB-8 | 7 | 2.595 | 0.924 3 |
| RB-16 | 8 | 2.514 | 0.838 0 |
| RB-30 | 12 | 3.195 | 0.891 3 |
| RB-60 | 13 | 3.455 | 0.933 7 |
| PB-8 | 4 | 1.180 | 0.589 9 |
| PB-16 | 6 | 1.488 | 0.575 7 |
| PB-30 | 11 | 3.019 | 0.872 7 |
| PB-60 | 10 | 2.758 | 0.830 3 |
| 注:BW (Blank water):空白组水样;RW (Raw water):自然挂膜组水样;PW (PCC water):附菌强化挂膜组水样;RB (Raw biofilm):自然挂膜组生物膜样;PB (PCC biofilm):附菌强化挂膜组生物膜样. -1、-8、-16、-30、-60:取样所对应的反应器运行天数. Note: BW: the water samples in Blank; RW represents the water samples in Raw; PW: the water samples in PCC; RB: the biofilm samples in Raw; PB: the biofilm samples in PCC. -1, -8, -16, -30, -60: the sampling time (day) of the reactors operation. |
|||
理论上不同营养条件下同一反应器中微生物群落结构不同,为更好地解析3组反应器在不同碳氮比环境下水体中微生物种群演变过程,依据Bray-curtis相似性(Bray-curtis similarity)计算不同组间微生物群落的差异度,并根据不同水样中T-RFs的峰面积进行NMDS分析。
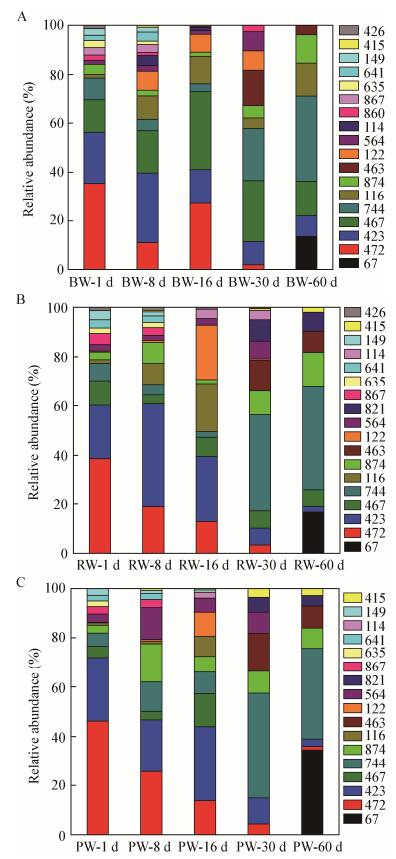
NMDS表明不同碳氮比条件下3个反应器中微生物群落组成差异较大(图 3和图 4)。初始运行阶段BW、RW及PW中的微生物群落构成相差不大,均主要来自经过驯化的凉水河水体和底泥,典型优势片段主要有472、423、122、116、467 bp等。第1天样品组BW-1和RW-1、BW-1和PW-1、RW-1和PW-1之间的差异度分别为8.89%、12.34%、10.60%。在第25天将C/N由1:1改为2:1后3个反应器水体中微生物种群结构开始发生显著变化(图 3),67、463、744、821、874 bp等T-RFs开始富集。30 d时样品组BW-30和RW-30、BW-30和PW-30、RW-30和PW-30的差异度相比于第1天时均显著增大,分别为33.06%、44.52%、27.65%,相较于空白组,自然成膜的生物膜反应器和预固定脱氮菌的生物膜反应器相似度更高,说明空白组比挂膜组水体中的微生物群落变动性更大。第60天时样品组BW-60和RW-60、BW-60和PW-60、RW-60和PW-60之间的差异度分别为21.52%、37.90%、22.70%,此时反应器运行稳定,相较于第30天时3个反应器水体中微生物群落结构差异显著性降低,差异性最大的是BW-60和PW-60,可能是因为PCC填料中最初所附的Paracoccus denitrificans PD1222对PW中的微生物群落组成有一定影响。此外,P. denitrificans PD1222所对应的片段长度为420 bp,但是在整个运行过程中P. denitrificans PD1222并未在PW中富集,该结果说明在生物修复中单纯的应用外源纯菌难以形成优势菌[18]。
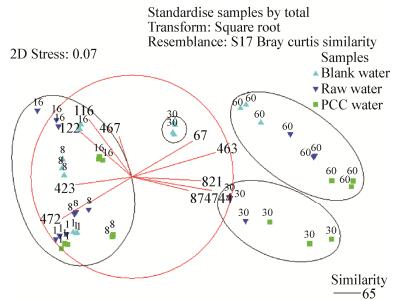
|
| 图 3 不同反应器水体中细菌群落NMDS分析 Figure 3 The NMDS plot of bacterial communities for water samples in different reactors |
|
|
|
| 图 4 不同反应器水体中T-RFs相对丰度 Figure 4 Relative abundance of T-RFs in the water niche of different reactors 注:A:空白反应器(Blank)运行过程中水体中T-RFs的相对丰度;B:自然成膜反应器(Raw)运行过程中水体中T-RFs的相对丰度;C:预附脱氮菌强化挂膜反应器(PCC)运行过程中水体中T-RFs的相对丰度. Note: A: The relative abundance of T-RFs in the water niche during the operation of Blank; B: The relative abundance of T-RFs in the water niche during the operation of Raw; C: The relative abundance of T-RFs in the water niche during the operation of PCC. |
|
|
结果表明,C/N是3个反应器水体中微生物群落结构发生演替的主要驱动因子。实验对3个反应器不同营养条件下水体中微生物群落进行了调查,根据不同反应器水体中T-RFs相对丰度(图 4)得出以下结论:423 bp (红细菌目,Rhodobacterales)和472 bp (伯克氏菌目,Burkholderiales)均为寡营养条件下的优势菌,但是在高碳氮比下其优势地位被取代;富营养条件下,744 bp (假单胞菌目,Pseudomonadales)、67 bp (放线菌纲,Actinobacteria)、874 bp (假单胞菌目,Pseudomonadales)和463 bp (硝化螺旋菌目,Nitrospirales)为3个反应器水体中同时富集的菌;116 bp (根瘤菌目,Rhizobiales)在BW-60时富集,相对丰度13.37%,但是在RW-60和PW-60中并未富集;821 bp (根瘤菌目,Rhizobiales)在RW和PW中被富集。对照C/N改变前后反应器的氨氮脱除效率,推测744、67、874、463 bp是在高C/N条件下具有异养硝化功能的细菌。
2.5 C/N对生物膜中微生物群落结构的影响反应器中的填料为微生物的定殖、成膜提供载体,生物膜形成过程受营养、温度、pH等因素影响。当环境条件改变时,不适应环境变化的微生物会从生物膜中分散脱离[19]。因此,实验将空载填料和固定有脱氮功能的P. denitrificans PD1222的填料浸没入反应器,研究其成膜过程及不同营养条件下生物膜中微生物群落变化规律。
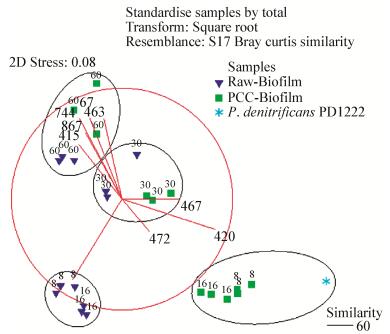
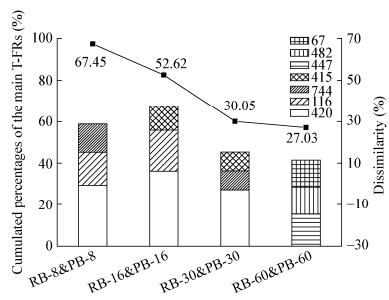
不同反应器生物膜中细菌群落NMDS表明,运行初期RB与PB中的微生物簇差异显著(图 5)。此时RB中的优势片段116 bp和472 bp等同时也是RW中的优势微生物(图 3),表明RB上的微生物主要来自RW。PB上的优势微生物主要为所附的P. denitrificans PD1222。第8天时RB-8和PB-8之间的差异度高达67.45% (图 6),其中420、116、744 bp对差异度的累计贡献为58.81%;第16天时RB-16和PB-16之间的差异度为52.62% (图 6),其中420、116、415 bp对差异度的累计贡献值为67.42%。在第25天将C/N由1:1改为2:1后RB与PB中细菌群落结构变化明显(图 5和图 7),相比于寡营养条件,样品组RB-30和PB-30及RB-60和PB-60的差异度显著下降,分别为30.05%和27.03%。高C/N下67、744、463、821、867、415 bp等T-RFs开始逐渐富集,第60天时PB中420 bp的相对丰度仅为2.46%,表明PB中P. denitrificans PD1222的优势地位被取代。同时生物膜成熟后PCC与Raw中生物膜上的微生物群落相似性较高(72.97%)。与Blank相比,富营养条件下67、744、463、821 bp在水体和生物膜中均得到了富集,而867 bp只在生物膜中得到了富集。
|
| 图 5 不同反应器生物膜中细菌群落NMDS分析 Figure 5 The NMDS plot of bacterial communities for biofilm samples in different reactors |
|
|
|
| 图 6 RB与PB的差异度趋势曲线及主要T-RFs的累计贡献 Figure 6 The dissimilarity of RB and PB and the cumulated contribution of the main T-RFs |
|
|
|
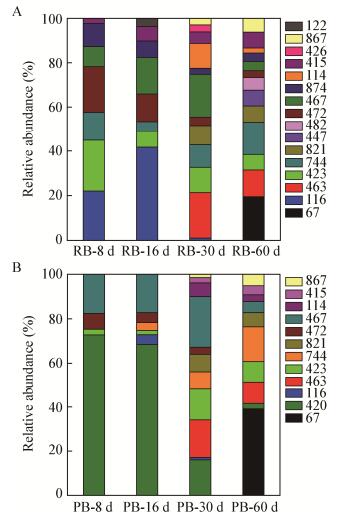
| 图 7 不同反应器生物膜中T-RFs相对丰度 Figure 7 Relative abundance of T-RFs in the biofilm niche of different reactors 注:A:自然成膜反应器(Raw)运行过程中生物膜中T-RFs的相对丰度;B:预附脱氮菌强化挂膜反应器(PCC)运行过程中生物膜中T-RFs的相对丰度. Note: A: The relative abundance of T-RFs in the biofilm niche during the operation of Raw; B: The relative abundance of T-RFs in the biofilm niche during the operation of PCC. |
|
|
为进一步研究不同营养条件下细菌群落的动态变化过程,对RB与PB中T-RFs的相对丰度进行了比较分析(图 7)。C/N=1:1条件下RB上所富集的优势片段116、423、472、874 bp等,在C/N=2:1条件下被67、744、463、821、447 (芽孢杆菌目,Bacillales)、867 bp (着色菌目,Chromatiales)等T-RFs所代表的菌群所演替;改变碳氮比后423 bp在RB中的优势地位被取代,其相对丰度由22.87% (RB-8) 变为7.03% (RB-60);447、482 bp (黄色单胞菌目,Xanthomonadales)为RB-60时富集的菌,而在PB中未富集。C/N=1:1条件下PB上的微生物结构较为单一,优势菌群主要为420、467、472 bp;在C/N=2:1条件下微生物多样性显著提高,67、744、463、821 bp等T-RFs大量富集成为优势菌(图 7B)。
3 讨论 3.1 C/N与氨氧化微生物群落结构之间的关系C/N、温度、pH、DO、污染物类型等因素是影响微生物群落结构的主要因子[20-22],实验中温度、pH、DO及污染物类型无显著差异,因此,C/N是影响微生物群落结构改变的一个重要因素。本实验中,不同生态位细菌群落具有相似的演替规律。C/N=1:1时优势菌主要为α-变形菌纲(423、116 bp)和β-变形菌纲(467、472 bp),C/N=2:1时优势菌主要为γ-变形菌纲(744、874、867 bp)、放线菌纲(67 bp)和硝化螺旋菌纲(463 bp)。
氨氧化是指在微生物作用下将氨氮氧化为亚硝态氮的过程,是硝化作用(NH3→NO2-→NO3-)的第一步,同时也是其限速步骤,典型的氨氧化过程被认为主要是由变形菌纲中β-变形菌纲(β-proteobacteria)等细菌类群进行的专性好氧化能自养过程,该类微生物统称为氨氧化细菌(AOB)[23-25]。本实验中高COD下3个反应器NH4+-N去除率均显著提高,但相应的功能微生物β-变形菌纲的丰度下降,原因可能是由于在反应器系统中硝化作用是由自养细菌和异养细菌协同完成的,充足的有机碳为异养硝化细菌的生长和作用提供了基质,促使假单胞菌目(744,874 bp)[26]、放线菌纲(67 bp)[27]、根瘤菌目(821 bp)[28]等潜在的异养硝化微生物快速富集,上述细菌在生物膜反应器脱氨氮过程中可能起主要作用。假单胞菌(Pseudomonas sp.)具有较强的环境适应能力,在地表水体、土壤等自然环境中具有普遍存在性[29]。施氏假单胞菌(Pseudomonas stutzeri)被认为是具有异养硝化-好氧反硝化特性的模式菌株,在有氧条件下能够同时去除氨氮及硝态氮[2, 30];Ji等[31]研究发现在高溶氧条件下Pseudomonas stutzeri对NO3--N的去除速率可达到5.7 mg/(L·h),且只有少量的NO2--N累积,并且对该菌株异养硝化-好氧反硝化过程中关键酶的编码基因napA、narG、nirS、norB、nosZ进行了验证;研究表明好氧反硝化菌株的终产物大多为温室气体N2O,Miyahara等[32]发现在反硝化过程中Pseudomonas stutzeri的N2O排放量较低,可以将N2O还原为N2。此外,门多萨假单胞菌(Pseudomonas mendocina)[33]、恶臭假单胞菌(Pseudomonas putida)[34]均表现出了一定的好氧反硝化能力。放线菌中的红球菌属(Rhodococcus sp.)等同样具有异养硝化-好氧反硝化潜能,Chen等[27]报道了一株从养殖废水中筛选的同时具有异养硝化和好氧反硝化能力的红球菌Rhodococcus sp. CPZ24,其反硝化产物中N2和N2O分别占了约99%和1%的量。根瘤菌目是一类重要的亚硝酸盐氧化菌(NOB),Okada等[35]从土壤中分离出了一株好氧反硝化菌Mesorhizobium sp. strain NH-14,在有氧或缺氧条件下均能将NO2--N还原为N2O。因此推测本实验室生物膜反应器的硝化作用可能主要由异养硝化菌贡献,高氨氮去除率、低AOB丰度的实验结果与之前的报道一致[23, 36-37]。此外,大部分具有异养硝化特性的微生物同时能进行好氧反硝化[38-40],本实验中随着NH4+-N的消减,NO3--N有一定程度的积累,因为不同异养硝化-好氧反硝化微生物进行反硝化作用对应的氧阈值(The oxygen threshold)不同[41-42],因此推测DO可能是影响好氧反硝化程度的关键因子。硝化螺旋菌为典型的亚硝酸盐氧化细菌(NOB),463 bp的富集可能解释了本实验中几乎无NO2-累积的原因。关于异养硝化在反应器脱氮过程中的贡献、作用条件及其作用机制仍需深入研究。
3.2 生物膜反应器硝化过程中的关键微生物本研究中假单胞菌目(Pseudomonadales)、根瘤菌目(Rhizobiales)、放线菌纲(Actinobacteria)在生物膜中具有较高的丰度。已有研究发现假单胞菌目(Pseudomonadales)、根瘤菌目(Rhizobiales)是微生物电池阴极生物膜及饮用水排水系统中的主要群落[43];同时,γ-变形菌纲被证实是形成生物膜的主要微生物[44-46]。以上研究与本实验结果一致,说明这些微生物具有较强的成膜能力。伯克氏菌目(Burkholderiales)被证明是生物膜中的主要菌群[47-48],然而本研究中伯克氏菌目(472 bp)随着反应器的运行其绝对优势地位被取代,该差异可能与实验运行条件有关。
研究表明,第30天时生物膜中的114 (浮霉菌目,Planctomycetales)、423、463、467 bp等T-RFs快速富集,相对丰度显著提高,但是,在反应器运行稳定后取样分析发现以上菌群的相对丰度明显下降,发生了演替。反应器生物膜形成过程是一个有序的过程,在成膜过程中,某些微生物首先构建生物膜的初期结构,另一些微生物受益于该结构而定殖繁殖,导致后期生物膜的形成。
运行初期相比于Blank和Raw,PCC脱除氨氮能力较好,PCC填料上所附的脱氮副球菌PD1222起主要作用,但是在反应器运行过程中该菌逐渐被演替,分析原因如下:(1) Paracoccus denitrificans PD1222为外源功能菌,可能与生态系统中的土著微生物存在竞争关系而不能形成优势菌,从而难以发挥其作用[49-51];(2) Paracoccus denitrificans PD1222分泌的胞外多聚物可能会促进PCC生物膜的形成,缩短成膜周期,但由于其自身成膜能力较差导致其优势地位被取代。
4 结论对实验室规模的不同成膜方式的生物膜反应器的运行效果及在氨氮消减过程中微生物群落结构多样性特征进行了分析。研究表明:(1) 可控条件下生物膜反应器Raw与PCC对氨氮(5 mg/L)的去除率均可达到95%以上,可达到《地表水环境质量标准(GB 3838-2002)》Ⅱ类水水质(0.5 mg/L)标准。(2) C/N是影响氨氮去除效果的关键因素之一,且不同营养环境下微生物群落结构差异较大,C/N=2:1条件下反应器运行稳定后优势菌群主要为γ-变形菌纲(744、874、867 bp)、放线菌纲(67 bp)和硝化螺旋菌纲(463 bp)。异养硝化微生物在氨氮转化过程中可能起主导作用。(3) PCC在运行初期相比于Blank和Raw氨氮转化效率较突出,反应器运行稳定后,Raw与PCC脱氮效率及微生物群落结构差异并不显著,说明PCC预附的Paracoccus denitrificans PD1222已被演替,土著微生物的污染物降解能力被充分放大。
总体而言,生物膜反应器本质是一种生物强化修复技术,结合生物膜技术抗逆性强、净化能力高、微生物群落结构稳定性好等优势,该技术可广泛应用于污水处理厂尾水深度处理,以及河流、湖泊、景观水体等地表水体的原位修复。基于受污环境水体的差异性和复杂性,筛选高效、易成膜的土著功能微生物,优化培养与生物膜联合应用技术,探究环境因素对原位生物膜修复的影响,建立并完善生物膜修复工程体系是该技术未来发展的侧重点。本实验采用的生物膜技术为污染水体原位微生物强化净化提供了一种思路。
| [1] |
Qin BQ, Xu PZ, Wu QL, et al. Environmental issues of lake Taihu, China[J]. Hydrobiologia, 2007, 581(1): 3-14. DOI:10.1007/s10750-006-0521-5 |
| [2] |
Guo LY, Chen QK, Fang F, et al. Application potential of a newly isolated indigenous aerobic denitrifier for nitrate and ammonium removal of eutrophic lake water[J]. Bioresource Technology, 2013, 142: 45-51. DOI:10.1016/j.biortech.2013.05.021 |
| [3] |
Tian WJ, Hao FH, Zhai JB. Elasticity plastic filller for purification of polluted streams in situ entering lake[J]. Environmental Science, 2008, 29(5): 1308-1312. (in Chinese) 田伟君, 郝芳华, 翟金波. 弹性填料净化受污染入湖河流的现场试验研究[J]. 环境科学, 2008, 29(5): 1308-1312. |
| [4] |
Wang RC, Wen XH, Jing YQ, et al. Remediation of polluted river water by using a suspended carrier biofilm reactor[J]. Environmental Science, 2004, 25(S1): 67-69. (in Chinese) 王荣昌, 文湘华, 景永强, 等. 悬浮载体生物膜反应器修复受污染河水试验研究[J]. 环境科学, 2004, 25(S1): 67-69. |
| [5] |
Song YW, Nie ZD, Nian YG, et al. Research of aeration with bio-film tecthnology to treat urban landscape water[J]. Environmental Science, 2008, 29(1): 58-62. (in Chinese) 宋英伟, 聂志丹, 年跃刚, 等. 城市景观水体曝气与生物膜联合净化技术研究[J]. 环境科学, 2008, 29(1): 58-62. |
| [6] |
Ma Q, Gao MY, Tan W, et al. Role of new type floating island in improving effluent quality and the characterization of microbe on biofilm carrier[J]. Environmental Science, 2011, 32(6): 1596-1601. (in Chinese) 马强, 高明瑜, 谭伟, 等. 新型生态浮岛在改善水质中的作用及生物膜载体微生物特征研究[J]. 环境科学, 2011, 32(6): 1596-1601. |
| [7] |
Marsh TL, Saxman P, Cole J, et al. Terminal restriction fragment length polymorphism analysis program, a web-based research tool for microbial community analysis[J]. Applied and Environmental Microbiology, 2000, 66(8): 3616-3620. DOI:10.1128/AEM.66.8.3616-3620.2000 |
| [8] |
Jia JT, Song LS, Li Y. T-RFLP technique and its application in research on microbial community structure[J]. Marine Sciences, 2004, 28(3): 64-68. (in Chinese) 贾俊涛, 宋林生, 李筠. T-RFLP技术及其在微生物群落结构研究中的应用[J]. 海洋科学, 2004, 28(3): 64-68. |
| [9] |
Paithankar K, Prasad KSN. Precipitation of DNA by polyethylene glycol and ethanol[J]. Nucleic Acids Research, 1991, 19(6): 1346. DOI:10.1093/nar/19.6.1346 |
| [10] |
Griffiths RI, Whiteley AS, O'Donnell AG, et al. Rapid method for coextraction of DNA and RNA from natural environments for analysis of ribosomal DNA-and rRNA-based microbial community composition[J]. Applied and Environmental Microbiology, 2000, 66(12): 5488-5491. DOI:10.1128/AEM.66.12.5488-5491.2000 |
| [11] |
Moeseneder MM, Arrieta JM, Muyzer G, et al. Optimization of terminal-restriction fragment length polymorphism analysis for complex marine bacterioplankton communities and comparison with denaturing gradient gel electrophoresis[J]. Applied and Environmental Microbiology, 1999, 65(8): 3518-3525. |
| [12] |
Wang XH, Wen XH, Criddle C, et al. Community analysis of ammonia-oxidizing bacteria in activated sludge of eight wastewater treatment systems[J]. Journal of Environmental Sciences, 2010, 22(4): 627-634. DOI:10.1016/S1001-0742(09)60155-8 |
| [13] |
Shyu C, Soule T, Bent SJ, et al. MiCA: a web-based tool for the analysis of microbial communities based on terminal-restriction fragment length polymorphisms of 16S and 18S rRNA genes[J]. Microbial Ecology, 2007, 53(4): 562-570. DOI:10.1007/s00248-006-9106-0 |
| [14] |
Hill TC, Walsh KA, Harris JA, et al. Using ecological diversity measures with bacterial communities[J]. FEMS Microbiology Ecology, 2003, 43(1): 1-11. DOI:10.1111/fem.2003.43.issue-1 |
| [15] |
Shannon CE. A mathematical theory of communication[J]. ACM SIGMOBILE Mobile Computing and Communications Review, 2001, 5(1): 3-55. DOI:10.1145/584091 |
| [16] |
Heip C. A new index measuring evenness[J]. Journal of the Marine Biological Association of the United Kingdom, 1974, 54(3): 555-557. DOI:10.1017/S0025315400022736 |
| [17] |
Clarke KR. Non-parametric multivariate analyses of changes in community structure[J]. Austral Ecology, 1993, 18(1): 117-143. DOI:10.1111/aec.1993.18.issue-1 |
| [18] |
Tyagi M, da Fonseca MMR, de Carvalho CCR. Bioaugmentation and biostimulation strategies to improve the effectiveness of bioremediation processes[J]. Biodegradation, 2011, 22(2): 231-241. DOI:10.1007/s10532-010-9394-4 |
| [19] |
Cogan N, Keener JP. The role of the biofilm matrix in structural development[J]. Mathematical Medicine and Biology, 2004, 21(2): 147-166. DOI:10.1093/imammb/21.2.147 |
| [20] |
Valentín-Vargas A, Toro-Labrador G, Massol-Deyá AA. Bacterial community dynamics in full-scale activated sludge bioreactors: operational and ecological factors driving community assembly and performance[J]. PLoS One, 2012, 7(8): e42524. DOI:10.1371/journal.pone.0042524 |
| [21] |
Wang XH, Hu M, Xia Y, et al. Pyrosequencing analysis of bacterial diversity in 14 wastewater treatment systems in China[J]. Applied and Environmental Microbiology, 2012, 78(19): 7042-7047. DOI:10.1128/AEM.01617-12 |
| [22] |
Shen CC, Xiong JB, Zhang HY, et al. Soil pH drives the spatial distribution of bacterial communities along elevation on Changbai Mountain[J]. Soil Biology and Biochemistry, 2013, 57: 204-211. DOI:10.1016/j.soilbio.2012.07.013 |
| [23] |
Ma Q, Qu YY, Shen WL, et al. Bacterial community compositions of coking wastewater treatment plants in steel industry revealed by Illumina high-throughput sequencing[J]. Bioresource Technology, 2015, 179: 436-443. DOI:10.1016/j.biortech.2014.12.041 |
| [24] |
Purkhold U, Pommerening-Röser A, Juretschko S, et al. Phylogeny of all recognized species of ammonia oxidizers based on comparative 16S rRNA and amoA sequence analysis: implications for molecular diversity surveys[J]. Applied and Environmental Microbiology, 2000, 66(12): 5368-5382. DOI:10.1128/AEM.66.12.5368-5382.2000 |
| [25] |
Li M, Cao HL, Hong YG, et al. Spatial distribution and abundances of ammonia-oxidizing archaea (AOA) and ammonia-oxidizing bacteria (AOB) in mangrove sediments[J]. Applied Microbiology and Biotechnology, 2011, 89(4): 1243-1254. DOI:10.1007/s00253-010-2929-0 |
| [26] |
Zhang JB, Wu PX, Hao B, et al. Heterotrophic nitrification and aerobic denitrification by the bacterium Pseudomonas stutzeri YZN-001[J]. Bioresource Technology, 2011, 102(21): 9866-9869. DOI:10.1016/j.biortech.2011.07.118 |
| [27] |
Chen PZ, Li J, Li QX, et al. Simultaneous heterotrophic nitrification and aerobic denitrification by bacterium Rhodococcus sp. CPZ24[J]. Bioresource Technology, 2012, 116: 266-270. DOI:10.1016/j.biortech.2012.02.050 |
| [28] |
Pai SL, Chong NM, Chen CH. Potential applications of aerobic denitrifying bacteria as bioagents in wastewater treatment[J]. Bioresource Technology, 1999, 68(2): 179-185. DOI:10.1016/S0960-8524(98)00140-0 |
| [29] |
Ueda A, Saneoka H. Characterization of the ability to form biofilms by plant-associated Pseudomonas species[J]. Current Microbiology, 2015, 70(4): 506-513. DOI:10.1007/s00284-014-0749-7 |
| [30] |
Huang TL, Guo L, Zhang HH, et al. Nitrogen-removal efficiency of a novel aerobic denitrifying bacterium, Pseudomonas stutzeri strain ZF31, isolated from a drinking-water reservoir[J]. Bioresource Technology, 2015, 196: 209-216. DOI:10.1016/j.biortech.2015.07.059 |
| [31] |
Ji B, Wang HY, Yang K. Tolerance of an aerobic denitrifier (Pseudomonas stutzeri) to high O2 concentrations[J]. Biotechnology Letters, 2014, 36(4): 719-722. DOI:10.1007/s10529-013-1417-x |
| [32] |
Miyahara M, Kim SW, Fushinobu S, et al. Potential of aerobic denitrification by Pseudomonas stutzeri TR2 to reduce nitrous oxide emissions from wastewater treatment plants[J]. Applied and Environmental Microbiology, 2010, 76(14): 4619-4625. DOI:10.1128/AEM.01983-09 |
| [33] |
Zhu L, Ding W, Feng LJ, et al. Characteristics of an aerobic denitrifier that utilizes ammonium and nitrate simultaneously under the oligotrophic niche[J]. Environmental Science and Pollution Research, 2012, 19(8): 3185-3191. DOI:10.1007/s11356-012-0822-3 |
| [34] |
Kim M, Jeong SY, Yoon SJ, et al. Aerobic denitrification of Pseudomonas putida AD-21 at different C/N ratios[J]. Journal of Bioscience and Bioengineering, 2008, 106(5): 498-502. DOI:10.1263/jbb.106.498 |
| [35] |
Okada N, Nomura N, Nakajima-Kambe T, et al. Characterization of the aerobic denitrification in Mesorhizobium sp. strain NH-14 in comparison with that in related Rhizobia[J]. Microbes and Environments, 2005, 20(4): 208-215. DOI:10.1264/jsme2.20.208 |
| [36] |
Ye L, Shao MF, Zhang T, et al. Analysis of the bacterial community in a laboratory-scale nitrification reactor and a wastewater treatment plant by 454-pyrosequencing[J]. Water Research, 2011, 45(15): 4390-4398. DOI:10.1016/j.watres.2011.05.028 |
| [37] |
Zhao YG, Huang J, Zhao H, et al. Microbial community and N removal of aerobic granular sludge at high COD and N loading rates[J]. Bioresource Technology, 2013, 143: 439-446. DOI:10.1016/j.biortech.2013.06.020 |
| [38] |
Padhi SK, Tripathy S, Sen R, et al. Characterisation of heterotrophic nitrifying and aerobic denitrifying Klebsiella pneumoniae CF-S9 strain for bioremediation of wastewater[J]. International Biodeterioration & Biodegradation, 2013, 78: 67-73. |
| [39] |
Joo HS, Hirai M, Shoda M. Characteristics of ammonium removal by heterotrophic nitrification-aerobic denitrification by Alcaligenes faecalis No.4[J]. Journal of Bioscience and Bioengineering, 2005, 100(2): 184-191. DOI:10.1263/jbb.100.184 |
| [40] |
Zhang Y, Shi Z, Chen MX, et al. Evaluation of simultaneous nitrification and denitrification under controlled conditions by an aerobic denitrifier culture[J]. Bioresource Technology, 2015, 175: 602-605. DOI:10.1016/j.biortech.2014.10.016 |
| [41] |
Ji B, Yang K, Zhu L, et al. Aerobic denitrification: a review of important advances of the last 30 years[J]. Biotechnology and Bioprocess Engineering, 2015, 20(4): 643-651. DOI:10.1007/s12257-015-0009-0 |
| [42] |
Su JJ, Liu BY, Liu CY. Comparison of aerobic denitrification under high oxygen atmosphere by Thiosphaera pantotropha ATCC 35512 and Pseudomonas stutzeri SU2 newly isolated from the activated sludge of a piggery wastewater treatment system[J]. Journal of Applied Microbiology, 2001, 90(3): 457-462. DOI:10.1046/j.1365-2672.2001.01265.x |
| [43] |
Wang ZJ, Zheng Y, Xiao Y, et al. Analysis of oxygen reduction and microbial community of air-diffusion biocathode in microbial fuel cells[J]. Bioresource Technology, 2013, 144: 74-79. DOI:10.1016/j.biortech.2013.06.093 |
| [44] |
Zhang K, Choi H, Dionysiou DD, et al. Application of membrane bioreactors in the preliminary treatment of early planetary base wastewater for long-duration space missions[J]. Water Environment Research, 2008, 80(12): 2209-2218. DOI:10.2175/106143008X325809 |
| [45] |
Lim SY, Kim S, Yeon KM, et al. Correlation between microbial community structure and biofouling in a laboratory scale membrane bioreactor with synthetic wastewater[J]. Desalination, 2012, 287: 209-215. DOI:10.1016/j.desal.2011.09.030 |
| [46] |
Miura Y, Watanabe Y, Okabe S. Membrane biofouling in pilot-scale membrane bioreactors (MBRs) treating municipal wastewater: impact of biofilm formation[J]. Environmental Science & Technology, 2007, 41(2): 632-638. |
| [47] |
Ivnitsky H, Katz I, Minz D, et al. Bacterial community composition and structure of biofilms developing on nanofiltration membranes applied to wastewater treatment[J]. Water Research, 2007, 41(17): 3924-3935. DOI:10.1016/j.watres.2007.05.021 |
| [48] |
Liu RY, Yu ZS, Guo HG, et al. Pyrosequencing analysis of eukaryotic and bacterial communities in faucet biofilms[J]. Science of the Total Environment, 2012, 435-436: 124-131. DOI:10.1016/j.scitotenv.2012.07.022 |
| [49] |
Boon N, Top EM, Verstraete W, et al. Bioaugmentation as a tool to protect the structure and function of an activated-sludge microbial community against a 3-chloroaniline shock load[J]. Applied and Environmental Microbiology, 2003, 69(3): 1511-1520. DOI:10.1128/AEM.69.3.1511-1520.2003 |
| [50] |
Boon N, Goris J, de Vos P, et al. Bioaugmentation of activated sludge by an indigenous 3-chloroaniline-degrading Comamonas testosteroni strain, I2gfp[J]. Applied and Environmental Microbiology, 2000, 66(7): 2906-2913. DOI:10.1128/AEM.66.7.2906-2913.2000 |
| [51] |
Quan XC, Shi HC, Liu H, et al. Removal of 2, 4-dichlorophenol in a conventional activated sludge system through bioaugmentation[J]. Process Biochemistry, 2004, 39(11): 1701-1707. DOI:10.1016/S0032-9592(03)00307-8 |
 2017, Vol. 44
2017, Vol. 44




