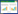扩展功能
文章信息
- 段小果, 李博, 贺银凤
- DUAN Xiao-Guo, LI Bo, HE Yin-Feng
- 乳酸菌与酵母菌共生机理的研究进展
- Progress in symbiotic mechanism between lactic acid bacteria and yeast
- 微生物学通报, 2017, 44(8): 1988-1995
- Microbiology China, 2017, 44(8): 1988-1995
- DOI: 10.13344/j.microbiol.china.160784
-
文章历史
- 收稿日期: 2016-10-28
- 接受日期: 2017-04-07
- 优先数字出版日期(www.cnki.net): 2017-04-26
基于乳酸菌自身是营养缺陷型菌株,早期研究乳酸菌与酵母菌之间存在的共生机理时,有研究者认为是乳酸菌与酵母菌的代谢产物之间存在互补机制,如酵母菌为乳酸菌提供营养因子,乳酸菌为酵母菌提供能量来源等。进一步的研究发现乳酸菌与酵母菌之间存在互作关系,如乳酸菌或酵母菌上清液能够减缓酸抑制作用促进目标活菌数的增加,且促进作用与上清液添加量有关;混合培养能够促进有机酸、游离氨基氮等含量的提高,更有利于风味代谢产物的产生[1-4]。经研究证实引起乳酸菌与酵母菌产生共生作用的关键物质是代谢产物中的某些蛋白类或大分子肽类物质[5]。这些能够在菌种间引起共生作用的物质在研究中还没有明确的信息,因此研究者开始从LuxS/AI-2介导的群体感应系统相关联的信号分子AI-2角度解释乳酸菌与酵母菌的共生机理。群体感应(Quorum sensing,QS)即细菌随着菌体密度的增大和生长周期的变化分泌出一种或几种化学信号分子,通过这些信号分子进行种内或种间交流,协调群体行为[6]。根据细菌合成感应机制和信号分子种类的不同,QS系统主要分为三类:革兰氏阴性菌(G-)的AHL (酰基高丝氨酸内酯)-LuxI/LuxR系统和扩散信号因子(DSF)参与的群体感应,由寡肽介导的革兰氏阳性菌(G+)双组分感应系统,不同种间细菌型LuxS/AI-2介导的QS系统[7]。研究表明LuxS/AI-2介导的QS系统对乳酸菌的耐酸特性、抑制病原微生物、降低肠毒力、对肠表皮细胞的黏附性、生物膜的形成以及在动物消化道中的存活性等具有介导作用[8-12]。在医学等领域应用LuxS/AI-2介导的群体感应系统也得到了很好的研究成果,如李洪东等[13]通过调节外源信号分子AI-2的剂量有效地治疗了由铜绿假单胞菌引起的生物膜感染;林琳等[14]发现信号分子AI-2参与调节耐辐射球菌与压力有关的基因,有助于耐辐射球菌的极强抗辐射和氧化胁迫特性。从LuxS/AI-2介导的群体感应角度出发研究酸马奶酒中乳酸菌与酵母菌的共生机理,不仅能够更加清楚地了解种间信息交流模式以及共生机理,更能够为其在食品工业、医学等领域的应用奠定基础。
1 从营养角度分析乳酸菌与酵母菌的共生机制 1.1 乳酸菌与酵母菌之间的互补机制乳酸菌与酵母菌共同发酵多发生于乳制品和含乳饮料中,二者的互补机制研究是从营养学的角度分析其代谢产物产生及消耗的情况,从而阐明了酵母菌为乳酸菌提供营养因子如丙酮酸盐、维生素及氨基酸等[15],乳酸菌为酵母菌提供能量来源[16]的机理。如在乳酪的生产过程中,酵母菌分泌的脂肪酶和蛋白酶分解基质物质为乳酸菌的生长提供了营养物质,同时代谢掉乳酸菌生长过程中产生的乳酸盐,生成芳香物质,促进乳酪的后熟[17]。Cheirsilp等[18]在牛乳中共同培养乳酸菌与酵母菌发现,酵母菌利用乳酸菌把乳糖降解成的半乳糖作为碳源,而酵母菌利用乳酸盐升高牛乳的pH值从而促进乳酸菌的生长。Mendes等[19]研究开费尔共生发酵的酿酒酵母(Saccharomyces cerevisiae)和保加利亚乳杆菌,不仅鉴定出保加利亚乳杆菌水解乳糖生成的半乳糖为酵母提供碳源的作用机制,还鉴定出在厌氧共培养中,酿酒酵母通过酒精发酵增加CO2的浓度并提供丙氨酸从而促进保加利亚乳杆菌的生长。
乙醛是由乳酸菌利用苏氨酸转化而来,是能够使酸奶具有特殊风味的主要物质,因乳酸菌水解蛋白和脂肪酸能力有限,大部分必需氨基酸则是从牛乳中获取[20-21],而酵母菌能够分泌苏氨酸,因此Rysstad等[22]提出假设,乳酸菌与酵母菌共培养时能够提高乙醛的产生量。Gadaga等[23]研究了自然发酵乳中的4株乳酸菌和9株酵母菌,结果表明乳酸菌与酵母菌混合培养能够产生更多的乙醛等物质,确证了两种菌株之间存在互补机制。
1.2 乳酸菌与酵母菌之间的互作关系研究发现乳酸菌与酵母菌之间相互添加发酵液能够促进目标菌株的生长及相关物质产量的增加。贺银凤等[1]相互添加酸马奶酒中的2株乳酸菌(坚强肠球菌W1和粪肠球菌W2)和2株酵母菌(厚壁酵母属J1和娄德酵母属J2)的发酵液发现,乳酸菌的发酵液经灭菌前和灭菌后对酵母菌菌落的生长均有促进作用;酵母菌发酵液只对粪肠球菌W2的生长及其产酸能力有促进作用,且随着发酵液添加量的增加,促进作用明显增加。这一试验结果证实了乳酸菌与酵母菌之间并非都能产生促进作用,有的相互之间并无影响;产生促进作用的物质耐高温,且促进作用在一定范围内会随着发酵液的添加量而增强。闫彬等[2]测定明串珠菌1-3-2和克勒克酵母6-4混合培养体系中的风味代谢产物,结果表明克勒克酵母6-4水解蛋白的能力更强,混合培养有利于更多风味物质的产生,且在任何时期混合培养基中甲酸、乙酸、丙酸的产量都高于单菌落培养过程中的生成量。确证了在风味代谢产物方面乳酸菌与酵母菌之间存在着互相促进作用的关系,这一结论与Álvarez-Martín等[3]试验结果相一致。
Álvarez-Martín等[3]通过混合培养研究了12株酵母菌和4株乳酸菌的生长活动及其相互作用,研究发现混合发酵不仅促进了活菌数的增加,且酵母菌的存在使得乳酸菌产生更多的乳酸,丙酸、乙酸、酪酸和丙酮酸的产生减少,这一现象从侧面体现了酸类产量和酵母菌及乳酸菌利用底物的程度;研究中还发现大多数乳酸球菌和酵母菌混合培养具有促进作用,而有些乳酸杆菌受到假丝酵母、汉逊德巴利酵母、乳酸克鲁维酵母、毕赤酵母4种酵母菌的抑制。但有研究者认为乳酸菌和酵母菌混合培养的抑制作用表现在乳酸菌产生的代谢产物如苯乳酸、4-羟基-苯乳酸、环肽类物质抑制了酵母菌的生长[24-25],也有研究者认为是酵母菌产生的脂肪酸有可能抑制了乳酸菌的生长[26-27]。但Mendes等[19]分析保加利亚乳杆菌的转录组显示,在混合培养过程中脂质代谢转录物过度表达,猜测有可能是两种微生物对脂肪酸的竞争或是对酿酒酵母产生乙醇的应激反应,乳酸菌本身水解脂肪酸的能力有限,因此在一定程度上酵母菌的存在有利于脂肪酸的水解。
Tiukova等[28]发现乳酸杆菌与酿酒酵母共培养过程中能够形成絮凝物,絮凝物的形成可以减少暴露于乙醇的表面,有利于乳酸杆菌在高乙醇含量条件下生存,从而具有高耐受乙醇的能力,且絮凝物结合细胞的大小和比例随着乙醇浓度的增加而增加,经观察发现絮凝物是由酵母细胞外层的细菌为核心组成的乳酸菌-酵母菌聚集体,进一步测试发现甘露糖参与了絮凝过程。相比单一培养,Sudun等[29]在研究马奶酒中乳酸菌和酵母菌的相互作用中也发现混合培养中都显示出高的活菌计数;同时也发现酿酒酵母4C仅在混合培养中通过利用乳酸菌产生的代谢产物从而产生大量的乙醇;而明串珠菌6B2081和瑞士乳杆菌130B4在与酵母菌混合培养过程中,产生的葡萄糖和半乳糖都被完全消耗,乳糖也显著降低,表明酵母菌能够利用乳酸菌产生的葡萄糖和半乳糖促进细胞生长。乳酸菌与酵母菌积极的相互作用为开发新的有益于酒精发酵的乳制品提供了理论依据。
刘敏敏[4]针对乳酸菌和酵母菌促进作用极为显著的屎肠球菌LC5和毕赤氏酵母YE4共生菌组,通过顺序培养的方式研究了代谢产物对另一菌株生长的影响,结果表明二者的代谢物能够促使另一菌株发酵液pH值降低速率减缓,减缓酸抑制作用,从而相对有利于甚至接近最适宜的生长条件,使得活菌数量增加;相对于粪肠球菌LC5单独发酵,毕赤氏酵母YE4代谢物一定程度上促进了粪肠球菌LC5产乳酸、乙醛等的量,苹果酸、酒石酸和柠檬酸36 h内增加速率减缓,但始终处于上升趋势;粪肠球菌LC5代谢物促进了毕赤氏酵母YE4的生长,使得毕赤氏酵母YE4发酵液中游离氨基氮含量较高。王美霞[5]在此基础上有了进一步的研究发现,对粪肠球菌LC5促进作用最好的毕赤氏酵母YE4代谢物添加量为40%,超滤发现促生物质分子量(M)在10 kD<M<30 kD范围,M>30 kD有一定的抑制作用;促生物质经过紫外扫描后出现两个最高峰值,将其认为是蛋白或肽类物质的混合物,该混合物经硫酸铵盐析得到的沉淀物对粪肠球菌LC5的促进作用效果最好,经层析、电泳得到一条位于鸡蛋清溶菌酶相对分子质量(Mr) 14.4 kD以下,5条分子量分别为15.2、17.6、19.1、21.3和23.8 kD的条带。此类物质可能是蛋白类、大分子肽类或者两者的混合物,具体组成成分及结构需要更进一步的研究。
Maligoy等[30]从基因分子角度研究二者混合培养时乳酸菌基因表达的变化,结果表明共培养时乳酸菌在mRNA水平上与多种新陈代谢有关的158个重要基因发生了较大程度地调整。Guerzoni等[31]通过改变培养环境如高氧、高酸、高渗透压等来研究乳酸菌与酵母菌之间的互作关系,比较乳酸菌与酵母菌单独培养和混合培养时产生乙醇及芳香物质的差异,结果发现酵母菌在高酸、高氧环境下会分泌出长链不饱和脂肪酸酯。这种现象有学者认为是酵母菌体的自我保护机制,而有的学者将酵母菌分泌的这种不饱和脂肪酸脂看作是其在特定环境下释放的信号分子[32]。因此将分泌物中的某种物质作为信号分子,并通过测量其代谢产物如乙醇等来观察二者之间的促生作用,从而研究乳酸菌与酵母菌之间的共生机制。
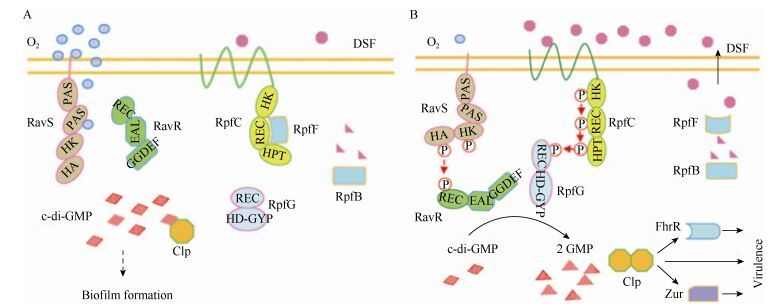
2 从信号分子AI-2角度分析乳酸菌与酵母菌的共生机理 2.1 信号分子简述信号分子可以分为4大类:寡肽(Oligopeptides)、酰基高丝氨酸内酯(Acyl-homoserine lactone,AHL)、自体诱导物2 (Autoinduction-2,AI-2) 和扩散信号因子(Diffusible signal factor,DSF)。哈维氏弧菌存在的信号分子分为自体诱导物1 (Autoinduction-1,AI-1) 和自体诱导物2 (Autoinduction-2,AI-2)。AI-1是酰基高丝氨酸内酯(AHL)类化合物,AI-2是LuxS/AI-2介导的QS系统中的AI-2,即种内和种间密度感应信号分子[33],在启动相关基因表达的前提下,细菌类型不同,需要的受体蛋白不同,4类信号分子对应的作用机制如图 1和图 2所示。
革兰氏阴性菌的AHL-LuxI/LuxR系统:LuxI自动诱导剂合酶产生AHL分子,其自由地扩散跨过细胞质膜,在临界浓度,AHL结合LuxR,产生活化反应调节器激活目标基因的转录(图 1a)。革兰氏阳性菌中以寡肽作为信号分子的双组分感应系统:ComC基因编码肽的前体物质,即通过专用ABC转运蛋白(ComAB)分泌的成熟信号肽(CSP),在临界浓度,CSP结合到细胞质膜上的同源组氨酸激酶(ComD)并诱导其自磷酸化(phosphorylates),该激酶随后磷酸化(P)并激活反应器(ComE) (图 1b)。哈维氏弧菌中的群体感应:两个群体感应系统经过相同的LuxU磷酸化反应,分别通过LuxN和LuxQ在细胞表面检测到AI-1和AI-2 (参与识别AI-2的周质LuxP蛋白为简单起见未示出),AI-1由LuxLM基因编码产物催化产生,相应的感应分子为LuxN,AI-2为一种新的信号分子,感应分子为LuxP和LuxQ,LuxN和LuxQ均通过LuxU来实现信号传递,LuxU是一种磷酸转移酶,活化后将信号传递至调节蛋白LuxO,并在另一蛋白LuxR的协助下,启动基因的表达(图 1c)[34]。
DSF参与的群体感应系统多存在于革兰氏阴性菌,在低细胞密度和在正常氧水平条件下,DSF传感器RpfC通过其受体结构域与DSF的合成酶RpfF形成复合物,限制基础水平的DSF生物合成,同样,RavS分子与氧分子的反应使RavS/RavR双组分系统保持在惰性状态,细胞内高含量的参与调节细菌中各种生物功能的信使c-di-GMP促进生物膜形成并通过形成蛋白质-配体复合物抑制Clp转录因子的活性(图 2A);在高细胞密度下,RpfC在感测累积的细胞外DSF信号时经历自磷酸化。通过保守的磷酸转移机制,RpfG被磷酸化,并且导致其c-di-GMP磷酸二酯酶活性的活化,c-diGMP的降解释放Clp,其直接或通过下游转录因子FhrR和Zur分别调节毒力基因的子集(图 2B);此外,传感器感知低氧压力促使RavS激活RavR的c-di-GMP磷酸二酯酶活性,促使c-di-GMP降解并通过Clp增强毒力基因表达[35]。
2.2 乳酸菌产信号分子AI-2的变化规律目前哈维氏弧菌(Vibrio harveyi,V. harveyi)的信号分子AI-2已被确定是一种呋喃硼酸二酯(Furanosyl borate diester)分子,研究乳酸菌与酵母菌种内种间的信息交流多采用V. harveyi BB170生物学方法检测。廉雪花[36]对分离自酸马奶酒中的11株乳酸菌产信号分子AI-2的活性进行了检测,通过V. harveyi BB170生物学方法检测结果显示粪肠球菌LC5、麦芽香乳杆菌2-1等9株乳酸菌均可产生信号分子AI-2,其中只有植物乳杆菌6-1-1在稳定期开始产生,其余8株都在对数期开始产生。产生AI-2信号分子的乳酸菌在4 h时产生的信号分子AI-2积累到了诱导发光的阈值,在10-19 h大部分乳酸菌上清液中的荧光强度逐渐达到峰值,只有少数是在19-25 h达到峰值,随后降低。
李博等[37]通过V. harveyi BB170生物学方法检测内蒙古锡盟地区的8株乳酸菌产生信号分子AI-2的活性,并对筛选出的高产信号分子AI-2的菌株2-1进行了分子生物学鉴定,结果表明酸马奶酒中部分乳酸菌产信号分子AI-2,其荧光强度随着菌体浓度的增加而逐渐增强;乳酸菌开始产生信号分子AI-2及达到峰值的时间有所不同,大多数产信号分子AI-2的乳酸菌在4 h开始产生,19 h达到峰值,之后随着时间的延长逐渐下降,这一结果验证了廉雪花[36]的结论;进一步筛选发现菌株1-3-2、2-1产信号分子AI-2的能力显著优于其他乳酸菌,其中菌株2-1最为显著,经鉴定为发酵乳杆菌。
Moslehi-Jenabian等[8]针对性地研究了4种乳酸杆菌Lactobacilli rhamnosus GG、Lactobacilli salivariusUCC118、Lactobacilli acidophilus NCYM及Lactobacilli johnsonii NCC533在不同酸性作用下LuxS/AI-2介导的群体感应现象,结果表明随着生长环境中pH强度的增加,4种乳酸杆菌在对数期或稳定期信号分子AI-2的活性增强,对应的LuxS基因转录随时间推移达到最高水平,随后下降;对数期初期4株益生乳酸杆菌均开始产生信号分子AI-2,对数生长中期随着生理环境中pH的降低,信号分子AI-2的量逐渐增加;Lactobacilli rhamnosus GG直到对数期末期信号分子AI-2的量达到最大值,而Lactobacilli salivarius UCC118、Lactobacilli acidophilus NCYM和Lactobacilli johnsonii NCC533到稳定期分别达到最大值。此结果表明益生乳酸杆菌LuxS基因在酸性应激反应中具有明确的作用,在一定程度上也说明具有耐酸特性的乳酸杆菌在环境胁迫下生长的优势,也为将菌体应用于胃肠道微生物等相关研究提供了选择依据。
多种乳酸菌信号分子AI-2活性的测定在一定程度上说明了信号分子AI-2产生时间、达到峰值以及降低的时间有所不同,且菌体生理环境的变化会影响信号分子AI-2产生以及到达活性峰值的时间。目前仅有文献报道了产信号分子AI-2的乳酸杆菌在不同pH生理环境下的研究,但在其他不同生理环境条件(如渗透压、温度等)下的影响效应还有待进一步的研究。
2.3 酵母菌对乳酸菌产信号分子AI-2的影响毕赤氏酵母YE4、克鲁克酵母J6、有孢圆酵母J11、厚壁孢酵母J23-1不能产生信号分子AI-2,但其代谢产物在一定时间内对粪肠球菌LC5信号分子AI-2的产生都有一定的促进作用,对麦芽香乳杆菌2-1,只有毕赤氏酵母YE4有促进作用,其余都表现出一定的抑制作用[36]。郭倩茹等[38]筛选出3组典型的乳酸菌与酵母菌共生组合:粪肠球菌LAB4与厚壁孢酵母YST2、植物乳杆菌LAB7与厚壁孢酵母YST2、干酪乳杆菌LAB9与厚壁孢酵母YST2,其中乳酸菌LAB4、LAB7、LAB9均能产生信号分子AI-2,厚壁孢酵母YST2代谢产物对粪肠球菌LAB4、干酪乳杆菌LAB9产生信号分子AI-2的促进作用极为显著,而整个共生过程中对植物乳杆菌LAB7产信号分子AI-2既有促进作用,也有抑制作用。产生促进作用可能是信号分子AI-2参与了调控共生环境中的相关物质,从而有利于乳酸菌的生长,而某些乳酸菌后期又表现出的抑制作用可能是酵母菌能够降解信号分子AI-2,也有可能是将其作为营养物质消化吸收,具体原因还需要进一步的研究。
酸马奶酒中共生菌组合的筛选证实了代谢产物中的某些物质能够促进或抑制目标菌株的生长,大量研究表明不是所有的乳酸菌和酵母菌都能够相互作用。部分酵母菌代谢产物对产信号分子AI-2的乳酸菌有一定的作用效果,产生促进或抑制作用与酵母菌代谢产物中的某些物质有关,也可能是不同酵母菌的代谢产物能为信号分子AI-2的产生创造有利或不利的环境。酵母菌促进或抑制乳酸菌产生信号分子AI-2的时间点以及产生量的不同,可能与乳酸菌感应生理环境变化并作出相应的自我调节应激反应速度有关,也可能与LuxS基因的表达量有关。
3 展望从营养角度研究酸马奶酒中乳酸菌与酵母菌的共生机理更倾向于分析共生体系中的互补机制或互作关系,其积极的相互作用有利于发展食品产业在实际应用中的多元化。然而从群体感应角度研究能够从分子水平或基因水平更深入地探讨种间信息交流模式。因此目前针对乳酸菌与酵母菌共生机理的研究已从早期的营养角度过度到了群体感应的角度。实时监测信号分子AI-2的变化规律,并深入探讨LuxS/AI-2介导的群体感应系统的影响力,对探究乳酸菌与酵母菌的共生机理有很大意义。现阶段对乳酸菌产信号分子AI-2的研究还处于菌体自分泌状态下的分析,存在干扰因素,而体外合成的纯度较高的信号分子AI-2对乳酸菌与酵母菌的共生系统影响还尚不清楚。因此体外合成信号分子AI-2结合生理环境条件的影响,是从群体感应角度研究乳酸菌与酵母菌共生机理的突破点。
| [1] |
He YF, Liu MM, Li YJ, et al. Interactions between lactic acid bacteria and yeasts from koumiss[J]. Food Science and Technology, 2011, 36(5): 32-36. (in Chinese) 贺银凤, 刘敏敏, 李燕军, 等. 酸马奶酒中乳酸菌和酵母菌互作关系的研究[J]. 食品科技, 2011, 36(5): 32-36. |
| [2] |
Yan B, He YF. Interactions between lactic acid bacteria and yeasts in kumiss: research on flavour metabolites[J]. China Dairy Industry, 2012, 40(11): 10-15. (in Chinese) 闫彬, 贺银凤. 酸马奶中一株乳酸菌与一株酵母菌共生关系和风味代谢产物的研究[J]. 中国乳品工业, 2012, 40(11): 10-15. DOI:10.3969/j.issn.1001-2230.2012.11.003 |
| [3] |
Álvarez-MartínP, FlórezA, Hernández-BarrancoA, 等. Interaction between dairy yeasts and lactic acid bacteria strains during milk fermentation[J]. Food Control, 2008, 19(1): 62-70. |
| [4] |
Liu MM. Study on potential symbiosis and fermentation characteristic between lactic acid bacteria and yeasts isolated from koumiss[D]. Hohhot: Master's Thesis of Inner Mongolia agricultural University, 2011(in Chinese) 刘敏敏. 酸马奶中乳酸菌和酵母菌潜在共生菌筛选及发酵特性研究[D]. 呼和浩特: 内蒙古农业大学硕士学位论文, 2011 http://cdmd.cnki.com.cn/Article/CDMD-10129-1011178400.htm |
| [5] |
Wang MX. Optimization medium of symbiotic lactic acid bacteria and yeasts and the separation of the promote biomass[D]. Hohhot: Master's Thesis of Inner Mongolia agricultural University, 2013(in Chinese) 王美霞. 具有共生作用乳酸菌与酵母菌培养基优化及促生物质的分离[D]. 呼和浩特: 内蒙古农业大学硕士学位论文, 2013 http://cdmd.cnki.com.cn/Article/CDMD-10129-1013245473.htm |
| [6] |
Melissa BM, Bassler BL. Quorum sensing in bacteria[J]. Annual Review of Microbiology, 2001, 5(1): 165-199. |
| [7] |
Shao CL, Sun ZK, Liao XR, et al. Research progress in quorum sensing and pathogenicity of pathogens[J]. Letters in Biotechnology, 2011, 22(3): 445-448. (in Chinese) 邵长林, 孙忠科, 廖祥儒, 等. 群体感应与病原菌致病性研究进展[J]. 生物技术通讯, 2011, 22(3): 445-448. |
| [8] |
Moslehi-Jenabian S, Gori K, Jespersen L. AI-2 signalling is induced by acidic shock in probiotic strains of Lactobacillus spp.[J]. International Journal of Food Microbiology, 2009, 135(3): 295-302. DOI:10.1016/j.ijfoodmicro.2009.08.011 |
| [9] |
Moslehi-Jenabian S, Vogensen FK, Jespersen L. The quorum sensing LuxS gene is induced in Lactobacillus acidophilus NCFM in response to Listeria monocytogenes[J]. International Journal of Food Microbiology, 2011, 149(3): 269-273. DOI:10.1016/j.ijfoodmicro.2011.06.011 |
| [10] |
Park H, Yeo S, Ji Y, et al. Autoinducer-2 associated inhibition by Lactobacillus sakei NR28 reduces virulence of enterohaemorrhagic Escherichia coli O157:H7[J]. Food Control, 2014, 45: 62-69. DOI:10.1016/j.foodcont.2014.04.024 |
| [11] |
Buck BL, Azcarate-Peril MA, Klaenhammer TR. Role of autoinducer-2 on the adhesion ability of Lactobacillus acidophilus[J]. Journal of Applied Microbiology, 2009, 107(1): 269-279. DOI:10.1111/jam.2009.107.issue-1 |
| [12] |
Sun ZK, He X, Brancaccio VF, et al. Bifidobacteria exhibit LuxS-dependent Autoinducer-2 activity and biofilm formation[J]. PLoS One, 2014, 9(2): e88260. DOI:10.1371/journal.pone.0088260 |
| [13] |
Li HD, Li XY, Yu JL, et al. Autoinducer-2 regulates Pseudomonas aeruginosa PAO1 biofilm formation and virulence production in a dose-dependent manner[J]. BMC Microbiology, 2015, 15: 192. DOI:10.1186/s12866-015-0529-y |
| [14] |
Lin L, Li T, Dai S, et al. Autoinducer-2 signaling is involved in regulation of stress‑related genes of Deinococcus radiodurans[J]. Archives of Microbiology, 2016, 198: 43-51. DOI:10.1007/s00203-015-1163-7 |
| [15] |
Roostita R, Fleet GH. The occurrence and growth of yeasts in Camembert and blue-veined cheeses[J]. International Journal of Food Microbiology, 1996, 28(3): 393-404. DOI:10.1016/0168-1605(95)00018-6 |
| [16] |
Loretan T. The diversity and technological properties of yeasts from indigenous traditional South African fermented milks[D]. University of the Orange Free State, 1999
|
| [17] |
Welthagen JJ, Viljoen BC. Yeast profile in Gouda cheese during processing and ripening[J]. International Journal of Food Microbiology, 1998, 41(3): 185-194. DOI:10.1016/S0168-1605(98)00042-7 |
| [18] |
Cheirsilp B, Shimizu H, Shioya S. Enhanced kefiran production by mixed culture of Lactobacillus kefiranofaciens and Saccharomyces cerevisiae[J]. Journal of Biotechnology, 2003, 100(1): 43-53. DOI:10.1016/S0168-1656(02)00228-6 |
| [19] |
Mendes F, Sieuwerts S, de Hulster E, et al. Transcriptome-based characterization of interactions between Saccharomyces cerevisiae and Lactobacillus delbrueckii subsp. bulgaricus in lactose-grown chemostat cocultures[J]. Applied and Environmental Microbiology, 2013, 79(19): 5949-5961. DOI:10.1128/AEM.01115-13 |
| [20] |
Narvhus JA, steraas K, Mutukumira T, et al. Production of fermented milk using a malty compound-producing strain of Lactococcus lactis subsp. lactis biovar. diacetylactis, isolated from Zimbabwean naturally fermented milk[J]. International Journal of Food Microbiology, 1998, 14(1): 73-80. |
| [21] |
Ayad EHE, Verheul A, de Jong C, et al. Flavour forming abilities and amino acid requirements of Lactococcus lactis strains isolated from artisanal and non-dairy origin[J]. International Dairy Journal, 1999, 9(10): 725-735. DOI:10.1016/S0958-6946(99)00140-5 |
| [22] |
Rysstad G, Knutsen WJ, Abrahamsen RK. Effect of threonine and glycine on acetaldehyde formation in goats milk yogurt[J]. Journal of Dairy Research, 1990, 57(3): 401-411. DOI:10.1017/S0022029900027059 |
| [23] |
Gadaga H, Mutukumira A, Narvhus JA. The growth and interaction of yeasts and lactic acid bacteria isolated from Zimbabwean naturally fermented milk in UHT milk[J]. International Journal of Food Microbiology, 2001, 68(1/2): 21-32. |
| [24] |
Broome MC, Thomas MP, Hi Her AJ, et al. The effect of linoleic acid on the growth and met a bo1ism of Streptococcus lactic[J]. Australian Journal of Dairy Technology, 1979, 34: 163-168. |
| [25] |
Nielsen MS, Frisvad JC, Nielsen PV. Protection by fungal starters against growth and secondary metabolite production of fungal spoilers of cheese[J]. International Journal of Food Microbiology, 1998, 42(1/2): 91-99. |
| [26] |
Bayrock DP, Ingledew WM. Inhibition of yeast by lactic acid bacteria in continuous culture: nutrient depletion and/or acid toxicity?[J]. Journal of Industrial Microbiology and Biotechnology, 2004, 31(8): 362-368. DOI:10.1007/s10295-004-0156-3 |
| [27] |
Ng WL, Bassler BL. Bacteria; quorum-sensing network architectures[J]. Annual Review of Genetics, 2009, 43(1): 197-222. DOI:10.1146/annurev-genet-102108-134304 |
| [28] |
Tiukova I, Eberhard T, Passoth V. Interaction of Lactobacillus vini with the ethanol-producing yeasts Dekkera bruxellensis and Saccharomyces cerevisiae[J]. Biotechnology and Applied Biochemistry, 2014, 61(1): 40-44. DOI:10.1002/bab.2014.61.issue-1 |
| [29] |
Sudun, Wulijideligen, Arakawa K, et al. Interaction between lactic acid bacteria and yeasts in airag, an alcoholic fermented milk[J]. Animal Science Journal, 2013, 84(1): 66-74. DOI:10.1111/asj.2013.84.issue-1 |
| [30] |
Maligoy M, Mercade M, Cocaign-Bousquet M, et al. Transcriptome analysis of Lactococcus lactis in coculture with Saccharomyces cerevisiae[J]. Applied and Environmental Microbiology, 2008, 74(2): 485-494. DOI:10.1128/AEM.01531-07 |
| [31] |
Guerzoni ME, Vernocchi P, Ndagijimana M, et al. Generation of aroma compounds in sourdough: effects of stress exposure and lactobacilli-yeasts interactions[J]. Food Microbiology, 2007, 24(2): 139-148. DOI:10.1016/j.fm.2006.07.007 |
| [32] |
Thoma I, Loeffler C, Sinha AK, et al. Cyclopentenone isoprostanes induced by reactive oxygen species trigger defense gene activation and phytoalexin accumulation in plants[J]. The Plant Journal, 2003, 34(3): 363-375. DOI:10.1046/j.1365-313X.2003.01730.x |
| [33] |
Jiang LM, Kang ZT, Liu CJ, et al. The research advances of AI-2/LuxS quorum sensing system mediating Lactobacillus probiotic properties[J]. Chinese Bulletin of Life Sciences, 2014, 26(4): 414-418. (in Chinese) 姜黎明, 康子腾, 柳陈坚, 等. AI-2/LuxS群体感应系统介导乳酸杆菌益生特性研究进展[J]. 生命科学, 2014, 26(4): 414-418. |
| [34] |
McNab R, Lamont RJ. Microbial dinner-party conversations: the role of LuxS in interspecies communication[J]. Journal of Medical Microbiology, 2003, 52(7): 541-545. DOI:10.1099/jmm.0.05128-0 |
| [35] |
Deng YY, Wu JE, Tao F, et al. Listening to a new language: DSF-based quorum sensing in gram-negative bacteria[J]. Chemical Reviews, 2011, 111(1): 160-173. DOI:10.1021/cr100354f |
| [36] |
Lian XH. Study of AI-2 signal molecule in lactic acid bacteria from koumiss[D]. Hohhot: Master's Thesis of Inner Mongolia agricultural University, 2014(in Chinese) 廉雪花. 酸马奶酒中乳酸菌产AI-2信号分子的研究[D]. 呼和浩特: 内蒙古农业大学硕士学位论文, 2014 http://cdmd.cnki.com.cn/Article/CDMD-10129-1014374056.htm |
| [37] |
Li B, Gu Y, Yan CL, et al. Screening and identification of AI-2 high-producing lactic acid bacteria[J]. Science and Technology of Food Industry, 2016, 37(2): 185-188. (in Chinese) 李博, 顾悦, 燕彩玲, 等. 高产信号分子AI-2乳酸菌的筛选及鉴定[J]. 食品工业科技, 2016, 37(2): 185-188. |
| [38] |
Guo QR, He YF. Screening on the symbiotic lactic acid bacteria and yeasts[J]. Science and Technology of Food Industry, 2015, 36(2): 203-206, 211. (in Chinese) 郭倩茹, 贺银凤. 具有共生关系的乳酸菌和酵母菌的筛选[J]. 食品工业科技, 2015, 36(2): 203-206, 211. |
 2017, Vol. 44
2017, Vol. 44