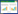扩展功能
文章信息
- 杨伟伟, 王智能, 郑露华, 丁冯玲, 梁小波
- YANG Wei-Wei, WANG Zhi-Neng, ZHENG Lu-Hua, DING Feng-Ling, LIANG Xiao-Bo
- Rib类似蛋白在三种乳酸杆菌中的分布
- Distribution of Rib like protein in three lactobacilli bacteria
- 微生物学通报, 2017, 44(8): 1866-1873
- Microbiology China, 2017, 44(8): 1866-1873
- DOI: 10.13344/j.microbiol.china.170232
-
文章历史
- 收稿日期: 2017-03-20
- 接受日期: 2017-04-19
- 优先数字出版日期(www.cnki.net): 2017-05-09
乳酸杆菌是乳酸菌中最大的一个属,与动物的关系最为紧密,广泛存在于动物和人类的消化道内,其中绝大多数被认为是安全的益生菌[1]。研究显示乳酸杆菌具有抵抗胃肠道感染、缓解腹泻、改善乳糖代谢、降低胆固醇、改善骨质疏松及免疫调节等作用[2-4],被广泛应用于食品发酵和生产中[5],具有广阔的工业和医学前景[6]。另外,还有研究发现乳酸杆菌能够长期定殖在肠道和泌尿生殖道内,调节机体菌群平衡,改善内源细菌特性,竞争性抑制病原菌黏附和繁殖[7-10]。罗伊氏乳杆菌作为重要的益生菌之一,广泛存在于哺乳动物及鸟类的肠道内[11]。近年来的研究表明罗伊氏乳杆菌具有改善人体机能、提高机体免疫力、促进人体健康的作用[12-14]。
益生菌在胃肠道中的黏附定殖对其发挥益生功效尤为重要,且黏附是定殖的先决条件,也是评价乳酸杆菌益生功能的重要指标之一[15-16]。研究发现细菌的表面蛋白在乳酸菌对肠道细胞的黏附中发挥重要作用[17]。乳酸杆菌表层蛋白具有黏附、抑制病原菌及非致病性的特性,因此在口服型活性疫苗中具有潜在的应用价值[18]。在B类链球菌中,Rib蛋白为细菌的一种表面蛋白,对胰蛋白酶具有相对抗性,可刺激机体产生抗体,起到免疫保护的作用[19]。
本文在罗伊氏乳杆菌ATCC55730基因组的序列分析中发现了编码类似Rib蛋白的基因,而且该蛋白可能主要分布在细胞表面,同时存在于其他乳酸杆菌中,并且参与乳杆菌对肠道上皮细胞的黏附与定殖。为证明该推测,本文通过基因扩增、特异性抗体的制备与免疫印迹的方法,检测Rib类似蛋白在罗伊氏乳杆菌、植物乳杆菌及干酪乳杆菌中的分布及其在细菌中的亚细胞分布,为进一步研究Rib类似蛋白在乳酸杆菌对肠道的黏附机制奠定基础,从而使乳酸杆菌与宿主相互作用及其益生功能的进一步研究具有更广泛的意义。
1 材料与方法 1.1 菌株、质粒和培养基大肠杆菌E. coli BL21(DE3) (乳糖诱导表达菌株)及pET27b质粒购自Novagen公司;BL21(DE3)/L2为本实验室构建的Rib蛋白表达的重组菌株;罗伊氏乳杆菌L. reuteri ATCC55730、L. reuteri CGMCC1.2838、L. reuteri CGMCC1.3222、L. reuteri CGMCC1.3264、植物乳杆菌L. plantarum CGMCC 1.1856、L. plantarum CGMCC1.2437、L. plantarum CGMCC1.3252、L. plantarum CGMCC1.6971、干酪乳杆菌L. casei均购自中国微生物菌种保藏管理中心;L. plantarum ZN02、L. plantarum ZN07由本实验室分离并鉴定[20-21]。
LB液体培养基(g/L):胰蛋白胨10.0,酵母膏5.0,氯化钠10.0,添加卡那霉素终浓度为50.0 mg/L。MRS液体培养基(g/L):细菌蛋白胨10.0,酵母膏5.0,牛肉膏10.0,柠檬酸三铵2.15,乙酸钠5.0,葡萄糖20.0,七水硫酸镁0.58,磷酸氢二钾2.0,一水硫酸锰0.19,吐温-80 1.0 mL,pH调节为6.2-6.4。
1.2 主要试剂和仪器 1.2.1 主要试剂: 限制性内切酶购自Thermo公司;PCR体系试剂购自TaKaRa公司;Rib蛋白抗血清由中国科学院遗传与发育研究所完成;HRP标记的羊抗鼠购自Biosharp公司;蛋白预染Marker、BCA试剂盒、ECL试剂盒购自Thermo公司;DNA marker购自Life Science公司;引物由生工生物工程(上海)股份有限公司合成;生化试剂为Sigma或Biosharp公司产品,其他为国产分析纯。缓冲液配方:PBS缓冲液(g/L):磷酸二氢钾0.2,磷酸氢二钠2.9,氯化钠8.0,氯化钾0.2;TEP缓冲液:10 mmol/L Tris,1 mmol/L EDTA,1% PMSF,pH 8.0;原生质体缓冲液:10 mmol/L Tris-HCl,2 mmol/L MgCl2,26%棉籽糖,1 mmol/L PMSF,pH 8.0。
1.2.2 主要仪器: PCR仪T100、蛋白电泳仪、蛋白转膜系统、蛋白纯化系统NGC,美国Bio-Rad公司;核酸电泳仪DYY-6C,北京市六一仪器厂;紫外可见光光度计UV-1800PC,上海美普达仪器有限公司;摇床TS-200B,上海天呈实验仪器制造有限公司。 1.3 实验方法 1.3.1 表达载体的构建: 以罗伊氏乳杆菌ATCC 55730基因组DNA为模板,使用Vector NTI软件对Rib蛋白非重复区(N末端)基因序列设计引物并进行PCR扩增,引物为F (5′-GGAATTCCATATGAGAGGAATGAATAAGGAA-3′,下划线表示的是NdeⅠ酶切位点),R (5′-CCGCTCGAGTTTATCAGC ATCAAGTTCTG-3′,下划线表示的是XhoⅠ酶切位点)。PCR反应体系(50 μL):dNTP mix 4 μL,10×buffer 5 μL,Taq酶0.25 μL,引物F (5 μmol/L) 2.5 μL,引物R (5 μmol/L) 2.5 μL,DNA 1 μL,ddH2O补充至50 μL。PCR反应条件:95;95 ℃ 30 s,54 ℃ 30 s,72 ℃ 2 min,30个循环;72 ℃ 10 min。将NdeⅠ和XhoⅠ双酶切后的pET27b质粒和PCR产物纯化回收,连接过夜。连接产物转化大肠杆菌BL21(DE3) 感受态细胞,筛选出具有卡那霉素抗性并PCR验证为阳性的克隆。对阳性克隆的质粒进行酶切和测序验证,测序由生工生物工程(上海)股份有限公司完成。将验证正确的菌株命名为BL21(DE3)/L2。
1.3.2 Rib蛋白诱导表达的初步优化与蛋白纯化: 接种BL21(DE3)/L2单菌落于5 mL LB液体培养基中,37 ℃、250 r/min振荡培养过夜,1:100稀释到新的LB培养基中,相同条件培养至对数期进行目标蛋白的诱导表达。实验中对不同IPTG终浓度(0.02、0.05、0.10、0.40、1.00 mmol/L)在诱导温度25、37℃进行优化,诱导时间为12 h,摇床转速为120 r/min。通过SDS-PAGE对表达产物进行分析评价,诱导条件重复3次。Rib蛋白的纯化使用Bio-Rad NGCTM Scout 10 Plus系统,使用Histrap HP层析柱(GE)采用线性洗脱方式进行亲和层析;使用HiLoad Superdex 16/600 75pg预装柱(GE)进行凝胶过滤;使用超滤管进行超滤浓缩得到纯化的蛋白,经SDS-PAGE凝胶电泳分析达到单一蛋白条带,经BCA蛋白定量试剂盒测定蛋白浓度。 1.3.3 多克隆抗体的制备: 多克隆抗体制备参照Harlow E和Lane D的方法[22],由中国科学院遗传与发育研究所实验动物中心协助完成。利用ELISA方法[23]对抗体效价进行测定,测定结果为1:2 000。 1.3.4 Rib蛋白亚细胞定位: 根据文献[24]报道的方法对罗伊氏乳杆菌ATCC55730菌液进行处理,以分别获得细菌细胞壁、细胞质和上清液组分。具体如下:5 mL菌液培养至对数期,5 000 r/min离心5 min,分别取上清液和菌体。对于上清液,加入2倍体积无水乙醇,-80 ℃沉淀30 min。13 000 r/min离心10 min后,70%乙醇洗涤一次,ddH2O溶解沉淀。对于菌体,PBS缓冲液洗涤一次,再用TEP缓冲液洗涤两次,然后用100 μL原生质体缓冲液重悬菌体,加入60 U溶菌酶,37 ℃保温30 min。13 000 r/min离心10 min后取上清液作为细胞壁组分。沉淀用100 μL原生质体缓冲液重悬作为细胞质组分。对不同组分的蛋白经过SDS-PAGE电泳后通过免疫印迹(Western blot)确定Rib蛋白在不同细胞组分中的分布。 1.3.5 Western blot: 为检测Rib蛋白在乳杆菌中的分布情况,选取11株不同乳杆菌进行免疫印迹。以罗伊氏乳杆菌ATCC55730为阳性对照进行SDS-PAGE以及免疫印迹。接种乳酸菌单菌落于5 ml MRS液体培养基中,37 ℃、5% CO2静置培养过夜。用新鲜MRS培养基调节细菌浓度使其一致后,取1 ml培养液于5 000 r/min离心5 min收集菌体,PBS洗涤一次,80 μL TEP缓冲液重悬,加入60 U溶菌酶,37 ℃保温30 min。加入20 μL 5×Loading buffer,煮沸10 min,离心取上清进行SDS-PAGE以及免疫印迹检测与Rib抗体特异性作用的蛋白。 2 结果与分析 2.1 Rib类似蛋白基因序列分析使用NCBI在线软件Protein BLAST对L. reuteri ATCC55730的Rib蛋白氨基酸序列进行同源性检索。结果显示与该Rib蛋白具有一定同源性的蛋白在多种来源的罗伊氏乳杆菌、巴氏乳酸菌、卷曲乳杆菌、植物乳杆菌、动物乳杆菌、鼠李糖乳杆菌等乳杆菌中都被发现,在肺炎链球菌、酿脓链球菌、无乳链球菌、轻型链球菌、粪肠球菌、铜绿假单胞菌等病原菌中也被发现。这表明Rib类似蛋白在肠道微生物中普遍存在(结果未展示)。
2.2 Rib类似蛋白基因PCR检测在罗伊氏乳杆菌ATCC55730中Rib蛋白N端为一非重复的未知功能区,中间部分为8个重复的结构域,C末端含有一个保守的LPXTG转肽酶信号序列(图 1)。

|
| 图 1 罗伊氏乳杆菌ATCC55730 Rib蛋白结构示意图 Figure 1 The structural diagram of Rib protein from L. reuteri ATCC55730 注:NR:非重复区;RP:重复区. Note:NR: Non repeat region; RP: Repeat region. |
|
|
为检测Rib类似蛋白的基因是否在多种来源的乳酸杆菌中存在,以相对保守的Rib蛋白C端重复区和N端非重复区基因序列为模板分别设计引物,以11株乳杆菌基因组DNA为模板进行PCR扩增。C端引物序列如下:F-C (5′-GCTATTTC GGCAGCCAAAAG-3′);R-C (5′-CCCGGTTGTTT CTTCCCTAA-3′)。PCR条件为:95 ℃ 30 s,48-58 ℃ 30 s,72 ℃ 3 min,30个循环。N端引物序列:F-N (5′-CGGGTGTATAATGAACTA-3′);R-N (5′-TCTGTCGTCATAACCGTC-3′)。PCR条件为:95 ℃ 30 s,40-58 ℃ 30 s,72 ℃ 1 min,30个循环。
PCR结果(图 2)显示两对引物均可以从11株乳酸杆菌基因组中扩增出类似的基因片段,不同的是C末端重复区(图 2A)的PCR扩增片段大小和扩增条带亮度均比较一致,而N末端非重复区(图 2B)的PCR扩增片段则大小不同,扩增效率也有差异。4株罗伊氏乳杆菌均扩增获得约3.0 kb大小的片段,而6株植物乳杆菌获得2.0 kb左右大小片段,干酪乳杆菌中则获得约6.0 kb和0.3 kb大小的两个片段。PCR检测结果表明11株乳杆菌中均含有类似Rib蛋白的基因,且基因的大小不同。要确认Rib类似蛋白是否存在,需要通过免疫印迹的方法直接对蛋白质进行检测。
|
| 图 2 乳酸杆菌中Rib类似蛋白的PCR分析 Figure 2 PCR analyses of Rib like protein in different Lactobacillus strains 注:A:重复区;B:非重复区. M:DNA分子量标品;1:罗伊氏乳杆菌ATCC55730;2:阴性对照;3:干酪乳杆菌L. casei;4:植物乳杆菌ZN02;5:植物乳杆菌ZN07;6:植物乳杆菌CGMCC1.1856;7:植物乳杆菌CGMCC1.2437;8:罗伊氏乳杆菌CGMCC1.2838;9:罗伊氏乳杆菌CGMCC1.3222;10:植物乳杆菌CGMCC1.3252;11:罗伊氏乳杆菌CGMCC1.3264;12:植物乳杆菌CGMCC1.6971. Note: A: Repeat region; B: Non repeat region. M: DNA ladder; 1: L. reuteri ATCC55730; 2: Negative control; 3: L. casei; 4: L. plantarum ZN02; 5: L. plantarum ZN07; 6: L. plantarum CGMCC1.1856; 7: L. plantarum CGMCC1.2437; 8: L. reuteri CGMCC1.2838; 9: L. reuteri CGMCC1.3222; 10: L. plantarum CGMCC1.3252; 11: L.reuteri CGMCC1.3264; 12: L. plantarum CGMCC1.6971. |
|
|
为制备Rib蛋白特异性抗体,通过基因重组表达的方法,构建了含有部分Rib基因的重组大肠杆菌BL21(DE3)/L2。该工程菌中含有乳糖启动子引导的Rib蛋白N末端非重复区基因,在IPTG的诱导下可表达相应蛋白。首先对不同温度下的表达量进行初步实验,发现37 ℃条件下诱导12 h时表达的蛋白多为不溶性包涵体,而在较低温度25 ℃下为可溶性蛋白(结果未展示)。在此基础上对IPTG浓度进行优化,分别选取IPTG浓度为0.02、0.05、0.10、0.40和1.00 mmol/L。结果如图 3所示,当IPTG浓度为0.02 mmol/L时,目标蛋白的表达量较低,当IPTG浓度大于0.05 mmol/L时,目标蛋白的表达量变化不大。因而研究中采用25℃、0.05 mmol/L IPTG诱导12 h表达的条件,依次经过了His亲和层析和凝胶过滤的方法进行纯化后,经BCA蛋白定量试剂盒测定浓度为1.06 g/L,纯化的蛋白用于Rib蛋白特异性抗体制备。

|
| 图 3 IPTG浓度对Rib蛋白表达的影响 Figure 3 Effect of IPTG concentrations on the expression of Rib protein in E. coli 注:M:蛋白质分子量标准品;1:大肠杆菌BL21(DE3);2:未诱导大肠杆菌BL21(DE3)/L2;3:大肠杆菌BL21(DE3)/L2 (0.02 mmol/L IPTG);4:大肠杆菌BL21(DE3)/L2 (0.05 mmol/L IPTG);5:大肠杆菌BL21(DE3)/L2 (0.10 mmol/L IPTG);6:大肠杆菌BL21(DE3)/L2 (0.40 mmol/L IPTG);7:大肠杆菌BL21(DE3)/L2 (1.00 mmol/L IPTG). Note: M: Protein molecular weight ladder; 1: E. coli BL21(DE3); 2: Uninduced E. coli BL21(DE3)/L2; 3: E. coli BL21(DE3)/L2 (0.02 mmol/L IPTG); 4: E. coli BL21(DE3)/L2 (0.05 mmol/L IPTG); 5: E. coli BL21(DE3)/L2 (0.10 mmol/L IPTG); 6: E. coli BL21(DE3)/L2 (0.40 mmol/L IPTG); 7: E. coli BL21(DE3)/L2 (1.00 mmol/L IPTG). |
|
|
由于通过Rib蛋白的结构表明其N末端具有典型的信号肽序列,C末端序列为具有典型的细胞壁锚定结构LPXTG及一段富含疏水氨基酸的尾巴。结构表明Rib蛋白可能是一个细胞表面蛋白,通过锚定酶分布在细菌细胞壁上。为证明该蛋白的亚细胞分布,将细胞培养后的培养基上清液、细胞壁和细胞质分离后再通过SDS-PAGE和免疫印迹进行检测(图 4)。结果显示在细胞壁蛋白部分能检测到约190 kD大小的特异蛋白条带,而在细胞质和培养基上清液中未能检测到相应条带,这表明Rib蛋白分布在细菌细胞的细胞壁上。

|
| 图 4 Rib蛋白在罗伊氏乳杆菌不同细胞组分中的分布 Figure 4 Subcellular distribution of Rib in L. reuteri 注:1:细胞壁;2:细胞质;3:培养基上清液. Note: 1: Cell wall; 2: Cytoplasm; 3: Cultural supernatant. |
|
|
为检测不同乳酸杆菌中Rib类似蛋白的存在,11株不同的菌株培养至对数期的细胞总蛋白经过SDS-PAGE和Western blot检测(图 5)。SDS-PAGE结果表明经过处理后的细胞总蛋白量比较一致,但罗伊氏乳杆菌(图 5A,泳道1-4) 与植物乳杆菌(图 5A,泳道5-10) 的蛋白谱有较大的差异,干酪乳杆菌(图 5A,泳道11) 的蛋白谱接近于植物乳杆菌。而Western blot结果表明所有乳酸杆菌中均能够检测到特异杂交条带。4株罗伊氏乳杆菌中所检测到的蛋白(图 5B,泳道1-4) 大小相似,约为190 kD,而6株植物乳杆菌中所检测到的蛋白(图 5B,泳道1-4) 大小也相似,约为100 kD,而干酪乳杆菌中的蛋白条带则与前两类不同,约为130 kD (图 5B,泳道11)。
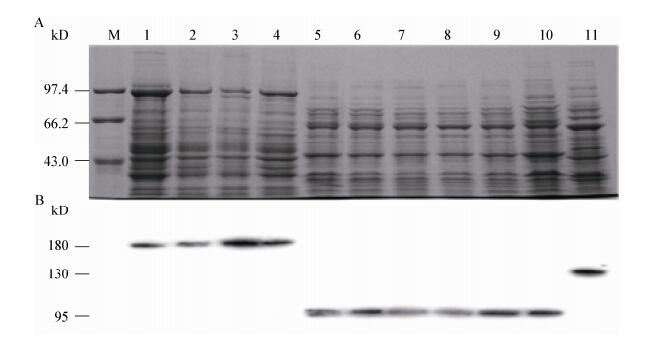
|
| 图 5 Rib蛋白在不同乳酸杆菌中的检测 Figure 5 Detection of Rib protein in different Lactobacilli strains 注:A:总蛋白的SDS-PAGE;B:Rib抗体的免疫印迹分析. M:蛋白质分子量标品;1:罗伊氏乳杆菌ATCC55730;2:罗伊氏乳杆菌CGMCC1.2838;3:罗伊氏乳杆菌CGMCC1.3222;4:罗伊氏乳杆菌CGMCC1.3264;5:植物乳杆菌ZN02;6:植物乳杆菌ZN07;7:植物乳杆菌CGMCC1.1856;8:植物乳杆菌CGMCC1.2437;9:植物乳杆菌CGMCC1.3252;10:植物乳杆菌CGMCC1.6971;11:干酪乳杆菌L. casei. Note: A: SDS-PAGE of the total protein; B: Western blot analyses against Rib antibody. M: Protein marker; 1: L. reuteri ATCC55730; 2: L. reuteri CGMCC1.2838; 3: L. reuteri CGMCC1.3222; 4: L. reuteri CGMCC1.3264; 5: L. plantarum ZN02; 6: L. plantarum ZN07; 7: L. plantarum CGMCC1.1856; 8: L. plantarum CGMCC1.2437; 9: L. plantarum CGMCC1.3252; 10: L. plantarum CGMCC1.6971; 11: L. casei. |
|
|
Rib蛋白是罗伊氏乳杆菌细胞表面的大分子蛋白,可能与细菌的黏附与定殖有关。Rib蛋白最先被报道是在Ⅲ型B类链球菌中[25],对其蛋白的氨基酸序列分析表明该蛋白N-末端区域具有174个残基的非重复区,C端为12个重复区域且含有一个保守的LPXTG转肽酶信号序列[26]。进一步研究发现Rib蛋白的抗体可以保护小鼠免受B类链球菌的侵染[25]。本研究分析罗伊氏乳杆菌基因组序列后发现了这个类似Rib蛋白的大分子蛋白,其结构与B类链球菌Rib蛋白相似。通过基因克隆与免疫印迹的方法分别在DNA和蛋白质水平检测到了Rib蛋白在罗伊氏乳杆菌中的存在,并且发现该蛋白分布在细菌的细胞壁上,为进一步研究其在细菌黏附与定殖中的功能奠定了基础。这是首次在乳酸杆菌中对Rib类似蛋白的研究。
Rib类似蛋白可能存在于多种乳酸杆菌中。应用罗伊氏乳杆菌Rib蛋白特异性抗体的免疫印迹结果显示,Rib类似蛋白不仅在罗伊氏乳杆菌中存在,而且在植物乳杆菌以及干酪乳杆菌中也可被检测到,并且其大小在同一种内相同,种间表现差异。在植物乳杆菌AY01基因组分析中也可发现相应大小类似结构的蛋白[27]。更多对类似蛋白的研究在肺炎双球菌、无乳链球菌、霍乱弧菌等常见致病菌中较为常见,并且对此类蛋白的免疫特性、基因、结构、功能等进行了大量研究,结果表明Rib类似蛋白参与了病原细菌与其宿主相互作用从而发挥其致病性的过程[19, 28-32]。在益生菌的研究中,大量研究表明细菌表面蛋白在益生菌的黏附与定殖中起着重要作用,如乳杆菌的S层蛋白Slp[33]和SlpA[34]、双歧杆菌的DnaK蛋白[35]、双歧杆菌中的BopA蛋白[36]、植物乳杆菌His-N2蛋白[37]。另外的研究发现细胞表面蛋白不仅参与了细胞的黏附,也与免疫细胞的激活相关[38-39]。罗伊氏乳杆菌是在肠道定殖力最强的乳酸菌之一,而对于其Rib类似蛋白功能的研究尚未见报道,本文的初步研究发现这种蛋白在不同的乳酸杆菌中广泛存在,因而对其功能的研究不仅有助于对罗伊氏乳杆菌肠道黏附机制的阐明,也会对更多具有Rib类似蛋白的乳酸杆菌的研究与应用提供有力的参考。
| [1] |
Michon C, Langella P, Eijsink VGH, et al. Display of recombinant proteins at the surface of lactic acid bacteria: strategies and applications[J]. Microbial Cell Factories, 2016, 15(1): 70. DOI:10.1186/s12934-016-0468-9 |
| [2] |
Hevia A, Delgado S, Sánchez B, et al. Molecular players involved in the interaction between beneficial bacteria and the immune system[J]. Frontiers in Microbiology, 2015, 6: 1285. |
| [3] |
McCabe L, Britton RA, Parameswaran N. Prebiotic and probiotic regulation of bone health: role of the intestine and its microbiome[J]. Current Osteoporosis Reports, 2015, 13(6): 363-371. DOI:10.1007/s11914-015-0292-x |
| [4] |
Thomas LV, Suzuki K, Zhao J. Probiotics: a proactive approach to health. A symposium report[J]. British Journal of Nutrition, 2015, 114(S1): S1-S15. DOI:10.1017/S0007114515004043 |
| [5] |
McKay LL, Baldwin KA. Applications for biotechnology: present and future improvements in lactic acid bacteria[J]. FEMS Microbiology Letters, 1990, 87(1/2): 3-14. |
| [6] |
Wei MQ, Rush CM, Norman JM, et al. An improved method for the transformation of Lactobacillus strains using electroporation[J]. Journal of Microbiological Methods, 1995, 21(1): 97-109. DOI:10.1016/0167-7012(94)00038-9 |
| [7] |
Redondo-Lopez V, Cook RL, Sobel JD. Emerging role of lactobacilli in the control and maintenance of the vaginal bacterial microflora[J]. Reviews of Infectious Diseases, 1990, 12(5): 856-872. DOI:10.1093/clinids/12.5.856 |
| [8] |
Wang B, Wei H, Yuan J, et al. Identification of a surface protein from Lactobacillus reuteri JCM1081 that adheres to porcine gastric mucin and human enterocyte-like HT-29 cells[J]. Current Microbiology, 2008, 57(1): 33-38. DOI:10.1007/s00284-008-9148-2 |
| [9] |
Chan RC, Reid G, Irvin RT, et al. Competitive exclusion of uropathogens from human uroepithelial cells by Lactobacillus whole cells and cell wall fragments[J]. Infection and Immunity, 1985, 47(1): 84-89. |
| [10] |
Tihole F. Possible treatment of AIDS patients with live lactobacteria[J]. Medical Hypotheses, 1988, 26(1): 85-88. DOI:10.1016/0306-9877(88)90119-3 |
| [11] |
Liu CJ, Bian X, Zhao SJ. Screening of Lactobacillus reuteri inhibiting spoilage organisms and its application to yoghourt production[J]. Food Science, 2016, 37(7): 157-162. (in Chinese) 刘春娟, 边鑫, 赵士举. 具有抑制腐败菌能力的罗伊氏乳杆菌筛选及在酸乳生产中的应用[J]. 食品科学, 2016, 37(7): 157-162. DOI:10.7506/spkx1002-6630-201607029 |
| [12] |
Liu YY, Fatheree NY, Dingle BM, et al. Lactobacillus reuteri DSM 17938 changes the frequency of Foxp3+ regulatory T cells in the intestine and mesenteric lymph node in experimental necrotizing enterocolitis[J]. PLoS One, 2013, 8(2): e56547. DOI:10.1371/journal.pone.0056547 |
| [13] |
Thomas CM, Hong T, van Pijkeren JP, et al. Histamine derived from probiotic Lactobacillus reuteri suppresses TNF via modulation of PKA and ERK signaling[J]. PLoS One, 2012, 7(2): e31951. DOI:10.1371/journal.pone.0031951 |
| [14] |
Dong HL, Rowland I, Yaqoob P. Comparative effects of six probiotic strains on immune function in vitro[J]. British Journal of Nutrition, 2012, 108(3): 459-470. DOI:10.1017/S0007114511005824 |
| [15] |
Liu L, Wu RY, Li J, et al. Adhesion ability of Lactobacillus paraplantarum L-ZS9 and influencing factors of its biofilm formation[J]. Food Science, 2016, 37(15): 136-143. (in Chinese) 刘蕾, 武瑞赟, 李军, 等. 类植物乳杆菌L-ZS9生物被膜态的黏附能力及形成影响因素[J]. 食品科学, 2016, 37(15): 136-143. |
| [16] |
Zhang YC, Xiang XL, Zhang LW, et al. A review on the structure and immune regulation function of Lactobacillus S-layer protein[J]. Food Science, 2016, 37(3): 229-234. (in Chinese) 张英春, 向鑫玲, 张兰威, 等. 乳酸杆菌S-层蛋白的结构及免疫调控功能研究进展[J]. 食品科学, 2016, 37(3): 229-234. DOI:10.7506/spkx1002-6630-201603040 |
| [17] |
Pang RP. Advances in Lactobacilli adhesion to human intestinal epithelium[J]. China Dairy Cattle, 2014(15): 46-48. (in Chinese) 庞瑞鹏. 乳酸杆菌黏附人肠道上皮功能的研究进展[J]. 中国奶牛, 2014(15): 46-48. DOI:10.3969/j.issn.1004-4264.2014.15.012 |
| [18] |
Hyn nen U, Palva A. Lactobacillus surface layer proteins: structure, function and applications[J]. Applied Microbiology and Biotechnology, 2013, 97(12): 5225-5243. DOI:10.1007/s00253-013-4962-2 |
| [19] |
Lindahl G, St lhammar-Carlemalm M, Areschoug T. Surface proteins of Streptococcus agalactiae and related proteins in other bacterial pathogens[J]. Clinical Microbiology Reviews, 2005, 18(1): 102-127. DOI:10.1128/CMR.18.1.102-127.2005 |
| [20] |
Saeedi M, Shahidi F, Mortazavi SA, et al. Isolation and identification of lactic acid bacteria in winter salad (local pickle) during fermentation using 16S rRNA gene sequence analysis[J]. Journal of Food Safety, 2015, 35(3): 287-294. DOI:10.1111/jfs.2015.35.issue-3 |
| [21] |
Guo LD, Liu Q, Jiang LQ, et al. Isolation and properties of Lactococcuslactis strain from kefir grains[J]. Modern Food Science and Technology, 2014, 30(9): 121-125. (in Chinese) 国立东, 刘倩, 江柳青, 等. 开菲尔粒中一株乳酸乳球菌的分离及性能研究[J]. 现代食品科技, 2014, 30(9): 121-125. |
| [22] |
Wright K. Antibodies: a laboratory manual, by E Harlow and D Lane. pp 726. Cold Spring Harbor Laboratory, 1988. $50, ISBN 0-87969-314-2[J]. Biochemical Education, 1989, 17(4): 20. |
| [23] |
Pappas MG, Hajkowski R, Hockmeyer WT. Standardization of the dot enzyme-linked immunosorbent assay (Dot-ELISA) for human visceral leishmaniasis[J]. American Journal of Tropical Medicine and Hygiene, 1984, 33(6): 1105-1111. DOI:10.4269/ajtmh.1984.33.1105 |
| [24] |
Liang XB, Chen YYM, Ruiz T, et al. New cell surface protein involved in biofilm formation by Streptococcus parasanguinis[J]. Infection and Immunity, 2011, 79(8): 3239-3248. DOI:10.1128/IAI.00029-11 |
| [25] |
St lhammar-Carlemalm M, Stenberg L, Lindahl G. Protein rib: a novel group B streptococcal cell surface protein that confers protective immunity and is expressed by most strains causing invasive infections[J]. The Journal of Experimental Medicine, 1993, 177(6): 1593-1603. DOI:10.1084/jem.177.6.1593 |
| [26] |
W stfelt M, St lhammar-Carlemalm M, Delisse AM, et al. Identification of a family of streptococcal surface proteins with extremely repetitive structure[J]. Journal of Biological Chemistry, 1996, 271(31): 18892-18897. DOI:10.1074/jbc.271.31.18892 |
| [27] |
Li XR, Gong FM, Zheng HJ, et al. Draft genome sequence of Lactobacillus plantarum strain AY01, isolated from the raw material of fermented goat milk cheese[J]. Genome Announcements, 2013, 1(5): e00737-13. |
| [28] |
Maeland JA, Afset JE, Lyng RV, et al. Survey of immunological features of the alpha-like proteins of Streptococcus agalactiae[J]. Clinical and Vaccine Immunology, 2015, 22(2): 153-159. DOI:10.1128/CVI.00643-14 |
| [29] |
Deng YQ, Wang KY, Wang J, et al. Cloning, identification and molecular characteristics analysis of α protein gene of Streptococcus agalactiae from Tilapia[J]. Sichuan Journal of Zoology, 2014, 33(5): 700-707. (in Chinese) 邓永强, 汪开毓, 王均, 等. 罗非鱼无乳链球菌α蛋白基因的克隆、鉴定及分子特性分析[J]. 四川动物, 2014, 33(5): 700-707. |
| [30] |
Almeida A, Villain A, Joubrel C, et al. Whole-genome comparison uncovers genomic mutations between group B streptococci sampled from infected newborns and their mothers[J]. Journal of Bacteriology, 2015, 197(20): 3354-3366. DOI:10.1128/JB.00429-15 |
| [31] |
Dangor Z, Kwatra G, Izu A, et al. HIV-1 is associated with lower Group B Streptococcus capsular and surface-protein IgG antibody levels and reduced transplacental antibody transfer in pregnant women[J]. Journal of Infectious Diseases, 2015, 212(3): 453-462. DOI:10.1093/infdis/jiv064 |
| [32] |
Kristian SA, Ota T, Bubeck SS, et al. Generation and improvement of effector function of a novel broadly reactive and protective monoclonal antibody against pneumococcal surface protein A of Streptococcus pneumoniae[J]. PLoS One, 2016, 11(5): e0154616. DOI:10.1371/journal.pone.0154616 |
| [33] |
Johnson-Henry KC, Hagen KE, Gordonpour M, et al. Surface-layer protein extracts from Lactobacillus helveticus inhibit enterohaemorrhagic Escherichia coli O157:H7 adhesion to epithelial cells[J]. Cellular Microbiology, 2007, 9(2): 356-367. DOI:10.1111/cmi.2007.9.issue-2 |
| [34] |
Buck BL, Altermann E, Svingerud T, et al. Functional analysis of putative adhesion factors in Lactobacillus acidophilus NCFM[J]. Applied and Environmental Microbiology, 2005, 71(12): 8344-8351. DOI:10.1128/AEM.71.12.8344-8351.2005 |
| [35] |
Candela M, Turroni S, Centanni M, et al. Relevance of Bifidobacterium animalis subsp. lactis plasminogen binding activity in the human gastrointestinal microenvironment[J]. Applied and Environmental Microbiology, 2011, 77(19): 7072-7076. DOI:10.1128/AEM.00413-11 |
| [36] |
Gleinser M, Grimm V, Zhurina D, et al. Improved adhesive properties of recombinant bifidobacteria expressing the Bifidobacterium bifidum-specific lipoprotein BopA[J]. Microbial Cell Factories, 2012, 11(1): 80. DOI:10.1186/1475-2859-11-80 |
| [37] |
Du LH, Zhang H, Shi RH, et al. In vitro exclusive inhibition of E. coli DH5α adhesion to porcine intestinal mucus by His-N2 protein[J]. Food Science, 2014, 35(11): 95-99. (in Chinese) 都立辉, 张虹, 施荣华, 等. His-N2蛋白体外排除抑制大肠杆菌DH5α黏附猪肠黏液蛋白的研究[J]. 食品科学, 2014, 35(11): 95-99. DOI:10.7506/spkx1002-6630-201411019 |
| [38] |
Li Q, Liu XL, Wang Y, et al. Surface properties and adhesion to Caco-2 cells of Lactobacillus planturam strains[J]. Food Science, 2015, 36(9): 97-101. (in Chinese) 李清, 刘小莉, 王英, 等. 植物乳杆菌表面性质及对Caco-2细胞的黏附[J]. 食品科学, 2015, 36(9): 97-101. DOI:10.7506/spkx1002-6630-201509018 |
| [39] |
Li HH, Yang ZQ, Wang XL, et al. Surface proteins of lactic acid bacteria induce the proliferation and inhibit the adherence of immunocytes[J]. Chinese Journal of Microecology, 2016, 28(1): 15-20. (in Chinese) 李海红, 杨振泉, 王晓霖, 等. 乳酸菌表面蛋白对免疫细胞的诱导增殖及粘附抑制效应[J]. 中国微生态学杂志, 2016, 28(1): 15-20. |
 2017, Vol. 44
2017, Vol. 44