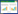扩展功能
文章信息
- 张欣, 刘静, 沈国平, 龙启福, 韩睿, 朱德锐
- ZHANG Xin, LIU Jing, SHEN Guo-Ping, LONG Qi-Fu, HAN Rui, ZHU De-Rui
- 基于高通量测序研究青藏高原茶卡盐湖微生物多样性
- Illumina-based sequencing analysis of microbial community composition in Chaka Salt Lake in Qinghai-Tibet Plateau
- 微生物学通报, 2017, 44(8): 1834-1846
- Microbiology China, 2017, 44(8): 1834-1846
- DOI: 10.13344/j.microbiol.china.160752
-
文章历史
- 收稿日期: 2016-10-19
- 接受日期: 2017-02-20
- 优先数字出版日期(www.cnki.net): 2017-03-16
2. 青海大学农林科学院 青海 西宁 810016
2. Academy of Agriculture and Forestry, Qinghai University, Xining, Qinghai 810016, China
我国的盐湖资源丰富,大小盐湖共计1 500多个,主要分布于新疆、青藏高原、内蒙古及其周边地区[1]。其中,青藏高原柴达木盆地(平均海拔为4 000 m)是高原湖泊聚集最密集的地区,分布着地球上面积最大(2×106 km2)、数量最多以及盐碱湖集中的特色高原盐湖群,如达布逊湖、托素湖、可鲁克湖、伊克柴达木湖、苏干湖、尕斯库勒湖、巴嘎柴达木湖、茶卡盐湖以及柯柯盐湖等。盐湖作为高盐浓度的特殊环境,水体中栖息着大量的极端嗜盐微生物[2],然而不同的盐湖又具有不同的水化学性质和自然环境条件,以此制约微生物的物种多样性和种质资源分布[3]。因此,全面了解我国西部盐湖的微生物群落多样性及结构特征,对盐湖资源的开发和合理利用具有重要的实际意义。
茶卡盐湖(99°04′41.67″E,36°45′0.05″N)位于我国西部的柴达木盆地,平均海拔为3 017 m,属于天然结晶盐湖,湖水总矿化度达322.4 g/L,是青藏高原重要的固液相石盐矿床之一。柴丽红等[4]采用DGGE (Denaturing gradient gel electrophoresis,DGGE)法和纯培养法对茶卡盐湖底泥及水样进行菌株分离,获得纯培养菌株16株,并利用DGGE和ARDRA (Amplified ribosomal DNA restriction analysis,ARDRA)结合分析了12株菌的系统发育关系。虽然分离培养技术有利于微生物资源的开发及利用,但仅通过传统纯培养技术只能获得1%左右甚至更低的可培养微生物,对于认识环境微生物的多样性及群落结构是远远不够的[5]。高通量测序技术具有低成本、高通量和高准确度等优势,应用广泛,已成为当前研究环境微生物多样性及群落结构差异的重要技术手段[6-7]。针对茶卡盐湖细菌和古菌群落结构及多样性的相关研究尚有不足,本研究采用高通量测序技术分析茶卡盐湖微生物的群落结构,旨在系统了解盐湖水体和底泥中细菌和古菌的多样性,从而为后续极端环境微生物资源的挖掘及嗜盐菌的开发提供理论依据。
1 材料与方法 1.1 样品采集及处理采样地点:青海省海西州乌兰县茶卡镇茶卡盐湖(海拔为3 017 m);采样时间:2015年7月下旬;样点位置:盐湖景区入口边缘区样点CSL1 (99°5′17.840 4″E,36°45′23.385 0″N),盐湖景区西侧深水样点CSL2 (99°5′9.934 8″E,36°45′15.865 2″N),盐湖景区东侧深水样点CSL3 (99°6′5.169 6″E,36°44′48.875 4″N)和莫河盐场深水样点CSL4 (99°10′22.976 4″E,36°42′53.294 4″N),取样点间距大于100 m;样品采集深度为20 cm,样品为茶卡盐湖水样和底泥混合物,浑浊状态。检测水体温度为21.2-22.6 ℃,氧含量为1.54-1.58 mg/L。样本储存于4 ℃车载冰箱,并立即运回实验室进行操作。各样本的理化性质测定(Na+、K+、Mg2+、Cl-等)由上海微谱化工技术公司完成,离子检测标准参照离子色谱分析方法通则(JY/T 020-1996)。
1.2 主要试剂和仪器DNA提取试剂盒(QIAamp Fast DNA Stool Mini Kit),上海赛百盛公司;微量检测仪Microplate Reader,美国MD公司;引物合成,上海美吉生物公司;TransStart Fastpfu DNA Polymerase (TransGen AP221-02),北京全式金生物公司;AxyPrep DNA凝胶回收试剂盒,AXYGEN公司。QuantiFluor™-ST蓝色荧光定量系统,Promega公司;ICS-1600型离子色谱仪,美国戴安公司;MiSeq PE300测序平台,美国Illumina公司;GeneAmp® 9700型PCR仪,美国ABI公司。
1.3 DNA的提取与PCR扩增将采集的茶卡盐湖4个样品(水样与底泥混合物)进行0.22 μm滤膜真空抽滤,将滤膜剪碎放入DNA试剂盒,参照试剂盒步骤提取基因组DNA,2.0%琼脂糖凝胶电泳分析样本DNA的完整性。采用微量检测仪Microplate Reader分析DNA的质量纯度,合格样品的总DNA保存于-80 ℃。采用细菌16S rRNA基因V3-V4区和古菌16S rRNA基因V3-V5区进行PCR扩增,细菌引物为338F (5′-ACT CCTACGGGAGGCAGCAG-3′)和806R (5′-GGACT ACHVGGGTWTCTAAT-3′);古菌引物为344F (5′-AC GGGGYGCAGCAGGCGCGA-3′)和915R (5′-GTGCT CCCCCGCCAATTCCT-3′)。PCR反应体系(20 μL):5×FastPfu buffer 4 μL,2.5 mmol/L dNTPs 2 μL,正、反向引物(浓度为5 μmol/L)各0.8 μL,TransStart FastPfu DNA Polymerase 0.4 μL,DNA模板2 μL,ddH2O补充至20 μL。PCR反应条件:95 ℃ 3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,细菌和古菌分别为27和32个循环;72 ℃ 10 min。每个样本3个重复,将同一样本的PCR产物混合后用2%琼脂糖凝胶电泳检测,采用DNA凝胶回收试剂盒切胶回收PCR产物,Tris-HCl洗脱。PCR产物采用蓝色荧光定量系统进行定量检测,按照每个样本的测序量要求进行相应比例的混合,委托上海美吉生物有限公司进行高通量测序。
1.4 测序数据优化处理MiSeq PE300测序平台获得双端序列数据,每个样本的数据量为两三万条序列。为了保证后续生物信息分析的准确性,对测序数据进行质量控制。基于PE reads之间的Overlap关系,将成对的Reads拼接成一条序列,同时对Reads的质量和Merge的效果进行质控过滤。根据序列首尾两端的Barcode和引物序列特征筛选有效序列,并校正序列方向。利用Trimmomatic和FLASH软件平台筛选拼接序列,过滤Read尾部质量值20以下的碱基,Overlap区允许的错配率低于0.2,Barcode错配数为0,最大引物错配数为2。
1.5 群落结构与多样性分析采用Usearch (version 7.1) 软件进行数据处理,基于97%相似度水平筛选OTU,然后根据各样品OTU与Silva数据库的相似性比对结果进行优势物种的OTU picking和物种注释分析,将所得结果采用Graphpad Prism 5软件绘制群落结构组分图(门Phylum、纲Class与属Genus水平)。根据分类学统计结果分析微生物的群落结构、物种组成比例及丰度,绘制Rank-Abundance曲线[8],以此反映样本所含物种的丰富度和均匀度。利用Mothur软件进行多样性指数(Alpha-diversity)、Chao1、ACE、香农指数(Shannon)和辛普森指数(Simpson)等计算分析。
1.6 样品间相似性聚类分析基于茶卡盐湖的细菌和古菌代表性序列数计算并绘制热图(Heatmap)[9],反映各样本在不同分类水平上群落组成的相似性和差异性。采用R语言Vegan包进行Vegdist距离计算和Hclust聚类分析,距离算法采用Bray-Curtis法,聚类方法为Complete。
1.7 环境因子相关性分析基于水质离子特征数据和OTU注释数据(Species和Sample数据,97%相似度),采用多元直接梯度回归法分析菌群与环境因子之间的相关性。采用R软件语言Vegan包进行典范对应分析(Canonical correlation analyses,CCA)和作图,Bioenv函数判断环境因子与样本群落分布差异的最大相关系数Pearson,Permutest判断CCA分析的显著性。
1.8 序列登录号本研究所得序列均已提交至GenBank数据库中,细菌BioSample登录号为SAMN04376877-SAMN049376880,古菌BioSample登录号为SAMN05356250-SAMN05356253。
2 结果与分析 2.1 高通量测序数据优化分析通过MiSeq测序平台对茶卡盐湖4个水体生境样本的16S rRNA基因V3-V5区进行微生物多样性检测分析,分别获得细菌总优化序列117 192条和古菌110 571条,碱基平均长度分别为449 bp和268 bp。利用Mothur软件做Rarefaction分析,制作稀释曲线(图 1)。由图 1可知,随着测序序列数目的增加,测序样本的曲线基本趋向平稳,表明测序数据量渐近合理。其中,样品CSL2/3/4来源于相似的盐湖深水区水样,细菌和古菌的测序深度可以覆盖样品中微生物的数量。样品CSL1来源盐湖边缘区域,OTU数量显著多于CSL2/3/4,可能因为采样地点位于盐湖边缘区,易受到降水、外界土壤微生物以及人类活动等因素的影响所致。

|
| 图 1 细菌和古菌稀释性曲线 Figure 1 Rarefaction curve of bacterial and archaeal sample 注:CSL1/2/3/4代表细菌稀释性曲线;CSL1a/2a/3a/4a代表古菌稀释性曲线. Note: CSL1/2/3/4 represent rarefaction curve of bacteria; CSL1a/2a/3a/4a represent rarefaction curve of archaea. |
|
|
在Silva数据库[10]进行相似性比对,采用RDP classifier贝叶斯算法进行97%相似水平的OTU序列分类,获得细菌OTU 421个和古菌OTU 317个。根据分类学注释结果,分别在门Phylum、纲Class和属Genus水平下进行样品物种丰度的统计分析(图 2)。从图 2A可知,茶卡盐湖细菌门类群多集中于厚壁菌门Firmicutes (68.37%)、变形菌门Proteobacteria (20.49%)和拟杆菌门Bacteroidetes (8.46%),而放线菌门Actinobacteria (1.25%)、其它及未分类细菌所占比例相对较少(0.49%和0.94%)。通过纲分类层次(图 2B)统计分析,表明茶卡盐湖以芽孢杆菌纲Bacilli (60.45%)为主,其次为γ-Proteobacteria (17.26%)、梭菌纲Clostridia (7.91%)和纤维粘网菌纲Cytophagia (5.20%),其他所占比例相对较少。通过种属分类水平上的统计分析(图 2C),确定茶卡盐湖的优势种群是芽孢杆菌属Bacillus (41.94%)、海洋芽孢杆菌属Oceanobacillus(8.03%)和假单胞菌属Pseudomonas (7.67%),然而因CSL1采样地点为盐湖边缘区,相对于盐湖深水区CSL2/3/4而言,更易受淡水补给、周边土壤微生物渗透及人为因素等外界条件的干扰,致使优势细菌种群的丰度比例有所差异。

|
| 图 2 茶卡盐湖微生物分类学水平的相对丰度统计分析 Figure 2 Statistical analysis of relative abundance at the microbial taxonomic levels of Chaka salt Lake 注:A:细菌门分类;B:细菌纲分类;C:细菌属分类;D:古菌门分类;E:古菌纲分类;F:古菌属分类. Note: A: Bacteria phylum; B: Bacteria class; C: Bacteria genus; D: Archaea phylum; E: Archaea class; F: Archaea genus. |
|
|
从图 2D可知,茶卡盐湖古菌类群多集中于广古菌门Euryarchaeota (96.50%)以及部分Unclassified archaea (2.16%)。通过纲分类层次(图 2E)统计,表明茶卡盐湖古菌以盐杆菌纲Halobacteria (94.57%)为主,而甲烷杆菌纲Methanobacteria (0.77%)、甲烷微菌纲Methanomicrobia (0.72%)和热源体纲Thermoplasmata (0.43%)等相对较少。从属分类水平(图 2F)分析,表明丰度较高且4个样品均有分布的主要属类群有Halonotius (17.21%)、Halorubrum (16.23%)、盐盒菌属Haloarcula (8.34%)、Halapricum (6.37%)、Halovenus、Halobellus、Halorubellus、嗜盐碱单胞菌属Natronomonas、盐盘菌属Haloplanus、Halomicroarcula、嗜盐球菌属Halococcus、Halolamina、盐棍菌属Halorhabdus、盐简菌属Halosimplex、盐微菌属Halomicrobium、Nanohaloarchaeota和盐杆菌科(Halobacteriaceae)中未确定分类地位的属等。其中CSL1a的优势菌为盐盒菌属Haloarcula,占到20.04%;CSL2a和CSL3a的优势种群为Halonotius,所占比例分别为38.27%和22.30%;CSL4a的优势菌为Halobacteriaceae中未确定分类地位的属,比例为18.39%。有些类群仅在特定的样品中出现,如CSL1a和CSL2a特有的菌群为盐杆菌属Halobacterium,CSL3a和CSL4a出现特有的候选KTK4A norank等,但这些特有菌群的相对丰度通常较低。此外,尚存在大量未确定分类地位(Unclassified)和未纯培养(Uncultured)的古菌类群,两者占比例分别为12.57%和6.25%。
2.3 茶卡盐湖微生物多样性分析将高通量测序结果进行比对,获得分类学地位明确的细菌有14门28纲170属,古菌有5门4纲34属,未确定分类地位(Unclassified)的细菌和古菌共计有10门24纲90属。此外,盐湖生境中还存在部分未指定分类(Unassigned)的微生物,至少在分类学上还未归属于细菌或古菌。利用Mothur软件进行多样性指数(Alpha-diversity)统计分析(表 1),表明多样性测序结果覆盖率均在99.57%以上,数据有效可靠。从整体来看,茶卡盐湖边缘区样点CLS1的细菌OTU丰富度、物种总数与群落多样性指数明显高于古菌,同时也高于盐湖中心深水样点CSL2/3/4。对比分析盐湖中心深水样CSL2/3/4,表明受中心区域饱和盐度的影响,古菌的群落种类多样性、OTU丰富度和物种总数均显著高于细菌。
| 分类 Classification | 样本 Sample | 有效读数 Effective reads | OTU | Shannon | Simpson | Coverage | Ace指数 Ace index | Chao1指数 Chao1 index |
| 细菌 Bacteria | CSL1 | 32 762 | 363 | 3.84 | 0.054 | 99.57 | 535 | 513 |
| CSL2 | 27 707 | 170 | 2.56 | 0.140 | 99.77 | 228 | 223 | |
| CSL3 | 31 365 | 86 | 2.39 | 0.152 | 99.93 | 98 | 110 | |
| CSL4 | 25 358 | 100 | 2.40 | 0.153 | 99.89 | 126 | 133 | |
| 古菌 Archaea | CSL1a | 28 204 | 245 | 4.01 | 0.045 | 99.80 | 260 | 263 |
| CSL2a | 26 057 | 197 | 3.41 | 0.078 | 99.77 | 219 | 222 | |
| CSL3a | 35 526 | 121 | 3.76 | 0.042 | 99.90 | 132 | 141 | |
| CSL4a | 20 784 | 136 | 4.22 | 0.021 | 99.93 | 144 | 140 |
根据Usearch软件分析每个有效序列OTU所占的丰度比例绘制Heatmap,结果如图 3所示。细菌聚类分析显示(图 3A),聚集分支丰度由高到低依次为:Firmicutes (Bacilli纲中的Bacillus、Oceanobacillus、Lactococcus及Clostriia纲中的Halanaerobium)、Proteobacteria (γ-Proteobacteria纲中的Pseudomonas、Stenotrophomonas、Psychrobacter及Halomonas)和Bacteroidetes (Cytophagia纲中的Salinibacter和Flavobacteria纲中的Myroides);而古菌的高丰度聚类分支集中在Euryarchaeota (Halobacteria纲中的Halonotius、Halorubrum、Haloarcula、Halapricum和Halovenus等) (图 3B),且物种丰度明显低于细菌。此外,茶卡盐湖CSL1/2/3/4四个样品聚类为2簇,其中CSL2/3/4聚为一簇,说明3个样本具有相似性,而CSL1单独为一簇。聚类结果显示CSL2/3/4的细菌丰度分布相似,但CSL1细菌的种类和数量均多于CSL2/3/4。各样品的古菌聚类以CSL1和CSL4聚为一簇,而CSL2和CSL3聚为一簇,表明CSL1与CSL4样本具有相似性,CSL2与CSL3具有相似性,但与细菌分布规律相比,优势度明显,组间差异相对较小。
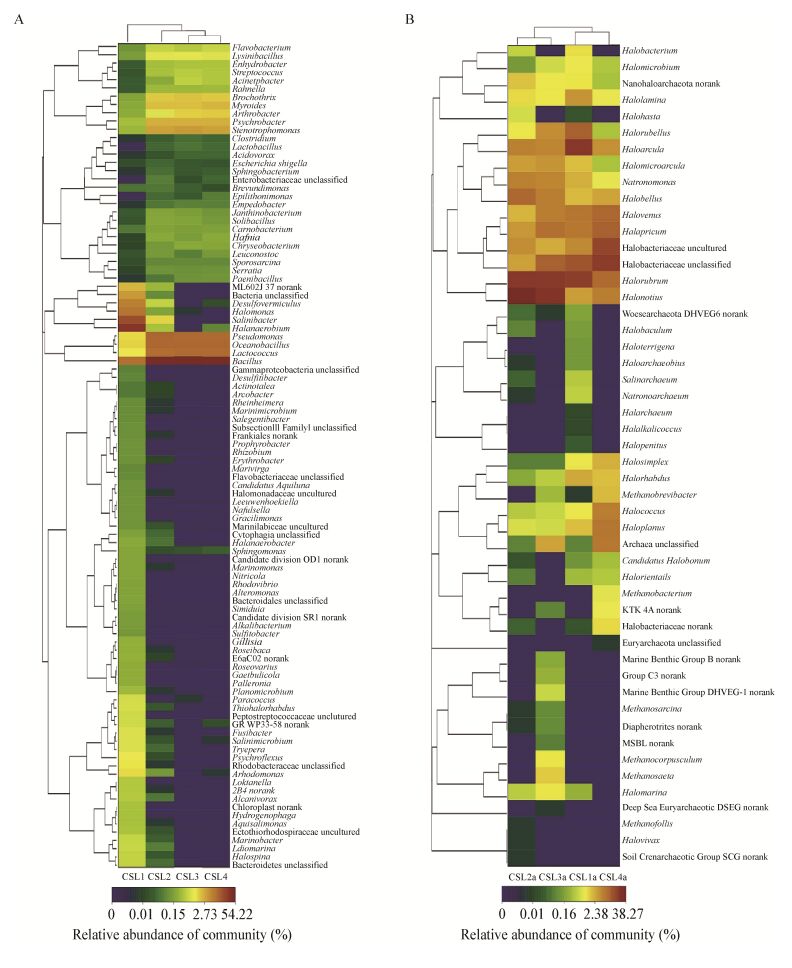
|
| 图 3 茶卡盐湖属分类水平下的多样本热图与相似性分析 Figure 3 Microbial community heatmap analysis and multiple samples similarity tree at the genus level 注:A:细菌属分类水平;B:古菌属分类水平. Note: A: Bacterial community at the genus level; B: Archaeal community at the genus level. |
|
|
采用离子色谱法分析4个样本的离子特征参数(表 2),表明茶卡盐湖中心区域总盐度为315.81-322.40 g/L,pH 7.20-7.81,属于极端盐湖。依据离子浓度特征常数结合盐湖水化学类型标准,茶卡盐湖以Na+、Mg2+、Cl-、SO42-为主要离子,属于硫酸镁亚型盐湖。就水体的有机碳和有机氮而言,盐湖边缘区样本CSL1的营养度要高于盐湖中心区域CSL2/3/4,而pH以及各离子浓度相对偏低。
| 样本 Sample | 矿化度 Total salinity (g/L) | 有机碳 TOC (%) | 有机氮 TN (%) | 离子特征参数Ion characteristic parameters (g/L) | |||||||
| pH | Na+ | K+ | Mg2+ | Ca2+ | Cl- | SO42- | CO32- | ||||
| CSL1 | 210.12 | 8.2 | 1.23 | 7.20 | 81.75 | 2.62 | 7.89 | 0.32 | 107.34 | 10.24 | 0.15 |
| CSL2 | 322.45 | 4.5 | 0.28 | 7.72 | 106.39 | 3.25 | 12.41 | 0.42 | 183.46 | 21.58 | 0.25 |
| CSL3 | 315.81 | 4.6 | 0.25 | 7.81 | 105.23 | 3.47 | 12.51 | 0.42 | 187.71 | 23.63 | 0.20 |
| CSL4 | 322.17 | 4.4 | 0.30 | 7.75 | 107.46 | 3.19 | 12.24 | 0.49 | 187.63 | 20.10 | 0.22 |
基于水质离子特征测定数据和Species-sample丰度数据,采用多元直接梯度回归分析采样点、菌群与环境因子之间关系,并构建典范对应CCA分析(图 4)。结果表明:样本CSL1与CSL2/3/4的总有机碳源(Total organic carbon,TOC)和总有机氮(Total nitrogen,TN)的属性值因采样点差异而有所不同,但CSL2/3/4组与总盐度(Total salinity,TS)、Na+/Mg2+浓度和pH的属性值相似,对样本的影响程度相当。茶卡盐湖CSL1样本中细菌优势菌属Desulfovermiculus、Halomonas、Halanaerobium、Paracoccus及Arhodomonas属群与TOC和TN因子明显呈正相关,而CSL2/3/4样本中的Acinetobacter、Arthrobacter、Bacillus、Chryseobacteriu、Enhydrobacter、Stenotrophomonas及Flavobacterium等细菌属群与TS、Ca2+、Na+/Mg2+浓度和pH等环境因子呈明显正相关,尤其是总盐度的制约影响。

|
图 4 茶卡盐湖微生物信息CCA分析
Figure 4 Canonical correspondence analysis of species information in the Chaka Salt Lake
注: :各样本组;▼:细菌物种; :各样本组;▼:细菌物种; :古菌物种;箭头:环境因子.
Note: Round dots ( :古菌物种;箭头:环境因子.
Note: Round dots ( ): different samples; Upper triangles (▼): bacteria species; Down triangles ( ): different samples; Upper triangles (▼): bacteria species; Down triangles ( ): archeae species; Arrows: environmental factors. ): archeae species; Arrows: environmental factors.
|
|
|
通过分析古菌与环境因子的制约关系,发现CSL1样本中的Haloarcula、Halobacterium、Halolamina、Halomicrobium、Halobaculum和Halorubellus属群与TOC和TN因子明显呈正相关;CSL2中的Methanosaeta、Natronomonas、Halomicroarcula和Halomarina等属群与K+/SO42-、Mg2+浓度等环境因子呈正相关;CSL3/4中的Methanobrevibacter、Halococcus、Haloplanus、Halapricum、Halosimplex、Halorientails及Halovenus属群与TS和pH因子呈正相关,且总盐度TS为主要制约因子,由此表明盐湖古菌的属群分布受到诸多因素的综合制约影响,且针对不同类群,其制约因素有所差异。
3 讨论 3.1 茶卡盐湖微生物优势类群大量研究发现,盐湖细菌类群多以Proteobacteria、Bacteroidetes、Firmicutes、酸杆菌门Acidobacteria和Actinobacteria等为主[11-12],但因各个盐湖水质化学特征差异,在群落结构和优势类群比例方面均有不同。张彦茹[13]在研究内蒙古桑根达来盐碱湖(pH 9.39,盐度为69.7 g/L)、达各淖盐湖(pH 9.20,盐度为100.0 g/L)、扎格斯台淖盐湖(pH 9.05,盐度为13.5 g/L)中的细菌群落时发现这些盐湖中主要以Proteobacteria、Actinobacteria为主,而Actinobacteria只有在富含钾盐及pH偏碱性的盐湖中方可被检出;Zaitseva等[14]研究外贝加尔咸水湖Beloe (pH 9.00-11.00,盐度为308.0 g/L)微生物群落结构结果显示,Proteobacteria、Bacteriodetes和Firmicutes为其主要优势菌门;杨丽等[15]对内蒙古姆州诺尔盐湖(硫酸盐型硫酸钠亚型盐湖,NaCl浓度为63.2 g/L)卤水样品进行研究,发现优势菌门为Proteobacteria和Bacteriodetes;时玉等[16]对青藏高原盐水湖阿翁错湖(盐度为87.1 g/L)的研究显示,Proteobacteria、Actinobacteria和Firmicutes为主要分布类群,而Cyanobacteria和栖热菌门Deinococcus-Thermus为其特有菌门。本研究中茶卡盐湖总盐度为322.4 g/L,pH 7.80,水体沉淀盐晶和盐华。细菌类群多集中于Firmicutes (68.37%)、Proteobacteria (20.49%)、Bacteroidetes (8.46%)和Actinobacteria (1.25%),与上述同类高盐环境相比,优势类群基本一致,仅丰度比例各有不同,反映出不同地区盐环境对微生物群落组成的制约影响。此外,本研究中也检出少量Actinobacteria,可能与盐湖的微碱性相关。
湖泊中的大多数古菌都属于Euryarchaeota和泉古菌门Crenarchaeota,但是在目或属的水平上,不同湖泊中的种类是不一致的[17]。如朱莉等[18]采用免培养法对新疆顿巴斯他乌盐湖(pH 8.80,盐度为97.7 g/L)沉积物古菌组成进行研究,结果显示古菌类群多集中于Euryarchaeota (92.0%)和Crenarchaeota (8.0%);邵冠军[19]对艾比湖(pH 8.60,盐度为44.2 g/L)的古菌多样性进行研究,发现古菌类群多集中在Euryarchaeota (96.6%),其次为纳古菌门Nanohaloarchaeota (1.1%)。本研究中,茶卡盐湖古菌类群多集中于Euryarchaeota (96.5%),而Crenarchaeota未被检出,且有未确定分类地位的古菌门(Archaeaunclassified,2.16%)和Nanohaloarchaeota (1.13%),与相关研究报道一致。
3.2 茶卡盐湖微生物优势种群我国青海、新疆、内蒙古等西部地区的盐湖细菌优势种群多以Bacillus、Halomonas、喜盐芽孢杆菌属Halobacillus和硝化球菌属Nitrococcus等为主[15, 20-21]。因不同盐湖的形成过程、盐分组成及气候特征差异,致使优势种群组成存在差异。内蒙古姆州诺尔盐湖[15](pH 7.00,盐度为63.16 g/L)优势菌属有Nitrococcus (8.29%)、Halomonas (2.64%)和海杆菌属Marinobacter (1.86%);新疆罗布泊盐湖[22](pH 7.50-10.00,盐度为229.78-689.03 g/L)的优势菌属依次为Halomonas (30.55%)、色盐杆菌属Chromohalobacter (20.37%)和Halobacillus (18.51%)等。本研究获得茶卡盐湖(pH 7.8,盐度为322.4 g/L)的细菌共计170个属,与内蒙古姆州诺尔盐湖、新疆罗布泊盐湖相比较,优势类群明显差异,以Bacillus(41.94%)为主,其次为Oceanobacillus (8.03%)。Bacillus作为茶卡盐湖细菌的第一优势类群,属于革兰阳性菌,好氧或兼性厌氧,具有能够产生对不利条件具有特殊抵抗力的芽孢等特点。Sylvan等[23]、Hong等[24]、Lei等[25]、Bagheri等[26]和Chen等[27]曾在高盐(碱)环境中筛选获得大量的Bacillus新种,由此说明Bacillus对高盐环境具有适应生存能力。Oceanobacillus作为第二大优势类群,属海洋微生物类群范畴,大多数在海洋环境中被检出,如Kim等[28]研究韩国东海微生物时检出Oceanobacillus profundus的新种;郁聪[29]采用多相分类法对南太平洋环流区表层、底层海水及地层沉积物分离出的菌株进行研究,发现有5株Oceanobacillus的新种。我国盐湖研究中关于Oceanobacillus的报道较少(如青海湖[3]),这类微生物在嗜盐或嗜盐碱生理生化、代谢功能和遗传基因表达等方面发生改变,从而适应盐(碱)环境得以生存,可能与青藏高原盐湖地质的历史演变有关[3]。
嗜盐古菌是极端环境中的微生物常见类群之一,目前已报道的盐湖古菌优势种群有Halorubrum、Haloterrigena、Natrinema、Haloarcula和Halonotius等[21, 30]。崔恒林等[30]采用富集培养和菌种分离的方法研究新疆艾比湖(pH 8.09,盐度为112.4 g/L)和艾丁湖(pH 7.49,盐度为336.47 g/L)的嗜盐古菌多样性,结果表明Natrinema(39.3%)、Haloterrigena (25%)和Halorubrum (19.6%)在两湖中均有分布且属于优势菌属。Xu等[31]在研究新疆阿牙克库木盐湖(盐度为157.4 g/L)可培养嗜盐古细菌时,结果显示优势菌属是Halorubrum和Natrinema,丰度比例分别为47%和24%。王璐[32]采用非培养技术对连云港地区盐水环境中古菌多样性进行研究,结果表明Halorubrum (53.56%)、Halonotius (22.22%)为优势菌属。本研究中,茶卡盐湖古菌的优势菌属依次为Halonotius(17.21%)、Halorubrum (16.23%)、Haloarcula (8.34%)、Halapricum (6.37%)、Halovenus、Halobellus和Halorubellus,均属于盐杆菌科,对比分析表明不同盐湖因环境化学特征各异,致使优势种群的群落分布和丰度比例有所差异。
3.3 茶卡盐湖微生物多样性在极端(如高盐、强碱或盐碱等)环境下,微生物多样性呈现偏低态势[16]。在盐度接近饱和的极端盐湖中,古菌是主要的嗜盐微生物菌群,而细菌只占很少一部分[33]。本研究利用高通量测序平台全面检测获得茶卡盐湖中心区域样本细菌OTU为86-170 (覆盖度99.57%-99.93%),古菌OTU为121-197 (覆盖度99.70%-99.93%)。多样性分析数据表明单个样本中古菌OTU丰富度、可观察物种和分布均匀度均明显高于细菌,生物种群信息量呈增大趋势,与相关研究报道相一致[4]。对比新疆赛里木咸水湖(盐度为27.14 g/L)和柴窝堡咸水湖(盐度为43.84 g/L)[34]、内蒙古桑根达来盐碱湖(盐度为69.70 g/L)、扎格斯台淖盐湖(盐度为13.50 g/L)和达各淖盐湖(盐度为100.00 g/L)[13]、陕西花马池(盐度为108.50 g/L)和苟池(盐度为156.77 g/L)[2]及柴达木盆地达布逊盐湖(盐度为371.05 g/L)[5],发现随着盐度的增加,细菌OTU数量呈减少趋势,而古菌则相反。此外,我们还发现采样点的水环境盐浓度的变化影响微生物多样性的差异,如在盐浓度较低的盐湖边缘区域,可能因为淡水的补给流入,周边土壤微生物的侵入以及牧区牛羊或生产生活造成碳氮源营养物质的汇集,致使微生物的种类结构趋于复杂,多样性(Shannon 3.84-4.01) 及丰度也趋高。
3.4 茶卡盐湖环境因子影响通常,盐湖微生物群落结构及多样性受到季节、温度、盐度、有机物比例以及水体富营养状况等多种因素的制约影响,其中盐度或矿化度是决定细菌群落结构的主要因素。盐湖细菌多样性可随湖泊盐度的变化而变化,而古菌群落结构则受多种环境因素的综合影响[17, 35]。李璐等[35]研究内蒙古巴丹吉林沙漠盐湖(pH 10.00以上,盐度为165-397 g/L)细菌群落和环境因子的相关性,发现总盐度与菌群结构呈最大正相关,相关系数高达0.98。Wu等[36]对青藏高原16个不同的盐浓度湖泊进行研究,发现盐度是影响湖泊中细菌群落组成和丰度的主要环境因子,Proteobacteria中不同纲分类细菌的相对丰度随含盐量的变化而改变。
本研究中,细菌的分布与环境因子之间的关系比较明显,TS是CSL2/3/4饱和盐度样本细菌群落结构差异的主要因素,对于Na+/Cl-、K+、SO42-、Mg2+和pH等单因素而言,细菌Acinetobacter、Arthrobacter、Bacillus、Chryseobacteriu、Enhydrobacter、Stenotrophomonas和Flavobacterium类群的制约影响程度基本一致。环境中TN和TOC是中盐度样本CSL1的细菌群落差异制约因素,尤以Desulfovermiculus、Halomonas、Halanaerobium、Paracoccus和Arhodomonas属群的分布差异明显。此外,古菌群落因不同样本的物化参数差异而呈现明显的分布差异,如CSL1中的Haloarcula、Halobacterium、Halolamina、Halomicrobium、Halobaculum与Halorubellus属群受制于TN和TOC;CSL2中的Natronomonas、Halomicroarcula、Halomarina及甲烷微菌纲中的Methanosaeta属与K+/SO42-、Mg2+等环境因子有关;CSL3/4中Halobacteriaceae种属(Halococcus、Haloplanus、Halapricum、Halosimplex和Halorientails等)的分布受制于TS的影响。
3.5 盐湖微生物多样性研究目前,分子生物学手段如基于分子杂交技术的荧光原位杂交(Fluorescence in situ hybridization,FISH),基于PCR技术的RFLP (Restriction fragment length polymorphism)与T-RFLP、DGGE与TGGE (Temperature gradient gel electrophoresis)以及16S rRNA基因保守区高通量测序等技术已广泛应用于湖泊生态系统中微生物多样性的研究[37]。其中16S rRNA基因高通量测序技术体系成熟、成本低廉,被广泛应用于揭示盐湖中微生物群落结构及多样性[38],但很少有研究者能考虑到细菌和古菌16S rRNA基因内不同可变区的基因差异性对测序结果的影响[39]。有文献报道[40]:基因差异性可能导致基于16S rRNA基因检测微生物多样性的方法存在类似OTU数目偏高等问题,从而使可靠性受到质疑。为了更全面、更深入地研究这一问题,Hong等[41]和Lee等[42]研究发现,当在97%水平下定义OTU时,测序区选择细菌V4-V5区产生的误差值最小(3%),V3区(5.2%)次之,而古菌的不同可变区差异不大,此为测序区域的选择提供了可信性参考依据。此外,研究还发现Firmicutes和Proteobacteria类群受基因差异性影响较大,而古菌Crenarchaeota类群几乎不受影响,这可能与它们的16S rRNA基因区域的结构不同有关[40]。因此,针对微生物16S rRNA基因结构的复杂性,需运用多种分子生物学手段相结合的方法研究微生物生态系统、种群群落及多样性,这势必会成为未来的研究趋势。
综上所述,茶卡盐湖生境中的细菌多以Firmicutes、Proteobacteria和Bacteroidetes为主,而古菌类群多集中于Euryarchaeota (96.5%),但仍存在大量未知的微生物,其中包括未指定分类、未确定门或科属分类地位的微生物,尚需深入研究。总体而言,盐湖中心区域的古菌群落种类多样性、OTU丰富度和物种总数均显著高于细菌,不同属类群的群落分布受到盐湖总盐度、有机氮与有机碳以及各种离子浓度的综合制约影响,其中总盐度是细菌群落组成和丰度制约的主要因素。借助高通量测序技术,我们初步研究青藏高原茶卡盐湖的细菌和古菌多样性,为微生物资源的挖掘和开发利用提供了一定的参考依据。
| [1] |
Zheng MP. On saline lakes of China[J]. Mineral Deposits, 2001, 20(2): 181-189. (in Chinese) 郑绵平. 论中国盐湖[J]. 矿床地质, 2001, 20(2): 181-189. |
| [2] |
Cao LF, Yin TT, Yuan ZY, et al. Analysis of the microbial community composition of Huama Lake and Gou Lake in Shannxi Province based on high-throughput sequencing[J]. Letters in Biotechnology, 2016, 27(3): 374-380. (in Chinese) 曹露芬, 殷婷婷, 袁振亚, 等. 基于高通量测序的陕西花马池和苟池微生物群落多样性分析[J]. 生物技术通讯, 2016, 27(3): 374-380. |
| [3] |
Zhu DR, Liu J, Han R, et al. Population diversity and phylogeny of halophiles in the Qinghai Lake[J]. Biodiversity Science, 2012, 20(4): 495-504. (in Chinese) 朱德锐, 刘建, 韩睿, 等. 青海湖嗜盐微生物系统发育与种群多样性[J]. 生物多样性, 2012, 20(4): 495-504. |
| [4] |
Chai LH, Cui XL, Peng Q, et al. Bacterial diversity of two salt lakes in Qinghai[J]. Acta Microbiologica Sinica, 2004, 44(3): 271-275. (in Chinese) 柴丽红, 崔晓龙, 彭谦, 等. 青海两盐湖细菌多样性研究[J]. 微生物学报, 2004, 44(3): 271-275. |
| [5] |
Zhao WY, Yang J, Dong HL, et al. Microbial diversity in the hypersaline Dabuxun Lake in Qaidam Basin, China[J]. Earth and Environment, 2013, 41(4): 398-405. (in Chinese) 赵婉雨, 杨渐, 董海良, 等. 柴达木盆地达布逊盐湖微生物多样性研究[J]. 地球与环境, 2013, 41(4): 398-405. |
| [6] |
Wu LY, Wen CQ, Qin YJ, et al. Phasing amplicon sequencing on Illumina MiSeq for robust environmental microbial community analysis[J]. BMC Microbiology, 2015, 15(1): 125. DOI:10.1186/s12866-015-0450-4 |
| [7] |
You J, Wu G, Ren FP, et al. Microbial community dynamics in Baolige oilfield during MEOR treatment, revealed by Illumina MiSeq sequencing[J]. Applied Microbiology and Biotechnology, 2016, 100(3): 1469-1478. DOI:10.1007/s00253-015-7073-4 |
| [8] |
Bates ST, Clemente JC, Flores GE, et al. Global biogeography of highly diverse protistan communities in soil[J]. The ISME Journal, 2012, 7(3): 652-659. |
| [9] |
Jami E, Israel A, Kotser A, et al. Exploring the bovine rumen bacterial community from birth to adulthood[J]. The ISME Journal, 2013, 7(6): 1069-1079. DOI:10.1038/ismej.2013.2 |
| [10] |
Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41(D1): 590-596. DOI:10.1093/nar/gks1219 |
| [11] |
Ventosa A, Fernández AB, León MJ, et al. The Santa Pola saltern as a model for studying the microbiota of hypersaline environments[J]. Extremophiles, 2014, 18(5): 811-824. DOI:10.1007/s00792-014-0681-6 |
| [12] |
Ghai R, Pašić L, Fernández AB, et al. New abundant microbial groups in aquatic hypersaline environments[J]. Scientific Reports, 2011, 1: 135. DOI:10.1038/srep00135 |
| [13] |
Zhang YR. Bacterial diversity of Haloalkaline Lakes in desert Hunshandake in Inner Mongolia[D]. Hohhot: Master's Thesis of Inner Mongolia Agricultural University, 2011(in Chinese). 张彦茹. 内蒙古浑善达克盐碱湖细菌多样性分析[D]. 呼和浩特: 内蒙古农业大学硕士学位论文, 2011 http://d.wanfangdata.com.cn/Thesis/D341287 |
| [14] |
Zaitseva SV, Abidueva EY, Namsaraev BB, et al. Microbial community of the bottom sediments of the Brackish Lake Beloe (Transbaikal Region)[J]. Microbiology, 2014, 83(6): 861-868. DOI:10.1134/S0026261714060216 |
| [15] |
Yang L, Yin TT, Yin X, et al. Analysis on the bacterial community composition of Muzhou Nuoer Salt Lake in Inner Mongolia based on high-throughput sequencing[J]. Sichuan Environment, 2015, 34(6): 34-38. (in Chinese) 杨丽, 殷婷婷, 尹馨, 等. 基于高通量测序的内蒙古姆州诺尔盐湖细菌群落组成分析[J]. 四川环境, 2015, 34(6): 34-38. |
| [16] |
Shi Y, Sun HB, Liu YQ, et al. Vertical distribution of bacterial community in sediments of freshwater lake Puma Yumco and saline lake AWongco on the Tibetan Plateau[J]. Microbiology China, 2014, 41(11): 2379-2387. (in Chinese) 时玉, 孙怀博, 刘勇勤, 等. 青藏高原淡水湖普莫雍错和盐水湖阿翁错湖底沉积物中细菌群落的垂直分布[J]. 微生物学通报, 2014, 41(11): 2379-2387. |
| [17] |
Gong SJ, Wu L, Li SG. Studying on lake microbial diversity[J]. Biotechnology Bulletin, 2008(4): 54-57. (in Chinese) 龚世杰, 吴兰, 李思光. 湖泊微生物多样性研究进展[J]. 生物技术通报, 2008(4): 54-57. |
| [18] |
Zhu L, Yang HM, Wang Y, et al. Archaeal diversity in sediment of Dunbasitawu salt lake Xinjiang, China, estimated by 16S rRNA gene sequence analyses[J]. Acta Microbiologica Sinica, 2012, 52(6): 769-775. (in Chinese) 朱莉, 杨红梅, 王芸, 等. 新疆顿巴斯他乌盐湖沉积物免培养古菌多样性[J]. 微生物学报, 2012, 52(6): 769-775. |
| [19] |
Shao GJ. Study on Biodiversity of Actinomycetes and Arch Aea in the Sediments of Ebinur Lake[D]. Urumqi: Master's Thesis of Xinjiang University, 2015(in Chinese). 邵冠军. 艾比湖湖底沉积物中放线菌和古菌菌群多样性研究[D]. 乌鲁木齐: 新疆大学硕士学位论文, 2015 http://cdmd.cnki.com.cn/Article/CDMD-10755-1015800092.htm |
| [20] |
Li M, Guo J, Shi ZG, et al. Bacterial community structure in lakes on the Northeastern Qinghai-Tibetan Plateau[J]. Chinese Journal of Applied and Environmental Biology, 2013, 19(5): 750-758. (in Chinese) 李明, 郭嘉, 石正国, 等. 春季青藏高原东北部湖泊细菌种类组成[J]. 应用与环境生物学报, 2013, 19(5): 750-758. |
| [21] |
Xu XW, Wu M, Dilber T, et al. Halophilic archaea diversity of Aibi Lake and Yiwu Lake in Xinjiang[J]. Biodiversity Science, 2006, 14(4): 359-362. (in Chinese) 许学伟, 吴敏, 迪丽拜尔·托乎提, 等. 新疆艾比湖和伊吾湖可培养嗜盐古菌多样性[J]. 生物多样性, 2006, 14(4): 359-362. |
| [22] |
Luo M, Han J, Jiang PA, et al. Diversity of culturable halophilic bacteria isolated from Lop Nur region in Xinjiang[J]. Biodiversity Science, 2009, 17(3): 288-295. (in Chinese) 罗明, 韩剑, 蒋平安, 等. 新疆罗布泊地区可培养嗜盐细菌多样性[J]. 生物多样性, 2009, 17(3): 288-295. |
| [23] |
Sylvan JB, Hoffman CL, Momper LM, et al. Bacillus rigiliprofundi sp. nov., an endospore-forming, Mn-oxidizing, moderately halophilic bacterium isolated from deep subseafloor basaltic crust[J]. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(6): 1992-1998. DOI:10.1099/ijs.0.000211 |
| [24] |
Hong SW, Kwon SW, Kim SJ, et al. Bacillus oryzaecorticis sp. nov., a moderately halophilic bacterium isolated from rice husks[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(8): 2786-2791. |
| [25] |
Lei ZC, Qiu P, Ye RY, et al. Bacillus shacheensis sp. nov., a moderately halophilic bacterium isolated from a saline-alkali soil[J]. The Journal of General and Applied Microbiology, 2014, 60(3): 101-105. DOI:10.2323/jgam.60.101 |
| [26] |
Bagheri M, Didari M, Amoozegar MA, et al. Bacillus iranensis sp. nov., a moderate halophile from a hypersaline lake[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(4): 811-816. |
| [27] |
Chen YG, Zhang YQ, He JW, et al. Bacillus hemicentroti sp. nov., a moderate halophile isolated from a sea urchin[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(12): 2950-2955. DOI:10.1099/ijs.0.026732-0 |
| [28] |
Kim YG, Choi DH, Hyun S, et al. Oceanobacillus profundus sp. nov., isolated from a deep-sea sediment core[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(2): 409-413. DOI:10.1099/ijs.0.64375-0 |
| [29] |
Yu C. Taxonomic analysis and related research of Oceanobacillus pacificus sp. nov. and other five novel bacteria from the South Pacific Gyre[D]. Qingdao: Master's Thesis of Ocean University of China, 2014(in Chinese). 郁聪. 太平洋海洋杆菌(Oceanobacillus pacificus sp. nov. )等5株南太平洋新菌的分类鉴定及相关特性分析[D]. 青岛: 中国海洋大学硕士学位论文, 2014 http://d.wanfangdata.com.cn/Thesis/D547828 |
| [30] |
Cui HL, Yang Y, Dilber T, et al. Biodiversity of halophilic archaea isolated from two salt lakes in Xin-jiang rejion of China[J]. Acta Microbiologica Sinica, 2006, 46(2): 171-176. (in Chinese) 崔恒林, 杨勇, 迪丽拜尔·托乎提, 等. 新疆两盐湖可培养嗜盐古菌多样性研究[J]. 微生物学报, 2006, 46(2): 171-176. |
| [31] |
Xu XW, Wu M, Wu YH, et al. Culturable halophilic archaeal diversity of Ayakekumu salt lake located in Xinjiang, China[J]. Acta Ecologica Sinica, 2007, 27(8): 3119-3123. DOI:10.1016/S1872-2032(07)60066-3 |
| [32] |
Wang L. Diversity of halophilic archaea isolated from different hypersaline lakes and salt fields[D]. Xuzhou: Master's Thesis of Jiangsu Normal University, 2014(in Chinese). 王璐. 不同盐湖、盐田之间嗜盐古菌多样性比较[D]. 徐州: 江苏师范大学硕士学位论文, 2014 http://cdmd.cnki.com.cn/Article/CDMD-10320-1015967821.htm |
| [33] |
Pašić L, Bartual SG, Ulrih NP, et al. Diversity of halophilic archaea in the crystallizers of an Adriatic solar saltern[J]. FEMS Microbiology Ecology, 2005, 54(3): 491-498. DOI:10.1016/j.femsec.2005.06.004 |
| [34] |
Deng LJ, Lou K, Zeng J, et al. Archaea diversity in water of two typical brackish lakes in Xinjiang[J]. Acta Ecologica Sinica, 2012, 32(21): 6811-6818. (in Chinese) 邓丽娟, 娄恺, 曾军, 等. 新疆两典型微咸水湖水体免培养古菌多样性[J]. 生态学报, 2012, 32(21): 6811-6818. |
| [35] |
Li L, Hao CB, Wang LH, et al. Microbial diversity of salt lakes in Badain Jaran desert[J]. Acta Microbiologica Sinica, 2015, 55(4): 412-424. (in Chinese) 李璐, 郝春博, 王丽华, 等. 巴丹吉林沙漠盐湖微生物多样性[J]. 微生物学报, 2015, 55(4): 412-424. |
| [36] |
Wu QL, Zwart G, Schauer M, et al. Bacterioplankton community composition along a salinity gradient of sixteen high-mountain lakes located on the Tibetan Plateau, China[J]. Applied and Environmental Microbiology, 2006, 72(8): 5478-5485. DOI:10.1128/AEM.00767-06 |
| [37] |
Li GQ, Xue LG, Mo TL, et al. New progress of research methods for microbial diversity in lake sediment[J]. Journal of Lanzhou Jiaotong University, 2015, 34(6): 12-16. (in Chinese) 李国强, 薛林贵, 莫天录, 等. 湖泊沉积物微生物多样性研究方法的新进展[J]. 兰州交通大学学报, 2015, 34(6): 12-16. |
| [38] |
Wang XC, Yang ZR, Wang M, et al. High-throughput sequencing technology and its application[J]. China Biotechnology, 2012, 32(1): 109-114. (in Chinese) 王兴春, 杨致荣, 王敏, 等. 高通量测序技术及其应用[J]. 中国生物工程杂志, 2012, 32(1): 109-114. |
| [39] |
Case RJ, Boucher Y, Dahll f I, et al. Use of 16S rRNA and rpoB genes as molecular markers for microbial ecology studies[J]. Applied and Environmental Microbiology, 2007, 73(1): 278-288. DOI:10.1128/AEM.01177-06 |
| [40] |
Sun DL, Jiang X, Wu QLL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19): 5962-5969. DOI:10.1128/AEM.01282-13 |
| [41] |
Hong PY, Wheeler E, Cann IKO, et al. Phylogenetic analysis of the fecal microbial community in herbivorous land and marine iguanas of the Galápagos Islands using 16S rRNA-based pyrosequencing[J]. The ISME Journal, 2011, 5(9): 1461-1470. DOI:10.1038/ismej.2011.33 |
| [42] |
Lee OO, Wang Y, Yang JK, et al. Pyrosequencing reveals highly diverse and species-specific microbial communities in sponges from the Red Sea[J]. The ISME Journal, 2011, 5(4): 650-664. DOI:10.1038/ismej.2010.165 |
 2017, Vol. 44
2017, Vol. 44