扩展功能
文章信息
- 马芮, 苏莉, 宋宇昊, 王莹莹
- MA Rui, SU Li, SONG Yu-hao, WANG Ying-ying
- 多聚磷酸盐:菌体内多功能调控子和环境压力守护者
- Inorganic polyphosphate: the multifunctional regulator and the guardian of environmental stresses in bacteria
- 微生物学通报, 2017, 44(7): 1736-1746
- Microbiology China, 2017, 44(7): 1736-1746
- DOI: 10.13344/j.microbiol.china.160880
-
文章历史
- 收稿日期: 2016-12-01
- 接受日期: 2017-02-27
- 优先数字出版日期(www.cnki.net): 2017-02-27
20世纪中叶,科研人员在对细胞的研究中发现一种致密性颗粒,又称为异染颗粒(“Metachromatic” or “volutin” granules),这个研究标志着细胞学与化学方法的结合,随后的研究发现在不利生长条件下细胞和微生物中都会产生这种物质——多聚磷酸盐[1]。多聚磷酸盐(Inorganic polyphosphate,polyP)是由三个到上百个磷酸基团组成的链状无机化合物,由磷酸酐键(同ATP的高能磷酸键)连接。这个大分子聚合物在自然环境中无处不在,在火山口和海洋深处都有它们的印记,细菌、真菌、植物以及哺乳动物细胞中均能检测到[1-2],并且在一些细胞中占有相当大的比重,所以它被看作是生命起源的生物分子,存在于生命之树的所有枝节。polyP是由磷酸酐键连接的高储能物质,被称作古老的能源资源和生物燃料,这些能量在微生物体内用来合成氨基酸、核碱基、糖类、脂肪等必需生命物质[3],它的合成代谢途径也渐渐被人们知晓。20世纪50年代,Kornberg团队开始了对polyP的研究,在大肠杆菌中发现一种酶,可以利用polyP来合成ATP[4]。近来,Albi等发现在固氮蓝藻中有多聚磷酸-葡萄糖磷酸转移酶,它严格地利用polyP上的磷酰基,并直接消耗其中的能量,而不是昂贵的ATP,这为糖激酶的进化起源提供了新的视角[5]。Kornberg团队五十多年的深入研究,发现了polyP的许多生理功能,诸如抵抗饥饿、热击、氧化和紫外暴露等[6]。polyP这个无处不在、无所不能的生物大分子引起了越来越多研究者的青睐。
polyP这种大分子链状聚合物及其相关的酶,对微生物在一些环境压力下的应激反应有着多种重要的调节作用,从代谢水平到基因水平[7-9]。除了作为磷源和能源库之外,polyP由于其高荷负电性以及链状的灵活性,一方面通过电性引力吸附结合细胞内的生命物质,另一方面使信号蛋白发生磷酸化作用从而进行信号传导。因此作为支架、桥梁、通讯工具和开关等,在生命活动中承担了众多任务。此外,由于多聚磷酸盐独特的结构、价格低、无毒害、可生物降解的特性,使其在工业、农业、食品、医学、环境方面发挥着越来越重要的作用[10]。早在20世纪50年代,国外就已经开始了对微生物体内的polyP作用的研究,发现了许多种不同类型的环境压力都可以引起其在菌体内的累积,并发挥着重要的调节作用,但是这一系列具体的调节机理和路径尚不明确,这个科学问题需要进一步大量、深入的研究。本文讲述了菌体内polyP的观察和测量的方法,重点介绍了以polyP为核心的抗环境压力机制,以及在寡营养环境下的调控机理。
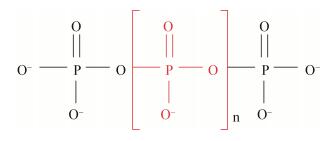
1 菌体内polyP的物理化学性质及定性定量方法 1.1 菌体内polyP的物理化学性质polyP是由磷酸酐键连接的正磷酸基团链状聚合物,是一种高荷负电、高储磷和储能物质。研究者在火山口和深海蒸汽中发现它的踪迹,所以推测它出现在早期地球环境中,是生命起源和进化过程中的关键大分子[11-12],高能和高磷含量使它被认为是DNA、RNA和蛋白质形成的先驱体。polyP的化学结构如图 1所示,是由若干个磷酸基团组成,由磷酸酐键连接的链状无机化合物,其中磷为五价,磷酸基团为四面体结构,通过共有的氧原子相互配位。多聚磷酸盐易溶于水,荷负电,常温下稳定性较高且结构灵活,在一定条件下会水解而释放能量,有着很强的持水性、缓冲能力、螯合能力。polyP在菌体内主要以颗粒的形式存在,polyP颗粒上会结合一些蛋白质、核酸、脂质和糖类等生物大分子物质和金属离子等荷正电无机物,除了储存能量和营养物质外,另一个重要的作用就是修饰其结合物的性能,它几乎出现在所有生命体细胞质、液泡、膜间质等部位,有着很高的生物活性,调控着错综复杂的生理代谢活动。此外,从polyP的结构(图 1)可以发现,它跟遗传物质DNA和RNA有一些共性:同为链状化合物、都含有正磷酸基团、均荷载大量的负电,这意味着它可以参与一些基因层面的调控行为。近年来研究人员发现的一种细胞器叫酸性钙体(Acidocalcisomes),主要含焦磷酸盐(Pyrophosphate,PPi)、多磷酸盐(Polyphosphate,polyP),还含有钙和其他阳离子,它是一种富含磷酸盐的致密颗粒,大量存在于真菌的液泡中[13-14],研究发现它的性质功能和细菌体内的polyP颗粒基本类似[15]。polyP的生物提取和纯化效率较低,但是化学合成工艺很成熟,廉价且纯度高[16],被广泛用于阻燃剂、缓蚀剂、食品保鲜剂、骨骼代替材料和抑菌剂等。polyP作为胞内天然合成的无机物,有着许多重要的生物作用,它们被越来越多的科学研究所发现,但是它的许多生物调控机理尚未完全明朗。如果化学合成的polyP以及其相关的酶可以用于生物学研究,比如新型抗生素的研发、ATP再生系统的创造和一些生物活性物质的合成[10],实现化学化工和生物学横向协作,必然会给食品、卫生以及环境保护带来更多的利益。
|
| 图 1 polyP结构图 Figure 1 The construction of polyp |
|
|
polyP作为异染颗粒,可以被Toluidine blue (甲苯胺蓝)、Neisser (奈瑟尔)、Nile red (尼罗红)、DAPI (4', 6-二脒基-2-苯基吲哚)和Tc (盐酸四环素)等染料染色后定性观察[17-20]。在这些染料中,前三种染色方法都是传统的无荧光方法,目前最常用的是DAPI荧光染色。DAPI (4', 6-Diamidino-2-phenylindole)是一种核酸荧光染料,低浓度(0.24-5 μmol/L)时染DNA,产生蓝色荧光,高浓度(5-50 μmol/L)时发生以polyP为媒介的异染反应,产生黄绿色荧光[21-23],所以推测DAPI跟DNA结合的特异性更强,当DAPI浓度高时,由于polyP与DNA化学结构的相似性,游离的DAPI会跟polyP结合,这两种结合物都有其专一的激发和发射波长,可以将其区分开来。在经过一些简单的前处理之后(在中性的HEPES或PBS缓冲液中进行离心冲洗),用100 mg/L的DAPI溶液避光染色10-30 min,便可通过荧光显微镜、共聚焦显微镜、扫描电镜或流式细胞仪进行观察[20-21, 24],由于polyP在微生物体内是高电子密度颗粒,所以也可以经过固定之后上电镜直接观察[11, 25]。考虑到DNA-DAPI结合物可能产生荧光干扰,新的研究发现盐酸四环素Tc由于其特殊的化学性质,跟polyP有着更强的特异性,可以减弱上述干扰,同时还可以避免金属阳离子的干扰,两者的结合物在365 nm的激发光下发出很强的绿色荧光[21]。此外,荧光原位杂交(FISH)法,比如16S rRNA基因探针或PPBD (Polyphosphate binding domain)探针,可以实现对polyP在超微结构水平上的非常精准的定性和定位观察[19, 26]。
polyP的量化方法最初是经过提取和纯化,然后经强酸或强碱水解为磷酸根,通过检测磷酸根的浓度来进行定量polyP,提取和纯化过程必然会导致一部分损失,使测量结果偏低,而一些非来源于polyP的磷酸基团的干扰,通常会导致测量结果偏高,所以这种方法的精准度较低。同样地,31P标记的核磁共振法(31P NMR)[11, 27-28],特异性更差(菌体内大量存在的核酸、磷脂等干扰),测得的polyP含量会更高地偏离实际。PPX酶水解法特异性强[18],但是由于酶需要生物合成纯化,所以耗时耗力。以DAPI染色技术为基础的荧光分光光度计定量方法,以其简捷、准确的优势逐渐被普遍应用[17, 22-24, 29-30]。使用荧光分光光度计时,要考虑到DAPI是一种核酸染料,DAPI-DNA的结合体也会产生荧光,为了提高对DAPI-polyP结合体荧光监测的专一性,需要调节仪器的激发光(400-430 nm,415 nm为最佳)和发射光(500-560 nm,550 nm为最佳)波长[17, 23, 29-31]。在此基础上,是否提取、是否纯化这些前处理步骤引起讨论,因为无论是提取还是纯化都会造成polyP的损失[17, 23, 29]。Kulakova等研究者实验证实了一些简单的前处理方法,比如加热、煮沸、冷冻、液氮速冻、超声等方法迫使菌体破裂以及在颗粒上结合的polyP释放,进而染色检测[17],对比得出液氮冻融这个处理方法最佳,但是经过这个简单的处理之后细胞内的其他物质可能对polyP的测量产生干扰。Martin等进一步排除结合着的蛋白质以及DNA(RNA)-DAPI的干扰,在进行了超声、煮沸、再超声的破菌过程后,增加了蛋白酶K、DNA酶、RNA酶的酶解过程[30],需要注意的是,煮沸会影响测量结果的准确性,因为酸性和高温会破坏polyP的结构[10]。研究者可以根据样品的特点和需求选择和优化出合适的测量方法,主要宗旨就是在不损害polyP的前提下(如使用pH 7.0的缓冲液,尽可能地避光和避免高温),完成破菌和polyP从结合状态的释放这两个过程,使其充分地与DAPI结合,从而实现快捷精准的量化。
2 polyP及其相关酶对菌体抵抗环境压力的调节在菌体内,参与polyP代谢的两类酶一类为多聚磷酸盐激酶PPK (Polyphosphate kinase),它是polyP合成酶,分为PPK1和PPK2,PPK1利用ATP末端的磷酸基团来合成polyP链:polyPn–1+nATP→polyPn+nADP,PPK2催化GDP合成GTP,这两个反应均为可逆反应[32]。另一类酶为polyP水解酶,外切聚磷酸酶PPX (Exopolyphosphatase)释放polyP链末端的磷酸基团:polyP n→polyPn–1+Pi→nPi[33-34],同时释放出能量,且具有核苷三磷酸酶NTPase的活性[35];内切聚磷酸酶PPN (Endopolyphosphatase),它的功能为裂解长链polyP为短链[32];鸟苷五磷酸水解酶pppGpp,它能协同菌体内许多基因的表达,是应激反应的警报器[36-37]。这两类酶控制着菌体内polyP的积累和消耗,同时也控制着许多催化反应,polyP的研究者从编码这两类酶的基因入手,敲除或沉默掉这两类基因,或者构建含有这两类基因的重组质粒使之过表达,以野生型为对照,通过对比表型变化来分析polyP及其酶在菌体内的调控作用[12, 33, 38-43]。
polyP和微生物体内的许多生理活动都有关系,比如运动性、生物膜的形成、群集反应等[33, 41-42],ppk和ppx基因敲除之后,菌体内polyP的含量都有所减少,ppk的突变体相对于野生型减少的尤为明显,并且严重影响了生物膜的形成、运动能力、微生物体的群集反应以及抗毒能力。这些丧失polyP的菌体都表现出了对环境压力敏感和脆弱,比如紫外照射、强氧化、缺氧、抗生素污染、高温、干燥等[11, 38-39, 44-45],在这些极端的环境下,polyP发挥了重要的作用。这些抗性看似独立,其实是相互联系的,细菌在不适宜的生存条件下会调动以polyP为核心的几种自我保护机制:(1) 形成生物膜[46]。生物膜是一个个细菌通过聚集,表面接触所形成的,聚集的过程细菌需要相互交流,传递信号(群集效应),这个信号可能就是关键的环境刺激因素,或群集运动逃逸危害,或形成生物膜。生物膜是菌体的一个保护隔离层,具有很高的耐受性,除了抗紫外照射、高温和干燥之外,对抗生素等杀菌剂以及宿主免疫防御机制有很强的抗性,生物膜被证实可以保护细菌体免遭单过硫酸钾、氯化铵、过氧化氢和戊二醛的危害,C. perfringens生物膜保护机制普遍应用于农场和食品加工环境[47]。究其原因,是由于polyP磷酸基团上连接的氢键有极强的亲水性,导致polyP结合大量的水,持水性很高,有助于悬浮和凝胶[10],同时,菌体产生的polyP可以和碳酸盐结合形成核壳结构的颗粒[48],进一步促进了生物膜的形成。(2) polyP作为蛋白伴侣,强氧化、高温、抗生素等压力会导致相应蛋白的伸展或聚合而产生对细菌的损害作用[49],polyP有类蛋白伴侣的特性,它和核蛋白的赖氨酸残基共价结合,通过非酶促的机理发生后翻译的多磷酸化作用Polyphosphorylation,它和其他的蛋白修饰作用(磷酸化和焦磷酸化)非常相似[50],从而阻止蛋白变性;凭借其灵活的链状大分子结构,以不依赖ATP的方式高亲和度地固定蛋白质的构造,从而抵抗蛋白毒性压力[51]。(3) 在毒素-抗毒素系统(Toxin antitoxin system,TAS)中,polyP大量积累,它不需要转录激活yefM/yoeB TAS,而是调节Lon蛋白酶降解抗毒素,抗毒素浓度的降低激活了毒素的增加[52],形成细菌的自我保护机制。
polyP作为负聚合电解质,可以大量结合H+和金属离子,所以担任了pH缓冲剂、渗透压平衡剂、金属离子的螯合剂的角色[11, 40, 46, 53-54],同时参与了跨膜运输,在化工工业、环境工程和医疗卫生上有不可替代的作用。Pavlov等发现,从细菌到真菌的细胞膜上都存在一个PHB/Ga2+/polyP的复合体,它在细胞膜的磷脂双分子层上形成一个通道,可以控制其电导率,对阳离子有选择透过性,控制体外遗传物质的进入[55],也能抵挡一些有毒物质进入细胞,起到了防御作用。polyP水解释放磷的过程中,磷的排出会增加跨膜质子梯度[10],有利于物质顺着电化学梯度进行跨膜转运。新的研究还发现Streptomyces lividans的PPX具有磷脂酶D (PLD)的活性[56],后者不仅是一类重要的磷脂水解酶,也是一类重要的跨膜信号传导酶。polyP和Mn2+螯合形成Mn2+-polyP复合体,进而被PPX酶水解成MnHPO4,实现对重金属Mn的解毒作用,与此同时,MnHPO4可以有效地抵抗O2-离子的氧化作用[50]。Gloeomargarita lithophora这株蓝藻在不影响自身生长速率的前提下积累碱土金属Sr和Ba,对这类元素的地球化学循环有着重要的意义,这得益于菌体内的polyP和碳酸盐颗粒,两者形成核壳结构[48],Sr和Ba固定于其中从而免于给细菌带来伤害。污水除磷系统中的高效聚磷菌,(Polyphosphate accumulating organisms,PAOs),它们可以大量积累polyP而除磷,同时菌体利用polyP的PO43--P进一步缺氧去除NOx,实现脱氮过程[57]。而且在酸性条件下,PAOs的聚磷活性更高,群落的宏基因组学分析发现了丰富的ppk、ppx基因[58],这是由于polyP在环境压力下会大量积累,所以推测在其他一些复杂水质的污水如高盐和重金属污染等,PAOs也会有同样的特性,从而给处理这些较难处理的污水带来便捷。总之,链状的灵活性以及拥有许多负电性引力结合位点这两个绝妙的优点,使得polyP在感应了外界不利于生存的环境变化之后大量积累,并和其它因子(如pppGpp等)联合反应,非常活跃地参与了诸多调节活动(表 1)。
| 菌种 Bacterial strain |
表型观察 Phenotypic observation |
文献 Reference |
| Pseudomonas aeruginosa | 在寡磷环境中不能生长丧失生物膜形成、运动和群集反应能力β-半乳糖活性降低、绿脓菌素等合成减少 | [42] |
| Lactobacillus spp. | 抵抗高盐、低pH、氧化压力的能力减小 | [41] |
| Pseudomonas putida | 对紫外照射、热击、抗生素、重金属变得敏感细胞膜形成和运动能力降低延迟稳定期的成活率降低 | [33] |
| Escherichia coli | 抗渗透性和酸性压力的能力降低 | [40] |
| 核糖体蛋白的降解速率翻了3倍 | [59] | |
| Streptomyces coeli | 放线菌紫素的合成量增加只能在富营养中生长且不能形成孢子对H2O2的毒效应变得敏感 | [38] |
| Campylobacter jejuni | 入侵宿主细胞和胞内存活能力减弱 | [60] |
| Mycobacterium smegmatis | 在氧化应激、表面压力(清洁剂)下存活率降低在缺氧条件下生存能力骤降 | [61] |
| Salmonella typhimurium | 对H2O2的氧化应激反应基本丧失 | [45] |
| Yeast | 失去了对胞内ATP和磷的平衡调节能力 | [43] |
| Rhodococcus erythropolis N9T-4 | 胞内海藻糖合成增加、糖原合成减少 | [62] |
| Helicobacter pylori | 饥饿环境下细菌过早死亡 | [63] |
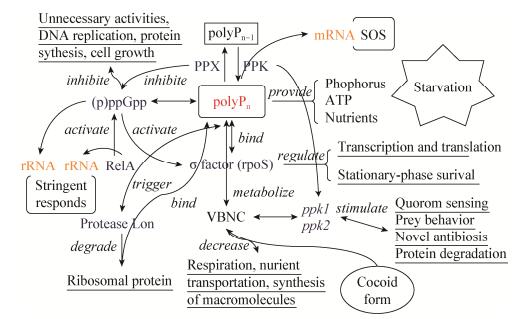
研究表明,polyP参与了细菌在延迟生长期(营养物质匮乏的状态下保持休眠但仍然是活的这种生理状态)的耐受性和抗性活动[33, 64]。首先,单从polyP自身来看,它可以为细菌提供P源和能量,polyP颗粒上结合的蛋白质和糖类等也可提供少量的营养物质。Albi研究发现,原核生物可以在光合作用中利用polyP合成葡萄糖-6-磷酸,并高效地从polyP中获得能量来代替昂贵的ATP[5]。polyP参与抗寡营养环境的过程是十分复杂的,需要相关的酶以及一些其他生物因子参与(图 2)。RelA介导的应激反应是细菌适应饥饿压力的关键,RelA整合几种环境诱因,可以快速地转变激活和灭活状态,使其对新的环境诱因感应变得敏感,并且合成信号素(p)ppGpp,它可以综合地重调转录、翻译和复制程序[65]。氨基酸浓度贫乏会唤起应激反应的主导信号因子(p)ppGpp酶,(p)ppGpp酶通过抑制PPX酶的活性来提高polyP的积累,还会抑制蛋白质的合成和DNA的复制进而抑制细胞的生长,控制细菌的生长率,它通过缩减非生长细胞的不必要的活性来调控基因的表达。除此之外,(p)ppGpp还会刺激RpoS在稳定期的积累[36],RpoS为稳定期调控子,许多稳定生长期中的基因都依赖于RpoS蛋白的表达,如氨基酸的运输和代谢基因、蛋白合成基因、糖代谢基因、核苷代谢基因以及铁的吸收和储存基因等[66],很大程度地调控着饥饿压力应答。非常类似的一个过程,在氮磷元素缺乏时,ppx的表达受代替σ因子(RpoS)控制[42],polyP积累并在饥饿压力应答这一复杂的过程中发挥作用,促使了稳定期σ因子(RpoS)的最佳表达[33, 67]。Yang等在研究人体幽门螺杆菌时发现,饥饿时期polyP的积累可以通过结合σ因子直接作用于转录过程,σ因子N端富含赖氨酸的区域荷正电,是和polyP的结合位点,因此,polyP被认为是幽门螺旋杆菌在饥饿压力下调节基因表达的信使[63]。还有一种饥饿压力存活机制是,polyP可以结合Lon蛋白酶(负责把损坏的蛋白分解清除掉)分解核糖体蛋白来供给氨基酸[59, 68]。
|
| 图 2 饥饿压力应答 Figure 2 Starvation stress response |
|
|
综上所述,当细菌处于饥饿状态或氨基酸缺乏时,以polyP为核心的信号因子便活跃起来,联合抵抗饥饿压力(图 2)。我们推测polyP在营养物质极度缺乏时的三种作用机制:(1) 直接提供能源和其身缠绕的营养物质;(2) 作为信号因子,和一些特定的酶、基因、RNA相互作用(图 2)完成复杂的调节过程以提高细菌的耐受性。例如在蛋白的翻译后修饰过程中,polyP的磷酸基团和目标蛋白的赖氨酸残基共价结合,完成多聚磷酸化作用[69],从而控制蛋白质的活性变化(比如使酶活性化或钝化),实现信号传导,调动和参与饥饿压力应答。此外,研究发现许多蛋白的亚结构组成中都还有荷正电的半通道,被推测为polyP结合蛋白的区域[70];(3) 改变了能量和营养物的代谢通路以适应低营养环境,如Yano等的研究发现,在极端寡营养环境下Rhodococcus erythropolis N9T-4体内的polyP合成和海藻糖、糖原的代谢途径有关,该菌敲除了ppx基因后,体内海藻糖的累积增加,而糖原却反之[62]。海藻糖具有稳定生物膜(细胞膜)和蛋白质结构及抗干燥的作用,糖原是菌体内的储备多糖,可以供能和供碳,它们和polyP一起应对饥饿压力。
3.2 寡营养菌和VBNC状态polyP参与了细菌VBNC (Viable but non-culturable)状态的形成,这个状态被广义地定义为细胞进入不可培养状态来抵御环境压力,但是仍然维持可观测的、降低的生命代谢活性(比如降低呼吸速率、营养物质转运和大分子的合成),此过程中RpoS感应并与一些特定的蛋白结合,使菌进入这个状态,同时,生活在寡营养状态下的极端微生物,如Campylobacter jejuni由于基因含量相对较小,可能缺乏一些典型的压力反应系统,通过一些特定的酶和polyP结合来投入这个状态以维持活性[71],以此整合和简化了压力反应。Vibrio cholerae也被研究证实在营养物质匮乏的条件下会进入VBNC状态,同时还会改变菌体形态,变为球形,增大了比表面积以便营养物质的吸收[72],而polyP恰好也与菌体的形变有关[46]。Hammes等在研究不同水环境中微生物体内ATP的含量时也发现,当处于饥饿环境时,菌体变小且ATP含量显著降低[73]。此外,Rao等的研究中发现3个现象:(1) 在饥饿条件下,细胞如同狼群效应一般发生群集反应,通过分泌细胞壁裂解酶和抗生素,聚成一个密实的球状多核子实体以及抗逆性的粘孢子;(2) 营养物质缺乏的情况下细菌存活的机制之一就是发生捕食行为,而成为捕食者还是被食饵取决于其是否有ppk1和polyP;(3) 对ppk1和ppk2的代谢研究中发现有新型抗生素的产生[11],增加了细菌的毒力。此外,在ppk突变体中以及磷缺乏的情况下也会产生抗生素[74],抗生素是细菌抵抗环境压迫的有力盾牌。
Wang等研究在自然界中,大部分微生物具有尺寸微小、不易或不可培养的生理特性,而由于检测方法的局限性,许多微生物不能通过传统的培养方法(如平板技术)所分离检测,人们对其认识还很有限,这类细菌被微生物学家称为“看不见的主体(Unseen majority)”,又称超微细菌[75]。超微细菌体积不足0.1 μm3,被认为是地球上最小的生命,在广阔的海洋中,超微细菌的浓度可达到(0.5-5)×105 cells/mL[76]。在淡水湖泊河流以及土壤中也有超微细菌的存在,并且为优势菌[77-78],这些环境通常营养物质浓度特别低,被称为寡营养环境,所以它们的另一个名字为寡营养细菌[79]。在营养物质缺乏的环境中,这类细菌通过流线型管理和基因缺失来简化营养物质的代谢方式,如避开一些不必要的酶,相应地减少这些酶的基因以及酶的合成[76, 80]。而且寡营养细菌单位体积活性比普通的大细菌活性更高,在营养物质浓度充足时,吸收效率很高且会把营养物质储存起来,在寡营养时的吸收系统缓慢但是高效,对一些高能量的营养物质有较强的亲和力和追踪能力[81],且它们可以利用更广泛的营养物质[82],生长代谢方式经济有效。Temperton等已经证实了在海洋微生物中普遍存在polyP代谢相关基因,且polyP代谢在寡营养环境中有很重要的作用[83]。海洋及其他淡水环境中超微细菌经常保持VBNC状态,简化遗传和代谢系统,减少对营养物质和能量消耗以及生物大分子的合成,降低呼吸速率和减缓细胞分裂。同时,寡营养菌属于一种极端微生物,除了能在寡营养环境中生长以外,还对其他的一些环境压迫有较强的适应性。而普遍存在于微生物体内的polyP对环境压力有一定的适应和调节能力,因此推定polyP在超微细菌适应寡营养环境及其他环境压力的过程中具有重要的作用。
4 应用和展望polyP存在于所有的生物中,且在一些细胞内占了相当大的体积(细胞质体积的37%)和干重(30%),研究者推断早在生命起源之前就有polyP的存在[11],它在细胞的代谢和环境压力胁迫下发挥了多种不可替代的调节作用。polyP活跃于细菌中,使细菌在大幅度的温度和pH值波动、紫外暴露、寡营养、高重金属浓度等极端环境下保持耐受性和活性[12, 46]。由于其独特的化学结构,polyP可以和酶、DNA、RNA等生物因子相互作用,在各个层面上调节生命活动,从简单的能量供给与物质代谢和转运,到信号传导,再到蛋白质的合成与基因的表达和修复,它会继续参与“物竞天择,适者生存”的生物进化过程。
大到海洋生态环境,小到生产生活的实际应用,polyP扮演了许多重要的角色:在磷浓度极低的马尾藻的研究中发现,光合浮游菌中富含polyP,海洋微生物的polyP积累和代谢是整个海洋环境中P元素循环的重要环节[84];作为阻燃剂、肥料、食品添加剂、防腐剂、凝血剂、骨骼替代材料、释药剂、洗衣液添加剂等[9, 85],它也渗入到人们日常生活的方方面面。研究人员把PPK等酶聚合到细菌的包涵体中,作为固定化生物催化酶广泛地应用于工业化学合成[86],polyP的微生物积累和化工生产之间如果可以相辅相成,联合应用,这个新的视角会带来更大的贡献。
polyP在环境治理领域具有广泛的应用潜力。Nagata等通过把细菌体内的ppx基因转移到烟草中,成功实现了对汞污染土壤的植物修复[54]。污水处理中,聚磷菌在高效除磷系统中承担了主要作用[24],对水体富营养化也有一定的作用。在生活污水的生物处理工艺中,常常因为一些重金属离子的存在而使一些参与有机物代谢的酶变性失活,可尝试投加适量的聚磷酸盐或是聚磷酸盐菌以改善此类状况。嗜极性微生物及其产生的polyP也将在环境修复上具有很好的前景,如对微污染水体或土壤的重金属解毒,饮用水的软化和除盐。工业废水中一些酸性、高温和高盐等极端环境也可以尝试通过它们净化,如在各种生物反应器中投入野生的或基因构建的聚磷酸盐菌,它们对这些难生物处理的水质有较强的适应性,保持活性和降解能力,而且当环境猛烈变化时细菌可以大量产生polyP,因此有极强的稳定性和较强的世代时间,抗水力负荷强,从而更经济、更广泛地净化废水。
另一方面,虽然某些致病菌产生的polyP由于其超强的抗性,是一些疾病难以治愈的元凶,但是它的代谢会引起一些新型抗生素的合成,这在医学和药物化学上具有重要的价值。polyP被证实具有HIV抗性[87],并且参与了哺乳动物体内癌细胞增殖[88],这有可能会推动对人体免疫缺陷病毒和癌症治疗的发展。有研究发现一种药物可以抑制PPK酶的活性,增强PPX酶的活性,以减少菌体内polyP的合成,从而降低菌体的抗性和毒理效应,来治疗诸如铜绿假单胞菌、霍乱弧菌等病原菌给人体带来的疾病[89],以polyP为核心的代谢体系会给新药物的研发提供靶点。总之,polyP具有超凡的生物活性,它的代谢调控和生物转化作用给医学卫生、环境保护和工业应用提供了更具吸引力的选择,以polyP为中心的环境压力调控网庞大而复杂,研究者可以通过基因工程和结构生物学等先进的科学技术继续深入阐明具体的调控脉络,为环境污染治理和医学领域提供更多的研究应用基础。
| [1] | Harold FM. Inorganic polyphosphates in biology: structure, metabolism, and function[J]. Bacteriological Reviews, 1966, 30(4) : 772–794. |
| [2] | Brown MRW, Kornberg A. Inorganic polyphosphate in the origin and survival of species[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(46) : 16085–16087. DOI:10.1073/pnas.0406909101 |
| [3] | Achbergerová L, Nahálka J. Polyphosphate-an ancient energy source and active metabolic regulator[J]. Microbial Cell Factories, 2011, 10 : 63. DOI:10.1186/1475-2859-10-63 |
| [4] | Kornberg SR. Adenosine triphosphate synthesis from polyphosphate by an enzyme from Escherichia coli[J]. Biochimica et Biophysica Acta, 1957, 26(2) : 294–300. DOI:10.1016/0006-3002(57)90008-2 |
| [5] | Albi T, Serrano A. Two strictly polyphosphate-dependent gluco(manno)kinases from diazotrophic Cyanobacteria with potential to phosphorylate hexoses from polyphosphates[J]. Applied Microbiology and Biotechnology, 2015, 99(9) : 3887–3900. DOI:10.1007/s00253-014-6184-7 |
| [6] | Kornberg A, Rao NN, Ault-Riché D. Inorganic polyphosphate: a molecule of many functions[J]. Annual Review of Biochemistry, 1999, 68 : 89–125. DOI:10.1146/annurev.biochem.68.1.89 |
| [7] | Kulaev I, Vagabov V, Kulakovskaya T. New aspects of inorganic polyphosphate metabolism and function[J]. Journal of Bioscience and Bioengineering, 1999, 88(2) : 111–129. DOI:10.1016/S1389-1723(99)80189-3 |
| [8] | Yang ZH, Peng L, Huang MF, et al. The role of polyphosphate in bacteria and mammalian cells[J]. Microbiology China, 2015, 42(11) : 2223–2228. (in Chinese) 杨正慧, 彭亮, 黄慕芳, 等. 多聚磷酸盐在细菌和哺乳动物细胞中的作用[J]. 微生物学通报, 2015, 42(11) : 2223–2228. |
| [9] | Levering J, Musters MWJM, Bekker M, et al. Role of phosphate in the central metabolism of two lactic acid bacteria-a comparative systems biology approach[J]. FEBS Journal, 2012, 279(7) : 1274–1290. DOI:10.1111/j.1742-4658.2012.08523.x |
| [10] | Kulakovskaya TV, Vagabov VM, Kulaev IS. Inorganic polyphosphate in industry, agriculture and medicine: Modern state and outlook[J]. Process Biochemistry, 2012, 47(1) : 1–10. DOI:10.1016/j.procbio.2011.10.028 |
| [11] | Rao NN, Gómez-García MR, Kornberg A. Inorganic polyphosphate: essential for growth and survival[J]. Annual Review of Biochemistry, 2009, 78 : 605–647. DOI:10.1146/annurev.biochem.77.083007.093039 |
| [12] | Orell A, Navarro CA, Rivero M, et al. Inorganic polyphosphates in extremophiles and their possible functions[J]. Extremophiles, 2012, 16(4) : 573–583. DOI:10.1007/s00792-012-0457-9 |
| [13] | Moreno-Sanchez D, Hernandez-Ruiz L, Ruiz FA, et al. Polyphosphate is a novel pro-inflammatory regulator of mast cells and is located in acidocalcisomes[J]. Journal of Biological Chemistry, 2012, 287(34) : 28435–28444. DOI:10.1074/jbc.M112.385823 |
| [14] | Seufferheld MJ, Kim KM, Whitfield J, et al. Evolution of vacuolar proton pyrophosphatase domains and volutin granules: clues into the early evolutionary origin of the acidocalcisome[J]. Biology Direct, 2011, 6 : 50. DOI:10.1186/1745-6150-6-50 |
| [15] | Seufferheld M, Vieira MCF, Ruiz FA, et al. Identification of organelles in bacteria similar to acidocalcisomes of unicellular eukaryotes[J]. Journal of Biological Chemistry, 2003, 278(32) : 29971–29978. DOI:10.1074/jbc.M304548200 |
| [16] | Renier ML, Kohn DH. Development and characterization of a biodegradable polyphosphate[J]. Journal of Biomedical Materials Research, 1997, 34(1) : 95–104. DOI:10.1002/(ISSN)1097-4636 |
| [17] | Kulakova AN, Hobbs D, Smithen M, et al. Direct quantification of inorganic polyphosphate in microbial cells using 4'-6-Diamidino-2-Phenylindole (DAPI)[J]. Environmental Science & Technology, 2011, 45(18) : 7799–7803. |
| [18] | Werner TP, Amrhein N, Freimoser FM. Novel method for the quantification of inorganic polyphosphate (iPoP) in Saccharomyces cerevisiae shows dependence of iPoP content on the growth phase[J]. Archives of Microbiology, 2005, 184(2) : 129–136. DOI:10.1007/s00203-005-0031-2 |
| [19] | Sukenik A, Kaplan-Levy RN, Welch JM, et al. Massive multiplication of genome and ribosomes in dormant cells (akinetes) of Aphanizomenon ovalisporum (Cyanobacteria)[J]. The ISME Journal, 2012, 6(3) : 670–679. DOI:10.1038/ismej.2011.128 |
| [20] | Jimenez-Nuñez MD, Moreno-Sanchez D, Hernandez-Ruiz L, et al. Myeloma cells contain high levels of inorganic polyphosphate which is associated with nucleolar transcription[J]. Haematologica, 2012, 97(8) : 1264–1271. DOI:10.3324/haematol.2011.051409 |
| [21] | Güenther S, Trutnau M, Kleinsteuber S, et al. Dynamics of polyphosphate-accumulating bacteria in wastewater treatment plant microbial communities detected via DAPI (4', 6'-Diamidino-2-Phenylindole) and tetracycline labeling[J]. Applied and Environmental Microbiology, 2009, 75(7) : 2111–2121. DOI:10.1128/AEM.01540-08 |
| [22] | Gomes FM, Ramos IB, Wendt C, et al. New insights into the in situ microscopic visualization and quantification of inorganic polyphosphate stores by 4ʹ, 6-diamidino-2-phenylindole (DAPI)-staining[J]. European Journal of Histochemistry, 2013, 57(4) : e34. DOI:10.4081/ejh.2013.e34 |
| [23] | Diaz JM, Ingall ED. Fluorometric quantification of natural inorganic polyphosphate[J]. Environmental Science & Technology, 2010, 44(12) : 4665–4671. |
| [24] | Mesquita DP, Luis Amaral A, Leal C, et al. Monitoring intracellular polyphosphate accumulation in enhanced biological phosphorus removal systems by quantitative image analysis[J]. Water Science and Technology, 2014, 69(11) : 2315–2323. DOI:10.2166/wst.2014.146 |
| [25] | Wolf SG, Rez P, Elbaum M. Phosphorus detection in vitrified bacteria by cryo-STEM annular dark-field analysis[J]. Journal of Microscopy, 2015, 260(2) : 227–233. DOI:10.1111/jmi.2015.260.issue-2 |
| [26] | Saito K, Ohtomo R, Kuga-Uetake Y, et al. Direct labeling of polyphosphate at the ultrastructural level in Saccharomyces cerevisiae by using the affinity of the polyphosphate binding domain of Escherichia coli exopolyphosphatase[J]. Applied and Environmental Microbiology, 2005, 71(10) : 5692–5701. DOI:10.1128/AEM.71.10.5692-5701.2005 |
| [27] | Diaz J, Ingall E, Benitez-Nelson C, et al. Marine polyphosphate: a key player in geologic phosphorus sequestration[J]. Science, 2008, 320(5876) : 652–655. DOI:10.1126/science.1151751 |
| [28] | Serafim LS, Lemos PC, Levantesi C, et al. Methods for detection and visualization of intracellular polymers stored by polyphosphate-accumulating microorganisms[J]. Journal of Microbiological Methods, 2002, 51(1) : 1–18. DOI:10.1016/S0167-7012(02)00056-8 |
| [29] | Aschar-Sobbi R, Abramov AY, Diao C, et al. High sensitivity, quantitative measurements of polyphosphate using a new DAPI-Based approach[J]. Journal of Fluorescence, 2008, 18(5) : 859–866. DOI:10.1007/s10895-008-0315-4 |
| [30] | Martin P, van Mooy BAS. Fluorometric quantification of polyphosphate in environmental plankton samples: extraction protocols, matrix effects, and nucleic acid interference[J]. Applied and Environmental Microbiology, 2013, 79(1) : 273–281. DOI:10.1128/AEM.02592-12 |
| [31] | Klauth P, Pallerla SR, Vidaurre D, et al. Determination of soluble and granular inorganic polyphosphate in Corynebacterium glutamicum[J]. Applied Microbiology and Biotechnology, 2006, 72(5) : 1099–1106. DOI:10.1007/s00253-006-0562-8 |
| [32] | Wei Z, Nie YH, Liu LT, et al. Progress in functional polyphosphate in prokaryotic and eukaryotic living organisms[J]. Progress in Physiological Sciences, 2009, 40(3) : 197–202. (in Chinese) 魏峥, 聂琰晖, 刘乐庭, 等. 多聚磷酸盐在原核和真核生物中的研究进展[J]. 生理科学进展, 2009, 40(3) : 197–202. |
| [33] | Nikel PI, Chavarría M, Martínez-García E, et al. Accumulation of inorganic polyphosphate enables stress endurance and catalytic vigour in Pseudomonas putida KT2440[J]. Microbial Cell Factories, 2013, 12 : 50. DOI:10.1186/1475-2859-12-50 |
| [34] | Whitehead MP, Eagles L, Hooley P, et al. Most bacteria synthesize polyphosphate by unknown mechanisms[J]. Microbiology, 2014, 160 : 829–831. DOI:10.1099/mic.0.075366-0 |
| [35] | Albi T, Serrano A. Two exopolyphosphatases with distinct molecular architectures and substrate specificities from the thermophilic green-sulfur bacterium Chlorobium tepidum TLS[J]. Microbiology, 2014, 160 : 2067–2078. DOI:10.1099/mic.0.080952-0 |
| [36] | Potrykus K, Cashel M. (p)ppGpp: still magical?[J]. Annual Review of Microbiology, 2008, 62 : 35–51. DOI:10.1146/annurev.micro.62.081307.162903 |
| [37] | Keasling JD, Bertsch L, Kornberg A. Guanosine pentaphosphate phosphohydrolase of Escherichia coli is a long-chain exopolyphosphatase[J]. Proceedings of the National Academy of Sciences of the United States of America, 1993, 90(15) : 7029–7033. DOI:10.1073/pnas.90.15.7029 |
| [38] | Camci IY, Doruk T, Avican U, et al. Deletion of polyphosphate kinase gene (ppk) has a stimulatory effect on actinorhodin production by Streptomyces coelicolor A3(2)[J]. Turkish Journal of Biology, 2012, 36(4) : 373–380. |
| [39] | Diaz JM, Ingall ED, Snow SD, et al. Potential role of inorganic polyphosphate in the cycling of phosphorus within the hypoxic water column of Effingham Inlet, British Columbia[J]. Global Biogeochemical Cycles, 2012, 26(2) : GB2040. |
| [40] | Peng L, Luo WY, Zhao T, et al. Polyphosphate kinase 1 is required for the pathogenesis process of meningitic Escherichia coli K1 (RS218)[J]. Future Microbiology, 2012, 7(3) : 411–423. DOI:10.2217/fmb.12.3 |
| [41] | Alcántara C, Blasco A, Zúñiga M, et al. Accumulation of polyphosphate in Lactobacillus spp. and its involvement in stress resistance[J]. Applied and Environmental Microbiology, 2014, 80(5) : 1650–1659. DOI:10.1128/AEM.03997-13 |
| [42] | Gallarato LA, Sánchez DG, Olvera L, et al. Exopolyphosphatase of Pseudomonas aeruginosa is essential for the production of virulence factors, and its expression is controlled by NtrC and PhoB acting at two interspaced promoters[J]. Microbiology, 2014, 160 : 406–417. DOI:10.1099/mic.0.074773-0 |
| [43] | Freimoser FM, Hürlimann HC, Jakob CA, et al. Systematic screening of polyphosphate (poly P) levels in yeast mutant cells reveals strong interdependence with primary metabolism[J]. Genome Biology, 2006, 7 : R109. DOI:10.1186/gb-2006-7-11-r109 |
| [44] | Luna MAC, Vieira ER, Okada K, et al. Copper-induced adaptation, oxidative stress and its tolerance in Aspergillus niger UCP1261[J]. Electronic Journal of Biotechnology, 2015, 18(6) : 418–427. DOI:10.1016/j.ejbt.2015.09.006 |
| [45] | Cheng YY, Sun BL. Polyphosphate kinase affects oxidative stress response by modulating cAMP receptor protein and rpoS expression in salmonella typhimurium[J]. Journal of Microbiology and Biotechnology, 2009, 19(12) : 1527–1535. DOI:10.4014/jmb |
| [46] | Seufferheld MJ, Alvarez HM, Farias ME. Role of polyphosphates in microbial adaptation to extreme environments[J]. Applied and Environmental Microbiology, 2008, 74(19) : 5867–5874. DOI:10.1128/AEM.00501-08 |
| [47] | Charlebois A, Jacques M, Boulianne M, et al. Tolerance of Clostridium perfringens biofilms to disinfectants commonly used in the food industry[J]. Food Microbiology, 2017, 62 : 32–38. DOI:10.1016/j.fm.2016.09.009 |
| [48] | Cam N, Benzerara K, Georgelin T, et al. Selective uptake of alkaline earth metals by cyanobacteria forming intracellular carbonates[J]. Environmental Science & Technology, 2016, 50(21) : 11654–11662. |
| [49] | Kampinga HH. Chaperoned by prebiotic inorganic polyphosphate molecules: an ancient transcription-independent mechanism to restore protein homeostasis[J]. Molecular Cell, 2014, 53(5) : 685–687. DOI:10.1016/j.molcel.2014.02.023 |
| [50] | Albi T, Serrano A. Inorganic polyphosphate in the microbial world. Emerging roles for a multifaceted biopolymer[J]. World Journal of Microbiology and Biotechnology, 2016, 32 : 27. DOI:10.1007/s11274-015-1983-2 |
| [51] | Gray MJ, Wholey WY, Wagner NO, et al. Polyphosphate is a primordial chaperone[J]. Molecular Cell, 2014, 53(5) : 689–699. DOI:10.1016/j.molcel.2014.01.012 |
| [52] | Ramisetty BCM, Ghosh D, Chowdhury MR, et al. What is the link between stringent response, endoribonuclease encoding type Ⅱ toxin-antitoxin systems and persistence?[J]. Frontiers in Microbiology, 2016, 7 : 1882. |
| [53] | Merroun ML, Selenska-Pobell S. Bacterial interactions with uranium: an environmental perspective[J]. Journal of Contaminant Hydrology, 2008, 102(3/4) : 285–295. |
| [54] | Nagata T, Kiyono M, Pan-Hou H. Engineering expression of bacterial polyphosphate kinase in tobacco for mercury remediation[J]. Applied Microbiology and Biotechnology, 2006, 72(4) : 777–782. DOI:10.1007/s00253-006-0336-3 |
| [55] | Pavlov E, Grimbly C, Diao CTM, et al. A high-conductance mode of a poly-3-hydroxybutyrate/calcium/polyphosphate channel isolated from competent Escherichia coli cells[J]. FEBS Letters, 2005, 579(23) : 5187–5192. DOI:10.1016/j.febslet.2005.08.032 |
| [56] | Esnault C, Leiber D, Toffano-Nioche C, et al. Another example of enzymatic promiscuity: the polyphosphate kinase of Streptomyces lividans is endowed with phospholipase D activity[J]. Applied Microbiology and Biotechnology, 2017, 101(1) : 139–145. DOI:10.1007/s00253-016-7743-x |
| [57] | Wang XX, Wang SY, Zhao J, et al. Combining simultaneous nitrification-endogenous denitrification and phosphorus removal with post-denitrification for low carbon/nitrogen wastewater treatment[J]. Bioresource Technology, 2016, 220 : 17–25. DOI:10.1016/j.biortech.2016.06.132 |
| [58] | Weerasekara AW, Jenkins S, Abbott LK, et al. Microbial phylogenetic and functional responses within acidified wastewater communities exhibiting enhanced phosphate uptake[J]. Bioresource Technology, 2016, 220 : 55–61. DOI:10.1016/j.biortech.2016.08.037 |
| [59] | Kuroda A, Nomura K, Ohtomo R, et al. Role of inorganic polyphosphate in promoting ribosomal protein degradation by the lon protease in E. coli[J]. Science, 2001, 293(5530) : 705–708. DOI:10.1126/science.1061315 |
| [60] | Drozd M, Gangaiah D, Liu Z, et al. Contribution of TAT system translocated PhoX to Campylobacter jejuni phosphate metabolism and resilience to environmental stresses[J]. PLoS One, 2011, 6(10) : e26336. DOI:10.1371/journal.pone.0026336 |
| [61] | Sureka K, Dey S, Datta P, et al. Polyphosphate kinase is involved in stress-induced mprAB-sigE-rel signalling in mycobacteria[J]. Molecular Microbiology, 2007, 65(2) : 261–276. DOI:10.1111/mmi.2007.65.issue-2 |
| [62] | Yano T, Funamizu Y, Yoshida N. Intracellular accumulation of trehalose and glycogen in an extreme oligotroph, Rhodococcus erythropolis N9T-4[J]. Bioscience, Biotechnology, and Biochemistry, 2016, 80(3) : 610–613. DOI:10.1080/09168451.2015.1107467 |
| [63] | Yang ZX, Zhou YN, Yang Y, et al. Polyphosphate binds to the principal sigma factor of RNA polymerase during starvation response in Helicobacter pylori[J]. Molecular Microbiology, 2010, 77(3) : 618–627. DOI:10.1111/mmi.2010.77.issue-3 |
| [64] | Kim KS, Rao NN, Fraley CD, et al. Inorganic polyphosphate is essential for long-term survival and virulence factors in Shigella and Salmonella spp.[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(11) : 7675–7680. DOI:10.1073/pnas.112210499 |
| [65] | English BP, Hauryliuk V, Sanamrad A, et al. Single-molecule investigations of the stringent response machinery in living bacterial cells[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(31) : E365–E373. DOI:10.1073/pnas.1102255108 |
| [66] | Wang SX, Wei JT, Li TB, et al. Research on regulation of protein RpoS expression and its function in bacteria[J]. Biotechnology Bulletin, 2011(10) : 24–31. (in Chinese) 王淑娴, 魏鉴腾, 李天保, 等. 细菌中RpoS蛋白的表达调控及其功能的研究进展[J]. 生物技术通报, 2011(10) : 24–31. |
| [67] | Shiba T, Tsutsumi K, Yano H, et al. Inorganic polyphosphate and the induction of rpoS expression[J]. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(21) : 11210–11215. DOI:10.1073/pnas.94.21.11210 |
| [68] | Gottesman S, Maurizi MR. Surviving starvation[J]. Science, 2001, 293(5530) : 614–615. DOI:10.1126/science.1063371 |
| [69] | Azevedo C, Saiardi A. The new world of inorganic polyphosphates[J]. Biochemical Society Transactions, 2016, 44 : 13–17. DOI:10.1042/BST20150210 |
| [70] | Wei ZZ, Vatcher G, Tin AHY, et al. Positively-charged semi-tunnel is a structural and surface characteristic of polyphosphate-binding proteins: an in-silico study[J]. PLoS One, 2015, 10(4) : e0123713. DOI:10.1371/journal.pone.0123713 |
| [71] | Kassem II, Chandrashekhar K, Rajashekara G. Of energy and survival incognito: a relationship between viable but non-culturable cells formation and inorganic polyphosphate and formate metabolism in Campylobacter jejuni[J]. Frontiers in Microbiology, 2013, 4 : 183. |
| [72] | Krebs SJ, Taylor RK. Nutrient-dependent, rapid transition of Vibrio cholerae to coccoid morphology and expression of the toxin co-regulated pilus in this form[J]. Microbiology, 2011, 157(10) : 2942–2953. DOI:10.1099/mic.0.048561-0 |
| [73] | Hammes F, Goldschmidt F, Vital M, et al. Measurement and interpretation of microbial adenosine tri-phosphate (ATP) in aquatic environments[J]. Water Research, 2010, 44(13) : 3915–3923. DOI:10.1016/j.watres.2010.04.015 |
| [74] | Rossa CA, White J, Kuiper A, et al. Carbon flux distribution in antibiotic-producing chemostat cultures of Streptomyces lividans[J]. Metabolic Engineering, 2002, 4(2) : 138–150. DOI:10.1006/mben.2001.0217 |
| [75] | Wang YY, Hammes F, Boon N, et al. Isolation and characterization of low nucleic acid (LNA)-content bacteria[J]. The ISME Journal, 2009, 3(8) : 889–902. DOI:10.1038/ismej.2009.46 |
| [76] | Williams TJ, Ertan H, Ting L, et al. Carbon and nitrogen substrate utilization in the marine bacterium Sphingopyxis alaskensis strain RB2256[J]. The ISME Journal, 2009, 3(9) : 1036–1052. DOI:10.1038/ismej.2009.52 |
| [77] | Liu J, Song YH, Zhao ZP, et al. Filterability of bacteria with low nucleic acid content in three freshwater environments[J]. Microbiology China, 2016, 43(9) : 1895–1901. (in Chinese) 刘杰, 宋宇昊, 赵志鹏, 等. 3种淡水环境中低核酸含量细菌的滤过性[J]. 微生物学通报, 2016, 43(9) : 1895–1901. |
| [78] | Liu J, Song YH, Ma R, et al. Correlation of LNA and HNA bacteria based on flow cytometric characteristics[J]. China Environmental Science, 2016, 36(3) : 865–874. (in Chinese) 刘杰, 宋宇昊, 马芮, 等. 基于流式细胞特性的LNA和HNA细菌相关性研究[J]. 中国环境科学, 2016, 36(3) : 865–874. |
| [79] | Liu J, Li L, Wang YY. Distribution and ecological role of small bacteria in aquatic environments-a review[J]. Microbiology China, 2014, 41(10) : 2100–2111. (in Chinese) 刘杰, 李蕾, 王莹莹. 水环境中微小细菌的分布及生态作用研究进展[J]. 微生物学通报, 2014, 41(10) : 2100–2111. |
| [80] | García-Fernández JM, de Marsac NT, Diez J. Streamlined regulation and gene loss as adaptive mechanisms in Prochlorococcus for optimized nitrogen utilization in oligotrophic environments[J]. Microbiology and Molecular Biology Reviews, 2004, 68(4) : 630–638. DOI:10.1128/MMBR.68.4.630-638.2004 |
| [81] | Salcher MM, Pernthaler J, Posch T. Seasonal bloom dynamics and ecophysiology of the freshwater sister clade of SAR11 bacteria 'that rule the waves' (LD12)[J]. The ISME Journal, 2011, 5(8) : 1242–1252. DOI:10.1038/ismej.2011.8 |
| [82] | Han DD, Hao ZY, Gao GH, et al. Ecological function of oligotrophic bacteria and their applications in the environment[J]. Microbiology China, 2012, 39(4) : 526–535. (in Chinese) 韩东东, 郝振宇, 高广海, 等. 寡营养细菌及其生态作用和应用的研究进展[J]. 微生物学通报, 2012, 39(4) : 526–535. |
| [83] | Temperton B, Gilbert JA, Quinn JP, et al. Novel analysis of oceanic surface water metagenomes suggests importance of polyphosphate metabolism in oligotrophic environments[J]. PLoS One, 2011, 6(1) : e16499. DOI:10.1371/journal.pone.0016499 |
| [84] | Martin P, Dyhrman ST, Lomas MW, et al. Accumulation and enhanced cycling of polyphosphate by Sargasso Sea plankton in response to low phosphorus[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(22) : 8089–8094. DOI:10.1073/pnas.1321719111 |
| [85] | Cini N, Ball V. Polyphosphates as inorganic polyelectrolytes interacting with oppositely charged ions, polymers and deposited on surfaces: fundamentals and applications[J]. Advances in Colloid and Interface Science, 2014, 209 : 84–97. DOI:10.1016/j.cis.2014.01.011 |
| [86] | Hrabárová E, Achbergerová L, Nahálka J. Insoluble protein applications: the use of bacterial inclusion bodies as biocatalysts[A]//García-Fruitós E. Insoluble Proteins[M]. New York: Springer, 2015: 411-422 |
| [87] | Lorenz B, Leuck J, Köehl D, et al. Anti-HIV-1 activity of inorganic polyphosphates[J]. Journal of Acquired Immune Deficiency Syndromes and Human Retrovirology, 1997, 14(2) : 110–118. DOI:10.1097/00042560-199702010-00003 |
| [88] | Dedkova EN, Blatter LA. Role of β-hydroxybutyrate, its polymer poly-β-hydroxybutyrate and inorganic polyphosphate in mammalian health and disease[J]. Frontiers in Physiology, 2014, 5 : 260. |
| [89] | Kornberg A. Reducing the amount of polyphosphate in a microorganism for preparing a composition for treating diseases caused by pathogenic microorganisms, e.g. Helicobacter pylori, Pseudomonas aeruginosa, Vibrio cholera or Shigella flexneri[P]. US2003162691-A1 |
 2017, Vol. 44
2017, Vol. 44




