扩展功能
文章信息
- 刘洋荧, 王尚, 厉舒祯, 邓晔
- LIU Yang-ying, WANG Shang, LI Shu-zhen, DENG Ye
- 基于功能基因的微生物碳循环分子生态学研究进展
- Advances in molecular ecology on microbial functional genes of carbon cycle
- 微生物学通报, 2017, 44(7): 1676-1689
- Microbiology China, 2017, 44(7): 1676-1689
- DOI: 10.13344/j.microbiol.china.160941
-
文章历史
- 收稿日期: 2016-12-22
- 接受日期: 2017-03-08
- 优先数字出版日期(www.cnki.net): 2017-04-05
2. 中国科学院大学资源与环境学院 北京 100049;
3. 大连理工大学环境学院 工业生态与环境工程教育部重点实验室 辽宁 大连 116024
2. College of Resources and Environment, University of Chinese Academy of Sciences, Beijing 100049, China;
3. Key Laboratory of Industrial Ecology and Environmental Engineering (Ministry of Education), School of Environmental Science and Technology, Dalian University of Technology, Dalian, Liaoning 116024, China
碳是构成有机体的必需元素,也是有机体中含量最多的元素之一,碳的生物地球化学循环(碳循环)是生态系统中最重要的物质循环之一。微生物具有生物量大、种类复杂、代谢功能多样、相互作用关系复杂等特点,介导了碳循环多个重要代谢过程(图 1),如碳固定(CO2转化成有机物的过程)、甲烷代谢(产甲烷和甲烷氧化过程)和碳降解(有机质的分解过程)等三个基本过程。具有相似功能的微生物类群构成微生物群落的基本功能单元,不同的功能群落共同调控和驱动碳循环各个过程,在响应全球气候变化、维持生态系统的功能和稳定方面有着不可替代的作用[1-3]。
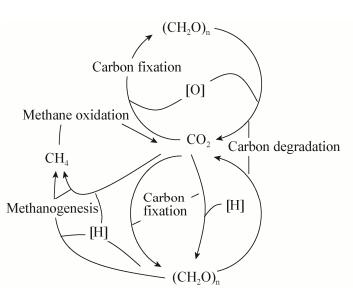
|
| 图 1 微生物驱动的碳循环过程 Figure 1 Carbon cycle driven by microorganisms |
|
|
Handelsman等于1998年首先提出了宏基因组(又称为元基因组,Metagenome)的概念[4]。宏基因组技术通过非培养的手段,基于环境样品中所有微生物的DNA来获得微生物群落遗传信息,因此可更加全面真实地反映微生物群落的结构[5-6]。宏基因组学技术的研究对象可以是基因组也可以是某个基因[7]。以基因组为中心的研究基于大规模宏基因组鸟枪法测序,测序完成后进行宏基因组序列拼接,并与已知基因序列数据库的相似性比对来进行微生物分类和功能的注释。其优势是可发现新的基因,缺点是目标不明确且分析工作量大[8]。以基因为中心的宏基因组学研究以微生物特定基因片段(如16S rRNA基因和功能基因等)为研究对象,减少了海量数据分析的限制,有针对性地对微生物群落的组成结构以及与生态过程密切相关的功能类群进行研究,极大地减少了工作量[9],同时随着分析流程的不断完善,也为后续生态过程的研究提供了保障。
针对微生物群落的宏基因组学技术主要包含PCR扩增、DNA指纹图谱、分子杂交、克隆文库以及高通量测序等技术,其中代表性方法主要包括PCR技术中的实时荧光定量PCR技术(qPCR),指纹图谱技术中的末端限制性片段长度多态性分析(T-RFLP)和变性梯度凝胶电泳(DGGE),分子杂交技术中的基因芯片技术,以及测序技术中的克隆文库和高通量测序(表 1)。
| 分类 Classification |
代表性技术 Representative techniques |
原理 Principle |
特点 Characteristics |
| PCR | qPCR[10-16] | PCR扩增过程中,通过荧光信号积累实时监测PCR进程,依据PCR扩增指数时期模板的Ct值和模板起始拷贝数的线性关系,从而实现初始基因拷贝数定量 | 与传统PCR技术相较,qPCR可定量目标序列,且操作简便、灵敏度高、稳定性和重复性好,在宏基因组学的研究中可用于对16S rRNA基因或特定功能基因等分子标记基因的定量,从而实现环境微生物总丰度或特定环境微生物类群丰度的定量 |
| DNA分子 指纹图谱 DNA-fingerprinting |
T-RFLP[17-19] | 利用荧光标记的特异性引物扩增宏基因组,扩增后产生的基因片段存在不同的酶切位点,经限制性内切酶酶切之后产生不同长度的限制性片段,使用测序仪检测带有荧光标记的末端片段,不同长度末端片段代表不同类型微生物物种 | 可直观、快速分析复杂的微生物群落物种区系,提供不同物种种类相对数量的信息;但无法检测低丰度微生物种类以及直接鉴定微生物功能,通常需结合其他分子技术来鉴定微生物种类以及功能 |
| DGGE[20-21] | 长度相似碱基序列不同的基因片段在不同浓度的变性剂中溶解温度不同,基因片段通过变性剂浓度呈梯度增加的凝胶时,基因片段迁移至某一点变性剂浓度恰好相当于此段DNA的低熔点区的Tm值,此区便开始熔解,而高熔点区仍为双链。局部解链的DNA分子迁移率发生改变,从而达到分离效果,形成的不同条带代表不同的物种 | ||
| 基因芯片 Microarray |
Geochip[22-24] | 在固相支持物上原位合成寡核苷酸或者直接将大量预先制备的DNA探针固定在支持物表面,然后与荧光标记样品杂交,通过对杂交信号的检测分析进而得到样品的物种和功能的遗传信息 | 可探测不同环境中微生物群落功能的多样性,检测通量高、速度快、灵敏性和特异性强;但基因芯片属于封闭系统,无法检测新基因 |
| 高通量测序 High-throughput sequencing |
Illumina[25-26] | 首先将目标DNA剪切为小分子片段,通过接头连接到固相表面进行扩增,产生大量拷贝,每复制一次碱基则输出一次荧光信号,经高分辨率成像系统检测最终信号,最后通过生物信息学分析通过原始数据确定物种遗传信息 | 高通量测序具有通量高、速度快、价格低廉等优势;但海量数据的产生对数据分析产生限制,PCR扩增引入误差,物种和基因丰度可能被高估,基于16S rRNA基因的分析仅能确定到属水平 |
| 克隆文库[18] Clone library |
将目标DNA片段通过PCR扩增,然后将其连接到载体中,进而转化到大肠杆菌内,得到阳性克隆后经一代测序分析即可构建目的基因克隆文库 | 构建克隆文库再对含目的基因片段的载体进行测序效率更高,可测片段更长。但是构建克隆文库再测序,操作过程繁琐且价格较高 |
目前已经发现6条微生物的碳固定途径:卡尔文循环、还原柠檬酸循环、还原乙酰辅酶A途径、3-羟基丙酸双循环、3-羟基丙酸/4-羟基丁酸循环、二羧酸/4-羟基丁酸循环[27]。其中卡尔文循环是光能自养生物和好氧化能自养生物固定CO2的主要途径,而其余5条固碳途径则因能量需求、还原物需求、必要金属、辅酶的使用及对O2的敏感程度等方面都有较大区别,因而这几条固定CO2的途径在微生物中分布不同。微生物固定碳的途径、关键酶以及编码关键酶重要亚基的功能基因如表 2所示。
| 固碳途径 Carbon fixtion pathways |
关键酶 Enzymes |
功能基因 Function genes |
| 卡尔文循环 Reductive pentose phosphate cycle (Calvin cycle) |
二磷酸核酮糖羧化酶 | cbbL (rbcL) cbbM |
| 还原三羧酸循环 Reductive tricarboxylic acid cycle |
丙酮酸:铁氧化还原蛋白酶 2-氧化戊二酸:铁氧化还原蛋白酶 ATP-柠檬酸裂解酶 |
porA/nifJ oorA aclB |
| 厌氧乙酰辅酶A途径 Reductive acetyl-CoA pathway (Wood-Ljungdahl pathway) |
一氧化碳脱氢酶 乙酰辅酶A合成酶 |
acsA (cooS) acsB |
| 3-羟基丙酸双循环 3-Hydroxypropionate bicycle |
丙二酰辅酶A还原酶 丙酰辅酶A合成酶 苹果酰辅酶A/β-甲基苹果酰辅酶A/柠苹酰辅酶A裂解酶 |
Pcc/Acc |
| 3-羟基丙酸/4-羟基丁酸循环 3-Hydroxypropionate/4-hydroxybutylate cycle |
乙酰辅酶A/丙酰辅酶A羧化酶 4-羟丁酰辅酶A脱氢酶 丙二酰辅酶A还原酶 |
accA hcd |
| 二羧酸/4-羟基丁酸循环 Dicarboxylate/4-hydroxybutyrate cycle |
磷酸烯醇丙酮酸羧化酶 丙酮酸合成酶 4-羟基丁酰辅酶A脱氢酶 |
名称未统一 |
卡尔文循环(Calvin-Benson-Bassham cycle)是自然界普遍存在于植物和藻类中的CO2固定途径,也是光能自养微生物固定CO2途径,对调节和响应全球CO2浓度具有关键作用。卡尔文循环中的关键酶是核酮糖-1, 5-二磷酸羧化/加氧酶,即RubisCO酶,它催化卡尔文循环中的第一步CO2固定反应[29]。已经发现的RubisCO酶有4种形式,而参与卡尔文循环的RubisCO酶主要有两种存在形式:Ⅰ型RubisCO和Ⅱ型RubisCO,分别对应的功能基因为cbbL (或者rbcL)和cbbM。对一些数量相对较少且使用16S rRNA基因检测比较困难的微生物物种,如果其体内存在RubisCO基因,可以通过功能基因cbbL和cbbM来进行检测。这两种功能基因保守性与长度适合,因而可代替16S rRNA基因用作另一种基因标记物来检测环境中的固碳微生物群落及进行进化分析[30-31]。目前RubisCO的功能基因cbbL和cbbM已经从水生生态系统、陆地生态系统以及极端环境等各个类型的生态系统中被检出。
水生生态系统,尤其是海洋生态系统是一个巨大的碳库。水生生态系统中存在的光合细菌、化能自养菌及微藻类等微生物是最主要的固碳生物,通过卡尔文循环来固定CO2,利用cbbL和cbbM作为分子标记可反映不同水生生态系统和同一水生生态系统不同深度的的固碳能力差异。目前已有大量关于利用cbbL和cbbM功能基因研究水生生态系统中固碳微生物类群的报道。Alfreider等[32]研究了厌氧和微氧蓄水层和地下水中RubisCO大亚基基因cbbL和cbbM的分布和进化多样性,验证了可固定CO2的自养微生物存在于所研究的两个地下水修复项目中的水系统中。Giri等[33]研究了美国加州莫诺湖不同氧化还原区域的cbbL和cbbM分布和进化多样性,他们在所有样本中都发现了cbbL,而没有发现cbbM,并对获得的所有RubisCO基因序列进行进化分析,结果表明一些序列与依赖硫酸盐和硝酸盐生长的微生物有关,而另一些序列与砷酸盐还原菌和硫化物氧化菌相关。
陆地生态系统中针对cbbL和cbbM的功能基因研究已有大量报道,但陆地生态系统的异质性相对于海洋更加强烈,因而不同环境中的固碳微生物群落差异受环境异质性影响强烈。Guo等[34]利用qPCR、T-RFLP和克隆文库等技术对青藏高原高山草甸土壤中的cbbL基因进行检测和分析,发现cbbL丰度处于较高的水平,并且随海拔梯度的增加而增加,而群落结构的梯度变化则主要由土壤温度、土壤含水量、营养以及植被类型等因素驱动。Xiao等[18]利用克隆文库、T-RFLP、qPCR等技术分析了5种水稻田土壤中三类RubisCO基因cbbLG、cbbLR、cbbM的多样性,结果表明cbbLG序列多样性较低,并且大多来源于硫杆菌属(Thiobacillus)。
除了水生生态系统和陆地生态系统,一些研究者在极端环境中也发现了cbbL和cbbM的存在,但极端环境中检测到cbbL和cbbM在进化方面与已有序列有所差异,但也由此表明固碳微生物多样性极其丰富,并且在自然界中有着广泛的分布。Tahon等[35]通过构建南极洲山地土壤中的一系列功能基因的克隆文库,发现非蓝藻IC型cbbL基因多样性较高,表明厌氧光合作用的非蓝藻型自养微生物是主要初级生产者,它们在南极贫营养的高海拔陆地环境中有重要的生态作用。Elsaied等[36]通过克隆文库技术研究了3个深海热液口(Fryer、Pika和Suiyo)微生物群落的RubisCO大亚基编码基因cbbL的多样性,结果表明古菌cbbL只存在于Fryer和Pika中。IA和Ⅱ型RubisCO存在所有样品中,而IB型RubisCO没有从任何样品中检测到。Fryer和Pika样品中古菌cbbL多样性最高,Pika样本中的cbbL进化多样性最高,但Fryer和Pika古菌cbbL文库存在显著的差异。
2.1.2 还原型三羧酸循环:还原型三羧酸循环是另一种重要的CO2固定途径,该途径是1966年Evans提出的存在于少数光合紫色细菌和绿硫细菌自养固定CO2的途径。在还原型三羧酸循环中丙酮酸:铁氧化还原蛋白氧化还原酶(Pyruvate:Ferredoxin oxidoreductase)、2-氧戊二酸:铁氧还原蛋白氧化还原酶(2-Oxoglutarate:Ferredoxin oxidoreductase)和柠檬酸裂解酶(Citrate lyase)是3个最关键的酶,编码基因分别为porCDAB/nifJ、oorDABC和aclAB。
利用还原型三羧酸循环固定CO2的微生物类群通常存在于厌氧与富含硫磺酸的极端环境中,如深海热液喷口和热泉环境中,有关还原三羧酸循环的功能基因研究集中于高温厌氧环境中,并且可能与硫循环过程相关。Campbell等[37]对深海热液口样品中的aclB、porA、oorA、nifJ等功能基因构建克隆文库,发现深海热液口环境中ε-变形菌(Epsilon-Proteobacteria)是主要微生物类群,是利用还原型三羧酸循环固定CO2的主要微生物类群。Hall等[38]对美国黄石公园一个热泉的微生物功能类群进行了研究,通过对热泉样品中的16S rRNA基因和aclB、cbbM等功能基因构建克隆文库和qPCR分析,发现了产水菌科(Aquificae)以及α-变形菌纲、β-变形菌纲、γ-变形菌纲(Alpha-、Beta-、Gamma-Proteobacteria)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)和异常球菌属-栖热菌门(Deinococcus-Thermus)等微生物类群。检测表明热泉样品中存在还原型三羧酸循环的功能基因aclB,进化分析表明所有的aclB序列与Sulfurihydrogenibium相关。
2.1.3 还原乙酰辅酶A途径:还原乙酰辅酶A途径是在可利用氢的化能自养厌氧菌(产乙酸菌、硫酸盐原菌和产甲烷菌等)中发现的CO2固定途径。还原乙酰辅酶A途径(Reductive acetyl-CoA pathway)有两个分支,一个分支固定CO2后通过一系列反应生成甲基化合物;另一条分支固定CO2以后生成羰基化合物。一氧化碳脱氢酶(CODH)和乙酰辅酶A合成酶(ACS)是还原乙酰辅酶A途径中的关键酶,编码这两种酶的功能基因分别为acsA和acsB。
还原乙酰辅酶A过程中存在的一氧化碳脱氢酶(CODH)对氧气非常敏感,因此该途径需要在严格厌氧的条件下进行[39]。Gagen等[40]使用克隆文库的方法对牛瘤胃和尤金袋鼠前胃中的还原乙酰辅酶A功能基因acsB和fhs进行研究,表明acsB和fhs序列都集中在产乙酸的毛螺菌科(Lachnospiraceae)和梭菌科(Clostridiaceae)之间。此外,两个生态系统功能基因acsB和fhs的多样性差异表明了两个生态系统产乙酸菌群落有显著差异。Li等[41]使用GeoChip技术研究了玉米根际微生物群落的结构和代谢潜力,在根际土壤和非根际土壤中发现了CODH基因、Pcc/Acc和RubisCO基因,它们主要存在于红假单胞菌属(Rhodopseudomonas)和斯塔普氏菌属(Stappia)中,并且在根际土壤中相对信号强度更高,表明根际土壤固定CO2潜力更高。
2.1.4 3-羟基丙酸双循环:3-羟基丙酸双循环(3-Hydroxypropionate bicycle)是1989年由Holo[42]在绿色非硫细菌绿屈挠菌科(Chloroflexus aurantiacus)中首次发现,但一直到2009年才被Zarzycki和Fuchs等完全确立。由于该途径包含2个循环过程,因此被称之为双循环。第1个循环过程中,两分子的碳酸氢盐被固定形成乙醛酸盐;在第2个循环过程中乙醛酸盐和丙酰辅酶A歧化生成乙酰辅酶A和丙酮酸。3-羟基丙酸双循环过程中包括3个关键的酶:丙二酰辅酶A还原酶(Malonyl-CoA reductase)、丙酰辅酶A合成酶(Propionyl-CoA synthase)和苹果酰辅酶A/β-甲基苹果酰辅酶A/柠苹酰辅酶A裂解酶(Malyl-CoA/β-methylmalyl-CoA/citramalyl-CoA lyase)[43]。
3-羟基丙酸双循环完全确立较晚,目前对于其关键酶以及功能基因的研究相对仍然有待进一步探索,而相较于卡尔文等循环的宏基因组研究则又极少,目前已有研究主要针对丙酰辅酶A/乙酰辅酶A羧化酶(Propionyl-CoA/acetyl-CoA carboxylase)的功能基因Pcc/Acc开展。He等[44]利用高通量测序和GeoChip技术研究了雪松湾生态系统自然保护区土壤微生物群落结构和功能对CO2浓度持续升高的响应,在碳固定的功能变化过程中通过功能基因Pcc/Acc来鉴定3-羟基丙酸双循环途径。结果表明在CO2增加的情况下,Pcc/Acc和RubisCO基因的信号强度随之增强。Diaz等[45]研究了来自巴哈马群岛3种差异明显的环境中鲕粒岩的微生物功能基因多样性和代谢潜力,结果表明3种环境中都检测出了Pcc/Acc和RubisCO基因,并且Pcc/Acc基因普遍存在于研究的鲕粒岩种,丰度是RubisCO的3倍,表明在鲕粒岩中3-羟基丙酸双循环是主要的碳固定方式。
2.1.5 3-羟基丙酸循环/4-羟基丁酸循环:3-羟基丙酸/4-羟基丁酸酯循环与3-羟基丙酸循环存在一些类似的中间产物,但涉及的酶在系统发育方面不相关,且该循环存在于严格厌氧的环境中。已经发现的3-羟基丙酸/4-羟基丁酸循环存在于硫化叶菌目(Sulfolobales)、勤奋金属球菌(Metallosphaera sedula)等泉古菌以及一些奇古菌中,关键酶是乙酰辅酶A/丙酰辅酶A羧化酶(Acetyl-CoA/propionyl-CoA carboxylase)、4-羟丁酰辅酶A脱氢酶(4-Hydroxybutyryl-CoA dehydratase)和丙二酰辅酶A还原酶(malonyl-CoA reductase)。
目前针对3-羟基丙酸循环/4-羟基丁酸循环的微生物功能基因研究开展极少,已经报道的3-羟基丙酸/4-羟基丁酸循环功能基因主要包括accA和hcd[46-47]。如Bergauer等[48]研究了热带大西洋中固定CO2的微生物情况,使用定量PCR方法对accA和amoA两个功能基因以及16S rRNA基因进行调查,结果发现从海洋亚表层到氧气最少区域的accA基因丰度增加,与古菌amoA丰度增加一致,说明黑暗条件下的CO2固定途径可能在海洋微生物中广泛存在。4-羟基丁酸辅酶A脱水酶是自养型奇古菌中由功能基因hcd编码的一种关键的功能酶,Offre等[49]利用qPCR和DGGE技术对江口、深海及2个农业土壤(乙炔处理土壤和未经乙炔处理土壤)中的hcd基因进行研究,调查了利用3-羟基丙酸/4-羟基丁酸循环自养奇古菌的多样性和丰度,结果发现硝化反应随hcd基因丰度的增加而增加,另外硝化反应受乙炔抑制,证明了土壤中利用3-羟基丙酸/4-羟基丁酸循环的奇古菌是自养氨氧化菌。
2.2 微生物群落中甲烷代谢功能基因研究甲烷代谢分为甲烷合成和甲烷氧化两类,甲烷代谢的关键酶和功能基因如表 3所示。
| 甲烷代谢 Methane metabolism |
关键酶 Key enzyme |
功能基因 Functional gene |
| 甲烷生成 Methanogenesis |
Ⅰ型甲基辅酶M还原酶 | mcrA |
| Ⅱ型甲基辅酶M还原酶 | mrtA | |
| 甲烷氧化 Methane oxidation |
颗粒性甲烷单加氧酶 | pmoA |
| 可溶性甲烷单加氧酶 | mmoX |
自然界的产甲烷过程由严格厌氧的产甲烷古菌完成,已发现的产甲烷菌均属于广古门菌门(Euryarchaeota)。已知的产甲烷菌分为7个目:甲烷杆菌目(Methanobacteriales)、甲烷球菌目(Methanococcales)、甲烷微菌目(Methanomicrobiales)、甲烷八叠球菌目(Methanosarcinales)、甲烷火球菌目(Methanopyrales)、甲烷胞菌目(Methanocellales)和Methanoplasmatales。产甲烷古菌可将CO2、H2和乙酸及一些甲基型物质如甲酸、甲醇和甲胺等物质经历水解、酸化、产乙酸等过程之后最终产生甲烷。产甲烷过程的关键酶是甲基辅酶M还原酶MCR (Methyl coenzyme M reductase)。MCR包含MCR-Ⅰ和MCR-Ⅱ两种形式,分别由mcrBDCGA操纵子和mrtBDGA操纵子编码,其中MCR-Ⅰ存在于所有的产甲烷菌中;而MCR-Ⅱ仅存在于甲烷球菌目(Methanococcales)和甲烷杆菌目(Methanobacteriales)中[50-51]。MCR有一定保守性,而且至今为止没有发现MCR的水平基因转移,mcrA可作为功能基因来检测特定环境中产甲烷菌的多样性[52-53]。
目前在泥炭地、垃圾填埋场、水稻田及牛的瘤胃等环境中都已经通过功能基因mcrA检测出多样性较高的产甲烷古菌类群。Galand等[54]通过对3个不同泥炭地生态系统中产甲烷功能基因mcrA的多样性研究,探究了产甲烷古菌群落的多样性,结果表明中富营养型沼泽地CO2还原型产甲烷菌的比例最低,而贫营养沼泽地中该类产甲烷菌最丰富。Luton等[53]构建了英国埃塞克斯和萨默塞特垃圾填埋场mcrA功能基因的克隆文库,结果表明产甲烷菌群多样性远远高于之前的估计。Seo等[55]使用分子生物学手段基于mcrA、pmoA和一些反硝化功能基因获得了稻田土壤中产甲烷菌和甲烷氧化菌及反硝化细菌的群落构成,结果表明甲烷氧化菌对氧气浓度和土壤理化性质比较敏感,在土壤表层附近氧气浓度高的地方丰度最高。Denman等[56]研究了氯溴甲烷处理对牛瘤胃甲烷产生菌多样性的影响,分析了氯溴甲烷处理组牛瘤胃和控制组牛瘤胃的mcrA数量及多样性,结果表明甲烷短杆菌(Methanobrevibacter)是牛瘤胃中最主要的产甲烷菌,氯溴甲烷可干扰牛瘤胃中产甲烷菌群落构成而使牛瘤胃中甲烷排放减少。
2.2.2 甲烷氧化:甲烷氧化菌对于调节全球气候变暖有潜在作用,目前已知的好氧甲烷氧化菌主要来自变形菌门(Proteobacteria)和疣微菌门(Verrucomicrobia)。疣微菌门中的甲烷氧化菌主要分布在一些嗜热嗜酸的极端环境中[57];变形菌根据碳同化方式、磷脂脂肪酸及膜结构的不同分为Ⅰ型和Ⅱ型两种类型[58]。好氧甲烷氧化菌的分类如表 4所示。
| 门 Phylum |
纲 Class |
科 Family |
属 Genus |
| 变形菌门 Proteobacteria |
γ-Proteobacteria (Ⅰ型甲烷氧化菌) |
Methylococcaceae (甲基球菌科) |
Methylobacter Methylomicrobium Methylomonas Methylosarcina Methylococcus Methylocaldum Methylosphaera Methylosoma Methylomarinum Methylovulum Methyogaea Methylothermus Methylohalobium Crenothrix Clonothrix |
| α-Proteobacteria (Ⅱ型甲烷氧化菌) |
Methylocystaceae (甲基孢囊菌科) Beijerinckiaceae (拜叶林克氏菌科) |
Methylocystis Methylosinus Methylocapsa Methylocella Methyloferula |
|
| 疣微菌门 Verrucomicrobia |
Methylacidiphilum (属) |
甲烷单加氧酶(Methane monooxygenase)是甲烷氧化过程最关键的酶,分为两种类型:颗粒甲烷单加氧酶pMMO (Particulate methane monooxygenase)和可溶性甲烷单加氧酶sMMO (Soluble methane monooxygenase)。几乎所有已知甲烷氧化菌体内都存在pMMO,而sMMO通常存在于绝大多数Ⅱ型甲烷氧化菌和甲基暖菌属(Methylocaldum)中[61]。编码pMMO蛋白β亚基的pmoA基因目前已被广泛应用于检测环境样品中甲烷氧化菌群。此外,mmoX编码sMMO的α亚基,在检测环境中Methylocella和Methyloferula丰富的样品时具有比较重要的作用[62],因为在这两种甲烷氧化菌中不含pMMO,因此选择mmoX来检测环境中的这两类甲烷氧化菌。
对于甲烷氧化菌群的功能基因研究集中在水稻田、泥炭地、湿地、垃圾填埋场、石油和煤丰富的地区等环境中。Gupta等[63]通过基于pmoA和mmoX两种功能基因的DGGE结果结合稳定同位素的方法研究了北美两个理化性质差异较大的泥炭地之间甲烷氧化菌群落多样性,结果表明两地甲烷氧化菌群落进化多样性存在差异,但甲烷氧化速率无明显的差异。Yun等[64]对三江湿地中分别种植3种不同植物的沼泽土壤的甲烷氧化菌丰度和多样性进行了研究,通过Sanger测序、焦磷酸测序及qPCR等技术分析了土壤样品中的16S rRNA基因和pmoA基因,结果表明毛苔草湿地土壤中的pmoA基因丰度最高,乌拉苔草湿地土壤次之,小叶章湿地土壤最少。Kong等[65]对不同三氯乙烯(TCE)浓度作用下的垃圾填埋覆盖土(LCS)和废物处理生物覆盖土(WBS)的甲烷氧化能力和TCE降解效率进行了研究,并通过功能基因pmoA和mmoX获得了两类土壤中甲烷氧化菌的多样性和丰度。结果发现甲基暖菌(Methylocaldum)和甲基杆菌(Methylobacter)是WBS中的主要物种,而Ⅱ型甲烷氧化菌甲基孢囊菌(Methylocystis)是LCS中主要物种,且WBS的pmoA丰度比LCS高,WBS中甲烷氧化活性和TCE降解率比LCS更高。
2.3 微生物群落中碳降解功能基因的研究微生物作为生态系统食物网中的分解者,能够分泌多种酶降解动植物残体及其它存在的有机物,如一些天然多聚物淀粉、半纤维素、纤维素、木质素等(表 5)。这些天然多聚物汇集了自然界中大量的碳元素,除淀粉之外的几类都属于难降解的有机物。微生物将这些物质中储存的碳进行转化与迁移,进而加速碳元素的生物地化循环。微生物分解作用对于碳循环有重要的促进作用,对于难降解有机质的生物降解不仅有重要的生态学意义[66],也具有广泛的实际应用价值[67]。
| 天然碳化合物 Carbon compounds |
结构 Construction |
关键酶 Key enzymes |
| 淀粉 Starch |
葡糖糖残基通过α-1, 4-糖苷键首尾相连形成;支链通过α-1, 6-糖苷键连接形成的多聚体 | α-淀粉酶 β-淀粉酶 葡糖糖化酶 α-葡糖苷酶 异淀粉酶 支链淀粉酶 环糊精糖基转移酶 |
| 半纤维素 Hemicellulose |
由木糖、阿拉伯糖和半乳糖连接形成的不均一的多聚糖 | 木聚糖酶 β-甘露糖酶 α-L-阿拉伯糖苷酶 α-D-葡萄糖苷酸酶 β-木糖苷酶 |
| 纤维素 Cellulose |
葡萄糖通过β-1, 4-糖苷键连接而形成的均聚物 | 内切葡聚糖酶 外切纤维素酶 β-葡糖苷酶 |
| 几丁质 Chitin |
由N-乙酰-α-氨基葡糖胺通过β-1, 4-糖苷键连接形成的多聚体 | 外切几丁质酶 内切几丁质酶 |
| 木质素 Lignin |
苯丙烷通过醚键或碳碳单键连接而成的三维网状结构 | 木质素过氧化物酶 锰过氧化物酶 漆酶 |
淀粉是一种高分子的葡萄糖聚合物,包括直链淀粉和支链淀粉两种形态。淀粉水解酶主要包括α-淀粉酶、β-淀粉酶、葡糖糖化酶、α-葡糖苷酶、异淀粉酶、支链淀粉酶和环糊精糖基转移酶等,这些酶不仅存在于植物中,在微生物中也被检出[68-69]。
目前有关淀粉的微生物降解多集中于工业微生物的研究中,而有关自然环境中淀粉降解的群落微生物功能基因的研究报道在基因芯片技术方向应用较多。Yang等[70]通过GeoChip 4.0技术研究了青藏高原高山草甸沿海拔梯度微生物群落基因多样性的变化情况,结果发现碳循环中与淀粉降解相关的异支链淀粉酶和外切葡聚糖酶功能基因在高海拔地区丰度更加高。Paula等[71]利用GeoChip 4.0研究了土地使用方式变化对亚马逊雨林土壤中微生物功能多样性的影响,发现与淀粉降解相关的两类基因——支链淀粉酶基因和异支链淀粉酶基因,表明牧场与森林相比不稳定的碳化合物相对更加丰富。Eisenlord等[72]利用GeoChip 4.0技术研究了氮沉降减缓森林生态系统碳循环和增强碳储存中微生物的介导机制,结果发现氮沉降显著减少了淀粉等碳化合物解聚基因的多样性和丰度。
2.3.2 纤维素:纤维素结构复杂,其降解主要通过内切葡聚糖酶(EG)、外切纤维素酶(CBH)和β-葡糖苷酶(BG)这3种酶的协同作用实现[73]。外切酶可以水解纤维素结晶区,(CBH I)从纤维素链的还原端或(CBH Ⅱ)非还原端开始持续水解,释放纤维二糖;内切葡聚糖主要作用于纤维素的非结晶区,随机水解纤维素链中的糖苷键,把纤维素长链切断,转化成大量不同聚合度的纤维素短链,降低纤维素分子聚合度,使可供外切酶作用的纤维素链末端数增加;β-葡萄糖苷酶则主要水解纤维二糖和可溶性纤维寡糖,最终将纤维素转化为可利用的葡萄糖[74]。
目前报道的纤维素降解菌主要包括真菌[木霉属(Trichoderma)、青霉属(Penicillium)、曲霉属(Aspergillus)、白腐菌(White rot fungi)等]和细菌[纤维杆菌属(Fibrobacter)、纤维单胞属(Cellulomonas)等]两大类[75]。纤维素降解菌存在于堆肥、反刍动物瘤胃、家蚕肠道、白蚁肠道及一些厌氧反应器。Štursová等[76]利用真菌ITS基因和纤维素降解基因cbhI研究了欧洲云杉森林生态系统土壤中的纤维素降解真菌群落的多样性,结果表明大部分纤维素降解真菌都属于子囊菌门(Ascomycota)。可降解纤维素的担子菌门(Basidiomycota)主要是毛孢子菌属(Trichosporon)和隐球菌属(Cryptococcus)。Voříšková等[77]利用真菌ITS和外切纤维素酶cbhI基因研究了以无梗花栎为优势树种的温带森林的真菌群落在24个月里对森林凋落物的降解情况,担子菌门(Basidiomycota)尤其是酵母的相对丰度随着时间的增加而增加。
2.3.3 半纤维素:半纤维素与纤维素通常连在一起,构成植物细胞壁,不可溶且水解过程缓慢,其降解需要多种酶的联合作用。已经发现的半纤维素和纤维素的微生物降解途径可分为3种:(1) 好氧微生物分泌不同胞外酶协同降解半纤维素,这些酶将半纤维素多聚物完全降解成单糖或二糖而被周围微生物利用;(2) 厌氧细菌如梭状芽胞杆菌(Clostridia)通过酶复合体——纤维小体来降解纤维素和半纤维素;(3) 在好氧微生物(Cytophaga hutchinsonii)和厌氧微生物(Fibrobacter succinogenes)中通过细胞结合型非复合体来降解半纤维素和纤维素[78]。
半纤维素的降解酶主要包括木聚糖酶、β-甘露糖酶、α-L-阿拉伯糖苷酶、α-D-葡萄糖苷酸酶、β-木糖苷酶及一些半纤维素酯酶等[79-80]。木聚糖酶是最主要的半纤维素降解酶,在很多环境中已被发现,Wang等[81]通过针对GH10和GH11木聚糖酶基因克隆文库对泰山高山冻土的木聚糖酶基因的多样性进行研究,结果表明即使在高山冻土中木聚糖酶基因依然有非常高的多样性。Hayashi等[82]通过对人体肠道中5种编码木聚糖酶的基因xyn10A、xyn10B、xyn10C、xyn10D和xyn10E进行克隆文库研究,证明了人体肠道中木聚糖酶的多样性很高,肠道中不仅存在已被鉴定过的木聚糖酶,也有新的木聚糖酶Xyn10A被发现。
2.3.4 几丁质:几丁质是自然界产量次于纤维素的有机聚合物,是一种没有分支的链状β-1, 4-N-乙酰葡糖胺多聚物,主要存在于虾、蟹等甲壳纲动物外壳以及真菌和藻类细胞壁中。几丁质主要分为三类:α-、β-、γ-几丁质[83]。几丁质降解酶根据作用位点的不同可分为外切几丁质酶和内切几丁质酶两类,而外切几丁质酶又进一步分为壳二糖酶和乙酰葡萄糖胺糖苷酶[84]。
主要的细菌几丁质降解酶属于GH18,并且又被分为ChiA、ChiB和ChiC三个种类[85]。ChiA被广泛应用于研究陆地系统和水系统中几丁质水解微生物的多样性及分布。目前有关几丁质水解微生物酶和功能基因多样性的研究已经在多种环境中开展,Lian等[86]设计了针对几丁质酶基因GH18的ChiA的特异性引物,联合RFLP技术研究了太平洋多金属结核区的几丁质酶和几丁质降解微生物多样性,结果发现了18个几丁质酶基因且大部分编码的水解酶与沙雷氏菌属(Serratia)相关。Xiao等[87]通过PCR和RFLP等分子技术对南极洲Ardley岛湖底沉积岩的几丁质酶降解基因进行研究,结果发现几丁质酶降解基因丰度与湖底磷和总有机碳相关,几丁质降解酶基因拷贝数目和湖中企鹅粪便数量相关。Metcalfe等[88]通过几丁质水解酶基因ChiA来探究石灰与处理过的活性污泥施用下的高山草场几丁质水解细菌群落多样性,结果表明污泥应用增加几丁质水解酶活性,但却降低了其多样性,进一步分析表明放线菌门(Actinobacteria)在几丁质降解功能中起主要作用。
2.3.5 木质素:木质素是植物的重要成分,也是最难降解的天然多聚物之一。木质素是一类具有三维网状结构的苯丙烷多聚物,由几种不同的碳碳单键或者醚键连接大量苯丙烷形成。在自然界中存在3种基本结构:愈创木基型、紫丁香基型和对羟基苯基型结构。木质素不易水解,易通过氧化方式降解,主要涉及3种酶:木质素过氧化物酶(LiP)、锰过氧化物酶(MnP)和漆酶(LA)[89-90]。
木质素的降解依赖细菌群落和真菌群落的协同作用,但研究表明真菌起主要作用,真菌中的担子菌门如黄孢原毛平革菌(Phanerochaete chrysosporium)、云芝(Coliorus versicolor)、脉射菌(Phlebia radiate)和杏鲍菇(Pleurotus eryngii)等白腐真菌可降解木质素[91]。木质素降解功能基因的研究也已经得到应用,Stuardo等[92]为了检测木腐菌丰富土壤中的木质素降解菌,利用PCR和克隆文库手段研究木腐菌丰富土壤系统中的木质素过氧化物酶基因lip和锰过氧化物酶基因mnp,结果表明这种分子技术适合用来检测土壤中模式生物的木质素降解基因存在及表达差异,但并不适合研究木质素降解或者检测新lip和mnp基因。Su等[93]利用454焦磷酸测序和GeoChip 4.0技术研究了22年使用不同化肥处理的水稻田土壤中微生物群落成分和功能结构的变化,发现木质素降解功能基因丰度不随化学施肥变化,而木质素可能在维持土壤碳稳定性方面有重要作用。Zhang等[94]通过高通量测序和GeoChip技术研究了自然成熟林和自然次生林的土壤微生物多样性和代谢潜力,发现与木质素及其它一些碳化合物降解相关的基因信号强度在次生林中比自然成熟林显著高,表明了在两种森林生态系统中土壤微生物代谢能力存在显著差异。
3 结论与展望微生物群落对碳循环的响应和反馈是近年来生态学研究的热点问题之一,微生物不同的代谢途径和生物周转过程影响了碳的生物地球化学循环过程。微生物群落的作用会对环境中有机物质转化分解和积累造成直接影响,从而影响土壤肥力的发展和演化;微生物群落对环境因子的响应甚至会对全球范围碳循环、温室气体的排放及气候变化产生影响。因此碳循环过程的微生物调控机制、碳循环功能微生物类群对环境因子响应与反馈机制、碳元素与其它元素耦合循环的微生物联动机制以及功能微生物类群的种间关系等,都将是未来碳循环研究的重要内容[95]。
通过宏基因组学技术如DNA分子指纹图谱、qPCR、基因芯片和高通量测序等技术进行的碳循环功能基因分子生态学研究进展迅速,极大地扩展研究者们对微生物功能多样性以及物种多样性的了解,成为微生物群落研究的高效工具。碳循环功能基因的分子生态学研究揭示了环境中参与碳循环的微生物功能类群的多样性及物种和环境因子之间的相互作用关系,为进一步深入探究碳循环中微生物调控机制奠定了基础。但基于碳循环过程中不同功能基因的分子生态学研究进展并不一致,如产甲烷过程和甲烷氧化过程中微生物功能类群研究较多,功能基因研究较为成熟;而碳降解过程中由于酶系繁杂,很多化合物降解酶系的关键功能基因尚未清楚和统一;碳固定过程中因不同途径完全确立的时间不同,因此研究程度也有所差别,卡尔文循环、还原乙酰辅酶A途径及还原三羧酸循环发现较早,因而关键酶及编码基因研究较丰富和成熟,而另外3种碳固定途径因发现时间较晚,酶和相关基因研究并不完善。未来有关碳循环过程的功能基因分子生态学研究仍然依赖于更精确分子工具的发展。
近年来以高通量测序和基因芯片为代表的宏基因组学技术得到了广泛应用,同转录组学、蛋白组学和代谢组学一起,实现了单一过程向微生物群落水平研究的转变,从而使得科学家可以在更高更复杂的整体水平研究微生物群落的系统功能[95]。但高通量测序同时也带来一些问题,如海量数据的分析[9]、测序数据的真伪辨识、基于16S rRNA基因的物种分辨问题及其只能鉴定物种而无法直接鉴定微生物功能等问题都制约了碳循环微生物群落研究的发展。相比较而言,若针对微生物功能基因宏基因组进行研究,则目的明确,能够大幅减少测序数据量以及数据分析的工作量,且可直接鉴定环境中微生物群落的功能。因而未来碳循环微生物驱动机制的宏基因组学研究的核心问题将是由大规模海量测序转向功能导向的微生物组学研究。
| [1] | Bardgett RD, Freeman C, Ostle NJ. Microbial contributions to climate change through carbon cycle feedbacks[J]. The ISME Journal, 2008, 2(8) : 805–814. DOI:10.1038/ismej.2008.58 |
| [2] | Zhou JZ, Xue K, Xie JP, et al. Microbial mediation of carbon-cycle feedbacks to climate warming[J]. Nature Climate Change, 2012, 2(2) : 106–110. |
| [3] | Madigan MT, Martinko JM, Bender KS, et al. Brock Biology of Microorganisms[M]. San Francisco: Pearson Education, 2015: 633 . |
| [4] | Handelsman J, Rondon MR, Brady SF, et al. Molecular biological access to the chemistry of unknown soil microbes: A new frontier for natural products[J]. Chemistry & Biology, 1998, 5(10) : R245–R249. |
| [5] | Streit WR, Schmitz RA. Metagenomics-the key to the uncultured microbes[J]. Current Opinion in Microbiology, 2004, 7(5) : 492–498. DOI:10.1016/j.mib.2004.08.002 |
| [6] | Deng Y, Feng K, Wei ZY, et al. Recent studies and applications of metagenomics in environmental engineering[J]. Chinese Jounal of Environmental Engineering, 2016, 10(7) : 3373–3382. (in Chinese) 邓晔, 冯凯, 魏子艳, 等. 宏基因组学在环境工程领域的应用及研究进展[J]. 环境工程学报, 2016, 10(7) : 3373–3382. DOI:10.12030/j.cjee.201501219 |
| [7] | He JZ, Yuan CL, Shen JP, et al. Methods for and progress in research on soil metagenomics[J]. Acta Pedologica Sinica, 2012, 49(1) : 155–164. (in Chinese) 贺纪正, 袁超磊, 沈菊培, 等. 土壤宏基因组学研究方法与进展[J]. 土壤学报, 2012, 49(1) : 155–164. DOI:10.11766/trxb201103180094 |
| [8] | Zhou JZ, He ZL, Yang YF, et al. High-throughput metagenomic technologies for complex microbial community analysis: open and closed formats[J]. Mbio, 2015, 6(1) : e02288–14. |
| [9] | Wei ZY, Jin DC, Deng Y. Bioinformatics tools and applications in the study of environmental microbial metagenomics[J]. Microbiology China, 2015, 42(5) : 890–901. (in Chinese) 魏子艳, 金德才, 邓晔. 环境微生物宏基因组学研究中的生物信息学方法[J]. 微生物学通报, 2015, 42(5) : 890–901. |
| [10] | Vanguilder HD, Vrana KE, Freeman WM. Twenty-five years of quantitative PCR for gene expression analysis[J]. Biotechniques, 2008, 44(5) : 619–626. |
| [11] | Turnbaugh PJ, Gordon JI. The core gut microbiome, energy balance and obesity[J]. The Journal of Physiology, 2009, 587(17) : 4153–4158. DOI:10.1113/jphysiol.2009.174136 |
| [12] | Frias-Lopez J, Shi YM, Tyson GW, et al. Microbial community gene expression in ocean surface waters[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(10) : 3805–3810. DOI:10.1073/pnas.0708897105 |
| [13] | Wakelin SA, Colloff MJ, Harvey PR, et al. The effects of stubble retention and nitrogen application on soil microbial community structure and functional gene abundance under irrigated maize[J]. FEMS Microbiology Ecology, 2007, 59(3) : 661–670. DOI:10.1111/fem.2007.59.issue-3 |
| [14] | Yergeau E, Hogues H, Whyte LG, et al. The functional potential of high Arctic permafrost revealed by metagenomic sequencing, qPCR and microarray analyses[J]. The ISME Journal, 2010, 4(9) : 1206–1214. DOI:10.1038/ismej.2010.41 |
| [15] | Staats M, Braster M, Röling WFM. Molecular diversity and distribution of aromatic hydrocarbon-degrading anaerobes across a landfill leachate plume[J]. Environmental Microbiology, 2011, 13(5) : 1216–1227. DOI:10.1111/emi.2011.13.issue-5 |
| [16] | Wakelin SA, Colloff MJ, Kookana RS. Effect of wastewater treatment plant effluent on microbial function and community structure in the sediment of a freshwater stream with variable seasonal flow[J]. Applied and Environmental Microbiology, 2008, 74(9) : 2659–2668. DOI:10.1128/AEM.02348-07 |
| [17] | Fredriksson NJ, Hermansson M, Wilén BM. Impact of T-RFLP data analysis choices on assessments of microbial community structure and dynamics[J]. Bmc Bioinformatics, 2014. DOI:10.1186/S12859-014-0360-8 |
| [18] | Xiao KQ, Bao P, Bao QL, et al. Quantitative analyses of ribulose-1, 5-bisphosphate carboxylase/oxygenase (RubisCO) large-subunit genes (cbbL) in typical paddy soils[J]. FEMS Microbiology Ecology, 2014, 87(1) : 89–101. DOI:10.1111/1574-6941.12193 |
| [19] | Horz HP, Rotthauwe JH, Lukow T, et al. Identification of major subgroups of ammonia-oxidizing bacteria in environmental samples by T-RFLP analysis of amoA PCR products[J]. Journal of Microbiological Methods, 2000, 39(3) : 197–204. DOI:10.1016/S0167-7012(99)00119-0 |
| [20] | Muyzer G. DGGE/TGGE a method for identifying genes from natural ecosystems[J]. Current Opinion in Microbiology, 1999, 2(3) : 317–322. DOI:10.1016/S1369-5274(99)80055-1 |
| [21] | Wartiainen I, Eriksson T, Zheng WW, et al. Variation in the active diazotrophic community in rice paddy-nifH PCR-DGGE analysis of rhizosphere and bulk soil[J]. Applied Soil Ecology, 2008, 39(1) : 65–75. DOI:10.1016/j.apsoil.2007.11.008 |
| [22] | He ZL, Deng Y, Van Nostrand JD, et al. GeoChip 3.0 as a high-throughput tool for analyzing microbial community composition, structure and functional activity[J]. The ISME Jounal, 2010, 4(9) : 1167–1179. DOI:10.1038/ismej.2010.46 |
| [23] | Tu QC, Yu H, He ZL, et al. GeoChip 4: a functional gene-array-based high-throughput environmental technology for microbial community analysis[J]. Molecular Ecology Resources, 2014, 14(5) : 914–928. |
| [24] | Cong J, Liu XD, Lu H, et al. Analyses of the influencing factors of soil microbial functional gene diversity in tropical rainforest based on GeoChip 5.0[J]. Genomics Data, 2015, 5 : 397–398. DOI:10.1016/j.gdata.2015.07.010 |
| [25] | Tseng CH, Tang SL. Marine microbial metagenomics: from individual to the environment[J]. International journal of molecular sciences, 2014, 15(5) : 8878–8892. DOI:10.3390/ijms15058878 |
| [26] | Ravin NV, Mardanov AV, Skryabin KG. Metagenomics as a tool for the investigation of uncultured microorganisms[J]. Russian Journal of Genetics, 2015, 51(5) : 431–439. DOI:10.1134/S1022795415050063 |
| [27] | Yuan HC, Qin HL, Liu SL, et al. Advances in research of molecular ecology of carbon fixation microorganism[J]. Scientia Agricultura Sinica, 2011, 44(14) : 2951–2958. (in Chinese) 袁红朝, 秦红灵, 刘守龙, 等. 固碳微生物分子生态学研究[J]. 中国农业科学, 2011, 44(14) : 2951–2958. DOI:10.3864/j.issn.0578-1752.2011.14.012 |
| [28] | Hügler M, Sievert SM. Beyond the calvin cycle: autotrophic carbon fixation in the ocean[J]. Annual Review of Marine Science, 2011, 3(1) : 261–289. DOI:10.1146/annurev-marine-120709-142712 |
| [29] | Berg IA. Ecological aspects of the distribution of different autotrophic CO2 fixation pathways[J]. Applied and Environmental Microbiology, 2011, 77(6) : 1925–1936. DOI:10.1128/AEM.02473-10 |
| [30] | Kovaleva OL, Tourova TP, Muyzer G, et al. Diversity of RuBisCO and ATP citrate lyase genes in soda lake sediments[J]. FEMS Microbiology Ecology, 2011, 75(1) : 37–47. DOI:10.1111/fem.2010.75.issue-1 |
| [31] | Liu JF, Mbadinga S, Sun XB, et al. Microbial communities responsible for fixation of CO2 revealed by using mcrA, cbbM, cbbL, fthfs, fefe-hydrogenase genes as molecular biomarkers in petroleum reservoirs of different temperatures[J]. Biogeosciences Discussions, 2015, 12(2) : 1875–1906. DOI:10.5194/bgd-12-1875-2015 |
| [32] | Alfreider A, Vogt C, Hoffmann D, et al. Diversity of ribulose-1, 5-bisphosphate carboxylase/oxygenase large-subunit genes from groundwater and aquifer microorganisms[J]. Microbial Ecology, 2003, 45(4) : 317–328. DOI:10.1007/s00248-003-2004-9 |
| [33] | Giri B J, Bano N, Hollibaugh JT. Distribution of RuBisCO genotypes along a redox gradient in Mono Lake, California[J]. Applied and Environmental Microbiology, 2004, 70(6) : 3443–3448. DOI:10.1128/AEM.70.6.3443-3448.2004 |
| [34] | Guo GX, Kong WD, Liu JB, et al. Diversity and distribution of autotrophic microbial community along environmental gradients in grassland soils on the Tibetan Plateau[J]. Applied Microbiology and Biotechnology, 2015, 99(20) : 8765–8776. DOI:10.1007/s00253-015-6723-x |
| [35] | Tahon G, Tytgat B, Stragier P, et al. Analysis of cbbL, nifH, and pufLM in soils from the s r rondane mountains, antarctica, reveals a large diversity of autotrophic and phototrophic bacteria[J]. Microbial Ecology, 2016, 71(1) : 131–149. DOI:10.1007/s00248-015-0704-6 |
| [36] | Elsaied HE, Kimura H, Naganuma T. Composition of archaeal, bacterial, and eukaryal RuBisCO genotypes in three Western Pacific arc hydrothermal vent systems[J]. Extremophiles, 2007, 11(1) : 191–202. DOI:10.1007/s00792-006-0025-2 |
| [37] | Campbell BJ, Cary SC. Abundance of reverse tricarboxylic acid cycle genes in free-living microorganisms at deep-sea hydrothermal vents[J]. Applied and Environmental Microbiology, 2004, 70(10) : 6282–6289. DOI:10.1128/AEM.70.10.6282-6289.2004 |
| [38] | Hall JR, Mitchell KR, Jackson-Weaver O, et al. Molecular characterization of the diversity and distribution of a thermal spring microbial community by using rRNA and metabolic genes[J]. Applied and Environmental Microbiology, 2008, 74(15) : 4910–4922. DOI:10.1128/AEM.00233-08 |
| [39] | Techtmann SM, Lebedinsky AV, Colman AS, et al. Evidence for horizontal gene transfer of anaerobic carbon monoxide dehydrogenases[J]. Frontiers in Microbiology, 2012, 3 : 132. |
| [40] | Gagen EJ, Denman SE, Padmanabha J, et al. Functional gene analysis suggests different acetogen populations in the bovine rumen and tammar wallaby forestomach[J]. Applied and Environmental Microbiology, 2010, 76(23) : 7785–7795. DOI:10.1128/AEM.01679-10 |
| [41] | Li XZ, Rui JP, Xiong JB, et al. Functional potential of soil microbial communities in the maize rhizosphere[J]. PLoS One, 2014, 9(11) : e112609. DOI:10.1371/journal.pone.0112609 |
| [42] | Holo H. Chloroflexus aurantiacus secretes 3-Hydroxypropionate, a possible intermediate in the assimilation of CO2 and acetate[J]. Archives of microbiology, 1989, 151(3) : 252–256. DOI:10.1007/BF00413138 |
| [43] | Zarzycki J, Brecht V, Müller M, et al. Identifying the missing steps of the autotrophic 3-hydroxypropionate CO2 fixation cycle in Chloroflexus aurantiacus[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(50) : 21317–21322. DOI:10.1073/pnas.0908356106 |
| [44] | He ZL, Xu MY, Deng Y, et al. Metagenomic analysis reveals a marked divergence in the structure of belowground microbial communities at elevated CO2[J]. Ecology Letters, 2010, 13(5) : 564–575. DOI:10.1111/j.1461-0248.2010.01453.x |
| [45] | Diaz MR, Van Norstrand JD, Eberli GP, et al. Functional gene diversity of oolitic sands from Great Bahama Bank[J]. Geobiology, 2014, 12(3) : 231–249. DOI:10.1111/gbi.2014.12.issue-3 |
| [46] | Dang HY, Zhou HX, Yang JY, et al. Thaumarchaeotal signature gene distribution in sediments of the Northern South China sea: an indicator of the metabolic intersection of the marine carbon, nitrogen, and phosphorus cycles?[J]. Applied and Environmental Microbiology, 2013, 79(7) : 2137–2147. DOI:10.1128/AEM.03204-12 |
| [47] | Zhang LM, Offre PR, He JZ, et al. Autotrophic ammonia oxidation by soil thaumarchaea[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(40) : 17240–17245. DOI:10.1073/pnas.1004947107 |
| [48] | Bergauer K, Sintes E, Van Bleijswijk J, et al. Abundance and distribution of archaeal acetyl-CoA/propionyl-CoA carboxylase genes indicative for putatively chemoautotrophic Archaea in the tropical Atlantic's interior[J]. FEMS Microbiology Ecology, 2013, 84(3) : 461–473. DOI:10.1111/femsec.2013.84.issue-3 |
| [49] | Offre P, Nicol GW, Prosser JI. Community profiling and quantification of putative autotrophic thaumarchaeal communities in environmental samples[J]. Environmental Microbiology Reports, 2011, 3(2) : 245–253. DOI:10.1111/emi4.2011.3.issue-2 |
| [50] | Friedrich MW. Methyl-coenzyme M reductase genes: Unique functional markers for methanogenic and anaerobic methane-oxidizing Archaea[J]. Methods in Enzymology, 2005, 397 : 428–442. DOI:10.1016/S0076-6879(05)97026-2 |
| [51] | Lueders T, Chin KJ, Conrad R, et al. Molecular analyses of methyl-coenzyme M reductase α-subunit (mcrA) genes in rice field soil and enrichment cultures reveal the methanogenic phenotype of a novel archaeal lineage[J]. Environmental Microbiology, 2001, 3(3) : 194–204. DOI:10.1046/j.1462-2920.2001.00179.x |
| [52] | Kröber M, Bekel T, Diaz NN, et al. Phylogenetic characterization of a biogas plant microbial community integrating clone library 16S-rDNA sequences and metagenome sequence data obtained by 454-pyrosequencing[J]. Journal of Biotechnology, 2009, 142(1) : 38–49. DOI:10.1016/j.jbiotec.2009.02.010 |
| [53] | Luton PE, Wayne JM, Sharp RJ, et al. The mcrA gene as an alternative to 16S rRNA in the phylogenetic analysis of methanogen populations in landfill[J]. Microbiology, 2002, 148 : 3521–3530. DOI:10.1099/00221287-148-11-3521 |
| [54] | Galand PE, Fritze H, Conrad R, et al. Pathways for methanogenesis and diversity of methanogenic archaea in three boreal peatland ecosystems[J]. Applied and Environmental Microbiology, 2005, 71(4) : 2195–2198. DOI:10.1128/AEM.71.4.2195-2198.2005 |
| [55] | Seo J, Jang I, Gebauer G, et al. Abundance of methanogens, methanotrophic bacteria, and denitrifiers in rice paddy soils[J]. Wetlands, 2014, 34(2) : 213–223. DOI:10.1007/s13157-013-0477-y |
| [56] | Denman SE, Tomkins NW, Mcsweeney CS. Quantitation and diversity analysis of ruminal methanogenic populations in response to the antimethanogenic compound bromochloromethane[J]. FEMS Microbiology Ecology, 2007, 62(3) : 313–322. DOI:10.1111/fem.2007.62.issue-3 |
| [57] | Dunfield PF, Yuryev A, Senin P, et al. Methane oxidation by an extremely acidophilic bacterium of the phylum Verrucomicrobia[J]. Nature, 2007, 450(7171) : 879–882. DOI:10.1038/nature06411 |
| [58] | Semrau JD, Dispirito AA, Yoon S. Methanotrophs and copper[J]. FEMS Microbiology Reviews, 2010, 34(4) : 496–531. DOI:10.1111/j.1574-6976.2010.00212.x |
| [59] | Yun JL, Wang YF, Zhang HX. Ecology of aerobic methane oxidizing bacteria (methanotrophs)[J]. Acta Ecologica Sinica, 2013, 33(21) : 6774–6785. (in Chinese) 贠娟莉, 王艳芬, 张洪勋. 好氧甲烷氧化菌生态学研究进展[J]. 生态学报, 2013, 33(21) : 6774–6785. |
| [60] | Deng YC, Cui XY, Lüke C, et al. Aerobic methanotroph diversity in Riganqiao peatlands on the Qinghai-Tibetan Plateau[J]. Environmental Microbiology Reports, 2013, 5(4) : 566–574. DOI:10.1111/1758-2229.12046 |
| [61] | Henckel T, Friedrich M, Conrad R. Molecular analyses of the methane-oxidizing microbial community in rice field soil by targeting the genes of the 16S rRNA, particulate methane monooxygenase, and methanol dehydrogenase[J]. Applied and Environmental Microbiology, 1999, 65(5) : 1980–1990. |
| [62] | Horz HP, Yimga MT, Liesack W. Detection of methanotroph diversity on roots of submerged rice plants by molecular retrieval of pmoA, mmoX, mxaF, and 16S rRNA and ribosomal DNA, including pmoA-based terminal restriction fragment length polymorphism profiling[J]. Applied and Environmental Microbiology, 2001, 67(9) : 4177–4185. DOI:10.1128/AEM.67.9.4177-4185.2001 |
| [63] | Gupta V, Smemo KA, Yavitt JB, et al. Active methanotrophs in two contrasting North American peatland ecosystems revealed using DNA-SIP[J]. Microbial Ecology, 2012, 63(2) : 438–445. DOI:10.1007/s00248-011-9902-z |
| [64] | Yun JL, Zhang HX, Deng YC, et al. Aerobic methanotroph diversity in Sanjiang wetland, Northeast China[J]. Microbial Ecology, 2015, 69(3) : 567–576. DOI:10.1007/s00248-014-0506-2 |
| [65] | Kong JY, Bai Y, Su Y, et al. Effects of trichloroethylene on community structure and activity of methanotrophs in landfill cover soils[J]. Soil Biology and Biochemistry, 2014, 78 : 118–127. DOI:10.1016/j.soilbio.2014.07.018 |
| [66] | Ljungdahl LG, Eriksson KE. Ecology of microbial cellulose degradation[A]//Marshall KC. Advances in Microbial Ecology[M]. New York: Springer, 1985, 8: 237-299 |
| [67] | Rubin EM. Genomics of cellulosic biofuels[J]. Nature, 2008, 454(7206) : 841–845. DOI:10.1038/nature07190 |
| [68] | Bernfeld P. Enzymes of starch degradation and synthesis[A]// Nord FF. Advances in Enzymology and Related Areas of Molecular Biology[M]. Bronx, New York: Interscience Publishers, Inc., 1951, 12: 379-428 |
| [69] | Bertoldo C, Antranikian G. Starch-hydrolyzing enzymes from thermophilic archaea and bacteria[J]. Current opinion in chemical biology, 2002, 6(2) : 151–160. DOI:10.1016/S1367-5931(02)00311-3 |
| [70] | Yang YF, Gao Y, Wang SP, et al. The microbial gene diversity along an elevation gradient of the Tibetan grassland[J]. The ISME Journal, 2014, 8(2) : 430–440. DOI:10.1038/ismej.2013.146 |
| [71] | Paula FS, Rodrigues JLM, Zhou JZ, et al. Land use change alters functional gene diversity, composition and abundance in Amazon forest soil microbial communities[J]. Molecular Ecology, 2014, 23(12) : 2988–2999. DOI:10.1111/mec.12786 |
| [72] | Eisenlord SD, Freedman Z, Zak DR, et al. Microbial mechanisms mediating increased soil C storage under elevated atmospheric n deposition[J]. Applied and Environmental Microbiology, 2013, 79(4) : 1191–1199. DOI:10.1128/AEM.03156-12 |
| [73] | van den Brink J, de Vries RP. Fungal enzyme sets for plant polysaccharide degradation[J]. Applied microbiology and biotechnology, 2011, 91(6) : 1477–1492. DOI:10.1007/s00253-011-3473-2 |
| [74] | Fang X, Qin YQ, Li XZ, et al. Rrogress on cellulase and enzymatic hydrolysis of lignocellulosic biomass[J]. Chinese Journal of Biotechnology, 2010, 26(7) : 864–869. (in Chinese) 方诩, 秦玉琪, 李雪芝, 等. 纤维素酶与木质纤维素生物降解转化的研究进展[J]. 生物工程学报, 2010, 26(7) : 864–869. |
| [75] | Beguin P. Molecular biology of cellulose degradation[J]. Annual Review in Microbiology, 1990, 44(1) : 219–248. DOI:10.1146/annurev.mi.44.100190.001251 |
| [76] | Štursová M, Žifčáková L, Leigh MB, et al. Cellulose utilization in forest litter and soil: identification of bacterial and fungal decomposers[J]. FEMS Microbiology Ecology, 2012, 80(3) : 735–746. DOI:10.1111/fem.2012.80.issue-3 |
| [77] | Voříšková J, Baldrian P. Fungal community on decomposing leaf litter undergoes rapid successional changes[J]. The ISME Journal, 2013, 7(3) : 477–486. DOI:10.1038/ismej.2012.116 |
| [78] | Wilson DB. Three microbial strategies for plant cell wall degradation[J]. Annals of the New York Academy of Sciences, 2008, 1125(1) : 289–297. DOI:10.1196/annals.1419.026 |
| [79] | Pérez J, Muñoz-Dorado J, De La Rubia T, et al. Biodegradation and biological treatments of cellulose, hemicellulose and lignin: an overview[J]. International Microbiology, 2002, 5(2) : 53–63. DOI:10.1007/s10123-002-0062-3 |
| [80] | Shallom D, Shoham Y. Microbial hemicellulases[J]. Current Opinion in Microbiology, 2003, 6(3) : 219–228. DOI:10.1016/S1369-5274(03)00056-0 |
| [81] | Wang GZ, Wang YR, Yang PL, et al. Molecular detection and diversity of xylanase genes in alpine tundra soil[J]. Applied Microbiology and Biotechnology, 2010, 87(4) : 1383–1393. DOI:10.1007/s00253-010-2564-9 |
| [82] | Hayashi H, Abe T, Sakamoto M, et al. Direct cloning of genes encoding novel xylanases from the human gut[J]. Canadian Journal of Microbiology, 2005, 51(3) : 251–259. DOI:10.1139/w04-136 |
| [83] | Cabib E. The synthesis and degradation of chitin[A]//Meister A. Advances in Enzymology and Related Areas of Molecular Biology[M]. New York: Wiley, 1987, 59: 59-101 |
| [84] | Souza CP, Almeida BC, Colwell RR, et al. The importance of chitin in the marine environment[J]. Marine Biotechnology, 2011, 13(5) : 823–830. DOI:10.1007/s10126-011-9388-1 |
| [85] | Krsek M, Wellington EMH. Assessment of chitin decomposer diversity within an upland grassland[J]. Antonie Van Leeuwenhoek, 2001, 79(3/4) : 261–267. DOI:10.1023/A:1012043401168 |
| [86] | Lian MZ, Lin S, Zeng RY. Chitinase gene diversity at a deep sea station of the east Pacific nodule province[J]. Extremophiles, 2007, 11(3) : 463–467. DOI:10.1007/s00792-006-0057-7 |
| [87] | Xiao X, Yin XB, Lin H, et al. Chitinase genes in lake sediments of Ardley Island, Antarctica[J]. Applied and Environmental Microbiology, 2005, 71(12) : 7904–7909. DOI:10.1128/AEM.71.12.7904-7909.2005 |
| [88] | Metcalfe AC, Krsek M, Gooday GW, et al. Molecular analysis of a bacterial chitinolytic community in an upland pasture[J]. Applied and Environmental Microbiology, 2002, 68(10) : 5042–5050. DOI:10.1128/AEM.68.10.5042-5050.2002 |
| [89] | Jeffries TW. Biodegradation of lignin and hemicelluloses[A]// Ratledge C. Biochemistry of Microbial Degradation[M]. Netherlands: Springer, 1994: 233-277 |
| [90] | Dong XQ, Yuan HL, Gao TG. Progress in studies of ligninolytic enzymes and genes[J]. Biotechnology Bulletin, 2014(11) : 62–72. (in Chinese) 董秀芹, 袁红莉, 高同国. 木质素降解酶及相关基因研究进展[J]. 生物技术通报, 2014(11) : 62–72. |
| [91] | Higuchi T. Microbial degradation of lignin: Role of lignin peroxidase, manganese peroxidase, and laccase[J]. Proceedings of the Japan Academy, Series B, 2004, 80(5) : 204–214. DOI:10.2183/pjab.80.204 |
| [92] | Stuardo M, Vásquez M, Vicuña R, et al. Molecular approach for analysis of model fungal genes encoding ligninolytic peroxidases in wood-decaying soil systems[J]. Letters in Applied Microbiology, 2004, 38(1) : 43–49. DOI:10.1046/j.1472-765X.2003.01442.x |
| [93] | Su JQ, Ding LJ, Xue K, et al. Long-term balanced fertilization increases the soil microbial functional diversity in a phosphorus-limited paddy soil[J]. Molecular Ecology, 2015, 24(1) : 136–150. DOI:10.1111/mec.13010 |
| [94] | Zhang YG, Cong J, Lu H, et al. An Integrated Study to Analyze Soil Microbial Community Structure and Metabolic Potential in Two Forest Types[J]. PLoS One, 2014, 9(4) : e93773. DOI:10.1371/journal.pone.0093773 |
| [95] | National Natural Science Foundation of China, Chinese Acedemy of Sciences. Soil Biolog[M]. Beijing: Science Press, 2016: 45 -53. (in Chinese) 国家自然科学基金委员会, 中国科学院. 土壤生物学[M]. 北京: 科学出版社, 2016: 45 -53. |
 2017, Vol. 44
2017, Vol. 44




