扩展功能
文章信息
- 伍朝亚, 孟宪刚, 李岩, 曲慧敏, 解志红
- Wu Chao-Ya, Meng Xian-Gang, Li Yan, Qu Hui-Min, Xie Zhi-Hong
- 渤海沉积物产脂肪酶细菌的筛选及其多样性分析
- Screening and diversity of lipase-producing bacteria in the sediments of Bohai Sea
- 微生物学通报, 2017, 44(7): 1655-1667
- Microbiology China, 2017, 44(7): 1655-1667
- DOI: 10.13344/j.microbiol.china.170034
-
文章历史
- 收稿日期: 2017-01-13
- 接受日期: 2017-05-16
- 优先数字出版日期(www.cnki.net): 2017-05-25
2. 中国科学院烟台海岸带研究所 山东 烟台 264003
2. Yantai Institute of Coastal Zone Research Chinese Academy of Sciences, Yantai, Shandong 264003, China
脂肪酶是甘油酯水解酶[1],隶属于羧基酯水解酶类,能够将甘油三酯水解成甘油和脂肪酸,广泛存在于动物、植物和微生物中[2-3]。动植物体内的脂肪酶含量少且活性低,使用受限[4]。微生物脂肪酶的种类多、来源广,具有比动植物脂肪酶更广的pH、温度作用范围和底物的专一性类型,以及较高的催化活性和稳定性[5],是当代酶工业最受瞩目的酶种之一[6]。脂肪酶已被广泛应用于食品、石油、化工、洗涤行业、日用化学合成、生物制药、造纸、废物处理[7-9]、生物柴油和皮革生产等行业[10],是重要的工业用酶。目前用于脂肪酶生产的微生物大多来源于陆地或淡水环境,而随着工业发展其不能满足对脂肪酶不同特征需求的多样性[11]。地球表面的2/3以上由海洋沉积物覆盖,这些沉积物中存在着大量的产多种酶及代谢产物的微生物。因此,寻找并开发海洋来源的产脂肪酶微生物,利于发现新型脂肪酶产生菌种并为酶工业的大量生产提供丰富的菌群资源。
海洋微生物作为产酶资源,正在成为一个新的研究热点。目前,国内外关于脂肪酶产生菌的报道多来自极地[12]和深海海泥中[13],脂肪酶微生物大都属于假单胞菌(Pseudomonas)、气单胞菌属(Aeromonas)[14-15]等,对于浅海沉积物中产脂肪酶微生物则关注较少。本研究根据渤海独特的地理、气候及环境特征,从其海底表面沉积物中筛选产脂肪酶的菌株,以期系统发掘海洋微生物资源,解决工业生产脂肪酶来源的问题。渤海位于中国大陆东部的最北端,是一个半封闭的陆缘浅海,平均水深小于20 m,海域面积77 284 km2,被富集的环渤海经济圈工业区包围,是我国重要的经济鱼虾蟹生产海洋系统[16]。渤海三面环陆,其海水交换能力较差,沉积物类型大部分为泥沙和软泥质,受人类活动影响较大,而水体环境的改变会引起渤海沉积物生物多样性的变化[17]。基于此,本文研究了渤海沉积物中可培养产脂肪酶细菌,并通过基于16S rRNA基因序列的系统发育关系分析,揭示这些细菌的分类地位和遗传多样性;利用对硝基苯酚法测定胞外脂肪酶活性,筛选出高效产脂肪酶菌株,增加人们对渤海生态系统中产脂肪酶菌多样性的认识,为海洋产脂肪酶微生物的挖掘提供菌群资源,对于开发海洋功能性微生物资源具有一定的借鉴作用。
1 材料与方法 1.1 材料 1.1.1 主要试剂和仪器:酵母提取物、蛋白胨,美国Thermo Fisher Scientific公司;对硝基苯酚棕榈酸酯,美国Sigma-Aldrich公司;细菌基因组DNA提取试剂盒,天根生化科技有限公司。PCR仪,杭州晶格科学仪器有限公司;电泳仪,北京市六一仪器厂;凝胶成像仪,北京友华照钦医疗器械有限公司Bio-Rad生命科学授权代理商;恒温培养摇床、电热恒温水槽,上海一恒科技有限公司;小型高速冷冻离心机,德国Thermo Fisher公司;酶标仪,深圳市华德维康科技有限公司。
1.1.2 培养基:初筛培养基(吐温-80筛选培养基,g/L):葡萄糖20.0,酵母提取物5.0,蛋白胨5.0,氯化钙1.0,吐温-80 10.0,琼脂15.0,天然陈海水1.0,pH自然,1.0×105 Pa灭菌20 min。三丁酸甘油酯培养基(g/L):三丁酸甘油酯10.00,NaCl 39.45,KNO3 1.00,MgSO4·7H2O 10.45,MgCl2·6H2O 6.90,KCl 2.10,CaCl2·6H2O 1.50,K2HPO4 0.05,NaHCO3 0.02,NaBr 0.05,痕量金属离子溶液2.00 mL,琼脂粉18.00,天然陈海水1.00,pH 7.2-7.5,1.0×105 Pa灭菌30 min。痕量金属离子溶液(g/L):EDTA·2Na 5.20,FeCl2·4H2O 1.50,CoCl2·6H2O 0.19,MgCl2·4H2O 0.10,ZnCl2 0.07,H3BO3 0.06,NaMoO4·2H2O 0.04,CuCl2·2H2O 0.02,柠檬酸铁铵3.00。2216E培养基(g/L):酵母提取物2.5,蛋白胨5.0,琼脂15.0 (固体培养基),天然陈海水1.0,pH自然,1.0×105 Pa灭菌20 min。发酵培养基(g/L):葡萄糖20.0,酵母提取物5.0,蛋白胨5.0,氯化钙1.0,吐温-80 10.0,天然陈海水1.0,pH自然,1.0×105 Pa灭菌20 min。
1.2 样品采集沉积物样品采集自2014年9月中国科学院烟台海岸带研究所渤海航次,去除表面可能被污染的样品,收集没有被污染的表层沉积样品。样品通过无菌操作储存在无菌自封袋内,表层沉积物温度和pH值原位测定。采样船上用干冰低温保存样品,返回实验之后,用于菌株筛选的样品于-4 ℃保存。
1.3 产胞外脂肪酶细菌的选择性分离及保藏称取1 g沉积物样品,用无菌海水以10倍稀释梯度稀释至10–6,混匀后取100 µL (10–2-10–6)的稀释样品涂布于吐温-80筛选培养基平板上,置于20 ℃培养,待脂肪酶筛选培养基上出现明显白色沉淀圈时,根据菌落形态、颜色、大小等表型差异,选择能够在吐温-80为底物的培养基中产生沉淀圈的不同形态的菌落,在新的筛选培养基平板上划线纯化,之后使用2216E液体培养基扩大培养至对数期,取1 mL菌液,加入终浓度为20%的甘油,保藏于-80 ℃[18-19]。
1.4 菌株基因组DNA的提取将所有纯化后的菌株接种至2216E液体培养基中,待生长至对数期后收集菌体,使用细菌基因组提取试剂盒提取所有菌株的基因组DNA,提取方法及流程按照试剂盒要求操作。提取后用NanoDrop检测DNA纯度,检测合格的DNA样品于-20 ℃冷冻保存待用。
1.5 16S rRNA基因的扩增及序列测定选用细菌16S rRNA基因通用引物27F/1492R扩增所有菌株的16S rRNA基因序列[11],引物序列、PCR反应体系及条件参照参考文献[18],PCR扩增产物经1.0%琼脂糖凝胶电泳检测合格后送北京奥科鼎盛生物科技有限公司测序[20-21]。测序得到的序列提交至NCBI数据库,登录号为:KY244042-KY244060及KY272021-KY272052。
1.6 菌株系统发育分析将测得的16S rRNA序列在NCBI进行BLAST程序在线比对,并从GenBank中下载相似性最高近缘物种的模式菌株序列,使用MEGA 5.0的Clustal程序比对后,并选择邻接法(Neighbor-Joining method)构建系统进化树,选择Kimura two-parameter model,自展值设定为1 000[22-23],并通过MEGA 5.0计算所有菌株之间及菌株与模式菌株之间的相似性。
1.7 脂肪酶酶活的测定 1.7.1 对硝基苯酚标准曲线的制作:称取对硝基苯酚(p-Nitrophenol,pNP)溶解于5 mL异丙醇中,使其终浓度为1 mmol/L,再用50 mmol/L Tris-HCl (pH 8.0) 配成不同浓度的对硝基苯酚(pNP)溶液,分别测定405 nm下吸光度,绘制吸光度-浓度关系标准曲线。
1.7.2 对硝基苯酚棕榈酸酯(p-nitrophenylpal-mitate,PNPP)底物溶液的配制:配制溶液A (30 mg PNPP溶解于10 mL异丙醇中)和溶液B (0.1 g阿拉伯树胶和0.4 mL Triton X-100溶解于90 mL 50 mmol/L Tris-HCl pH 8.0缓冲液中),将溶液A和溶液B混合搅拌,直到完全溶解[24]。
1.7.3 脂肪酶活力的测定:使用对硝基苯酚法测定脂肪酶酶活。分别将活化后的菌体接种到发酵培养基中,20 ℃、180 r/min摇床培养,2 d后取适量发酵液12 000 r/min离心15 min,上清即为粗酶液[25],向300 µL粗酶液中加入60 ℃预热的700 µL PNPP底物溶液,60 ℃反应20 min,于405 nm下测吸光度。脂肪酶活力单位(U)定义为:在以上条件下,每分钟释放1 µmol对硝基苯酚(pNP)所需要的酶量为1 U。
1.7.4 菌株在吐温-80平板上沉淀圈的测定:将各菌株在2216E固体活化培养基上进行划线,倒置于28 ℃培养24 h,然后挑取单菌落点种至吐温-80平板上,20 ℃培养6 d,观察脂肪酶沉淀圈的大小,分别测量沉淀圈直径和菌落直径的大小,并计算其比值。
1.7.5 菌株在三丁酸甘油酯平板上水解圈的测定:将各菌株在2216E固体活化培养基上进行划线,倒置于28 ℃培养24 h,然后挑取单菌落点种至三丁酸甘油酯平板上,25 ℃培养3 d,观察脂肪酶水解圈的大小,分别测量水解圈直径和菌落直径的大小,并计算其比值。
1.7.6 模式菌株Marinobacter sp. EF10[5]的阳性对照实验:为了突出本实验筛选脂肪酶菌株在酶活力上的优势,在实验中增加了阳性对照实验,选取的阳性对照模式菌株Marinobacter sp. EF10[5]是分离自南大西洋深海沉积物中的一株产脂肪酶细菌,由哈尔滨工业大学威海分校海洋学院实验室提供。分别利用对硝基苯酚法和吐温-80平板沉淀圈法对模式菌株Marinobacter sp. EF10[5]进行酶活力测定。
1.7.7 温度对产脂肪酶菌株70623的酶活影响:以对硝基苯酚棕榈酸酯(PNPP)为底物,将产脂肪酶菌株70623粗酶液置于20-90 ℃温度梯度,以10 ℃为间隔测酶活,确定不同反应温度对酶活力的影响。
2 结果与分析 2.1 渤海海底表层样品的特点8个站点海底表层沉积物样品的采集地点、海水深度、温度和pH如图 1和表 1所示。8个站点中水深最深的是N4 (36 m),从表 1可以看出温度与水深有关系,水深越深温度越低,最低温度为10.66 ℃,各样品的pH均是中性偏碱,最大pH为8.19。

|
| 图 1 渤海沉积物位点分布图 Figure 1 Geographic location of sampled sediment stations in Bohai Sea |
|
|
| 站点 Station |
站点坐标 Station location (E, N) |
深度 Depth (m) |
温度 Temperature (℃) |
pH |
| BH25 | 117.95°, 38.95° | 8.10 | 22.18 | 8.19 |
| BH12 | 117.95°, 38.55° | 9.90 | 21.62 | 8.17 |
| BH8 | 118.55°, 38.35° | 14.80 | 19.91 | 8.09 |
| N4 | 120.83°, 39.21° | 36.00 | 10.66 | 7.92 |
| R6 | 120.63°, 38.23° | 25.00 | 16.83 | 7.98 |
| P2 | 119.49°, 37.96° | 15.00 | 19.66 | 8.07 |
| PLB1 | 120.15°, 38.36° | 15.00 | 17.12 | 7.98 |
| E6 | 121.50°, 37.70° | 18.00 | 19.50 | 8.05 |
每个沉积物样品取3个重复,分菌前将3个样品合并再称量样品进行分离。在初筛培养基稀释度为10-1-10-4的涂布平板上培养后长出大量的菌落,根据菌落形态、颜色、大小等表型差异,选择能以吐温-80为底物培养基中产生脂肪酶生成脂肪酸钙盐沉淀圈的不同菌落,经分离纯化后得到51株产脂肪酶细菌。
2.3 产脂肪酶细菌多样性将测序得到的菌株的16S rRNA基因序列在NCBI进行BLAST程序在线比对后,可以得出细菌的分类,结果显示,51株菌分别隶属于Bacteroidetes、Proteobacteria和Firmicutes三个门的8个属。除了菌株70642属于Bacteroidetes的Salegentibacter,菌株70201、70204、70208、70212、70617、70618、70646和菌株70619、70634属于Firmicutes的Bacillus和Staphylococcus之外,其余的菌株都隶属于Proteobacteria的Pseudoalteromonas、Marinobacter、Sulfitobacter、Photobacterium、Shewanella,其中Pseudoalteromonas (35.2%)、Marinobacter (23.5%)和Sulfitobacter (17.6%)是优势菌群。对分离自各个站点菌株的分类地位统计如图 2所示,18株属于Pseudoalteromonas的菌株在7个不同站点(除站点E6外)的沉积物样品中均有分布,分离自站点BH8、N4、E6的细菌各只有一株,分别属于Shewanella、Photobacterium、Salegentibacter属。在不同站点和不同水深的沉积物里的细菌数量是不同的,在较深的站点P2 (15 m)和PLB1 (15 m),产脂肪酶细菌的物种多样性较为丰富,其中站点P2的14株属于Bacillus (1株)、Staphylococcus (2株)、Sulfitobacter (3株)、Marinobacter (3株)、Pseudoalteromonas (5株),在所有站点中细菌物种多样性最丰富,站点PLB1有11株,菌株数量仅次于站点P2,其菌株分布于Bacillus、Sulfitobacter、Marinobacter、Pseudoalteromonas四个属。在最浅的站点BH25 (8.1 m)和最深的站点N4 (36 m),主要的产脂肪酶细菌属是Pseudoalteromonas,另外各有一株属于Bacillus和Photobacterium。分离自站点E6的6株分布于Salegentibacter、Sulfitobacter、Marinobacter三个属,站点R6的4株属于Bacillus、Marinobacter、Pseudoalteromonas三个属,站点BH8的4株属于Bacillus、Shewanella、Pseudoalteromonas三个属,站点BH12菌株物种多样性最小,其菌株分布于两个属:Marinobacter和Pseudoalteromonas。

|
| 图 2 渤海沉积物中的产脂肪酶细菌的百分比丰度 Figure 2 Relative percentage abundances of the phylogenetic groups of cultivable lipase-producing bacteria recovered from sediment samples of Bohai Sea |
|
|
基于菌株的16S rRNA基因序列构建的系统发育树如图 3所示,共有18株菌属于Pseudoalteromonas,其中11株之间相似性为100%,它们与Pseudoalteromonas halopanktis LMG 2852T的相似性最高为100%;70206和70207与Pseudoalteromonas spongiae JCM 12884T相似性为100%,70214与70206和70207进化关系非常亲近,处于同一分支上,与Pseudoalteromonas spongiae JCM 12884T相似性为99.9%;70211与Pseudoalteromonas distincta KMM 638T相似性为100%;70203与Pseudoalteromonas prydzensis ACAM 620T在进化树上聚为一个小分支,其相似性为99.6%;这些Pseudoalteromonas属的细菌进化关系非常接近,形成了最大的细菌类群。70609与Photobacterium angustum 68T相似性为99.8%;70202与Shewanella aquimarn SW-120T相似性为100%;有12株属于Marinobacter属,它们的进化关系接近,形成了Marinobacter sediminum R65T和Marinobacte rguineae M3BT、Marinobacter lipolyticus SM19T这三个细菌种的姊妹群;有9株属于Sulfitobacter属的细菌的进化距离很近,与Sulfitobacte rpontiacus ChLG10T的相似性较高,为99.8%。细菌70634和70619与Staphylococcus epidermidis LMG 10474T相似性为100%;属于Salegentibacter的细菌70642与Salegentibacter mishustinae KMM 6049T相似性最高为99.9%;属于Bacillus属的细菌有7株,其进化关系接近,其中70204与Bacillus simplex LMG 1160T相似性为99.8%;70201和70208与Bacillus malacitensis CR-95T相似性为100%;70646与Bacillus aquimaris TF-12T相似性最高为99.4%;70212与Bacillus megaterium NBRCT相似性最高为99.9%; 70617与70618相似性为100%,其与Bacillus halosaccharovorans E33T相似性最高为99.8%。

|
| 图 3 渤海沉积物中产脂肪酶细菌的16S rRNA基因NJ系统发育树 Figure 3 Neighbor-Joining tree of the lipase-producing bacteria isolated from eight sampled sediment stations in the Bohai Sea based on their 16S rRNA gene sequences 注:粗体是本文筛选的产脂肪酶菌株;序列的登录号位于圆括号内;系统发育树分支点处的数字表示置信度( > 50%);标尺表示100个核苷酸中有5个被替换. Note: The boldfaced strains are lipase-producing bacteria isolated in this study; The accession number is shown in parenthesis; Numbers at the branch points indicated the bootstrap values ( > 50%); The scale bar cooresponds to 0.05 substitution per nucleotide position. |
|
|
根据方法制作对硝基苯酚标准曲线,得到标准曲线方程为:y=0.022 5x+0.056 5;对51株菌进行液体发酵培养,通过对硝基苯酚法测得粗酶液与PNPP底物作用后在405 nm下的吸光度,再通过标准曲线方程换算酶活值,如表 2所示。筛选到菌株的酶活在2.6-42.4 U/mL的范围内,多数Pseudoalteromonas和Marinobacter属细菌的酶活相对较高,酶活值超过了10 U/mL,Pseudoalteromonas属细菌70621的酶活值为21.93 U/mL,是该属细菌酶活最高菌株;Marinobacter属的两株细菌70604、70623的酶活分别为37.23 U/mL和42.4 U/mL,是所有筛选到的产脂肪酶细菌中酶活值较高和最高的菌株。其它属的菌株酶活相对较低,其酶活值都低于10 U/mL,其中,Staphylococcus属的细菌酶活最低,最低酶活值为2.6 U/mL。
| 门 Phylum |
属 Genus |
菌株 Strain |
登录号 Accession number |
酶活 Enzyme activity (U/mL) |
沉淀圈直径/菌落直径P/C | 水解圈直径/菌落直径H/C |
| Proteobacteria | Pseudoalteromonas | 70203 | KY244044 | 5.35 | 3.14 | 2.00 |
| 70206 | KY244046 | 5.47 | 3.37 | 1.19 | ||
| 70207 | KY244047 | 7.05 | 3.38 | 1.60 | ||
| 70210 | KY244050 | 12.01 | 5.80 | 1.67 | ||
| 70211 | KY244051 | 8.60 | 2.67 | 2.00 | ||
| 70213 | KY244053 | 10.33 | 4.60 | 1.67 | ||
| 70214 | KY244054 | 12.54 | 3.56 | 1.43 | ||
| 70600 | KY244055 | 20.91 | 2.70 | 1.59 | ||
| 70605 | KY244059 | 11.08 | 2.8 | 1.71 | ||
| 70606 | KY244060 | 11.65 | 2.56 | 1.57 | ||
| 70607 | KY272021 | 10.98 | 2.40 | 2.80 | ||
| 70612 | KY272025 | 20.29 | 3.57 | 1.17 | ||
| 70621 | KY272034 | 21.93 | 3.88 | 1.43 | ||
| 70631 | KY272037 | 13.02 | 2.78 | 1.29 | ||
| 70632 | KY272038 | 14.04 | 3.50 | 1.57 | ||
| 70639 | KY272043 | 10.67 | 2.36 | 1.83 | ||
| 70643 | KY272047 | 21.88 | 3.63 | 2.40 | ||
| 70645 | KY272049 | 17.03 | 3.13 | 2.50 | ||
| Marinobacter | 70209 | KY244049 | 5.83 | 4.14 | 1.43 | |
| 70601 | KY244056 | 11.50 | 2.80 | 1.27 | ||
| 70602 | KY2440657 | 9.82 | 2.25 | 2.00 | ||
| 70604 | KY244058 | 37.23 | 3.83 | 1.43 | ||
| 70608 | KY272022 | 15.09 | 3.00 | 1.63 | ||
| 70610 | KY272024 | 11.43 | 2.00 | 2.00 | ||
| 70623 | KY272035 | 42.40 | 3.4 | 2.67 | ||
| 70637 | KY272041 | 16.36 | 2.6 | 1.75 | ||
| 70638 | KY272042 | 18.85 | 2.17 | 2.50 | ||
| 70640 | KY272044 | 15.20 | 2.67 | 2.20 | ||
| 70644 | KY272048 | 11.72 | 1.56 | 1.75 | ||
| 70648 | KY272052 | 10.93 | 2.83 | 1.50 | ||
| Sulfitobacter | 70613 | KY272026 | 3.40 | 1.43 | 2.00 | |
| 70614 | KY272027 | 6.09 | 1.40 | 2.40 | ||
| 70615 | KY272028 | 3.45 | 1.88 | 2.00 | ||
| 70616 | KY272029 | 3.46 | 1.50 | 1.38 | ||
| 70620 | KY272033 | 3.50 | 1.86 | 1.25 | ||
| 70627 | KY272036 | 3.46 | 1.38 | 1.67 | ||
| 70635 | KY272040 | 3.44 | 2.00 | 2.50 | ||
| 70641 | KY272045 | 3.41 | 2.00 | 1.67 | ||
| 70647 | KY272051 | 3.82 | 1.14 | 1.56 | ||
| Shewanella | 70202 | KY244043 | 6.53 | 2.29 | 1.33 | |
| Photobacterium | 70609 | KY272023 | 5.83 | 4.14 | 1.71 | |
| Firmicutes | Bacillus | 70201 | KY244042 | 5.19 | 1.17 | 1.27 |
| 70204 | KY244045 | 4.24 | 1.11 | 1.86 | ||
| 70208 | KY244048 | 3.76 | 1.17 | 1.12 | ||
| 70212 | KY244052 | 2.89 | 1.13 | 3.25 | ||
| 70617 | KY272030 | 3.44 | 1.67 | 1.43 | ||
| 70618 | KY272031 | 5.30 | 2.00 | 1.54 | ||
| 70646 | KY272050 | 4.66 | 1.4 | 1.28 | ||
| Staphylococcus | 70619 | KY272032 | 2.60 | 1.33 | 1.20 | |
| 70634 | KY272039 | 2.76 | 1.20 | 2.33 | ||
| Bacteroidetes | Salegentibacter | 70642 | KY272046 | 6.72 | 1.33 | 1.67 |
| 注:沉淀圈直径/菌落直径:产脂肪酶菌株在吐温-80平板上沉淀圈菌落直径比值;水解圈直径/菌落直径:产脂肪酶菌株在三丁酸甘油酯平板上水解圈菌落直径比值. Note: P/C: The ratio of the precipitation zone diameter versus the colony diameter of a colony on tween 80 plate; H/C: The ratio of the hydrolytic zone diameter versus the colony diameter of a colony on three butyric acid glyceride plate. | ||||||
产脂肪酶菌株在2216E培养基平板划线纯化后得到单菌落,单菌落点种至吐温-80培养基平板,6 d后观察到明显的脂肪酶沉淀圈,说明菌株分泌的脂肪酶分解了吐温-80,沉淀圈越大说明酶解能力越强。测量所有菌株产生沉淀圈直径和菌落直径大小,并计算其比值(P/C)(表 2)。
由于初筛培养基是吐温-80培养基,因此经筛选分离到的51株产脂肪酶细菌均能在吐温-80平板上产生沉淀圈,但所筛选到的菌株分泌的脂肪酶对吐温-80的分解能力不尽相同。从表 2可以得出,有18株细菌对吐温-80的分解能力较低,其P/C低于2,这些菌株主要是Bacillus、Sulfitobacter、Photobacterium、Salegentibacter、Staphylococcus属;多数菌株对吐温-80具有较强的分解能力,其P/C大于2,这些细菌主要属于Pseudoalteromonas和Marinobacter这两个属,其中Pseudoalteromonas属的70210对吐温-80的分解能力最强,其P/C最大,为5.8。
2.6 产脂肪酶细菌的水解圈产脂肪酶菌株在2216E培养基平板划线纯化后得到单菌落,单菌落点种至三丁酸甘油酯培养基平板,3 d后观察到明显的脂肪酶水解圈,说明菌株分泌的脂肪酶分解了三丁酸甘油酯,水解圈越大说明酶解能力越强。测量所有菌株产生水解圈直径和菌落直径大小,并计算其比值(H/C)(表 2)。经筛选分离到的51株产脂肪酶细菌均能在三丁酸甘油酯平板上产生水解圈,说明其具有降解三丁酸甘油酯的能力。同样,所筛选到的菌株分泌的脂肪酶对三丁酸甘油酯的分解能力不尽相同。多数菌株对三丁酸甘油酯的分解能力较低,其H/C低于2,这些菌株主要属于Pseudoalteromonas、Photobacterium、Sulfitobacter、Bacillus和Salegentibacter属,有10株细菌对三丁酸甘油酯的分解能力较高,其H/C大于2,这些菌主要属于Marinobacter属,少数分布于Pseudoalteromonas、Sulfitobacter、Bacillus和Salegentibacter属,其中Marinobacter属的70623对三丁酸甘油酯的分解能力较强,其H/C较大,为2.67;Bacillus属的70212对三丁酸甘油酯的分解能力最强,其H/C最大,为3.25。
2.7 模式菌株Marinobacter sp. EF10[5]的酶活利用对硝基苯酚法和吐温-80平板沉淀圈法相结合测定模式菌株Marinobacter sp. EF10[5]的脂肪酶活性。结果显示,模式菌株Marinobacter sp. EF10[5]在吐温-80平板上能够产生白色沉淀圈,测得其沉淀圈直径与菌落直径比值(P/C)为1.23,利用对硝基苯酚法测定其酶活为6.04 U/mL,与本文分离到的菌株酶活对比,其比本文分离到的同一属(Marinobacter)产脂肪酶细菌的酶活低,相对于本文Pseudoalteromonas属的脂肪酶菌株的酶活也较低,相对本文其他属的大部分产脂肪酶的菌株酶活也较低,说明本文从渤海沉积物中分离筛选到的产脂肪酶菌株的酶活力相对较高,对于开发海洋微生物脂肪酶资源具有一定的借鉴作用。
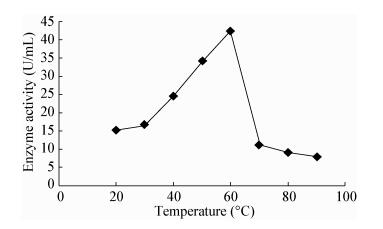
2.8 产脂肪酶菌株70623在不同温度条件下的酶活以对硝基苯酚棕榈酸酯(PNPP)为底物,将产脂肪酶菌株Marinobacter sp. 70623粗酶液置于20-90 ℃温度梯度,以10 ℃为间隔测酶活,确定不同反应温度对酶活力的影响,结果如图 4所示,菌株70623在20-60 ℃条件下随温度的增加酶活逐渐增强,在60 ℃时酶活最高,之后随温度升高酶活迅速下降。
|
| 图 4 温度对脂肪酶产生菌70623酶活力的影响 Figure 4 Effect of temperatures on enzyme activity of the lipase-producing strain of 70623 |
|
|
细菌脂肪酶是一类广泛应用于工业领域的生物催化剂,具有重要的应用价值。脂类水解酶广泛存在于动物、植物和微生物中,由于动植物脂肪酶作用底物的范围窄,体外活性和产量都很低,所以现在工业上所用的脂肪酶都来源于微生物[26]。据统计,大约有65个种属的微生物可以产生脂肪酶,包括细菌、酵母、放线菌及其他真菌[27]。微生物脂肪酶的商业化生产主要是通过Rhizopus delemar、Aspergillus niger、Geotrichum candidum、Candida rugosa和Chromabacterium viscosum等真菌、酵母和细菌的发酵获得[28-29]。海洋中蕴含着丰富的微生物资源,海洋微生物逐渐成为新型酶制剂的重要来源,多种海洋微生物如细菌、放线菌和真菌能够产生活性酶[30]。
本文选用吐温-80选择培养基,从8个渤海沉积物样品中筛选出51株产脂肪酶菌株,通过对其16S rRNA基因的系统发育分析,51株细菌属于3个门的8个属,包括Proteobacteria的Pseudoalteromonas、Marinobacter、Sulfitobacter、Photobacterium、Shewanella,Firmicutes的Bacillus、Staphylococcus,Bacteroidetes的Salegentibacter,其中Pseudoalteromonas (35.2%)、Marinobacter (23.5%)和Sulfitobacter (17.6%)是优势菌群。说明吐温-80平板用于筛选作用于较短碳链底物的脂肪酶和酯酶,产作用于短碳链的脂肪酶的菌株多样性丰富。徐宇丽等用吐温-80选择培养基从新疆天山冻土筛选到17株产低温脂肪酶细菌,分布于Pseudomonas、Duganella、Brevundimonas、Chryseobacterium、Arthrobacter和Rhodococcus六个属[1],与本文筛选到的产脂肪酶细菌在属的水平上完全不相同,可能是陆地和海洋中的各种环境条件的差异,导致了陆源与海洋生态系统中物种的多样性不同。本文筛选到产脂肪酶优势菌群主要是Pseudoalteromonas细菌与Zhang等从南太平洋环流区深海海水中分离到产脂肪酶优势菌群为Pseudoalteromonas和Alteromonas属[31]相似,它们都属于Gammaproteobacteria细菌。据报道,Gammaproteobacteria是各种酶主要的产生菌[31],国内外同行的研究表明Proteobacteria,特别是Gammaproteobacteria细菌是亚南极地区乔治王岛和中国南海、胶州湾沉积物中产蛋白酶细菌的优势菌,同时Pseudoalteromonas菌株也在这些样点被分离到,而且也是中国南海沉积物中的优势菌群[18-19, 32]。这些研究说明了Pseudoalteromonas产酶细菌广泛存在于海洋的不同环境中。Zhang等分离到的Shewanella、Marinobacter、Bacillus、Staphylococcus产脂肪酶细菌[31]在本文中也被分离到,而本文分离到的Sulfitobacter、Photobacterium、Salegentibacter产脂肪酶细菌之前未见报道。
通过比较8个不同取样点细菌,分离自站点BH25、BH12、BH8、E6的脂肪酶菌株多样性相对较少,可能是这些站点位于渤海的近岸海域,受人类活动的影响及环渤海经济圈的工业化不断密集产生大量污染物质排入渤海沿海海域,造成了严重的环境污染,引起近岸水体和沉积物环境发生富营养化,生物多样性降低[33]。位于渤海较中央处P2、PLB1站点,其分离到的细菌在属水平上多样性较为丰富,这可能是该站点受渤海与南黄海物质与能量交换较大,而养殖业等人类干预的影响较少造成产脂肪酶细菌的多样化。而在水深较深、温度最低的两个站点(N4、R6) 分离到的脂肪酶菌株主要是Pseudoalteromonas细菌,与之前报道的产酶Pseudoalteromonas细菌具有较强的适冷性相关[34]。
通过对筛选到的菌株的胞外脂肪酶酶活测定,结果得出多数Pseudoalteromonas和Marinobacter属细菌的酶活相对较高,酶活值超过了10 U/mL,其在吐温-80平板上产生的沉淀圈与菌落直径比值也较大,其P/C大于2;其它6个属的菌株产生的酶活相对较低,酶活值都低于10 U/mL,其在吐温-80平板上产生的沉淀圈与菌落直径比值也相对较小,除了Shewanella的菌株70202的P/C为2.29外,其余的P/C均小于2,表明了Pseudoalteromonas和Marinobacter是渤海沉积物中主要的产脂肪酶细菌。近几年来,有越来越多的研究报道从海洋环境中分离得到产脂肪酶菌株,如郝文惠等从南极深海沉积物中分离到产脂肪酶芽孢杆菌XZ18的最高产酶量为4.65 U/mL[27];邵铁娟等从2 000多份渤海海区海水海泥样品中分离获得一株新型脂肪酶高产菌株Yarrowia lipolytica bohaisea-9145,最高酶活达25.6 U /mL[35];王全富等从鄂霍次克海域沉积物样品(lv39-06-06-04-18H)分离筛选到菌株Bacillus sp. 130产脂肪酶活性最高,其活性达到42 U/mL[36],而本文筛选到的Marinobacter属的细菌70623产脂肪酶酶活最高,达到42.4 U/mL,在20-60 ℃条件下可以检测出随温度的增加酶活逐渐增强,最适产酶温度为60 ℃,确定其为中温脂肪酶产生菌,有较好的开发应用价值。目前新的一项研究报道来自于Bakir等[37]从土耳其的天然温泉中筛选到22株能够产脂肪酶菌株,其中有一株属于中温脂肪酶产生菌株Anoxybacillus flavithermus HBB 134的产酶能力最高。作者采用同样的方法,即对硝基苯酚法,在相同的条件下测定菌株Anoxybacillus flavithermus HBB-134在30-80 ℃不同温度下的酶活,结果得到,在温度30-45 ℃条件下随温度的增加酶活逐渐增强;在温度为45 ℃时酶活最高,为79.96 U/mg;45 ℃之后的酶活逐渐降低,在60 ℃时的酶活降为40.52 U/mg。与本文筛选到的高效产脂肪酶菌株Marinobacter sp. 70623相比,它们的共同点是均为中温脂肪酶产生菌株,不同点是两者的最优产酶温度不同,Anoxybacillus flavithermus HBB-134的最适产酶温度为45 ℃,Marinobacter sp. 70623的最适产酶温度为60 ℃。此外,两者在最适产酶温度下的最高酶活也不同,Anoxybacillus flavithermus HBB-134的最高酶活(79.96 U/mg)高于Marinobacter sp. 70623的最高酶活(42.4 U/mL),但在温度为60 ℃时,Anoxybacillus flavithermus HBB-134的酶活(40.52 U/mg)低于Marinobacter sp. 70623的酶活(42.4 U/mL),表明不同来源的中温脂肪酶菌株代谢产酶的能力有所差异,但它们的酶活相对较高,具有良好的开发应用前景。
本文采用3种方法测定脂肪酶活力,对硝基苯酚法测定脂肪酶活力是以对硝基苯酚棕榈酸酯(PNPP)为底物,使发酵粗酶液与底物发生显色反应,然后采用分光光度计法进行测定。以对硝基苯酚作为标准物,绘制标准曲线,根据测出的光密度值和标准曲线计算出酶活值。该方法的优点是能够精确地、定量地测定脂肪酶活力值,是测定脂肪酶活性一种常用的方法,缺点是该方法实验周期长,实验中所用到药品试剂多、成本高。三丁酸甘油酯水解圈法是以三丁酸甘油酯为碳源,脂肪酶能够以三丁酸甘油酯为底物,在具有分泌脂肪酶菌落的周围形成透明的水解圈,达到鉴别目的。该方法的优点是可以直接观察水解圈的有无来确定菌株是否具有脂肪酶活性,方便又快捷,缺点是配制三丁酸甘油酯培养基所要的药品试剂多,价格成本高。吐温-80平板沉淀圈法测定脂肪酶活力的原理是在Ca2+存在的条件下,脂肪酶能够以吐温-80为底物,在具有分泌脂肪酶菌落的周围形成白色的Ca2+复合白色沉淀颗粒,从而与非分泌的脂肪酶菌落相区分,达到鉴别效果。该方法的优点是吐温-80培养基不使用植物油和指示剂,可以替代相关的油剂培养基使用,在成本上较为经济。缺点是该方法仅适合初筛脂肪酶使用,只能定性鉴别出脂肪酶活性,不能测定酶活力的大小。因此,通常情况下,采用吐温-80平板沉淀圈法和对硝基苯酚法结合测定脂肪酶活力,吐温-80平板沉淀圈法用于脂肪酶的初步定性鉴别,对硝基苯酚法则可以进一步地测定其酶活力的大小。此外,通过模式菌株的阳性对照实验表明:本文从渤海沉积物中分离筛选到的产脂肪酶菌株的酶活力相对较高,对于开发海洋微生物脂肪酶资源具有一定的借鉴作用。
海洋的高盐、高压、寡营养和无光照等特殊生态环境决定了海洋微生物种类的多样性和特殊性,因而造成了具有独特的代谢途径和遗传背景,并且能够产生具有独特结构和功能的酶类物质。本文从渤海8个沉积物站点分离到51株产脂肪酶细菌,属于3个门的8个属,其中Pseudoalteromonas (35.2%)、Marinobacter (23.5%)和Sulfitobacter (17.6%)是优势菌群,而筛选到的Sulfitobacter、Photobacterium、Salegentibacter产脂肪酶细菌为首次报道,这些研究结果丰富了浅海微生物的脂肪酶资源,增加了研究者们对渤海生态系统中产脂肪酶细菌多样性的认识和了解,为工业生产脂肪酶提供了重要的菌群资源。本文获得了一株高效产脂肪酶菌株Marinobacter sp. 70623,为挖掘海洋产脂肪酶细菌及新脂肪酶具有一定的借鉴作用。
| [1] | Xu YL, Wang DW, Shi XW, et al. Selective isolation and diversity of cold-adapted lipase-producing strains from permafrost soil at the terminus of a glacier in the Tianshan Mountains[J]. Acta Microbiological Sinica, 2011, 51(2) : 233–240. (in Chinese) 徐宇丽, 王大伟, 史学伟, 等. 天山冻土产低温脂肪酶菌株的筛选及其多样性分析[J]. 微生物学报, 2011, 51(2) : 233–240. |
| [2] | Zhang Y, Sun XC, Wang KF, et al. Study on the character of a lipase produced by a bacterium[J]. Journal of Hubei Agricultural Sciences, 2011, 50(1) : 53–55. (in Chinese) 张勇, 孙新城, 王开放, 等. 一株细菌产生的脂肪酶性质研究[J]. 湖北农业科学, 2011, 50(1) : 53–55. |
| [3] | Zhong GF, Yuan HX, Shi WT, et al. Screening of lipase producing strains and studying on enzymatic properties of lipase[J]. Science and Technology of Food Industry, 2013, 34(20) : 221–224. (in Chinese) 钟桂芳, 袁海晓, 史文婷, 等. 产碱性脂肪酶菌株的筛选及酶学性质研究[J]. 食品工业科技, 2013, 34(20) : 221–224. |
| [4] | Yang JF. Isolation of alkaline lipase producing strain and purification and characterization of alkaline lipase[D]. Nanjing: Master's Thesis of Nanjing University of Science and Technology, 2004 (in Chinese) 杨军方. 产脂肪酶细菌的筛选和酶学性质研究[D]. 南京: 南京理工大学硕士学位论文, 2004 http://cdmd.cnki.com.cn/article/cdmd-10288-2004108098.htm |
| [5] | Wang K, Liu HG, Jiang K, et al. Study on classification and enzymatic propertis of deep-sea bacteria producing lipase[J]. Current Biotechnology, 2015, 5(3) : 246–249. (in Chinese) 王凯, 刘洪国, 姜昆, 等. 产脂肪酶深海细菌的筛选鉴定及酶学性质研究[J]. 生物技术进展, 2015, 5(3) : 246–249. |
| [6] | Hasan F, Shah AA, Hameed A. Industrial applications of microbial lipases[J]. Enzyme and Microbial Technology, 2006, 39(2) : 235–251. DOI:10.1016/j.enzmictec.2005.10.016 |
| [7] | Jaeger KE, Dijkstra BW, Reetz MT. Bacterial biocatalysts: molecular biology, three-dimensional structures, and biotechnological applications of lipases[J]. Annual Review of Microbiology, 1999, 53(1) : 315–351. DOI:10.1146/annurev.micro.53.1.315 |
| [8] | Alquati C, de Gioia L, Santarossa G, et al. The cold-active lipase of Pseudomonas fragi. Heterologous expression, biochemical characterization and molecular modeling[J]. European Journal of Biochemistry, 2002, 269(13) : 3321–3328. DOI:10.1046/j.1432-1033.2002.03012.x |
| [9] | Haki GD, Rakshit SK. Developments in industrially important thermostable enzymes: a review[J]. Bioresource Technology, 2003, 89(1) : 17–34. DOI:10.1016/S0960-8524(03)00033-6 |
| [10] | Wang JY, Wu QJ, Bao YM, et al. Screening of lipase producing alkalophiles bacteria and study of enzymatic properties[J]. Biotechnology, 2010, 20(6) : 57–61. (in Chinese) 王静云, 武秋娟, 包永明, 等. 产脂肪酶嗜碱细菌的筛选及酶学性质研究[J]. 生物技术, 2010, 20(6) : 57–61. |
| [11] | Chen MX, Li HY, Chen WW, et al. Isolation, identification and characterization of 68 protease-producing bacterial strains from the Arctic[J]. Acta Microbiologica Sinica, 2013, 53(7) : 702–709. (in Chinese) 陈明霞, 李和阳, 陈维维, 等. 68株北极产蛋白酶菌株的筛选、鉴定以及部分酶学性质[J]. 微生物学报, 2013, 53(7) : 702–709. |
| [12] | And OK, Christensen MW. Lipases from Candida antarctica: unique biocatalysts from a unique origin[J]. Organic Process Research and Development, 2002, 6(4) : 446–451. DOI:10.1021/op0200165 |
| [13] | Bowman JP, Mccammon SA, Brown MV, et al. Diversity and association of psychrophilic bacteria in Antarctic sea ice[J]. Applied and Environmental Microbiology, 1997, 63(8) : 3068–3078. |
| [14] | Rashid N, Shimada Y, Ezaki S, et al. Low-temperature lipase from psychrotrophic Pseudomonas sp. strain KB700A[J]. Applied and Environmental Microbiology, 2001, 67(9) : 4064–4069. DOI:10.1128/AEM.67.9.4064-4069.2001 |
| [15] | Arpigny JL, Lamotte J, Gerday C. Molecular adaptation to cold of an Antarctic bacterial lipase[J]. Journal of Molecular Catalysis B: Enzymatic, 1997, 3(1/4) : 29–35. |
| [16] | Yang FL. Response and degradation potential of bacterial communities against petroleum hydrocarbons in the surface-sediments of Bohai Sea, China[D]. Beijing: Doctoral Dissertation of China Agricultural University, 2015 (in Chinese) 杨凤龙. 渤海表层沉积物中细菌群落对石油烃污染的响应及其降解潜力研究[D]. 北京: 中国农业大学博士学位论文, 2015 http://cdmd.cnki.com.cn/Article/CDMD-10019-1015717663.htm |
| [17] | Wang YT, Zhang CB, Qi L, et al. Diversity and antimicrobial activities of cultivable bacteria isolated from Jiaozhou Bay[J]. Acta Microbiologica Sinica, 2016, 56(12) : 1892–1900. (in Chinese) 王怡婷, 张传波, 齐麟, 等. 胶州湾沉积物可培养细菌的多样性及其抑菌活性[J]. 微生物学报, 2016, 56(12) : 1892–1900. |
| [18] | Zhou MY, Chen XL, Zhao HL, et al. Diversity of both the cultivable protease-producing bacteria and their extracellular proteases in the sediments of the South China Sea[J]. Microbial Ecology, 2009, 58(3) : 582–590. DOI:10.1007/s00248-009-9506-z |
| [19] | Zhang XY, Han XX, Chen XL, et al. Diversity of cultivable protease-producing bacteria in sediments of Jiaozhou Bay, China[J]. Frontiers in Microbiology, 2015, 6 : 1021. |
| [20] | Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors[J]. Proceedings of the National Academy of Sciences of the United States of America, 1977, 74(12) : 5463–5467. DOI:10.1073/pnas.74.12.5463 |
| [21] | Li Y, Li XY, Liu YJ, et al. Genetic diversity and community structure of rhizobia nodulating Sesbania cannabina in saline-alkaline soils[J]. Systematic and Applied Microbiology, 2016, 39(3) : 195–202. DOI:10.1016/j.syapm.2016.02.004 |
| [22] | Tamura K, Peterson D, Peterson N, et al. MEGA 5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10) : 2731–2739. DOI:10.1093/molbev/msr121 |
| [23] | Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987, 4(4) : 406–425. |
| [24] | Massadeh MI, Sabra FM. Production and characterization of lipase from Bacillus stearothermophilus[J]. African Journal of Biotechnology, 2011, 10(61) : 13139–13146. |
| [25] | Chen XL, Zhang YZ, Gao PJ, et al. Two different proteases produced by a deep-sea psychrotrophic bacterial strain, Pseudoaltermonas sp. SM9913[J]. Marine Biology, 2003, 143(5) : 989–993. DOI:10.1007/s00227-003-1128-2 |
| [26] | Cieśliński H, Kur J, Białkowska A, et al. Cloning, expression, and purification of a recombinant cold-adapted β-galactosidase from antarctic bacterium Pseudoalteromonas sp. 22b[J]. Protein Expression and Purification, 2005, 39(1) : 27–34. DOI:10.1016/j.pep.2004.09.002 |
| [27] | Hao WH, Wang FY, Guo Y, et al. Screening of cold-active lipase producing strains from Antarctic sediments and cloning of the gene[J]. Journal of Applied Oceanography, 2014, 33(3) : 306–311. (in Chinese) 郝文惠, 王凡羽, 郭玉, 等. 南极深海沉积物中产低温脂肪酶菌株的筛选与基因克隆[J]. 应用海洋学学报, 2014, 33(3) : 306–311. |
| [28] | Gandhi NN. Applications of lipase[J]. Journal of the American Oil Chemists' Society, 1997, 74(6) : 621–634. DOI:10.1007/s11746-997-0194-x |
| [29] | Chen GY, Ji XL, Lin LB, et al. Isolation and identification of a cold-adapted lipase producing bacterium, study on fermentation conditions and lipase properties[J]. Journal of Yunnan University (Natural Science Edition), 2010, 32(1) : 108–113. (in Chinese) 陈贵元, 季秀玲, 林连兵, 等. 低温脂肪酶产生菌筛选与鉴定、产酶条件及酶学性质研究[J]. 云南大学学报:自然科学版, 2010, 32(1) : 108–113. |
| [30] | Shi CJ, Yan PS, Zhao RX, et al. Advances on enzymes from marine microorganisms[J]. Current Biotechnology, 2015, 5(3) : 185–190. (in Chinese) 史翠娟, 闫培生, 赵瑞希, 等. 海洋微生物酶研究进展[J]. 生物技术进展, 2015, 5(3) : 185–190. |
| [31] | Zhang L, Wang Y, Liang J, et al. Degradation properties of various macromolecules of cultivable psychrophilic bacteria from the deep-sea water of the South Pacific Gyre[J]. Extremophiles, 2016, 20(5) : 663–671. DOI:10.1007/s00792-016-0856-4 |
| [32] | Zhou MY, Wang GL, Li D, et al. Diversity of both the cultivable protease-producing bacteria and bacterial extracellular proteases in the coastal sediments of king george island, antarctica[J]. PLoS One, 2013, 8(11) : e79668. DOI:10.1371/journal.pone.0079668 |
| [33] | Tian F. The study of ammonia-oxidizing bacteria from sediments in Bohai Sea[D]. Beijing: Master's Thesis of China University of Petroleum (East China), 2012 (in Chinese) 田方. 渤海沉积环境硝化过程氨氧化细菌的研究[D]. 北京: 中国石油大学(华东)硕士学位论文, 2012 http://cdmd.cnki.com.cn/Article/CDMD-10425-1014017387.htm |
| [34] | Yu ZC, Zhao DL, Ran LY, et al. Development of a genetic system for the deep-sea psychrophilic bacterium Pseudoalteromonas sp. SM9913[J]. Microbial Cell Factories, 2014, 13(1) : 133–139. DOI:10.1186/s12934-014-0133-0 |
| [35] | Shao TJ, Sun M, Zheng JS, et al. Study on low-temperature alkaline lipase from bohaisea-9145[J]. Acta Microbiologica Sinica, 2004, 44(6) : 789–793. (in Chinese) 邵铁娟, 孙谧, 郑家声, 等. Bohaisea-9145海洋低温碱性脂肪酶研究[J]. 微生物学报, 2004, 44(6) : 789–793. |
| [36] | Wang QF, Hou YH, Lin YF, et al. Screening and classification of deep-sea microbes producing low-temperature lipase and characterization of the lipase[J]. Food Research and Development, 2012, 33(11) : 180–183. (in Chinese) 王全富, 侯艳华, 蔺一飞, 等. 产低温脂肪酶深海菌株的筛选、鉴定及酶学性质研究[J]. 食品研究与开发, 2012, 33(11) : 180–183. |
| [37] | Bakir ZB, Metin K. Screening for industrially important enzymes from thermophilic bacteria; selection of lipase-producing microorganisms and optimization of culture conditions[J]. European Journal of Biotechnology and Bioscience, 2015, 3(6) : 43–48. |
 2017, Vol. 44
2017, Vol. 44




