扩展功能
文章信息
- 乔铖, 任磊, 樊双虎, 贾阳, 赵百锁, 闫艳春
- QIAO Cheng, REN Lei, FAN Shuang-Hu, JIA Yang, ZHAO Bai-Suo, YAN Yan-Chun
- 联苯利用节杆菌YC-RL1中2, 3-二羟基联苯-1, 2-双加氧酶BphC功能验证
- Characterization of 2, 3-dihydroxybiphenyl-1, 2-dioxygenase BphC from biphenyl degrader Arthrobacter sp. YC-RL1
- 微生物学通报, 2017, 44(7): 1639-1648
- Microbiology China, 2017, 44(7): 1639-1648
-
文章历史
- 收稿日期: 2017-02-17
- 接受日期: 2017-05-17
- 优先数字出版日期(www.cnki.net): 2017-05-25
近几十年来,科研工作者长期致力于改善环境污染问题,证明了生物降解法在污染物清除方面较通常的物理或化学性策略具有更好的应用潜力。由于一些传统的物理或化学降解法通常需要更多花费且会形成一些技术性难题,而应用微生物对污染物进行降解则不仅具有无二次污染、成本低廉的特点,而且具有生态安全及环境友好等优势[1-3],因而生物降解作为可替代传统修复手段的新兴技术已成为广受青睐的环境污染治理方法。
多氯联苯是一类有毒的持久性环境有机污染物,广泛存在于自然环境中。这一类化合物由于具有优良的化学稳定性、较强的绝缘性及高导热性能而被大量应用于工业原料及除草剂等的生产中[4]。然而,多氯联苯对生物体具有致癌、致畸和致突变的“三致效应”,可在自然环境中引起严重的污染问题[5]。有研究报道称[6-7],自1929年首次被人工合成以来,迄今全球已有约200万t多氯联苯生产量,尽管20世纪70年代多氯联苯已被世界范围内禁止使用,然而地球上部分地区仍存在较高浓度水平的多氯联苯污染物。因此,对这类化合物的生物降解研究仍然具有至关重要的意义。
联苯作为重要的多氯联苯前体物,其生物降解已得到了广泛的研究,为了解多氯联苯的降解提供了有效的科学基础。通常认为,细菌是经由联苯降解途径来完成对多氯联苯的好氧性代谢的[8]。已有研究报道[9-10],联苯的上游降解途径(图 1)中,其代谢终产物分别是苯甲酸与2-羟基戊-2, 4-二烯酸,参与其中的四类酶主要为联苯双加氧酶(BphA)、二氢二醇脱氢酶(BphB)、2, 3-二羟基联苯-1, 2-双加氧酶(BphC)以及2-羟基-6-氧-6-苯基-2, 4-己二烯水解酶(BphD),其相应的中间产物分别为2, 3-二氢二醇联苯、2, 3-二羟基联苯(2, 3-DHBP)、2-羟基-6-氧-6-苯基-2, 4-己二烯(HOPDA)以及苯甲酸等。其中,羟基联苯类双加氧酶通常根据其裂解苯环上C-C键位置的不同被分为内二醇与外二醇类双加氧酶。外二醇类双加氧酶根据其独立进化单元又可分为三种类型,据报道[11],多数外二醇类双加氧酶属于第一种类型,即为邻氧螯合超家族;第二种类型属于非邻氧螯合超家族且含有同源寡聚或杂低聚复合物;第三种类型则属于Cupin超家族。在联苯降解的几类酶中,2, 3-二羟基联苯-1, 2-双加氧酶多为外二醇类双加氧酶,是实现多氯联苯完全降解的关键酶,对环境中多氯联苯以及一些芳香族化合物的生物降解具有至关重要的作用[12-13]。目前,相关报道中多氯联苯降解菌主要有红球菌属[14]、伯克氏菌属[15]、假单胞菌属[16]等,节杆菌则鲜有报道。因此,对该酶的研究将有助于进一步理解多氯联苯的微生物代谢途径。

|
| 图 1 联苯的生物降解上游途径 Figure 1 The upstream biodegradation pathway of biphenyl 注:有氧条件下,联苯的上游降解途径分别由联苯双加氧酶(BphA)、二氢二醇脱氢酶(BphB)、2, 3-二羟基联苯-1, 2-双加氧酶(BphC)以及2-羟基-6-氧-6-苯基-2, 4-己二烯水解酶(BphD)在不同条件下催化进行,其相应产物分别为2, 3-二氢二醇联苯、2, 3-二羟基联苯、2-羟基-6-氧-6-苯基-2, 4-己二烯以及苯甲酸等. Note: The upstream biodegradation pathway of biphenyl was catalyzed by BphA, BphB, BphC and BphD under aerobic conditions while the metabolites were 2, 3-dihydrodiol-biphenyl, 2, 3-dihydroxybiphenyl, 2-hydroxy-6-oxo-phenyl-2, 4-hexadienoic acid, benzoic acid and 2-hydroxypenta-2, 4-dienoate, respectively. |
|
|
从长期受芳香族化合物污染的活性淤泥中分离并筛选到了一株可高效利用联苯为唯一碳源和能源生长的细菌,通过全基因组测序手段鉴定其为节杆菌属,命名YC-RL1,其16S rRNA基因序列已递交至GenBank,登录号为EU266494[17]。从YC-RL1基因组中经PCR扩增获得2, 3-二羟基联苯-1, 2-双加氧酶的编码基因bphC,利用携带有6个组氨酸标签的pET32a(+)质粒构建表达载体并在E. coli BL21(DE3) 中进行了原核表达。根据亲和凝胶层析法利用ÄKTA avant25蛋白质纯化系统将该基因的表达产物BphC从细胞粗提物中进行纯化,研究了不同pH、温度及金属离子等对该酶活性的影响,进一步对其酶促动力学特性进行了探究。研究表明,该酶对2, 3-DHBP具有较强的催化活性,能够在体外环境中对多氯联苯化合物的生物降解发挥至关重要的作用,对污染环境的生物修复具有较高的应用潜力。
1 材料与方法 1.1 材料 1.1.1 菌株及培养基:节杆菌YC-RL1由本实验室分离并保存;pET32a(+)载体与E. coli DH5α和E. coli BL21(DE3) 为本实验室保存,分别用以载体构建与蛋白质表达,在37 ℃生长于含100 mg/mL氨苄青霉素(Amp)的LB培养基[18]中。节杆菌YC-RL1细胞在30 ℃生长于无机盐培养基BM[19]或LB培养基中。固体BM与LB中均添加15 g/L琼脂。所有新鲜配制的培养基均在1×105 Pa灭菌30 min后备用。
1.1.2 主要试剂和仪器:联苯与2, 3-二羟基联苯购自美国Sigma公司,溶于甲醇后分别制成终浓度为2×104 mg/L和100 mg/mL的母液,经0.22 μm滤器过滤除菌后备用。所有DNA操作中的酶均购自大连TaKaRa生物技术有限公司。甲醇与乙腈购自美国Fisher公司,均为色谱级纯,其他药品均为分析纯,购自国药集团。PCR仪购自美国Bio-Rad公司,高效液相色谱仪1200购自美国Agilent公司,分光光度计Nifinite 200购自瑞士Tecan公司,ÄKTA avant25蛋白质纯化仪购自美国GE公司。
1.2 方法 1.2.1 bphC的基因克隆与生物信息学分析:采用细菌基因组提取试剂盒获得菌株YC-RL1基因组DNA,以此为模板进行PCR扩增目的基因片段。基于基因组测序结果[17],利用Primer Premier 6.0软件设计PCR扩增引物序列,分别为:bphC-F:5′-GCGCGGATCCATGACTCACATCCGTGGACTT-3′;bphC-R:5′-GCGCCTCGAGGTTTGCCGCGGCGAC-3′;其中下划线部分分别为BamH I和Xho I相应的酶切位点。25 μL反应体系:Premix TaqTM 12.5 μL,引物bphC-F/bphC-R (10 μmol/L)各1 μL,模板1 μL,补加超纯水至25 μL。PCR反应条件为:95 ℃ 3 min;95 ℃ 30s,60 ℃ 30s,72 ℃ 1 min,35个循环;72 ℃ 5 min。PCR扩增产物根据DNA片段纯化试剂盒操作步骤进行纯化。利用BamH I与Xho I酶对DNA纯化产物与pET32a(+)质粒分别进行双酶切后16 ℃连接过夜,连接产物经42 ℃转化至E. coli DH5α,筛选重组质粒并经双酶切后经生工生物工程(上海)股份有限公司测序验证,测序正确后将重组质粒转入E. coli BL21(DE3) 感受态细胞,在含100 mg/mL Amp的LB固体培养基中挑选阳性克隆,用于下一步原核表达。
同时,根据该基因所编码的氨基酸序列,通过BLASTp程序(https://blast.ncbi.nlm.nih.gov/)进行同源序列比对,并使用MEGA 6.0软件基于Neighbor-Joining方法[20],将bootstrap值设为1 000,构建系统发育树。此外,以BphC_2zyq.1.A[21]的3D结构为模板,利用SWISS-MODEL及PyMOL软件对该酶的三维结构进行同源建模分析。
1.2.2 基因表达与BphC酶的纯化:重组E. coli BL21(DE3) 细胞37 ℃在含100 mg/mL Amp的LB培养基中生长至OD600为0.8时转接1%菌液至400 mL LB中,37 ℃培养4 h后添加诱导剂异丙基-β-D-硫代吡喃半乳糖苷(Isopropy-β-D-thiogalactoside,IPTG)至终浓度为2 mmol/L并在30 ℃诱导表达4 h,固定摇床转速均为180 r/min。收集菌体并在4 ℃、8 000 r/min离心10 min后弃上清,用适量结合缓冲液(20 mmol/L磷酸钠,0.5 mol/L氯化钠,30 mmol/L咪唑,pH 7.4) 冲洗沉淀3次后重悬浮,悬浮细胞经超声破碎至澄清,4 ℃、14 000×g离心30 min,弃沉淀即得可溶性重组蛋白粗提液。进一步通过蛋白质纯化仪ÄKTA avant25利用His-TrapTM HP (1 mL)镍柱根据亲和层析方法对含有6个组氨酸标签的重组蛋白进行纯化,纯化过程中上样量50 mL,系统流速1 mL/min,等度洗脱体积10 mL,洗脱收集液每管1 mL,洗脱缓冲液组成为:20 mmol/L磷酸钠,0.5 mol/L氯化钠,500 mmol/L咪唑,pH 7.4,纯化后所得酶液4 ℃保存备用,保存时间不超过48 h。根据Bradford法[22]利用BCA蛋白检测试剂盒以牛血清白蛋白为标准品对所得蛋白质进行浓度测定。通过SDS-PAGE (12%分离胶和5%浓缩胶)检测相应蛋白的纯度与大小,利用考马斯亮蓝R-250对蛋白胶进行染色。
1.2.3 酶活实验:BphC的酶活特性测定是以2, 3-DHBP为底物,利用分光光度计检测2, 3-DHBP裂解产物HOPDA在λmax=434 nm [摩尔消光系数=13 200 L/(mol·cm)]时的吸光值所得[11]。酶活反应是在30 ℃、1 mL 50 mmol PBS缓冲液(pH 7.4) 中进行,反应由体系中加入20 μg 30 ℃预热10 min的BphC酶起始,其中反应底物2, 3-DHBP起始浓度为5 mmol/L。一个酶活单位被定义为该酶在30 ℃每分钟催化2, 3-DHBP裂解形成1 μmol产物HOPDA时的用量。
通过分光光度法在50 mmol/L不同pH的缓冲液[HAc-NaAc (pH 3.6-4.5),Na2HPO4-KH2PO4 (pH 4.2-6.6),PBS (pH 6.2-7.4),Tris-HCl (pH 7.0-8.8) 和Na2CO3-NaHCO3 (pH 8.0-10.5)]中对该酶的最适pH进行测定,同时在最适pH条件下检测该酶在10-50 ℃范围内每5 ℃的相对酶活,确定该酶最适温度。金属离子对酶活的影响则是在最适pH与温度条件下,向反应体系中添加不同金属离子(Fe2+,Fe3+,Zn2+,Mg2+,Ca2+,Mn2+,Co2+,Cd2+,Ni2+)至终浓度为2 mmol/L时测得,由于EDTA作为金属离子螯合剂可能会去除酶活中心的金属离子,因此也检测了其对酶活的影响。以上实验反应时间均为10 min,至少3次重复。
1.2.4 酶促动力学实验:为研究该酶的酶促动力学特性,计算了其在30 ℃、1 mL 50 mmol/L的PBS (pH 7.4) 缓冲体系中以2, 3-DHBP底物浓度范围为0.5-10 mmol/L时反应1 min后的底物降解率,体系中加入10 μmol/L NaOH以终止反应。底物降解率利用高效液相色谱仪基于外标法进行检测所得,其中,HPLC色谱柱为Zorbax Eclipse Plus C18 (4.6 mm×150 mm×5 μm),流动相为甲醇与水(70:30,体积比),流速为1 mL/min,进样量10 μL,检测波长为260 nm。
该酶的动力学参数Km、Vmax、kcat及kcat/Km根据Lineweaver-Burk双倒数法[23]利用BphC对不同浓度底物的降解计算所得。相关酶活反应均如上所述为BphC酶30 ℃温育10 min后进行。以上所有实验至少重复3次。
2 结果与分析 2.1 bphC的基因克隆与生物信息学分析 2.1.1 bphC的基因克隆与验证:以菌株YC-RL1的基因组DNA为模板进行PCR扩增,扩增产物经1%的琼脂糖凝胶电泳检测,结果如图 2所示,得到与预期大小相符的DNA目的片段(930 bp)。扩增产物纯化后经BamH I与Xho I酶切,连接到同样处理的pET32a(+)载体上。连接产物转化E. coli DH5α,筛选重组质粒并进行测序验证,测序结果与GenBank (登录号:CP013297.1) 中报道的序列大小一致,可用于下一步重组蛋白表达。

|
| 图 2 bphC扩增基因琼脂糖凝胶电泳 Figure 2 Agarose gel electrophoresis of PCR amplification of bphC gene 注:M:Marker DL2000;bphC:PCR克隆基因. Note: M: Marker DL2000; bphC: The gene cloned by PCR. |
|
|
菌株YC-RL1中的bphC基因编码309个氨基酸,通过在线程序(http://web.expasy.org/protparam/)计算其分子量为34.14 kD,pI值为4.97。利用其氨基酸序列通过BLASTp程序(https://blast.ncbi.nlm.nih.gov/)进行同源序列比对,基于Neighbor-Joining方法,使用MEGA 6.0软件构建系统发育树如图 3所示。结果表明,菌株YC-RL1中BphC的氨基酸序列在进化水平上处于一个相对独立的分支,且与一株节杆菌LS16中的外二醇类双加氧酶聚为一支,二者具有较高的相似性,因此推测该酶的编码序列是较为新颖的一段基因。

|
| 图 3 BphC的系统发育树 Figure 3 Phylogenetic analysis of BphC 注:括号内数字为GenBank登录号;分支点上数字代表每1 000次抽样分析的置信度;标尺代表碱基替代率. Note: The numbers in the bracket denote the GenBank accession number; Numbers at branch points represent the confidence level of each 1 000 sampling analysis; Scale represents base substitution rate. |
|
|
此外,以BphC_2zyq.1.A的3D结构为模板,利用SWISS-MODEL及PyMOL软件对该酶的三维结构进行同源建模。结果如图 4A所示,BphC酶具有5个α-螺旋(以红色显示)与14个β-折叠(以黄色显示)二级结构,其中17Ile-28Leu、72Gln-85Ala、94Glu-100Arg、154Met-165Leu及219Leu-232Met氨基酸残基形成5个α-螺旋结构,均分布在蛋白质外侧,其余部分构成β-折叠结构以及Loop结构(以绿色显示)等。其中,在该酶活性中心位置处含有一个Fe(Ⅱ)原子(图 4中绿色圆点),该原子被8个β-折叠结构所包围(图 4B)。据报道[24],绝大多数内二醇类双加氧酶活性中心含有一个非血红素Fe(Ⅲ),催化邻位碳碳键断裂;外二醇类双加氧酶活性中心则通常含有一个非血红素Fe(Ⅱ),催化苯环上间位碳碳键的断裂。因此推测该酶为Fe(Ⅱ)依赖型外二醇类双加氧酶。
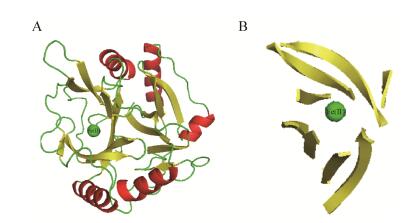
|
| 图 4 BphC酶三维结构预测与分析 Figure 4 3D structure predicted and analysis of BphC 注:A:以BphC_2zyq.1.A为模板BphC的三维预测结构;B:BphC酶活中心Fe(Ⅱ)原子及其他二级结构. Note:A: The 3D structure of BphC predicted using BphC_2zyq.1.A as a template; B: The positions of Fe(Ⅱ) in the active site with other secondery structures. |
|
|
菌株YC-RL1中的bphC基因被克隆至含有6个组氨酸标签的pET32a(+)载体上,转入E. coli BL21(DE3) 细胞后经2 mmol/L的IPTG进行原核表达。重组蛋白经超声破碎法后离心,再利用ÄKTA avant25蛋白质纯化仪进一步对细胞裂解物进行亲和层析纯化,详细纯化步骤参照操作指南进行适当调整。纯化所得蛋白经SDS-PAGE凝胶电泳分析,结果显示两次洗脱蛋白条带均出现在50-60 kD之间(图 5A),与该酶本身大小为34.14 kD且携带有6个组氨酸标签的结果相一致。同时,细胞外液中未检测到蛋白质条带,经SignalP 4.1 Server (http://www.cbs.dtu.dk/services/SignalP/)程序分析,该酶不含信号肽结构,表明其为胞内分泌蛋白。第二次洗脱所得蛋白含量明显高于第一次,且第二次洗脱后蛋白质纯度更高,经检测第二次纯化后蛋白浓度约为3 000 μg/mL,是第一次纯化所得蛋白浓度的1.7倍。

|
| 图 5 BphC酶的SDS-PAGE结果与蛋白纯化在线检测图谱 Figure 5 SDS-PAGE for BphC and online detection of protein purification from ÄKTA avant25 注:A为BphC酶的SDS-PAGE结果,M:蛋白质Marker;CL:细胞裂解液;FT:流穿液;W:洗涤液;EC:细胞外液;E1,E2:洗脱液1和洗脱液2;上样量为5 μL. B为ÄKTA avant25蛋白质纯化曲线,蓝色曲线为280 nm时蛋白质检测曲线;红色部分为收集液位置;其中3A1-3A5为细胞裂解液上样过程,3B1-3C4为蛋白质洗脱液收集过程. Note:A is SDS-PAGE for BphC. M: Protein marker; CL: Cell lysate; FT: Flow-through fraction; W: Wash fractions; EC: Extracellular fraction; E1, E2: Elution fractions 1, 2; 5 μL of each fraction was loaded on to the gel. B is online detection of BphC from ÄKTA avant25 in which blue curves are detection of protein at 280 nm while red ones are for the positions of collected fractions, where 3A1-3A5 are sampling process and 3B1-3C4 are collections for elution fractions. |
|
|
实验中ÄKTA avant25在线检测图谱如图 5B所示,其中,蓝色曲线为280 nm时蛋白质检测曲线,红色部分为收集液对应的收集管位置,其中3A1-3A5对应于粗酶液上样过程,3B1-3C4为蛋白质洗脱液收集过程,洗脱液收集体积为每管1 mL,可见,事实上纯化后较高浓度的蛋白质主要集中在3B3-3B4收集管中,280 nm时的最高吸收值约为2 000 mAU,为上样总蛋白的62.5%,蛋白纯化效率较高,有利于进一步的实验研究。
2.3 酶活实验 2.3.1 BphC酶活的最适反应pH与温度:BphC酶与底物2, 3-DHBP反应生成的产物HOPDA呈黄色(图 6A),这为实验结果的检测提供了良好的筛选标记。同时,经HPLC检测显示,不完全反应时,在260 nm处可同时检测到2, 3-DHBP和HOPDA的吸收峰(图 6B),后者保留时间较2, 3-DHBP提前约1.41 min,两种物质具有良好的分离度,且随着反应的进行HOPDA含量明显提高,2, 3-DHBP含量则逐渐减少。

|
| 图 6 BphC与2, 3-DHBP反应结果及HPLC检测图谱 Figure 6 Results of reaction for BphC and 2, 3-DHBP and HPLC detection 注:A:2, 3-DHBP在BphC作用下生成黄色产物,图中S代表底物与缓冲液混合物,E为酶与缓冲液混合物,S+E为完整的反应体系;B:不完全反应时HPLC检测结果. Note: A is for 2, 3-DHBP turned to be yellow under BphC, where S is for substrate with buffer, E is for the enzyme with buffer while S+E is for the complete system of the reaction; B is for the HPLC results for the incomplete reation. |
|
|
以2, 3-DHBP为底物在不同条件下分别测定BphC的酶活并确定其最适反应pH与温度。根据各溶液的缓冲能力不同设置了不同的pH缓冲体系,各体系间pH均设有重复区域。结果显示,在相对较小的范围内不同缓冲液pH的变化未引起BphC酶活的明显改变,且不同pH缓冲液对酶活的影响基本与图 7A中黑色虚线所示的趋势一致。因此推测,不同pH的缓冲体系并非影响酶活的主要因素。实验表明,BphC在pH 7.4时表现出最高酶活力,在pH 6.6-8.8时其相对酶活均高于85%,表明该酶可在较宽的pH范围内实现对底物2, 3-DHBP的环裂解作用。然而,pH低于4.5或高于9.2时其酶活明显下降。事实上,当pH低于4.5或高于9.2时,反应体系中加入酶液后迅速有白色沉淀出现,推测可能是过酸或过碱条件下蛋白质已发生变性,从而失活,不能催化底物降解。

|
| 图 7 BphC与2, 3-DHBP反应时pH和温度对酶活的影响 Figure 7 Effects of pH and temperatures on BphC activity 注:A:30 ℃时BphC在不同pH范围内的酶活;B:pH 7.4时BphC在不同温度下的酶活. Note:A: The realative activity of BphC at 30 ℃ in different pH; B: The relative activity at different temperatures in pH 7.4. |
|
|
在pH 7.4时,以2, 3-DHBP为底物在不同温度条件下分别测定BphC的酶活并确定其最适反应温度。结果显示,30 ℃时该酶表现出最高酶活力,当反应温度高于或低于30 ℃时酶活均呈现下降趋势(图 7B)。然而,当温度在25−40 ℃范围内时,其相对酶活仍保持在70%以上,表明该酶具有较宽的耐受温度范围。
2.3.2 金属离子对BphC酶活的影响:通常,外二醇类双加氧酶活性中心含有非血红素Fe(Ⅱ),然而一定条件下部分该类酶中的Fe(Ⅱ)可被其他金属离子所取代,从而影响酶的催化活性。为探究不同金属离子对BphC酶活力的影响,在其最适反应条件下反应前分别加入不同金属离子至2 mmol/L,以不加任何金属离子时酶活为100%作对照,利用分光光度计检测其相对酶活。结果如表 1所示,添加Fe2+、Cu2+与Cd2+均可提高BphC酶的催化活力,其中Cu2+对该酶活力稍有提高,Fe2+与Cd2+对BphC酶活促进作用明显,约提高2.4-3.2倍。Fe3+则对该酶活力影响不大,其余金属离子均明显抑制该酶活性。同时,EDTA抑制酶活力作用明显,推测EDTA对酶活中心的Fe(Ⅱ)具有较强的螯合作用。事实表明,BphC酶活力受其他金属离子及金属离子螯合剂等影响较为明显,且该酶的催化活性需要Fe2+参与。同时,根据实验结果推测,在部分金属离子(如Cu2+、Cd2+等)污染环境中,该酶仍可能发挥较好的酶促作用,在环境污染物的修复作用中具有一定的应用价值。
| 金属离子 Metal ions |
相对酶活 Relative activity (%) |
| Fe2+ | 322.87±7.62 |
| Fe3+ | 98.56±10.43 |
| Cu2+ | 178.72±4.32 |
| Mg2+ | 71.15±1.25 |
| Ca2+ | 59.19±2.43 |
| Mn2+ | 54.77±5.87 |
| Zn2+ | 65.34±6.90 |
| Ni2+ | 62.14±0.95 |
| Cd2+ | 239.86±2.16 |
| Co2+ | 49.09±4.58 |
| EDTA | 43.52±6.02 |
由于HOPDA不易从2, 3-DHBP酶解产物中直接提取或进行检测,因此利用HPLC法首先测得2, 3-DHBP经BphC酶解后的降解率,再根据酶解反应式转换为HOPDA的生成率。利用不同浓度的2, 3-DHBP与BphC在最适条件下反应1 min后,根据Lineweaver-Burk双倒数法,以1/[S]为横坐标、1/[v]为纵坐标计算BphC酶的酶促动力学参数。结果显示,BphC酶的米氏常数Km为8.67 mmol/L,表明该酶对底物具有较高的亲和力。当底物浓度[S]达到一定值时其反应速度可达最大值为27.32 μmol/s,且当酶被底物所饱和时,其每秒钟可转换底物2, 3-DHBP的分子数为15.55;同时,该酶催化效率kcat/Km较部分已报道同类酶的催化效率更高[25-26],如表 2所示。
| 菌株 Strains |
Km (mmol/L) |
Vmax (μmol/s) |
kcat (s-1) |
kcat/Km (L/(mmol·s)) |
| BphC_YC-RL1 | 8.67 | 27.32 | 15.55 | 1 790 |
| BphC_P6 | 29.40 | - | 9.80 | 330 |
| BphC_R04 | 60.00 | - | 17.00 | 280 |
| 注:-:参考文献中未给出数据. Note: -: no data given in the references. | ||||
微生物在环境污染治理中扮演着非常重要的角色,因此对环境中尤其是多氯联苯化合物的生物修复作用被认为是最有前景的一项应用技术[27]。目前,已有诸多报道从多氯联苯污染环境中分离到的降解菌株可经过联苯降解的上游途径实现对污染物的清除。其中,2, 3-二羟基联苯-1, 2-双加氧酶在这一过程中起到了至关重要的作用[28-30],可使羟基苯环发生断裂从而进入下游代谢途径,该酶是微生物对联苯等有氧代谢的关键限速酶。然而,目前多氯联苯类化合物的生物降解研究仍存在降解菌种单一、降解基因不够丰富及降解底物谱不够广泛等问题[31],因此对该类化合物的生物降解研究仍然具有重要意义。
本实验利用节杆菌YC-RL1通过相关基因克隆与蛋白质的原核表达获得了重组蛋白,通过亲和层析等方法对其进行纯化并检测了相关的酶活特性。结果表明,该酶与底物2, 3-DHBP作用时的最适反应条件为pH 7.4、30 ℃,且其活性受不同金属离子的影响明显,其中Fe2+、Cu2+及Cd2+均可明显促进其酶反应活性。此外,生物信息学等分析显示,该酶是一类含有一个非血红素Fe(Ⅱ)为单一辅因子的外二醇类双加氧酶,对其作用底物具有较高的亲和性。
研究表明,菌株YC-RL1中bphC基因对于多氯联苯类化合物的生物降解具有至关重要的作用,其编码的BphC酶是重要的芳香环裂解酶,不仅在环境污染治理中具有良好的应用前景,同时对于该类化合物降解途径等的深入研究也具有重要的参考和指导意义。
| [1] | Pandey G, Chauhan A, Samanta SK, et al. Chemotaxis of a Ralstonia sp. SJ98 toward co-metabolizable nitroaromatic compounds[J]. Biochemical and Biophysical Research Communications, 2002, 299(3) : 404–409. DOI:10.1016/S0006-291X(02)02601-3 |
| [2] | Hu JX, Wang YL, Su XM, et al. Effects of RAMEB and/or mechanical mixing on the bioavailability and biodegradation of PCBs in soil/slurry[J]. Chemosphere, 2016, 155 : 479–487. DOI:10.1016/j.chemosphere.2016.04.084 |
| [3] | Stella T, Covino S, Čvančarová M, et al. Bioremediation of long-term PCB-contaminated soil by white-rot fungi[J]. Journal of Hazardous Materials, 2017, 324 : 701–710. DOI:10.1016/j.jhazmat.2016.11.044 |
| [4] | Leewis MC, Uhlik O, Leigh MB. Synergistic processing of biphenyl and benzoate: carbon flow through the bacterial community in polychlorinated-biphenyl-contaminated soil[J]. Scientific Reports, 2016, 6 : 22145. DOI:10.1038/srep22145 |
| [5] | Yang JM, Salmon AG, Marty MA. Development of TEFs for PCB congeners by using an alternative biomarker-thyroid hormone levels[J]. Regulatory Toxicology and Pharmacology, 2010, 56(2) : 225–236. DOI:10.1016/j.yrtph.2009.12.008 |
| [6] | Leewis MC, Reynolds CM, Leigh MB. Long-term effects of nutrient addition and phytoremediation on diesel and crude oil contaminated soils in subarctic Alaska[J]. Cold Regions Science and Technology, 2013, 96 : 129–137. DOI:10.1016/j.coldregions.2013.08.011 |
| [7] | Zhao QQ, Bai JH, Lu QQ, et al. Polychlorinated biphenyls (PCBs) in sediments/soils of different wetlands along 100-year coastal reclamation chronosequence in the Pearl River Estuary, China[J]. Environmental Pollution, 2016, 213 : 860–869. DOI:10.1016/j.envpol.2016.03.039 |
| [8] | Pieper DH. Aerobic degradation of polychlorinated biphenyls[J]. Applied Microbiology and Biotechnology, 2005, 67(2) : 170–191. DOI:10.1007/s00253-004-1810-4 |
| [9] | Gilbert ES, Crowley DE, et al. Plant compounds that induce polychlorinated biphenyl biodegradation by Arthrobacter sp. strain B1B[J]. Applied and Environmental Microbiology, 1997, 62(5) : 1933–1938. |
| [10] | Dhindwal S, Gomez-Gil L, Neau DB, et al. Structural basis of the enhanced pollutant-degrading capabilities of an engineered biphenyl dioxygenase[J]. Journal of Bacteriology, 2016, 198(10) : 1499–1512. DOI:10.1128/JB.00952-15 |
| [11] | Kotake T, Matsuzawa J, Suzuki-Minakuchi C, et al. Purification and partial characterization of the extradiol dioxygenase, 2′-carboxy-2, 3-Dihydroxybiphenyl 1, 2-dioxygenase, in the fluorene degradation pathway from Rhodococcus sp. strain DFA3[J]. Bioscience, Biotechnology, and Biochemistry, 2016, 80(4) : 719–725. DOI:10.1080/09168451.2015.1123605 |
| [12] | Vaillancourt FH, Haro MA, Drouin NM, et al. Characterization of extradiol dioxygenases from a polychlorinated biphenyl-degrading strain that possess higher specificities for chlorinated metabolites[J]. Journal of Bacteriology, 2003, 185(4) : 1253–1260. DOI:10.1128/JB.185.4.1253-1260.2003 |
| [13] | Ishida T, Senda T, Tanaka H, et al. Single-turnover kinetics of 2, 3-dihydroxybiphenyl 1, 2-dioxygenase reacting with 3-formylcatechol[J]. Biochemical and Biophysical Research Communications, 2005, 338(1) : 223–229. DOI:10.1016/j.bbrc.2005.08.218 |
| [14] | Hauschild JE, Masai E, Sugiyama K, et al. Identification of an Alternative 2, 3-dihydroxybiphenyl 1, 2-dioxygenase in Rhodococcus sp. strain RHA1 and Cloning of the Gene[J]. Applied and Environmental Microbiology, 1996, 62(8) : 2940–2946. |
| [15] | Yi L, Meggo R, Hu DF, et al. Enhanced polychlorinated biphenyl removal in a switchgrass rhizosphere by bioaugmentation with Burkholderia xenovorans LB400[J]. Ecological Engineering, 2014, 71 : 215–222. DOI:10.1016/j.ecoleng.2014.07.046 |
| [16] | Xiong F, Shuai JJ, Jin XF, et al. Expression and characterization of a recombinant 2, 3-dihydroxybiphenyl-1, 2-dioxygenase from Pseudomonas[J]. Molecular & Cellular Toxicology, 2012, 8(4) : 375–382. |
| [17] | Ren L, Jia Y, Ruth N, et al. Complete genome sequence of an aromatic compound degrader Arthrobacter sp. YC-RL1[J]. Journal of Biotechnology, 2015, 219 : 34–35. |
| [18] | Sambrock J, Russel DW. Molecular Cloning: A Laboratory Manual[M].3rd Edition. New York: Cold Spring Harbor Laboratory Press, 2001 . |
| [19] | Qiao C, Ren L, Jia Y, et al. Chemotaxis of Arthrobacter sp. CN2 towards p-nitrophenol[J]. Journal of Agro-Environment Science, 2016, 35(10) : 1945–1952. (in Chinese) 乔铖, 任磊, 贾阳, 等. 节杆菌(Arthrobacter sp.) CN2对对硝基苯酚的趋化性研究[J]. 农业环境科学学报, 2016, 35(10) : 1945–1952. DOI:10.11654/jaes.2016-0528 |
| [20] | Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987, 4(4) : 406–425. |
| [21] | Yam KC, D'Angelo I, Kalscheuer R, et al. Studies of a ring-cleaving dioxygenase illuminate the role of cholesterol metabolism in the pathogenesis of Mycobacterium tuberculosis[J]. PLoS Pathogens, 2009, 5(3) : e1000344. DOI:10.1371/journal.ppat.1000344 |
| [22] | Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72(1/2) : 248–254. |
| [23] | Ghim YS, Chang HN. Diffusional falsification of kinetic constants on Lineweaver-Burk plots[J]. Journal of Theoretical Biology, 1983, 105(1) : 91–102. DOI:10.1016/0022-5193(83)90426-5 |
| [24] | Vaillancourt FH, Bolin JT, Eltis LD. The ins and outs of ring-cleaving dioxygenases[J]. Critical Reviews in Biochemistry and Molecular Biology, 2006, 41(4) : 241–267. DOI:10.1080/10409230600817422 |
| [25] | Mckay DB, Prucha M, Reineke W, et al. Substrate specificity and expression of three 2, 3-dihydroxybiphenyl 1, 2-dioxygenases from Rhodococcus globerulus strain P6[J]. Journal of Bacteriology, 2003, 185(9) : 2944–2951. DOI:10.1128/JB.185.9.2944-2951.2003 |
| [26] | Yang XQ, Xie FH, Zhang GQ, et al. Purification, characterization, and substrate specificity of two 2, 3-dihydroxybiphenyl 1, 2-dioxygenase from Rhodococcus sp. R04, showing their distinct stability at various temperature[J]. Biochimie, 2008, 90(10) : 1530–1538. DOI:10.1016/j.biochi.2008.05.020 |
| [27] | Field JA, Sierra-Alvarez R. Microbial transformation and degradation of polychlorinated biphenyls[J]. Environmental Pollution, 2008, 155(1) : 1–12. DOI:10.1016/j.envpol.2007.10.016 |
| [28] | Fukuda K, Hosoyama A, Tsuchikane K, et al. Complete genome sequence of polychlorinated biphenyl degrader Comamonas testosteroni TK102 (NBRC 109938)[J]. Genome Announcements, 2014, 2(5) : e00865-14. DOI:10.1128/genomeA.00865-14 |
| [29] | Sun Y, Qian SJ. Characterization and identification of a biphenyl degrading strain[J]. Microbiology China, 2004, 31(6) : 23–26. (in Chinese) 孙艳, 钱世钧. 一株联苯降解菌的特性及鉴定[J]. 微生物学通报, 2004, 31(6) : 23–26. |
| [30] | Li C, Yuan G, Wu G, et al. Cloning of three 2, 3-dihydroxybiphenyl-1, 2-dioxygenase genes from Achromobacter sp. BP3 and the analysis of their roles in the biodegradation of bipheny[J]. Journal of Hazardous Materials, 2013, 261 : 246–252. DOI:10.1016/j.jhazmat.2013.07.019 |
| [31] | Li A, Qu YY, Zhou JT, et al. Enzymatic biodegradation of 2, 3-dihydroxybiphenyl and amplification of encoding enzyme gene[J]. Journal of Dalian University of Technology, 2009, 49(4) : 494–498. (in Chinese) 李昂, 曲媛媛, 周集体, 等. 2, 3-二羟基联苯酶促降解及编码酶基因扩增[J]. 大连理工大学学报, 2009, 49(4) : 494–498. DOI:10.7511/dllgxb200904005 |
 2017, Vol. 44
2017, Vol. 44




