扩展功能
文章信息
- 戎振, 霍颖异, 徐曹狄, 简书令, 王春生, 许学伟
- RONG Zhen, HUO Ying-Yi, XU Cao-Di, JIAN Shu-Ling, WANG Chun-Sheng, XU Xue-Wei
- 来源于海洋细菌Altererythrobacter epoxidivorans CGMCC 1.7731T的新型碱性酯酶E22的酶学性质
- Characterization of a novel alkaline esterase (E22) from a marine bacterium Altererythrobacter epoxidivorans CGMCC 1.7731T
- 微生物学通报, 2017, 44(7): 1622-1630
- Microbiology China, 2017, 44(7): 1622-1630
- DOI: 10.13344/j.microbiol.china.170167
-
文章历史
- 收稿日期: 2017-02-28
- 接受日期: 2017-05-31
- 优先数字出版日期(www.cnki.net): 2017-06-06
广义上的酯酶(EC 3.1.1.X)是指能催化酯键水解和形成的酶类,又称脂类水解酶;狭义的酯酶(EC 3.1.1.2) 是指可催化水解短链酰基甘油的酶类[1]。通过对数据库中微生物来源的脂类水解酶氨基酸序列和催化作用特点的分析,Arpigny和Jaeger将脂类水解酶分为8个家族,不同家族通常有其独特的保守基序。作为一种α/β水解酶,酯酶通常具有由丝氨酸、组氨酸和天冬氨酸3个氨基酸残基组成的催化三联体,在催化酯键断裂或还原的反应中作为活性中心[2]。酯酶因其较广的底物谱、良好的有机溶剂耐受性和独特的手性选择性等特点而广泛应用于食品、纺织、制药以及化妆品等工业生产领域[3]。为了适应生产过程中的一些相对极端条件,例如高温、高盐、极酸、极碱或高浓度有机溶剂,寻找能够适应极端条件的新型酯酶已成为研究热点之一[4]。
酯酶存在于绝大多数生物体内,而目前作为工业催化剂的酯酶多分离于真菌或细菌[3, 5],这不仅是因为微生物来源酯酶具有产量高、成本低和操作简便等优点[6],更重要的是,从适应极端条件的微生物中更容易获得具有独特性质的酯酶。海洋是一个天然的微生物资源宝库,海洋来源的酯酶通常具有与海洋环境相关的优良性质,例如温度稳定性、耐盐性、耐碱性、耐低温、以及优异的手性选择性等[7],从海洋微生物中筛选具有独特性质的酯酶已成为开发新型工业用酶的一个重要方向。随着研究深入,越来越多海洋来源的酯酶被人们发现[8-10]。
本研究通过对海洋来源菌株Altererythrobacter epoxidivorans CGMCC 1.7731T的全基因组序列分析[11],筛选出一个假定的酯酶基因e22。以pET-28a为载体将其转入大肠杆菌BL21(DE3) 细胞中异源表达,纯化并研究重组酶的底物特异性、最适反应条件、热稳定性、变性剂和有机溶剂耐受等酶学性质。
1 材料与方法 1.1 菌株、质粒和培养基菌株Altererythrobacter epoxidivorans CGMCC 1.7731T分离自日本鹿儿岛海湾冷泉区沉积物[12],购买于中国普通微生物菌种保藏管理中心(China General Microbiological Culture Collection Center,CGMCC)。质粒pET-28a、菌株Escherichia coli BL21(DE3) 和E. coli DH5α为本实验室保藏。海洋培养基2216[13]用于培养菌株A. epoxidivorans CGMCC 1.7731T,LB培养基用于培养E. coli[14]。
1.2 主要试剂和仪器限制性内切酶、T4 DNA连接酶,New England Biolabs公司(NEB);Prime STAR GXL DNA聚合酶,宝生物工程有限公司(TaKaRa);分子克隆所需试剂盒,OMEGA公司;对硝基苯酚酯、对硝基苯酚标准溶液,Sigma-Aldrich公司;亲和层析柱及填料,美国GE Healthcare公司;其他相关色谱级或分析级试剂,生工生物工程(上海)股份有限公司。PCR基因扩增仪(XP cycles),杭州博日科技有限公司;微型离心机(MICROCL 17) 和低温落地式中型离心机(SL40R),赛默飞世尔科技(中国)有限公司;超声波细胞破碎仪(650Y),上海比朗仪器制造有限公司;核酸蛋白分析仪(DU800),贝克曼库尔特商贸(中国)有限公司。
1.3 序列分析筛选菌株A. epoxidivorans CGMCC 1.7731T全基因组注释结果[11],获得一个假定的酯酶基因e22。使用BLASTp程序(http://blast.ncbi.nlm.nih.gov/)将其氨基酸序列与同源酯酶序列进行比对分析。相对分子质量和等电点(Isoelectric point,pI)通过ExPASy数据库(http://www.expasy.org/proteomics)预测。信号肽采用SignalP 4.1 Server (http://www.cbs.dtu.dk/services/SignalP/)预测。多序列比对分析后,采用MEGA 7.0[15]基于Neighbor-Joining方法构建系统发育树。
1.4 克隆与表达以A. epoxidivorans CGMCC 1.7731T基因组DNA为模板,通过PCR扩增目的基因。PCR体系(50 μL):DNA模板1 μL;Prime STAR GXL DNA聚合酶2 μL;2.5 mmol/L dNTPs 4 μL;10 μmol/L上下游引物各1 μL;5×Prime STAR GXL Buffer 10 μL;ddH2O 31 μL。PCR扩增条件:98℃ 10 min;98℃ 15 s,60℃ 15 s,68℃ 35 s,30个循环;68℃ 10 min。引物序列为:上游引物5′-TCGCGAGCTC ATGGCTGTTGGCAGCTGGTCG-3′和下游引物5′-TCCGGCGGCCGCTCAATCGAGCGCTTC-3′,其中限制酶切位点Sac Ⅰ和Not Ⅰ分别由下划线标出。将扩增获得的目的基因与质粒pET-28a同时用Sac Ⅰ和Not Ⅰ限制酶消化后连接,得到的重组质粒pET-28a-e22转化入E. coli DH5α细胞并扩增,提取质粒并测序验证是否为正确克隆。序列验证后,将重组质粒转化入E. coli BL21(DE3) 细胞中培养表达。待培养液OD600达到0.6-0.8,加入0.5 mmol/L IPTG诱导,16℃、200 r/min培养20 h。4℃、5 000×g离心15 min收集细胞。
1.5 酯酶纯化收集的菌体采用Buffer A (500 mmol/L氯化钠,10 mmol/L咪唑,20 mmol/L Tris-HCl,pH 8.0) 重悬,并采用超声波破胞。破胞液于4℃、12 000×g高速离心20 min后,通过Ni-NTA亲和层析柱纯化蛋白。纯化过程为:采用Buffer A平衡亲和层析柱后,将离心后的上清液加入层析柱与Ni介质结合,再采用Buffer B (500 mmol/L氯化钠,50 mmol/L咪唑,20 mmol/L Tris-HCl,pH 8.0) 和Buffer C (500 mmol/L氯化钠,250 mmol/L咪唑,20 mmol/L Tris-HCl,pH 8.0) 依次洗脱蛋白。使用浓度为12.5%的SDS-PAGE检测洗脱液中蛋白量及纯度,并用Bradford[16]方法测定蛋白浓度。
1.6 酶活性质研究酯酶催化水解对硝基苯酚酯生成对硝基苯酚,采用核酸蛋白分析仪(Beckman DU800) 测定2 min内生成的对硝基苯酚数值,根据其在一定波长下的吸光值变化计算酯酶活力值。反应的标准体系为:10 μL 0.1 mol/L对硝基苯酚丁酸酯,980 μL 100 mmol/L Ches-NaOH缓冲液(pH 9.5) 和10 μL纯化后的酶液,总体系为1 ml;反应温度为35℃,测量光波长为405 nm[17](若无另外说明,反应均在此波长和温度下测量)。每次测量设置3个平行实验组,并以加入灭活酶的体系作为对照组。一个酶活力单位定义为:每分钟从对硝基苯酚酯催化产生1 μmol对硝基苯酚需要的酶量。用100 mmol/L Ches-NaOH缓冲液(pH 9.5) 配置对硝基苯酚标准品0.01-0.1 mmol的浓度梯度,分别测量其在35℃及405 nm波长下的吸光值,绘制标准曲线并计算得到对硝基苯酚的消光系数为14.1 L/(mmol·cm)[18]。数据处理及图形绘制利用软件GraphPad Prism 6[19]完成。
1.6.1 底物特异性:在55℃和pH 10.5条件下,反应体系中加入终浓度为1 mmol/L不同链长度的对硝基苯酚酯,具体包括:乙酸酯,C2;丁酸酯,C4;己酸酯,C6;辛酸酯,C8;癸酸酯,C10;十二酸酯,C12;十四酸酯,C14;十六酸酯,C16。
1.6.2 最适反应pH:在35℃条件下(避免高温对不同缓冲液pH值的影响)连续测定不同pH条件348 nm波长(对硝基苯酚非pH依赖的等吸光点)[17]的吸光值。所用缓冲液包括:100 mmol/L磷酸二氢钾-磷酸氢二钾缓冲液(pH 7.0-7.5)、100 mmol/L Tris-HCl缓冲液(pH 7.5-9.0) 和100 mmol/L Ches-NaOH缓冲液(pH 9.0-10.5)。
1.6.3 最适反应温度与热稳定性:分别将标准体系置于不同温度(15-60℃,间隔5℃)下反应,连续测定其吸光值。将纯化后的酶液分别置于40、50和60℃孵育1 h和2 h,在最适反应温度55℃条件下分别测定剩余活性。
1.6.4 金属离子及EDTA耐受性:为认识不同金属离子对酶的活力影响,在标准反应体系中分别加入10 mmol/L的Sr2+、Mg2+、Ca2+、Ba2+或乙二胺四乙酸(EDTA)[20],测定酶活力。对照组为不加金属离子的标准反应体系。
1.6.5 有机溶剂及去垢剂耐受性:为认识不同有机溶剂及去垢剂对酶的活力影响,在标准反应体系中分别加入1%的异丙醇(Isopropanol)、乙腈(Acetonitrile)、甲醇(Methanol)、乙醇(Ethanol)、丙酮(Acetone)、二甲基亚砜(Dimethyl sulfoxide,DMSO)、二甲基甲酰胺(Dimethyl formamide,DMF)或甘油(Glycerol)等有机溶剂,0.1%的十二烷基硫酸钠(Sodium dodecyl sulfate,SDS)或1%的聚乙二醇辛基苯基醚(Triton X-100)、吐温-20 (Tween-20) 或吐温-80 (Tween-80) 等去垢剂,测定酶活力。对照组为不加有机溶剂及去垢剂的标准反应体系。
1.7 核苷酸序列登录号A. epoxidivorans CGMCC 1.7731T的酯酶基因e22核苷酸序列的GenBank登录号为WP 061923136中的Locus_tag AMC99_00842,其氨基酸序列登录号为ALE16145。
2 结果与分析 2.1 序列分析通过对A. epoxidivorans CGMCC 1.7731T基因组注释,筛选获得的e22基因,其为一个693 bp的开放阅读框,编码230个氨基酸,预测E22其相对分子质量为25 kD,pI为4.26。SignalP 4.1 Server分析结果显示,E22具有一个信号肽,切割位点位于序列中的17和18位,期望值为0.579。通过NCBI上BLASTp比对,结果显示E22的氨基酸序列与来自菌株Erythrobacter sp. AP23的假定酯酶arylesterase (WP 067695227) 序列一致性最高,达到64%。将E22与不同家族酯酶的氨基酸序列进行比对后构建系统发育树,结果显示E22属于酯酶第二家族(Family Ⅱ) (图 1),ClustalW比对结果表明,它们具有相同的代表第二家族酯酶特征的GDSL基序(E22的145-148位氨基酸)[2]。

|
| 图 1 E22与其他脂类水解酶的系统发育树 Figure 1 Phylogenetic tree of E22 and other lipolytic enzymes 注:括号内数字为GenBank登录号;各个分支处数值表示基于1 000次重复取样的Bootstrap支持率;标尺表示氨基酸替换率;罗马数字代表分支上的蛋白所属的家族. Note: The numbers in the bracket denote the GenBank accession number; bootstrap values are based on 1 000 replicates; bar scale indicates 0.20 amino acid substitutions per site; and the numbers represent family of lipolytic enzyme which proteins on the branch belong to. |
|
|
酯酶基因e22与pET-28a连接后,重组质粒转化入E. coli DH5α扩增并验证测序。而后将重组质粒转入E. coli BL21(DE3) 中诱导表达,蛋白纯化后的SDS-PAGE结果显示,重组蛋白E22的相对分子质量约为25 kD,与预测相符,如图 2所示。

|
| 图 2 酯酶E22的SDS-PAGE图 Figure 2 SDS-PAGE of E22 注:1-3:10、50或250 mmol/L咪唑溶液洗脱的流出液;M:标准分子量蛋白Marker. Note: 1-3: Elution of 10, 50 or 250 mmol/L imidazole; M: Marker. |
|
|
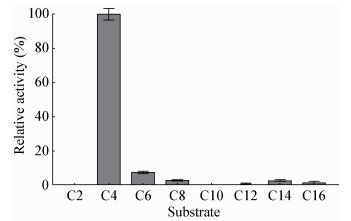
选用不同侧链长度的对硝基苯酚酯(C2-C16) 作为底物,分析酯酶E22的底物特异性,结果如图 3所示。酯酶E22主要对短链底物如对硝基苯酚丁酸酯(C4) 具有显著催化水解作用,其水解反应活性最强(酶活力值为25 U/mg),约为C6作底物时的10倍。E22对短链底物具有的高水解能力表明其是一个酯酶而不是脂肪酶[1]。
|
| 图 3 酯酶E22的底物特异性 Figure 3 Substrate specificity of E22 注:在55℃和pH 10.5条件下测定酶活力值,以对硝基苯酚丁酸酯(C4) 为底物时酶活为100%. Note: The esterase activity of the purified recombinant enzyme E22 towards various chain lengths of p-NP esters was assayed at 55℃ and pH 10.5. The highest level of activity with p-nitrophenol butyrate was taken as 100%. |
|
|
图 4显示E22是一个碱性酯酶,在pH为10.5的情况下酶活力依然在增长。选用了Britton-Robinson缓冲液[21]测定pH 11.0、11.5和12.0条件下的酶活力,但由于底物在高pH条件下底物对硝基苯酚丁酸酯自降解速度快导致无法测量光吸收值。因此在可测定的pH范围内,E22在pH 10.5条件下具有最高水解活性。

|
| 图 4 pH对E22活性的影响 Figure 4 Effect of different pH on the activity of E22 注:以对硝基苯酚丁酸酯为底物,35℃条件下测定不同pH时的酶活力值. Note: The activity was determined using p-nitrophenol butyrate as substrate. The assay was performed under 35℃. |
|
|
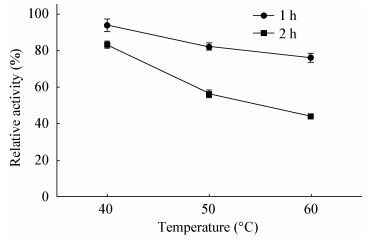
最适反应温度的实验结果如图 5所示,E22在15-60℃范围内均有酶活,且在55℃条件下发挥最高水解活性。热稳定性的实验结果表明,在40-60℃条件下孵育1 h对E22酶活力影响不大,仍具有80%以上的酶活力;30℃和40℃孵育2 h后酶活力仍具有80%以上,60℃孵育2 h后具有45%的酶活力,说明酯酶E22具有良好的热稳定性(图 6)。

|
| 图 5 温度对E22活性的影响 Figure 5 The activity of E22 under different temperatures 注:以对硝基苯酚丁酸酯为底物,在标准条件下测定不同温度下的酶活力值,将55℃条件下测得的酶活作为100%. Note: Enzyme activity was determined with a series of temperatures at pH 9.5 using p-nitrophenol butyrate as substrate. The value obtained at 55℃ was defined as 100%. |
|
|
|
| 图 6 E22的热稳定性研究 Figure 6 Thermostability of E22 |
|
|
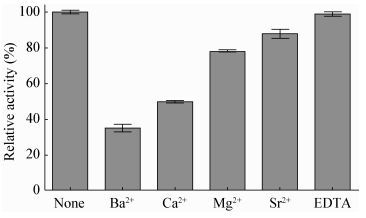
二价金属阳离子的加入对E22的酶活力有不同程度的抑制作用(图 7)。相比而言,Mg2+和Sr2+对酯酶活性的影响较小,E22能够保留78%以上的活力;Ba2+强烈抑制E22的催化水解能力,仅保留34%左右的酶活力。有机溶剂耐受性实验中(图 8),加入异丙醇后底物水解反应速度快,导致无法准确测量数值,其他有机溶剂的测量过程均正常,其中仅乙腈、二甲基甲酰胺和甘油对E22有轻微的抑制作用,丙酮、乙醇和二甲基亚砜对酶活力无影响,而甲醇将E22活性提高了30%以上。去垢剂影响实验中(图 8),0.1%的SDS和1%的Triton X-100可显著提高酶活力值。结果表明,E22具有较好的有机溶剂及去垢剂耐受性。
|
| 图 7 二价金属阳离子和EDTA对E22活性的影响 Figure 7 Effect of metal ions and EDTA on activity of E22 |
|
|

|
| 图 8 有机溶剂及去垢剂对E22活性的影响 Figure 8 Effect of detergents and organic solvents on activity of recombinant E22 |
|
|
近年来,围绕新型酯酶的研究工作取得了长足进步,以海洋微生物为研究对象,研究者们从中筛选出了许多性质各异的酯酶[22-24]。尤其是海洋中一些极端环境促进了生物的某些定向进化,使得孕育其中的微生物往往能够表达具有极端性质的酯酶[24-25]。以海洋极端环境中的微生物作为研究对象已成为获取新型酯酶重要方向之一。
在本研究中,我们以分离自海底沉积物的赤杆菌科菌株A. epoxidivorans CGMCC 1.7731T为研究对象,克隆表达了一个新型酯酶基因e22,并研究了其酶学性质。氨基酸序列分析结果表明,E22属于脂类水解酶第二家族,该家族酯酶的催化机制与其他家族相比较为独特,其活性位点在与不同底物的结合时可以改变构象,具有较高灵活性,这使得不同的底物能以最佳构象与酶结合进行催化水解反应[2]。这一特性对于理解具有多重底物或多功能特性的酶催化原理具有重要意义。酶学性质的鉴定结果表明,酯酶E22具有一些优良特性。首先,催化反应最适pH实验结果表明,E22是一种新型碱性酯酶,其对碱性的偏好在同源的Family Ⅱ酯酶中并不常见(表 1)。将E22与近年来已公布的海洋来源碱性酯酶相比(表 2),它具有较高的催化反应最适pH值。E22在耐碱性方面的独特性质,使其在碱性条件下的相关催化过程中具有应用潜力[36]。其次,海底低温的生长环境通常会造成酶能适应低温环境[37],但仍有些酶的最适温度较高且热稳定性差[38];来源于海底冷泉区沉积物的E22具有较高的催化反应最适温度(55℃)和良好的热稳定性(60℃孵育2 h后检测到51%剩余活性),将其与同为海底沉积物来源的Est_p1[31]、EstF[32]、EM2L8[33]、Est12[34]以及Est4[35]相比,结果显示E22在热稳定性方面具有较大优势。第三,E22具有良好的有机溶剂及去垢剂耐受性。实际工业生产中,酯酶催化酯键水解、合成或交换反应常在非纯水性环境中进行,酶在有机溶剂存在下的高失活率限制了其在工业生产中的应用[39]。有机溶剂影响实验表明,在一些有机溶剂(1%)存在情况下E22酶活力不受或仅受轻微影响,甚至部分有机溶剂能够促进其酶活力提高。此外,E22还表现出耐受去垢剂SDS和Triton X-100的能力,这也增加了它在未来工业应用中的潜力。
| 名称 Name |
菌株来源 Resource of strain |
最适底物 Optimal substrate |
底物谱 Substrate range |
最适温度 Optimal temperature (℃) |
最适pH Optimal pH |
60℃孵育2 h剩余活性 Residual activity after incubated for 2 h (%) |
| E22 | A. epoxidivorans | C4 | C4-C16 | 55 | 10.5 | 46 |
| EstSL3[26] | Alkalibacterium sp. SL3 | C2 | C2-C6 | 30 | 9.0 | <10 |
| EstS9N[27] | Pseudomonas sp. S9 | C4 | C2-C8 | 35 | 9.0 | 95-100 |
| EstL5[28] | Geobacillus thermodenitrificans T2 | C4 | C2-C8 | 60 | 8.0 | 75-80 |
| EstHE1[29] | Unidentified bacterium | C2 | C2-C6 | 40 | 7.4 | 80 |
| EstA[30] | Serratia liquefaciens MG1 | C4 | C2-C6 | NR | 7.0 | NR |
| 注:酯酶剩余活性均在最适条件下测定;NR:文献中缺少相关资料. Note: Residual activity of esterases were measured under optimal conditions; NR: Not record in the literature. | ||||||
| 名称 Name |
菌株来源 Resource of strain |
最适底物 Optimal substrate |
最适温度 Optimal temperature (℃) |
最适pH Optimal pH |
热稳定性 Thermostability |
|||
| 孵育温度 Incubation temperature (℃) |
孵育时间 Incubation time (h) |
反应条件 Reaction condition |
剩余活性 Residual activity (%) |
|||||
| E22 | A. epoxidivorans | C4 | 55 | 10.5 | 40 | 2 | 55℃ pH 10.5 |
88 |
| 60 | 2 | 46 | ||||||
| RhLip[24] | Rhodococcus sp. | C4 | 30 | 11.0 | 50 | 2 | 30℃ pH 10.0 |
20 |
| Est_p1[31] | Marine sediment metagenome | C4 | 40 | 8.57 | 40 | 2 | 40℃ pH 8.57 |
<40 |
| EstF[32] | Marine sediment metagenome | C4 | 50 | 9.0 | 40 | 1 | 45℃ pH 8.0 |
85-90 |
| 60 | 1 | 0 | ||||||
| EM2L8[33] | Marine sediment metagenome | C4 | 50 | 10.0 | NR | NR | NR | NR |
| Est12[34] | Marine sediment metagenome | C6 | 50 | 9.0 | 40 | 1 | 40℃ pH 9.0 |
85-90 |
| 60 | 1 | 0 | ||||||
| Est4[35] | Marine mud metagenome | C4 | 45 | 8.0 | 40 | 2 | 30℃ pH 8.0 |
90-95 |
| 60 | 2 | <30 | ||||||
| 注:NR:文献中缺少相关资料. Note: NR: Not record in the literature. | ||||||||
综上所述,酯酶E22具有作为工业用酶的潜力。随着研究深入,其功能与结构的内在机理将可能更清晰,通过对其催化机制认识将可促进工业酯酶的理性设计,为酯酶的定向改造提供理论指导。
| [1] | Arpigny JL, Jeager KE. Bacterial lipolytic enzymes: classification and properties[J]. Biochemical Society, 1999, 343(1) : 177–183. DOI:10.1042/bj3430177 |
| [2] | Akoh CC, Lee GC, Liaw YC, et al. GDSL family of serine esterases/lipases[J]. Progress in Lipid Research, 2004, 43(6) : 534–552. DOI:10.1016/j.plipres.2004.09.002 |
| [3] | Bornscheuer UT. Microbial carboxyl esterases: classification, properties and application in biocatalysis[J]. FEMS Microbiology Reviews, 2002, 26(1) : 73–81. DOI:10.1111/j.1574-6976.2002.tb00599.x |
| [4] | Gupta MN, Roy I. Enzymes in organic media. Forms, functions and applications[J]. European Journal of Biochemistry, 2004, 271(13) : 2575–2583. DOI:10.1111/(ISSN)1432-1033 |
| [5] | Jaeger KE, Dijkstra BW, Reetz MT. Bacterial biocatalysts: molecular biology, three-dimensional structures, and biotechnological applications of lipases[J]. Annual Review of Microbiology, 1999, 53(1) : 315–351. DOI:10.1146/annurev.micro.53.1.315 |
| [6] | Houde A, Kademi A, Leblanc D. Lipases and their industrial applications[J]. Applied Biochemistry and Biotechnology, 2004, 118(1/3) : 155–170. |
| [7] | Antranikian G, Egorova K. Extremophiles, a unique resource of biocatalysts for industrial biotechnology//Gerday C, Glansdorff N. Physiology and Biochemistry of Extremophiles[M]. Washington: ASM Press, 2007: 361-406 |
| [8] | Jiang H, Zhang SW, Gao HF, et al. Characterization of a cold-active esterase from Serratia sp. and improvement of thermostability by directed evolution[J]. BMC Biotechnology, 2016, 16 : 7. DOI:10.1186/s12896-016-0235-3 |
| [9] | Zheng JY, Liu YY, Luo WF, et al. Biocatalytic resolution of Rac-α-Ethyl-2-Oxo-Pyrrolidineacetic acid methyl ester by immobilized recombinant Bacillus cereus esterase[J]. Applied Biochemistry and Biotechnology, 2015, 178(8) : 1471–1480. |
| [10] | Rahman MA, Culsum U, Tang WH, et al. Characterization of a novel cold active and salt tolerant esterase from Zunongwangia profunda[J]. Enzyme and Microbial Technology, 2016, 85 : 1–11. DOI:10.1016/j.enzmictec.2015.12.013 |
| [11] | Li ZY, Wu YH, Huo YY, et al. Complete genome sequence of a benzo[a]pyrene-degrading bacterium Altererythrobacter epoxidivorans CGMCC 1. 7731T[J]. Marine Genomics, 2016, 25 : 39–41. DOI:10.1016/j.margen.2015.11.009 |
| [12] | Kwon KK, Woo JH, Yang SH, et al. Altererythrobacter epoxidivorans gen. nov., sp. nov., an epoxide hydrolase-active, mesophilic marine bacterium isolated from cold-seep sediment, and reclassification of Erythrobacter luteolus Yoon et al. 2005 as Altererythrobacter luteolus comb. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(10) : 2207–2011. DOI:10.1099/ijs.0.64863-0 |
| [13] | Zobell CE. Studies on marine bacteria. I. The cultural requirements of heterotrophic aerobes[J]. Journal of Marine Research, 1941, 4 : 42–75. |
| [14] | Sezonov G, Joseleau-Petit D, D'ari R. Escherichia coliphysiology in Luria-Bertani broth[J]. Journal of Bacteriology, 2007, 189(23) : 8746–8749. DOI:10.1128/JB.01368-07 |
| [15] | Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7) : 1870–1874. DOI:10.1093/molbev/msw054 |
| [16] | Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72(1/2) : 248–254. |
| [17] | Manco G, Gennaro SD, Rosa MD, et al. Purification and characterization of a thermostable carboxylesterase from the thermoacidophilic eubacterium Bacillus acidocaldarius[J]. European Journal of Biochemistry, 1994, 221(3) : 965–972. DOI:10.1111/ejb.1994.221.issue-3 |
| [18] | Quinn DM, Shirai K, Jackson RL, et al. Lipoprotein lipase catalyzed hydrolysis of water-soluble p-nitrophenyl esters. Inhibition by apolipoprotein C-Ⅱ[J]. Biochemistry, 1982, 21(26) : 6872–6879. DOI:10.1021/bi00269a038 |
| [19] | Swift ML. GraphPad prism, data analysis, and scientific graphing[J]. Journal of Chemical Information and Computer Sciences, 1997, 37(2) : 411–412. DOI:10.1021/ci960402j |
| [20] | Naika GS, Tiku PK. Influence of ethylenediaminetetraacetic acid (EDTA) on the structural stability of endoglucanase from Aspergillus aculeatus[J]. Journal of Agricultural and Food Chemistry, 2011, 59(13) : 7341–7345. DOI:10.1021/jf103889m |
| [21] | Alghamdi AF, Alkhalaf AS, Alkhalaf AS. High perfomance liquid chromatographic determantion of glimepiride and timolol in pharmaceutical formulations using Britton-Robinson buffer in mobile phases[J]. Moroccan Journal of Chemistry, 2016, 4(2) : 454–460. |
| [22] | Dukunde A, Schneider D, Lu MJ, et al. A novel, versatile family IV carboxylesterase exhibits high stability and activity in a broad pH spectrum[J]. Biotechnology Letters, 2017, 39(4) : 577–587. DOI:10.1007/s10529-016-2282-1 |
| [23] | Hu YF, Fu CZ, Huang YP, et al. Novel lipolytic genes from the microbial metagenomic library of the South China Sea marine sediment[J]. FEMS Microbiology Ecology, 2010, 72(2) : 228–237. DOI:10.1111/fem.2010.72.issue-2 |
| [24] | de Santi C, Tedesco P, Ambrosino L, et al. A new alkaliphilic cold-active esterase from the psychrophilic marine bacterium Rhodococcus sp.: functional and structural studies and biotechnological potential[J]. Applied Biochemistry and Biotechnology, 2014, 172(6) : 3054–3068. DOI:10.1007/s12010-013-0713-1 |
| [25] | Zarafeta D, Moschidi D, Ladoukakis E, et al. Metagenomic mining for thermostable esterolytic enzymes uncovers a new family of bacterial esterases[J]. Scientific Reports, 2016, 6 : 38886. DOI:10.1038/srep38886 |
| [26] | Wang GZ, Wang QH, Lin XJ, et al. A novel cold-adapted and highly salt-tolerant esterase from Alkalibacterium sp. SL3 from the sediment of a soda lake[J]. Scientific Reports, 2016, 6 : 19494. DOI:10.1038/srep19494 |
| [27] | Wicka M, Wanarska M, Krajewska E, et al. Cloning, expression, and biochemical characterization of a cold-active GDSL-esterase of a Pseudomonas sp. S9 isolated from Spitsbergen island soil[J]. Acta Biochimica Polonica, 2016, 63(1) : 117–125. DOI:10.18388/abp.2015_1074 |
| [28] | Yang ZX, Zhang Y, Shen TT, et al. Cloning, expression and biochemical characterization of a novel, moderately thermostable GDSL family esterase from Geobacillus thermodenitrificans T2[J]. Journal of Bioscience and Bioengineering, 2013, 115(2) : 133–137. DOI:10.1016/j.jbiosc.2012.08.016 |
| [29] | Okamura Y, Kimura T, Yokouchi H, et al. Isolation and characterization of a GDSL esterase from the metagenome of a marine sponge-associated bacteria[J]. Marine Biotechnology, 2010, 12(4) : 395–402. DOI:10.1007/s10126-009-9226-x |
| [30] | Riedel K, Talker-Huiber D, Givskov M, et al. Identification and characterization of a GDSL esterase gene located proximal to the swr quorum-sensing system of Serratia liquefaciens MG1[J]. Applied and Environmental Microbiology, 2003, 69(7) : 3901–3910. DOI:10.1128/AEM.69.7.3901-3910.2003 |
| [31] | Peng Q, Zhang X, Shang M, et al. A novel esterase gene cloned from a metagenomic library from neritic sediments of the South China Sea[J]. Microbial Cell Factories, 2011, 10 : 95. DOI:10.1186/1475-2859-10-95 |
| [32] | Fu CZ, Hu YF, Xie F, et al. Molecular cloning and characterization of a new cold-active esterase from a deep-sea metagenomic library[J]. Applied Biochemistry and Biotechnology, 2011, 90(3) : 961–970. |
| [33] | Park HJ, Jeon JH, Kang SG, et al. Functional expression and refolding of new alkaline esterase, EM2L8 from deep-sea sediment metagenome[J]. Protein Expression and Purification, 2007, 52(2) : 340–347. DOI:10.1016/j.pep.2006.10.010 |
| [34] | Hu YF, Liu YH, Li J, et al. Structural and functional analysis of a low-temperature-active alkaline esterase from South China Sea marine sediment microbial metagenomic library[J]. Journal of Industrial Microbiology & Biotechnology, 2015, 42(11) : 1449–1461. |
| [35] | Gao WY, Wu K, Chen LF, et al. A novel esterase from a marine mud metagenomic library for biocatalytic synthesis of short-chain flavor esters[J]. Microbial Cell Factories, 2016, 15 : 41. DOI:10.1186/s12934-016-0435-5 |
| [36] | López-López O, Cerdán ME, González Siso MI. New extremophilic lipases and esterases from metagenomics[J]. Current Protein & Peptide Science, 2014, 15(5) : 445–455. |
| [37] | Wei XL, Jiang XW, Ye LD, et al. Cloning, expression and characterization of a new enantioselective esterase from a marine bacterium Pelagibacterium halotolerans B2T[J]. Journal of Molecular Catalysis B: Enzymatic, 2013, 97 : 270–277. DOI:10.1016/j.molcatb.2013.09.002 |
| [38] | Sheridan PP, Panasik N, Coombs JM, et al. Approaches for deciphering the structural basis of low temperature enzyme activity[J]. Biochimica et Biophysica Acta (BBA)-Protein Structure and Molecular Enzymology, 2000, 1543(2) : 417–433. DOI:10.1016/S0167-4838(00)00237-5 |
| [39] | Dordick JS. Enzymatic catalysis in monophasic organic solvents[J]. Enzyme and Microbial Technology, 1989, 11(4) : 194–211. DOI:10.1016/0141-0229(89)90094-X |
 2017, Vol. 44
2017, Vol. 44




