扩展功能
文章信息
- 李志杰, 郭长城, 石杰, 林匡飞, 曹国民, 崔长征
- LI Zhi-Jie, GUO Chang-Cheng, SHI Jie, LIN Kuang-Fei, CAO Guo-Min, CUI Chang-Zheng
- 高通量测序解析多环芳烃污染盐碱土壤翅碱蓬根际微生物群落多样性
- Diversity of bacterial community in Suaeda roots rhizosphere growth in PAHs-contaminated saline soil estimated by high throughput sequencing method
- 微生物学通报, 2017, 44(7): 1602-1612
- Microbiology China, 2017, 44(7): 1602-1612
- DOI: 10.13344/j.microbiol.china.170164
-
文章历史
- 收稿日期: 2017-02-28
- 接受日期: 2017-05-23
- 优先数字出版日期(www.cnki.net): 2017-06-02
2. 天津师范大学 水环境与水资源重点实验室 天津 300387
2. Tianjin Key Laboratory of Water Resources and Environment, Tianjin Normal University, Tianjin 300387, China
多环芳烃(Polycyclic aromatic hydrocarbons,PAHs)是一类广泛存在于环境中的具有“三致”效应的持久性有机污染物,研究表明,土壤至少承担了90% PAHs污染[1]。其中,采油区原油污染导致的土壤PAHs污染存在浓度高、分布广等特点,其潜在风险备受关注[2-3]。
生物修复技术因其成本低、条件温和、二次污染效应小等优点,是最有潜力的土壤污染治理技术[4]。然而,研究表明盐碱化和PAHs污染的双重胁迫改变了土壤理化性质、微生物群落结构及多样性,导致非嗜盐的PAHs高效降解菌难以在盐碱化PAHs污染土壤修复中发挥有效作用[5-6]。翅碱蓬是滨海盐碱地和河口地区常见的植物,已用于研究海滨湿地保护和盐碱土生态修复[7]。有研究表明,翅碱蓬对盐碱化土壤有良好的适应能力,同时能富集土壤中PAHs[8]。另外,植物根际是土壤-植物-根际微生物相互作用的重要区域,根际促生菌、PAHs降解菌和植物之间良好的平衡状态能有效提高土壤修复效率[9]。近年来已有报道筛选出了能在盐碱条件下降解PAHs的细菌,如Ochrobactrum sp. VA1[10]、Martelella sp. AD-3[11]、Planococcus sp. B-1[12]等。上述微生物资源的不断发现和研究,提高了运用生物修复方法治理PAHs污染盐碱土壤的可行性。因此,探究PAHs污染盐碱土壤的翅碱蓬根际细菌多样性、群落结构及其相互作用关系,对PAHs污染土壤的植物-微生物联合修复研究和实践,具有重要的现实意义,然而目前鲜有相关报道。
传统的富集培养和平板培养方法所能分离获得的土壤微生物不超过总数的1%,大部分细菌无法培养鉴别。通过现代生物技术提取并分析DNA,如高通量测序、PCR-DGGE等技术,能更准确地反映微生物群落结构,已被广泛应用于环境微生物相关研究。细菌降解PAHs主要通过PAHs起始双加氧酶作用于苯环,在苯环上加入两个氧原子后逐渐开环降解,该步骤是PAHs降解的限速步骤。PAHs起始双加氧酶是由多个亚基组成多聚体蛋白质,其中α亚基(RHDα)相对保守,通常采用实时荧光定量PCR (Real-time PCR,RT-PCR)测定该功能基因的含量来反映功能基因的丰度[13]。
本研究以胜利油田油井周边PAHs污染土壤为研究对象,分别采集翅碱蓬生长区域和无翅碱蓬生长区域污染土壤样品,在分析比较其土壤含盐量及其他理化性质的基础上,利用高通量测序技术对不同土壤中的微生物群落结构进行分析,鉴别盐碱化PAHs污染土壤中的优势菌群,分析翅碱蓬和根际微生物之间的相互作用关系,采用实时荧光定量PCR测定并比较不同土壤中PAHs双加氧酶基因的含量,为植物-微生物联合修复盐碱化PAHs污染土壤提供依据。
1 材料与方法 1.1 土壤样品采集供试土样于2015年10月采自胜利油田的油井周围(37°26′56″N,118°22′37″E),分别选择距井口5 m处的碱蓬生长区域和无碱蓬生长区域的土壤进行样品采集,所选的不同采样区域中心相距30-50 cm。采集表层(0-20 cm)土壤作为样品,每个区域采集3个平行样,将其充分混匀作为一个样品,编号分别为样品1 (根际土壤)和样品2 (裸地土壤)。其中,碱蓬根际土壤采集时将根系挖出,轻轻抖落根系外围的大土块,只收集附着在根系上的土壤。样品采集后分成两部分,一部分4℃保存用于PAHs含量和理化性质分析,另一部分-20℃保存用于生物分析。
1.2 主要试剂和仪器FastDNA® Spin Kit for Soil试剂盒,MP Biomedicals;2×Taq PCR Master Mix、SYBR Premix Ex TaqTMⅡ、pMD18-T连接试剂盒,宝生物工程(大连)有限公司。
Fastprep24样品均质系统,MP Biomedicals公司;Bio-Rad IQ5 Real-time PCR Detection System,Bio-Rad公司。
1.3 土壤PAHs含量和理化性质测定将采集的土壤样品进行处理,经自然风干、研磨过筛(1 mm)后,测定土壤的理化性质和PAHs含量[14]。土壤的含水量、pH、可溶性盐含量、有机质以及氮磷钾含量等基本理化性质采用标准分析方法分析[9]。
1.4 土壤微生物DNA提取和高通量测序根据FastDNA® Spin Kit for Soil试剂盒的操作步骤提取土壤微生物总DNA,Miseq高通量测序由生工生物工程(上海)股份有限公司完成。PCR所用的引物为已经融合了Miseq测序平台的V3-V4通用引物,341F引物为:5′-CCTACGGGNGGCWGCAG-3′;805R引物为:5′-GACTACHVGGGTATCTAATCC-3′。
1.5 Real-time PCR分析实验所用革兰氏阴性细菌(Gram-negative,GN)和革兰氏阳性细菌(Gram-positive,GP) PAH-RHDα基因引物分别为PAH-RHDα GN F/PAH-RHDα GN R和PAH-RHDα GP F/PAH-RHDα GP R (GP),16S rRNA基因引物为968F/1401R,各引物序列如表 1所示。
实验对GN和GP两种PAH-RHDα基因进行PCR退火温度的优化,退火温度范围为47-58℃。根据Cébron报道的相关PCR程序进行条件的优化且将最终优化条件用于RT-PCR[13]。普通PCR反应体系为(50 μL):2×Taq PCR Master Mix 25 μL,模板DNA 1 μL,引物10 μmol/L各1 μL,补加ddH2O至总体积为50 μL。RT-PCR体系为(20 μL):10 μL SYBR Premix Ex TaqTM Ⅱ,引物10 μmol/L各0.4 μL,DNA模板1 μL,加ddH2O至总体积为20 μL。PCR反应条件为:95℃ 5 min;95℃ 30 s,PAH-RHDα GN 50℃ 30 s/16S rRNA 55℃ 30 s,72℃ 30 s,30个循环;72℃ 5 min。RT-PCR的循环数为40。每个样品设3个平行。利用pMD18-T载体构建双加氧酶标准质粒和16S rRNA标准质粒[16]。
将双加氧酶标准质粒和16S rRNA标准质粒分别稀释为10-1-10-8梯度浓度,作为制作标准曲线DNA模板,每一个梯度设置3个平行,将配置好的体系放入Bio-Rad iQ5实时荧光定量PCR仪扩增目的基因,以梯度稀释液中DNA的拷贝数的对数作为横坐标,以Ct值作为纵坐标制作标准曲线。
1.6 数据分析和数据处理将所得序列数据用FLASH 1.2.3和Prinseq 0.20.4软件进行质量控制,再通过Mothur 1.30.1软件进行校正,去除嵌合体及靶区域外序列,最后采用Uclust 1.1.579软件进行OTU (Operational taxonomic units)聚类分析,采用RPD classifier软件贝叶斯算法对97%相似度水平的OTU代表序列进行分类学分析,统计各样品的菌落组成,分类可信度用bootstrap方法估算。分析所用数据库有RDP classifier数据库(16S rRNA基因)、Sliva数据库(16S rRNA基因)和Unite ITS数据库。样品丰富度稀疏分析图利用Mothur 1.30.1软件绘制。
2 结果与分析 2.1 土壤的理化性质和PAHs含量不同土壤样品的理化性质和PAHs含量如表 2所示。
| 样品编号 Sample code |
PAHs含量 PAHs content (μg/kg) |
含水率 Moisture (%) |
pH | 盐含量 Salinity (g/kg) |
有机质 Organic matter (g/kg) |
全氮 Total nitrogen (g/kg) |
有效磷 Available P (mg/kg) |
有效钾 Available K (mg/kg) |
碳氮比 C/N |
| 1 | 1 065.33 | 15.90 | 8.20 | 22.51 | 24.41 | 1.59 | 29.07 | 571.88 | 15 |
| 2 | 1 309.02 | 10.55 | 8.22 | 40.03 | 18.80 | 0.71 | 38.94 | 434.19 | 26 |
| 注:样品编号1:碱蓬根际土壤样品;样品编号2:无碱蓬生长区域土壤样品(裸地). Note: Sample 1: Soil samples in the seepweed rhizosphere; Sample 2: Soil samples in the nudation. | |||||||||
根际土壤中PAHs含量低于裸地土壤,两种土壤中PAHs含量均高于1 000 μg/kg,参照Maliszewska-kordybach对欧洲农业土壤PAHs含量与分布所划分的4个等级,这两种土壤的PAHs污染水平均为严重污染[17]。
两种土壤样品的pH和可溶性盐含量表明其均为盐碱土壤,对比发现不同土壤的pH值差别不大,但翅碱蓬根际土壤的盐含量显著低于裸地土壤。翅碱蓬根际土壤有机质和总氮含量分别高于裸地土壤,且C/N值为15,低于裸地土壤样品的26,说明翅碱蓬根际土壤C/N值更适于PAHs降解微生物发挥作用。根际土壤的有效磷含量略低于裸地土壤,而有效钾的含量相对较高。
2.2 序列数据预处理及OTU聚类分析由Miseq双端测序所得样品数据经质量控制,去除非靶区域序列、嵌合体、短片段序列等,使得质控后序列长度大部分分布在400-600 bp之间,满足序列分析要求。质控后,根际土壤高质量序列数为53 854条,高于裸地土壤的高质量序列数30 312条。
根据Barcode标签对样品进行序列拆分,去冗余处理后获得16S rRNA Unique reads,并在97%相似度下将其聚类为用于物种分类的OTU,统计得到各个样品在不同OTU中的丰度信息,根际土壤产生5 934个OTU,裸地土壤产生4 262个OTU。
2.3 不同样品Alpha多样性分析Alpha多样性分析指标包括文库覆盖率(Coverage)、Chao 1指数、Ace指数、Shannon指数和Simpson指数,各指数的值如表 3所示。
| 样品编号 Sample code |
文库覆盖率 Coverage |
Chao 1指数 Chao 1 index |
ACE指数 ACE index |
Shannon指数 Shannon index |
Simpson指数 Simpson index |
| 1 | 0.944 | 11 461 | 15 555 | 7.01 | 0.002 9 |
| 2 | 0.927 | 8 262 | 11 186 | 6.77 | 0.004 2 |
样品1和2的文库覆盖率均大于92.7%,说明样本中序列没有被测出的概率低,本次测序结果能够代表样本的真实情况。根际土壤的Chao 1、ACE和Shannon指数值均高于裸地土壤,而Simpson指数值低于裸地土壤,表明两种土壤中的物种丰度和多样性存在明显差异。
OTU水平下,结果显示根际土壤包含5 934个OTU,高于裸地土壤的4 262个OTU,不同样品中共有和特有的OTU数目用Venn图显示,如图 1所示。由图 1可知,有2 250个OTU是根际土壤和裸地土壤中共有的,分别特有3 684和2 012个OTU,两种土壤样品总共有7 946个OTU,共有OTU数目占全部的28.32%,相同的OTU所占比例较低,说明两种样品之间存在较大差异性。

|
| 图 1 不同样品中共有和独有的OTU数目Venn图 Figure 1 Venn analysis for bacterial species (OTUs) of different samples 注:序列按相似度高于0.97进行OTU聚类. Note: OTUs: the sequences are clustered according to the similarity above 0.97. |
|
|
根际土壤测序获得32门(亚门) 64纲128目241科758属土壤细菌,裸地土壤测得28门(亚门) 58纲118目228科676属土壤细菌,不同土壤中门(亚门)分类水平下菌群组成和丰度情况如表 4所示。由表 4可知,变形菌门(Proteobacteria)在两种土壤样品中均是优势菌门,在根际土壤和裸地土壤中的相对丰度分别是55.22%和46.96%,远高于次优势门。根际土壤和裸地土壤的次优势门同为拟杆菌门(Bacteroidetes),相对丰度分别为10.64%和13.07%。
| 菌群(门水平分类) bacteria groups (phylum level) |
土壤样品1 Sample code 1 |
土壤样品2 Sample code 2 |
相对丰度比值 relative abundance ratio |
|||
| sequence数 sequence number |
相对丰度 relative abundance (%) |
sequence数 sequence number |
相对丰度 relative abundance (%) |
|||
| Proteobacteria | 29 738 | 55.22 | 14 233 | 46.96 | 1.18 | |
| Bacteroidetes | 5 732 | 10.64 | 3 961 | 13.07 | 0.81 | |
| Actinobacteria | 4 531 | 8.41 | 1 561 | 5.15 | 1.63 | |
| Firmicutes | 4 088 | 7.59 | 2 336 | 7.71 | 0.98 | |
| Chloroflexi | 3 053 | 5.67 | 2 308 | 7.61 | 0.75 | |
| Planctomycetes | 1 512 | 2.81 | 405 | 1.34 | 2.10 | |
| Gemmatimonadetes | 1 336 | 2.48 | 1 159 | 3.82 | 0.65 | |
| Verrucomicrobia | 958 | 1.78 | 283 | 0.93 | 1.91 | |
| Cyanobacteria | 897 | 1.67 | 2 935 | 9.68 | 0.17 | |
| Spirochaetae | 414 | 0.77 | 292 | 0.96 | 0.80 | |
| Acidobacteria | 399 | 0.74 | 141 | 0.47 | 1.57 | |
| Chlorobi | 220 | 0.41 | 180 | 0.59 | 0.69 | |
| Nitrospirae | 173 | 0.32 | 32 | 0.11 | 2.91 | |
| Tenericutes | 160 | 0.30 | 123 | 0.41 | 0.73 | |
| Deinococcus-Thermus | 154 | 0.29 | 66 | 0.22 | 1.32 | |
| Chlamydiae | 134 | 0.25 | 19 | 0.06 | 4.17 | |
| Deferribacteres | 90 | 0.17 | 29 | 0.10 | 1.70 | |
| Euryarchaeota | 65 | 0.12 | 114 | 0.38 | 0.32 | |
| Candidate_division_TM7 | 50 | 0.09 | 36 | 0.12 | 0.75 | |
| Lentisphaerae | 37 | 0.07 | 14 | 0.05 | 1.40 | |
| Elusimicrobia | 31 | 0.06 | 16 | 0.05 | 1.20 | |
| Candidate_division_WS3 | 25 | 0.05 | 10 | 0.03 | 1.67 | |
| unclassified | 17 | 0.03 | 23 | 0.08 | 0.38 | |
| Candidate_division_OD1 | 11 | 0.02 | 18 | 0.06 | 0.33 | |
| Aquificae | 9 | 0.02 | - | - | - | |
| Fibrobacteres | 5 | 0.01 | 1 | 0 | - | |
| Crenarchaeota | 5 | 0.01 | 1 | 0 | - | |
| Thermotogae | 3 | 0.01 | - | - | - | |
| Synergistetes | 3 | 0.01 | 14 | 0.05 | 0.20 | |
| Dictyoglomi | 2 | 0 | - | - | - | |
| Armatimonadetes | 1 | 0 | - | - | - | |
| Fusobacteria | 1 | 0 | 1 | 0 | - | |
| Candidate_division_KB1 | - | - | 1 | 0 | - | |
| 注:0:相对丰度小于0.01%;-:未在该种类样品中检测到对应菌群;Unclassified:未分类序列. Note: 0: Relative abundance<0.01%; -: Not detected; Unclassified: Not classified. | ||||||
不同样品中菌群相对丰度不同,表明样品的群落结构存在差异,计算各类细菌的相对丰度比值(细菌在根际土壤中相对丰度与裸地土壤中相对丰度之比),比值越接近于1,则说明该类细菌在不同样品中的存在情况越相似,厚壁菌门(Firmicutes)在根际土壤中的相对丰度为7.59%,在裸地土壤中为7.71%,相对丰度高且接近,比值为0.98,表明厚壁菌门(Firmicutes)在两种土壤环境中均有较强的适应性,实验用的两种土壤环境的不同不会影响该类细菌在土壤的含量。另一方面,如蓝细菌门(Cyanobacteria)在根际土壤中的相对丰度为1.67%,而在裸地土壤中为9.68%,相对丰度是在根际土壤中的5.80倍,差异明显,说明蓝细菌门(Cyanobacteria)更适应地表裸露的土壤环境。相对丰度比值小于0.5或大于2.0表明菌群在不同土壤样品中存在显著差异,不同门类细菌在根际土壤中显著高(比值大于2.0) 的有浮霉菌门(Planctomycetes)、疣微菌门(Verrucomicrobia)、硝化螺旋菌门(Nitrospirae)、衣原体门(Chlamydiae),显著低(比值小于0.5) 的有蓝藻细菌门(Cyanobacteria)、广古菌门(Euryarchaeota)。
在属分类水平下,根际土壤中测得758属细菌,其中优势菌属(相对丰度大于1%)有23个属,其中包括嗜盐碱或耐盐碱优势菌属如Thioalkalispira (3.35%)、Marinilabiaceae (2.44%)、Salinimicrobium (1.76%)等。裸地土壤中测得676属细菌,比根际土壤少82个属,其优势菌属有24个,其中嗜盐碱或耐盐碱优势菌属有Salinimicrobium (3.08%)、Marinilabiaceae (2.61%)、Halothiobacillus (2.31%)、Desulfobacteraceae (2.09%)、Caenispirillum (1.83%)等。根际土壤中丰度最高的前50个菌属相对丰度总和为61.47%,裸地土中为63.65%,说明丰度最高的前50个菌属已包含了土壤中的大部分细菌。对两种土壤中相对丰度最高的前50个菌属进行比较,发现有32个菌属相同。
根据菌属在不同土壤样品中的相对丰度之比,选择丰度较高(sequence数大于100) 且差异性较大(相对丰度之比小于0.5或大于2.0) 的53个菌属进行比较分析,结果如图 2所示。从图 2中可见,不同土壤样品中的优势菌属(相对丰度大于1%)分布不同,群落结构差异较大。比较发现,在根际土壤中有36个菌属的相对丰度显著高于其在裸地土壤中的相对丰度,而另外17个菌属的相对丰度显著低于裸地土样品中的。其中Thioalkalispira在根际土壤中的相对丰度为3.35%,是该土壤中的次优势菌属,而此菌属在裸地土壤中的相对丰度仅为0.82%,相差4倍。另外在根际土壤中的优势菌属,如Coleofasciculus (1.42%)、Fusibacter (1.12%)、Desulfobulbus (1.14%)等均不能在裸地土中成为优势菌;而在裸地土壤中的优势菌属,如Phormidium (2.48%)、Lyngbya (2.42%)、Halothiobacillus (2.31%)、Caenispirillum (1.83%)、Alteromonadaceae (1.29%)等均不是根际土壤中的优势菌,并且没有测序得到Lyngbya菌属序列。菌属Denitromonas在根际土壤中的相对丰度为0.19%,对比裸地土壤中的相对丰度0.01%,相差19倍;Leptolyngbya、Phormidium在根际土壤和裸地土壤中的相对丰度差异分别是50和100倍。
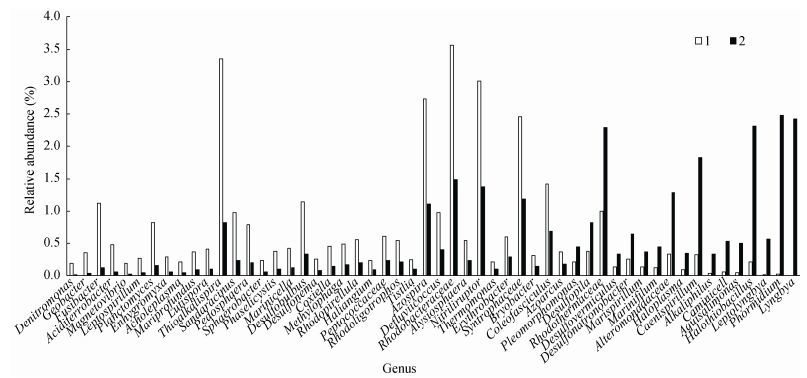
|
| 图 2 属水平分类下不同土壤样品中菌属丰度对比 Figure 2 Comparison of bacteria groups at genus level 注:虚线表示相对丰度等于1%. Note: The imaginary line: Relative abundance=1%. |
|
|
在退火温度为47-58℃时,扩增得到PAH-RHDα GN基因,但未扩增到PAH-RHDα GP基因。根据扩增退火温度条带清晰度,选取50℃作为扩增PAH-RHDα GN基因的退火温度。利用PAH-RHDα GN基因反映土壤中微生物PAH-RHDα基因的情况,所以下文中的PAH-RHDα基因代表的是微生物中PAH-RHDα GN基因的情况。不同土壤样品中PAH-RHDα和16S rRNA基因拷贝数如表 5所示,根际土壤样品的PAH-RHDα基因拷贝数和16S rRNA基因拷贝数(2.12×103 copies/g和3.34×106 copies/g)均高于裸地土壤(未检出和9.21×105 copies/g),说明根际土壤中PAHs降解菌的降解作用优于裸地土壤,并且其细菌数量也多于裸地土壤,与高通量测序结果一致。
| 样品编号 Sample code |
PAH-RHDα基因拷贝数 PAH-RHDα genes copy (copies/g) |
16S rRNA基因拷贝数 16S rRNA genes copy (copies/g) |
| 1 | 2.12×103 | 3.34×106 |
| 2 | - | 9.21×105 |
| 注:-:未检测到. Note: -: Not detected. | ||
样品pH和可溶性盐含量表明,本研究中的两种土壤均为盐碱土壤,与胜利油田盐碱土壤的污染特征类似[12]。高世珍等[8]研究发现翅碱蓬对PAHs有吸收和富集作用,且根部的吸收能力最强达到1.5 μg/g;杨红军等[18]研究发现翅碱蓬对100 mg/kg菲污染土壤有42.86%的去除率。翅碱蓬对PAHs的富集作用,可能是根际土壤中PAHs含量低于裸地土壤的原因之一。
翅碱蓬通过改善根际土壤盐碱环境,增加土壤含N量,降低C/N比值,促进PAHs降解微生物发挥作用。张立宾等[19]研究发现种植翅碱蓬能有效降低盐碱土壤中的含盐量。土壤中C/N比为12时利于土壤微生物对石油污染物的分解利用[20]。本研究中两种土壤C/N比都大于12,推测油田开采导致的石油污染增加了土壤中的碳源,另一方面土壤中降解石油烃、PAHs的微生物在降解过程中消耗N源,但C源过剩,从而使土壤C/N比升高[2]。翅碱蓬根际土壤中的C/N比值为15,有利于根际降解微生物发挥作用,是根际土壤的PAHs含量低于裸地土壤的主要原因。
3.2 土壤样品微生物多样性分析原核生物基因组内的16S rRNA基因多样性会导致群落多样性测定结果偏高,这种影响与16S rRNA基因的检测位置有关。Sun等[21]研究发现在97%的序列相似度下,V6区域的检测结果受影响程度最高,达12.9%,V4-V5区域受影响程度最低为3.0%。本研究利用MiSeq测序技术对16S rRNA的V3-V4区域进行测序分析,保证了16S rRNA基因多样性对测序结果的影响程度最小。对比不同土壤中的菌群信息发现,翅碱蓬根际土壤中的微生物种类和数量均高于相同地理环境中的裸地土壤。推测其原因,一方面在于翅碱蓬对PAHs污染盐碱土壤的改良作用,另一方面在于翅碱蓬与其根际微生物之间的相互作用促进了降解微生物的生长,提高了修复效果。高乃媛等[22]利用翅碱蓬修复石油污染盐碱土壤得到类似结果。
属分类发现根际土壤中固氮螺菌属(Azospira)的相对丰度高达2.73%,裸地土壤中仅为1.11%,这类菌具有固氮的作用,所以根际土壤环境的N含量高于裸地土壤,与所测结果一致。属分类显示,Halothiobacillus、Thiohalophilus、Halobacteroidaceae、Halomonas、Thiohalomonas、Thiohalobacter、Thioalkalispira、Alkaliphilus、Geoalkalibacter等有过嗜盐或嗜碱报道的微生物为两类土壤中的优势菌属,但在不同土壤中的相对丰度存在明显差异。比较发现,翅碱蓬根际土壤中的盐含量明显低于裸地土壤,微生物对土壤含盐量的适应能力不同,导致了上述细菌在两种土壤样品中的丰度分布存在差异,差异较大的菌属如Rhodobacteraceae、Thioalkalispira,在根际土壤中相对丰度3.56%和3.35%,裸地土壤中仅为1.48%和0.82%。Gutierrez等[23]研究发现Rhodobacteraceae与低分子量PAHs的降解有关。
添加功能菌可提高PAHs污染的修复效果,但常规的功能菌难以在高盐碱土壤环境中发挥有效作用,而翅碱蓬对此类土壤的适应能力和修复作用大大提高了运用生物修复方法治理此类污染的可行性。从整体上来看,根际土壤里的微生物丰度和种类均比裸地土壤中的高,说明翅碱蓬根际能够为微生物提供良好的生长环境,微生物资源丰富,有待于进一步研究。在鉴别出的菌属中,发现一些相对丰度较高的功能菌属,如海旋菌(Thalassospira)、鞘氨醇单胞菌属(Sphingomonas)、假单胞菌(Pseudomonas)、寡养单胞菌(Stenotrophomonas)、芽孢杆菌属(Bacillus)等,它们在根际土壤样品中相对丰度依次为0.79%、0.46%、0.65%、0.41%、0.31%。上述菌属中,分别有多种菌株已被文献报道,发现其有降解PAHs的功能并被筛选出来,如Kodama等[24]分离到具有PAHs降解功能的新型菌株Thalassospira tepidiphila sp.;Schuler等[25]发现Sphingomonas sp. strain LH128具有降解菲和苯并[a]蒽的功能;Ling等[26]报道了Bacillus vallismortis JY3A降解芘的相关研究;Santos等[27]研究了Pseudomonas aeruginosa 312A对蒽的降解作用。
本研究对土壤样品中微生物的16S rRNA基因进行检测,对比孙佳杰等[28]对天津滨海盐碱土壤微生物生态特征的研究结果,发现本研究所得盐碱土壤细菌数量偏低,这是由于土壤环境的不同导致。本研究土壤样品的含盐量为22.51-40.03 g/kg,显著高于孙佳杰等研究所用样品的含盐量0.49-18.48 g/kg,而周宁一[29]指出细菌数量与土壤盐含量呈显著负相关,土壤盐害程度越高,微生物数量越少。对土壤样品中微生物降解PAHs的PAH-RHDα基因的检测结果显示翅碱蓬根际土壤中存在着PAH-RHDα基因,而在裸地土壤中没有检测到PAH-RHDα基因,说明了根际土壤中的翅碱蓬在富集PAH的过程中也能够保留微生物降解PAH能力。由于细菌主要通过PAHs起始双加氧酶对PAHs进行开环降解,PAH-RHDα基因的存在可推测此土壤样品中存在PAHs降解菌的可能性。
综上所述,PAHs污染盐碱土壤中蕴含着丰富的微生物资源,通过生物刺激盐碱土壤土著微生物或生物强化添加嗜盐PAHs高效降解菌,将在PAHs污染盐碱土壤的生物修复中发挥重要作用。
4 结论(1) Thioalkalispira、Halothiobacillus、Thiohalophilus等嗜盐碱或耐盐碱微生物是胜利油田PAHs污染盐碱土壤中的优势菌属,存在丰富的嗜盐碱或耐盐碱PAHs降解微生物资源;有些菌属可能与PAH降解或耐盐碱相关,有待进一步研究。
(2) 翅碱蓬作为石油开采区盐碱土壤的优势植物,能有效降低盐碱化土壤中的含盐量,增加土壤N、P、K的含量并改善C/N值,增加微生物群落结构多样性,提高PAHs关键功能基因的丰度,有助于促进嗜盐PAHs降解微生物在PAHs污染盐碱土壤的生物修复中发挥作用。
| [1] | Cao YZ, Liu XJ, Xie YF, et al. Patterns of PAHs concentrations and components in surface soils of main areas in China[J]. Acta Scientiae Circumstantiae, 2012, 32(1) : 197–203. (in Chinese) 曹云者, 柳晓娟, 谢云峰, 等. 我国主要地区表层土壤中多环芳烃组成及含量特征分析[J]. 环境科学学报, 2012, 32(1) : 197–203. |
| [2] | Liu J, Song XY, Sun RL, et al. Petroleum pollution and the microbial community structure in the soil of Shengli Oilfield[J]. Chinese Journal of Applied Ecology, 2014, 25(3) : 850–856. (in Chinese) 刘健, 宋雪英, 孙瑞莲, 等. 胜利油田采油区土壤石油污染状况及其微生物群落结构[J]. 应用生态学报, 2014, 25(3) : 850–856. |
| [3] | Li XC, Hu Y, Du XY, et al. Simulated model of the fate and transfer for PAHs in the multimedia environment for a onshore oilfield[J]. Journal of Safety and Environment, 2011, 11(6) : 5–9. (in Chinese) 李兴春, 胡艳, 杜显元, 等. 陆地石油开采区PAHs环境多介质分布及迁移转化模拟[J]. 安全与环境学报, 2011, 11(6) : 5–9. |
| [4] | Zou DX, Luo YM, Xu FH, et al. Microbial degradation of polycyclic aromatic hydrocarbons in soil environment and combined bioremediation of PAHs contaminated soils[J]. Soils, 2007, 39(3) : 334–340. (in Chinese) 邹德勋, 骆永明, 徐凤花, 等. 土壤环境中多环芳烃的微生物降解及联合生物修复[J]. 土壤, 2007, 39(3) : 334–340. |
| [5] | Gao YC, Wang JN, Guo SH, et al. Effects of salinization and crude oil contamination on soil bacterial community structure in the Yellow River Delta region, China[J]. Applied Soil Ecology, 2015, 86 : 165–173. DOI:10.1016/j.apsoil.2014.10.011 |
| [6] | Cheung PY, Kinkle BK. Mycobacterium diversity and pyrene mineralization in petroleum-contaminated soils[J]. Applied and Environmental Microbiology, 2001, 67(5) : 2222–2229. DOI:10.1128/AEM.67.5.2222-2229.2001 |
| [7] | Li CF, Ge BM, Jiang SH, et al. Review on remedial effect of Suaeda salsa on saline and polluted soils[J]. Chinese Journal of Soil Science, 2014, 45(4) : 1014–1019. (in Chinese) 李超峰, 葛宝明, 姜森颢, 等. 碱蓬对盐碱及污染土壤生物修复的研究进展[J]. 土壤通报, 2014, 45(4) : 1014–1019. |
| [8] | Gao SZ, Zhao XR, Cui SM, et al. Distribution characteristics of the typical persistent organic pollutants (POPs) in Suaeda heteroptera[J]. Environmental Science, 2010, 31(10) : 2456–2461. (in Chinese) 高世珍, 赵兴茹, 崔世茂, 等. 典型持久性有机污染物在翅碱蓬中的分布特征[J]. 环境科学, 2010, 31(10) : 2456–2461. |
| [9] | Wang XX, Bai ZH, Jin DC, et al. Bacterial diversity and halotolerant petroleum-degrading bacteria of the rhizosphere of Suaeda salsa (L.) in petroleum-contaminated saline-alkali soil[J]. Microbiology China, 2011, 38(12) : 1768–1777. (in Chinese) 王新新, 白志辉, 金德才, 等. 石油污染盐碱土壤翅碱蓬根围的细菌多样性及耐盐石油烃降解菌筛选[J]. 微生物学通报, 2011, 38(12) : 1768–1777. |
| [10] | Arulazhagan P, Vasudevan N. Biodegradation of polycyclic aromatic hydrocarbons by a halotolerant bacterial strain Ochrobactrum sp. VA1[J]. Marine Pollution Bulletin, 2011, 62(2) : 388–394. DOI:10.1016/j.marpolbul.2010.09.020 |
| [11] | Cui CZ, Feng TC, Yu YQ, et al. Isolation, charcaterization of an anthracene degrading bacterium Martelella sp. AD-3 and cloning of dioxygenase gene[J]. Environmental Science, 2012, 33(11) : 4062–4068. (in Chinese) 崔长征, 冯天才, 于亚琦, 等. 降解蒽嗜盐菌AD-3的筛选、降解特性及加氧酶基因的研究[J]. 环境科学, 2012, 33(11) : 4062–4068. |
| [12] | Song LC, Li PJ, Liu W, et al. Isolation, identification and degradation characteristics of a PAHs-degrading bacteria from salt-alkaline soil[J]. Microbiology China, 2011, 38(2) : 282–287. (in Chinese) 宋立超, 李培军, 刘宛, 等. 盐碱土壤PAHs降解菌的筛选鉴定及其降解特性[J]. 微生物学通报, 2011, 38(2) : 282–287. |
| [13] | Cébron A, Norini MP, Beguiristain T, et al. Real-time PCR quantification of PAH-ring hydroxylating dioxygenase (PAH-RHDα) genes from Gram positive and Gram negative bacteria in soil and sediment samples[J]. Journal of Microbiological Methods, 2008, 73(2) : 148–159. DOI:10.1016/j.mimet.2008.01.009 |
| [14] | Zhang J, Zhang HW, Li XY, et al. Bacterium and mycobacterium diversity in paddy soils under PAHs stress[J]. Chinese Journal of Eco-Agriculture, 2008, 16(2) : 405–410. (in Chinese) 张晶, 张惠文, 李新宇, 等. 多环芳烃胁迫下稻田土壤细菌及分支杆菌种群多样性研究[J]. 中国生态农业学报, 2008, 16(2) : 405–410. |
| [15] | Felske A, Akkermans ADL, de Vos WM. Quantification of 16S rRNAs in complex bacterial communities by multiple competitive reverse transcription-PCR in temperature gradient gel electrophoresis fingerprints[J]. Applied and Environmental Microbiology, 1998, 64(11) : 4581–4587. |
| [16] | Tang JZ, Feng TC, Liu SY, et al. Functional verification of a dual functional consortium: degrading phenanthrene and oxidizing arsenite simultaneously[J]. Journal of East China University of Science and Technology (Natural Science Edition), 2013, 39(4) : 450–456. (in Chinese) 汤军芝, 冯天才, 刘世友, 等. 菌群降解菲和氧化三价砷的双功能验证[J]. 华东理工大学学报:自然科学版, 2013, 39(4) : 450–456. |
| [17] | Maliszewska-Kordybach B. Polycyclic aromatic hydrocarbons in agricultural soils in Poland: preliminary proposals for criteria to evaluate the level of soil contamination[J]. Applied Geochemistry, 1996, 11(1/2) : 121–127. |
| [18] | Yang HJ, Xie WJ, Chen ZY, et al. Decontamination of six grass species to phenanthrene in soil[J]. Chinese Journal of Soil Science, 2012, 43(5) : 1242–1246. (in Chinese) 杨红军, 谢文军, 陈志英, 等. 六种野草对土壤中菲的降解研究[J]. 土壤通报, 2012, 43(5) : 1242–1246. |
| [19] | Zhang LB, Xu HL, Zhao GX. Salt tolerance of Suaeda salsa and its soil ameliorating effect on coastal saline soil[J]. Soils, 2007, 39(2) : 310–313. (in Chinese) 张立宾, 徐化凌, 赵庚星. 碱蓬的耐盐能力及其对滨海盐渍土的改良效果[J]. 土壤, 2007, 39(2) : 310–313. |
| [20] | Płaza G, Nałęcz-Jawecki G, Ulfig K, et al. The application of bioassays as indicators of petroleum-contaminated soil remediation[J]. Chemosphere, 2005, 59(2) : 289–296. DOI:10.1016/j.chemosphere.2004.11.049 |
| [21] | Sun DL, Jiang X, Wu QL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19) : 5962–5969. DOI:10.1128/AEM.01282-13 |
| [22] | Gao NY, Liu XB, Zhao XR. Influence of oil in soil on growth and physiological indexes of suaeda heteroptera and plant-microbial remediation[J]. Chinese Journal of Environmental Engineering, 2013, 7(4) : 1578–1582. (in Chinese) 高乃媛, 刘宪斌, 赵兴茹. 石油烃对翅碱蓬生理特性的影响及植物-微生物联合降解[J]. 环境工程学报, 2013, 7(4) : 1578–1582. |
| [23] | Gutierrez T, Singleton DR, Aitken MD, et al. Stable isotope probing of an algal bloom to identify uncultivated members of the Rhodobacteraceae associated with low-molecular-weight polycyclic aromatic hydrocarbon degradation[J]. Applied and Environmental Microbiology, 2011, 77(21) : 7856–7860. DOI:10.1128/AEM.06200-11 |
| [24] | Kodama Y, Stiknowati LI, Ueki A, et al. Thalassospira tepidiphila sp. nov. a polycyclic aromatic hydrocarbon-degrading bacterium isolated from seawater[J]. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(3) : 711–715. DOI:10.1099/ijs.0.65476-0 |
| [25] | Schuler L, Jouanneau Y, Chadhain SMN, et al. Characterization of a ring-hydroxylating dioxygenase from phenanthrene-degrading Sphingomonas sp. strain LH128 able to oxidize benz[a]anthracene[J]. Applied Microbiology and Biotechnology, 2009, 83(3) : 465–475. DOI:10.1007/s00253-009-1858-2 |
| [26] | Ling JY, Zhang GY, Sun HB, et al. Isolation and characterization of a novel pyrene-degrading Bacillus vallismortis strain JY3A[J]. Science of the Total Environment, 1994, 409(10) : 1994–2000. |
| [27] | Santos EC, Jacques RJS, Bento FM, et al. Anthracene biodegradation and surface activity by an iron-stimulated Pseudomonas sp.[J]. Bioresource Technology, 2008, 99(7) : 2644–2649. DOI:10.1016/j.biortech.2007.04.050 |
| [28] | Sun JJ, Yin JD, Xie YH, et al. Microbial ecological characteristics of saline-alkali soil in coastal area of Tianjin[J]. Journal of Nanjing Forestry University (Natural Science Edition), 2010, 34(3) : 57–61. (in Chinese) 孙佳杰, 尹建道, 解玉红, 等. 天津滨海盐碱土壤微生物生态特性研究[J]. 南京林业大学学报:自然科学版, 2010, 34(3) : 57–61. |
| [29] | Zhou NY. Microbial diversity in saline-alkali soil[J]. Microbiology China, 2012, 39(7) : 1030. (in Chinese) 周宁一. 盐碱地微生物类群的多样性[J]. 微生物学通报, 2012, 39(7) : 1030. |
 2017, Vol. 44
2017, Vol. 44




