扩展功能
文章信息
- 范习贝, 梁前勇, 牛明杨, 余甜甜, 王寅炤, 王风平
- FAN Xi-bei, LIANG Qian-yong, NIU Ming-yang, YU Tian-tian, WANG Yin-zhao, WANG Feng-ping
- 中国南海北部陆坡沉积物古菌多样性及丰度分析
- The diversity and richness of archaea in the northern continental slope of South China Sea
- 微生物学通报, 2017, 44(7): 1589-1601
- Microbiology China, 2017, 44(7): 1589-1601
- DOI: 10.13344/j.microbiol.china.170159
-
文章历史
- 收稿日期: 2017-02-28
- 接受日期: 2017-05-05
2. 上海交通大学 微生物代谢国家重点实验室 上海 200240;
3. 上海交通大学 海洋工程国家重点实验室 上海 200240;
4. 中国地质调查局广州海洋地质调查局 广东 广州 510760
2. State Key Laboratory of Microbial Metabolism, Shanghai Jiao Tong University, Shanghai 200240, China;
3. State Key Laboratory of Ocean Engineering, Ocean and Civil Engineering, Shanghai Jiao Tong University, Shanghai 200240, China;
4. Guangzhou Marine Geological Survey, China Geological Survey, Guangzhou, Guangdong 510760, China
海洋沉积物覆盖地球表面约2/3,广泛分布着活跃的微生物(细菌和古菌)。全球海洋沉积物的微生物数高达2.9×1029个细胞,约等于海水微生物数量[1]。这些微生物参与海底碳元素的转化和其它生物地球化学循环[2]。自Woese在1990年[3]提出三域分类系统以来,古菌作为一种独特的生命形式,引发了科学家广泛的关注。早期古菌被认为是极端微生物,仅存在于极端环境中,后来更多的研究发现其生境广泛,不仅包括一些极端环境例如盐湖、热泉等,还包括土壤、淡水河、海水、沉积物等[4-5]。近年来随着高通量测序技术的发展,自然界大量未培养古菌被发现[6-8],古菌系统进化树也在不断地扩充和更新[9]。目前,古菌类群可分为3个超级亚门:TACK、DPANN和Euryarchaeota类群[10],其中,TACK代表Thaumarchaeota、Crenarchaeota、Bathyarchaeota[11]、Korarchaeota、Geoarchaeota[12]和Aigarchaeota[7]等;DAPNN代表Diapherotrites、Parvarchaeota、Aenigmarchaeota (DSEG)和Woesearchaeota (DHVEG-6) 等,还有更多的古菌新类群正在被不断发现。海洋沉积物中拥有大量古菌,其中一些具有独特代谢功能例如厌氧甲烷氧化古菌(ANME),仍还有许多古菌的生理和代谢功能未知,认识和理解古菌的代谢和生态学功能需要我们首先对它们的分布和多样性进行调查和分析[13]。
南海又称“南中国海”,是世界上第三大边缘海,仅次于珊瑚海和阿拉伯海,为西太平洋的一部分。海域面积约356万km2,平均水深约为1 212 m,最深处为中部的深海平原,达5 567 m左右[14]。南海地处低纬度地域,属于热带深海,蕴藏着丰富的天然气水合物,具有非常重要的经济和政治意义。南海沉积物中含有丰富多样的微生物资源,探索南海沉积物中微生物的多样性和丰度对认识海洋沉积物元素循环具有重要的意义。随着全球气候变暖,二氧化碳(CO2)、甲烷(CH4)和其他温室气体的含量不断增加,导致极地冰川融化和海平面上升,显著影响全球降雨量及降雪量[15],对全球生态系统造成严重威胁。海洋沉积物中蕴含丰富的天然气水合物(主要成分为CH4),是一种新型的绿色能源,可作为煤、石油等传统能源的补充能源,但是其导致的温室效应约是同分子量CO2的25倍,分解和释放的少量CH4就能对地球表面的温度产生巨大的影响[16]。同时,海洋沉积物中甲烷厌氧氧化(Anaerobic oxidation of methane,AOM)过程是微生物介导代谢消耗甲烷的主要途径[17-18],是减少海洋沉积物中CH4释放的重要过程,可以屏蔽90%以上的甲烷释放[19]。目前,甲烷厌氧氧化菌(Anaerobic methanotrophic archaea,ANME)暂分为三类:ANME-1 (a,b,c) (Methanomicrobiales和Methanosarcinales类群)、ANME-2 (a,b,c) (Methanosarcinales的一个分支)以及ANME-3 (Methanococcoides)[20-21]。另外,有报道显示Bathyarchaeota类群的部分古菌也具有氧化甲烷的潜在能力[22],古菌Syntrophoarchaeum类群具有新的丁烷厌氧氧化途径[23]。甲烷代谢保守功能基因mcrA (methyl coenzyme-M reductase A)[24]作为研究甲烷代谢菌的功能基因,是由于其编码还原辅酶M存在于所有已知的甲烷产生菌和甲烷氧化菌中[25-26]。mcrA基因的定量可以反映甲烷代谢类群的丰度。了解南海沉积物潜在水合物区厌氧甲烷氧化类群的多样性及丰度可以为评估南海沉积物中碳循环速率提供数据支持。
我国自1998年开始在南海进行天然气水合物调查,并于2007年、2012年、2015年和2016年分别在南海北部陆坡进行了4次天然气水合物钻探,均获得天然气水合物样品,证明南海北部陆坡的东部、西部和神狐海域蕴含丰富的天然气水合物[27-29]。本研究选取3个海域海底表层沉积物样品,对其古菌类群进行研究,结合高通量测序的方法分析16S rRNA基因,同时运用定量PCR对南海3个海域的细菌和古菌数量进行定量。对南海深海沉积物中古菌群落结构特征的研究,是深入了解南海沉积物中古菌代谢与功能的重要基础。
1 材料与方法 1.1 样品的采集和保存搭载中国地质调查局广州海洋地质调查局2013-2015年的相关调查航次。采集样品为南海北部陆坡东部、西部和神狐海域潜在水合物区的表层沉积物样品。研究使用的7个沉积样品分别来自于南海北部陆坡东部海域的DS07-2013、DS12-2013、DS16-2013、DS08-2014和DS16-2014站位,南海北部陆坡西部海域的QDN22-2015站位和神狐海域的CHB03-2014站位。本次采集的样品(表 1)为陆坡边缘向深部延伸的位置。水深范围为766 m-1 553 m (平均水深为1 099 m)。采集深度范围为226 cm-850 cm (平均采样深度为469 cm)。采集方法有重力柱(Gravity core)和活塞柱(Piston core)两种。航次中样品的初步采集在甲板上,后期处理在无菌操作箱中进行。步骤如下:沉积柱的分样,前1 m每5 cm分割,1 m后每10 cm分割。用50 mL的无菌离心管保藏于液氮罐中。航次结束后,用液氮罐运回实验室并转移到-80℃,为后续进行微生物分子多样性分析做准备。
| 采样位点 Sites |
深度 depth (m) |
样品深度 Sampling depth (cmbsf**) |
采样方式 Sampling method |
| DS07-2013 | 766 | 350 | Gravity core |
| DS12-2013 | 785 | 291 | Gravity core |
| DS16-2013 | 1 302 | 226 | Gravity core |
| DS08-2014 | 877 | 298 | Gravity core |
| DS16-2014 | 1 007 | 453 | Gravity core |
| CHB03-2014 | 1 553 | 812 | Piston core |
| QDN22-2015 | 1 400 | 850 | Piston core |
| Note: South China Sea*: SCS; cmbsf**: centimeters below seafloor. | |||
BSA溶液,MP Biomedicals,LLC.;ExTaq酶、pMD18-T载体、大肠杆菌DH5α化学感受态细胞、荧光染料SYBR Green Ⅱ,TaKaRa公司;DNA提取试剂盒PowerSoil DNA Isolation kit,美国MoBio公司;胶回收试剂盒E.Z.N.A.® Cycle Pure kit (200)、质粒提取试剂盒,美国Omega生物技术公司;扩增引物,生工生物工程(上海)股份有限公司。GeneAmp® PCR系统9700 PCR仪、7500 Real-Time PCR System荧光定量PCR仪,美国Applied Biosystems公司;离心机Pico17,美国Thermo Scientific公司;超净工作台SW-CJ-2D,苏州净化设备有限公司;DNA浓度测定仪Qubit 3.0 Fluorometer,美国Life Technologies公司。
1.3 核酸提取和PCR扩增沉积物样品约0.5 g,用DNA提取试剂盒提取环境样品总DNA,溶于灭菌的50 μL蒸馏水中。提取操作步骤参照厂家说明书。保存于-20℃。
1.4 古菌16S rRNA基因高通量测序对古菌的16S rRNA基因V4区进行PCR扩增,设置3个平行,引物为Uni519F (5′-CAGYMGCCRC GGKAAHACC-3′)和Arch806R (5′-GGACTACNSG GGTMTCTAAT-3′),并加有Barcode标记。PCR体系(50μL)为:10×ExTaq buffer (Mg2+ plus) 5 μL,0.1 mg/μL的BSA溶液,2.5 mmol/L dNTP mixture 4 μL,5 U/μL ExTaq酶0.5 μL,10 μmol/L引物各1 μL,DNA 1 ng,用双蒸水补充至50 μL。PCR条件:95℃ 10 min;95℃ 45 s,55℃ 1 min,72℃ 1.5 min,35个循环;72℃ 7 min。3个平行的PCR产物混合,经1%琼脂糖凝胶电泳质量检验后,用2%的琼脂糖切胶回收目的条带,操作步骤见厂家说明书。用Gubit 3.0对胶纯化回收后的DNA测定。根据Illumina文库准备的步骤上机测序。
1.5 测序结果与分析测序样品总数为50个,其中南海北部陆坡东部海域的样品数29个,南海北部陆坡西部海域的样品数11个,神狐海域的样品数10个。测序reads总量为2 210 626。其中古菌的序列为1 918 786,占总量的86.8%,说明扩增的样品具有有效性和准确性。将非古菌序列去除,不同的样品用不同的Barcode进行区分,用QIIME软件[30]对原始数据去噪和修整处理,同时去掉Barcode、引物序列、嵌合体和不合格序列[31]。在进行下游分析之前对测序深度标准化(每个样品取12 271条序列),OTU (可分类操作单元)是基于97%的相似性划分的,并在QIIME软件[32]上生成相应的表格。结果中删除了OTU中的单拷贝序列,以去除噪音。序列在QIIME软件[32]上进行α多样性分析。α多样性用Chao1值、Shannon指数和Simpson指数来评估,其中,Chao1值用来估计物种总量[33],平台期的Chao1值越大代表物种总量越多。用Shannon指数和Simpson指数来反映样品中微生物多样性,指数值越大,说明群落多样性越高。基于OTU计算每个样品中各个类群所占百分比,及基于各个类群在总样品的平均数计算各个类群所占总样品的百分比。数据已上传到NCBI的SRA数据库,序列登录号为SRP104332。
1.6 甲烷代谢类群mcrA功能基因系统进化树的构建对潜在水合物区DS08-2014站位次表层(100 cm)样品中mcrA基因构建克隆文库,引物为ME1 (5′-GCMATGCARATHGGWATGTC-3′)和ME2 (5′-TCATKGCRTAGTTDGGRTAGT-3′)[34],PCR体系为:10×ExTaq buffer 5 μL,BSA溶液4 μL,dNTP mixture 4μL,ExTaq酶0.25 μL,10 μmol/L的正、反向引物各1 μL,100 ng的DNA模板,最后用适量的去离子水补充至50μL。PCR条件为:98℃ 2 min;98℃ 10 s,55℃ 30 s,72℃ 1 min,35个循环;72℃ 7 min。产物经1%琼脂糖凝胶电泳质量检验后切胶回收。将扩增的长度约为760 bp的片段连接到pMD18-T载体上,将质粒转化至大肠杆菌DH5α化学感受态细胞上,经过含有氨苄青霉素抗性的LB固体培养基(g/L:胰蛋白胨10.0,酵母粉5.0,NaCl 10.0,琼脂15.0) 的筛选以及菌落PCR的阳性克隆检验,挑取57个阳性克隆送生工生物工程(上海)股份有限公司进行测序。
采用ClustalW[35]软件比对序列;Seaview软件[36]手动修整序列;比对修整后的数据基于近似最大似然(approximately-maximum-likelihood)系统进化树FastTree软件[37]分析;用MEGA 7.0[38]软件对系统进化树修饰。所产生的mcrA基因序列的代表序列提交至GenBank数据库,并获得序列号(KY966976-KY966994)。
1.7 荧光定量PCR (Real-time PCR) 1.7.1 标准品的制备:用定量PCR的方法对南海陆坡沉积物3个海域7个站位(DS07-2013、DS08-2014、DS12-2013、DS16-2013、DS16-2014、CHB03-2014和QDN22-2015站位)的沉积物中16S rRNA基因和潜在水合物区(DS08-2014、DS12-2013、DS16-2013和DS16-2014站位)的mcrA基因进行定量分析。方法如下:对古菌的16S rRNA基因进行扩增,引物为Arch21F (5′-TTCCGGTTGATCCYGCCGGA-3′)和Arch958R (5′-YCCGCGTTGAMTCCAATT-3′)[39];对细菌的16S rRNA基因进行扩增,引物为Bac27F (5′-AGAGTTTGATCCTGGCTCAG-3′)和Bac1492R (5′-GGTTACCTTGTTACGACTT-3′)[40];对甲烷代谢菌的关键基因mcrA进行扩增,引物为mlas (5′-GGTGGTGTMGGDTTCACMCARTA-3′)和mcrA-rev (5′-CGTTCATBGCGTAGTTVGGRTAGT-3′)[41],利用琼脂糖凝胶回收试剂盒纯化和回收PCR产物,连接到pMD18-T载体,转移到Escherichia coli的DH5α感受态细胞中,以氨苄青霉素(100μg/ml)抗性和菌落PCR筛选阳性克隆子,用质粒提取试剂盒回收阳性克隆质粒,用EcoR Ⅰ酶切质粒,用2%的琼脂糖凝胶回收质粒,作为标准品。
1.7.2 标准曲线的构建:标准品经过10倍梯度稀释后用带有荧光染料(SYBR Green Ⅱ)的试剂在荧光定量PCR仪上扩增,每个样品3个重复,构建标准曲线。其中,转化效率在95%-105%之间,R2值在0.99以上,且所有样品的溶解曲线中没有双峰出现。
1.7.3 样品定量:将待测样品和10倍梯度稀释标准品(101-109/μL)的DNA为模板同时进行PCR扩增,得到并转换成待测样品的湿重每克的拷贝数。定量PCR的体系:SYBR® Premix ExTaqTM Ⅱ 10 μL,ROX Ⅱ 0.4 μL,扩增目的片段所用的引物(细菌Bac341F/Bac519R[42-43],古菌Uni519F/Arch908R[43-44]和mcrA基因mlas/mcrA-rev[41])各1 μL,取1 μL原始DNA,用双蒸水补齐到20 μL。定量PCR反应的体系如下:1 μL的DNA模板,10 μmol/L的正反向引物各1.2 μL,10 μL的SYBR Green Ⅱ,0.4 μL的ROX,最后将适量的ddH2O补充总体积至20 μL。PCR的扩增条件为:95℃ 15 min;95℃ 30 s,退火30 s,72℃ 45 s,40个循环。其中退火温度:细菌为58℃,古菌为60℃,mcrA基因为55℃。
2 结果与分析 2.1 南海北部陆坡沉积物古菌主要类群结合高通量测序,对南海北部陆坡沉积物中古菌16s rRNA基因进行测序。基于OTU数、Chao1值、Shannon指数和Simpson指数来反映微生物α多样性。对于南海北部陆坡沉积物3个海域7根柱子的50个样品,Chao1值的变化范围为90.77-118.37,获得的OTU数变化范围为88.2-113.1,Shannon指数的变化范围为1.48-5.73,Simpson指数的变化范围为0.31-0.97。在7根沉积柱中,α多样性最高的为DS1613_195 (Chao1值为118.37),而DS0713_270的α多样性最低(Chao1值为90.77)。
结果表明,南海北部陆坡沉积物中古菌类群主要为:Bathyarchaeota、Thermoplasmata (Marine Benthic Group D)、Woesearchaeota (DHVEG-6)、Thaumarchaeota (Marine Group Ⅰ)、Lokiarchaeota (Marine Benthic Group B)和Marine Hydrothermal Vent Group (MHVG),所占比例依次是36%、17%、16%、12%、11%和3%。除此之外,还存在少量的AK8、Marine Benthic Group A、Terrestrial Hot Spring Crenarchaeota Group (THSCG)等,所占比例小于1%。每个站位古菌类群随深度变化情况如图 1所示。其中,Bathyarchaeota是QDN22-2015站位(图 1A)、CHB03-2014站位(图 1B)、DS08-2014站位(图 1C)、DS12-2013站位(图 1D)和DS16-2013站位(图 1E)的最主要古菌群落;Woesearchaeota (DHVEG-6) 是DS16-2014站位(图 1F)的最主要古菌群落;Thermoplasmata是DS07-2013站位(图 1G)的最主要古菌群落。古菌Bathyarchaeota类群百分含量在次表层达到最大值,如DS12-2013_150 (76.2%,古菌百分比)。Woesearchaeota (DHVEG-6)、Thermoplasmata、Lokiarchaeota的百分比含量随深度变化不明显,如DS16-2013站位和DS16-2014站位。DS08-2014站位和DS12-2013站位的次表层中存在大量具有甲烷厌氧氧化菌的ANME-1类群,如DS08-2014_50 (0.96%)、DS08-2014_100 (7.3%)和DS12-2013_200 (8.81%)。
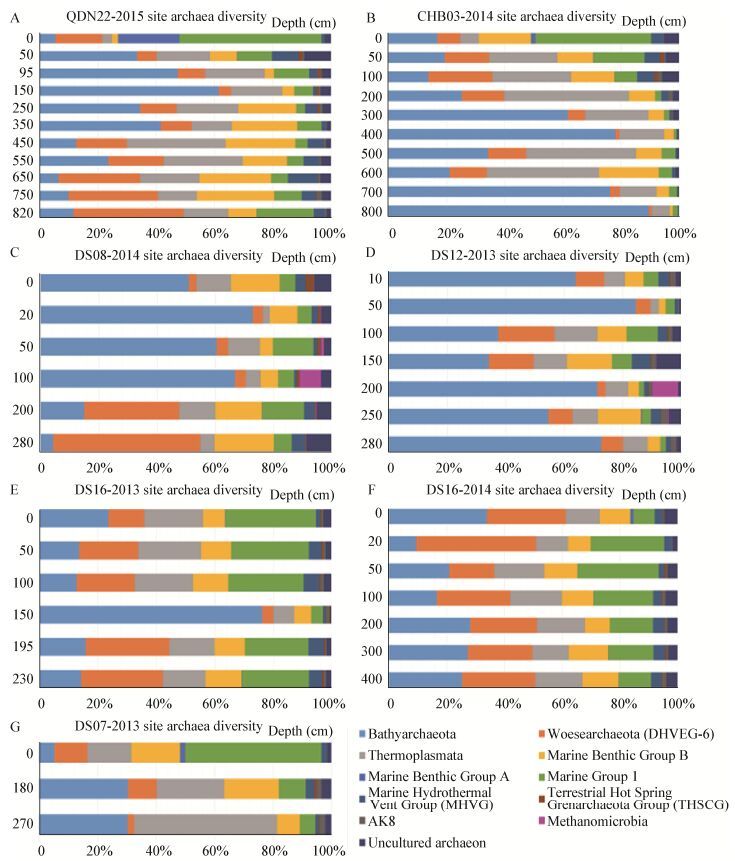
|
| 图 1 南海北部陆坡沉积物7个站位古菌主要类群随深度变化情况 Figure 1 Seven sites sediment archaea diversity changing with deep increase at the northern slope of SCS |
|
|
主成分分析(Principal components analysis,PCA)是一种非约束性排序方法的统计学方法。基于OTU所占百分比的主成分分析(PCA)[45],对中国南海北部陆坡沉积物中的古菌进行类群分析(图 2),可知在沉积物表层Marine Group Ⅰ和Marine Benthic Group A类群含量较高,如QDN22-2015_0 (48.61%,21.14%)、DS07-2013_0 (46.52%,1.83%)和CHB03-2014_0 (39.67%,1.65%);在潜在水合物区Methanomicrobia和Bathyarchaeota类群有较高含量,如DS12-2013_200 (8.81%,71.33%)、DS08-2014_100 (7.30%,66.76%)和DS08-2014_050 (0.96%,60.35%)等。

|
| 图 2 南海北部陆坡沉积物(7个站位)基于OTU所占百分比的PCA分析 Figure 2 PCA analysis based on OTU percent at the northern slope of SCS (7 sites) |
|
|
对潜在水合物区DS08-2014站位的甲烷代谢类群进一步分析。构建16S rRNA基因系统进化树,可知DS08-2014站位的甲烷代谢类群为ANME-1 (99.7%)和ANME-2c (0.3%)。其中,ANME-1是主要的甲烷代谢类群。同时基于mcrA基因克隆文库分析,挑取DS08-2014站位次表层(DS08-2014_100,57条序列)甲烷代谢类群,构建系统进化树,可知DS0814站位的甲烷代谢类群为:ANME-1 (84.2%),ANME-2ab (10.5%)和ANME-2c (5.3%) (图 3)。结合16S rRNA基因和mcrA基因的系统进化树,可知南海北部陆坡沉积物潜在水合物区DS08-2014站位的主要甲烷代谢类群为ANME-1、少量ANME-2ab和ANME-2c类群。

|
| 图 3 潜在水合物区DS08-2014站位次表层(100 cm) mcrA基因的系统发育树 Figure 3 Phylogenetic tree showing the relationship of mcrA gene sequences derived from potential gas hydrate zone DS08-2014 site subsurface (100 cm) sediment 注:基于最大似然算法,Bootstrap值选自1 000重复,黑点标志的序列为本实验数据,最后的数字为序列的条数,其余为参考序列,参考序列选自NCBI数据库. Note: Based on maximum likelihood analysis, numbers on branches are the supporting percentage by 1 000 replicates. Sequences retrieved in this study are marked by black circle, the last number is sequence's number, the other are get from NCBI database. |
|
|
采用荧光定量PCR的方法对来自3个海域的潜在水合物区(DS12-2013、DS16-2013、DS08-2014和DS16-2014站位) (图 4)和对照区(DS07-2013、CHB03-2014、QDN22-2015站位)沉积物中的细菌和古菌的16S rRNA基因和mcrA基因进行定量分析,发现细菌16S rRNA基因的丰度范围为(5.52±0.38)×105-(2.86±0.14)×107 copies/g (湿重)。随着深度的增加,细菌16S rRNA基因的丰度趋于减少。古菌16S rRNA基因的丰度相对于细菌低1-2个数量级,其丰度范围为(1.61±0.20)×104-(6.72±0.73)×106 copies/g (湿重)。整体而言,随着深度的增加,古菌的丰度趋于减少,但在某些层位出现陡增的情况,如DS12-2013_100和DS16-2013_150等。mcrA基因作为甲烷代谢菌的标记基因,其丰度可暗示潜在水合物的强弱。在潜在水合物区,mcrA基因的丰度范围为(1.03±0.16)×103-(7.10±0.78)×105 copies/g。其中,在DS08-2014站位(图 4A)从表层到深层均能检测到mcrA基因的存在,丰度变化范围为(1.15±0.20)×103-(7.10±0.78)×105 copies/g (湿重)。DS12-2013站位(图 4B)和DS16-2013站位(图 4C)在次表层有mcrA基因的陡增,最大值分别为(3.01±0.30)×105 copies/g (湿重)和(1.99±0.73)× 104 copies/g (湿重),DS16-2014站位(图 4D)只有表层检测到mcrA基因,丰度变化范围为(1.41±0.48)× 103-(3.82±0.29)×103 copies/g (湿重),mcrA基因的定量结果可提供间接证据证明该区域含有丰富的甲烷代谢类群。对照区没有检测到mcrA基因。
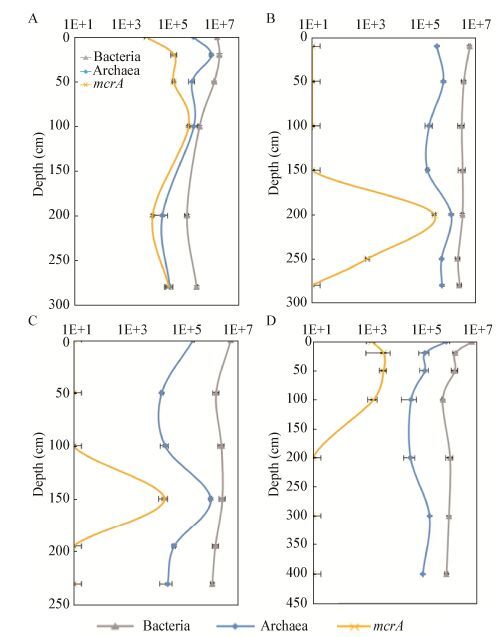
|
| 图 4 潜在水合物区细菌、古菌和mcrA基因的定量数据 Figure 4 Quantification data of bacteria, archaea and mcrA gene in potential gas hydrate zone 注:A:DS08-2014站位的微生物定量结果;B:DS12-2013站位的微生物定量结果;C:DS16-2013站位的微生物定量结果;D:DS16-2014站位的微生物定量结果;单位为copies/g. Note: A: DS08-2014 site microbial abundance; B: DS12-2013 site microbial abundance; C: DS16-2013 site microbial abundance; D: DS16-2014 site microbial abundance; Unit is copies/g. |
|
|
本研究调查了南海北部陆坡3个海域(东部、西部和神狐海域)沉积物中的古菌类群。采集了7个沉积柱共50个样品。结合高通量测序的方法,可知南海北部陆坡沉积物覆盖了古菌的3大超级门:“TACK”超级亚门,“DPANN”超级亚门和Euryarchaeota门。其中,“TACK”超级亚门主要为Bathyarchaeota、Thaumarchaeota类群,而“DPANN”超级亚门主要为Woesearchaeota (DHVEG-6) 类群和少量Marine Hydrothermal Vent Group (MHVG)类群,Euryarchaeota门主要为Thermoplasmata (MBGD)类群和Methanomicrobiales类群(图 1)。
作为南海沉积物中的最主要类群,Bathyarchaeota类群广泛存在于各种环境,如深海沉积物、热泉、盐碱地等,且至少可被分为17个亚群,宏基因组学和单细胞测序的结果推测其具有分解蛋白质和降解苯环化合物的功能[11, 46]。近些年的研究表明,Bathyarchaeota类群可能具有自养产乙酸和参与甲烷代谢的功能,是兼性异氧代谢类型[22, 47]。在Nankai Trough水合物层位中也发现了大量的Bathyarchaeota类群,并在Bathyarchaeota的基因组中发现了完整的甲烷代谢途径,暗示其在甲烷代谢过程可能发挥作用[48]。本研究的潜在水合物区DS08-2014站位中,特别是含有ANME-1类群的层位,Bathyarchaeota类群均是古菌群落的主要组成成分(图 2),推测Bathyarchaeota在潜在水合物区碳循环中发挥着重要的作用。
南海北部陆坡3个海域沉积物中古菌的主要类群为“TACK”超级亚门的Bathyarchaeota (36%),Euryarchaeota的Thermoplasmata (17%)和“DPANN”超级亚门的Woesearchaeota (16%),而焦露等[49]在中国南海北部神狐海域沉积物古菌多样性研究表明,Euryarchaeota的Halobacteriales (42%)、“TACK”超级亚门的Bathyarchaeota (25.1%)、“DPANN”超级亚门的Wosesarchaeota (5.2%)为主要沉积物古菌类群,与本研究的结果不同。分析其原因:一方面是由测序方法和采集柱自身因素不同造成的。本研究采用的是高通量测序的方法,比构建克隆文库方法[49]的测序能获得更大数据量,更能反映原位的古菌类群的多样性和丰度信息,特别是低丰度和特殊环境的沉积物样品。此外,同一区域不同站位的微生物多样性受到多种因素的影响,如化学参数,是否为水合物区等因素。另一方面,随着近些年来古菌的系统进化树不断的更新,如新基因组的发现和16S rRNA基因测序结果的填补和归类[10, 50-51],都是造成古菌多样性结果不同的原因。综上所述,南海北部陆坡沉积物中的古菌类群主要参与碳元素的生物地球化学循环。沉积物中古菌类群的生理生态学意义正在逐渐被理解。
3.2 南海北部陆坡沉积物微生物的丰度南海北部陆坡沉积物(7个站位)中细菌16S rRNA基因的丰度范围为105-107 copies/g (湿重);古菌的16S rRNA基因的丰度范围为103-106 copies/g (湿重)。在潜在水合物区,mcrA基因的丰度范围约103-105 copies/g (湿重) (图 4)。本研究的结果显示细菌丰度在表层最高,与其它南海沉积物中的微生物丰度变化相似[49, 52]。海底沉积物表层的有机物含量相对较高,为微生物的生长提供丰富的底物,随着深度的增加,有机物含量减少,细菌丰度逐渐减少。古菌的丰度随着深度的变化波动较大,推测与某些古菌类群独特的生理特性有关,如潜在水合物区DS08-2014站位,古菌丰度随深度变化波动较大,同时检测到较高的mcrA基因。结合PCA分析,Methanomicrobia类群和Bathyarchaeota类群在DS08-2014站位具有较高的丰度(图 1和2),暗示古菌在该站位的地球化学元素循环上起到重要作用。大部分样品中,细菌16S rRNA基因拷贝数高出细菌1-2个数量级。部分层位中,古菌和细菌的16S rRNA基因的丰度同处一个数量级,如DS08-2014_100,可能与细菌和古菌不同的代谢特征有关。对南海北部陆坡沉积物中细菌和古菌的16S rRNA基因和mcrA基因进行定量分析,不仅为了解南海北部陆坡沉积物中微生物资源的储量提供基础信息,且为后续深入研究古菌和细菌在参与全球海洋沉积物生物地球化学循环提供重要依据。值得一提的是,本实验中古菌多样性的引物选用的是古菌16S rRNA基因V4区,相比于全长和其它分区能降低古菌基因组内16S rRNA基因多样性造成的影响[53],但在对微生物的丰度分析时,细菌的定量引物选用的是16S rRNA基因的V3区,古菌的定量引物选用的是16S rRNA基因的V4区,由于细菌和古菌基因组内存在16S rRNA基因的多样性,因此对细菌和古菌的丰度分析存在高估,且细菌的高估情况较古菌明显[53]。
3.3 南海北部陆坡沉积物DS08-2014站位的甲烷代谢类群南海北部陆坡沉积物中潜在水合物区来源的DS08-2014站位含有丰富的甲烷代谢类群,结合高通量测序结果和构建的克隆文库,对16S rRNA基因和mcrA基因的系统进化树进行分析,可知南海北部陆坡潜在水合物区DS08-2014站位的甲烷代谢类群主要为甲烷厌氧氧化古菌ANME-1,同时还存在少量ANME-2ab和ANME-2c。同一海域DS12-2013站位也存在类似的甲烷代谢群落,可见南海北部陆坡东部海域潜在水合物区沉积物中的主要甲烷类群为ANME-1、ANME-2ab和ANME-2c类群。相比李涛等[54]对南海南部陆坡表层沉积物的古菌多样性研究:甲烷代谢类群主要是甲烷杆菌(Methanobacteriales),本研究南海北部陆坡沉积物的甲烷代谢类群主要为Methanomicrobia纲的ANME-1类群。推测可能与垂直取样深度及古菌不同的生态位有关。综上所述,南海北部陆坡沉积物中甲烷代谢类群的多样性为指示沉积物中甲烷厌氧氧化活动提供重要依据。
4 结论本研究通过高通量测序的方法,对中国南海北部陆坡3个海域(东部、西部和神狐海域)沉积物中微生物进行分析,结合定量分析,获得南海北部陆坡沉积物中古菌多样性和微生物丰度的基本数据。南海北部陆坡沉积物古菌的主要类群为:Bathyarchaeota、Thermoplasmata、Woesearchaeota (DHVEG-6)、Thaumarchaeota (Marine Group Ⅰ)、Lokiarchaeota和Marine Hydrothermal Vent Group (MHVG)等。对含有丰富甲烷代谢类群的DS08-2014站位,结合16s rRNA基因和mcrA基因的系统发育树进一步分析,发现ANME-1、ANME-2ab和ANME-2c类群为南海北部陆坡东部海域潜在水合物区沉积物的甲烷代谢类群。为深入了解中国南海北部陆坡沉积物古菌多样性及甲烷代谢类群提供数据支持。
致谢: 感谢“海洋四号”船员协助采集样品。| [1] | Kallmeyer J, Pockalny R, Adhikari RR, et al. Global distribution of microbial abundance and biomass in subseafloor sediment[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(40) : 16213–16216. DOI:10.1073/pnas.1203849109 |
| [2] | Dʼhondt S, J rgensen BB, Miller DJ, et al. Distributions of microbial activities in deep subseafloor sediments[J]. Science, 2004, 306(5705) : 2216–2221. DOI:10.1126/science.1101155 |
| [3] | Woese CR, Kandler O, Wheelis ML. Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya[J]. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(12) : 4576–4579. DOI:10.1073/pnas.87.12.4576 |
| [4] | Auguet JC, Casamayor EO. A hotspot for cold crenarchaeota in the neuston of high mountain lakes[J]. Environmental Microbiology, 2008, 10(4) : 1080–1086. DOI:10.1111/j.1462-2920.2007.01498.x |
| [5] | Liu YC, Whitman WB. Metabolic, phylogenetic, and ecological diversity of the methanogenic archaea[J]. Annals of the New York Academy of Sciences, 2008, 1125(1) : 171–189. DOI:10.1196/annals.1419.019 |
| [6] | Lipp JS, Morono Y, Inagaki F, et al. Significant contribution of Archaea to extant biomass in marine subsurface sediments[J]. Nature, 2008, 454(7207) : 991–994. DOI:10.1038/nature07174 |
| [7] | Guy L, Ettema TJG. The archaeal ʻTACKʼ superphylum and the origin of eukaryotes[J]. Trends in Microbiology, 2011, 19(12) : 580–587. DOI:10.1016/j.tim.2011.09.002 |
| [8] | Spang A, Hatzenpichler R, Brochier-Armanet C, et al. Distinct gene set in two different lineages of ammonia-oxidizing archaea supports the phylum Thaumarchaeota[J]. Trends in Microbiology, 2010, 18(8) : 331–340. DOI:10.1016/j.tim.2010.06.003 |
| [9] | Rinke C, Schwientek P, Sczyrba A, et al. Insights into the phylogeny and coding potential of microbial dark matter[J]. Nature, 2013, 499(7459) : 431–437. DOI:10.1038/nature12352 |
| [10] | Hug LA, Baker BJ, Anantharaman K, et al. A new view of the tree of life[J]. Nature Microbiology, 2016, 1(5) : 16048. DOI:10.1038/nmicrobiol.2016.48 |
| [11] | Meng J, Xu J, Qin D, et al. Genetic and functional properties of uncultivated MCG archaea assessed by metagenome and gene expression analyses[J]. The ISME Journal, 2014, 8(3) : 650–659. DOI:10.1038/ismej.2013.174 |
| [12] | Kozubal MA, Romine M, de Jennings R, et al. Geoarchaeota: a new candidate phylum in the Archaea from high-temperature acidic iron mats in Yellowstone National Park[J]. The ISME Journal, 2013, 7(3) : 622–634. DOI:10.1038/ismej.2012.132 |
| [13] | Teske A. Archaea in deep marine subsurface sediments[A]//Kallmeyer J, Wagner D. Microbial Life of the Deep Biosphere[M]. Berlin, Germany: De Gruyter, 2014: 143-160 |
| [14] | Groves DG, Hunt LM. Ocean world encyclopedia[M]. New York: McGraw-Hill, 1980 |
| [15] | Boucher O, Friedlingstein P, Collins B, et al. The indirect global warming potential and global temperature change potential due to methane oxidation[J]. Environmental Research Letters, 2009, 4(4) : 044007. DOI:10.1088/1748-9326/4/4/044007 |
| [16] | Cho H, Hyun JH, You OR, et al. Microbial community structure associated with biogeochemical processes in the Sulfate-Methane Transition Zone (SMTZ) of gas-hydrate-bearing sediment of the Ulleung Basin, East Sea[J]. Geomicrobiology Journal, 2016, 34(3) : 207–219. |
| [17] | Niew hner C, Hensen C, Kasten S, et al. Deep sulfate reduction completely mediated by anaerobic methane oxidation in sediments of the upwelling area off Namibia[J]. Geochimica et Cosmochimica Acta, 1998, 62(3) : 455–464. DOI:10.1016/S0016-7037(98)00055-6 |
| [18] | Moore TS, Murray RW, Kurtz AC, et al. Anaerobic methane oxidation and the formation of dolomite[J]. Earth and Planetary Science Letters, 2004, 229(1/2) : 141–154. |
| [19] | Knittel K, Boetius A. Anaerobic oxidation of methane: progress with an unknown process[J]. Annual Review of Microbiology, 2009, 63(1) : 311–334. DOI:10.1146/annurev.micro.61.080706.093130 |
| [20] | Borrel G, O'Toole PW, Harris HMB, et al. Phylogenomic data support a seventh order of methylotrophic methanogens and provide insights into the evolution of methanogenesis[J]. Genome Biology and Evolution, 2013, 5(10) : 1769–1780. DOI:10.1093/gbe/evt128 |
| [21] | Fang XY, Li JB, Rui JP, et al. Research progress in biochemical pathways of methanogenesis[J]. Chinese Journal of Applied and Environmental Biology, 2015, 21(1) : 1–9. (in Chinese) 方晓瑜, 李家宝, 芮俊鹏, 等. 产甲烷生化代谢途径研究进展[J]. 应用与环境生物学报, 2015, 21(1) : 1–9. |
| [22] | Evans PN, Parks DH, Chadwick GL, et al. Methane metabolism in the archaeal phylum Bathyarchaeota revealed by genome-centric metagenomics[J]. Science, 2015, 350(6259) : 434–438. DOI:10.1126/science.aac7745 |
| [23] | Laso-Pérez R, Wegener G, Knittel K, et al. Thermophilic archaea activate butane via alkyl-coenzyme M formation[J]. Nature, 2016, 539(7629) : 396–401. DOI:10.1038/nature20152 |
| [24] | Friedrich MW. Methyl-coenzyme M reductase genes: Unique functional markers for methanogenic and anaerobic methane-oxidizing Archaea[J]. Methods in Enzymology, 2005, 397 : 428–442. DOI:10.1016/S0076-6879(05)97026-2 |
| [25] | Hallam SJ, Putnam N, Preston CM, et al. Reverse methanogenesis: testing the hypothesis with environmental genomics[J]. Science, 2004, 305(5689) : 1457–1462. DOI:10.1126/science.1100025 |
| [26] | Shima S, Krueger M, Weinert T, et al. Structure of a methyl-coenzyme M reductase from Black Sea mats that oxidize methane anaerobically[J]. Nature, 2012, 481(7379) : 98–101. |
| [27] | Gong YH, Wu SG, Zhang GX, et al. Relation between gas hydrate and geologic structures in Dongsha islands sea area of South China Sea[J]. Marine Geology & Quaternary Geology, 2008, 28(1) : 99–104. (in Chinese) 龚跃华, 吴时国, 张光学, 等. 南海东沙海域天然气水合物与地质构造的关系[J]. 海洋地质与第四纪地质, 2008, 28(1) : 99–104. |
| [28] | Wu NY, Yang SX, Wang HB, et al. Gas-bearing fluid influx sub-system for gas hydrate geological system in Shenhu Area, Northern South China Sea[J]. Chinese Journal of Geophysics, 2009, 52(6) : 1641–1650. (in Chinese) 吴能友, 杨胜雄, 王宏斌, 等. 南海北部陆坡神狐海域天然气水合物成藏的流体运移体系[J]. 地球物理学报, 2009, 52(6) : 1641–1650. |
| [29] | Wu DD, Ye Y, Wu NY, et al. Early diagenesis records and chemical composition abnormalities in pore water for methane-seep in sediments from the southern Qiongdong Basin[J]. Acta Oceanologica Sinica, 2009, 31(2) : 86–96. (in Chinese) 邬黛黛, 叶瑛, 吴能友, 等. 琼东南盆地与甲烷渗漏有关的早期成岩作用和孔隙水化学组分异常[J]. 海洋学报, 2009, 31(2) : 86–96. |
| [30] | Caporaso JG, Kuczynski J, Stombaugh J, et al. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5) : 335–336. DOI:10.1038/nmeth.f.303 |
| [31] | Edgar RC, Haas BJ, Clemente JC, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16) : 2194–2200. DOI:10.1093/bioinformatics/btr381 |
| [32] | Kuczynski J, Stombaugh J, Walters WA, et al. Using QIIME to analyze 16S rRNA gene sequences from microbial communities[J]. Current Protocols in Microbiology, 2012. DOI:10.1002/9780471729259.mc01e05s27 |
| [33] | Chao A. Nonparametric estimation of the number of classes in a population[J]. Scandinavian Journal of Statistics, 1984, 11(4) : 265–270. |
| [34] | Hales BA, Edwards C, Ritchie DA, et al. Isolation and identification of methanogen-specific DNA from blanket bog peat by PCR amplification and sequence analysis[J]. Applied and Environmental Microbiology, 1996, 62(2) : 668–675. |
| [35] | Larkin MA, Blackshields G, Brown NP, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21) : 2947–2948. DOI:10.1093/bioinformatics/btm404 |
| [36] | Gouy M, Guindon S, Gascuel O. SeaView version 4: a multiplatform graphical user interface for sequence alignment and phylogenetic tree building[J]. Molecular Biology and Evolution, 2010, 27(2) : 221–224. DOI:10.1093/molbev/msp259 |
| [37] | Price MN, Dehal PS, Arkin AP. FastTree 2-approximately maximum-likelihood trees for large alignments[J]. PLoS One, 2010, 5(3) : e9490. DOI:10.1371/journal.pone.0009490 |
| [38] | Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7) : 1870–1874. DOI:10.1093/molbev/msw054 |
| [39] | DeLong EF. Archaea in coastal marine environments[J]. Proceedings of the National Academy of Sciences of the United States of America, 1992, 89(12) : 5685–5689. DOI:10.1073/pnas.89.12.5685 |
| [40] | Weisburg WG, Barns SM, Pelletier DA, et al. 16S ribosomal DNA amplification for phylogenetic study[J]. Journal of Bacteriology, 1991, 173(2) : 697–703. DOI:10.1128/jb.173.2.697-703.1991 |
| [41] | Steinberg LM, Regan JM. Phylogenetic comparison of the methanogenic communities from an acidic, oligotrophic fen and an anaerobic digester treating municipal wastewater sludge[J]. Applied and Environmental Microbiology, 2008, 74(21) : 6663–6671. DOI:10.1128/AEM.00553-08 |
| [42] | Ishii K, Fukui M. Optimization of annealing temperature to reduce bias caused by a primer mismatch in multitemplate PCR[J]. Applied and Environmental Microbiology, 2001, 67(8) : 3753–3755. DOI:10.1128/AEM.67.8.3753-3755.2001 |
| [43] | Ovre s L, Forney L, Daae FL, et al. Distribution of bacterioplankton in meromictic Lake Saelenvannet, as determined by denaturing gradient gel electrophoresis of PCR-amplified gene fragments coding for 16S rRna[J]. Applied and Environmental Microbiology, 1997, 63(9) : 3367–3373. |
| [44] | Jurgens G, Lindstr m K, Saano A. Novel group within the kingdom Crenarchaeota from boreal forest soil[J]. Applied and Environmental Microbiology, 1997, 63(2) : 803–805. |
| [45] | Lai J S, Mi X C. Ordination analysis of Ecological data using Vegan package in R[J]. Advances in Biodiversity Conservation and Research in China, 2005 : 332–343. |
| [46] | Lloyd KG, Alperin MJ, Teske A. Environmental evidence for net methane production and oxidation in putative ANaerobic MEthanotrophic (ANME) archaea[J]. Environmental Microbiology, 2011, 13(9) : 2548–2564. DOI:10.1111/j.1462-2920.2011.02526.x |
| [47] | He Y, Li M, Perumal V, Feng X, Fang J, Xie J, Sievert S, Wang F. Genomic and enzymatic evidence for acetogenesis among multiple lineages of the archaeal phylum Bathyarchaeota widespread in marine sediments[J]. Nature Microbiology, 2016, 1(6) : 16035. DOI:10.1038/nmicrobiol.2016.35 |
| [48] | Katayama T, Yoshioka H, Takahashi HA, et al. Changes in microbial communities associated with gas hydrates in subseafloor sediments from the Nankai Trough[J]. FEMS Microbiology Ecology, 2016, 92(8) : fiw093. DOI:10.1093/femsec/fiw093 |
| [49] | Jiao L, Su X, Chen F, et al. Microbial diversity in sediments of core HS-PC 500 from Shenhu Area, northern South China Sea[J]. Acta Microbiologica Sinica, 2011, 51(7) : 876–890. (in Chinese) 焦露, 苏新, 陈芳, 等. 南海北部陆坡神狐海域HS-PC500岩心微生物多样性[J]. 微生物学报, 2011, 51(7) : 876–890. |
| [50] | Castelle CJ, Wrighton KC, Thomas BC, et al. Genomic expansion of domain archaea highlights roles for organisms from new phyla in anaerobic carbon cycling[J]. Current Biology, 2015, 25(6) : 690–701. DOI:10.1016/j.cub.2015.01.014 |
| [51] | Ortiz-Alvarez R, Casamayor EO. High occurrence of Pacearchaeota and Woesearchaeota (Archaea superphylum DPANN) in the surface waters of oligotrophic high-altitude lakes[J]. Environmental Microbiology Reports, 2016, 18(2) : 210–217. |
| [52] | Inagaki F, Nunoura T, Nakagawa S, et al. Biogeographical distribution and diversity of microbes in methane hydrate-bearing deep marine sediments on the Pacific Ocean Margin[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(8) : 2815–2820. DOI:10.1073/pnas.0511033103 |
| [53] | Sun DL, Jiang X, Wu QL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19) : 5962–5969. DOI:10.1128/AEM.01282-13 |
| [54] | Li T, Wang P, Wang PX. Bacterial and archaeal diversity in surface sediment from the south slope of the South China Sea[J]. Acta Microbiologica Sinica, 2008, 48(3) : 323–329. (in Chinese) 李涛, 王鹏, 汪品先. 南海南部陆坡表层沉积物细菌和古菌多样性[J]. 微生物学报, 2008, 48(3) : 323–329. |
 2017, Vol. 44
2017, Vol. 44




