扩展功能
文章信息
- 鲁辛辛, 冯乐, 于永慧, 罗声栋, 孙志会, 王涛, 端青, 王宁利, 宋立华
- LU Xin-Xin, FENG Le, YU Yong-Hui, LUO Sheng-Dong, SUN Zhi-Hui, WANG Tao, DUAN Qing, WANG Ning-Li, SONG Li-Hua
- 青海藏区B型沙眼衣原体的分离培养与鉴定
- Isolation and characterization of ocular Chlamydia trachomatis from Qinghai China
- 微生物学通报, 2017, 44(6): 1444-1450
- Microbiology China, 2017, 44(6): 1444-1450
- DOI: 10.13344/j.microbiol.china.160676
-
文章历史
- 收稿日期: 2016-09-21
- 接受日期: 2017-01-13
- 优先数字出版日期(www.cnki.net): 2017-01-17
2. 病原微生物生物安全国家重点实验室 军事医学科学院微生物流行病研究所 北京 100071;
3. 北京市眼科研究所 北京 100005
2. State Key Laboratory of Pathogen and Biosecurity, Institute of Microbiology and Epidemiology, Academy of Military Medical Sciences, Beijing 100071, China;
3. Beijing Institute of Ophthalmology, Beijing 100005, China
沙眼是由眼型沙眼衣原体感染眼上皮组织导致的一种极古老的人类特有疾病,历史上曾严重危害国人健康。在欠发达国家和地区,沙眼是首位的可预防型致盲病因[1]。在我国,沙眼已不再是重要的致盲因素,但沙眼仍在很多地区流行,例如甘肃、湖南、湖北、辽宁、宁夏、河北、内蒙古等地区近年来都有沙眼流行病学报道[2-6]。在沙眼病原学领域,汤飞凡和张晓楼等在1956年首次报道了沙眼病原的分离培养[7],随后十年间有46株衣原体在北京、广东、济南等地被分离到[8],由于各种原因这些菌株多数已经失传。
眼型沙眼衣原体包括A、B、Ba、C等血清型或ompA基因型。汤飞凡的早期分离株TE55被称为汤氏株,为C型[2, 8]。北京市眼科研究所在1991年和2007年的流行病学调查发现,我国华北地区的沙眼流行株为B型和C型,二者病例数的比例约为4:1[2, 9]。我国台湾省的流行株也限于B型和C型,代表菌株有B/TW5和C/TW3[10]。由于培养困难等原因,近30年来我国还没有沙眼病原的分离培养报道。我国幅员辽阔,绝大多数地区的沙眼病原还有待调查研究。
沙眼是一种有望被彻底消灭的疾病。世界卫生组织制定了SAFE策略,提出了2020年全球消灭致盲性沙眼的目标[11]。SAFE策略取得了显著成绩,但由于缺乏沙眼疫苗,很多科学家认为缺乏可持续性。本研究对青海藏区45份活动性沙眼患者的临床样本进行了衣原体分离培养,共获得15株基因B型沙眼衣原体。ompA基因序列分析发现,该地区的眼型沙眼衣原体存在微进化,共有3个ompA流行株,但都有UGT (urogenital tract,泌尿生殖道)型密码子特征。本研究对我国的沙眼防治和理解沙眼进化有重要意义。
1 材料与方法 1.1 沙眼标本采集北京同仁医院王宁利、鲁辛辛带领医生先后于2015年5月25日对青海省循化县尕楞乡建设堂小学和2015年12月31日对尕楞乡中心小学学生进行沙眼调查及眼部采样,标本采集方法参照文献[12]。样本采集后保存在1 mL Copan样本保护培养基中,至冰盒中暂存,空运至北京同仁医院实验室,样本分装后转运至军事医学科学院微生物流行病研究所立克次体室进行沙眼衣原体分离培养。
1.2 沙眼衣原体的分离培养在每份拟用于培养的样本中加入等体积SPG (0.01 mol/L磷酸钠,0.25 mol/L蔗糖,5 mmol/L L-谷氨酸)缓冲液,-80冻存。取100 µL处理后样本(相当于总样本量的1/20) 加入至96孔BGM细胞板单孔中,800×g离心30 min,弃接种物,补加100 µL DMEM (含1 mg/L放线菌酮、100 mg/L万古霉素、5 mg/L两性霉素B、10 mg/L庆大霉素)[13],置37培养箱培养3 d,连续传代3次。每代培养48 h后均用相差显微镜观察培养结果。
1.3 免疫荧光染色接种BGM细胞至盖玻片,感染沙眼衣原体,感染36 h后用甲醇固定,PBS洗3次,加入衣原体属特异性LPS单抗(美国Santa Cruz公司),37反应1 h,PBS洗3次,加入Alexa488标记的抗鼠二抗(美国Invitrogen公司),PBS洗3次,用DAPI染色DNA,共聚焦荧光显微镜观察成像。
1.4 基因序列分析用蛋白酶K消化,酚氯仿异戊醇抽提法纯化衣原体DNA[14]。用引物5′-CGCGGATCCatgaaaaaactctt gaaatcggtattagtattt-3′和5′-TTTTCCTTTTGCGGCCG Cttagaagcggaattgtgcatttacg-3′及Phusion高保真酶(NEB公司)扩增ompA基因。BamHⅠ和NotⅠ双酶切后连接至载体pGEX-4T-1,转化Escherichia coli DH5α,挑取单菌落送生工生物工程(上海)股份有限公司测序,至少测3个克隆。用Mega 7.0软件进行序列比对和系统进化分析[15]。进化树(Bootstrap consensus tree)的构建采用邻接法和1 000次Bootstrap检验[16-17]。使用最大组成似然模型(Maximum composite likelihood)估算遗传距离[18]。
1.5 统计分析采用Excel 2013软件对数据进行χ2检验统计学分析,P<0.05为差异有统计学意义。
2 结果与分析 2.1 标本采集与处理青海省循化县尕楞乡建设堂小学和尕楞乡中心小学共有45名学生被诊断为疑似沙眼患者(至少有一只眼有一个以上沙眼滤泡),共采集了115份拭子标本(分别采集左、右眼结膜标本,个别患者只采单眼或将双眼采至一个样本管,部分患者同时采集结膜囊样本,或将左右眼结膜囊样本分别采集)。对全部样本进行qPCR检测质粒基因、ompA序列分析和病原分离培养,其中qPCR与样本的ompA测序由北京同仁医院完成,部分结果参见文献[12]。
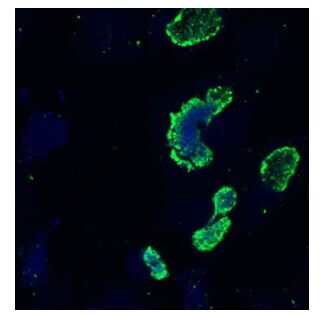
2.2 沙眼衣原体分离培养结果在BGM细胞上,青海沙眼衣原体菌株可以形成典型、致密的包涵体,经衣原体属特异性单抗免疫荧光染色后可观察到形状不规则的包涵体(图 1)。共有15份标本培养阳性,将15株新分离沙眼衣原体分别编号为:QH111R、QH111L、QH113R、QH118L、QH118S、QH120S、QH123R、QH123S1、QH123S2、QH155R、QH155L、QH155S、QH159R、QH159L和QH159S。这里QH代表青海(Qing Hai),QH后数字为患者编号,L代表来自左眼上睑结膜标本,R代表来自右眼上睑结膜标本,S代表来自结膜囊标本。
|
| 图 1 沙眼衣原体QH111R感染BGM细胞36 h后的免疫荧光染色结果 Figure 1 Immunofluorescence of BGM cells infected with Chlamydia trachomatis QH111R at 36 h post-infection |
|
|
将分离培养结果与qPCR检测结果进行比较。从表 1可以看出,115份临床诊断为滤泡性沙眼的样本中有54份样本为qPCR阳性,15份样本为分离培养阳性且这些样本均为qPCR阳性,分离培养法的检测灵敏度显著低于qPCR (χ2=19.5,P<0.01),这可能与实验中使用了少量样本进行分离培养有关。qPCR结果更好地反映了样本中的菌体载量与沙眼的病程发展有关。120份当地小学生的正常结膜拭子均为qPCR阴性,说明滤泡的存在与衣原体感染直接相关(χ2=73.2,P<0.01) (表 2)。但45份滤泡个数≥5的标本中仅有21份qPCR阳性(46.7%),则说明滤泡个数与菌体载量没有必然关联(χ2=0.002,P=0.96) (表 3)。以上结果也说明两种沙眼病原检测方法在临床应用中均存在较高的假阴性。
| 分离培养法检测结果 Culture results |
标本数 Sample counts |
|||
| qPCR检测结果: qPCR results |
+ | - | 合计 Total |
|
| + | 15 | 0 | 15 | |
| - | 39 | 61 | 100 | |
| 合计 Total | 54 | 61 | 115 | |
| 眼部有无滤泡 With (+) or without (-) follicles |
标本数 Sample counts |
|||
| qPCR检测结果: qPCR results |
+ | - | 合计 Total |
|
| + | 54 | 61 | 115 | |
| - | 0 | 120 | 120 | |
| 合计 Total | 54 | 181 | 235 | |
| 眼部滤泡个数 Follicle counts |
标本数 Sample counts |
|||
| qPCR检测结果: qPCR results |
+ | - | 合计 Total |
|
| ≥5 | 21 | 24 | 45 | |
| 1-4 | 33 | 37 | 70 | |
| 合计 Total | 54 | 61 | 115 | |
主要外膜蛋白MOMP与其基因ompA分别是衣原体血清型和基因型分型标志[19]。Li等报道了37份样本ompA分析的部分结果[12]。这里,我们对37份样本的ompA进行深度再分析,发现共有3种ompA序列,其中30份样本的ompA序列完全相同(选择111R为代表样本),另有6份样本(选118L为代表样本)和1份样本(111L)的ompA与111R相比分别在可变区887位和511位各有一个不同的非同义碱基差异。115份沙眼样本的背景信息、检测结果和ompA分析结果见表 4。编号111患者的左右眼样本111L和111R有不同的ompA,它们有一个碱基差异。与111R的ompA序列(GenBank登录号:KU737520) 相比,111L的ompA在511位发生了非同义碱基改变(G>A)。重要的是,QH111L和QH111R分离株的ompA测序结果与样本分析结果相一致,说明QH111L可能是新出现的ompA突变体。
| 患者编号 Patient ID |
性别 Gender (F/M) |
年龄 Age (Y) |
滤泡数目 Follicle counts |
qPCR检测结果 qPCR results |
分离培养结果 Culture results |
|||||||
| OD | OS | OD | OS | CS | OD | OS | CS | |||||
| 101 | F | 9 | 1 | 2 |  |
ND | - | - | ND | |||
| 102 | F | 11 | >5 | >5 |  |
ND | - | - | ND | |||
| 103 | M | 10 | 1 | 3 |  |
ND | - | - | ND | |||
| 104 | M | 10 | 2 | >5 |  |
ND | - | - | ND | |||
| 105 | F | 9 | 4 | >5 | - | - | ND | - | - | ND | ||
| 106 | F | 9 | 2 | >5 | - |  |
ND | - | - | ND | ||
| 107 | F | 8 | 3 | 4 | - | - | ND | - | - | ND | ||
| 108 | M | 8 | 2 | 4 | - | - | ND | - | - | ND | ||
| 109 | F | 8 | 3 | 2 | - | - | ND | - | - | ND | ||
| 110 | M | 12 | 0 | 2 | ND | - | ND | ND | - | ND | ||
| 111 | M | 11 | >5 | >5 |  |
ND | QH111R | QH111L | ND | |||
| 112 | M | 13 | 5 | 5 |  |
ND | - | - | ND | |||
| 113 | M | 10 | 5 | 5 |  |
ND | QH113R | - | ND | |||
| 114 | F | 11 | 5 | >5 | - | - | ND | - | - | ND | ||
| 118 | F | 7 | 3 | 4 |  |
+ | - | QH118L | QH118S | |||
| 119 | F | 10 | 5 | 3 |  |
+ | - | - | - | |||
| 120 | M | 8 | >5 | 5 |  |
+ | - | - | QH120S | |||
| 121 | M | 8 | 1 | 1 | - | - | + | - | - | - | ||
| 122 | F | 7 | 5 | 5 | - | - | + | - | - | - | ||
| 123 | M | 7 | 5 | 5 |  |
- | + | QH123R | - | QH123S1, -S2 | ||
| 124 | M | 9 | 1 | 3 | - |  |
+ | - | - | - | ||
| 125 | M | 8 | >5 | >5 | - | - | - | - | - | - | ||
| 126 | F | 8 | 4 | 4 |  |
- | + | - | - | - | ||
| 127 | F | 7 | 1 | 3 | - | - | + | - | - | - | ||
| 128 | M | 9 | 5 | >5 | - | - | - | - | - | - | ||
| 129 | F | 10 | 3 | 1 | - | - | - | - | - | - | ||
| 130 | M | 11 | 1 | 2 | - | - | - | - | - | - | ||
| 131 | F | 9 | 5 | >5 | - | - | - | - | - | - | ||
| 132 | F | 9 | >5 | 4 | - | - | - | - | - | - | ||
| 133 | M | 8 | >5 | >5 | - | - | - | - | - | - | ||
| 134 | M | 8 | 2 | >5 | - | - | - | - | - | - | ||
| 151 | M | 9 | 5 | 5 | - | - | - | - | - | - | ||
| 152 | M | 7 | 5 | 5 |  |
- | + | - | - | - | ||
| 153 | F | 12 | 5 | 3 | - | - | - | - | - | - | ||
| 154 | F | 12 | >5 | >5 | - | - | - | - | - | - | ||
| 155 | F | 11 | 2 | 3 |  |
+ | QH155R | QH155L | QH155S | |||
| 157 | F | 10 | 1 | 2 | - | - | - | - | - | - | ||
| 158 | F | 8 | 1 | 1 | - | - | - | - | - | - | ||
| 159 | M | 12 | 5 | 5 |  |
+ | QH159R | QH159L | QH159S | |||
| 160 | F | 7 | 2 | 2 |  |
+ | - | - | - | |||
| 161 | F | 8 | 5 | 5 |  |
+ | - | - | - | |||
| 162 | M | 9 | 5 | 5 | - |  |
+ | - | - | - | ||
| 163 | F | 6 | 2 | 3 | - |  |
+ | - | - | - | ||
| 164 | F | 9 | >5 | >5 | - |  |
+ | - | - | - | ||
| 165 | M | 10 | 1 | 2 | - |  |
- | - | - | - | ||
| Note: OD: Right eye; OS: Left eye; CS: Conjunctiva sac; ND: No data; Red, blue, yellow colors, three different ompA variants. | ||||||||||||
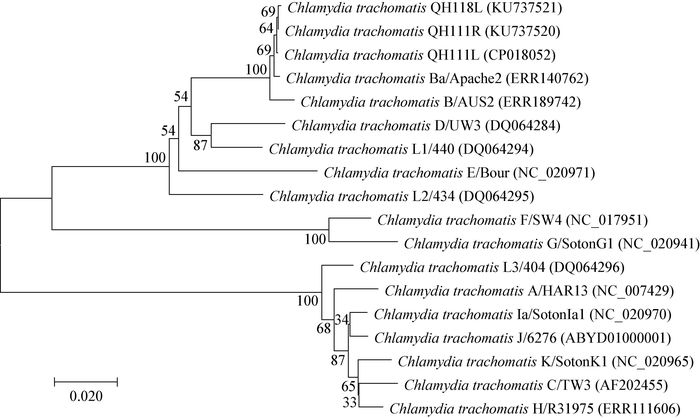
ompA是甄别眼型与UGT型沙眼衣原体的标志基因。对青海菌的ompA进行系统进化分析发现,3种ompA与已知的B和Ba型位于同一分支(图 2)。目前已知的B或Ba型ompA序列高度保守,不同B型分离株ompA基因序列之间只有有限的SNP差异。将3种ompA序列与已有的B和Ba型ompA进行比对分析,发现在760位碱基与B型相同。760位是B和Ba型的甄别位点,说明3种ompA均属B型,而不是Ba型。37份样本的ompA在可变区271位均为G,而已有B和Ba型ompA为A。将3种ompA与其它型沙眼衣原体ompA进行比较,发现271位的密码子GCT此前只存在于LGV、F和G等UGT型ompA中。
|
| 图 2 新分离青海沙眼衣原体与不同基因型沙眼衣原体ompA的系统进化关系 Figure 2 Evolutionary relationships of ompA genes of Chlamydia trachomatis strains from Qinghai and other ompA genovars 注:括号内的数字为GenBank序列号;节点处数字为Bootstrap值. Note: Numbers in parenthesis represented GenBank accession No.; Numbers at the branch points indicated the bootstrap values. |
|
|
随着生活卫生条件的不断提高,我国的沙眼发病率已大幅下降,“十人九沙”已成为历史。人是沙眼衣原体的唯一自然宿主,继天花、脊髓灰质炎之后,人类有望彻底消灭沙眼甚至沙眼衣原体,沙眼疫苗被认为是消灭沙眼最有效的工具。由于培养困难等原因,老一辈科学家在20世纪培养的沙眼病原体多数没有得到有效保存。本文首次报道从青海藏区分离培养15株眼型沙眼衣原体,为研究沙眼发病机制和研制沙眼疫苗奠定了基础。
当前的沙眼诊断依据是WHO制定的沙眼简化分级标准。衣原体分离培养是诊断沙眼的金标准方法,但操作难度大、灵敏度低。qPCR等分子诊断方法提高了检测的灵敏度,但在实际应用中也存在较高的假阴性率,这是因为病原体载量与炎症反应间的相关性差。在食蟹猴沙眼模型或人志愿者感染实验中,当病原体检测阴性时,滤泡等沙眼症状仍将持续数周至数月的时间[20-21]。在个别沙眼流行区,沙眼病原的检测阳性率很低,特别是那些经过大规模抗生素治疗的区域[22]。其它病原体如肺炎链球菌、流感嗜血杆菌等可能与持续的滤泡等炎症反应有关[23]。
依据传统的ompA基因分型方法,青海藏区的眼型沙眼衣原体均为基因B型。青海地区是否存在其它基因型有待调查,很可能还有C型菌流行。这是因为早期的流行病学调查均表明我国的北京、河南、河北、宁夏、台湾等不同地区均同时存在B型和C型,且均以B型为主[2, 9]。沙眼在亚洲广泛流行,流行的基因型是B型和C型,B型为优势型。最近Sharifi-Rad等报道了伊朗边境农村地区存在沙眼流行[24],但沙眼衣原体的型别不清楚。
本研究对理解沙眼衣原体的ompA微进化有特殊意义。首先,青海沙眼流行株ompA的UGT型密码子提示青海菌可能有独特的进化地位。另外,我们首次发现同一患者左右眼样本中的沙眼衣原体ompA有一个碱基差异。此前Li等报道了2种ompA突变体[12],在他们进行样本测序分析时,来自同一患者的样本111L和111R的ompA有一个SNP差异,由于该SNP仅存在于样本111L,被误认为是测序错误。沙眼衣原体分离株QH111L的ompA序列分析证实了第3种ompA突变体的存在。3种青海菌ompA突变体间的差异均位于可变区,这些变异可能与逃逸保护性体液免疫或细胞免疫有关。这与Nunes等报道的“MOMP进化趋势是在特定结构域产生点突变”相一致[25]。青海藏区相对封闭,该地区沙眼病原的分离培养为下一步全基因组序列分析等奠定了基础,将有助于了解我国沙眼的发生、演变和传播。
| [1] | Whitcher JP, Srinivasan M, Upadhyay MP. Corneal blindness: a global perspective[J]. Bulletin of the World Health Organization, 2001, 79(3) : 214–221. |
| [2] | Zhou YM, Wang ZQ, Sun XG. Detection and genotyping of Chlamydia trachomatis in the primary school children of two districts of north China[J]. Chinese Ophthalmic Research, 2007, 25(6) : 465–468. (in Chinese)周玉梅, 王智群, 孙旭光. 我国北方两地区小学生沙眼衣原体检测及基因分型研究[J]. 眼科研究, 2007, 25(6) : 465–468. |
| [3] | Dong YS, Liu XD. Trachoma survey in Tibetan boarding middle schools in Gannanzhou, Gansu province[J]. Gansu Medical Journal, 2013, 32(4) : 304–305. (in Chinese)董永生, 刘晓东. 甘南州寄宿制藏族中学生沙眼患病率调查[J]. 甘肃医药, 2013, 32(4) : 304–305. |
| [4] | Wang XH. Trachoma survey in primary school students of Yueyang city, Hunan province[J]. Practical Preventive Medicine, 2012, 19(2) : 210–211. (in Chinese)王霞辉. 岳阳市某小学学生沙眼患病及沙眼卫生常识知晓情况调查[J]. 实用预防医学, 2012, 19(2) : 210–211. |
| [5] | Zhao Q. Investigation and analysis of prevalence of trachoma in 1654 children[J]. Journal of Baotou Medical College, 2015, 31(2) : 19–20. (in Chinese)赵嫱. 1654例儿童沙眼患病情况调查分析[J]. 包头医学院学报, 2015, 31(2) : 19–20. |
| [6] | Yu CH, Gong CR, Shen H, et al. The situation survey analysis and thinking of trachoma of boarding senior middle schools students of Shiyan[J]. Chinese Primary Health Care, 2014, 28(3) : 87–88. (in Chinese)喻成华, 龚晨睿, 沈红, 等. 十堰市某寄宿制高级中学学生沙眼发生情况调查分析及思考[J]. 中国初级卫生保健, 2014, 28(3) : 87–88. |
| [7] | Tang FF, Zhang XL, Huang YT, et al. Studies on the etiology of trachoma Ⅳ. Attempt to isolate the virus in the embryonated hens eggs[J]. Acta Microbiologica Sinica, 1956, 4(2) : 189–210. (in Chinese)汤飞凡, 张晓楼, 黄元桐, 等. 沙眼病原研究Ⅳ.接种鸡胚, 分离病毒[J]. 微生物学报, 1956, 4(2) : 189–210. |
| [8] | Zhang XL. Research advances of trachoma[J]. Guowai Yixue Cankao Ziliao Yankexue Fence, 1977(1) : 1–6. (in Chinese)张晓楼. 沙眼研究进展[J]. 国外医学参考资料·眼科学分册, 1977(1) : 1–6. |
| [9] | Zhang L, Zhang XL, Jin XY. Chlamydia trachomatis immunotypes in trachoma epidemic areas of north China[J]. Chinese Journal of Ophthalmology, 1991, 27(2) : 67–70. (in Chinese)张力, 张晓楼, 金秀英. 我国华北沙眼流行区患者抗沙眼衣原体抗体的检测及其分型[J]. 中华眼科杂志, 1991, 27(2) : 67–70. |
| [10] | Stephens RS, Sanchez-Pescador R, Wagar EA, et al. Diversity of Chlamydia trachomatis major outer membrane protein genes[J]. Journal of Bacteriology, 1987, 169(9) : 3879–3885. DOI:10.1128/jb.169.9.3879-3885.1987 |
| [11] | Cook JA. Eliminating blinding trachoma[J]. New England Journal of Medicine, 2008, 358(17) : 1777–1779. DOI:10.1056/NEJMp0708546 |
| [12] | Li X, Zhang SY, Liang QF, et al. Molecular characteristics of the ompA gene of serotype B Chlamydia trachomatis in Qinghai Tibetan primary school students[J]. Science China Life Sciences, 2016, 59(6) : 561–570. DOI:10.1007/s11427-016-5059-9 |
| [13] | Caldwell HD, Kuo CC, Kenny GE. Antigenic analysis of Chlamydiae by two-dimensional immunoelectrophoresis. I. Antigenic heterogeneity between C. trachomatis and C. psittaci[J]. The Journal of Immunology, 1975, 115(4) : 963–968. |
| [14] | Carlson JH, Porcella SF, McClarty G, et al. Comparative genomic analysis of Chlamydia trachomatis oculotropic and genitotropic strains[J]. Infection and Immunity, 2005, 73(10) : 6407–6418. DOI:10.1128/IAI.73.10.6407-6418.2005 |
| [15] | Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7) : 1870–1874. DOI:10.1093/molbev/msw054 |
| [16] | Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987, 4(4) : 406–425. |
| [17] | Sanderson MJ, Wojciechowski MF. Improved bootstrap confidence limits in large-scale phylogenies, with an example from Neo-Astragalus (Leguminosae)[J]. Systematic Biology, 2000, 49(4) : 671–685. DOI:10.1080/106351500750049761 |
| [18] | Tamura K, Nei M, Kumar S. Prospects for inferring very large phylogenies by using the neighbor-joining method[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(30) : 11030–11035. DOI:10.1073/pnas.0404206101 |
| [19] | Li P, Duan Q, Song LH. Recent advances on chlamydial taxonomy and identification[J]. Chinese Journal of Zoonoses, 2014, 30(12) : 1262–1266. (in Chinese)李鹏, 端青, 宋立华. 衣原体最新分类体系与分类鉴定方法研究进展[J]. 中国人兽共患病学报, 2014, 30(12) : 1262–1266. DOI:10.3969/cjz.j.issn.1002-2694.2014.12.019 |
| [20] | Kari L, Whitmire WM, Carlson JH, et al. Pathogenic diversity among Chlamydia trachomatis ocular strains in nonhuman primates is affected by subtle genomic variations[J]. The Journal of Infectious Diseases, 2008, 197(3) : 449–456. DOI:10.1086/586912 |
| [21] | Ramadhani AM, Derrick T, Macleod D, et al. The relationship between active trachoma and ocular Chlamydia trachomatis infection before and after mass antibiotic treatment[J]. PLoS Neglected Tropical Diseases, 2016, 10(10) : e0005080. DOI:10.1371/journal.pntd.0005080 |
| [22] | Burr SE, Hart JD, Edwards T, et al. Association between ocular bacterial carriage and follicular trachoma following mass azithromycin distribution in the Gambia[J]. PLoS Neglected Tropical Diseases, 2013, 7(7) : e2347. DOI:10.1371/journal.pntd.0002347 |
| [23] | Butcher RMR, Sokana O, Jack K, et al. Low prevalence of conjunctival infection with Chlamydia trachomatis in a treatment-na ve trachoma-endemic region of the Solomon Islands[J]. PLoS Neglected Tropical Diseases, 2016, 10(9) : e0004863. DOI:10.1371/journal.pntd.0004863 |
| [24] | Sharifi-Rad J, Fallah F. Trachoma prevalence in rural areas of eastern Iran[J]. New Microbes and New Infections, 2016, 11 : 82–83. DOI:10.1016/j.nmni.2016.03.001 |
| [25] | Nunes A, Borrego MJ, Nunes B, et al. Evolutionary dynamics of ompA, the gene encoding the Chlamydia trachomatis key antigen[J]. Journal of Bacteriology, 2009, 191(23) : 7182–7192. DOI:10.1128/JB.00895-09 |
 2017, Vol. 44
2017, Vol. 44




