扩展功能
文章信息
- 李静, 张金羽, 张琪, 李德生, 黄治, 李倜, 赵珂
- LI Jing, ZHANG Jin-Yu, ZHANG Qi, LI De-Sheng, HUANG Zhi, LI Ti, ZHAO Ke
- 大熊猫肠道放线菌的种群组成及多样性分析
- Diversity and community structure of gut actinobacteria of giant panda
- 微生物学通报, 2017, 44(5): 1138-1148
- Microbiology China, 2017, 44(5): 1138-1148
- DOI: 10.13344/j.microbiol.china.160477
-
文章历史
- 收稿日期: 2016-06-25
- 接受日期: 2016-09-14
- 优先数字出版日期(www.cnki.net): 2016-09-28
2. 中国大熊猫研究保护中心 四川 都江堰 623006
2. China Conservation and Research Center of Giant Panda, Dujiangyan, Sichuan 623006, China
放线菌种类繁多、代谢功能各异,是分泌天然药物最多的一类微生物。目前临床和农业生产使用的抗生素有3/4是用放线菌生产的[1]。然而动物肠道系统作为一个典型的、独特的环境,放线菌是大多数哺乳动物肠道中的优势菌群[2]。动物肠道放线菌在与宿主长期的协同进化过程中,与宿主进行着物质、能量及基因的互动作用,构成了宿主小生境特定的微生态系统[3]。随着对肠道微生物研究的深入,发现肠道内放线菌类群丰富,所产生的代谢产物如抗生素、酶抑制剂、免疫调节剂等在动物体内起着至关重要的作用,具有促进机体的营养吸收和抵御病害等多种生物学功能,一些肠道放线菌可以产生具有抗菌、抗肿瘤等不同功能的新结构化合物[4-6],这为寻找新的化合物资源提供了新的来源[7]。
大熊猫是我国特有的珍稀濒危动物,也是世界野生动物保护的旗舰动物[8],其数量十分稀少,主要栖息在我国四川、陕西、甘肃三省交界的山区,属于哺乳动物纲,食肉动物目,主要以竹子为食。较之其它草食动物,大熊猫具有简单的胃、退化的和短直的结肠等,这符合典型肉食性哺乳动物的消化系统,并具有类似于肉食性动物的肠道微生物种群结构[9]。目前关于大熊猫肠道微生物菌群的研究报道较多[7, 10-12],但大熊猫肠道放线菌种群组成及多样性研究报道却非常少见,且研究方法主要采用纯培养法。由于分离方法、营养成分、培养条件等因素的影响,动物肠道中有2/3的微生物目前是不能被培养的[13],因此纯培养方法难以全面了解大熊猫肠道放线菌的多样性与种群结构。
基于16S rRNA基因建立的多种生物学方法已成为现代微生物生态学研究的重要手段,并在研究动物肠道微生物种群结构上得到了广泛的应用[9, 14-16]。因此,本研究采用变性梯度凝胶电泳(DGGE)技术结合DNA测序技术对大熊猫肠道内放线菌的多样性及群落结构进行分析,探究不同年龄、不同性别大熊猫肠道放线菌多样性的差异,深入了解大熊猫肠道内的微生物资源,为筛选潜在产生活性化合物的放线菌资源提供理论依据。
1 材料与方法 1.1 实验材料2015年5月从中国大熊猫保护研究中心都江堰基地采集7只大熊猫的新鲜粪便,放入无菌塑料袋中,冰盒保存带回实验室立即提取总DNA。7只大熊猫均生活在相同的环境中,以竹子为主食,苹果、胡萝卜等为辅食,健康状况良好,采集样品的具体信息如表 1所示。
| 熊猫名字 Name | 性别 Gender | 出生日期(年份) Date of birth (Year) | 年龄 Age |
| 园欣Yuanxin (F1) | 雌性 | 2012 | 3岁(亚成体) |
| 苏珊Susan (M1) | 雄性 | 2011 | 4岁(亚成体) |
| 美茜Messi (F2) | 雌性 | 2006 | 9岁(成年) |
| 泰山Mount Tai (M2) | 雄性 | 2005 | 10岁(成年) |
| 紫霞Zixia (F3) | 雌性 | 1995 | 20岁(老年) |
| 白雪White snow (F4) | 雌性 | 1990 | 25岁(老年) |
| 强强Qiang (M3) | 雄性 | 1987 | 28岁(老年) |
土壤FastDNA® SPIN试剂盒,美国MPBIO公司;PCR引物、凝胶回收试剂盒,上海生工生物科技有限公司;PCR Mix,天根生化科技有限公司;pMD19-T,大连宝生物工程有限公司。5804R冷冻高速离心机,德国Eppendorf公司;PCR仪、凝胶成像仪、电泳仪、DCode DGGE System,美国Bio-Rad公司。
1.3 样品总DNA提取和放线菌PCR扩增对采集的每个样品随机挑取3份作为重复,采用土壤FastDNA® SPIN试剂盒提取总DNA,于-20保存备用。采用巢氏PCR对目标片段进行扩增。第一轮PCR扩增引物为F243 (5′-GGATGAG CCCGCGGCCTA-3′)和R1186 (5′-CTTCCTCCGAG TTGACCC-3′)[17],反应体系为:Mix缓冲液10 μL,引物(10 μmol/L)各0.2 μL,模板DNA 1 μL,ddH2O补足20 μL。第二轮所用的引物为F907GC (5′-CGC CCGGGGCGCGCCCCGGGCGGGGCGGGGGCACGGGGGGAAACTCAAAGGAATTGACGG-3′)和R1186 (5′-CTTCCTCCGAGTTGACCC-3′)[17],反应体系为:Mix缓冲液25 μL,引物(10 μmol/L)各0.5 μL,第1轮PCR产物1 μL,ddH2O补足50 μL。为减少非特异性产物,采用降落PCR (Touch-down)程序扩增,参照李小林等[18]的方法略有改进,第一、二轮反应程序均为:95;94,65-55每个循环温度降低0.5,72,20个循环;94,55,72 45 s,25个循环;72;产物于4恒温保存。最后将PCR产物用1.0%琼脂糖凝胶电泳检测片段大小,约为270 bp。
1.4 DGGE凝胶电泳分析大熊猫肠道放线菌的多样性DGGE凝胶电泳在DCode系统上进行。取第二轮扩增的PCR产物20 μL进行DGGE电泳分析。变性剂梯度浓度范围为40%-65%,其变性方向与电泳方向一致。在1×TAE缓冲液中,50 V预进样30 min,然后180 V、60电泳7 h。电泳完毕后将剥离的凝胶使用银染法进行染色[19],之后在凝胶成像系统下成像并拍照。
1.4.1 DGGE图谱及遗传多样性分析: DGGE指纹图谱用Bio-Rad公司的Quantity One分析软件进行分析,通过检测条带,匹配条带,扣除背景强度,对目的条带进行标记,利用DGGE图谱的数字化结果计算大熊猫肠道放线菌群落的多样性指数(H')、丰度(S)和均匀度(EH),以此来评价大熊猫肠道放线菌群落结构多样性的变化。多样性指数和均匀度计算公式如下:
H'=-ΣPi·lnPi;
EH=H′/lnS
公式中,Pi是某个动物样品中单一条带的强度在该样品中所有条带强度的比率,S是某个动物样品中所有条带数目的总和[20]。
1.4.2 肠道放线菌群落相似性分析: 使用Quantity One分析软件计算群落相似系数(Sc)并导出放线菌群落结构UPGMA聚类分析图,分析不同大熊猫个体肠道放线菌组成的相似性及亲缘关系。 1.4.3 DGGE条带测序和系统发育分析: 将DGGE条带中的亮度较大的优势条带和不同位置出现的特异性条带切割下后,用无菌去离子水清洗3次,并用20 μL去离子水浸泡过夜,再以浸液为模板进行PCR扩增。用第二次PCR扩增时用的引物(无GC发夹),相同的体系和程序进行PCR扩增。PCR产物用UNIQ-10 DNA纯化试剂盒进行纯化,之后用大连宝生物公司的pMD19-T Vector试剂盒进行克隆。挑选白色菌落进一步测序,测序由上海生工生物工程公司完成。将所测的序列,通过BLAST程序在GenBank获取相似度较高的序列,ClustalX (version 1.81) 进行全序列比对[21],采用Neighbor-Joining方法在Mega 5.0中进行系统发育分析[22]。将测序得到的序列提交GenBank数据库,登录号为KX431053-KX431079,KX579056。 1.4.4 主成分分析: 根据Quantity One导出的相对亮度值计算得出DGGE各条带的相对百分亮度值,并使用Canoco 5.0软件[23]进行主成分分析,了解大熊猫肠道放线菌的群落结构。 1.5 数据分析采用SPSS 13.0统计软件对数据进行单因素方差分析、标准误差和相关性分析,采用Excel 2013软件作图。
2 结果与分析 2.1 大熊猫肠道放线菌DGGE图谱及多样性分析微生物群落的遗传多样性在一定程度上可以通过DGGE条带的多少反映出来[24]。大熊猫肠道放线菌DGGE图谱显示(图 1),各样品经DGGE后分离出数目不等的条带,而且不同样品各条泳道的强度和迁移率不相同,同一样品的3个重复的泳道强度和迁移率相似度很高。对大熊猫肠道放线菌DGGE条带进行比较可以发现F1和M3的条带较为复杂,M1和F4的条带较为单一,表明不同个体大熊猫肠道放线菌的分布存在差异。
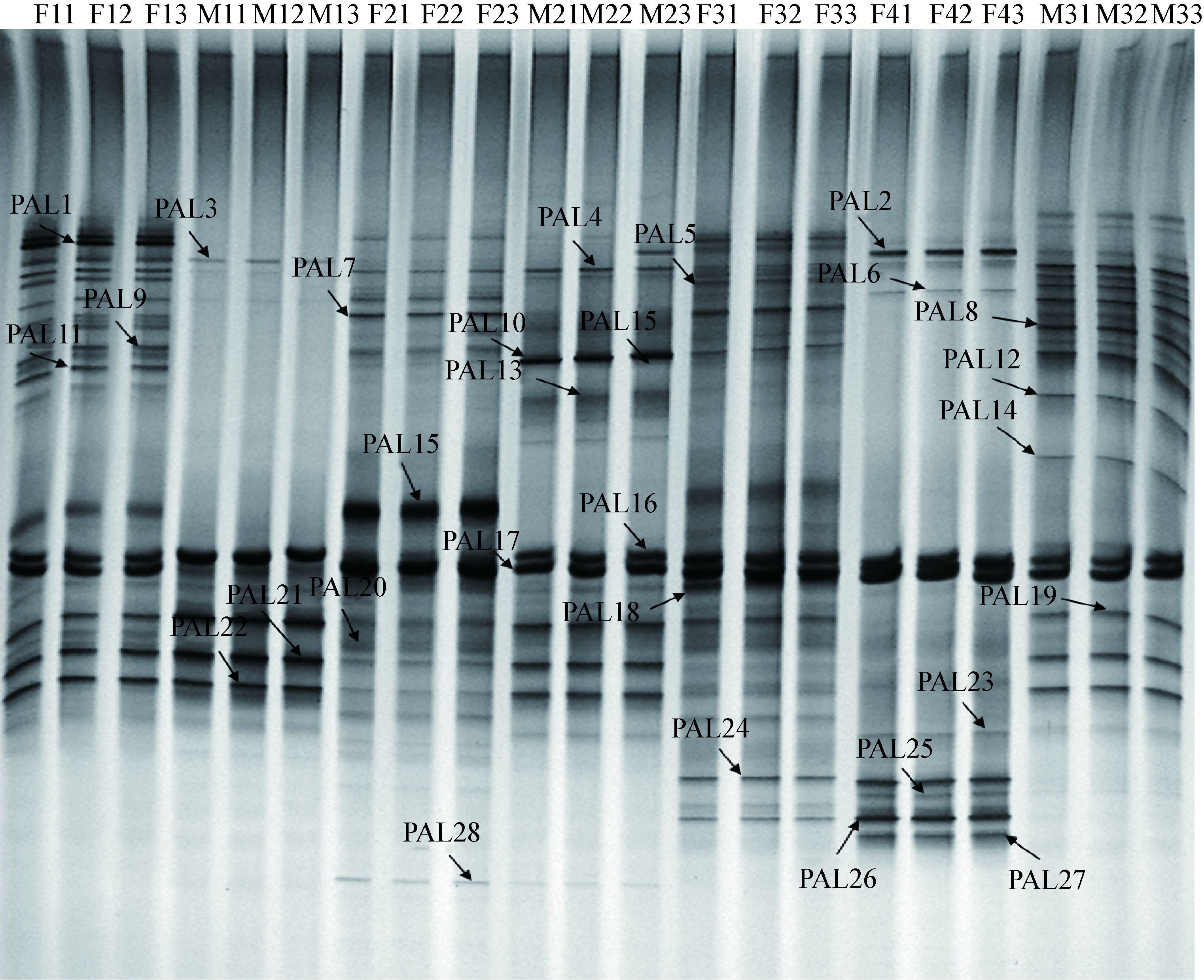
|
| 图 1 大熊猫肠道放线菌DGGE图谱 Figure 1 DGGE profile of the enteric canal actinobacteria of giant pandas 注:F11-F13:园欣粪便样品;M11-M13:苏珊粪便样品;F21-F23:美茜粪便样品;M21-M23:泰山粪便样品;F31-F33:紫霞粪便样品;F41-F43:白雪粪便样品;M31-M33:强强粪便样品;PAL1-PAL28:克隆条带. Note: F11-F13: faeces of Yuanxin; M11-M13: faeces of Susan; F21-F23: faeces of Messi; M21-M23: faeces of Mount Tai; F31-F33: faeces of Zixia; F41-F43: faeces of White snow; M31-M33: faeces of Qiang; PAL1-PAL28: Excised bands. |
|
|
依据电泳图谱中条带的数量与强度,转化成多样性指数(H')、丰富度(S)和均匀度(EH),并采用单因素方差分析法比较不同大熊猫肠道放线菌的遗传多样性。结果如图 2所示,大熊猫个体间肠道放线菌的各个指标都存在着不同程度的差异。然而多样性指数与丰度显示的结果一致,F1肠道内放线菌的多样性指数(H')与丰度(S)皆为最高,达到了3.20和24,M1肠道内的多样性指数(H')与丰度(S)最低,仅为2.42和11 (图 2A、B)。而均匀度(EH)最高和最低的分别是F4和M1,为0.999 5和0.998 3 (图 2C)。综合各项指标来看,大熊猫肠道放线菌的分布呈现出性别差异,雌性大熊猫肠道内放线菌的多样性为F1 > F3 > F2 > F4,雄性大熊猫肠道内放线菌的多样性为M3 > M2 > M1。对不同大熊猫肠道内放线菌多样性的比较结果表明,年龄小的雌性大熊猫肠道放线菌多样性明显高于年龄大的,而雄性大熊猫年龄大的肠道放线菌的多样性明显高于年龄小的,这表明在大熊猫这一群体中,肠道放线菌的多样性和群落结构受年龄与性别的双重影响。

|
| 图 2 大熊猫粪便样品中DGGE条带多样性指数(A)、丰度(B)和均匀度(C) Figure 2 Shannon-weaver index (H') (A), Richness (S) (B), and Evenness (EH) (C) for the test faeces samples of giant pandas |
|
|
不同大熊猫粪便样品的电泳图谱中条带数目的差异反映各样品之间的相似性,通过计算群落相似系数(Sc)可以得到不同大熊猫个体间肠道放线菌组成的相似性及亲缘关系。结果如图 3所示,当Sc值小于0.48,大熊猫M1、M3、M2肠道放线菌相似度较高,聚为一支,且均为雄性;大熊猫F2、F3、F1肠道内放线菌相似度较高,聚为一支,且均为雌性;而大熊猫F4肠道放线菌与其它6只大熊猫相似度均不是很高,单独聚为一支。结果表明,性别是引起大熊猫肠道放线菌群落结构差异的主要因素。此外,性别相同的大熊猫由于年龄的差距,也会造成肠道放线菌群落结构的不同。
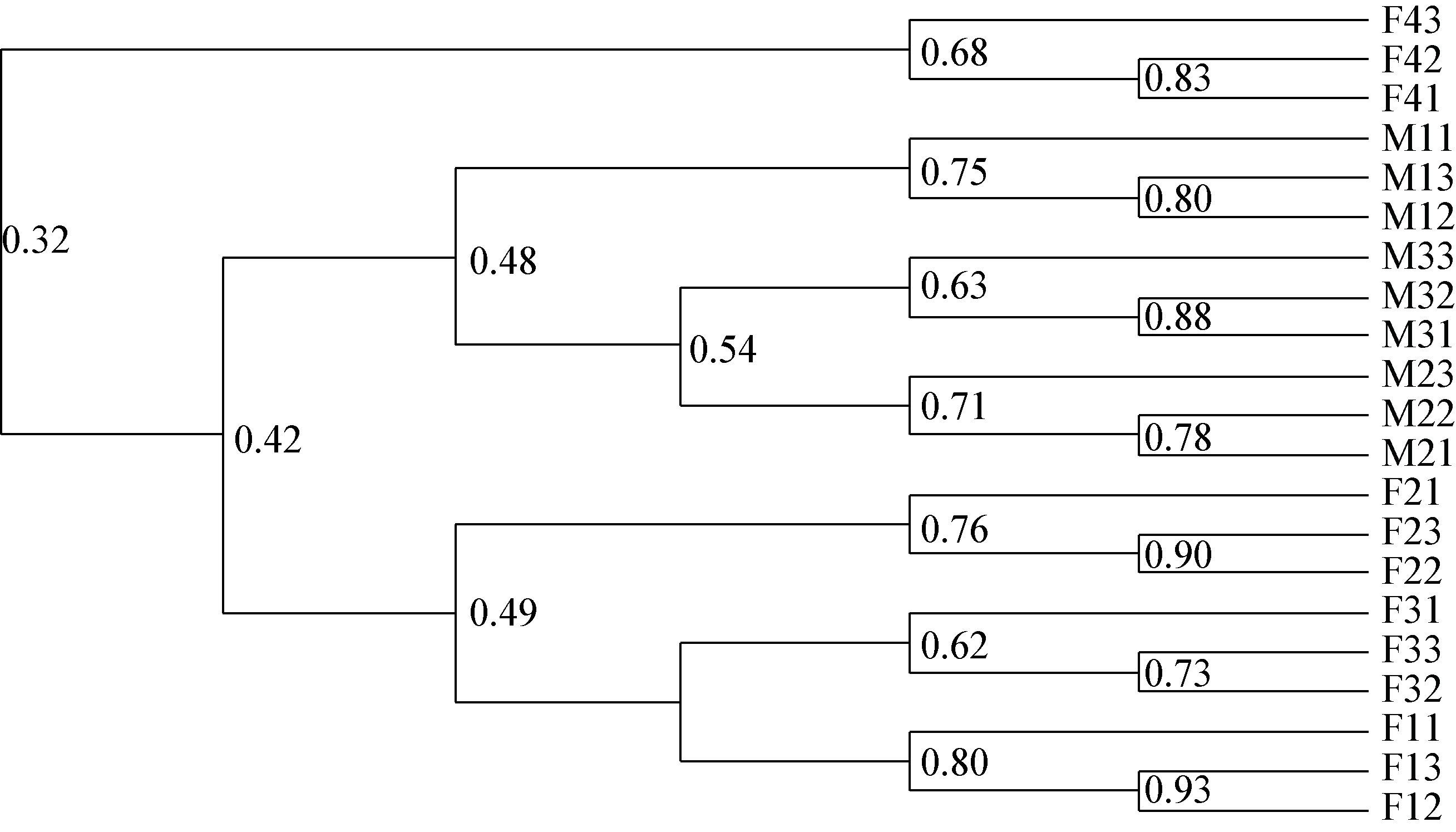
|
| 图 3 大熊猫肠道放线菌DGGE指纹图谱聚类分析图 Figure 3 Cluster analysis of DGGE profiles for the enteric canal actinobacteria of pandas 注:F11-F13:园欣粪便样品;M11-M13:苏珊粪便样品;F21-F23:美茜粪便样品;M21-M23:泰山粪便样品;F31-F33:紫霞粪便样品;F41-F43:白雪粪便样品;M31-M33:强强粪便样品. Note: F11-F13: faeces of Yuanxin; M11-M13: faeces of Susan; F21-F23: faeces of Messi; M21-M23: faeces of Mount Tai; F31-F33: faeces of Zixia; F41-F43: faeces of White snow; M31-M33: faeces of Qiang. |
|
|
运用Canoco软件对不同大熊猫肠道放线菌群落进行主成分分析,提取2个主成分因子作载荷图,结果如图 4所示。由图 4可以看出,成分2和性别变量存在很强的相关性,贡献率为20.49%,在成分2的基础上分析,可以看出成分1和年龄变量存在一定的相关性,解释了27.27%的物种组成变化。综合分析主成分1和2可以发现,雌性大熊猫肠道内的放线菌分群全部聚集在成分2的正轴上,雄性全部聚集在成分2的负轴上,而年龄最小的雄性大熊猫M1和年龄最大的雌性大熊猫F4分别与相同性别的其他样品较分散。主成分分析结果表明,不同年龄大熊猫肠道放线菌群落结构差异较大,但同一性别大熊猫肠道群落结构组成具有一定的相似性。

|
| 图 4 大熊猫肠道放线菌DGGE图谱的主成分(PCA)分析 Figure 4 Analysis of principal components of Actinobacteria communities in the gut of pandas 注:F11-F13:园欣粪便样品;M11-M13:苏珊粪便样品;F21-F23:美茜粪便样品;M21-M23:泰山粪便样品;F31-F33:紫霞粪便样品;F41-F43:白雪粪便样品;M31-M33:强强粪便样品. Note: F11-F13: faeces of Yuanxin; M11-M13: faeces of Susan; F21-F23: faeces of Messi; M21-M23: faeces of Mount Tai; F31-F33: faeces of Zixia; F41-F43: faeces of White snow; M31-M33: faeces of Qiang. |
|
|
DGGE电泳后,对28条主要特征条带进行切胶回收克隆测序后(图 1),将结果与Genbank数据库中的序列进行Blast比对。序列同源性结果显示,所有回收条带的序列与数据库中的最似模式菌株一致性为96%-100%。结果如表 2所示,红球菌属(Rhodococcus)和链霉菌属(Streptomyces)在所有供试大熊猫肠道中均有分布,其他放线菌属在不同大熊猫肠道中的分布不同。芽生球菌属(Blastococcus)在F1、F3、F4和M3的粪便中均有检出;布登堡菌属(Beutenbergia)在F2和M2的粪便中检出,北里孢菌属(Kitasatospora)和棒杆菌属(Corynebacterium)分别在F1和M2的粪便中检出;链嗜酸菌属(Streptacidiphilus)在F1、F2、F3、M2和M3的粪便中均有检出。上述结果表明,不同大熊猫肠道内的放线菌群落结构存在差异。
 |
28条序列被分别归属于5个亚目,分别为链霉菌亚目(Streptomycineae)、棒杆菌亚目(Corynebacterineae)、丙酸杆菌亚目(Propinibacterineae)、微球菌亚目(Micrococcineae)和弗兰克氏菌亚目(Frankinese)[25](表 2);6个科分别为链霉菌科(Strerptomycetaceae)、棒杆菌科(Corynebacteriaceae)、类诺卡氏菌科(Nocardioidaceae)、布登堡菌科(Beutenbergiaceae)、微杆菌科(Microbacterium)和地嗜皮菌科(Geodermatophilaceae);10个属分别为链霉菌属(Streptomyces)、北里孢菌属(Kitasatospora)、红球菌属(Rhodococcus)、棒杆菌属(Corynebacterium)、迪茨氏菌属(Dietziaceae)、大理石雕菌属(Marmoricola)、布登堡菌属(Beutenbergia)、微杆菌属(Microbacterium)、链嗜酸菌属(Streptacidiphilus)和芽生球菌属(Blastococcus) (图 5)。其中分属于链霉菌属的序列有13个,占总数的46%,为优势菌属;非链霉菌属有15个,占总数的54%,说明大熊猫肠道放线菌的群落结构具有丰富的多样性。

|
| 图 5 大熊猫肠道放线菌DGGE条带N-J系统发育分析 Figure 5 Phylogenetic analysis of 16S rRNA gene sequences of the gut actinobacteria of giant pandas from DGGE bands Note: The numbers at the nodes indicate the levels of bootstrap support (%) based on 1 000 resampled data sets; Only values above 50% are given; The scale bar corresponds to 0.02 substitutions per nucleotide position; Numbers in parentheses indicate accession numbers in Genbank; Bacillus badius ATCC 14574 was used as an out group. |
|
|
近年来,对于动物肠道放线菌的研究逐渐增多,主要集中在海洋动物[26-28]、肉食动物[29]和反刍动物[30-31]等的研究基础方面,有关大熊猫肠道放线菌多样性的研究目前关注还较少,仅见于曹艳茹[6]、姜怡等[32]采用可培养方法对大熊猫肠道放线菌的研究。他们从大熊猫肠道中分离出了链霉菌属(Streptomyces)、红球菌属(Rhodococcus)、节杆菌属(Arthrobacter)等11个属的放线菌,其中链霉菌属(Streptomyces)和红球菌属(Rhodococcus)为优势菌属。本研究首次运用DGGE方法对不同年龄、不同性别的大熊猫肠道放线菌的种群结构进行分析,结果发现,大熊猫粪便中分布最为广泛的放线菌也是链霉菌属(Streptomyces)和红球菌属(Rhodococcus),这与前人研究结果相一致,说明这两个属的放线菌在大熊猫肠道中稳定存在。曹艳茹[6]从大熊猫肠道内分离获得的具有纤维素降解潜力的纤维微菌属(Cellulosimicrobium)和纤维单孢菌属(Cellulomonas)菌株的序列未能在本次实验中检出,主要原因可能是分析手段的不同和实验材料的差异引起的。本研究所检测出的芽生球菌属(Blastococcus)分属于地嗜皮菌科(Geodermatophilaceae),这一类菌多存在于岩石表面、海洋基质、草地土壤等环境中[33-36],目前在肠道微生物的研究中尚未发现,而在粪便中检出可能是其附着于竹子等食物上随大熊猫进食进入肠道,后随粪便排出,这一类菌属于非常住肠道菌群,被称为过路菌[37]。综上,在环境微生物的检测中,往往需将可培养和免培养技术相结合,以获得更详尽的科学数据。
Tun等[38]采用高通量测序技术对成年和老年大熊猫肠道微生物种群结构研究结果发现,不同年龄大熊猫肠道微生物的种群结构差异明显,但仅在成年大熊猫肠道中检测到了分属于放线菌属(Actinomyces)、微杆菌属(Microbacterium)、气单孢菌属(Aeromonas)三个属的放线菌,而在老年的大熊猫肠道中均未检测到放线菌。然而本研究DGGE图谱分析显示,大熊猫肠道中分布着种群结构多样的放线菌,且其放线菌群落组成与年龄及性别具有较强的相关性,年龄较大的雄性大熊猫样品分群聚集明显,年龄较小的雌性大熊猫样品分群聚集则较为明显,而随着年龄的增长,雌性大熊猫肠道内放线菌的多样性变少,雄性大熊猫肠道内放线菌的多样性却更加丰富,表明年龄与性别的差异是影响大熊猫肠道菌群结构与组成的重要因素。有关其它动物以及人肠道微生物种群结构的研究结果也进一步证明了年龄与性别的差异对肠道微生物的影响,简平等[39]在川金丝猴肠道菌群的研究中发现川金丝猴肠道菌群生物多样性伴随年龄增长不断变化,呈现先增加后减少的趋势;周振金等[40]对不同性别猪粪样菌群结构及粪臭素产生相关菌群的研究发现猪粪样中的菌群组成存在一定的性别差异;徐洁[41]对不同年龄健康人肠道菌群的比较研究表明,处于不同生理年龄时期的健康人肠道菌群存在差异;Mueller等[42]对来自欧洲不同地区、不同年龄及性别的健康人肠道菌群比较研究表明,地域、年龄、性别等因素对健康人肠道菌群结构均有影响。此外,大熊猫肠道微生物群落结构的多样性与其是否圈养、饮食结构等均具有较大的关系[9, 43]。因此,各个学者所研究的大熊猫的生长环境、饮食结构的差异、采样季节性的不同,都会引起大熊猫肠道放线菌多样性的差异,进而导致研究结果的差异。本研究和前人的研究互为补充,既检测出部分已被分离或检出的放线菌,又检测出此前未被分离或检出的放线菌,进一步丰富了大熊猫肠道放线菌的资源,为大熊猫肠道放线菌种群结构的研究提供了一定的参考依据。本实验的样品数量有限,性别和年龄的代表性具有一定的局限,但所研究的大熊猫的生活环境、饮食结构和喂养方式是一致的,该结果仍具有一定的参考价值。
与土壤、海洋、极端条件、植物等环境相比,动物肠道是一个独特的环境,其中存在着大量未知的放线菌,是开发新型抗生素、抗肿瘤药物、酶抑制剂等的重要资源[44],而且从动物粪便放线菌获得的天然产物,对人畜具有更高的安全性[32]。因此,研究大熊猫肠道内的放线菌多样性,有助于了解不同年龄、不同性别的大熊猫肠道放线菌的遗传多样性及系统发育情况,丰富了对大熊猫肠道放线菌资源的了解,为进一步开发和挖掘新的放线菌资源提供了理论依据。
致谢: 感谢中国大熊猫研究保护中心在本研究工作上提供的帮助,特此表示诚挚的谢意!| [1] | Jiang Y, Cao YR, Han L, et al. Diversity and bioactivity of culturable actinobacteria from animal feces[J]. Acta Microbiologica Sinica, 2012, 52(10) : 1282–1289. (in Chinese) 姜怡, 曹艳茹, 韩力, 等. 五种动物粪便纯培养放线菌的多样性及生物活性[J]. 微生物学报, 2012, 52(10) : 1282–1289. |
| [2] | Ley RE, Hamady M, Lozupone C, et al. Evolution of mammals and their gut microbes[J]. Science, 2008, 320(5883) : 1647–1651. DOI:10.1126/science.1155725 |
| [3] | Jin HZ, Li KB. Progress in the study of human intestinal microecosystem[J]. Nature Magazine, 2004, 26(2) : 88–91. (in Chinese) 金红芝, 李堃宝. 人肠道微生态系统的研究进展[J]. 自然杂志, 2004, 26(2) : 88–91. |
| [4] | Gandhimathi R, Arunkumar M, Selvin J, et al. Antimicrobial potential of sponge associated marine actinomycetes[J]. Journal de Mycologie Médicale/Journal of Medical Mycology, 2008, 18(1) : 16–22. DOI:10.1016/j.mycmed.2007.11.001 |
| [5] | Matsui T, Tanaka J, Namihira T, et al. Antibiotics production by an actinomycete isolated from the termite gut[J]. Journal of Basic Microbiology, 2012, 52(6) : 731–735. DOI:10.1002/jobm.v52.6 |
| [6] | Cao YR. Study on the diversity and biological activity of fecal actinobacteria[D]. Yangling: Doctoral Dissertation of Northwest Agriculture and Forestry University, 2012 (in Chinese) 曹艳茹. 动物粪便放线菌多样性及生物活性研究[D]. 杨凌: 西北农林科技大学博士学位论文, 2012 |
| [7] | Sun YY, Jia XH. Prospect of research on animal intestinal actinomycetes[J]. Tianjin Agricultural Sciences, 2014, 20(7) : 25–28. (in Chinese) 孙玉宇, 贾晓慧. 动物肠道放线菌的研究展望[J]. 天津农业科学, 2014, 20(7) : 25–28. |
| [8] | Zhan XJ, Li M, Zhang ZJ, et al. Molecular censusing doubles giant panda population estimate in a key nature reserve[J]. Current Biology, 2006, 16(12) : R451–R452. DOI:10.1016/j.cub.2006.05.042 |
| [9] | Xue ZS, Zhang WP, Wang LH, et al. The bamboo-eating giant panda harbors a carnivore-like gut microbiota, with excessive seasonal variations[J]. MBio, 2015, 6(3) : e00022–15. |
| [10] | Bao N, Liu CJ, Tan Z, et al. Changes of intestinal microflora in the giant panda returned to wild[J]. Animal Husbandry & Veterinary Medicine, 2005, 37(8) : 10–13. (in Chinese) 鲍楠, 刘成君, 谭志, 等. 野外放归大熊猫肠道菌群变化的研究[J]. 畜牧与兽医, 2005, 37(8) : 10–13. |
| [11] | Zhu LF, Wu Q, Dai JY, et al. Evidence of cellulose metabolism by the giant panda gut microbiome[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(43) : 17714–17719. DOI:10.1073/pnas.1017956108 |
| [12] | Zhou JL, Li FP, Liu L, et al. Identification and analysis of the culturable bacterium in the feces of captive giant pandas in high-altitude: Taking yunnan wildlife park in kunming in spring for example[J]. Sichuan Journal of Zoology, 2015, 34(6) : 852–858. (in Chinese) 周杰珑, 李非平, 刘丽, 等. 高海拔地区圈养大熊猫粪便可培养细菌分析——以云南野生动物园春季为例[J]. 四川动物, 2015, 34(6) : 852–858. |
| [13] | Suau A, Bonnet R, Sutren M, et al. Direct analysis of genes encoding 16S rRNA from complex communities reveals many novel molecular species within the human gut[J]. Applied & Environmental Microbiology, 1999, 65(11) : 4799–4807. |
| [14] | Gao L, Xiong HS, Peng L, et al. Application of some culture-Independent detection approaches based on 16S rRNA in the research of gastrointestinal tract microbiota[J]. Chinese Journal of Animal Science, 2008, 44(15) : 56–60. (in Chinese) 高岚, 熊浩山, 彭莉, 等. 以16S rRNA为基础的检测技术在肠道菌群研究中的应用[J]. 中国畜牧杂志, 2008, 44(15) : 56–60. |
| [15] | Wang XY, Yuan T, Liao H, et al. Construction of the 16S rDNA clone library of intestinal flora of the captive elderly giant panda[J]. China Animal Husbandry & Veterinary Medicine, 2015, 42(6) : 1402–1408. (in Chinese) 王晓艳, 袁听, 廖虹, 等. 圈养老年大熊猫肠道菌群16S rDNA克隆文库的建立[J]. 中国畜牧兽医, 2015, 42(6) : 1402–1408. |
| [16] | Wei FW, Wang X, Wu Q. The giant panda gut microbiome[J]. Trends in Microbiology, 2015, 23(8) : 450–452. DOI:10.1016/j.tim.2015.06.004 |
| [17] | Sch fer J, J ckel U, K mpfer P. Development of a new PCR primer system for selective amplification of Actinobacteria[J]. FEMS Microbiology Letters, 2010, 311(2) : 103–112. DOI:10.1111/fml.2010.311.issue-2 |
| [18] | Li XL, Gu YF, Zhang XP, et al. Nitrobacteria community and diversity in the rhizosphere soil of mature tobacco[J]. Scientia Agricultura Sinica, 2011, 44(12) : 2462–2468. (in Chinese) 李小林, 辜运富, 张小平, 等. 烟草成熟期根际硝化细菌种群的结构及其多样性[J]. 中国农业科学, 2011, 44(12) : 2462–2468. DOI:10.3864/j.issn.0578-1752.2011.12.007 |
| [19] | Sanguinetti CJ, Dias Neto E, Simpson A. Rapid silver staining and recovery of PCR products separated on polyacrylamide gels[J]. Biotechniques, 1994, 17(5) : 914–921. |
| [20] | Eichner CA, Erb RW, Timmis KN, et al. Thermal gradient gel electrophoresis analysis of bioprotection from pollutant shocks in the activated sludge microbial community[J]. Applied & Environmental Microbiology, 1999, 65(1) : 102–109. |
| [21] | Larkin MA, Blackshields G, Brown NP, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21) : 2947–2948. DOI:10.1093/bioinformatics/btm404 |
| [22] | Tamura K, Peterson D, Peterson N, et al. MEGA 5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10) : 2731–2739. DOI:10.1093/molbev/msr121 |
| [23] | ŠmilauerP, LepšJ. Multivariate Analysis of Ecological Data Using CANOCO 5[M]. Cambridge: Cambridge University Press, 2003 . |
| [24] | Gu YF, Zhang XP, Tu SH, et al. Soil microbial biomass, crop yields, and bacterial community structure as affected by long-term fertilizer treatments under wheat-rice cropping[J]. European Journal of Soil Biology, 2009, 45(3) : 239–246. DOI:10.1016/j.ejsobi.2009.02.005 |
| [25] | Ruan JS. Bergey's Manual of Systematic Bacteriology (Second Edition) Volume 5 and the study of Actinomycetes systematic in China[J]. Acta Microbiologica Sinica, 2013, 53(6) : 521–530. (in Chinese) 阮继生. "伯杰氏系统细菌学手册(第二版)"第5卷与我国的放线菌系统学研究[J]. 微生物学报, 2013, 53(6) : 521–530. |
| [26] | Kim TK, Garson MJ, Fuerst JA. Marine actinomycetes related to the 'Salinospora' group from the Great Barrier Reef sponge Pseudoceratina clavata[J]. Environmental Microbiology, 2005, 7(4) : 509–518. DOI:10.1111/emi.2005.7.issue-4 |
| [27] | Ward AC, Bora N. Diversity and biogeography of marine actinobacteria[J]. Current Opinion in Microbiology, 2006, 9(3) : 279–286. DOI:10.1016/j.mib.2006.04.004 |
| [28] | Zhang HT, Lee YK, Zhang W, et al. Culturable actinobacteria from the marine sponge Hymeniacidon perleve: isolation and phylogenetic diversity by 16S rRNA gene-RFLP analysis[J]. Antonie van Leeuwenhoek, 2006, 90(2) : 159–169. DOI:10.1007/s10482-006-9070-1 |
| [29] | Zhao HX. Study on the diversity and function of intestinal bacteria flora from different wild animals[D]. Changchun: Master's Thesis of Jilin Agricultural University, 2015 (in Chinese) 赵晗旭. 不同野生动物肠道微生物多样性分析及功能初步研究[D]. 长春: 吉林农业大学硕士学位论文, 2015 |
| [30] | Zeng HY. Diversity of symbiotic anctinobacteria from healthy ruminant faeces[D]. Guangzhou: Master's Thesis of Sun Yat-Sen University, 2012 (in Chinese) 曾华颖. 反刍动物肠道共生放线菌类群多样性分析[D]. 广州: 中山大学硕士学位论文, 2012 |
| [31] | Tan HM, Deng QL, Cao LX. Ruminant feces harbor diverse uncultured symbiotic actinobacteria[J]. World Journal of Microbiology and Biotechnology, 2014, 30(3) : 1093–1100. DOI:10.1007/s11274-013-1529-4 |
| [32] | Jiang Y, Cao YR, Cai XF, et al. Diversity and bioactivities of cultured actinomycete isolated from animal feces and Xylocopa dissimilis[J]. Acta Microbiologica Sinica, 2009, 49(9) : 1152–1157. (in Chinese) 姜怡, 曹艳茹, 蔡祥凤, 等. 三种动物粪便及一种虫体可培养放线菌的多样性及其生物活性[J]. 微生物学报, 2009, 49(9) : 1152–1157. |
| [33] | Essoussi I, Ghodhbane-Gtari F, Amairi H, et al. Esterase as an enzymatic signature of Geodermatophilaceae adaptability to Sahara desert stones and monuments[J]. Journal of Applied Microbiology, 2010, 108(5) : 1723–1732. DOI:10.1111/jam.2010.108.issue-5 |
| [34] | Ivanova N, Sikorski J, Jando M, et al. Complete genome sequence of Geodermatophilus obscures type strain (G-20T)[J]. Standards in Genomic Sciences, 2010, 2(2) : 158–167. DOI:10.4056/sigs.711311 |
| [35] | Zhang YQ, Chen J, Liu HY, et al. Geodermatophilus ruber sp. nov., isolated from rhizosphere soil of a medicinal plant[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(1) : 190–193. DOI:10.1099/ijs.0.020610-0 |
| [36] | Jin L, Lee HG, Kim HS, et al. Geodermatophilus soli sp. nov. and Geodermatophilus terrae sp. nov., two actinobacteria isolated from grass soil[J]. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(7) : 2625–2629. |
| [37] | Chen LX, Hu T, Xu K, et al. The influences of probiotics on intestinal microflora in piglet[J]. China Animal Husbandry & Veterinary Medicine, 2013, 40(1) : 172–174. (in Chinese) 陈丽仙, 胡婷, 许可, 等. 益生菌剂对仔猪肠道菌群的影响[J]. 中国畜牧兽医, 2013, 40(1) : 172–174. |
| [38] | Tun HM, Mauroo NF, Yuen CS, et al. Microbial diversity and evidence of novel homoacetogens in the gut of both geriatric and adult giant pandas (Ailuropoda melanoleuca)[J]. PLoS One, 2014, 9(1) : e79902. DOI:10.1371/journal.pone.0079902 |
| [39] | Jian P, Wang Q, Wang J, et al. Difference analysis of gut microbiome of Rhinopithecus roxellana in Different Ages[J]. Chinese Journal of Animal Nutrition, 2015, 27(4) : 1302–1309. (in Chinese) 简平, 王强, 王剑, 等. 不同年龄段川金丝猴肠道菌群结构差异分析[J]. 动物营养学报, 2015, 27(4) : 1302–1309. |
| [40] | Zhou ZJ, Du HL, Shang WW, et al. Study the pig feces microflora in relation to gender and skatole[A]//The National Symposium on Feed Nutrition[C]. Zhengzhou: China Animal Husbandry and Veterinary Institute, 2014 (in Chinese) 周振金, 杜环利, 尚伟伟, 等. 不同性别猪粪样菌群结构及粪臭素产生相关菌群的研究[A]//第七届中国饲料营养学术研讨会论文集[C]. 郑州: 中国畜牧兽医学会, 2014 |
| [41] | Xu J. Comparative study on intestinal microflora of healthy People in different ages in Sichuan province[D]. Hohhot: Master's Thesis of Inner Mongolia Agricultural University, 2012 (in Chinese) 徐洁. 四川地区不同年龄健康人肠道菌群的比较研究[D]. 呼和浩特: 内蒙古农业大学硕士学位论文, 2012 |
| [42] | Mueller S, Saunier K, Hanisch C, et al. Differences in fecal microbiota in different European study populations in relation to age, gender, and country: a cross-sectional study[J]. Applied and Environmental Microbiology, 2006, 72(2) : 1027–1033. DOI:10.1128/AEM.72.2.1027-1033.2006 |
| [43] | Tan Z. The study on the normal intestinal microflora of the giant panda returned to wild and the giant panda in captivity[D]. Chengdu: Master's Thesis of Sichuan University, 2004 (in Chinese) 谭志. 野外放归大熊猫和圈养大熊猫肠道正常菌群的研究[D]. 成都: 四川大学硕士学位论文, 2004 |
| [44] | Jiang Y, Han L, Chen X, et al. Diversity and bioactivity of cultivable animal fecal actinobacteria[J]. Advances in Microbiology, 2013, 3(1) : 1–13. DOI:10.4236/aim.2013.31001 |
 2017, Vol. 44
2017, Vol. 44