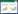扩展功能
文章信息
- 王鑫鑫, 杨珍珍, 周婉柔, 张美玲, 高红亮, 卞黎霞, 张冬梅
- WANG Xin-Xin, YANG Zhen-Zhen, ZHOU Wan-Rou, ZHANG Mei-Ling, GAO Hong-Liang, Bian Li-Xia, ZHANG Dong-Mei
- 水仙根围细菌群落组成
- Identification of rhizosphere bacterial communities of Narcissus tazetta
- 微生物学通报, 2017, 44(5): 1081-1088
- Microbiology China, 2017, 44(5): 1081-1088
- DOI: 10.13344/j.microbiol.china.160436
-
文章历史
- 收稿日期: 2016-06-05
- 接受日期: 2016-08-29
- 优先数字出版日期(www.cnki.net): 2016-09-07
2. 崇明县林业站 上海 202150;
3. 上海市园林科学规划研究院 上海 200232
2. Chongming County Forestry Station, Shanghai 202150, China;
3. Shanghai Academy of Landscape Architecture Science and Planning, Shanghai 200232, China
水仙(Narcissus tazetta)属于石蒜科水仙属,崇明水仙与漳州水仙是中国水仙的两大栽培类型。崇明水仙和漳州水仙均属于多花水仙(Narcissus tazetta L.),但崇明水仙商品球鳞茎小、少侧脚,花葶、花朵数少,而漳州水仙鳞茎肥大、多侧脚,花葶、花朵数多[1-2]。通过ISSR (Inter-simple sequence repeat)标记可以发现崇明水仙和漳州水仙平均遗传相似系数(GS)值为0.965,遗传距离0.003 5,二者相似度很高[1]。陈林姣等的研究结果也表明崇明水仙和漳州水仙亲缘关系很近[2],针对两种水仙在生长特性上的差异,有研究提出由于上百年的地理环境胁迫,以及经历的不同栽培方法,使得崇明水仙和漳州水仙在表型上产生一些变异,这种变异通过无性繁殖延续下来,而DNA序列上并没有产生变化[1]。植物根围微生物与植物的生长、生理状态、病害等有着较为密切的关系[3-4],刘丽等的研究表明根围微生物组成与大豆的单株分枝数、单株粒数和空荚数相关[5],随着微生物分子生态学技术的发展,植物生长状况与根围微生物的关系是目前国内外植物病理学及微生物学研究的热点之一[6]。然而目前对水仙的研究主要关注于内生真菌和次生代谢产物的发掘及育种上[7],对于水仙根围微生物与水仙生长特性之间关系的研究尚未见报道。
植物、植物内生物和植物根围微生物可以视为一个超级生物体[8],根围微生物的密度远远大于植物内的细胞密度,具有极高的生物多样性。植物根围细菌包括有益菌和有害菌(病原菌和非寄生性有害细菌)[9],有益菌可以促进植物吸收矿物质,同时产生的次生代谢产物也能促进植物生长,而有害菌会导致植物生长缓慢、植株矮小等[10]。因此,除了栽培方式和地理位置不同,根围微生物的组成与丰度是否与水仙的生长性状密切相关是本研究关注的主要问题。本研究分别采集了在崇明和漳州培育的崇明水仙和漳州水仙,观察其鳞茎球围径的大小、侧枝的多少以及根围微生物组成的差异,为阐明水仙根围微生物的组成以及根围微生物和与水仙生长性状之间的相关性提供基础数据。
1 材料与方法 1.1 材料 1.1.1 样地概况: (1) 崇明样地:实验样地位于上海向化镇向光村,崇明岛位于上海市东北部,处北亚热带,气候温和,年平均降水量一般在800-1 600 mm。(2) 漳州样地:实验样地位于漳州市九湖镇百花村,漳州属南亚热带海洋性季风气候,四季湿润,日照充分,年平均降水量一般为1 000-1 700 mm。 1.1.2 土壤样品收集: 漳州水仙土样于2015年11月6日采集于漳州样地,随机选择3个采样点,每个采样点分别采集一株水仙,用Riley和Barber的抖落法采集土样[11],挖取具有根系的土体,轻轻抖落大块不含根系的土壤,然后用力将根表面附着的土壤全部抖落下来,从而获得根围土壤,之后放置-20℃ 保存。崇明水仙土样于2015年11月7日采集于崇明样地,采集方法同上。 1.1.3 主要试剂和仪器: DNA抽提、纯化试剂盒及引物合成,上海美吉生物医药科技有限公司;其余PCR试剂购于生工生物工程(上海)股份有限公司;其他化学药品均为进口或国产分析纯。PCR仪购于TaKaRa宝生物工程有限公司;电泳仪购于Bio-Rad公司。 1.2 研究方法 1.2.1 直接提取土壤微生物的总DNA: 按试剂盒说明书直接提取土壤微生物的总DNA,然后用DNA纯化试剂盒对粗提的DNA进行纯化。 1.2.2 16S rRNA基因的PCR扩增: 根据Sun等[12]文献扩增16S rRNA基因Ⅴ4-Ⅴ5区。上游引物序列:5′-GAGAGTTTGATCCTGGCTCAG-3′,下游引物序列:5′-CTACGGCTACCTTGTTACG A-3′。反应体系:1.5 mmol/L 10×PCR缓冲液(含Mg2+) 5 μL,10 mmol/L dNTPs 1 μL,上下游引物(10 μmol/L)分别2μL,DNA模板(25 mg/L) 2 μL,20 g/l的2% BSA 2 mL,用无菌蒸馏水补至50 μL。反应条件:95℃ 90 s;95℃ 30 s,60℃30 s,72℃ 4 min,5个循环;95℃ 30 s,55℃ 30 s,72℃ 4 min,5个循环;95℃30 s,50℃ 30 s,72℃4 min,25个循环;72℃ 10 min,60℃ 10 min结束反应。 1.2.3 测序: 将总PCR产物送至上海美吉生物医药科技有限公司进行MiSeq高通量测序,通过对崇明水仙根围土壤(C,n=3) 和漳州水仙根围土壤(Z,n=3) 16S rRNA基因高通量测序(GenBank登录号:PR JNA322729),测定水仙根围土壤共生细菌的组成与分布。 1.2.4 数据分析: 用UPARSE (version 7.1,http://drive5.com/uparse/)根据97%的序列相似度将所有序列进行同源比对并聚类成操作分类单元(OTU),接着使用UCHIME确定和去除嵌合体[13],然后与数据库GreenGenes (version gg_13_8) 进行比对。稀释度曲线由Mothur分析得出,用于评价测序量是否足以覆盖所有类群,并间接反映样品中物种的丰富程度。以OTU丰度为基础,用ACE和Chao1反映群落丰富度,用辛普森指数[14]和香农-威纳指数[15]估计物种多样性。制作水仙根围细菌主要种系水平分布图时定义丰富度 > 1%的为主要种系。每组(C组或Z)中3个样品中2个及以上存在一类OTU时就定义该组中存在此类OTU,通过比较样品之间OTUs获得两组样品共有和特有的OTUs韦恩图。主成分分析(Principal component analysis,PCA)是一种多变量统计学方法,通过一系列的特征值和特征向量进行排序后,选择主要排在前几位的特征值,两点之间距离越近表明两者的群落构成差异越小。PCA分析基于OTU的分布及丰度数据。用R language制作热图来分析样品的差异,根据测序结果在目水平做热图分析,选取的细菌符合以下原则:(1) 优势菌种,即在两组或一组中的丰度 > 0.1%;(2) 在两组中的含量通过Student t检验呈现显著差异(P < 0.05),作图之前对每一组数据进行Z标准化。 2 结果与分析 2.1 两样地的理化特点及水仙鳞茎球的形态特征表 1所示为崇明和漳州土壤采集样地的理化特性。图 1为崇明水仙和漳州水仙鳞茎球的大小,从图 1中可以明显看出漳州水仙鳞茎球围径大于崇明水仙鳞茎球围径,崇明水仙鳞茎球围径平均值为5.03±0.15 cm,漳州水仙鳞茎球围径平均值为6.50±0.43 cm。
| 样品来源 Sample source |
pH | 有机质 organic matter (g/kg) |
全氮 total nitrogen (g/kg) |
全磷 total phospho- rus (%) |
全钾 total potassium (%) |
有效磷 Olsen-P (mg/kg) |
速效钾 Olsen-K (mg/kg) |
缓效钾 slowlyrele ased K (mg/kg) |
有效硼 Olsen-B (mg/kg) |
有效硫 Olsen-S (mg/kg) |
速效钾+缓效钾 Olsen-K+slowly released K (mg/kg) |
| 崇明样地 Chongming sample area |
8.01 | 13.69 | 1.234 | 0.161 | 1.29 | 33.14 | 58 | 569 | 0.487 | 62.96 | 627 |
| 漳州样地 Zhangzhou sample area |
6.09 | 15.68 | 1.520 | 0.136 | 2.15 | 12.22 | 59 | 426 | 0.962 | 9.87 | 485 |

|
| 图 1 崇明水仙鳞茎球(A)和漳州水仙鳞茎球(B) Figure 1 Bulbus of Chongming Narcissus (A) and Zhangzhou Narcissus (B) |
|
|
测序结果(表 2)共获得134 753条序列,其中C组序列条数为22 251-24 811,平均序列条数为23 195;Z组序列条数为18 590-27 144,平均序列条数为21 723,相比之下Z组内差异较C组大。按照序列97%的相似度进行OTUs划分,在C组平均获得1 703个OTUs,而在Z组平均获得1 521个OTUs。6个样品中的OTUs数量从1 466到1 719不等,且两组间OTUs分布差异显著(p < 0.05)。利用已测得的16S rRNA基因序列中已知的各种OTUs的相对比例计算稀释曲线(图 2),可看出6个样本稀释曲线已达到或将要达到平台期,表明测序深度已基本覆盖样品中所有的细菌类型。
| 组别 Groups |
总序列数 total sequence |
OTUs (97%) |
门 phylum |
目 order |
属 genus |
Chao1 | ace | 香农-威纳指数 Shannon-wiener index |
辛普森指数 Simpson’s diversity index |
|
| C | 1 | 22 251 | 1 689 | 33 | 179 | 491 | 1 885 | 1 868 | 6.42 | 0.003 7 |
| 2 | 22 522 | 1 719 | 33 | 185 | 501 | 1 910 | 1 895 | 6.40 | 0.004 2 | |
| 3 | 24 811 | 1 701 | 32 | 176 | 486 | 1 975 | 1 919 | 6.32 | 0.004 5 | |
| Z | 1 | 18 590 | 1 556 | 27 | 170 | 465 | 1 799 | 1 778 | 6.29 | 0.004 5 |
| 2 | 19 435 | 1 466 | 30 | 175 | 445 | 1 698 | 1 647 | 6.13 | 0.006 4 | |
| 3 | 27 144 | 1 541 | 31 | 176 | 454 | 1 709 | 1 678 | 6.32 | 0.003 9 | |
|
| 图 2 OTUs的稀释曲线 Figure 2 Rarefaction curves for OTUs |
|
|
为了测定和对比每个土壤样品中的菌群多样性,从每个土壤样品的OTUs中计算获得多样性指数,包括Chao1、ACE、辛普森指数和香浓-威纳指数。C组香浓-威纳指数为6.32-6.42,而Z组的值为6.13-6.32,说明两种水仙根围环境中细菌组成的多样性没有显著差异。
2.3 崇明水仙和漳州水仙根围细菌群落的组成在崇明水仙根围细菌群落中检测到30种不同门类的细菌,其中,12个门的细菌丰度高于总序列的1% (图 3A);在漳州水仙根围细菌群落中检测到33种不同门类的细菌,其中,12个门的细菌丰富度高于总序列的1% (图 3B)。崇明水仙和漳州水仙根围细菌群落虽然所含细菌门类数目不同,但主要的门类相同。两处样品的优势菌都为Proteobacteria、Chloroflexi、Bacteroidetes和Acidobacteria。

|
| 图 3 崇明水仙和漳州水仙根围细菌群丰富度对比图 Figure 3 Relative abundance of rhizosphere bacteria at phylum level in Chongming Narcissus and Zhangzhou Narcissus 注:A:崇明水仙根围细菌群主要种系门水平分布图;B:漳州水仙根围细菌群主要种系门水平分布图. Note: A: Relative abundance of the dominant bacterial phyla in rhizosphere bacteria in Chongming Narcissus; B: Relative abundance of the dominant bacterial phyla in rhizosphere bacteria in Zhangzhou Narcissus. |
|
|
利用韦恩图展示崇明水仙和漳州水仙根围细菌群落共有及特有的主要菌群,结果表明崇明水仙根围细菌群落与漳州水仙根围细菌群落共有762个OTUs (图 4),且共有的OTUs中所含序列分别占崇明水仙和漳州水仙根围细菌群落的43.22%和48.72%。有1 001个OTUs仅存在于崇明水仙根围土壤中,占崇明水仙根围土壤中细菌群落总序列的56.78%;802个OTUs仅存在于漳州水仙根围土壤中,占漳州水仙根围土壤中细菌群落总序列的51.28%。该部分结果表明虽然崇明水仙和漳州水仙根围细菌群落有一定量的共有菌群,但是它们均含有各自特有的根围细菌群落组成。

|
| 图 4 崇明水仙和漳州水仙根围微生物OTUs分布韦恩图 Figure 4 Venn diagram showing the distribution of all OTUs shared by Chongming Narcissus and Zhangzhou Narcissus 注:百分比表明每组OTUs在组内序列中的比例. Note: The percentages in the Venn diagram indicate the ratios of the sequences that are associated with the OTUs in total sequences in each group. |
|
|
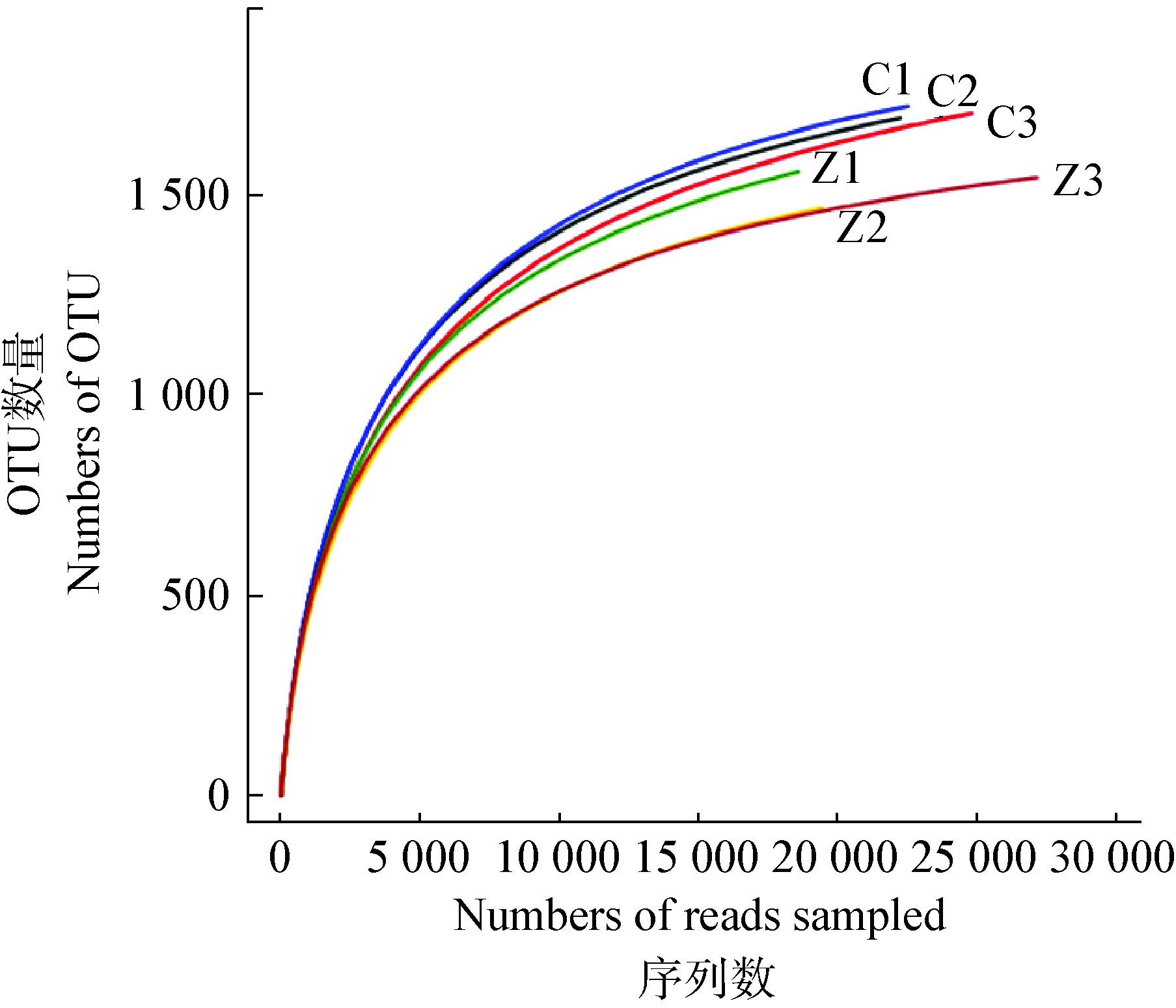
将两类水仙样品中根围细菌群落的OTU组成进行主成分分析(PCA),可以发现漳州水仙根围细菌群落与崇明水仙根围细菌群落被明显地分为两组,表明崇明水仙和漳州水仙根围细菌群落的组成有较大的差异(图 5)。
|
| 图 5 崇明水仙(红)和漳州水仙(蓝)根围细菌群主成分分析(PCA) Figure 5 Principal component analysis of the rhizosphere bacterial populations of Chongming Narcissus (red) and Zhangzhou Narcissus (blue) |
|
|
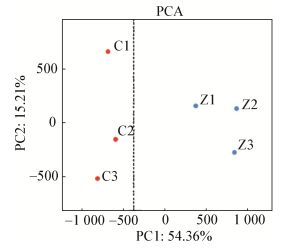
为进一步展示崇明水仙和漳州水仙根围细菌组成的差异,根据菌群组成的差异,在目(order)的水平上进行热图(heatmap)分析。与PCA结果吻合,崇明水仙和漳州水仙样品被清晰地分为两组(图 6)。两组样品中细菌群落都以Anaerolineales、Planctomycetales、Xanthomonadales ( > 3%)为主。在这涵盖50个目的细菌中,C组中发现所占比例较大而在Z组中所占比例很小(P < 0.05) 的细菌有Flavobacteriales、Phycisphaerales、Desulfuromonadales和Caldilineales共4目。在Z组中丰度较高,在C组中所占比例较小的细菌有Ignavibacteriales、Acidobacteriales、Chloroflexales、Gaiellales和Oligoflexales共5目。
|
| 图 6 崇明水仙和漳州水仙根围细菌丰富度热图 Figure 6 Heatmap of the bacterial abundance in the rhizosphere of Chongming Narcissus and Zhangzhou Narcissus |
|
|
崇明水仙和漳州水仙DNA差异不显著,但二者的性状如鳞茎球大小及花葶、花朵数量有差异,有研究猜测可能是由于表观遗传因素所致[1],而之前的研究表明植物根围微生物与植物的生长性状关系密切。根围微生物组就像是植物的第二套基因组[7],因此,研究植物根围微生物对于进一步了解植物的生长特性具有重要的意义。在本研究中,为了检测水仙根围细菌群落的组成与多样性,从而初步研究根围细菌与崇明水仙和漳州水仙生长特性的相关性,用高通量测序的方法对崇明和漳州两地的水仙根围土壤里的细菌群落进行测序。
从细菌门水平上的组成信息来看,崇明水仙和漳州水仙根围最为优势的细菌为Proteobacteria,占主要种系细菌的比例约为40%,这与之前其他植物根围细菌结构组成的研究结果相一致,例如,有研究表明马铃薯[16]、甜菜[17]、三七[18]、水葫芦[19]的根围细菌菌落中Proteobacteria均为优势菌。在这些植物中,Actinomycetes通常也是优势菌,然而在水仙根围细菌群落中,Actinobacteria所占比例却很小( < 1%),但在这些植物根围细菌群落中所占比例不高的Chloroflexi却是水仙根围细菌群落的优势菌,这也表明不同种植物所具有的根围微生物可能不同[20]。
在细菌目水平的组成分布上,可以发现Planctomycetales (P < 0.05) 和Xanthomonadales (P < 0.01) 在崇明水仙根围分布显著多于漳州水仙。Planctomycetales中的Anammoxaceae大多数为厌氧氨氧化菌,厌氧氨氧化菌可以把土壤中的亚硝酸根转化为氮气[21],研究发现厌氧氨氧化菌对水稻根围氮气产生量的贡献率为31%-41%[22],从而导致土壤氮肥的流失。然而何炎森等[23]和惠楠等[24]研究表明氮肥可以促进水仙种球围径的增加。崇明水仙根围细菌中这类细菌的含量较多,可能与其鳞茎球瘦小及花葶、花朵数少等生长特性有关。另外,已有研究表明Xanthomonas的细菌已经引起124种单子叶植物和268种双子叶植物致病[25],这可能是崇明水仙易感染的原因,虽然本研究中所用的水仙均为健康植株,但是崇明水仙根围某些细菌的组成可能与其易染病的特性有关。同时,一些光合细菌,如Chloroflexales (P < 0.01)、Chlorobiales (P < 0.05) 在漳州水仙根围的丰度明显高于崇明水仙。Chloroflexales、Chlorobiaceae二者都是光合细菌,前者被称为绿色非硫细菌[26],而后者被称为绿色硫细菌[27]。研究表明光合细菌能够分解硫化物、农药等多种有害物质,促进植物固氮,增加土壤肥力[28-29],促进植物生长,同时也可以改善植物根围微生物菌群,减少植物发病概率[30]。研究证明用109个/mL浓度绿硫细菌(Chlobrobiumlimicola)液处理黄瓜幼苗时,黄瓜幼苗的株高、茎粗和鲜重都有明显的增加[31],因此光合细菌组成的差异可能是漳州水仙生长特性优于崇明水仙的另一个原因。
本研究结合高通量测序技术和生物学信息方法分析崇明水仙和漳州水仙根围微生物组成,结果表明与其它植物相比,水仙根围具有其特有的微生物群落,而根围微生物的种类和丰度也可能与水仙生长特性密切相关,通过调节植株根围微生物的种类和组成,如提高光合细菌在水仙根围的定殖量、降低厌氧氨氧化菌的含量可能是提高水仙抗病力和促进水仙生长的有效措施。
| [1] | Zeng Y, Zhou YG, Bian LX, et al. 4 kinds of narcissus germplasm resources of ISSR and AFLP analysis of genetic diversity[J]. Fenzi Zhiwu Yuzhong (online), 2011, 9 : 1759–1765. (in Chinese) 曾艳, 周永刚, 卞黎霞, 等. 4种水仙种质资源的ISSR和AFLP遗传多样性分析[J]. 分子植物育种(网络版), 2011, 9 : 1759–1765. |
| [2] | Chen LJ, Miao Y, Chen DH, et al. Analysis of Germplasmresources of Narcissus tazetta L. var. chinensis by RAPD[J]. Journal of Xiamen University (Natural Science), 2002, 41(6) : 810–814. (in Chinese) 陈林姣, 缪颖, 陈德海, 等. 中国水仙种质资源的遗传多样性分析[J]. 厦门大学学报:自然科学版, 2002, 41(6) : 810–814. |
| [3] | Zhang L, Xu HM, Zhu BL. Association of rhizosphere soil microbiome with the occurrence and development of replant disease-a review[J]. Acta Microbiologica Sinica, 2016, 56(8) : 1234–1241. (in Chinese) 张蕾, 徐慧敏, 朱宝利. 根际微生物与植物再植病的发生发展关系[J]. 微生物学报, 2016, 56(8) : 1234–1241. |
| [4] | van der Ent S, van Wees SCM, Pieterse CMJ. Jasmonate signaling in plant interactions with resistance-inducing beneficial microbes[J]. Phytochemistry, 2009, 70(13/14) : 1581–1588. |
| [5] | Liu L, Ma MC, Jiang X, et al. Effect of rhizobia and PGPR co-inoculant on soybean characteristics and soil enzyme activities[J]. Plant Nutrition and Fertilizer Science, 2015, 21(3) : 644–654. (in Chinese) 刘丽, 马鸣超, 姜昕, 等. 根瘤菌与促生菌双接种对大豆生长和土壤酶活的影响[J]. 植物营养与肥料学报, 2015, 21(3) : 644–654. DOI:10.11674/zwyf.2015.0311 |
| [6] | Berendsen RL, Pieterse CMJ, Bakker PAHM. The rhizosphere microbiome and plant health[J]. Trends in Plant Science, 2012, 17(8) : 478–486. DOI:10.1016/j.tplants.2012.04.001 |
| [7] | Chen DF, Gao J, Peng ZH. Advances and perspectives in studies on narcissus plants[J]. Scientia Silvae Sinicae, 2008, 44(3) : 140–146. (in Chinese) 陈段芬, 高健, 彭镇华. 水仙属植物研究进展[J]. 林业科学, 2008, 44(3) : 140–146. DOI:10.11707/j.1001-7488.20080327 |
| [8] | Mendes R, Kruijt M, de Bruijn I, et al. Deciphering the rhizosphere microbiome for disease-suppressive bacteria[J]. Science, 2011, 332(6033) : 1097–1100. DOI:10.1126/science.1203980 |
| [9] | Wang XC, Tong XR, Wen XS, et al. Progress on the relationship between plant and rhizospheric microorganism[J]. Shandong Science, 2007, 20(6) : 40–44, 50 . (in Chinese) 王学翠, 童晓茹, 温学森, 等. 植物与根际微生物关系的研究进展[J]. 山东科学, 2007, 20(6) : 40–44, 50 . |
| [10] | Hu JC, Xue DL, Ma CX, et al. Research advances in plant growth-promoting rhizobacteria and its application prospects[J]. Chinese Journal of Applied Ecology, 2004, 15(10) : 1963–1966. (in Chinese) 胡江春, 薛德林, 马成新, 等. 植物根际促生菌(PGPR)的研究与应用前景[J]. 应用生态学报, 2004, 15(10) : 1963–1966. DOI:10.3321/j.issn:1001-9332.2004.10.052 |
| [11] | Riley D, Barber SA. icarbonate accumulation and pH changes at the soybean (Glycine max (L.) Merr.) root-soilinterface[J]. Soil Science Society of America Journal, 1969, 33(6) : 905–908. DOI:10.2136/sssaj1969.03615995003300060031x |
| [12] | Sun DL, Jiang X, Wu QL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryoticdiversity[J]. Applied and Environmental Microbiology, 2013, 79(19) : 5962–5969. DOI:10.1128/AEM.01282-13 |
| [13] | Edgar RC, Haas BJ, Clemente JC, et al. UCHIME improves sensitivity and speed of chimeradetection[J]. Bioinformatics, 2011, 27(16) : 2194–2200. DOI:10.1093/bioinformatics/btr381 |
| [14] | Sagar R, Sharma GP. Measurement of alpha diversity using Simpson index (1/Lamda): the jeopardy[J]. Environmental Skeptics and Critics, 2012, 1(1) : 23–24. |
| [15] | di Nardo A, Yamasaki K, Dorschner RA, et al. Mast cell cathelicidin antimicrobial peptide prevents invasive group A Streptococcus infection of the skin[J]. The Journal of Immunology, 2008, 180(11) : 7565–7573. DOI:10.4049/jimmunol.180.11.7565 |
| [16] | Weinert N, Piceno Y, Ding GC, et al. PhyloChip hybridization uncovered an enormous bacterial diversity in the rhizosphere of different potato cultivars: many common and few cultivar–dependenttaxa[J]. FEMS Microbiology Ecology, 2011, 75(3) : 497–506. DOI:10.1111/fem.2011.75.issue-3 |
| [17] | Mendes R, Kruijt M, de Bruijn I, et al. Deciphering the rhizosphere microbiome for disease-suppressive bacteria[J]. Science, 2011, 332(6033) : 1097–1100. DOI:10.1126/science.1203980 |
| [18] | Lu XJ, Guan HL, Zhang ZY, et al. Microbial distribution and 16S rRNA diversity in the rhizosphere soil of Panaxnotoginseng[J]. Acta Microbiologica Sinica, 2015, 55(2) : 205–213. (in Chinese) 陆晓菊, 官会林, 张正芸, 等. 三七连作根际土壤微生物区系的16S rRNA系统遗传多样性[J]. 微生物学报, 2015, 55(2) : 205–213. |
| [19] | Zheng LJ, Fu MH. Analysis of the diversity of bacterial community structure in the rhizosphere of Eichhornia crassipes[J]. Microbiology China, 2015, 42(11) : 2115–2125. (in Chinese) 郑李军, 傅明辉. 水葫芦根际细菌群落结构多样性分析[J]. 微生物学通报, 2015, 42(11) : 2115–2125. |
| [20] | Hartmann A, Schmid M, Wenzel W, et al. Rhizosphere 2004-perspectives and challenges –a tribute to Lorenz Hiltner[A]//GSF-Bericht, GSF-National[M]. Munich, Germany: Research Center for Environment and Health, 2004 |
| [21] | van de Graff AA, Mulder A, de Bruijn P, et al. Anaerobic oxidation of ammonium is a biologically mediated process[J]. Applied and Environmental Microbiology, 1995, 61(4) : 1246–1251. |
| [22] | Nie SA, Li H, Yang XR, et al. Nitrogen loss by anaerobic oxidation of ammonium in rice rhizosphere[J]. The ISME Journal, 2015, 9(9) : 2059–2067. DOI:10.1038/ismej.2015.25 |
| [23] | He YS, Li RM, Wu WJ, et al. Effect of nitrogen application rate on Narcissus growth and bulb quality[J]. Acta Agriculturae Jiangxi, 2006, 18(5) : 118–120. (in Chinese) 何炎森, 李瑞美, 吴维坚, 等. 不同施氮水平对水仙花生长和鳞茎球质量的影响[J]. 江西农业学报, 2006, 18(5) : 118–120. |
| [24] | Hui N, Qin J, Hu YH. Effect of different levels mineral elements on bulbs of Chongming Narcissus (Narcissus tazetta var. chinensis)[A]//Advances in Ornamental Horticulfure of China[C]. Beijing: Chinese Society for Horticultural Science, 2010: 361-365 (in Chinese) 惠楠, 秦俊, 胡永红. 不同矿质元素水平对崇明水仙种球围径的影响[A]//中国观赏园艺研究进展[C]. 北京: 中国园艺学会, 2010: 361-365 |
| [25] | Saddler GS, Bradbury JF. Xanthomonadales ord. nov.[A]// Brenner DJ, Krieg NR, Staley JT, et al. Bergey's Manual® of Systematic Bacteriology[M]. US: Springer, 2005: 63-122 |
| [26] | Hanada S. The phylum chloroflexi, the family Chloroflexaceae, and the related phototrophic families Oscillochloridaceaeand Roseiflexaceae[A]//Rosenberg E, DeLong EF, Lory S, et al. The Prokaryotes[M]. Berlin Heidelberg: Springer, 2014: 515-532 |
| [27] | Imhoff JF. The family Chlorobiaceae[A]//Rosenberg E, DeLong EF, LoryS, et al. The Prokaryotes[M]. Berlin Heidelberg: Springer, 2014: 501-514 |
| [28] | Ludden PW, Roberts GP. Nitrogen fixation by photosynthetic bacteria[J]. Photosynthesis Research, 2002, 73(1/3) : 115–118. DOI:10.1023/A:1020497619288 |
| [29] | Wu SB, Liang WZ. Inhibition of inorganic sulphocompounds in photosynthetic bacteria biotreamentprocess[J]. Transaction of China Pulp and Paper, 1996, 11(S1) : 57–60. (in Chinese) 武书彬, 梁文芷. 无机含硫化合物对光合细菌生物处理过程抑制作用的研究[J]. 中国造纸学报, 1996, 11(S1) : 57–60. |
| [30] | Marrs BL. The early history of the genetics of photosynthetic bacteria: a personal account[A]//Govindjee, Beatty JT, Gest H, et al. Discoveries in Photosynthesis[M]. Netherlands: Springer, 2005: 55-58 |
| [31] | Shao S, Cui XD. Effect of photosynthetic bacteria on the growth of cucumber seedlings[J]. Journal of Shenyang Institute of Chemical Technology, 2010, 24(3) : 215–218. (in Chinese) 邵双, 崔心栋. 光合细菌促进黄瓜幼苗生长作用的研究[J]. 沈阳化工大学学报, 2010, 24(3) : 215–218. |
 2017, Vol. 44
2017, Vol. 44