扩展功能
文章信息
- 汤丽燕, 杨乐, 邢新婧, 柏桂珍, 张克勤, 杨金奎
- TANG Li-Yan, YANG Le, XING Xin-Jing, BAI Gui-Zhen, ZHANG Ke-Qin, YANG Jin-Kui
- 食线虫真菌致病相关丝氨酸蛋白酶的研究进展
- Advances in pathogenicity-related serine proteases in nematophagous fungi
- 微生物学通报, 2017, 44(4): 911-919
- Microbiology China, 2017, 44(4): 911-919
- DOI: 10.13344/j.microbiol.china.160344
-
文章历史
- 收稿日期: 2016-04-28
- 接受日期: 2016-06-24
- 优先数字出版日期(www.cnki.net): 2016-08-01
线虫属于线虫动物门 (Aschelminthes) 线虫纲 (Nematoda),是动物界中数量最多的种类之一,寄生于动植物,或自由生活于土壤、淡水和海水环境中,绝大多数营自由生活,只有极少部分寄生于人体并引起疾病[1]。作物病原线虫危害非常严重,全球每年因作物病原线虫所引起的农作物经济损失高达1570亿美元[2]。传统的化学农药毒性高,长期不合理使用易造成过度依赖、农药残留等问题,使得农产品和环境污染现象日益突出[3]。食线虫真菌作为线虫的天敌之一,是自然界中控制、平衡和调节线虫种群数量的重要生物因子,也是研究开发生物杀线虫制剂的主要来源[4]。依据侵害线虫的习性差异可以把它们分为捕食线虫真菌 (Trapping fungi)、内寄生真菌 (Endoparasitic fungi)、机会真菌 (Opportunistic fungi) 和产毒真菌 (Toxin-producing fungi) 四大类群[5]。食线虫真菌侵染线虫主要包括以下步骤:(1) 吸引和识别;(2) 捕捉和黏附;(3) 侵入;(4) 固定和消解线虫[6]。侵染过程不仅需要真菌自身的机械作用,同时还有胞外水解酶的参与,包括蛋白酶 (尤其是枯草杆菌素丝氨酸蛋白酶)、几丁质酶和胶原酶等[7]。
线虫表皮主要由蛋白质组成,是它与体外环境隔绝的屏障,也是食线虫真菌侵染线虫时需要突破的第一道关卡,因此蛋白酶在食线虫真菌侵染线虫过程中显得尤为重要[8]。前期的研究发现,食线虫真菌产生的丝氨酸蛋白酶在侵染线虫的过程中发挥着重要的作用,它们能够降解线虫的体壁和消化线虫[7, 9]。1990年从线虫内寄生真菌萨克拉普可尼亚菌 (Verticillium suchlasporium, syn. Pochonia suchlasporia) 中分离得到第一个致病相关丝氨酸蛋白酶P32[10]。随后,更多的致病相关丝氨酸蛋白酶也陆续在其它食线虫真菌中被分离和鉴定[11],同时丝氨酸蛋白酶的结构和功能研究也不断地得到深入[12-13],本文对食线虫真菌致病相关丝氨酸蛋白酶的生物化学性质和功能进行了系统的总结,尤其对最近几年的研究进展包括丝氨酸蛋白酶的晶体结构和作用机制,以及丝氨酸蛋白酶的分子进化和正选择作用等进行了探讨。
1 食线虫真菌致病相关丝氨酸蛋白酶的生物化学性质和功能 1.1 丝氨酸蛋白酶的生物化学性质丝氨酸蛋白酶是一类重要的蛋白酶家族,它们的活性中心由丝氨酸、组氨酸和天冬氨酸组成,能够水解大分子蛋白质的肽键,使之断裂成为小分子的多肽和氨基酸。丝氨酸蛋白酶的生物学功能多样,同时它们还是细菌和真菌病原对抗昆虫、线虫甚至脊椎动物的重要致病因素[14]。丝氨酸蛋白酶是一种重要的胞外酶,大量产生于病原真菌,如植物病原真菌棉花黄萎病菌 (Verticillium dahliae)[15]和昆虫病原真菌金龟子绿僵菌 (Metarhizium anisopliae)[16]。它通常被划分为两个蛋白超家族:胰凝乳蛋白酶超家族 (Chymotrypsin superfamily) 和枯草杆菌素超家族 (Subtilase superfamily)。根据序列同源性和功能相似性,每个超家族又被划分为不同的蛋白家族[14]。目前已经从多种食线虫真菌中分离得到了蛋白酶K家族 (Protease K family) 的丝氨酸蛋白酶。蛋白酶K家族是枯草杆菌素丝氨酸蛋白酶超家族中一类重要的蛋白家族[17]。
1990年,从萨克拉普可尼亚菌 (P.suchlasporia) 中鉴定出首个致病相关丝氨酸蛋白酶P32[10]。随后,又分别从捕食线虫真菌少孢节丛孢 (Arthrobotrys oligospora) 和线虫内寄生真菌厚垣普可尼亚菌 (Pochonia chlamydosporia) 中分离和鉴定出2种丝氨酸蛋白酶PII[18]和VCP1[19]。目前,已从不同的食线虫真菌包括淡紫拟青霉 (Paecilomyces lilacinus)、刀孢轮枝菌 (Lecanicillium psalliotae)、粉红螺旋聚孢霉 (Clonostachys rosea)、中华被毛孢 (Cordyceps sinensis) 和洛斯里被毛孢 (Hirsutella rhossiliensis) 等中鉴定出20多种致病相关丝氨酸蛋白酶 (如pSP-3、Ver112、PrC、Csp1和Hnsp等)[9, 20]。表 1显示了不同食线虫真菌来源的致病相关丝氨酸蛋白酶的部分生物化学性质。
| 蛋白酶Proteases | 食线虫真菌Nematophagous fungi | 分子质量Molecular weights (kD) | 抑制剂Inhibitors | 最适pH Optimal pH | 等电点Isoelectric point (pI) | 参考文献Refrences |
| PII | 少孢节丛孢Arthrobotrys oligospora | 35.0 | PMSF, Chymostatin | 7.0–9.0 | 4.6 | [18] |
| Aoz1 | 少孢节丛孢A.oligospora | 38.0 | PMSF, SSI | 6.0–8.0 | 4.9 | [21] |
| rexAoz1 | 少孢节丛孢A.oligospora | 50.0 | – | 8.5 | – | [22] |
| reP186 | 少孢节丛孢A.oligospora | 33.0 | PMSF | 8.0 | – | [23] |
| Ac1 | 圆锥节丛孢A.conoides | 35.0 | PMSF | 7.0 | – | [24] |
| AmSP1 | 弯孢节丛孢A.musiformis | 41.9 | – | 6.5 | − | [25] |
| Mlx | 小舟单顶孢Monacrosporium microscaphoides | 39.0 | PMSF | 9.0 | 6.8 | [26] |
| Mc1 | 囊孢单顶孢M.cystosporium | 38.0 | PMSF | 7.0–9.0 | – | [27] |
| Mt1 | 奇妙单顶孢M.thaumasium | 40.0 | PMSF | 7.0–8.0 | – | [28] |
| Ds1 | 狮子山隔指孢Dactylella shizishanna | 32.0 | PMSF | 10.0 | − | [29] |
| Dv1 | 异形隔指孢D.varietas | 30.0 | PMSF | 8.0 | – | [11] |
| PrD1 | Dactylellina cionopaga | 45.0 | – | 5.0 | – | [30] |
| Df1 | Duddingtonia flagrans | 38.0 | PMSF | 8.0 | – | [31] |
| P32 | 萨克拉普可尼亚菌Pochonia suchlasporia | 32.0 | PMSF, pCMB | 8.5 | − | [10] |
| VCP1 | 厚垣普可尼亚菌P.chlamydosporia | 33.0 | PMSF | 10.2 | 10.2 | [19] |
| pSP-3 | 淡紫拟青霉Paecilomyces lilacinus | 33.5 | PMSF | 10.3 | 10.2 | [32] |
| Ver112 | 刀孢轮枝菌Lecanicillium psalliotae | 32.0 | PMSF | 10.0 | 10.2 | [33] |
| PrC | 粉红螺旋聚孢霉Clonostachys rosea | 33.0 | PMSF | 9.0–10.0 | 10.0 | [34] |
| Csp1 | 中华被毛孢Cordyceps sinensis | 36.0 | PMSF | 7.0 | – | [35] |
| Csp2 | 中华被毛孢C.sinensis | 35.0 | PMSF | 7.0 | – | [35] |
| Hnsp | 洛斯里被毛孢Hirsutella rhossiliensis | 32.0 | PMSF | 7.0 | – | [36] |
| Hasp | 洛斯里被毛孢H.rhossiliensis | 33.0 | PMSF | 9.0 | – | [37] |
| 注:PMSF:苯甲基磺酰氟,丝氨酸蛋白酶的专一性抑制剂;SSI:链霉菌枯草杆菌蛋白酶抑制剂;pCMB:对氯汞苯甲酸. −:文献中没有报道. | ||||||
| Note: PMSF: phenylmethanesulfonyl fluoride; SSI: Streptomyces subtilisin inhibitor; pCMB: 4-chloromercuribenzoic acid. −: no reported in the references. | ||||||
不同食线虫真菌来源的致病相关蛋白酶对丝氨酸蛋白酶专一性抑制剂PMSF具有高度的敏感性,它们具有相近的分子量 (约32.0−45.0 kD) (表 1),能利用多种蛋白质作为底物,如酪蛋白、明胶和线虫表皮等[9, 16]。同时,从捕食线虫真菌中分离的蛋白酶PII (A.oligospora)[18]、Aoz1 (A.oligospora)[21]、Mlx (M.microscaphoides)[26]和Dsl (D.shizishanna)[29]的最适pH和等电点 (pI) 值相对较低,而从内寄生真菌分离的蛋白酶P32 (P.rubescens)[10]、VCP1 (P.chlamysporia)[19]、pSP-3 (P.lilacinus)[32]、Ver112 (L.psalliotae)[33]和PrC (C.rosea)[34]的最适pH和pI值较高。近期的研究发现,较高的pI值有利于蛋白酶自身的水解活性,并在酶与线虫表皮结合过程中起到了重要的作用[20]。根据生物化学性质可以将来源不同的致病相关丝氨酸蛋白酶分为两大类:一类是捕食线虫真菌产生的蛋白酶,另一类是内寄生真菌产生的蛋白酶[20]。一致性比较发现,捕食线虫真菌产生的丝氨酸蛋白酶之间的氨基酸一致性高于捕食线虫真菌与内寄生真菌或机会病原真菌所产生蛋白酶的序列一致性[9, 20]。
近期的研究发现,食线虫真菌对不同种类的线虫表现出不同的杀线虫活性,同时来源于不同真菌的致病相关丝氨酸蛋白酶对不同线虫的作用效果也不一致,说明真菌对线虫的侵染在一定程度上依赖于所产生的丝氨酸蛋白酶的性质[20, 26],依据这一特性,可以选择不同的食线虫真菌防治不同的线虫病害,提高生防制剂的杀线虫效率。
1.2 丝氨酸蛋白酶的功能食线虫真菌侵染线虫时必须首先突破线虫体壁这一天然屏障,前期的研究表明丝氨酸蛋白酶等胞外酶作为一种重要的毒力因子在降解线虫体壁的过程中发挥着非常重要的作用[9, 20]。少孢节丛孢 (A.oligospora) 是一种典型的捕食线虫真菌,产生一种特殊的捕食器官三维菌网捕捉线虫,当菌丝黏附于线虫体表时,菌丝会分泌胞外酶降解线虫表皮[18, 38]。1994年,Tunlid等证实了胞外丝氨酸蛋白酶PII不仅能够固定全齿复活线虫 (Panagrellus redivius),而且能够水解线虫表皮的蛋白质[18]。2002年,hman等构建了含有多拷贝PII蛋白酶编码基因的突变株,检测结果表明PII基因的多拷贝突变株具有更高的杀线虫能力,说明在少孢节丛孢侵染线虫过程中,丝氨酸蛋白酶PII起到了重要的作用[39-40]。最近,云南大学杨金奎等对少孢节丛孢的全基因组进行了测序,发现了24个基因编码枯草杆菌素丝氨酸蛋白酶,其中有20个属于蛋白酶K家族,蛋白酶K家族又可细分为4个亚家族,其中丝氨酸蛋白酶P186、P233和P12与PII聚类形成一个进化分支。这些蛋白酶在少孢节丛孢侵染线虫过程中可能具有相似的生物学功能[41]。为了进一步验证以上的推测,利用同源重组技术敲除蛋白酶P186的编码基因并进行生物测定,发现在侵染初期 (24 h以前) 突变菌株的杀线虫速率下降,表明除了PII,其它的蛋白酶如P186等在捕食线虫真菌致病过程中也发挥着重要的作用[41]。最近,Zhao等扩增得到少孢节丛孢丝氨酸蛋白酶P186的基因序列,插入pPIC9K载体在毕赤酵母中成功表达,纯化后的蛋白酶reP186能够降解包括酪蛋白等多种蛋白质底物,并能有效降解线虫表皮层,破坏线虫组织器官结构,甚至杀死线虫[23]。同时,Wang等从少孢节丛孢中扩增得到丝氨酸蛋白酶Aoz1的编码基因,并在毕赤酵母中表达。这种重组蛋白酶XAoz1对秀丽隐杆线虫 (Caenorhabditis elegans) 和寄生于动物的捻转血矛线虫 (Haemonchus contortus) 均表现出较强的杀线虫活性,它能够黏附于线虫表皮,固定线虫并且降解线虫的体壁,有助于菌丝的侵入[22]。以上研究都进一步证实了丝氨酸蛋白酶PII和Aoz1等在真菌侵染线虫的过程中发挥着十分重要的作用。
内寄生真菌和机会病原真菌通过寄生线虫的卵、孢囊和游离线虫实现对线虫的侵染[20]。1995年,从淡紫拟青霉 (P.lilacinus) 的发酵液中纯化得到丝氨酸蛋白酶pSP-3,pSP-3严重影响线虫卵的孵化率,对不成熟的线虫卵的作用更为明显,但对已孵化的线虫没有明显的影响,说明pSP-3可能在侵染线虫卵的过程中起作用。透射电镜观察发现pSP-3和淡紫拟青霉产生的几丁质酶单独作用或协同作用均能改变线虫卵壳结构,蛋白酶处理使得线虫卵壳的内脂质层解体,中间几丁质层变薄[32]。刀孢轮枝菌 (L.psalliotae) 是一种重要的内寄生真菌,从该菌中分离得到一种丝氨酸蛋白酶Ver112,与其它的丝氨酸蛋白酶相似,Ver112能够降解多种蛋白质底物,但Ver112纯酶的水解活性高于PII。分别用丝氨酸蛋白酶Ver112和PII处理全齿复活线虫,前者12 h的致死率高达81%,后者20−22 h后仅有71%的线虫被固定[13, 33]。洛斯里被毛孢 (H.rhossiliensis) OWVT-1能产生两种胞外丝氨酸蛋白酶Hasp和Hnsp,它们都能杀死大豆孢囊线虫 (Heterodera glycines) 的幼虫并且能够降解线虫的体壁蛋白[36-37]。
2008年,Zhang等从中华被毛孢 (C.sinensis) 中克隆得到两个丝氨酸蛋白酶 (csp1和csp2),并在毕赤氏酵母中表达,生防活性测定实验表明csp1和csp2能够降解蝙蝠蛾 (Hepialus sp.) 幼虫的体壁蛋白[35]。拟康氏木霉 (T.pseudokoningii) SMF2可有效抑制植物病原真菌的生长,从SMF2发酵液中纯化得到一种分子量为31 kD的丝氨酸蛋白酶 (SprT),最适反应pH和温度分别为8.5和60−65 ℃。SprT可降解牛血清白蛋白、溶菌酶和明胶等的蛋白质底物;同时,它还具有较强的杀线虫活性,线虫的表皮经过蛋白酶SprT处理后明显皱缩,SprT还能够杀死根结线虫的幼虫,并且能够抑制线虫卵的孵化[42]。
最近,云南大学邹成钢等对粉红螺旋聚孢霉 (C.rosea) 的胞外丝氨酸蛋白酶PrC的表达调控进行了较为深入的研究。发现PrC基因受到环境pH的调控,在碱性pH下PrC基因的表达上调,而在酸性pH下PrC基因的表达被抑制。在PrC基因的启动子上有两个pH转录因子PacC的结合元件,敲除PacC基因导致粉红螺旋聚孢霉对线虫的侵染能力下降,说明pH转录因子PacC在真菌侵染线虫的过程中起正调节作用[43]。同时,PrC基因的表达还受到氮源的调控,谷氨酸等容易被真菌利用的氮源抑制PrC基因的表达,线虫表皮和表皮降解产物能促进PrC基因的表达[44]。此外,PrC基因的表达还受到氧化压力等环境条件的调控,氧化剂和热激处理能显著诱导PrC基因的表达。氧化剂和热激通过诱导氧化应激,抑制真菌孢子萌发,在线虫体壁的存在下,这种抑制作用被部分解除。进一步研究表明,在线虫体壁的存在下,氧化剂和热激处理的孢子中的活性氧自由基水平显著降低,这可能是由于线虫体壁的降解产物能够通过除去活性氧发挥保护功能[45]。以上研究不仅说明蛋白酶PrC能够降解线虫表皮帮助真菌侵染线虫,也揭示了食线虫真菌适应环境压力的一种新的生存策略。
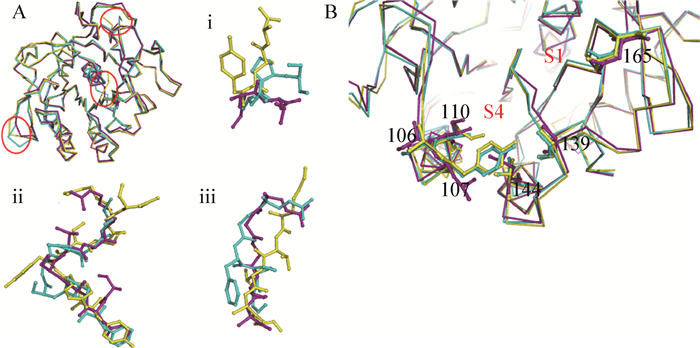
2 食线虫真菌致病相关丝氨酸蛋白酶的晶体结构和催化机制研究一些病原真菌不仅可以侵染昆虫和线虫,同时也能感染人类,如淡紫拟青霉 (P.lilacinus)[46]。因此,研究食线虫真菌丝氨酸蛋白酶晶体结构及其作用机制不仅为提高食线虫真菌的线虫生防效率,同时也可以促进人体病原菌的致病机制研究,在临床治疗上还能提供对抗病原真菌的治疗药物靶位点[46]。近年来,云南大学梁连铭等对两种食线虫真菌丝氨酸蛋白酶Ver112 (L.psalliotae) 和PL646 (P.lilacinus) 的晶体结构、底物偏好性和催化机制进行了深入的研究[12]。通过悬滴气相扩散结晶法获得蛋白酶Ver112和PL646的晶体,并通过浸泡法获得蛋白酶和抑制剂的复合物结晶。X-衍射结果表明,蛋白酶Ver112和PL646的分辨率分别达到1.6 (Ver112) 和2.1 (PL646)。两个蛋白酶都是球形蛋白,具有6个α螺旋,一个7股平行β折叠片,两个2股反平行β折叠片组成,另外Ver112有一个3/10螺旋。Ver112的底物结合区主要由两段氨基酸残基103−107和135−139组成。Asp41、His72和Ser227组成了Ver112的催化三聚体。此外,它们都含有一个保守的Ca2+结合位点,Ca2+的存在可增强蛋白酶的热稳定性[12]。
对食线虫真菌来源的体壁降解丝氨酸蛋白酶PL646 (ABO32256)、Ver112 (AAU01968)、VCP1 (CAD20580)、Pr1A (P29138) 和白色念球菌 (Tritirachium album) 蛋白酶K (ProK,P06873) 进行了序列比对,发现它们具有较高的序列相似性 ( > 60%)[12]。将Ver112、PL646和ProK的结构进行重叠比对,发现它们具有相似的折叠结构,非重合区仅存在于一些表面环区 (图 1a)。同时,对3种蛋白酶的底物结合位点进行比较,发现主要的差异在于底物结合区的S1口袋和S4口袋的氨基酸残基 (图 1b)。对于S1口袋,其165位 (序号对应PL646) 氨基酸在ProK中是Asn,而在Ver112和PL646是Asp。通过比较Asn和Asp在ProK、PL646和Ver112三维结构中的构象,发现二者的侧链具有相似的立体化学取向,即侧链均指向S1口袋内部。Asp在中性环境中携带两个负电荷,因此使得Ver112和PL646的S1口袋表现出更强的负电特征,这一负电特征可能与其功能相关。对于S4口袋,氨基酸残基变化发生在106、107、110、139和144位 (PL646序号)。ProK的106位是Gln,而在Ver112和PL646是Ser,Ser的侧链尺寸小于Asn侧链,因此Ver112和PL646相比ProK而言,其S4口袋更大[12]。为了比较以上3种蛋白酶的催化特点,梁连铭等在经典蛋白酶底物Suc-AAPF-pNA的基础上突变单个氨基酸获得了一些新的四肽底物,分别测定了3个蛋白酶对这些底物的酶动力学参数。实验结果证实了底物结合位点氨基酸残基的变化确实影响蛋白酶对不同多肽底物的催化活性。同时,这些蛋白酶具有较大的底物结合口袋,且S1口袋为疏水性口袋,因此对P1位点为大型疏水残基 (如苯丙氨酸) 的底物较为偏好。此外,S4口袋虽然比S1口袋小,但它仍然能够容纳较大的残基侧链。原因可能是大的P4残基侧链能够与S4口袋残基形成更多的非共价键作用,如氢键、疏水作用和范德华力[12-13]。
|
| 图 1 体壁降解蛋白酶的结构比较 Figure 1 Structural comparison of cuticle-degrading proteases[12] 注:a:Ver112 (红色)、PL646 (青色) 和ProK (黄色) 的超折叠结构比较;这3种酶的结构非常相似,除了一些暴露于表面的环结构,如N、C末端 (i),这些结构位于β4与α3之间 (ii),以及α6与α7之间 (iii). b:Ver112 (红色)、PL646 (青色)和ProK (黄色) S1和S4口袋残基的差异,PL646中氨基酸变化的位置已被标记出来,相应的残基以“棒”状结构表现. Note: a: superposition of the structures of Ver112 (magenta), PL646 (cyan), and ProK (yellow); Structures of the three enzymes are essentially identical, except for some surface-exposed loops such as the N and C termini (i), and those located between β4 and α3 (ii), and between α6 and α7 (iii). b: differences in residues in the S1 and S4 pockets between Ver112, PL646, and ProK. Positions involved in amino acid variations are labeled (PL646 numbering); corresponding residues are shown as sticks. |
|
|
在Ver112和PL646等碱性蛋白酶中,有5个保守的半胱氨酸 (Cys),其中4个组成了两个二硫键。在中性蛋白酶中只有一个保守的Cys,不能形成二硫键。因此,源于线虫/昆虫寄生真菌的碱性蛋白酶有两个保守的二硫键,而源于捕食线虫真菌的中性蛋白酶没有二硫键。蛋白质分子中的二硫键一般是起稳定蛋白构象的作用。为了检验碱性蛋白酶中二硫键所起的作用,构建了3个Ver112的突变体,分别打断了一个或同时打断两个二硫键;并对PII、Ver112以及它的3个突变体进行了动力学模拟,结果表明二硫键能够稳定其周围的局部构象;而中性蛋白酶PII不像Ver112具有二硫键,因此需要其它结构因素来稳定蛋白的局部构象。从对模拟的动态轨迹分析中发现,PII具有比Ver112更多的氢键和天然连接数 (number of natural contacts,NNC)。因此氢键和NNC可能是PII等中性蛋白酶中稳定局部构象的主要因素。进一步的研究发现二硫键还能够增加底物结合区S1和S4口袋的柔性,该结果可以解释为什么碱性蛋白酶比中性蛋白酶有更好的底物亲和力和催化活性[13]。梁连铭等还进一步对食线虫真菌丝氨酸蛋白酶的分子表面电势进行了分析,结果表明中性蛋白酶活性中心主要带负电荷,而碱性蛋白酶活性中心主要带正电荷。以前的研究表明线虫体壁带有大量的负电荷,且不易被中和[47]。因此,蛋白酶表面的带电性质可能直接影响到线虫体壁对蛋白酶分子的吸引和催化活性。Ver112和PL646等碱性蛋白酶带有较多的正点荷,因此它们可能更易受到线虫体壁的吸引,导致它们降解体壁的效率比中性蛋白酶更高。
3 食线虫真菌致病相关丝氨酸蛋白酶的进化分析通常,食线虫真菌和昆虫病原真菌产生的致病相关丝氨酸蛋白酶统称为体壁降解蛋白酶,它们都参与到侵染、消化线虫和昆虫体壁的过程[9, 20]。系统进化分析表明,这些不同来源的蛋白酶聚类形成两个分支,捕食线虫真菌产生的丝氨酸蛋白酶聚类形成一个独立的分支;而线虫内寄生真菌和昆虫病原真菌产生的丝氨酸蛋白酶聚在一起形成一个独立的分支[17, 20]。尽管昆虫病原真菌和食线虫真菌存在种属间差异,但昆虫病原真菌和内寄生线虫真菌所分泌的胞外丝氨酸蛋白酶都已经被证实具有降解宿主体壁功能,并且两者具有较高的序列相似性[9, 39-40]。2010年,Li等用昆虫病原真菌和食线虫真菌分别对线虫卵和昆虫卵进行侵染实验,结果显示这两类病原真菌都能侵染线虫和昆虫的体壁[17]。此外,用内寄生线虫真菌淡紫拟青霉 (P.lilacinus) 产生的枯草杆菌素丝氨酸蛋白酶PSP-3进行了生防活性测定实验,结果表明PSP-3能够降解昆虫和线虫体壁[17],说明内寄生线虫真菌和昆虫病原真菌对线虫和昆虫的侵染机制相类似。
最近,李娟等对来源于少孢节丛孢的15条丝氨酸蛋白酶进行了适应性进化分析,结果表明捕食线虫真菌致病相关丝氨酸蛋白酶至少在进化的早期受到了正选择压力的作用[17],据此推测枯草杆菌素丝氨酸蛋白酶基因曾在正选择的作用下快速进化出降解体壁的功能,以帮助捕食线虫真菌更好地完成对线虫的侵染。尽管捕食线虫真菌和线虫内寄生真菌都能产生体壁降解丝氨酸蛋白酶,但是它们却聚类在不同的系统进化分支上[9, 17, 20]。捕食线虫真菌侵染线虫时,菌丝能够特化形成捕食器官帮助真菌固定线虫,同时分泌丝氨酸蛋白酶对线虫体壁的蛋白质进行降解,从而获得营养并帮助真菌侵染线虫[9, 17];线虫内寄生真菌不能产生捕食器官,它们主要依赖于产生的胞外酶 (如丝氨酸蛋白酶) 作为毒力因子去侵染和消化线虫的表皮,因此胞外丝氨酸蛋白酶和捕食器官的协同作用可能使捕食线虫真菌能更快地固定和侵染线虫。此外,在捕食线虫真菌的进化过程中,丝氨酸蛋白酶与捕食器官的协同进化使得丝氨酸蛋白酶经历特殊的选择压力[17, 48]。通过Branch-site model的方法,从捕食线虫真菌丝氨酸蛋白酶序列中检测到了许多受到正选择作用的氨基酸位点,这些氨基酸位点对蛋白酶功能的分化可能起到了重要作用;同时,这部分受到正选择的氨基酸位点可能是导致蛋白酶自身底物特异性改变的关键位点[17, 48]。综上,正选择压力的作用使捕食线虫真菌丝氨酸蛋白酶进化为毒力因子,具备了降解线虫体壁的能力,帮助捕食线虫真菌更好地适应环境。
4 展望食线虫真菌在陆地和水生生态系统中普遍存在,它们对于维持自然生态环境中线虫种群数量的动态平衡起着非常重要的作用,同时作为对抗植物病原线虫的生防制剂,具有重要的生态和经济价值[1, 4]。高效生防制剂的开发依赖于对微生物侵染线虫的分子机理的了解。食线虫真菌产生的胞外丝氨酸蛋白酶是真菌侵染线虫的重要毒力因子[4, 9, 20],通过对食线虫真菌丝氨酸蛋白酶的性质、功能、晶体结构和作用机制等方面的系统研究,不仅可为开发高效的线虫生防制剂奠定良好的基础,同时也可以拓展丝氨酸蛋白酶在工业领域中的应用。
近年来,越来越多的致病相关丝氨酸蛋白酶在食线虫真菌中被分离和鉴定,同时对于丝氨酸蛋白酶的晶体结构和催化机制的研究也取得了较大的进展[9, 12-13, 20]。通过对食线虫真菌丝氨酸蛋白酶的性质和功能比较可以发现这些蛋白酶按照生物化学性质可以分为两类,一类是来源于捕食线虫真菌的中性丝氨酸蛋白酶,另一类是来源于内寄生真菌的碱性丝氨酸蛋白酶,这两类蛋白酶分别聚类在不同的进化分支[17, 20]。同时,通过对食线虫真菌丝氨酸蛋白酶的分子表面电势分析,可以看到这两类蛋白酶活性中心的分子表面电势呈现酸性到碱性逐渐过渡的现象;而对食线虫真菌丝氨酸蛋白酶的晶体结构比较,发现它们的结构和ProK的结构很相近,主要的差别在于表面的一些环区以及底物结合区的结合口袋 (如S1和S4)[12]。进一步的分析发现存在于碱性丝氨酸蛋白酶中的二硫键不仅能够稳定蛋白酶的局部构象,同时也能够增强底物结合区底物结合口袋的柔性;而中性丝氨酸蛋白酶分子中不存在二硫键,它们主要通过氢键和NNC来维持蛋白酶的局部构象,增强蛋白酶分子结构的稳定性[13, 47]。以上的这些重要的生物化学性质和结构特性对于丝氨酸蛋白酶的结构改造和筛选高毒力菌株具有非常重要的理论指导意义。
目前,已经有3株捕食线虫真菌和一株内寄生真菌的基因组被测序,分析结果表明在这些真菌的基因组中存在数量较多的丝氨酸蛋白酶,如少孢节丛孢的基因组中存在24种丝氨酸蛋白酶,其中有13种蛋白酶独立聚类形成一个分支[41, 48]。通过对这些蛋白酶的功能研究,人们将能更好地理解食线虫真菌侵染线虫的分子机制,为高效线虫生防制剂的开发提供理论基础,对病原线虫的生物防控也具有重要的理论和实践意义。
| [1] | Zuckerman BM, Rohde RA. Plant parasitic nematodes[M]. Amsterdam: Elsevier, 2012 . |
| [2] | Abad P, Gouzy J, Aury JM, et al. Genome sequence of the metazoan plant-parasitic nematode Meloidogyne incognita[J]. Nature Biotechnology, 2008, 26(8) : 909–915. DOI:10.1038/nbt.1482 |
| [3] | Kerry BR. Rhizosphere interactions and the exploitation of microbial agents for the biological control of plant-parasitic nematodes[J]. Annual Review of Phytopathology, 2000, 38(4) : 423–441. |
| [4] | Nordbring-Hertz B, Jansson HB, Tunlid A. Nematophagous fungi[A]//Encyclopedia of Life Science[M]. Basingtoke: Macmillam Publishers Ltd, 2011 |
| [5] | Zhang Y, Li GH, Zhang KQ. A review on the research of nematophagous fungal species[J]. Mycosystema, 2011, 30 : 836–845. |
| [6] | Tunlid A, Jansson S. Proteases and their involvement in the infection and immobilization of nematodes by the nematophagous fungus Arthrobotrys oligospora[J]. Applied and Environmental Microbiology, 1991, 57(10) : 928–935. |
| [7] | Cruz DG, Costa LM, Rocha LO, et al. Serine proteases activity is important for the interaction of nematophagous fungus Duddingtonia flagrans with infective larvae of trichostrongylides and free-living nematodes Panagrellus spp[J]. Fungal Biology, 2015, 119(8) : 672–678. DOI:10.1016/j.funbio.2015.03.005 |
| [8] | Cox GN, Kusch M, Edgar RS. Cuticle of Caenorhabditis elegans: its isolation and partial characterization[J]. The Journal of Cell Biology, 1981, 90(1) : 7–17. DOI:10.1083/jcb.90.1.7 |
| [9] | Yang JK, Tian BY, Liang LM, et al. Extracellular enzymes and the pathogenesis of nematophagous fungi[J]. Applied Microbiology and Biotechnology, 2007, 75(1) : 21–31. DOI:10.1007/s00253-007-0881-4 |
| [10] | Lopez-Llorca LV. Purification and properties of extracellular proteases produced by the nematophagous fungus Verticillium suchlasporium[J]. Canadian Journal of Microbiology, 1990, 36(8) : 530–537. DOI:10.1139/m90-093 |
| [11] | Yang JK, Liang LM, Zhang Y, et al. Purification and cloning of a novel serine protease from the nematode-trapping fungus Dactylellina varietas and its potential roles in infection against nematodes[J]. Applied Microbiology and Biotechnology, 2007, 75(3) : 557–565. DOI:10.1007/s00253-007-0839-6 |
| [12] | Liang LM, Meng ZH, Ye FP, et al. The crystal structures of two cuticle-degrading proteases from nematophagous fungi and their contribution to infection against nematodes[J]. The FASEB Journal, 2010, 24(5) : 1391–1400. DOI:10.1096/fj.09-136408 |
| [13] | Liang LM, Liu SQ, Yang JK, et al. Comparison of homology models and crystal structures of cuticle-degrading proteases from nematophagous fungi: structural basis of nematicidal activity[J]. The FASEB Journal, 2011, 25(6) : 1894–1902. DOI:10.1096/fj.10-175653 |
| [14] | Siezen RJ, Leunissen JAM. Subtilases: the superfamily of subtilisin-like serine proteases[J]. Protein Science, 1997, 6(3) : 501–523. |
| [15] | Dobinson KF, Lecomte N, Lazarovits G. Production of an extracellular trypsin-like protease by the fungal plant pathogen Verticillium dahliae[J]. Canadian Journal of Microbiology, 1997, 43(3) : 227–233. DOI:10.1139/m97-031 |
| [16] | St Leger RJ, Joshi L, Roberts D. Ambient pH is a major determinant in the expression of cuticle-degrading enzymes and hydrophobin by Metarhizium anisopliae[J]. Applied and Environmental Microbiology, 1998, 64(2) : 709–713. |
| [17] | Li J, Yu L, Yang JK, et al. New insights into the evolution of subtilisin-like serine protease genes in Pezizomycotina[J]. BMC Evolutionary Biology, 2010, 10(5) : 1–14. |
| [18] | Tunlid A, Rosen S, Ek B, et al. Purification and characterization of an extracellular serine protease from the nematode-trapping fungus Arthrobotrys oligospora[J]. Microbiology, 1994, 140(7) : 1687–1695. DOI:10.1099/13500872-140-7-1687 |
| [19] | Segers R, Butt TM, Kerry BR, et al. The nematophagous fungus Verticillium chlamydosporium produces a chymoelastase like protease which hydrolyses host nematode proteins in situ[J]. Microbiology, 1994, 140(10) : 2715–2723. DOI:10.1099/00221287-140-10-2715 |
| [20] | Yang JK, Liang LM, Li J, et al. Nematicidal enzymes from microorganisms and their applications[J]. Applied Microbiology and Biotechnology, 2013, 97(16) : 7081–7095. DOI:10.1007/s00253-013-5045-0 |
| [21] | Zhao ML, Mo MH, Zhang KQ. Characterization of a neutral serine protease and its full-length cDNA from the nematode-trapping fungus Arthrobotrys oligospora[J]. Mycologia, 2004, 96(1) : 16–22. DOI:10.1080/15572536.2005.11832991 |
| [22] | Wang JW, Meng QL, Qiao J. The recombinant serine protease XAoz1 of Arthrobotrys oligospora exhibits potent nematicidal activity against Caenorhabditis elegans and Haemonchus contortus[J]. FEMS Microbiology Letters, 2013, 344(1) : 53–59. DOI:10.1111/femsle.2013.344.issue-1 |
| [23] | Zhao HL, Qiao J, Meng QL, et al. Expression of serine proteinase P186 of Arthrobotrys oligospora and analysis of its nematode-degrading activity[J]. Antonie Van Leeuwenhoek, 2015, 108(6) : 1485–1494. DOI:10.1007/s10482-015-0595-z |
| [24] | Yang JK, Li J, Liang LM, et al. Cloning and characterization of an extracellular serine protease from the nematode-trapping fungus Arthrobotrys conoides[J]. Archives of Microbiology, 2007, 188(2) : 167–174. DOI:10.1007/s00203-007-0233-x |
| [25] | Tzean Y, Chou TH, Hsiao CC, et al. Cloning and characterization of cuticle-degrading serine protease from nematode-trapping fungus Arthrobotrys musiformis[J]. Mycoscience, 2016, 57(2) : 136–143. DOI:10.1016/j.myc.2015.12.003 |
| [26] | Wang M, Yang JK, Zhang KQ. Characterization of an extracellular protease and its cDNA from the nematode-trapping fungus Monacrosporium microscaphoides[J]. Canadian Journal of Microbiology, 2006, 52(2) : 130–139. DOI:10.1139/w05-110 |
| [27] | Yang JK, Ye FP, Mi QL, et al. Purification and cloning of an extracellular serine protease from the nematode-trapping fungus Monacrosporium cystosporium[J]. Journal of Microbiology and Biotechnology, 2008, 18(5) : 852–858. |
| [28] | Soares FE, Braga FR, Araújo JV, et al. In vitro activity of a serine protease from Monacrosporium thaumasium fungus against first-stage larvae of Angiostrongylus vasorum[J]. Parasitology Research, 2012, 110(6) : 2423–2427. DOI:10.1007/s00436-011-2781-x |
| [29] | Wang RB, Yang JK, Lin C, et al. Purification and characterization of an extracellular serine protease from the nematode-trapping fungus Dactylella shizishanna[J]. Letters in Applied Microbiology, 2006, 42(6) : 589–594. |
| [30] | Yu HY, Xue W, Duan JX. Expression of PrD1 of Dactylellina cionopaga in Aspergillus niger[J]. Biotechnology, 2012, 22(3) : 38–43. (in Chinese) 于寒颖, 薛伟, 段俊欣. 黑曲霉外源表达系统对Dactylellina cionopaga基因PrD1的表达[J]. 生物技术, 2012, 22(3) : 38–43. |
| [31] | Braga FR, Araújo JV, Soares FEF, et al. An extracellular serine protease of an isolate of Duddingtonia flagrans nematophagous fungus[J]. Biocontrol Science and Technology, 2012, 22(10) : 1131–1142. DOI:10.1080/09583157.2012.713912 |
| [32] | Bonants PJM, Fitters PFL, Thijs H, et al. A basic serine protease from Paecilomyces lilacinus with biological activity against Meloidogyne hapla eggs[J]. Microbiology, 1995, 141(4) : 775–784. DOI:10.1099/13500872-141-4-775 |
| [33] | Yang JK, Huang XW, Tian BY, et al. Isolation and characterization of a serine protease from the nematophagous fungus, Lecanicillium psalliotae, displaying nematicidal activity[J]. Biotechnology Letters, 2005, 27(15) : 1123–1128. DOI:10.1007/s10529-005-8461-0 |
| [34] | Li J, Yang JK, Huang XW, et al. Purification and characterization of an extracellular serine protease from Clonostachys rosea and its potential as a pathogenic factor[J]. Process Biochemistry, 2006, 41(4) : 925–929. DOI:10.1016/j.procbio.2005.10.006 |
| [35] | Zhang YJ, Liu XZ, Wang M. Cloning, expression, and characterization of two novel cuticle-degrading serine proteases from the entomopathogenic fungus Cordyceps sinensis[J]. Research in Microbiology, 2008, 159(6) : 462–469. DOI:10.1016/j.resmic.2008.04.004 |
| [36] | Wang B, Wu WP, Liu XZ. Purification and characterization of a neutral serine protease with nematicidal activity from Hirsutella rhossiliensis[J]. Mycopathologia, 2007, 163(3) : 169–176. DOI:10.1007/s11046-007-0100-y |
| [37] | Wang B, Liu X, Wu WP, et al. Purification, characterization and gene cloning of an alkaline serine protease from a highly virulent strain of the nematode-endoparasitic fungus Hirsutella rhossiliensis[J]. Microbiological Research, 2009, 164(6) : 665–673. DOI:10.1016/j.micres.2009.01.003 |
| [38] | Niu XM, Zhang KQ. Arthrobotrys oligospora: a model organism for understanding the interaction between fungi and nematodes[J]. Mycology, 2011, 2(2) : 60–78. |
| [39] | hman J, Ek B, Rask L, et al. Sequence analysis and regulation of a gene encoding a cuticle-degrading serine protease from the nematophagous fungus Arthrobotrys oligospora[J]. Microbiology, 1996, 142(7) : 1605–1616. DOI:10.1099/13500872-142-7-1605 |
| [40] | hman J, Johansson T, Olsson M, et al. Improving the pathogenicity of a nematode-trapping fungus by genetic engineering of a subtilisin with nematotoxic activity[J]. Applied and Environmental Microbiology, 2002, 68(7) : 3408–3415. DOI:10.1128/AEM.68.7.3408-3415.2002 |
| [41] | Yang JK, Wang L, Ji XL, et al. Genomic and proteomic analyses of the fungus Arthrobotrys oligospora provide insights into nematode-trap formation[J]. PLoS Pathogens, 2011, 7(9) : e1002179. DOI:10.1371/journal.ppat.1002179 |
| [42] | Chen LL, Liu LJ, Shi M, et al. Characterization and gene cloning of a novel serine protease with nematicidal activity from Trichoderma pseudokoningii SMF2[J]. FEMS Microbiology Letters, 2009, 299(2) : 135–142. DOI:10.1111/fml.2009.299.issue-2 |
| [43] | Zou CG, Tu HH, Liu XY, et al. PacC in the nematophagous fungus Clonostachys rosea controls virulence to nematodes[J]. Environmental Microbiology, 2010, 12(7) : 1868–1877. DOI:10.1111/j.1462-2920.2010.02191.x |
| [44] | Zou CG, Tao N, Liu WJ, et al. Regulation of subtilisin-like protease prC expression by nematode cuticle in the nematophagous fungus Clonostachys rosea[J]. Environmental Microbiology, 2010, 12(12) : 3243–3252. DOI:10.1111/emi.2010.12.issue-12 |
| [45] | Zou CG, Xu YF, Liu WJ, et al. Expression of a serine protease gene prC is up-regulated by oxidative stress in the fungus Clonostachys rosea: implications for fungal survival[J]. PLoS One, 2010, 5(10) : e13386. DOI:10.1371/journal.pone.0013386 |
| [46] | Lott ME, Sheehan DJ, Davis LS. Paecilomyces lilacinus infection with a sporotrichoid pattern in a renal transplant patient[J]. Journal of Drugs in Dermatology, 2007, 6(4) : 436–439. |
| [47] | Liu SQ, Liang LM, Tao Y, et al. Structural and dynamic basis of serine proteases from nematophagous fungi for cuticle degradation, pesticides[A]//Pesticides in the modern world-Pests control and pesticides exposure and toxicity assessment[M]. Stoytcheva M. Croatia: Intech, 2011: 333-346 |
| [48] | Li J. Molecular evolution of fungal subtilase superfamily[D]. Kunming: Doctoral Dissertation of Yunnan University, 2010 李娟. 真菌枯草杆菌素丝氨酸蛋白酶超家族分子进化研究[D]. 昆明: 云南大学博士学位论文, 2010 |
 2017, Vol. 44
2017, Vol. 44