扩展功能
文章信息
- 张真, 李晔, 张西轩, 胡双艳, 阮海华
- ZHANG Zhen, LI Ye, ZHANG Xi-Xuan, HU Shuang-Yan, RUAN Hai-Hua
- 蜡样芽孢杆菌胶原酶基因colR75E在毕赤酵母中的重组表达
- Recombinant expression of Bacillus cereus collagenase colR75E in Pichia pastoris
- 微生物学通报, 2017, 44(4): 807-817
- Microbiology China, 2017, 44(4): 807-817
- DOI: 10.13344/j.microbiol.china.160354
-
文章历史
- 收稿日期: 2016-05-03
- 接受日期: 2016-06-03
- 优先数字出版日期(www.cnki.net): 2016-06-28
胶原酶 (Collagenase) 指的是在生理pH与温度条件下特异性降解胶原纤维的一类蛋白酶[1-2]。目前已知的胶原酶按照来源可分为微生物胶原酶和动物胶原酶两大类,主要应用于食品与饲料的加工、化妆品以及药物的合成与加工领域。其中,微生物胶原酶以底物范围广、酶切位点多、生产成本低的优点得到了更加广泛的应用[3]。例如,在食品工艺中微生物胶原酶可作为肉类嫩化剂[4];在医疗中微生物胶原酶可作为骨质增生类疾病的治疗药物等[5]。目前,溶组织梭状芽孢杆菌 (Clostridium histolyticum) 产的胶原酶G (Collagenase G,ColG) 应用最广泛,研究背景最清楚[6]。但这些胶原酶产品多为梭状芽孢杆菌培养物直接冻干而得,其成分复杂、纯度普遍较低,限制了胶原酶产品的应用范围。因此,获得高纯度的胶原酶将具有重要的理论及工业应用价值。
Nagano等[7]在2000年建立了一种利用四步柱层析法从枯草芽孢杆菌FS-2 (Bacillus subtilis FS-2) 中纯化胶原酶的方法,但这种方法工艺复杂,且最终得到的胶原酶比活力只有0.71 U/mg,比活力偏低。本实验室在前期研究中筛选到一株产胶原酶的蜡样芽孢杆菌Bacillus cereus R75E,克隆到编码该胶原酶蛋白的基因colR75E (GenBank登录号:KP987200)[8],序列全长2 898 bp,其与胶原酶ColG具有69%的一致性,同属于Ⅰ型胶原酶[8-9],因其具有良好的温度及酸碱稳定性而具有很强的工业应用价值[9]。李晔等[10]建立了从该菌中获得高纯度胶原酶ColR75E的方法,虽然该方法较Nagano等[7]的方法简化了步骤,但依旧需要采用柱层析的方法,其配套的层析设备复杂而昂贵[11],不适合大规模工业生产;张西轩等[9]将ColR75E在大肠杆菌中表达并获得比活力较高的胶原酶产品。但是,利用大肠杆菌原核表达系统制备外源蛋白的过程中存在毒力蛋白、内毒素等对人体健康存在威胁的物质;同时,表达所得的外源蛋白易形成包涵体,不仅给后续的纯化带来困难,还会造成胶原酶的失活,限制了大肠杆菌所产胶原酶在食品以及药品领域的应用。基于此,本文采用巴斯德毕赤酵母 (Pichia pastoris) 表达系统表达生产胶原酶ColR75E,该表达系统具有继代稳定、可以高密度发酵、在发酵过程中无毒素分泌、自身分泌的杂蛋白少、外源蛋白表达量高以及分泌型表达等优点,而且有利于产物的纯化,非常适宜扩大为工业规模[12-15]。为获得能够直接应用于食品工业等领域的无毒性胶原酶ColR75E的生产提供材料基础与理论依据。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒: 蜡样芽胞杆菌R75E (Bacillus cereus R75E) 由本实验室分离、鉴定并保藏于中国微生物菌种保藏管理委员会普通微生物中心 (简称CGMCC),保藏编号为CGMCC 8614。感受态大肠杆菌Trans10及毕赤酵母X-33菌株均购自美国Promega公司;原核表达载体pET28a及真核表达载体pPICZαA均购自德国Novagen公司。 1.1.2 主要试剂和仪器: 博来霉素 (Zeocin),美国Invitrogen公司;KOD DNA聚合酶、dNTPs、T4 DNA连接酶、限制性内切酶Dpn Ⅰ等,大连TaKaRa公司;DNA回收试剂盒、质粒提取试剂盒、酵母基因组提取试剂盒、DNA marker,天根生化科技 (北京) 有限公司;蛋白预染Marker,立陶宛Fermentas公司;牛跟腱来源的Ⅰ型胶原蛋白、牛血清白蛋白,美国Sigma-Aldrich公司。PCR仪、电转化仪,美国伯乐公司;超净工作台、生化培养箱,哈尔滨东联电子;紫外分光光度仪,日本岛津公司。
引物合成以及DNA测序由上海生工生物工程有限公司完成。LB、YPD、BMGY、BMMY培养基均按照美国Invitrogen公司操作手册推荐方法自行配制。其中,BMGY培养基:1%酵母浸出物,2%蛋白胨,100 mmol/L磷酸钾 (pH 6.0),1.34%无氨基酵母氮源,4×10-5%生物素,1%甘油;BMMY培养基:将BMGY培养基中1%甘油替换为1%无水甲醇。
1.2 方法 1.2.1 PCR扩增colR75E基因: 用SignalP 4.0 Server软件分析colR75E胶原酶基因 (GenBank登录号:KP987200) 的DNA全序列,去除colR75E基因N端信号肽序列 (1-30位氨基酸),设计colR75E-Xho Ⅰ-F和colR75E-Not Ⅰ-R引物对,序列见表 1。其中下划线所示部分为Xho Ⅰ和Not Ⅰ的酶切位点,上游引物中斜体的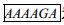
| 引物名称 Primer name |
引物序列 Primer sequences (5′→3′) |
| colR75E-Xho Ⅰ-F | CGC CTCGAG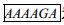 GAAGAAAAAGTACCGTATAACGTG
GAAGAAAAAGTACCGTATAACGTG |
| colR75E-Not Ⅰ-R | CGCGCGGCCGCTCATTTTACTGACAATTCGTATGTTCC |
| 1267-F | GTAAAGGCACAGTATCACCG |
| 2013-R | TCATAGGCAACGTATCACTA |
(1) 诱导时间对重组胶原酶表达的影响:重组酵母菌转入BMMY (pH 6.0) 诱导培养后,每24 h追加终浓度1%甲醇 (体积比),分别于诱导12、24、36、48、60、72、96 h后,各取1 mL样品,室温12 000 r/min离心3 min收集表达上清,测定胶原酶活力。
(2) 甲醇浓度对重组胶原酶表达的影响:重组酵母菌诱导24 h后,按0.5%、1.0%、1.5%、2.0%、2.5%的终浓度追加甲醇 (体积比),于72 h后室温12 000 r/min离心3 min收集表达上清,测定胶原酶活力。
(3) pH对重组胶原酶表达的影响:用H3PO4或KOH调节磷酸盐缓冲液的pH,将BMMY培养液的pH分别调节为5.5、6.0、6.5、7.0、7.5和8.0。重组酵母菌在不同pH的BMMY培养液中表达72 h后,室温12 000 r/min离心3 min收集表达上清,测定胶原酶活力。
1.2.6 重组胶原酶蛋白的纯化: 利用优化后的诱导条件进行胶原酶的诱导表达,将所得胶原酶液中加入硫酸铵粉末至饱和度为80%,在0条件下沉淀过夜,次日12 000 r/min离心20 min后用20 mmol/L PBS (含500 mmol/L NaCl,1 mmol/L PMSF,pH 7.4) 缓冲液重悬细胞沉淀,在冰水浴中超声破碎,12 000 r/min离心30 min后取上清,脱盐处理后进行His标签亲和层析Ni-NTA纯化,以备测定纯化后的胶原酶的活力。 1.2.7 重组胶原酶SDS-PAGE检测及胶原酶谱检测: 将得到的胶原酶液分别采用SDS-PAGE电泳及胶原酶谱法检测目标胶原酶蛋白[16]。其中胶原酶谱法是在明胶酶谱法的基础上改进的,即在聚丙烯酰胺凝胶中加入0.1%的Ⅰ型胶原蛋白,电泳结束后,含有目标胶原酶的凝胶经过复性、染色、脱色,目标胶原酶呈现负染条带[17]。 1.2.8 胶原酶的活力测定: 胶原酶活力测定采用通用的Mandl测定方法[18]。该方法中将1个活力单位 (U) 定义为:每毫升胶原酶与牛跟腱来源的Ⅰ型胶原蛋白在37、pH 7.4的条件下反应,在5 h内每生成1 mol游离氨基酸即对应1个活力单位 (1 U)。蛋白浓度测定采用考马斯亮蓝法,以牛血清白蛋白为标准品。 1.2.9 重组胶原酶底物特异性研究: 为了分析重组胶原酶对Ⅰ型胶原蛋白的降解作用,采用胃蛋白酶提取法提取草鱼鱼鳞中的Ⅰ型胶原蛋白[19],按照40:1的体积比将提取得到的Ⅰ型胶原蛋白溶液 (1 g/L) 与ColR75E酶液 (62.14 mg/L) 混合,置于37条件下进行反应,并在不同的时间点取样。将取得的样品进行浓度为10%的SDS-PAGE电泳检测,观察草鱼鱼鳞Ⅰ型胶原蛋白的降解情况。在此基础上进行重组胶原酶底物特异性分析,在37条件下将纯化后的胶原酶分别与胶原蛋白、酪蛋白、牛血清蛋白和溶菌酶进行反应,同时设置阴性对照,待反应结束后取样进行SDS-PAGE电泳检测底物蛋白的降解情况。
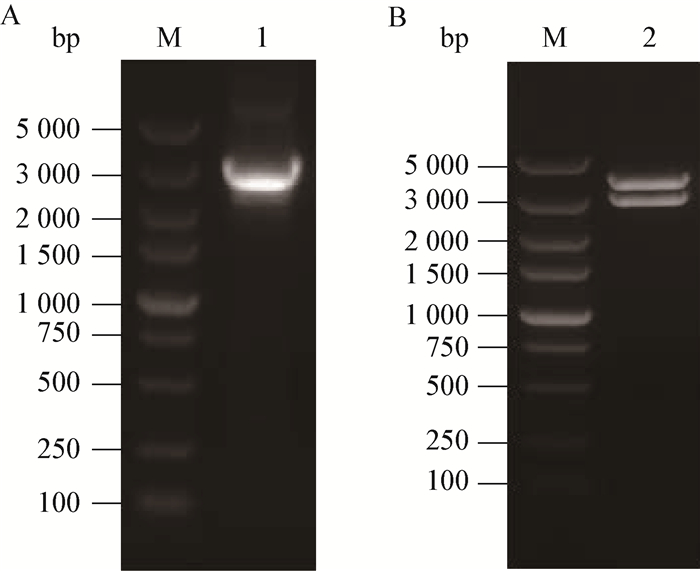
2 结果与分析 2.1 毕赤酵母表达载体的构建以蜡样芽孢杆菌R75E基因组gDNA为模板,经PCR扩增得到分子量为2 800 bp左右的DNA片段,如图 1A所示,该片段大小与预期的colR75E基因片段 (2 831 bp) 相吻合,表明PCR扩增成功。将该PCR片段酶切连接后获得转化子,挑取转化子提取质粒并进行酶切鉴定。结果如图 1B所示:转化子质粒经Xho Ⅰ和Not Ⅰ双酶切后得到两条条带,一条为pPICZαA质粒片段,其分子量为3.6 kb;另一条与目的基因colR75E片段大小 (2 831 bp) 相符,获得阳性重组质粒pPICZαA/colR75E。将该质粒进行测序验证后显示colR75E基因序列正确。
|
| 图 1 colR75E基因PCR扩增产物电泳图 (A) 及重组质粒pPICZαA/colR75E双酶切鉴定图 (B) Figure 1 Agarose gel electrophosis of Bacillus cereus colR75E PCR products (A) and recombinant pPICZαA/colR75E plasmid cut with Xho Ⅰ and Not Ⅰ (B) 注:M:DNA分子量标准;1:PCR产物;2:重组质粒双酶切产物. M: DNA marker; 1: PCR products; 2: Products of recombinant pPICZαA/colR75E plasmid cut with Xho Ⅰ and Not Ⅰ. |
|
|
用SacⅠ对获得的重组质粒pPICZαA/colR75E进行线性化处理,电转化至毕赤酵母X-33感受态细胞,用含100 mg/L Zeocin的YPD培养基筛选阳性转化子。随机挑取6个阳性转化子提取基因组gDNA。分别以gDNA作为模板,利用colR75E特异性引物1267-F和2013-R (表 1) 进行PCR鉴定。若重组质粒pPICZαA/colR75E整合至酵母基因组上,理论上应该扩增出分子量大小约为750 bp的DNA片段;若整合失败,则无法扩增出750 bp的DNA片段。从图 2结果可以看出,挑取的6个阳性转化子均扩增出约750 bp的片段,与预期的片段大小相符。表明线性化的pPICZαA/colR75E整合至毕赤酵母X-33菌株的基因组中。

|
| 图 2 线性化pPICZαA/colR75E质粒片段整合至毕赤酵母X-33基因组的PCR鉴定 Figure 2 PCR identification of linear pPICZαA/colR75E integration into Pichia pastoris X-33 genomic DNA |
|
|
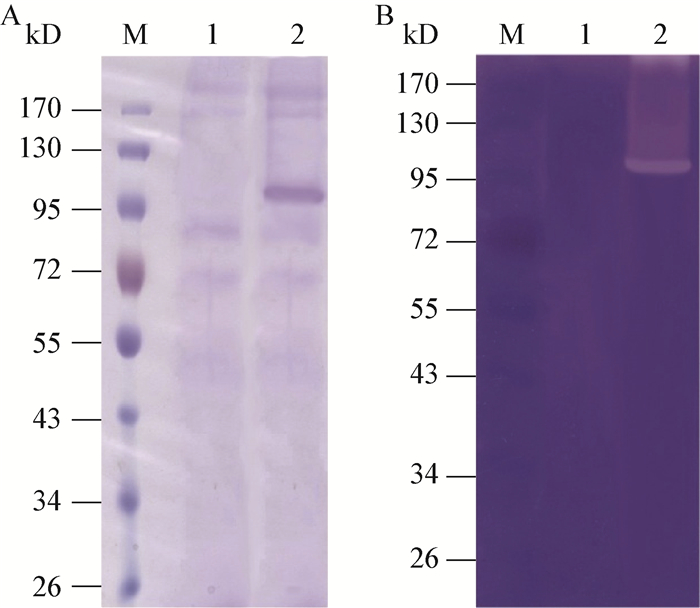
|
| 图 3 重组胶原酶ColR75E表达的SDS-PAGE (A) 以及胶原酶谱 (B) 分析 Figure 3 Determination of expressed ColR75E collagenase by SDS-PAGE (A) and zymography (B) 注:M:蛋白质分子量标准;1:未经甲醇诱导的酶液;2:经甲醇诱导的酶液. M: Protein marker; 1: Precipitated protein from supernatant of Pichia pastoris X-33 fermentation without induction by methanol; 2: ColR75E collagenase containing precipitated protein from supernatant of Pichia pastoris X-33 induced by methanol. |
|
|
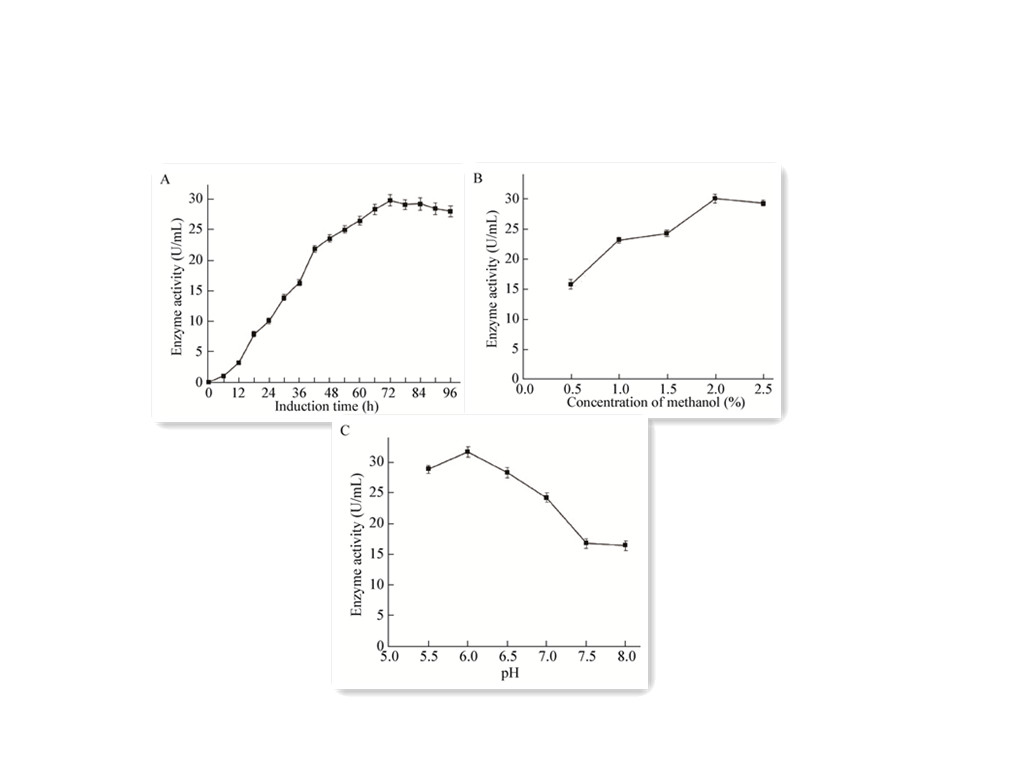
|
| 图 4 诱导时间 (A)、甲醇浓度 (B) 和pH (C) 对重组胶原酶ColR75E表达的影响 Figure 4 Effects of induction time (A), methanol concentration (B) and pH (C) on the expression of recombinant ColR75E in Pichia pastoris X-33 |
|
|
| 试验号 Number |
A诱导时间 A Induced time (h) |
B甲醇浓度 B Concentration of methanol (%) |
C pH | D空列 D Vacant column |
酶活力 Enzyme activity (U/mL) |
| 1 | 1(58) | 1(1.5) | 1(5.5) | 1 | 16.04 |
| 2 | 1 | 2(2.0) | 2(6.0) | 2 | 17.71 |
| 3 | 1 | 3(2.5) | 3(6.5) | 3 | 15.21 |
| 4 | 2(72) | 1 | 2 | 3 | 28.87 |
| 5 | 2 | 2 | 3 | 1 | 20.53 |
| 6 | 2 | 3 | 1 | 2 | 24.76 |
| 7 | 3(96) | 1 | 3 | 2 | 18.86 |
| 8 | 3 | 2 | 1 | 3 | 22.20 |
| 9 | 3 | 3 | 2 | 1 | 25.15 |
| K1 | 16.32 | 21.26 | 21.00 | 20.57 | T=252.42 |
| K2 | 24.72 | 20.14 | 23.91 | 20.44 | |
| K3 | 22.07 | 21.71 | 18.20 | 22.09 | |
| R | 8.40 | 1.56 | 5.71 | 1.65 |
表达条件的正交试验:采用L9(34) 正交试验研究诱导条件对重组酵母产胶原酶的影响,具体试验设计及结果见表 2。
由表 2可知,各因素影响重组酵母表达胶原酶酶的主次顺序为:A > C > B。其最佳诱导条件为A2B3C2,即诱导时间为72 h,甲醇终浓度为2.5%,pH值为6.0,按该条件重复发酵后所得粗酶液的活力值提高至29.79 U/mL,优化效果显著。
2.4 重组胶原酶蛋白的纯化及检测将最优条件下诱导的毕赤酵母重组胶原酶按照1.2.6方法分离纯化后进行SDS-PAGE电泳,结果如图 5A所示:凝胶上可观察到高纯度的ColR75E胶原酶蛋白条带,表明含有His标签的重组ColR75E胶原酶蛋白纯化成功。进一步对得到的重组胶原酶进行胶原酶谱检测,结果如图 5B所示,可在含有Ⅰ型胶原蛋白的凝胶上观察到由于重组胶原酶对底物的降解而产生的透明的负染条带 (泳道1-7),表明经Ni-NTA镍柱纯化得到的蛋白确为重组ColR75E胶原蛋白酶。将所得重组蛋白集中在一起进行透析去除洗脱液中的咪唑等盐类,用所得的纯化产物测定胶原酶活力,其比活力高达4.977 U/mg,与纯化前的1.037 U/mg相比,比活力提高了近4.8倍。
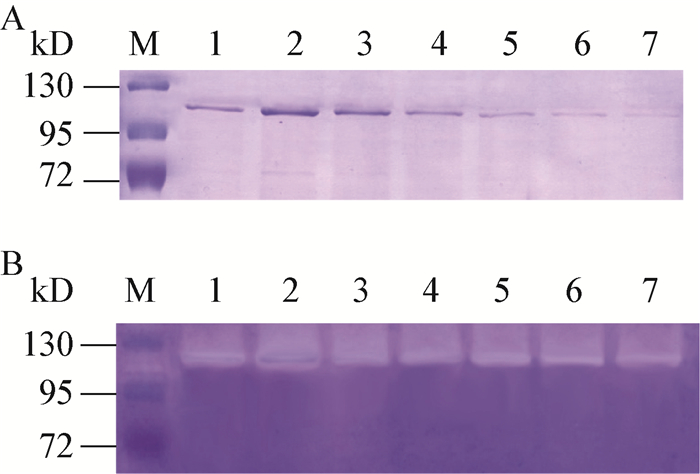
|
| 图 5 毕赤酵母重组ColR75E胶原酶纯化后的SDS-PAGE电泳 (A) 以及胶原酶谱检测结果 (B) Figure 5 Analysis of SDS-PAGE (A) and collagen zymography (B) of purified recombinant ColR75E collagenase in Pichia pastoris X-33 |
|
|
利用草鱼鱼鳞Ⅰ型胶原蛋白作为底物,通过SDS-PAGE电泳检测酵母菌重组ColR75E胶原酶的降解能力。结果如图 6A所示,泳道1为草鱼鱼鳞Ⅰ型胶原蛋白对照,在该泳道中可以清晰地观察到Ⅰ型胶原蛋白3种肽链:α1链 (分子量为126 kD)、α2链 (分子量为116 kD) 以及一条高分子量的β链 (α链的二聚体);泳道2-6分别为重组ColR75E胶原酶与草鱼鱼鳞Ⅰ型胶原蛋白反应2、4、6、8、10 h后的降解产物。结果表明随着反应时间的逐渐增加,草鱼鱼鳞Ⅰ型胶原蛋白的α1、α2、β链均逐渐减少,而分子量较小的水解肽段的比例逐渐增加;当反应进行至10 h时,Ⅰ型胶原蛋白中的α1、α2、β链几乎完全被降解,而且肽段也被进一步水解为分子量更小、难于在该胶上显示的肽段。研究表明,从酵母菌中诱导表达的重组ColR75E胶原酶能够将从鱼鳞中提取的具有天然构象的Ⅰ型胶原纤维完全降解为短链肽段。
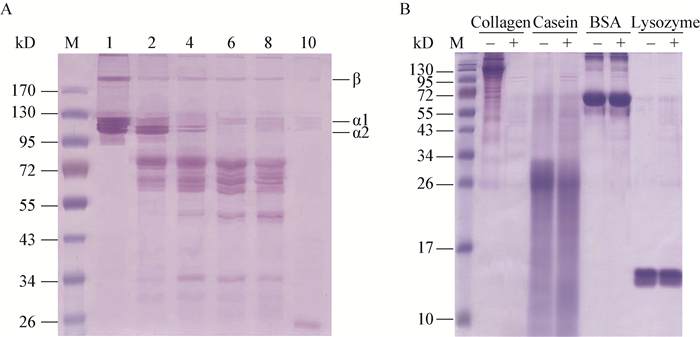
|
| 图 6 重组胶原酶ColR75E对草鱼鱼鳞中Ⅰ型胶原蛋白水解的时间进程检测 (A) 及底物特异性分析 (B) Figure 6 Hydrolysis phase of type Ⅰ collagen extracted from grass carp by recombinant collagenase ColR75E with incubation time (A) and analysis of substrate specificity of recombinant collagenase ColR75E (B) |
|
|
进一步以胶原蛋白、酪蛋白、牛血清蛋白和溶菌酶作底物,对纯化后的胶原酶进行底物特异性研究,结果如图 6B所示,胶原蛋白与胶原酶反应10 h后几乎完全被降解,而胶原酶对其他3种蛋白均未发生明显的降解作用,与不加胶原酶的对照组相比基本无明显变化,表明重组胶原酶对于胶原蛋白有特异性降解作用;同时,也表明通过硫酸铵沉淀、脱盐以及亲和层析后,获得的重组胶原酶纯度高,没有其他蛋白酶的污染。
3 结论与讨论在本研究的前期工作中证实,胶原酶ColR75E与目前应用最为广泛的溶组织梭状芽孢杆菌胶原酶ColG编码基因有69%的相似性[6, 8],表现出优良的pH稳定性和热稳定性,具有很强的工业应用价值[9],因此,胶原酶ColR75E可以用来开发新型胶原酶产品。在此背景之下,我们首次尝试采用毕赤酵母 (Pichia pastoris) 表达系统来表达胶原酶ColR75E,并对表达条件进行优化,得出重组酵母在pH 6.0的条件下,每24 h添加甲醇至终浓度为2.5%,诱导72 h可获得胶原酶ColR75E的高效表达。
进一步通过SDS-PAGE和胶原酶谱分析发现毕赤酵母分泌表达的胶原酶纯度高、生物活性高,利用硫酸铵沉淀及亲和层析得到了高纯度的蛋白,其比活力可达到4.977 U/mg。与市场上商品化的梭状芽胞杆菌Ⅰ型胶原酶标准品 (Sigma公司,货号:c0130) 的0.417 U/mg的比活力值高出11.9倍。比活力提高的原因主要是梭状芽胞杆菌Ⅰ型胶原酶标准品是未经过分离纯化的粗制品,而本研究中的重组胶原酶经过纯化去除了大量的杂蛋白,酶的纯度提高,进而比活力显著提高。同时,与本实验室从原核表达系统大肠杆菌中制备的重组胶原酶比活力3.62 U/mg相比,提高了近37.5%。由于无论是大肠杆菌还是毕赤酵母表达系统,我们制备的重组胶原酶的蛋白纯度都超过95%,所以导致两种表达系统中胶原酶比活力差异的原因主要是酶活力的差异,而酶活力的差异应该与蛋白质的翻译和组装有关。在酵母真核表达系统中,胶原酶ColR75E的诱导时间达到72 h,蛋白的翻译和组装速度慢,更容易获得结构组装完整进而活力高的蛋白;然而大肠杆菌中表达的蛋白,由于表达速度过快可能导致部分蛋白来不及组装或者组装的不好,导致酶活力低。因此,通过比较发现,利用毕赤酵母真核表达系统获得了具有更高比活力的酶,具有更好的市场应用价值。
此外,通过测定不同浓度 (0.5、1.0、2.0、4.0、6.0、8.0、10.0 g/L) 下Ⅰ型胶原酶蛋白的反应体系中重组ColR75E胶原酶的反应初速度,绘制出如图 7所示的Lineweaver-Bürk双倒数曲线图。由图 7可知,毕赤酵母重组ColR75E胶原酶对于Ⅰ型胶原酶蛋白的最大反应速度Vmax值为6.45 mmol/(g·min),显著高于大肠杆菌中重组胶原酶的最大反应速度Vmax值5.71 mmol/(g·min)。米氏常数Km值为2.75 g/L,比活力值与大肠杆菌中获得的重组ColR75E胶原酶的Km值2.93 g/L相仿[9],没有显著区别。由毕赤酵母真核表达所得的ColR75E胶原酶具有比原核表达系统所得胶原酶更高的最大反应速度和比活力,但是具有相似的Km值,表明不同表达系统改变了酶的反应速度,但是并未改变酶对底物的亲和力。底物特异性分析发现ColR75E仅对Ⅰ型胶原蛋白具有水解能力,而对其他蛋白例如溶菌酶、酪蛋白、BSA等不表现水解能力,这与报道的胶原酶特异性地水解天然胶原蛋白的三维螺旋结构相符合[1-2]。
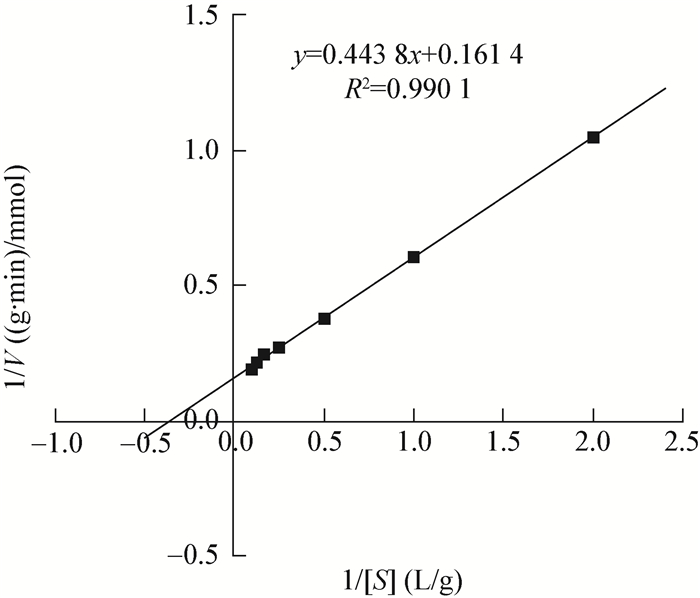
|
| 图 7 重组ColR75E胶原酶对于Ⅰ型胶原蛋白底物的Lineweaver-Bürk双倒数曲线图 Figure 7 Lineweaver-Bürk plot of recombinant ColR75E collagenase to type Ⅰ collagen substrate |
|
|
采用毕赤酵母表达系统操作更加简单,且菌种易于培养、生长速度快、外源表达的蛋白纯度高,大大降低了胶原酶的生产成本[20]。与原核表达工艺相比,毕赤酵母表达系统属于甲醇营养型表达系统[21],酵母表达载体pPIZαA为含有醇氧化酶 (AOX) 启动子的整合载体,可通过同源重组而将外源基因整合到酵母染色体中[22],这样的表达系统继代稳定,能够将外源蛋白分泌到细胞外,不但提高表达蛋白的活性,而且有利于产物的纯化,更加适用于大规模工业生产[23]。
毕赤酵母对密码子的偏好性会影响到外源基因的表达水平[24]。通过对比毕赤酵母和蜡样芽孢杆菌两种菌株对密码子的偏好比例 (图 8) 可知,两种微生物对密码子的偏好性有一定区别,在colR75E基因中非毕赤酵母偏好的密码子占密码子总数的比例高达38.41%,其中有40个Leu、32个Thr、29个Val和9个Ala是由非毕赤酵母偏好密码子编码,这可能是影响毕赤酵母表达量的关键因素[25]。因此,在后续的研究中可以尝试对colR75E进行密码子优化,使其编码序列更符合毕赤酵母的密码子偏爱性用法,从而提高ColR75E在毕赤酵母中的表达量。
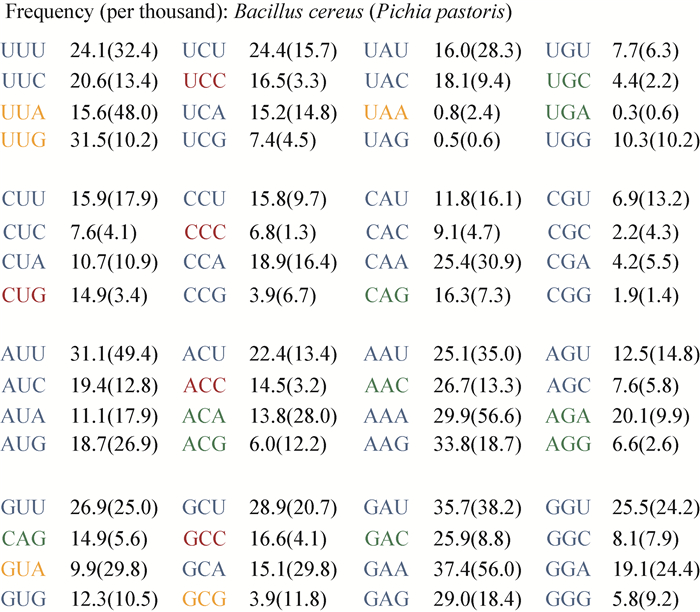
|
| 图 8 毕赤酵母与蜡样芽孢杆菌密码子偏好性比较 Figure 8 Comparison of codon preference between Pichia pastoris X-33 and Bacillus cereus |
|
|
总之,本研究利用毕赤酵母作为表达系统获得了高活性的蜡样芽菌胶原酶ColR75E,为胶原酶在毕赤酵母中的工业化生产进一步奠定了理论基础。
| [1] | Bauer R, Janowska K, Taylor K, et al. Structures of three polycystic kidney disease-like domains from Clostridium histolyticum collagenases ColG and ColH[J]. Acta Crystallographica Section D Biological Crystallography, 2015, 71(3) : 565–577. DOI:10.1107/S1399004714027722 |
| [2] | Jin M, Li JW, Wang ZY. Researches on collagenase secreted by microbes[J]. Amino Acids & Biotic Resources, 2003, 25(1) : 3–7. (in Chinese) 金敏, 李君文, 王忠彦. 微生物胶原酶研究进展[J]. 氨基酸和生物资源, 2003, 25(1) : 3–7. |
| [3] | Tokmina-Roszyk M, Tokmina-Roszyk D, Bhowmick M, et al. Development of a F rster resonance energy transfer assay for monitoring bacterial collagenase triple-helical peptidase activity[J]. Analytical Biochemistry, 2014, 453 : 61–69. DOI:10.1016/j.ab.2014.02.024 |
| [4] | Takagi H, Kondou M, Hisatsuka T, et al. Effects of an alkaline elastase from an alkalophilic Bacillus strain on the tenderization of beef meat[J]. Journal of Agricultural and Food Chemistry, 1992, 40(12) : 2364–2368. DOI:10.1021/jf00024a008 |
| [5] | Malaponte G, Hafsi S, Polesel J, et al. Tumor microenvironment in diffuse large B-cell lymphoma: matrix metallo proteinases activation is mediated by osteopontin overexpression[J]. Biochimica et Biophysica Acta (BBA)-Molecular Cell Research, 2016, 1863(3) : 483–489. DOI:10.1016/j.bbamcr.2015.09.018 |
| [6] | Eckhard U, Schönauer E, Nüss D, et al. Structure of collagenase G reveals a chew-and-digest mechanism of bacterial collagenolysis[J]. Nature Structural & Molecular Biology, 2011, 18(10) : 1109–1114. |
| [7] | Nagano H, To KA. Purification of collagenase and specificity of its related enzyme from Bacillus subtilis FS-2[J]. Bioscience, Biotechnology, and Biochemistry, 2000, 64(1) : 181–183. DOI:10.1271/bbb.64.181 |
| [8] | Zhang XX, Li Y, Wang SY, et al. Identification of a collagenase produced by Bacillus cereus R75E isolated from human colostrum[J]. Applied Biochemistry and Microbiology, 2015, 51(5) : 511–521. DOI:10.1134/S000368381505018X |
| [9] | Zhang XX, Li Y, Wang YH, et al. Expression, purification and enzymatic characterization of ColR75E collagenase of Bacillus cereus R75E[J]. China Biotechnology, 2015, 35(10) : 44–52. (in Chinese) 张西轩, 李晔, 王亚航, 等. 蜡样芽孢杆菌ColR75E胶原酶的表达、纯化及酶学性质研究[J]. 中国生物工程杂志, 2015, 35(10) : 44–52. |
| [10] | Li Y, Zhang XX, Cao GX, et al. Optimization of fermentation conditions for collagenase production from Bacillus cereus and its purification[J]. Microbiology China, 2016, 43(7) : 1419–1428. (in Chinese) 李晔, 张西轩, 曹广秀, 等. 产胶原酶的蜡样芽胞杆菌发酵条件优化及酶的分离纯化[J]. 微生物学通报, 2016, 43(7) : 1419–1428. |
| [11] | Pu Y, Wang ZX. Advancement of ion exchange chromatography and hydrophobic interaction chromatography media application in protein chromatography[J]. Chinese Journal of Biotechnology, 2004, 20(6) : 975–982. (in Chinese) 浦宇, 王芝祥. 蛋白质层析用离子交换和疏水作用层析介质的发展概况[J]. 生物工程学报, 2004, 20(6) : 975–982. |
| [12] | Damasceno LM, Huang CJ, Batt CA. Protein secretion in Pichia pastoris and advances in protein production[J]. Applied Microbiology and Biotechnology, 2012, 93(1) : 31–39. DOI:10.1007/s00253-011-3654-z |
| [13] | Sugiki T, Ichikawa O, Miyazawa-Onami M, et al. Isotopic labeling of heterologous proteins in the yeast Pichia pastoris and Kluyveromyces lactis[A]//Shekhtman A, Burz DS. Protein NMR Techniques[M]. New York: Humana Press, 2012, 831: 19-36 |
| [14] | Daly R, Hearn MTW. Expression of heterologous proteins in Pichia pastoris: a useful experimental tool in protein engineering and production[J]. Journal of Molecular Recognition, 2005, 18(2) : 119–138. DOI:10.1002/(ISSN)1099-1352 |
| [15] | Tang YJ, Yu BS. Yeast Pichia pastoris expression system[J]. World Notes on Antibiotics, 2002, 23(6) : 246–250. (in Chinese) 唐元家, 余柏松. 巴斯德毕赤酵母表达系统[J]. 国外医药:抗生素分册, 2002, 23(6) : 246–250. |
| [16] | Sheen H. Quantitation of yeast total proteins in sodium dodecyl sulfate-polyacrylamide gel electrophoresis sample buffer for uniform loading[J]. Analytical Biochemistry, 2016, 498 : 95–97. DOI:10.1016/j.ab.2016.01.002 |
| [17] | Kocabiyik S, Erdem B. Intracellular alkaline proteases produced by thermo acidophiles: detection of protease heterogeneity by gelatin zymography and polymerase chain reaction (PCR)[J]. Bioresource Technology, 2002, 84(1) : 29–33. DOI:10.1016/S0960-8524(02)00019-6 |
| [18] | Mandl I, MacLennan JD, Howes EL, et al. Isolation and characterization of proteinase and collagenase from Cl. histolyticum[J]. Journal of Clinical Investigation, 1953, 32(12) : 1323–1329. DOI:10.1172/JCI102861 |
| [19] | Liu DS, Liang L, Regenstein JM, et al. Extraction and characterisation of pepsin-solubilised collagen from fins, scales, skins, bones and swim bladders of bighead carp (Hypophthalmichthys nobilis)[J]. Food Chemistry, 2012, 133(4) : 1441–1448. DOI:10.1016/j.foodchem.2012.02.032 |
| [20] | Yao J, Wu ZJ, Ren J. Progress of Pichia pastoris expression system[J]. Jiangsu Agricultural Sciences, 2012, 40(3) : 35–38. (in Chinese) 姚晶, 吴正钧, 任婧. 巴氏毕赤酵母表达系统的研究进展[J]. 江苏农业科学, 2012, 40(3) : 35–38. |
| [21] | Sui SF, Chen SL. Recent advances and character of methylotrophic yeast Pichia pastoris expression system[J]. Biotechnology Bulletin, 2004(3) : 1–4. (in Chinese) 隋少飞, 陈松林. 巴氏毕赤酵母表达系统的特点及其研究进展[J]. 生物技术通报, 2004(3) : 1–4. |
| [22] | Katrolia P, Yan QJ, Jia HY, et al. Molecular cloning and high-level expression of a β-galactosidase gene from Paecilomyces aerugineus in Pichia pastoris[J]. Journal of Molecular Catalysis B: Enzymatic, 2011, 69(3/4) : 112–119. |
| [23] | Li RR, He ZQ, Bao ED, et al. Expression of the chicken antibacterial peptide fowlicidin-3 in Pichia pastoris and identification of biological activity[J]. Scientia Agricultura Sinica, 2010, 43(21) : 4529–4535. (in Chinese) 李荣荣, 和祯泉, 鲍恩东, 等. 鸡源抗菌肽Folicidin-3在毕赤酵母中的分泌表达及其生物学活性鉴定[J]. 中国农业科学, 2010, 43(21) : 4529–4535. DOI:10.3864/j.issn.0578-1752.2010.21.024 |
| [24] | Zhao X, Huo KK, Li YY. Synonymous codon usage in Pichia pastoris[J]. Chinese Journal of Biotechnology, 2000, 16(3) : 308–311. (in Chinese) 赵翔, 霍克克, 李育阳. 毕赤酵母的密码子用法分析[J]. 生物工程学报, 2000, 16(3) : 308–311. |
| [25] | Ma ZH, Zhao QX, Gao X, et al. Expression of hFGF-21 with optimized codon in P. pastoris[J]. Chinese Journal of Biologicals, 2010, 23(1) : 43–46. (in Chinese) 马忠辉, 赵巧香, 高新, 等. 密码子优化的人成纤维细胞生长因子-21在毕赤酵母中的表达[J]. 中国生物制品学杂志, 2010, 23(1) : 43–46. |
 2017, Vol. 44
2017, Vol. 44