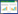扩展功能
文章信息
- 梁晨, 张萌, 喻其林, 邢来君, 李明春
- LIANG Chen, ZHANG Meng, YU Qi-Lin, XING Lai-Jun, LI Ming-Chun
- 巴斯德毕赤酵母Orm1蛋白在细胞生长和内质网功能维持方面的功能
- Function of Pichia pastoris Orm1 in cell growth and endoplasmic reticulum function maintenance
- 微生物学通报, 2017, 44(4): 755-765
- Microbiology China, 2017, 44(4): 755-765
- DOI: 10.13344/j.microbiol.china.160737
-
文章历史
- 收稿日期: 2016-10-14
- 接受日期: 2017-01-03
- 优先数字出版日期(www.cnki.net): 2017-01-10
鞘脂是构成细胞膜结构的重要组成成分,是细胞中三大膜脂 (磷脂、鞘脂、固醇) 之一,是细胞的重要结构组分和信号分子,可调节膜的流动性。鞘脂合成的稳态对细胞正常生长代谢过程至关重要[1]。Orosomucoids (ORM,α-酸性糖蛋白,血清类黏蛋白) 是ORM和ORMDL (ORM-like) 基因编码的内质网膜蛋白,它们属于一类进化上高度保守的内质网整合膜蛋白家族,广泛存在于真核生物中[2]。在酿酒酵母中,基因相互作用图谱表明ORM基因的产物是鞘脂合成的负调控因子[3],其作用机制是通过与鞘脂从头合成的限速酶丝氨酸棕榈酰基转移酶 (Serine palmitoyltransferase,SPT) 直接结合,对该酶起负调控作用,抑制鞘氨醇的从头合成[3]。酿酒酵母中有两个Orm蛋白,分别为Orm1和Orm2,均定位于内质网,二者有重叠功能,它们与SPT酶的三个亚基Lcb1、Lcb2、Tsc3和磷酸肌醇磷酸酶Sac1组成SPOTS复合物[3]。酿酒酵母中Orm蛋白的活性和含量受到细胞的精密调节,主要是表达水平和磷酸化调节[4]。Orm蛋白通过介导鞘脂稳态而影响膜结构的生物发生以及胞内的信号转导、营养应答等过程,在细胞的生长中起重要作用。
内质网压力可诱导非折叠蛋白应答 (Unfolded protein response,UPR)。脂类稳态的改变是诱导UPR的原因之一,脂类稳态失衡可在不引起蛋白质错误折叠的情况下诱导UPR[5]。酿酒酵母中的UPR激活完全依赖于Ire1途径:Ire1p受到激活后,具有Ser/Thr激酶活性的C端胞质结构域可互相聚合,激活C端另一个RNase结构域;活化的RNase结构域剪切HAC1的前体mRNA中一个252 bp的特殊内含子,再由Rlg1p将剪切后的HAC1 mRNA拼接起来,形成具有翻译活性的Hac1imRNA;由Hac1imRNA翻译而来的Hac1ip是转录活化因子,可进入细胞核与靶基因中诱导转录必需的未折叠蛋白反应元件序列结合,启动KAR2、PDI1等靶基因的转录[6-8]。除UPR途径之外,酵母细胞中由钙调神经磷酸酶 (Calcineurin,CaN) 介导的钙细胞存活 (Calcium cell survival,CCS) 途径也是应答内质网压力的一条重要途径,细胞钙稳态的变动会影响CCS途径,影响钙应答基因的表达[9]。
在细胞中活性氧 (Reactive oxygen species,ROS) 有两个主要来源:一是线粒体呼吸,二是内质网中UPR调控的氧化折叠机制。UPR的持续激活将通过内质网中的氧化蛋白折叠反应导致胞内ROS积累[10]。作为分泌蛋白加工的重要场所,内质网可通过二硫键的氧化和还原产生ROS:在二硫键形成过程中,通过一系列巯基-二硫键交换反应,电子将从底物蛋白的巯基转移到蛋白二硫异构酶Pdi1p,接着转移到Ero1p,最后传递给分子氧。在分子氧的还原过程中ROS往往作为副产物产生[11]。
巴斯德毕赤酵母作为一种重要的真核外源蛋白表达载体,具有重要的工业价值,也是一种重要的真核模式生物。脂类的组成与含量与细胞代谢息息相关,但目前有关巴斯德毕赤酵母的研究主要集中在如何提高外源蛋白表达产量上,对其自身的脂类代谢调控网络及相关调控因子的研究仍有空白,仅有的鞘脂合成相关研究也主要集中在酿酒酵母所缺乏的复杂鞘脂葡萄糖基神经酰胺的合成上[12]。在酿酒酵母中Orm1和Orm2蛋白参与了胞内多种代谢[3-4],因此,对巴斯德毕赤酵母Orm1蛋白功能的研究有助于人们了解ORMDL家族蛋白在毕赤酵母生长代谢中的作用,从而更好地发挥其工业应用价值,同时补充了真核微生物的鞘脂代谢方面的研究。
本研究通过序列比对在巴斯德毕赤酵母 (Pichia pastoris) 基因组数据库中发现了酿酒酵母Orm1和Orm2的同源蛋白,命名为Orm1,并将其编码基因命名为ORM1。进而探究了Orm1蛋白在毕赤酵母生长、内质网压力应答、细胞钙稳态和氧化压力应答方面发挥的作用,为进一步研究ORM1基因及其所调控的鞘脂代谢对细胞生长和压力应答的影响奠定理论基础。
1 材料与方法 1.1 实验菌株、质粒和引物巴斯德毕赤酵母 (P. pastoris) 野生型菌株GS115 (his4-,Mut+),购自Invitrogen;大肠杆菌 (Escherichia coli) DH5α菌株作为亚克隆宿主菌,由本实验室保存;本研究中以PCR介导的同源重组法构建orm1Δ缺失菌株,将回补质粒pIB1-ORM1转入orm1Δ缺失菌株以获得ORM1基因回补菌株orm1Δ+ORM1。本研究所用引物见表 1。
| Primers | Sequence (5′→3′) | Size (bp) |
| ORM1-5DR | GGAAAGATTGGAGCCAGTGAG | 21 |
| ORM1-1 | GAAGCTATGGTGTGTGGGGCTGTGTTAAGGATATATCG | 38 |
| ORM1-5ZEO | AGCCCCACACACCATAGCTTC | 21 |
| ORM1-3ZEO | CTGCACCACAGCTTGCAAATTAAAGCC | 27 |
| ORM1-2 | GGCTTTAATTTGCAAGCTGTGGTGCAGTCTGTGTTGAC | 38 |
| ORM1-3DR | CGCTACTGATACCTGTGGAC | 20 |
| ORM1-3inner | CTGAAAATCGGGAATAGATCGT | 22 |
| ORM1-5COM | CGGAATTCACAGACAAAGTATTTATTTATTG | 31 |
| ORM1-3COM | CCGCTCGAGATGTACCCAGGTTCATA | 26 |
| Real-PPACT-F | GGTTCCCACTTATTTCCCAG | 20 |
| Real-PPACT-R | TCCTTCAGTTTTTCCGTCTC | 20 |
| IRE1-5RT | AGCCCTTGGTTTCTGTTGC | 19 |
| IRE1-3RT | ATCTCTCCAGTCCATCCCGA | 20 |
| INO1-5RT | TGACAAGGCAAGATACACCG | 20 |
| INO1-3RT | GACCTACCAGCAACAAACCC | 20 |
| PMT4-5RT | GTCTCGTTCTGTGACTTCAACC | 22 |
| PMT4-3RT | GCCAACACCAGCAAATAATAGC | 22 |
| PDI1-5RT | GAACTTGTTTCTGCTGCCG | 19 |
| PDI1-3RT | CAATGCTTTGGCTCTGTCTT | 20 |
| KAR2-5RT | AAGTCGGGTCGTGTAGAAAT | 20 |
| KAR2-3RT | CGCTTCAAGTCTCTTTGGA | 19 |
| CCH1-5RT | TCAGAAACAAATCAGCCCG | 19 |
| CCH1-3RT | AGTGGGCATTGTCCTTGAC | 19 |
| CNB1-5RT | TTTGACGAAGATGGCGGT | 18 |
| CNB1-3RT | TCAGATTCCCACCAACCA | 18 |
| PMR1-5RT | TCTGTTACTGATGGGTTCCG | 20 |
| PMR1-3RT | GCCTCCAATGATTTCTCGG | 19 |
| PMC1-5RT | CCGTCCACACTATCAAGTCTG | 21 |
| PMC1-3RT | AGTATGTTCTCTCCGTATCTCTCG | 24 |
| CRZ1-5RT | CTTCCTCCAAACGCTTCTTT | 20 |
| CRZ1-3RT | GTATTGGTTGAAGTCTTGGGC | 21 |
| RT-cSOD-5 | CGAACAATCCTCCGAAAG | 18 |
| RT-cSOD-3 | ACCCTTGGCAACACCTTCA | 19 |
| RT-mSOD-5 | AAACAAGGAGGTGGAGAGC | 19 |
| RT-mSOD-3 | CAAAGGGACCAAACTACC | 18 |
| RT-CAT-5 | GCTACTAACCTGAAGGACGC | 20 |
| RT-CAT-3 | TTGAAGTTTACGACACCCAG | 20 |
| RT-GPX1-5 | CCCATTAGATAAGAAAGGCG | 20 |
| RT-GPX1-3 | CCAAACTGGTTACAGGGAA | 19 |
| GLR1-5RT | AACTTCGCCCAACCGTAT | 18 |
| GLR1-3RT | TCTCAATCGCCAAGGACT | 18 |
| TRR1-5RT | TTATTGCCACTGGTGCCT | 18 |
| TRR1-3RT | CCTCCAACAACTGCCAAAG | 19 |
| RT-YAP1-5 | GGAAGCCAAGCACAAGGTA | 19 |
| RT-YAP1-3 | TCCATACAGAGCCCATCAAT | 20 |
多球壳菌素 (Myriocin),生工生物工程 (上海) 有限公司;衣霉素 (Tunicamycin),Bio Basic公司;Eastep总RNA提取试剂盒、M-MLV反转录酶,普洛麦格 (北京) 生物技术有限公司;Oligo-dT、RNA酶抑制剂,Takara公司;SYBR-Green qPCR扩增试剂盒,TransGen公司;过氧化氢酶 (CAT) 酶活性检测试剂盒、超氧化物歧化酶 (SOD) 酶活性检测试剂盒、谷胱甘肽 (GSH) 含量测定试剂盒,南京建成生物工程研究所;Fluo-3 AM、DCFH-DA、焦炭酸二乙酯 (DEPC) 等,北京鼎国生物技术有限责任公司。其余试剂及药品皆为国产分析纯。
恒温培养箱,天津天市机电有限公司;恒温摇床,江苏太仓市实验设备厂;紫外-可见分光光度仪,瑞典Pharmacia Biotech公司;多功能酶标仪,PerkinElmer公司;Eppendorf实时荧光定量PCR仪,美国Bio-Rad公司。
1.3 培养基YPD培养基 (g/L):酵母提取物10.0,蛋白胨20.0,葡萄糖20.0;SC培养基 (g/L):酵母无氨基酸氮源6.7,葡萄糖20.0,各种氨基酸混合物2.0。按要求在YPD固体平板中添加不同浓度Myriocin、H2O2、CaCl2、EGTA。
1.4 实验方法 1.4.1 生长曲线测定: 将过夜培养活化的GS115、orm1Δ和orm1Δ+ORM1菌株转接至50 mL YPD培养基中,调整OD600为0.1,30 ℃、160 r/min振荡培养,每2 h取样一次测定OD600,绘制生长曲线图。 1.4.2 药物敏感性测定: 将过夜培养的GS115、orm1Δ和orm1Δ+ORM1菌株转接至YPD培养基中,调整OD600=0.1,按每组5个平行管、每管200 μL分装于灭菌的干燥1.5 mL离心管内,实验组加入2、3、4 mg/L的衣霉素处理,对照组加入等量无菌水,置于30 ℃培养箱中静置培养24 h,测定OD600,绘制比生长率图。 1.4.3 RNA的提取和基因相对表达量的测定: 将培养过夜的GS115、orm1Δ和orm1Δ+ORM1菌株转接至YPD或SC培养基中,培养至对数期,收集菌体,无菌水洗涤之后液氮速冻。以Eastep总RNA提取试剂盒提取毕赤酵母细胞总RNA,并反转录为cDNA。以SYBR-Green qPCR扩增试剂盒进行实时荧光定量PCR检测,以β-actin内参基因校正表达量,基因相对表达量以2-ΔΔCt方法计算。 1.4.4 胞质钙含量测定: 将培养过夜的GS115、orm1Δ和orm1Δ+ORM1菌株转接至YPD培养基中,调整OD600=0.1,培养至对数期收集菌体,用HBSS洗涤3次后,加入5 μL钙离子荧光探针Fluo-3 AM染料 (1 g/L),避光孵育1 h;收集细胞,HBSS洗涤3次,以酶标仪测定荧光强度,所得结果与菌液OD600的比值即代表胞质钙含量。 1.4.5 ROS含量测定: 将培养过夜的GS115、orm1Δ和orm1Δ+ORM1菌株转接至YPD培养基中,调整OD600=0.1,培养至对数期,实验组加入2 mg/L衣霉素,对照组加入等量无菌水,处理2 h,收集菌体,用PBS洗涤3次后,加入5 μL 2′, 7′-二氯二氢荧光黄二乙酸酯 (2′, 7′-Dichlorodihydro fluorescein diacetate,DCFH-DA) 染料 (1 g/L),避光孵育30 min;收集细胞,PBS洗涤3次,以酶标仪测定荧光强度,所得结果与菌液OD600的比值即代表胞内ROS水平。 1.4.6 CAT酶活性、SOD酶活性、GSH含量测定: 将培养过夜的GS115、orm1Δ和orm1Δ+ORM1菌株转接至YPD培养基中,调整OD600=0.1,培养至对数期,收集菌体,加入玻璃微珠、50 mmol/L Tris-HCl (pH 7.5)、1 mmol/L苯甲基磺酰氟,以液氮研磨法提取胞内总蛋白,测定CAT酶活性、SOD酶活性、GSH含量。 1.4.7 Folch方法测定酵母提取物中的总脂类含量: 称取一定质量酵母提取物 (Yeast extract),每1 g样品中加入20 mL体积比为2:1的氯仿/甲醇,室温下振荡15-20 min。过滤,取滤液,加入16 ml (0.2倍体积) 生理盐水洗涤2-3次。2 000 r/min离心10 min,取下层有机相,真空蒸发溶剂。称取所得物质量,计算产率。 2 结果与分析 2.1 毕赤酵母Orm1蛋白的鉴定酿酒酵母Orm1和Orm2蛋白属于一个保守的真核生物内质网膜蛋白家族 (ORMDL),该家族蛋白主要参与了细胞鞘脂合成稳态的调控[2-3]。从结构上看,ORMDL家族蛋白在其氨基酸序列的中部一般含3-4个跨膜结构域,与动物、植物相比,酵母 (包括酿酒酵母、粟酒裂殖酵母等) 中的Orm蛋白N端多出20-60个氨基酸残基,该家族蛋白虽无已知的功能结构域,但其序列高度保守[2]。从功能方面来看,酿酒酵母orm1Δorm2Δ双缺失菌株的生长受到严重抑制、对二硫苏糖醇 (DTT)、衣霉素、CaCl2、EGTA、SDS等试剂敏感[2]。通过序列比对在巴斯德毕赤酵母基因组数据库中找到一个与酿酒酵母Orm1和Orm2同源的蛋白。用DNAMAN软件进行序列比对表明,该蛋白与酿酒酵母ScOrm1的氨基酸序列具有较高相似性 (68.29%),此外与酿酒酵母ScOrm2的氨基酸序列具有62.58%的相似性 (图 1A)。因此,将毕赤酵母中该蛋白命名为Orm1,相应的编码基因命名为ORM1。
|
| 图 1 巴斯德毕赤酵母Orm1蛋白序列分析 Figure 1 Sequence analysis of Pichia pastoris Orm1 protein 注:A:DNAMAN软件对巴斯德毕赤酵母Orm1蛋白进行序列分析;B:用TMpred在线工具对毕赤酵母Orm1蛋白进行跨膜结构域分析. Note: A: Sequence analysis of Pichia pastoris Orm1 by DNAMAN; B: Transmembrane domain analysis of Pichia pastoris Orm1 via online TMpred tools. |
|
|
此外,通过TMpred在线工具 (http://www.ch. embnet.org/cgi-bin/TMPRED_form_parser) 对Orm1蛋白序列进行跨膜结构分析 (图 1B),发现Orm1蛋白有4个可能的跨膜结构域,分别是69-88,97-113,148-166,168-187。与酿酒酵母Orm1蛋白 (84-107,112-130,163-181,185-205) 和Orm2蛋白 (79-99,104-121,153-173,179-200) 跨膜区数量一致,位置相近。综上,毕赤酵母Orm1蛋白与酿酒酵母Orm1和Orm2同源程度高,具有多个相似的保守氨基酸位点和跨膜结构域,与ScOrm1和ScOrm2属于同一保守的内质网整合膜蛋白家族。
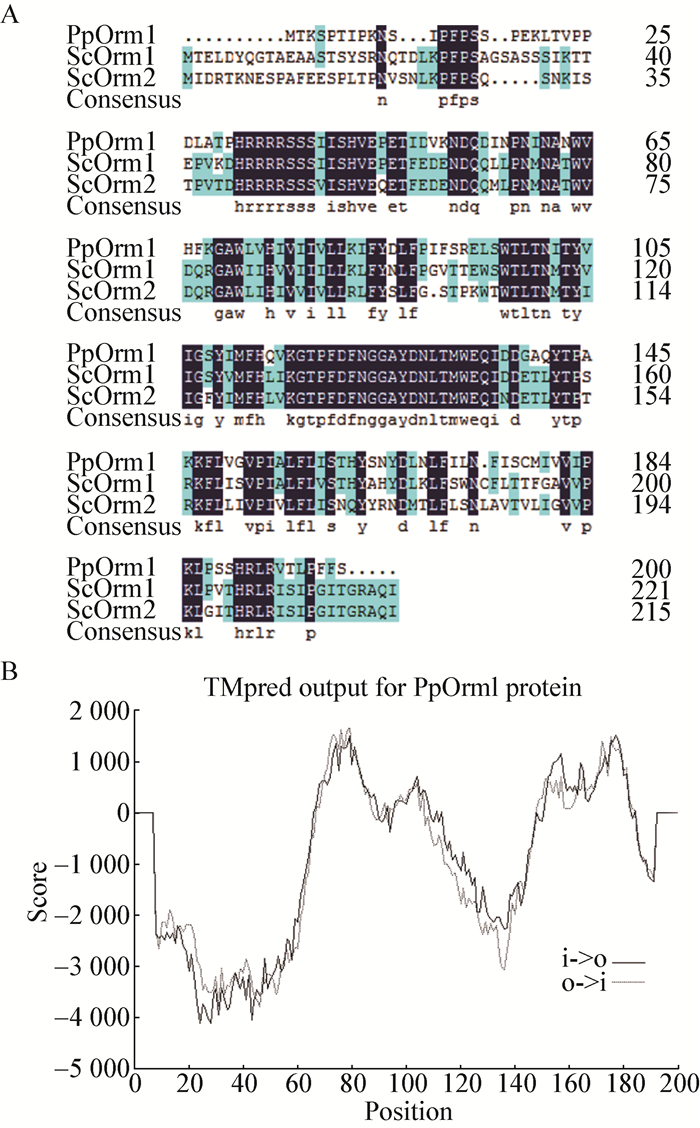
2.2 ORM1基因缺失影响毕赤酵母生长在YPD液体培养基中,与野生型菌株及回补菌株相比,orm1Δ缺失菌株表现出生长缓慢的特点 (图 2A)。与此相似,在YPD固体平板上,orm1Δ缺失菌株形成的菌落偏小 (图 2B)。为了进一步研究orm1Δ缺失菌株生长缓慢是否由胞内鞘脂合成紊乱所致,在固体YPD培养基中添加多球壳菌素 (Myriocin),分析其对上述菌株生长的影响。该药物是鞘氨醇的结构类似物,含量极微时即可抑制SPT酶的活性[13]。如图 2B所示,在加入50 μg/L Myriocin的固体平板上,orm1Δ缺失菌株的生长得以恢复,表明orm1Δ缺失菌株生长缓慢与胞内鞘脂从头合成紊乱相关。
|
| 图 2 ORM1基因缺失对毕赤酵母生长的影响 Figure 2 Effect of ORM1 deletion on the growth of P. pastoris 注:A:在30 ℃ YPD液体培养基培养条件下GS115、orm1Δ和orm1Δ+ORM1的生长情况;B:在YPD和YPD+50 μg/L myriocin固体培养基上GS115、orm1Δ和orm1Δ+ORM1的生长情况. Note: A: Growth situation of GS115, orm1Δ and orm1Δ+ORM1 in liquid YPD medium at 30 ℃; B: Serial dilution strain of GS115, orm1Δ and orm1Δ+ORM1 were spotted onto plates of YPD and YPD plus 50 μg/L myriocin. |
|
|
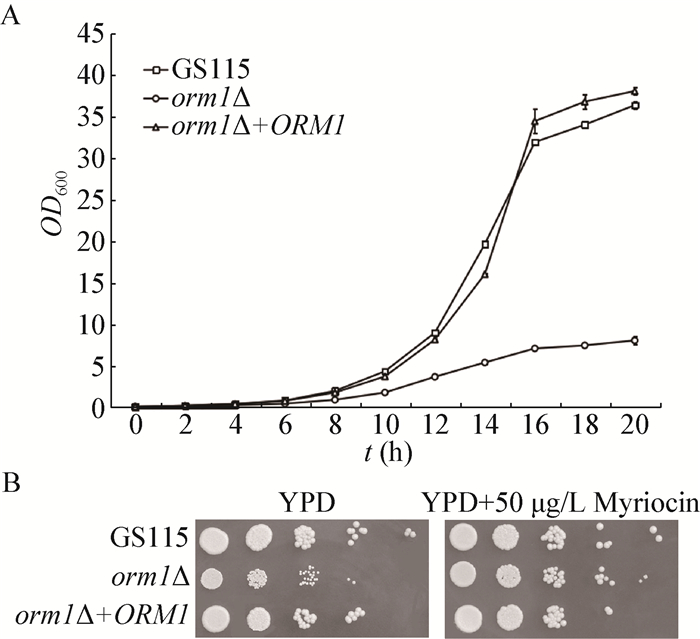
内质网是真核生物细胞内蛋白质和脂类的主要合成场所,脂类合成紊乱可导致内质网压力,激活非折叠蛋白反应 (Unfolded protein response,UPR)[5]。我们推测ORM1基因缺失可能造成的鞘脂稳态紊乱会进一步增加内质网压力,因此研究了ORM1基因缺失对细胞内质网功能造成的影响。如图 3A所示,在不同浓度衣霉素 (Tunicamycin) 处理条件下,与野生型相比,orm1Δ缺失菌株的生长受到明显抑制,并且具有剂量依赖的特点。

|
| 图 3 ORM1基因缺失对细胞内质网功能的影响 Figure 3 Effect of ORM1 deletion on ER function 注:A:orm1Δ对衣霉素的敏感性具有剂量依赖性;B、C:实时荧光定量PCR法分析UPR基因相对表达量,菌株分别培养于YPD培养基 (B) 或SC合成培养基 (C). *:orm1Δ和WT之间差异性显著 (P < 0.05). Note: A: Sensitivity of orm1Δ to tunicamycin is dose-dependent; B, C: Expression level changes of UPR genes were analyzed by real-time PCR. Cells were cultured in YPD (B) or SC (C) medium. *: Significant difference between orm1Δ and WT (P < 0.05). |
|
|
我们进而研究了毕赤酵母中ORM1基因缺失对UPR应答基因表达的影响。如图 3B所示,在YPD培养条件下仅INO1基因表达量显著升高。在酿酒酵母和白念珠菌中,YPD和SC合成培养基由于其营养成分的不同,将对所培养菌株的代谢、表型、基因表达造成不同程度的影响[14-15]。因此,针对上述结果,考虑到YPD中的营养成分较为丰富,推测其中可能含有大量脂类营养物,这部分脂类营养物干扰了培养于其中的毕赤酵母的UPR基因表达。因此,提取了YPD培养基组分之一——酵母提取物 (Yeast extract) 中的总脂类成分并分析其含量。从4.00 g酵母提取物中提取得到0.60 g淡黄色固体粉末状物质,产率为15.00%。这表明YPD培养基中的确存在脂类营养物质,且含量较高。
因此,为了排除这部分干扰,检测了不含脂类营养物的SC合成培养基中各UPR基因的表达情况,结果发现与对照菌株相比,orm1Δ缺失菌株中IRE1、INO1、PDI1、KAR2四种UPR基因的表达量均显著升高 (图 3C)。
综上所述,推测Orm1蛋白在毕赤酵母内质网功能维持方面发挥作用,ORM1基因缺失将造成内质网功能紊乱,进而激活UPR途径。
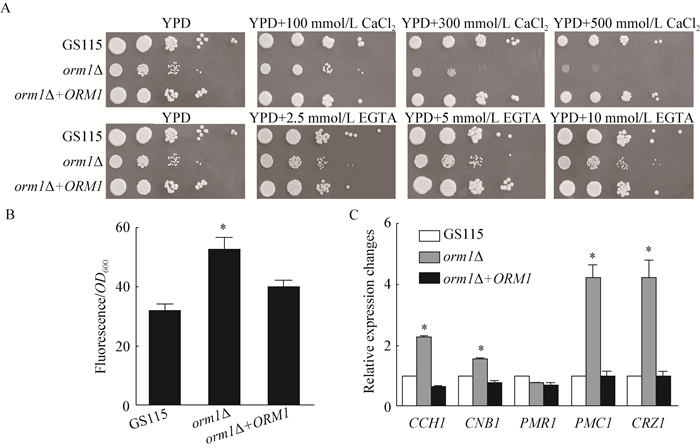
2.4 Orm1蛋白参与胞内钙稳态调控在酿酒酵母中,Orm2蛋白的表达受到钙调神经磷酸酶 (Calcineurin,CaN) 介导的钙细胞存活 (Calcium cell survival,CCS) 途径的调控[4]。本研究继而探讨了ORM1基因与钙信号途径的关系。结果发现,orm1Δ缺失菌株对CaCl2造成的高钙条件表现出剂量依赖的敏感性 (图 4A)。同时,其在YPD培养条件下所测定的胞质钙含量明显高于野生型 (图 4B)。此外,在orm1Δ菌株中,潜在的CCS途径应答基因CCH1、CNB1、CRZ1、PMC1的表达量均有显著升高 (图 4C)。以上结果表明,Orm1蛋白在一定程度上参与了细胞的钙稳态调节。由于内质网是细胞中Ca2+离子储存的重要场所,联系orm1Δ缺失菌株对内质网压力的敏感性,ORM1基因缺失对细胞钙稳态的影响可能与该基因的缺失影响了内质网功能有关。
|
| 图 4 ORM1基因缺失对细胞钙稳态的影响 Figure 4 Effect of ORM1 deletion on calcium homeostasis 注:A:orm1Δ缺失菌株对高钙 (CaCl2)、低钙 (EGTA) 条件的敏感性;B:GS115、orm1Δ和orm1Δ+ORM1菌株在YPD培养条件下测定的胞质钙含量;C:实时荧光定量PCR法分析钙应答基因相对表达量. *:差异显著 (P < 0.05). Note: A: Sensitivity of orm1Δ to high calcium (CaCl2) and low calcium (EGTA); B: Cytoplasmic calcium contents were measured in GS115, orm1Δ and orm1Δ+ORM1 strain under YPD culture condition; C: The expression level changes of calcium response genes were measured by real-time PCR. *: Significant difference between orm1Δ and WT (P < 0.05). |
|
|
大量研究表明,UPR可通过造成ROS积累进而促发细胞凋亡[10]。为了探究ORM1基因缺失对胞内ROS水平的影响,以DCFH-DA染色法对毕赤酵母胞内ROS水平进行了分析。结果表明,在YPD培养条件下,与野生型菌株相比,orm1Δ缺失菌株胞内ROS含量明显升高;在2 mg/L衣霉素处理条件下,胞内ROS含量进一步升高 (图 5A)。据此,研究了orm1Δ缺失菌株对H2O2的敏感性。然而,随着H2O2浓度的升高,与对照菌株相比,orm1Δ缺失菌株并未表现出对H2O2的敏感性,即ORM1基因的缺失并不影响毕赤酵母对H2O2的耐受性 (图 5B)。

|
| 图 5 ORM1基因缺失对胞内ROS水平和氧化压力敏感性的影响 Figure 5 Effects of ORM1 deletion on ROS level and sensitivity to oxidative stress 注:A:GS115、orm1Δ和orm1Δ+ORM1菌株在YPD和YPD+2 mg/L衣霉素处理下测定的胞内ROS水平;B:orm1Δ菌株对H2O2的敏感性. *:差异显著 (P < 0.05). Note: A: Cellular ROS levels were measured in GS115, orm1Δ and orm1Δ+ORM1 strain under YPD with or without 2 mg/L tunicamycin culture condition; B: Sensitivity of orm1Δ to H2O2. *: Significant difference between orm1Δ and WT, and orm1Δ with or without tunicamycin (P < 0.05). |
|
|
根据上述结果,推测ORM1基因的缺失激活了毕赤酵母细胞抗氧化系统,因而进一步测定了orm1Δ缺失菌株中抗氧化系统基因表达和主要酶活性的变化。结果发现orm1Δ缺失菌株中,胞内参与ROS清除和氧化压力应答基因的表达量 (图 6A),过氧化氢酶 (CAT) 活性 (图 6B)、超氧化物歧化酶 (SOD) 活性 (图 6C)、谷胱甘肽 (GSH) 含量 (图 6D) 均显著上升。这表明orm1Δ缺失菌株中抗氧化系统可能并未受到损伤,这在一定程度上能够解释虽然orm1Δ缺失菌株中的ROS显著升高,但其对H2O2造成的外源氧化压力耐受性并未减弱的原因。
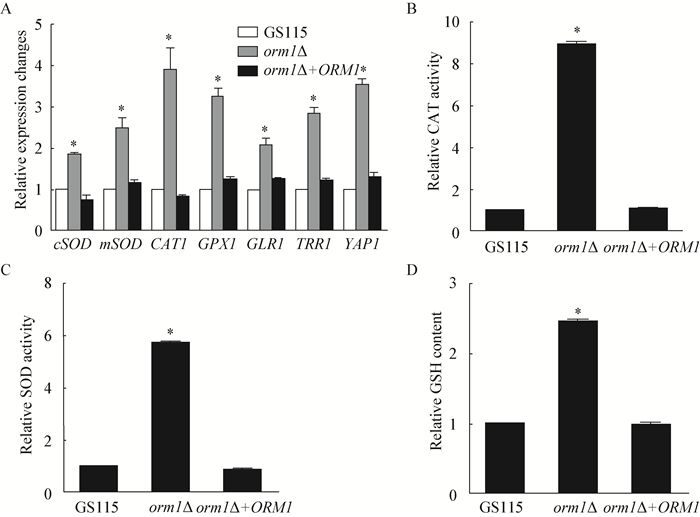
|
| 图 6 ORM1基因缺失对抗氧化系统活性的影响 Figure 6 Effects of ORM1 deletion on activity of antioxidative system 注:A:实时荧光定量PCR法分析抗氧化基因相对表达量;B-D:ORM1基因缺失导致抗氧化系统激活. YPD培养条件下,菌株培养至对数期,分别测定各菌株中CAT活性 (B)、SOD活性 (C) 和GSH含量 (D). *:差异显著P < 0.05. Note: A: The expression level changes of anti-oxidant genes were perceived by real-time PCR; B–D: Cellular antioxidant system was actived in orm1Δ cells. Cells were cultured in YPD medium to log-phase, and cellular CAT activity (B), SOD activity (C) and GSH content (D) were analyzed. *: Significant difference between orm1Δ and WT (P < 0.05). |
|
|
在酿酒酵母中,Orm1和Orm2蛋白可作为TOR (Target of rapamycin) 信号途径与CaN信号途径共同的下游应答基因,对营养状况、细胞鞘脂代谢、质膜状态等作出应答,协调细胞的生长与压力应答[4, 16]。本研究通过序列比对在巴斯德毕赤酵母中找到酿酒酵母Orm1和Orm2的同源蛋白及其编码基因,将该蛋白命名为Orm1,将该蛋白的编码基因命名为ORM1。本研究发现,与野生型和回补菌株相比,orm1Δ菌株的生长十分缓慢,并且这种缓慢与orm1Δ菌株中鞘脂合成紊乱有关。
在酿酒酵母中,orm1Δorm2Δ菌株中UPR组成型激活[17]。脂类稳态失衡时,Ire1可直接被触发而发生二聚化并激活,从而起始下游的信号途径[18]。鞘脂代谢的紊乱也会诱导UPR发生。有研究表明,C16-神经酰胺的堆积会诱发UPR[19]。抗惊厥药2-丙基戊酸钠也能通过提高胞内神经酰胺的水平诱导UPR[20]。本研究发现在以SC培养基进行培养时,UPR途径的应答基因IRE1、INO1、PDI1、KAR2表达量升高,但在高脂类含量的YPD培养基中培养时,仅INO1基因的表达量升高。上述结果表明毕赤酵母中ORM1基因的缺失可导致UPR激活,而UPR的激活程度则受到细胞可利用的脂类营养成分及含量的影响[5]。因此,ORM1基因缺失对毕赤酵母UPR的激活作用应该与细胞中脂类代谢失衡有关,并受到培养基中脂类成分的影响。
在酿酒酵母中INO1基因编码的肌醇-3-磷酸合酶是细胞内肌醇从头合成途径的限速酶,该基因在从人类到酵母等多种真核生物体内保守。肌醇是磷脂酰肌醇和酵母细胞中的复杂鞘脂磷酸肌醇神经酰胺 (Inositol phospharylceramide,IPC) 头部基团的重要组分,肌醇水平的改变将影响多种脂类的合成[21]。在酿酒酵母中,肌醇匮乏可直接诱导UPR,而INO1基因的表达受UPR的诱导[22]。肌醇营养缺陷对酿酒酵母orm1Δorm2Δ菌株而言是致死的[17],而在野生型菌株中,培养基中肌醇匮乏可诱导ORM2基因的转录[4]。在真核细胞中,磷脂和鞘脂的合成由于其共同的中间产物而相互交叉,因此有人提出鞘脂代谢受到磷酸肌醇信号途径的调控[23]。本研究发现,ORM1基因的缺失会导致INO1基因表达显著上调,表明ORM1基因缺失引起的鞘脂合成紊乱可能对磷脂等其他脂类的合成造成影响,这一点有待于继续研究。
内质网是细胞钙稳态调节的重要细胞器。有研究表明,在酿酒酵母中内质网压力通过激活CaN途径提高了Orm2的蛋白水平[4]。本研究发现ORM1基因缺失导致细胞对高钙条件敏感、导致胞质钙离子浓度升高、激活了部分CCS途径应答基因的表达。这表明毕赤酵母中ORM1基因的缺失可对细胞钙稳态造成一定影响。在酿酒酵母中,内质网压力激活质膜Cch1-Mid1钙通道蛋白引起钙内流,导致胞质钙含量增加;增加的胞质Ca2+离子与钙调蛋白 (Calmodulin,CaM) 结合激活CaN,后者通过转录因子Crz1激活钙应答基因的表达[9]。由于ORM1基因缺失后会导致内质网压力加剧,推测在巴斯德毕赤酵母中也存在类似机制。内质网压力可激活多种CaN途径的下游靶基因,使其参与到膜的重构中,其中就包括影响膜脂组分合成的基因,如编码复杂鞘脂甘露糖基磷酸肌醇神经酰胺 (Mannosyl inositol phospharylceramide,MIPC) 合酶催化亚基、调节亚基的SUR1、CSG2,与固醇合成有关的ERG26等[24]。因此ORM1基因对细胞钙稳态的影响可能与其对鞘脂合成的调控作用有关。
UPR的激活会使得内质网中的氧化蛋白质发生折叠,从而导致ROS大量产生。本研究发现ORM1基因的缺失导致胞内ROS升高,并且在衣霉素造成的内质网压力条件下ROS水平升高更为明显。然而,orm1Δ缺失菌株并未对过氧化氢所营造的氧化压力条件表现出明显的敏感性,推测与ORM1基因缺失并未破坏细胞的抗氧化系统有关。有报道称ROS可激活参与调控并维持细胞鞘脂稳态的TORC2-Ypk1信号途径[25],因此鞘脂稳态的失衡与ROS在胞内的积累有着密切联系。鉴于ORM1基因缺失并未造成明显的氧化压力表型,推测orm1Δ菌株内ROS积累在很大程度上来源于UPR所诱导的内质网蛋白质氧化折叠。
总之,ORM1基因所编码的Orm1蛋白作为细胞鞘脂从头合成的重要调控因子,在维持菌体生长、内质网功能、细胞钙稳态及胞内ROS水平方面扮演着重要角色,表明鞘脂在细胞生长及压力应答过程中发挥了极为重要的作用。
| [1] | Hannun YA, Obeid LM. Principles of bioactive lipid signalling: lessons from sphingolipids[J]. Nature Reviews Molecular Cell Biology, 2008, 9(2) : 139–150. DOI:10.1038/nrm2329 |
| [2] | Hjelmqvist L, Tuson M, Marfany G, et al. ORMDL proteins are a conserved new family of endoplasmic reticulum membrane proteins[J]. Genome Biology, 2002, 3(6) : research0027.1–0027.16. |
| [3] | Breslow DK, Collins SR, Bodenmiller B, et al. Orm family proteins mediate sphingolipid homeostasis[J]. Nature, 2010, 463(7284) : 1048–1053. DOI:10.1038/nature08787 |
| [4] | Gururaj C, Federman RS, Chang A. Orm proteins integrate multiple signals to maintain sphingolipid homeostasis[J]. Journal of Biological Chemistry, 2013, 288(28) : 20453–20463. DOI:10.1074/jbc.M113.472860 |
| [5] | Hou NS, Gutschmidt A, Choi DY, et al. Activation of the endoplasmic reticulum unfolded protein response by lipid disequilibrium without disturbed proteostasis in vivo[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(22) : E2271–E2280. DOI:10.1073/pnas.1318262111 |
| [6] | Wu HX, Ng BSH, Thibault G. Endoplasmic reticulum stress response in yeast and humans[J]. Bioscience Reports, 2014, 34(4) : e00118. |
| [7] | Pineau L, Colas J, Dupont S, et al. Lipid-induced ER stress: synergistic effects of sterols and saturated fatty acids[J]. Traffic, 2009, 10(6) : 673–690. DOI:10.1111/tra.2009.10.issue-6 |
| [8] | Cox JS, Chapman RE, Walter P. The unfolded protein response coordinates the production of endoplasmic reticulum protein and endoplasmic reticulum membrane[J]. Molecular and Cellular Biology, 1997, 8(9) : 1805–1814. DOI:10.1091/mbc.8.9.1805 |
| [9] | Bonilla M, Nastase KK, Cunningham KW. Essential role of calcineurin in response to endoplasmic reticulum stress[J]. The EMBO Journal, 2002, 21(10) : 2343–2353. DOI:10.1093/emboj/21.10.2343 |
| [10] | Haynes CM, Titus EA, Cooper AA. Degradation of misfolded protein prevents ER-Derived oxidative stress and cell death[J]. Molecular Cell, 2004, 15(5) : 767–776. DOI:10.1016/j.molcel.2004.08.025 |
| [11] | Tu BP, Weissman JS. Oxidative protein folding in eukaryotes: mechanisms and consequences[J]. Journal of Cell Biology, 2004, 164(3) : 341–346. DOI:10.1083/jcb.200311055 |
| [12] | Ternes P, Wobbe T, Schwarz M, et al. Two pathways of sphingolipid biosynthesis are separated in the yeast Pichia pastoris[J]. Journal of Biological Chemistry, 2011, 286(13) : 11401–11414. DOI:10.1074/jbc.M110.193094 |
| [13] | Miyake Y, Kozutsumi Y, Nakamura S, et al. Serine palmitoyltransferase is the primary target of a sphingosine-like immunosuppressant, ISP-1/myriocin[J]. Biochemical and Biophysical Research Communications, 1995, 211(2) : 396–403. DOI:10.1006/bbrc.1995.1827 |
| [14] | Klose C, Surma MA, Gerl MJ, et al. Flexibility of a eukaryotic lipidome-insights from yeast lipidomics[J]. PLoS One, 2012, 7(4) : e35063. DOI:10.1371/journal.pone.0035063 |
| [15] | Mahto KK, Singh A, Khandelwal NK, et al. An assessment of growth media enrichment on lipid metabolome and the concurrent phenotypic properties of Candida albicans[J]. PLoS One, 2014, 9(11) : e113664. DOI:10.1371/journal.pone.0113664 |
| [16] | Liu M, Huang CJ, Polu SR, et al. Regulation of sphingolipid synthesis through Orm1 and Orm2 in yeast[J]. Journal of Cell Science, 2012, 125(10) : 2428–2435. DOI:10.1242/jcs.100578 |
| [17] | Han S, Lone MA, Schneiter R, et al. Orm1 and Orm2 are conserved endoplasmic reticulum membrane proteins regulating lipid homeostasis and protein quality control[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(13) : 5851–5856. DOI:10.1073/pnas.0911617107 |
| [18] | Volmer R, Ron D. Lipid-dependent regulation of the unfolded protein response[J]. Current Opinion in Cell Biology, 2015, 33 : 67–73. DOI:10.1016/j.ceb.2014.12.002 |
| [19] | Epstein S, Kirkpatrick CL, Castillon GA, et al. Activation of the unfolded protein response pathway causes ceramide accumulation in yeast and INS-1E insulinoma cells[J]. The Journal of Lipid Research, 2012, 53(3) : 412–420. DOI:10.1194/jlr.M022186 |
| [20] | Jadhav S, Russo S, Cottier S, et al. Valproate induces the unfolded protein response by increasing ceramide levels[J]. Journal of Biological Chemistry, 2016, 291(42) : 22253–22261. DOI:10.1074/jbc.M116.752634 |
| [21] | Gaspar ML, Aregullin MA, Jesch SA, et al. Inositol induces a profound alteration in the pattern and rate of synthesis and turnover of membrane lipids in Saccharomyces cerevisiae[J]. Journal of Biological Chemistry, 2006, 281(32) : 22773–22785. DOI:10.1074/jbc.M603548200 |
| [22] | Chang HJ, Jones EW, Henry SA. Role of the unfolded protein response pathway in regulation of INO1 and in the sec14 bypass mechanism in Saccharomyces cerevisiae[J]. Genetics, 2002, 162(1) : 29–43. |
| [23] | Brice SE, Alford CW, Cowart LA. Modulation of sphingolipid metabolism by the phosphatidylinositol-4-phosphate phosphatase Saclp through regulation of phosphatidylinositol in Saccharomyces cerevisiae[J]. Journal of Biological Chemistry, 2009, 284(12) : 7588–7596. DOI:10.1074/jbc.M808325200 |
| [24] | Cyert MS. Calcineurin signaling in Saccharomyces cerevisiae: how yeast go crazy in response to stress[J]. Biochemical and Biophysical Research Communications, 2003, 311(4) : 1143–1150. DOI:10.1016/S0006-291X(03)01552-3 |
| [25] | Niles BJ, Joslin AC, Fresques T, et al. TOR Complex 2-Ypk1 signaling maintains sphingolipid homeostasis by sensing and regulating ROS accumulation[J]. Cell Reports, 2014, 6(3) : 541–552. DOI:10.1016/j.celrep.2013.12.040 |
 2017, Vol. 44
2017, Vol. 44