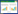扩展功能
文章信息
- 张博, 姚学萍, 王印, 杨泽晓, 曹随忠, 张鹏飞
- ZHANG Bo, YAO Xue-Ping, WANG Yin, YANG Ze-Xiao, CAO Sui-Zhong, ZHANG Peng-Fei
- CRISPR-Cas系统调控细菌生理功能的机制研究进展
- Advances in the mechanism of CRISPR-Cas system for regulating bacterial physiological function
- 微生物学通报, 2017, 44(12): 3045-3052
- Microbiology China, 2017, 44(12): 3045-3052
- DOI: 10.13344/j.microbiol.china.170504
-
文章历史
- 收稿日期: 2017-07-07
- 接受日期: 2017-09-04
- 优先数字出版日期(www.cnki.net): 2017-09-07
2. 动物疫病与人类健康四川省重点实验室 四川 成都 611130
2. Key Laboratory of Animal Disease and Human Health of Sichuan Province, Chengdu, Sichuan 611130, China
细菌基因组中规律成簇的间隔短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR)及附属的蛋白质(Cas)组成了CRISPR-Cas系统[1]。它的发现,最初源于人们对大肠杆菌K-12的酸性磷酸酶基因(AphA)进行研究时,发现一段存在于AphA的编码区附近且由简单重复序列(24-37 bp)组成的特殊DNA序列,但当时并没有得到研究者们的广泛重视[2]。随后,相似且广泛存在于细菌和古细菌基因组的结构序列也被研究者们发现,除此之外发现在单个重复序列之间还插入了其他序列,被称之为间隔序列。这种间隔序列片段的长度相似且具有回文结构,这样规律成簇的间隔短回文重复序列被命名为CRISPR序列[3]。该序列及相关的Cas蛋白共同组成了存在于细菌和古细菌中的CRISPR-Cas系统。当外源DNA入侵细菌时,细菌和古细菌识别入侵的DNA后,以CRISPR序列为模板转录出RNA来阻止外源DNA的入侵。间隔序列可以识别并结合特定的噬菌体DNA序列,Cas核酸酶将入侵的DNA进行切割使其降解,因此称为细菌的获得性免疫防御[4]。
1 细菌CRISPR-Cas系统的基本结构CRISPR-Cas系统是一段特殊的DNA重复序列,主要由用于切割目的基因的cas基因组成,其中cas基因又分为cas1、cas2、cas9等多种基因类型以及用于识别特异序列的识别序列组成。识别序列中包含了前导序列(Leader)、重复序列(direct repeats)、间隔序列(spacers) (图 1)。CRISPR序列中,间隔序列将重复序列分割开来形成许多不连续的重复序列,并且大部分的重复序列都有回文结构和稳定的二级结构。因物种之间的差异,CRISPR序列中重复序列的数量也有所不同[5]。细菌CRISPR-Cas系统广泛分布于基因组的不同区域,但大多数情况下存在于染色体上,仅少数存在于质粒中[6]。
2 细菌CRISPR-Cas系统的结构分型及主要成分的功能根据CRISPR-Cas系统中cas基因种类的多样性(表 1)以及Cas蛋白作用机理的不同,通常将该系统划分为3种不同的类型:Type Ⅰ、Type Ⅱ和Type Ⅲ。其中Type Ⅱ (CRISPR-Cas9)结构最为简单,研究最为深入[12]。
三种类型中,Ⅰ型系统比较复杂,存在于大部分的细菌和古细菌中,该系统发生作用时需要多种Cas蛋白,其中Cas3蛋白在CRISPR系统的功能作用中起至关重要的作用。Caseade (CRISPR-assoicated complex for antiviral defense)-crRNA复合物在ATP、二价镁离子的参与下,外源DNA被结合在特定的靶位点,随后Cas3核酸酶将外源DNA进行切割,使其降解[7]。
Ⅲ型CRISPR系统主要存在于古细菌中,系统较为复杂,主要有A、B两种类型,两种类型均需要多个Cas核酸酶才能切割目的DNA,此类型CRISPR系统中Cas6蛋白有重要的作用,主要参与crRNA的成熟,Cas6核酸酶与crRNA结合并识别入侵的DNA,将目标DNA降解[10]。
相比Ⅰ、Ⅲ型CRISPR-Cas系统,Ⅱ型CRISPR-Cas系统发挥免疫防御作用仅需Cas9蛋白参与即可,且易与Ⅰ、Ⅱ型免疫防御系统区分,cas9基因作为该系统的一个标志性元件,专一性地与CRISPR位点相联系。基于cas基因的多样性,Ⅱ型CRISPR-Cas系统又可以进一步细分为A、B、C三种类型[8]。在C型CRISPR系统中主要有cas9、cas1和cas2基因以及CRISPR RNA (crRNA)和反式激活crRNA (tracrRNA)。然而在A型和B型的CRISPR系统中还分别有相关的附加信号基因csn2或是cas4。C型CRISPR系统中的cas9核酸内切酶行使DNA的切割功能;tracrRNA是一段非蛋白编码RNA,作为CRISPR-Cas9系统的第二个重要组成成分,crRNA的成熟以及随后DNA的剪切就依靠该结构发挥作用[9];crRNA用于捕获外源DNA序列,CRISPR-Cas9系统中的间隔序列就存在于该结构中。当外源DNA进入细菌或古细菌机体内时,它们会特异性地识别并将外源DNA插入到前导序列和重复序列之间。当然外源DNA的插入也并不多是随机插入的,需要间隔序列随后的一段保守核苷酸序列即间隔序列临近基序——PAM (protospacer adjacent motif)结构帮助Cas9确定切割位点,以便随后的Cas9蛋白行使切割、剪切的功能(各成分及功能如表 2所示),因此研究中将其改造为人工核酸内切酶基因编辑系统[13]。
| 主要成分 main ingredients | 结构特点 structural features | 功能 Functions |
| Cas3蛋白Cas3 Protein | HD功能域 | 切割DNA[7] |
| Cas6蛋白Cas6 Protein | PDB、2XLJ | 参与crRNA的成熟[10, 14] |
| Cas9蛋白Cas9 Protein | 含4种Rucv核酸酶和一种HNH核酸酶 | 识别、切割、产生crRNA[15-16] |
| Cas10蛋白Cas10 Protein | 聚合酶和环化酶类功能域 | 切割ssDNA[14] |
| 反式激活RNA tracrRNA | 非蛋白编码的RNA | 促进crRNA的成熟[9] |
| CRISPR成熟RNA crRNA | 含前导区和一系列重复-间隔-重复序列 | 捕获外源DNA[9, 17] |
| 间隔序列临近基序PAM | 紧随间隔序列后的NGG序列 | 帮助Cas9核酸酶确定切割位点[13] |
| 前导序列Leader | 长度100-500 bp,不保守,含转录起始位点 | 启动CRISPR重复序列转录[11] |
| 重复序列Direct repeats | 位于两重复序列间,来自于噬菌体或质粒 | 使细菌获得对应噬菌体或质粒的免疫防御能力[18] |
| 间隔序列Spacers | 位于23-55 bp之间,序列多样性呈部分回文对称结构 | 识别并结合特定的噬菌体DNA序列[19-20] |
基于此,我们课题组以广西巴马小型猪为研究载体,利用CRISPR-Cas9为研究工具开展引导RNA的筛选,进行瓦登伯格氏综合症(Waardenburg syndrome,WS)小型猪MITF (Microphthalmia-associated transcription factor)小眼相关转录基因缺失模型的构建,构建了一个高效引导RNA快速筛选的平台,实现了大动物中利用CRISPR-Cas9进行基因组定点插入进行基因编辑的可行性[21]。该研究成果也体现了利用Ⅱ型CRISPR-Cas系统实现了目的基因特定位点的基因断裂,为该系统用于基因组的定点编辑奠定了基础。
3 细菌CRISPR-Cas系统的免疫识别及防御机理细菌CRISPR-Cas系统发挥免疫防御时以Ⅱ型防御系统最为广泛,该系统中Cas9核酸酶以向导RNA为靶标,然后通过碱基互补配对的方式去识别外源DNA[22]。当外源基因进入细菌后,细菌通过识别特异的PAM序列,将外源DNA加工成大小合适的间隔序列,插入到CRISPR序列中。此时CRISPR介导免疫需要crRNA和tracrRNA互补配对,激活RNase Ⅲ和Csn I,对CRISPR RNA进行剪切,使之成为成熟的crRNA[17]。Cas9蛋白包含4种Rucv核酸酶(Rucv Ⅰ-Ⅳ)和一种HNH核酸酶。Rucv I和HNH核酸酶主要降解目标DNA。随后Cas9识别目标基因中的PAM序列,crRNA识别与PAM互补的DNA序列。继而,Cas9蛋白中的核酸酶行使切割功能,导致靶位点的切割,基因组双键断裂[15-16](图 2)。
相关研究不仅剖析了是否该防御系统会将自身内部基因剪切而降解,还对它是如何识别自己和异己成分的机制进行了研究与分析。Marraffini等[23]通过构建表皮葡萄球菌CRISPR位点的间隔重复序列以及该序列上下游各200个碱基对的pC194重组质粒pCRISPR (wt),而后再转染CRISPR缺失型及野生型细胞,结果表明crRNA的非间隔序列成分与重复序列的碱基配对不完全或特定位点个别碱基配对失配,可导致crRNA干扰细菌自身的基因组。由此可见,crRNA和CRISPR DNA重复序列之间的碱基配对可阻止自身免疫的发生,同时也证明了crRNAs介导了细菌对于自己和异己成分的免疫[24]。
4 细菌CRISPR-Cas系统对细菌生理功能的调控 4.1 细菌CRISPR-Cas系统对细菌耐药性的调控作用细菌耐药性(Resistance to drug)又称抗药性,指细菌对于抗菌药物作用的耐受性,耐药性一旦产生,药物的杀菌作用就明显下降。细菌耐药性的产生大多是由像转座子、质粒等可移动元件的转移获得,编码这些可移动元件的基因作为耐药基因。耐药基因的水平转移(Horizontal gene transfer,HGT)使细菌产生耐药性,也是细菌产生耐药性的主要原因[25]。细菌CRISPR系统赋予了它们对外源入侵基因的获得性免疫防御能力,这种获得性免疫防御作用限制了耐药基因的水平转移,从而使细菌对抗菌药物产生一定的抵抗作用[26]。
Sampson等[27]对弗朗西斯菌所含的CRISPR-Cas9系统研究发现,该系统可通过调节该菌的胞膜渗透性使其产生相应耐药性。其他种属细菌中也有很多关于细菌耐药性与CRISPR-Cas系统关系的研究,例如对于金黄色葡萄球菌的研究中发现,CRISPR-Cas系统能够限制细菌耐药基因的水平转移,且有阻止耐药基因在葡萄球菌之间传播的作用[28];Burley等[29]对于粪肠球菌中的CRISPR相关的蛋白基因cas3、cas1检测发现,cas3或cas1的存在与该菌多重耐药的获得关系相反。
不难发现,CRISPR-Cas系统与某些细菌维持耐药性且稳定存在或遗传有潜在相关性,对细菌耐药性有重要影响作用。基于CRISPR-Cas系统耐药性方面的研究,研究者可构建出能表达细菌CRISPR-Cas免疫系统的活性质粒,以深入研究细菌耐药基因水平转移。除此之外,若能将表达细菌CRISPR-Cas免疫系统的活性质粒通过载体转入细菌中特异性地剪切致病性和耐药性基因,从而选择性地失活细菌的有害基因,一定能对细菌的耐药性研究提供崭新平台[30]。不仅如此,细菌耐药性研究也应从细菌耐药基因的水平转移方面入手。刘五高等[31]基于嗜热链球菌LMD-9菌株上有完整的CRISPR-Cas系统组件,并有阻止噬菌体和质粒入侵自身系统的功能。利用PCR技术分三段扩增嗜热链球菌LMD-9的CRISPR-Cas基因序列,并依次接入pACYCDuet-1质粒,成功构建了细菌CRISPR-Cas免疫系统质粒,将该质粒再转入嗜热链球菌,质粒中的gRNA就可特异性地剪切致病性和耐药性基因,为今后用于细菌耐药基因水平转移打下了基础。
4.2 细菌CRISPR-Cas系统对细菌毒力的调控作用细菌的毒力是细菌的重要生物学特性,在细菌致病过程中发挥非常重要的作用。细菌的毒力基因可以编码在质粒、致病岛或噬菌体上,如化脓性链球菌、棒状杆菌、肉毒梭菌、霍乱弧菌、链球菌和金黄色葡萄球菌等均含有编码毒力因子的基因[32]。CRISPR-Cas系统与细菌的毒力密切相关,且对细菌的毒力有调控作用,相关研究近年来层出不穷。
Nozawa等[33]研究发现化脓性链球菌中的CRISPR-Cas系统可以使噬菌体毒力基因转入,从而使这些菌株有了相应的致病性。空肠弯曲菌编码Ⅱ型CRISPR-Cas系统,在缺失CRISPR位点的菌株中表达Cas9蛋白能增强细菌毒力,这表明Cas9蛋白能独立于CRISPR转录子发挥功能。此外,空肠弯曲菌的Cas9突变株在感染人源细胞时其运动能力增强,而细胞毒性减弱[34]。Bikard等[35]研究发现CRISPR-Cas系统的干扰作用可以阻断人肺炎链球菌体内毒力的获得。因干扰作用的存在导致含有荚膜基因(肺炎链球菌的毒力基因)的质粒进入无毒菌株而受阻,不含荚膜基因的无毒菌株因此不能变为含荚膜基因的有毒菌株,最终抑制了毒力株的出现。CRISPR-Cas系统也可干扰细菌获取毒力基因,对细菌毒力有双重调控作用,原噬菌体序列与细菌的CRISPR间隔序列存在着不兼容性,会相互排斥,CRISPR-Cas系统此时可在抵抗毒性噬菌体过程中干扰毒力因子在致病菌之间的传播。噬菌体之间的相互作用也是构成细菌毒力的一个重要因素,噬菌体的感染同时也是细菌具有毒力的重要因素[36]。
大多数致病菌均含有CRISPR-Cas系统,通过对多种致病菌CRISPR序列及毒力编码基因的研究,表明尤其以Type Ⅱ CRISPR-Cas系统中的cas9基因在细菌毒力调节过程中起至关重要的作用[36]。
目前关于CRISPR-Cas系统与致病菌的毒力基因共同进化关系的探究仍有欠缺。因而,本课题组预以四川多地区的弯曲菌分离株为研究对象,针对其所含CRISPR-Cas系统、毒力基因的检测并聚类分析与比对,以探究弯曲菌中CRISPR-Cas系统与其毒力基因的共同进化关系。相信结合相关研究及实验条件的不断优化能为弯曲菌所含毒力基因与CRISPR-Cas系统的共同进化及调控关系提供基础数据。
4.3 细菌CRISPR-Cas系统对细菌生物膜形成的调控作用在生物膜形成方面,CRISPR-Cas系统通过部分碱基互补配对机制进行基因调控,这一结论在铜绿假单胞菌生物膜形成的研究中得到证实[37]。Zegans等[38]发现噬菌体DMS3感染铜绿假单胞菌产生的溶原性细菌无法形成完整的生物膜,还发现其生物膜形成受抑制需要宿主体内CRISPR的存在。此外,另有研究表明,CRISPR特异性插入序列是抑制铜绿假单胞菌生物膜形成所必需的,因该特异性插入序列转录形成反义RNA,沉默铜绿假单胞菌的生物膜形成基因,从而也就抑制了生物膜的形成[39]。
生物膜性能的改变会导致细菌膜的渗透性改变,从而会使细菌的毒力、耐药性、致病性都有所改变。可见,CRISPR-Cas系统对细菌生物膜的形成方面有重要的调节作用。基于此,可以构建表达细菌CRISPR-Cas免疫系统的活性质粒转入细菌,特异性地插入参与生物膜形成的序列中而抑制细菌生物膜的形成,以影响其多种性能。
5 结语与展望综上所述,细菌CRISPR-Cas系统在细菌耐药基因水平转移、生物膜形成、致病性调控等方面均具有调控作用。从近几年相关研究成果来看,主要研究方向趋向于CRISPR-Cas系统与细菌生理功能的相关性、基于该系统改造的CRISPR-Cas9基因组编辑技术的应用与研究。实际上,基于细菌CRISPR-Cas系统衍生的基因组编辑技术及生理功能探究,均源于对细菌基因组中CRISPR基因(如间隔序列的溯源、CRISPR序列的插入或缺失)与其毒力、耐药、生物膜形成基因的相关性研究分析,以及人为构建的具有活性的CRISPR-Cas质粒转入真核细胞或原核基因组中而实现的基因组定点插入、缺失等。
目前,多数研究所涉及的仍是CRISPR-Cas系统衍生的基因组编辑技术在真核细胞领域的研究,如基因治疗、疾病模型构建、植物学研究、昆虫学研究等方面,而在细菌领域内的应用略显匮乏,这或因与细菌所含内源CRISPR-Cas9系统的活性不同、外源引入的CRISPR-Cas9系统对其宿主的天然免疫防御能力的干扰作用有差异,以及许多细菌本身缺乏NHEJ (Nonhomologous end joining)机制或虽存在该机制但不能有效地将经Cas9蛋白诱导下断裂的DNA双链修复,进而影响到对细菌基因组的编辑等因素有关。但真核细胞领域的研究策略同样能指引研究者在细菌学领域开展相关研究,如进行CRISPR抗菌剂、基因抑制、缺失等方面的研究[40-41]。
随着生物信息学的不断发展,研究者在更多的生物基因组中寻找到了CRISPR-Cas系统的存在,而且还做了大量的相关研究。如Burstein等[42]通过对酸性矿井废水中的纳米古生菌的基因组分析发现了新的CRISPR系统,该新系统中含有两种新的蛋白CasY以及CasX,研究结果扩充了研究者对于CRISPR系统的认知领域;贺金荣等[43]对鼠疫菌菌株中CRISPR位点间遗传多态性进行分析,为鼠疫菌的起源、传播、进化研究奠定了基础;Zhang等[44]利用CRISPR-Cas9技术激活链霉菌中沉默的基因簇,促使细菌细胞制造这些基因簇编码的天然产物,为实现新型抗菌药物的研发打下基础;程希等[45]基于沙门氏菌中的CRISPR而建立的一种新型鉴定及显示细菌基因组CRISPR的方法,为搜寻致病菌中CRISPR及其它重复序列的相关研究提供了支持。回首诸多研究,而今研究者可以预见的是随着细菌CRISPR-Cas系统研究的不断深入,必将给研究病原微生物的分类、生物学功能、基因溯源带来新的启发,以便为更好地应用于筛选与控制耐药菌株、生物医药的研发奠定基础,应用前景将是大放异彩。
| [1] |
Marchfelder A. Special focus CRISPR-Cas[J]. RNA Biology, 2013, 10(5): 655-658. DOI:10.4161/rna.24687 |
| [2] |
Guo SC, Lv Y, Lin YX, et al. CRISPR/Cas9 systems: the next generation gene targeted editing tool[J]. Proceedings of the National Academy of Sciences, India Section B: Biological Sciences, 2015, 85(2): 377-387. DOI:10.1007/s40011-014-0362-3 |
| [3] |
Liu L, Fan XD. CRISPR-Cas system: a powerful tool for genome engineering[J]. Plant Molecular Biology, 2014, 85(3): 209-218. DOI:10.1007/s11103-014-0188-7 |
| [4] |
Wiedenheft B. In defense of phage: viral suppressors of CRISPR-mediated adaptive immunity in bacteria[J]. RNA Biology, 2013, 10(5): 886-890. DOI:10.4161/rna.23591 |
| [5] |
Wei CX, Liu JY, Yu ZS, et al. TALEN or Cas9—rapid, efficient and specific choices for genome modifications[J]. Journal of Genetics and Genomics, 2013, 40(6): 281-289. DOI:10.1016/j.jgg.2013.03.013 |
| [6] |
Koonin EV, Makarova KS. CRISPR-Cas: evolution of an RNA-based adaptive immunity system in prokaryotes[J]. RNA Biology, 2013, 10(5): 679-686. DOI:10.4161/rna.24022 |
| [7] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [8] |
Banfield JF. CRISPRs in the microbial community context[A]//Barrangou R, van der Oost J. CRISPR-Cas Systems[M]. Berlin, Heidelberg: Springer, 2013: 287-291
|
| [9] |
Shalem O, Sanjana NE, Hartenian E, et al. Genome-scale CRISPR-Cas9 knockout screening in human cells[J]. Science, 2014, 343(6166): 84-87. DOI:10.1126/science.1247005 |
| [10] |
Staals RHJ, Brouns SJJ. Distribution and mechanism of the type I CRISPR-Cas systems[A]//Barrangou R, van der Oost J. CRISPR-Cas Systems[M]. Berlin, Heidelberg: Springer, 2013: 145-169
|
| [11] |
Pougach KS, Lopatina AV, Severinov KV. CRISPR adaptive immunity systems of prokaryotes[J]. Molecular Biology, 2012, 46(2): 175-182. DOI:10.1134/S0026893312020136 |
| [12] |
Chen YL, Huang HR, Tang PP, et al. Genome editing techniques—CRISPR/Cas system[J]. Journal of Hangzhou Normal University (Natural Science Edition), 2015, 14(1): 60-65. 陈勇龙, 黄华荣, 唐平平, 等. 基因组编辑技术——CRISPR/Cas系统[J]. 杭州师范大学学报:自然科学版, 2015, 14(1): 60-65. |
| [13] |
Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering[J]. Cell, 2014, 157(6): 1262-1278. DOI:10.1016/j.cell.2014.05.010 |
| [14] |
Niewoehner O, Jinek M, Doudna JA. Evolution of CRISPR RNA recognition and processing by Cas6 endonucleases[J]. Nucleic Acids Research, 2014, 42(2): 1341-1353. DOI:10.1093/nar/gkt922 |
| [15] |
Sampson TR, Saroj SD, Llewellyn AC, et al. A CRISPR/Cas system mediates bacterial innate immune evasion and virulence[J]. Nature, 2013, 497(7448): 254-257. DOI:10.1038/nature12048 |
| [16] |
Yin LJ, Hu SQ, Guo F. The application of CRISPR-Cas9 gene editing technology in viral infection diseases[J]. Hereditas (Beijing), 2015, 37(5): 412-418. 殷利眷, 胡斯奇, 郭斐. CRISPR-Cas9基因编辑技术在病毒感染疾病治疗中的应用[J]. 遗传, 2015, 37(5): 412-418. |
| [17] |
Heidrich N, Vogel J. CRISPRs extending their reach: prokaryotic RNAi protein Cas9 recruited for gene regulation[J]. The EMBO Journal, 2013, 32(13): 1802-1804. DOI:10.1038/emboj.2013.141 |
| [18] |
Feng H, Dong G, Yong B, et al. Function and organization of CRISPR-Cas systems and application in biotechnology[J]. Journal of Sichuan Normal University (Natural Science), 2014, 37(2): 268-281. 冯红, 董阁, 雍彬, 等. 原核CRISPR-Cas系统的结构功能及应用[J]. 四川师范大学学报:自然科学版, 2014, 37(2): 268-281. |
| [19] |
Davison M, Bhaya D. Creation and analysis of a virome: using CRISPR spacers[A]//Lundgren M, Charpentier E, Fineran P. CRISPR[M]. New York, NY: Humana Press, 2015: 307-316
|
| [20] |
Deng KB, Liu F, Gu CT, et al. Networking clusters and sequence characteristics of clustered regularly interspaced short palindromic repeats (CRISPR) direct repeats and their evolutionary comparison with cas1 genes in lactic acid bacteria[J]. African Journal of Microbiology Research, 2012, 6(23): 5062-5073. |
| [21] |
Wang XL, Zhou JW, Cao CW, et al. Efficient CRISPR/Cas9-mediated biallelic gene disruption and site-specific knockin after rapid selection of highly active sgRNAs in pigs[J]. Scientific Reports, 2015, 5: 13348. DOI:10.1038/srep13348 |
| [22] |
Zhang Y, Heidrich N, Ampattu BJ, et al. Processing-independent CRISPR RNAs limit natural transformation in Neisseria meningitidis[J]. Molecular Cell, 2013, 50(4): 488-503. DOI:10.1016/j.molcel.2013.05.001 |
| [23] |
Marraffini LA, Sontheimer EJ. Self versus non-self discrimination during CRISPR RNA-directed immunity[J]. Nature, 2010, 463(7280): 568-571. DOI:10.1038/nature08703 |
| [24] |
Ma YB, Chang HY. A review on immune system of the bacteria and its self versus non-self discrimination[J]. Chinese Veterinary Science, 2012, 42(6): 657-660. 马延滨, 常惠芸. 细菌的CRISPR/cas免疫及免疫识别[J]. 中国兽医科学, 2012, 42(6): 657-660. |
| [25] |
Esvelt KM, Mali P, Braff JL, et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing[J]. Nature Methods, 2013, 10(11): 1116-1121. DOI:10.1038/nmeth.2681 |
| [26] |
Datsenko KA, Pougach K, Tikhonov A, et al. Molecular memory of prior infections activates the CRISPR/Cas adaptive bacterial immunity system[J]. Nature Communications, 2012, 3: 945. DOI:10.1038/ncomms1937 |
| [27] |
Sampson TR, Napier BA, Schroeder MR, et al. A CRISPR-Cas system enhances envelope integrity mediating antibiotic resistance and inflammasome evasion[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(30): 11163-11168. DOI:10.1073/pnas.1323025111 |
| [28] |
Toro M, Cao GJ, Ju WT, et al. Association of clustered regularly interspaced short palindromic repeat (CRISPR) elements with specific serotypes and virulence potential of shiga toxin-producing Escherichia coli[J]. Applied and Environmental Microbiology, 2014, 80(4): 1411-1420. DOI:10.1128/AEM.03018-13 |
| [29] |
Burley KM, Sedgley CM. CRISPR-Cas, a prokaryotic adaptive immune system, in endodontic, oral, and multidrug-resistant hospital-acquired Enterococcus faecalis[J]. Journal of Endodontics, 2012, 38(11): 1511-1515. DOI:10.1016/j.joen.2012.07.004 |
| [30] |
Li H, Shi ZD. Research progress of gene targeting technology of CRISPR/Cas9 system[J]. Jiangsu Journal of Agricultural Sciences, 2013, 29(4): 907-911. 李辉, 施振旦. CRISPR/Cas9新型基因打靶系统的研究进展[J]. 江苏农业学报, 2013, 29(4): 907-911. |
| [31] |
Liu WG, Ding YF, Liu AX, et al. Recombinant plasmid construction and functional identification of Bacterial CRISPR/Cas9 system[J]. Chinese Journal of Microecology, 2015, 27(9): 1031-1033. 刘五高, 丁友法, 刘爱霞, 等. 细菌CRISPR/Cas免疫系统的构建及功能鉴定[J]. 中国微生态学杂志, 2015, 27(9): 1031-1033. |
| [32] |
Ilyina TS. Filamentous bacteriophages and their role in the virulence and evolution of pathogenic bacteria[J]. Molecular Genetics Microbiology and Virology, 2015, 30(1): 1-9. DOI:10.3103/S0891416815010036 |
| [33] |
Nozawa T, Furukawa N, Aikawa C, et al. CRISPR inhibition of prophage acquisition in Streptococcus pyogenes[J]. PLoS One, 2011, 6(5): e19543. DOI:10.1371/journal.pone.0019543 |
| [34] |
Louwen R, Horst-Kreft D, de Boer AG, et al. A novel link between Campylobacter jejuni bacteriophage defence, virulence and Guillain-Barré syndrome[J]. European Journal of Clinical Microbiology & Infectious Diseases, 2013, 32(2): 207-226. |
| [35] |
Bikard D, Hatoum-Aslan A, Mucida D, et al. CRISPR interference can prevent natural transformation and virulence acquisition during in vivo bacterial infection[J]. Cell Host & Microbe, 2012, 12(2): 177-186. |
| [36] |
Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems[J]. Nature Reviews Microbiology, 2015, 13(11): 722-736. DOI:10.1038/nrmicro3569 |
| [37] |
Wietz M, Millán-Aguiñaga N, Jensen PR. CRISPR-Cas systems in the marine actinomycete Salinispora: linkages with phage defense, microdiversity and biogeography[J]. BMC Genomics, 2014, 15(1): 936. DOI:10.1186/1471-2164-15-936 |
| [38] |
Zegans ME, Wagner JC, Cady KC, et al. Interaction between bacteriophage DMS3 and host CRISPR region inhibits group behaviors of Pseudomonas aeruginosa[J]. Journal of Bacteriology, 2009, 191(1): 210-219. DOI:10.1128/JB.00797-08 |
| [39] |
Palmer KL, Whiteley M. DMS3-42: the secret to CRISPR-dependent biofilm inhibition in Pseudomonas aeruginosa[J]. Journal of Bacteriology, 2011, 193(14): 3431-3432. DOI:10.1128/JB.05066-11 |
| [40] |
Xiao J, Zhang ZD. Progress of harnessing CRISPR-Cas9 system in bacteria[J]. International Journal of Respiration, 2017, 37(6): 450-456. 肖婧, 张宗德. CRISPR-Cas9系统在细菌学领域内的应用研究进展[J]. 国际呼吸杂志, 2017, 37(6): 450-456. |
| [41] |
Gomaa AA, Klumpe HE, Luo ML, et al. Programmable removal of bacterial strains by use of genome-targeting CRISPR-Cas systems[J]. mBio, 2014, 5(1): e00928-13. |
| [42] |
Burstein D, Harrington LB, Strutt SC, et al. New CRISPR-Cas systems from uncultivated microbes[J]. Nature, 2017, 542(7640): 237-241. |
| [43] |
He JR, Zhang YJ, Wang YM, et al. Polymorphism analysis of clustered regularly interspaced short palindromic repeat (CRISPR) loci of 58 Yersinia pestis strains isolated from Xinjiang, China[J]. Chinese Journal of Vector Biology and Control, 2017, 28(3): 233-237. 贺金荣, 张渝疆, 王宇萌, 等. 新疆地区58株鼠疫耶尔森菌规律聚集的间隔短回文重复位点多态性分析[J]. 中国媒介生物学及控制杂志, 2017, 28(3): 233-237. DOI:10.11853/j.issn.1003.8280.2017.03.009 |
| [44] |
Zhang MM, Wong FT, Wang YJ, et al. CRISPR-Cas9 strategy for activation of silent Streptomyces biosynthetic gene clusters[J]. Nature Chemical Biology, 2017, 13: 607-609. DOI:10.1038/nchembio.2341 |
| [45] |
Cheng X, Guo ZR, Hu YM, et al. A method to predict clustered repeats in Salmonella genomes[J]. Basic & Clinical Medicine, 2017, 37(2): 150-155. 程希, 郭志荣, 胡跃明, 等. 一种新的沙门氏菌成簇重复序列的预测方法[J]. 基础医学与临床, 2017, 37(2): 150-155. |
 2017, Vol. 44
2017, Vol. 44