扩展功能
文章信息
- 何珂, 程昌勇, 方春, 宋厚辉, 方维焕
- He Ke, Cheng Chang-Yong, Fang Chun, Song Hou-Hui, Fang Wei-Huan
- 单核细胞增生李斯特菌抗酸应激机制
- Acid tolerance of Listeria monocytogenes
- 微生物学通报, 2017, 44(12): 3015-3023
- Microbiology China, 2017, 44(12): 3015-3023
- DOI: 10.13344/j.microbiol.china.170567
-
文章历史
- 收稿日期: 2017-07-22
- 接受日期: 2017-10-27
- 优先数字出版日期(www.cnki.net): 2017-11-02
2. 浙江大学动物预防医学研究所 浙江 杭州 310058
2. Institute of Preventive Veterinary Medicine, Zhejiang University, Hangzhou, Zhejiang 310058, China
李斯特菌属(Listeria genus)是低G+C%含量的革兰氏阳性无芽孢杆菌,目前发现有17个种[1],其中以单核细胞增生李斯特菌(Listeria monocytogenes,简称单增李斯特菌)、韦氏李斯特菌(Listeria grayi)、伊氏李斯特菌(Listeria ivanovii)、赛氏李斯特菌(Listeria welshimeri)和无害李斯特菌(Listeria innocua)较为常见。一般认为只有单增李斯特菌和伊氏李斯特菌为致病菌,而且仅有单增李斯特菌是该属中唯一的一种人兽共患病原[2-3]。单增李斯特菌广泛存在于环境(包括食品加工环境),人或动物一般通过摄入细菌污染的食物、水源等被感染[4]。
我国学者在2000-2009年间报道了新生儿单增李斯特菌感染25例,其中56% (14例)死亡[5]。2011年,美国发生甜瓜污染单增李斯特菌引起的食源性感染,涉及28个州147人患病,其中33人死亡,是美国历史上最严重的一次食源性疾病暴发[6]。2014年,27个欧盟成员国报告2 161例人的单增李斯特菌感染,相当于每10万人0.52个病例,其中死亡210例,在确诊感染病例中的死亡率为15%,2009-2014年平均每年有163个死亡病例[7]。
1 单增李斯特菌与抗酸应激酸应激是低pH和环境弱酸的综合生物学效应,单增李斯特菌在青贮饲料、发酵食品、宿主胃内以及巨噬细胞吞噬体内都会遭遇到酸应激。与其他食源性病原一样,单增李斯特菌的最适pH为6.0-7.0[8-9],但该菌具有很强的抗酸能力[10]。本实验室比较了参考菌株10403S和EGDe、临床菌株Lm850658和食品分离株M7的抗酸能力,在pH 2.5的BHI中应激处理1 h后,菌株10403S的存活能力显著高于菌株EGDe,Lm850658显著高于M7[11],表明抗酸能力存在菌株间的差异。菌体细胞内pH (pHi)分析结果显示,即便在胞外pH 4.5条件下(pHex,盐酸调节)处理1 h,4个菌株中有3个的pHi仍可维持在6.5-7.5之间,并能缓慢生长。相对于无机酸,用乳酸、醋酸等有机酸处理(将pHex值调整到4.5),4个受试菌株的pHi均下降到5.5左右,细菌不再生长,并且出现部分死亡。提示在同样pHex条件下,有机酸具有更强的抑菌作用。在pHex3.5 (无机酸或有机酸调pHex)条件下处理1 h,所有菌株的pHi降低到5.5或以下,部分细胞出现死亡(不同菌株的存活率在40%-85%之间)[12]。当pHi高于pHex时,弱酸更容易以非离子化的形式扩散进入细胞内。进入胞内后解离释放质子,酸化细胞质。pHex越低,则弱酸性化合物越容易扩散进入细胞质,从而影响pHi。因此,抗酸应激能力(保持细胞内的pH稳态,使之接近中性状态)是单增李斯特菌等细菌在酸性环境下存活,并通过消化道建立感染的重要前提[13]。
2 单增李斯特菌的酸应激适应性单增李斯特菌具有酸应激适应性,即酸耐受反应(acid tolerance response,ATR)。ATR是指细菌短时间暴露于温和酸应激,可显著增强其对更强酸应激(甚至是致死性酸应激)条件下的耐受能力。其机制是温和酸应激诱导了细菌抗酸作用相关蛋白的表达,这些蛋白的合成可被氯霉素抑制,进而阻止ATR的发生[14-15]。
将生长至对数早期的单增李斯特菌LO28菌株暴露于非致死性酸环境(pH 5.5乳酸)中适应1 h,然后置于致死性酸环境(pH 3.5乳酸)中应激1 h,发现经适应性酸应激的细菌比没有适应的细菌存活率高25%[15]。
单增李斯特菌不同菌株间的ATR存在差异:Faleiro等研究了8株来源于奶酪、肉和鱼的不同菌株在pH 5.5乳酸中应激诱导产生最高ATR所需时间:6株的ATR为2 h,在pH 3.5乳酸中处理1 h后细菌存活率介于0.06%-49.60%不等(平均18.3%);另外2株需要诱导4 h才能获得最大ATR,经pH 3.5乳酸同样处理后存活率分别达15.4%和85.7%[16]。
ATR与细菌生长阶段有关:Ferreira等研究了不同生长阶段(对数早期、中期以及稳定期)的10403S菌株在人工胃液(pH 2.5)中应激60 min后的存活能力差异:对数早期细菌于pH 4.5的BHI培养基中适应1 h,再转移至pH 2.5的人工胃液中,其存活能力是未适应细菌的2.5倍;对数中期的细菌用同样方法处理后,存活力提高约3倍;稳定期细菌,无论是否经pH 4.5酸适应,其在人工胃液中的存活率均为100%[17]。以上研究结果表明处于稳定期的细菌具备“天然”的抗酸应激能力(可能是细菌生长过程中产生的代谢物对其自身的应激耐受),无需酸适应就能耐受致死性酸环境[15]。
ATR不仅能为后续酸应激提供保护,还能交叉保护其他环境应激,如不同类型的弱酸、低浓度酒精、过氧化氢等应激[18-20]。因此,深入了解单增李斯特菌抗酸应激的分子机制,有利于评估其在易污染食物中的生长和增殖能力及其对消费者感染的风险,并为设计消除该菌污染的有效方法提供依据。
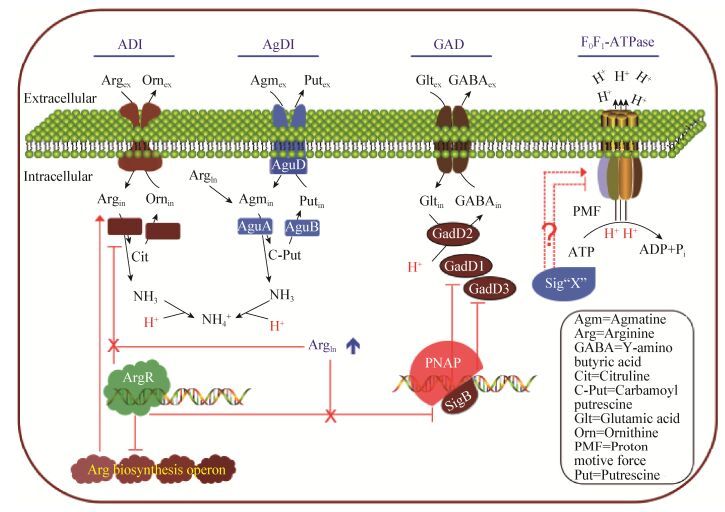
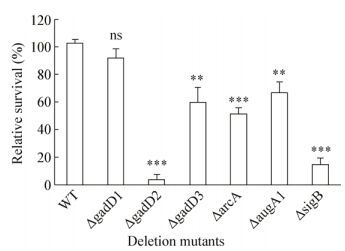
3 单增李斯特菌的抗酸应激系统及其作用机制单增李斯特菌有多种维持pHi稳态的机制(图 1),如F0F1-ATPase系统[21]、谷氨酸脱羧酶系统(glutamate decarboxylase system,GAD)[22]、精氨酸脱亚胺酶(arginine deiminase,ADI)和鲱氨酸脱亚胺酶(agmatine deiminase,AgDI)系统[23-24]。结合基因缺失,综合比较单增李斯特菌10403S不同抗酸系统的作用,我们发现抗酸作用最强的是GadD2,其次是SigB (sigB缺失),再次是ADI (arcA缺失),AgDI (augA1缺失)和GadD3抗酸作用相似(图 2) (F0F1-ATPase系统疑为其缺失具有致死性,无法获得相关组分的缺失菌株进行比较[25])。
|
| 图 1 单核细胞增生李斯特菌的主要抗酸应激系统及其可能的调控机制 Figure 1 Main acid resistance systems of Listeria monocytogenes and the proposed regulatory mechanisms 注:黑色箭头:代谢途径;红色箭头:正调控;红色“T”型:负调控;红色虚线:疑似调控机制;红色“X”:抑制. Note: Black arrows: Metabolic pathways; Red arrows: Positive regulation; Red "T-shaped" symbols: Negative regulation; Red "X": Inhibition. |
|
|
|
| 图 2 单核细胞增生李斯特菌的主要抗酸应激系统缺失株及其亲本菌株在pH 2.5 BHI中的存活能力比较 Figure 2 Comparison of the deletion mutants involved in acid resistance of Listeria monocytogenes in pH 2.5 BHI medium expressed as survival relative to their parent strain 注:WT:亲本菌株,其存活率设定为100%;ns:无显著差异(P > 0.05);**: P < 0.01;***:P < 0.001. Note: WT: parent strain, its survival set as 100%; ns: not significant (P > 0.05);**: P < 0.01; ***: P < 0.001. |
|
|
F0F1-ATPase由多个亚基组成,偶联了质子跨膜转运的ATP合成/水解酶,包括两个结构域(分别为催化结构域F1以及跨膜结构域F0)。F1由α、β、γ、δ和ε亚基组成,能够催化合成或水解ATP;F0包括a、b和c亚基,嵌合在内膜上形成一个跨膜质子通道[21]。F0F1-ATPase参与维持细胞pHi稳态,在质子顺电化学梯度流动时该酶可以催化ADP和Pi合成ATP;反过来当没有氢离子浓度梯度时,F0F1-ATPase能够催化水解ATP并将质子泵出细胞外。在有氧条件下,ATPase能够介导质子进入细胞而合成ATP;在无氧条件下F0F1-ATPase水解ATP供能,排出质子,产生质子动力(PMF)。PMF能够进一步促使细胞内质子排出,从而使处于酸性环境中细菌胞内pH升高[26]。
利用二环己基碳二亚胺(N, N′-Dicyclohexylcarbodiimide,DCCD)作为F0F1-ATPase抑制剂可以分析ATPase所起的作用。Datta等的研究发现,DCCD处理的对数期单增李斯特菌LS2菌株在pH 3.0 (HCl)中应激1 h后存活率下降数倍,但不影响其在pH 7.3条件下的存活,提示细菌在抵抗酸应激过程中伴随质子的泵出[27]。Cotter等发现,DCCD处理处于对数期的单增李斯特菌LO28株,先在pH 5.5乳酸中预处理1 h,后置于pH 3.5乳酸中继续应激2 h,存活的细菌数比没有DCCD处理的对照组细菌低3个log。说明DCCD抑制F0F1-ATPase可以显著降低细菌在致死性酸作用条件(pH 3.5)下的存活能力,进一步证实F0F1-ATPase作为质子泵在细菌抵抗酸应激过程中发挥重要作用[25]。
3.2 谷氨酸脱羧酶系统GAD系统在包括单增李斯特菌在内的多种细菌中发挥重要的抗酸应激作用[22],其抗酸作用主要通过谷氨酸脱羧酶的脱羧反应来实现,该反应通过消耗1 mol谷氨酸和1 mol H+,产生1 mol γ-氨基丁酸(GABA),进而提高pHi以维持胞内稳态。生成的GABA通过跨细胞膜的谷氨酸逆向转运子(glutamate antiporter,GadT)转运至胞外,并交换等摩尔数的谷氨酸。由于谷氨酸常用作食品增鲜剂和防腐剂,此类食品若有单增李斯特菌污染,显然有助于细菌发挥抗酸作用[22]。
相比其他细菌,单增李斯特菌GAD系统有多个组分,包括GadD1/GadD2/GadD3和GadT1/GadT2。但它们的抗酸作用并不完全相同:Karatzas等将它们分为胞外GAD系统(GADo,由GadT1/GadD1和GadT2/GadD2组成)和胞内GAD系统(GADi,即GadD3),并且认为在菌株LO28中GADo发挥主要抗酸作用[28]。Feehily等在菌株10403S中发现类似现象,但菌株EGDe不能输出GABA,其抗酸作用主要依赖GADi。提示GAD系统的抗酸作用存在菌株间差异[29-30]。
本实验室研究结果显示,菌株10403S的gadD2基因缺失可显著降低其在pH 2.5条件下的存活率,gadD3基因缺失后的存活率也显著下降,但不如gadD2基因缺失株显著,而gadD1基因缺失不影响细菌存活(图 2)。以上结果表明GadD2是该菌株的主要抗酸因子。qPCR结果显示:gadT2/gadD2转录水平最高,gadD3次之,gadD1/gadT1最低[11]。不同菌株GAD系统转录水平的分析结果表明,无论是在酸应激(pH 4.5)还是中性培养条件下,10403S的gadT2/gadD2转录水平显著高于EGDe,gadD3次之,gadD1/gadT1转录水平最低;生长稳定期,菌株M7和Lm850658的gadD2转录水平与10403S相当。免疫印迹显示,在pH 4.5或pH 7.0培养条件下,菌株EGDe未检测到GadD2表达,M7菌株仅有微量表达,而菌株10403S和Lm850658的GadD2表达量相当,且GadD2的表达量显著高于GadD3。上述结果提示,GAD系统中不同组分的抗酸作用与其表达水平紧密相关,尤其是GadT2/GadD2的表达量是决定不同菌株间抗酸应激能力差异的主要因素。
3.3 精氨酸脱亚胺酶系统单增李斯特菌有参与抗酸应激的ADI系统[23, 31],该系统由精氨酸脱亚胺酶(ADI,由arcA编码)、鸟氨酸氨甲酰基转移酶(ornithine carbamoyltransferase,OCT,由arcB编码)和氨甲酸激酶(carbamate kinase,CK,由arcC编码) 3个酶组成,可将1 mol精氨酸转化为2 mol氨(NH3)和1 mol鸟氨酸(ornithine),并产生1 mol ATP。1 mol氨可结合1 mol质子形成1 mol铵离子(NH4+),进而升高pHi,发挥抗酸应激作用。鸟氨酸则由逆向转运子(antiporter,由arcD编码)输出至胞外,交换等量精氨酸。本实验室研究发现,单增李斯特菌谱系I和谱系Ⅱ菌株中存在一个基因簇lmo0036-lmo0043,编码ADI系统相关基因及AgDI系统2个基因,依次为arcB-arcD-aguA1-arcC-aguA2-aguR-lmo0042-arcA,而谱系Ⅲ菌株缺失lmo0036-lmo0041基因簇[32-33]。ArcA具有ADI活性,在酸应激条件下acrA转录和表达水平显著上调,缺失arcA导致10403S菌株在人工胃液和小鼠胃中的存活能力显著下降,并降低其对小鼠的致病力[23]。
3.4 鲱精氨脱亚胺酶系统AgDI与ADI系统同处lmo0036-lmo0043基因簇,双拷贝的aguA (aguA1和aguA2)分别编码鲱精氨脱亚胺酶AguA1和AguA2。鲱精氨脱亚胺酶AgDI能够催化精氨酸的脱羧产物鲱精胺水解成氨甲酰腐胺(N-carbamoyl putrescine)和氨,氨甲酰腐胺在腐胺氨甲酰转移酶(Putrescine carbamoyltransferase,PTC)的催化下转变成腐胺(putrescine)和氨甲酰磷酸(carbamoyl phosphate),最后氨甲酰激酶(carbamate kinase,CK)水解腐胺和ADP生成ATP、氨和CO2。整个通路中底物鲱精胺和终产物腐胺通过转运蛋白AguD进行等量交换。我们的研究发现,尽管aguA1和aguA2在酸应激条件下的转录和表达水平都显著上调,且AguA1和AguA2具有高度相似的模拟三级结构,氨基酸相似性67.7%,但仅AguA1发挥抗酸应激作用,并与小鼠的毒力密切相关;AguA2没有抗酸能力[24]。定点突变AguA1和AguA2差异关键位点,我们发现决定二者功能差异的位点为第157位甘氨酸(Gly),将AguA1的157位由Gly突变为Cys后,AguA1即失去活性;而将AguA2的157位由Cys突变为Gly后AguA2便具有活性[24]。
4 单增李斯特菌抗酸应激的调控机制 4.1 应激调控因子σB单增李斯特菌除了含有上述抗酸应激相关系统外,其酸耐受反应ATR还受SigB (σB)调控。单增李斯特菌σB因子与革兰氏阴性菌的RpoS同属于Sigma70家族,能够全局性调控细菌众多基因的表达,并对胞外的环境信号产生应答,从而成为细菌抵抗环境应激的调控枢纽[34]。σB因子作为RNA聚合酶全酶(RNAP)的一个蛋白亚单位,主要负责特定启动子序列的识别和转录的启动。当细菌遇到应激时,σB激活产生调控级联反应,感受并传递应激信号,然后启动受σB调控基因(σB调控子)的转录[34]。单增李斯特菌σB已被证实与酸、温度、渗透压、氧化等应激调控相关[35, 17]。多达150个基因受到σB调控[36]。Bacillus subtilis的sigB操纵子中各组分之间互作和σB激活机制的研究比较深入[37],单增李斯特菌σB的激活网络同样由位于sigB操纵子内的8个蛋白(RsbR-RsbS-RsbT-RsbU-RsbV-RsbW-SigB-RsbX)组成,这些蛋白可能通过磷酸化与去磷酸化机制,以相关蛋白间的结合和解离来调控sigB的表达[38]。
Ferreira等研究了处于不同生长阶段、酸预应激或未应激的10403S sigB基因缺失株和亲本株在pH 2.5的人工胃液或BHI中的存活率,他们发现:(1) sigB基因缺失株的存活率显著低于亲本菌株:对数生长中期的存活率低4个log,而稳定期则低2个log。(2)处于稳定期的sigB基因缺失株和亲本株比对数期细菌的存活率高6个log。(3) pH 4.5的BHI预应激处于对数生长期的细菌,可显著提高ΔsigB株和亲本菌株的存活率(4个log以上);而在稳定期,预应激对ΔsigB株的存活率可提高1个log,但对亲本菌株的存活率影响不显著。这些结果提示,σB不仅与单增李斯特菌的抗酸应激有关,而且还贡献其ATR反应[17]。后续研究也证实了σB参与调节对数期和稳定期细菌的抗酸能力[36]。
Chaturongakul等研究了10403S菌株的调控蛋白RsbT和RsbV对σB依赖型抗酸应激能力的影响,发现对数中期的缺失株ΔsigB、ΔrsbT和ΔrsbV在pH 2.5人工胃液中应激10 min后的存活率较亲本株低3个log,而亲本株只下降了1.5个log。缺失株ΔrsbT和ΔrsbV在pH 2.5的人工胃液或BHI中的存活能力与sigB缺失株相当。说明细菌在受到酸应激时,RsbT和RsbV参与激活σB[39]。本实验室研究了RsbX在单增李斯特菌抗应激中的作用,设计了应激-恢复生长-再应激试验方案,发现rsbX基因缺失株在恢复生长0.5-1.5 h后抵抗二次应激的能力显著高于亲本株。qPCR、启动子活性和免疫印迹试验证实,rsbX基因缺失株在首次应激后恢复生长阶段σB的表达水平高于亲本株,表明RsbX在应激后恢复期负调控σB,将其从高活力状态下调至正常水平。通过比较△rsbX株和亲本株在对数生长期和平台期培养物对高盐应激的差异,我们发现rsbX基因缺失不影响对数生长期细菌对15% NaCl应激的存活率,但其在平台期对高盐应激的存活率显著高于亲本株,平台期△rsbX株的σB表达水平也显著高于亲本株。这些表明RsbX对σB的负调控作用不仅在应激后恢复期,也表现在细菌生长平台期[40]。
σB的激活可以保护单增李斯特菌应对多种环境应激,而这些应激因素又常存在于食品生产过程。因此,van Schaik等提出了以σB为靶标筛选抑制剂,阻止其激活,进而抑制该病原的抗应激能力,为减少单增李斯特菌在食品加工过程中的存活和传播提供一种新的策略[34]。Palmer等通过高通量筛选,发现氟化苯乙烯磺胺(fluoro-phenyl-styrene-sulfonamide,FPSS)可以有效下调受σB调控的大部分基因(IC50=3.5 μmol/L,208个下调基因中的75%受σB调控)[41]。他们的后续研究表明,FPSS并不直接与σB结合抑制其活性,而是可能通过抑制σB与抗SigmaB因子RsbW的释放发挥作用[42]。
4.2 精氨酸抑制因子ArgR单增李斯特菌精氨酸抑制因子ArgR属于ArgR/AhrC转录调控因子家族,参与精氨酸的代谢调控。ArgR与细菌的酸应激存活能力密切相关,10403S的△argR株在pH 3.5 (乳酸调节)应激2-3 h后,其存活率显著高于亲本株。凝胶迁移试验证实ArgR能够与arcA、sigB、argC和argG启动子区的Arg-box发生不同程度的结合,但结合能力存在明显差异,即PargC≥PargG > PsigB > ParcA。氨基酸置换(S42A和R43A)后的ArgR与被调控基因启动子的结合能力明显降低或丧失,证实这两个位点为ArgR的关键氨基酸。在不外源添加精氨酸的应激条件下(pH 5.5),argR缺失株的sigB和arcA转录及蛋白表达水平均显著上调,表明ArgR负调控σB和ArcA。但外源添加精氨酸可以解除这种负调控作用[43]。这些结果提示,ArgR不仅能够反馈负调控精氨酸合成代谢通路基因argC和argG,在酸应激条件下通过精氨酸依赖途径调控arcA和sigB的转录及表达,进而影响单增李斯特菌的抗酸应激作用(图 1)。由于精氨酸和谷氨酸在合成和分解代谢途径紧密相关,ArgR抑制或缺失是否影响细胞内的谷氨酸水平,进而影响GAD系统的抗酸作用也有待研究。
4.3 其他可能的调控机制F0F1-ATPase作为质子跨膜转运系统在抗酸应激中起着重要作用,但这一系统在酸应激条件下是否存在调控机制研究甚少。Barriuso-Iglesias等的研究表明,谷氨酸棒状杆菌(Corynebacterium glutamicum)的SigH因子可以通过与atpB的启动子区结合,调控其在不同pH条件下的表达,进而介导F0F1-ATPase系统对胞内pH的稳态[44]。单增李斯特菌除了SigB,还拥有SigH、SigC和SigL,这3个Sigma因子可能介导冷应激[45]。而这些因子是否介导抗酸应激、通过何种途径介导抗酸应激等问题也有待研究。
Bowman等通过蛋白质组学分析,发现在单增李斯特菌Scott A中gadD2并不响应酸应激调控,认为GadD2为组成型表达[46]。我们的试验也发现,酸应激(pH 4.5)也不显著影响gadD1、gadD2和gadD3的转录和表达,也提示这些基因可能为组成型表达。五个预测的GAD系统疑似调控子gadR缺失也不影响pH 2.5酸应激条件下的存活,表明这些因子并不介导抗酸应激[11]。Kazmierczak等通过转录组学分析,发现sigB缺失仅显著下调gadD3转录水平,提示SigB可能参与调控gadD3[47]。对于gadD1则因菌株而异,可能部分受SigB调控[48]。由于SigB并不调控单增李斯特菌GAD中起主要作用的gadD2,其对gadD1和gadD3调控在抗酸中的作用可能并不重要(图 1)。
经典的双元调控系统由膜蛋白组氨酸激酶(K)和胞质应答调控子(R)组成,组氨酸激酶感受特异性环境应激,并通过磷酸化改变胞质应答调控子的构象,从而激活或抑制受调控目标基因的表达[49]。Cotter等发现单增李斯特菌LO28菌株的lisRK基因位点编码一个双元调控系统,对数期的ΔlisRK菌株在pH 3.5中应激45 min,其抗酸能力显著低于缺失株[50]。但单增李斯特菌还拥有多种双元调控因子,如liaSR (lmo1021/1022)、virSR (lmo1741/1745)[51],这些双元调控因子是否调节该菌的抗酸应激也有待探索。20世纪80年代以来,已经发生了至少4起酸性即食食品引起的单增李斯特菌感染事件,死亡56人[52]。因此,酸性发酵食品生产过程中要严格控制单增李斯特菌的污染,以降低消费者的感染风险。
| [1] |
Orsi RH, Wiedmann M. Characteristics and distribution of Listeria spp., including Listeria species newly described since 2009[J]. Applied Microbiology and Biotechnology, 2016, 100(12): 5273-5287. DOI:10.1007/s00253-016-7552-2 |
| [2] |
Vázquez-Boland JA, Domínguez-Bernal G, González-Zorn B, et al. Pathogenicity islands and virulence evolution in Listeria[J]. Microbes and Infection, 2001, 3(7): 571-584. DOI:10.1016/S1286-4579(01)01413-7 |
| [3] |
Liu DY. Identification, subtyping and virulence determination of Listeria monocytogenes, an important foodborne pathogen[J]. Journal of Medical Microbiology, 2006, 55(6): 645-659. DOI:10.1099/jmm.0.46495-0 |
| [4] |
Schlech III WF, Lavigne PM, Bortolussi RA, et al. Epidemic listeriosis-evidence for transmission by food[J]. New England Journal of Medicine, 1983, 308(4): 203-206. DOI:10.1056/NEJM198301273080407 |
| [5] |
Feng YF, Ran L, Zhang LS. Listeriosis cases reported in medical literatures in China, 2000-2009[J]. Disease Surveillance, 2011, 26(8): 654-659. 冯延芳, 冉陆, 张立实. 2000-2009年中国李斯特菌病文献报告病例分析[J]. 疾病监测, 2011, 26(8): 654-659. |
| [6] |
US Center for Disease Control and Prevention. Multistate outbreak of listeriosis linked to whole cantaloupes from Jensen Farms, Colorado (Final update)[EB/OL]. 2012-08-27. http://www.cdc.gov/listeria/outbreaks/cantaloupes-jensen-farms/index.html
|
| [7] |
European Food Safety Authority, European Centre for Disease Prevention and Control. The European Union summary report on trends and sources of zoonoses, zoonotic agents and food-borne outbreaks in 2014[J]. EFSA Journal, 2015, 13(12): 4329. |
| [8] |
Abee T, Wouters JA. Microbial stress response in minimal processing[J]. International Journal of Food Microbiology, 1999, 50(1/2): 65-91. |
| [9] |
Hill C, O'Driscoll B, Booth I. Acid adaptation and food poisoning microorganisms[J]. International Journal of Food Microbiology, 1995, 28(2): 245-254. DOI:10.1016/0168-1605(95)00060-7 |
| [10] |
Phan-Thanh L, Montagne A. Physiological and biochemical aspects of the acid survival of Listeria monocytogenes[J]. The Journal of General and Applied Microbiology, 1998, 44(3): 183-191. DOI:10.2323/jgam.44.183 |
| [11] |
Fang C. Comparative genomics and pathogenicity difference mechanisms of Listeria monocytogenes lineage Ⅲ virulent and low virulent strains[D]. Hangzhou: Doctoral Dissertation of Zhejiang University, 2015 (in Chinese) 方春. 单核细胞增生李斯特菌谱系Ⅲ强毒株与弱毒菌株比较基因组及致病力差异机制[D]. 杭州: 浙江大学博士学位论文, 2015 http://cdmd.cnki.com.cn/Article/CDMD-10335-1015643555.htm |
| [12] |
Cheng CY, Yang YC, Dong ZM, et al. Listeria monocytogenes varies among strains to maintain intracellular pH homeostasis under stresses by different acids as analyzed by a high-throughput microplate-based fluorometry[J]. Frontiers in Microbiology, 2015, 6: 15. |
| [13] |
Cotter PD, Hill C. Surviving the acid test: responses of gram-positive bacteria to low pH[J]. Microbiology and Molecular Biology Reviews, 2003, 67(3): 429-453. DOI:10.1128/MMBR.67.3.429-453.2003 |
| [14] |
O'Driscoll B, Gahan C, Hill C. Two-dimensional polyacrylamide gel electrophoresis analysis of the acid tolerance response in Listeria monocytogenes LO28[J]. Applied and Environmental Microbiology, 1997, 63(7): 2679-2685. |
| [15] |
O'Driscoll B, Gahan CG, Hill C. Adaptive acid tolerance response in Listeria monocytogenes: isolation of an acid-tolerant mutant which demonstrates increased virulence[J]. Applied and Environmental Microbiology, 1996, 62(5): 1693-1698. |
| [16] |
Faleiro ML, Andrew PW, Power D. Stress response of Listeria monocytogenes isolated from cheese and other foods[J]. International Journal of Food Microbiology, 2003, 84(2): 207-216. DOI:10.1016/S0168-1605(02)00422-1 |
| [17] |
Ferreira A, Sue D, O'Byrne CP, et al. Role of Listeria monocytogenes σB in survival of lethal acidic conditions and in the acquired acid tolerance response[J]. Applied and Environmental Microbiology, 2003, 69(5): 2692-2698. DOI:10.1128/AEM.69.5.2692-2698.2003 |
| [18] |
Lou YQ, Yousef AE. Adaptation to sublethal environmental stresses protects Listeria monocytogenes against lethal preservation factors[J]. Applied and Environmental Microbiology, 1997, 63(4): 1252-1255. |
| [19] |
Skandamis PN, Yoon Y, Stopforth JD, et al. Heat and acid tolerance of Listeria monocytogenes after exposure to single and multiple sublethal stresses[J]. Food Microbiology, 2008, 25(2): 294-303. DOI:10.1016/j.fm.2007.10.008 |
| [20] |
Bonnet M, Rafi MM, Chikindas ML, et al. Bioenergetic mechanism for nisin resistance, induced by the acid tolerance response of Listeria monocytogenes[J]. Applied and Environmental Microbiology, 2006, 72(4): 2556-2563. DOI:10.1128/AEM.72.4.2556-2563.2006 |
| [21] |
Sebald W, Friedl P, Schairer HU, et al. Structure and genetics of the H+-conducting F0 portion of the ATP synthase[J]. Annals of the New York Acadamy of Sciences, 1982, 402: 28-44. DOI:10.1111/nyas.1982.402.issue-1 |
| [22] |
Cotter PD, Gahan CG, Hill C. A glutamate decarboxylase system protects Listeria monocytogenes in gastric fluid[J]. Molecular Microbiology, 2001, 40(2): 465-475. DOI:10.1046/j.1365-2958.2001.02398.x |
| [23] |
Cheng CY, Chen JS, Shan Y, et al. Listeria monocytogenes ArcA contributes to acid tolerance[J]. Journal of Medical Microbiology, 2013, 62(6): 813-821. |
| [24] |
Cheng CY, Chen JS, Fang C, et al. Listeria monocytogenes aguA1, but not aguA2, encodes a functional agmatine deiminase: Biochemical characterization of its catalytic properties and roles in acid tolerance[J]. Journal of Biological Chemistry, 2013, 288(37): 26606-26615. DOI:10.1074/jbc.M113.477380 |
| [25] |
Cotter PD, Gahan CGM, Hill C. Analysis of the role of the Listeria monocytogenes F0F1-ATPase operon in the acid tolerance response[J]. International Journal of Food Microbiology, 2000, 60(2/3): 137-146. |
| [26] |
Krulwich TA, Sachs G, Padan E. Molecular aspects of bacterial pH sensing and homeostasis[J]. Nature Reviews Microbiology, 2011, 9(5): 330-343. DOI:10.1038/nrmicro2549 |
| [27] |
Datta AR, Benjamin MM. Factors controlling acid tolerance of Listeria monocytogenes: effects of nisin and other ionophores[J]. Applied and Environmental Microbiology, 1997, 63(10): 4123-4126. |
| [28] |
Karatzas KAG, Suur L, O'Byrne CP. Characterization of the intracellular glutamate decarboxylase system: analysis of its function, transcription, and role in the acid resistance of various strains of Listeria monocytogenes[J]. Applied and Environmental Microbiology, 2012, 78(10): 3571-3579. DOI:10.1128/AEM.00227-12 |
| [29] |
Feehily C, Finnerty A, Casey PG, et al. Divergent evolution of the activity and regulation of the glutamate decarboxylase systems in Listeria monocytogenes EGD-e and 10403S: roles in virulence and acid tolerance[J]. PLoS One, 2014, 9(11): e112649. DOI:10.1371/journal.pone.0112649 |
| [30] |
Karatzas KAG, Brennan O, Heavin S, et al. Intracellular accumulation of high levels of γ-aminobutyrate by Listeria monocytogenes 10403S in response to low pH: uncoupling of γ-aminobutyrate synthesis from efflux in a chemically defined medium[J]. Applied and Environmental Microbiology, 2010, 76(11): 3529-3537. DOI:10.1128/AEM.03063-09 |
| [31] |
Ryan S, Begley M, Gahan CG, et al. Molecular characterization of the arginine deiminase system in Listeria monocytogenes: regulation and role in acid tolerance[J]. Environmental Microbiology, 2009, 11(2): 432-445. DOI:10.1111/emi.2009.11.issue-2 |
| [32] |
Chen JS, Jiang LL, Chen QM, et al. Lmo0038 is involved in acid and heat stress responses and specific for Listeria monocytogenes lineages Ⅰ and Ⅱ, and Listeria ivanovii[J]. Foodborne Pathogens and Disease, 2009, 6(3): 365-376. DOI:10.1089/fpd.2008.0207 |
| [33] |
Chen JS, Cheng CY, Xia Y, et al. Lmo0036, an ornithine and putrescine carbamoyltransferase in Listeria monocytogenes, participates in arginine deiminase and agmatine deiminase pathways and mediates acid tolerance[J]. Microbiology, 2011, 157(11): 3150-3161. DOI:10.1099/mic.0.049619-0 |
| [34] |
van Schaik W, Abee T. The role of σB in the stress response of Gram-positive bacteria-targets for food preservation and safety[J]. Current Opinion in Biotechnology, 2005, 16(2): 218-224. DOI:10.1016/j.copbio.2005.01.008 |
| [35] |
Ferreira A, O'Byrne CP, Boor KJ. Role of σB in heat, ethanol, acid, and oxidative stress resistance and during carbon starvation in Listeria monocytogenes[J]. Applied and Environmental Microbiology, 2001, 67(10): 4454-4457. DOI:10.1128/AEM.67.10.4454-4457.2001 |
| [36] |
Raengpradub S, Wiedmann M, Boor KJ. Comparative analysis of the σB-dependent stress responses in Listeria monocytogenes and Listeria innocua strains exposed to selected stress conditions[J]. Applied and Environmental Microbiology, 2008, 74(1): 158-171. DOI:10.1128/AEM.00951-07 |
| [37] |
Hecker M, Pané-Farré J, Uwe V. SigB-dependent general stress response in Bacillus subtilis and related gram-positive bacteria[J]. Annual Review of Microbiology, 2007, 61: 215-236. DOI:10.1146/annurev.micro.61.080706.093445 |
| [38] |
Shin JH, Brody MS, Pricet CW. Physical and antibiotic stresses require activation of the RsbU phosphatase to induce the general stress response in Listeria monocytogenes[J]. Microbiology, 2010, 156(9): 2660-2669. DOI:10.1099/mic.0.041202-0 |
| [39] |
Chaturongakul S, Boor KJ. RsbT and RsbV contribute to σB-dependent survival under environmental, energy, and intracellular stress conditions in Listeria monocytogenes[J]. Applied and Environmental Microbiology, 2004, 70(9): 5349-5356. DOI:10.1128/AEM.70.9.5349-5356.2004 |
| [40] |
Xia Y, Xin YP, Li XL, et al. To modulate survival under secondary stress conditions, Listeria monocytogenes 10403S employs RsbX to down-regulate σB activity in the post-stress recovery stage or stationary phase[J]. Applied and Environmental Microbiology, 2016, 82(4): 1126-1135. DOI:10.1128/AEM.03218-15 |
| [41] |
Palmer ME, Chaturongakul S, Wiedmann M, et al. The Listeria monocytogenes σB regulon and its virulence-associated functions are inhibited by a small molecule[J]. mBio, 2011, 2(6): e00241-11. |
| [42] |
Ringus DL, Gaballa A, Helmann JD, et al. Fluoro-phenyl-styrene-sulfonamide, a novel inhibitor of σB activity, prevents the activation of σB by environmental and energy stresses in Bacillus subtilis[J]. Journal of Bacteriology, 2013, 195(11): 2509-2517. DOI:10.1128/JB.00107-13 |
| [43] |
Cheng CY, Dong ZM, Han X, et al. Listeria monocytogenes 10403S arginine repressor ArgR finely tunes arginine metabolism regulation under acidic conditions[J]. Frontoers in Microbiology, 2017, 8: 145. |
| [44] |
Barriuso-Iglesias M, Barreiro C, Sola-Landa A, et al. Transcriptional control of the F0F1-ATP synthase operon of Corynebacterium glutamicum: SigmaH factor binds to its promoter and regulates its expression at different pH values[J]. Microbial Biotechnology, 2013, 6(2): 178-188. DOI:10.1111/1751-7915.12022 |
| [45] |
Chan YC, Hu YW, Chaturongakul S, et al. Contributions of two-component regulatory systems, alternative σ factors, and negative regulators to Listeria monocytogenes cold adaptation and cold growth[J]. Journal of Food Protection, 2008, 71(2): 420-425. DOI:10.4315/0362-028X-71.2.420 |
| [46] |
Bowman JP, Hages E, Nilsson RE, et al. Investigation of the Listeria monocytogenes Scott A acid tolerance response and associated physiological and phenotypic features via whole proteome analysis[J]. Journal of Proteome Research, 2012, 11(4): 2409-2426. DOI:10.1021/pr201137c |
| [47] |
Kazmierczak MJ, Mithoe SC, Boor KJ, et al. Listeria monocytogenes σB regulates stress response and virulence functions[J]. Journal of Bacteriology, 2003, 185(19): 5722-5734. DOI:10.1128/JB.185.19.5722-5734.2003 |
| [48] |
Cotter PD, Ryan S, Gahan CGM, et al. Presence of GadD1 glutamate decarboxylase in selected Listeria monocytogenes strains is associated with an ability to grow at low pH[J]. Applied and Environmental Microbiology, 2005, 71(6): 2832-2839. DOI:10.1128/AEM.71.6.2832-2839.2005 |
| [49] |
Gao R, Stock AM. Biological insights from structures of two-component proteins[J]. Annual Review of Microbiology, 2009, 63: 133-154. DOI:10.1146/annurev.micro.091208.073214 |
| [50] |
Cotter PD, Emerson N, Gahan CGM, et al. Identification and disruption of lisRK, a genetic locus encoding a two-component signal transduction system involved in stress tolerance and virulence in Listeria monocytogenes[J]. Journal of Bacteriology, 1999, 181(21): 6840-6843. |
| [51] |
Pöntinen A, Lindström M, Skurnik M, et al. Screening of the two-component-system histidine kinases of Listeria monocytogenes EGD-e. LiaS is needed for growth under heat, acid, alkali, osmotic, ethanol and oxidative stresses[J]. Food Microbiology, 2017, 65: 36-43. DOI:10.1016/j.fm.2017.01.018 |
| [52] |
Paudyal R, Karatzas KAG. Stress adaptation of Listeria monocytogenes in acidic ready-to-eat products[J]. Amsterdam: Elsevier, 2016, 167-182. |
 2017, Vol. 44
2017, Vol. 44