扩展功能
文章信息
- 杨登辉, 孔里程, 孙建和, 严亚贤
- YANG Deng-Hui, KONG Li-Cheng, SUN Jian-He, YAN Ya-Xian
- 密度感应系统:对细菌致病力的自行调控
- Quorum sensing: an auto regulator for bacterial pathogenicity
- 微生物学通报, 2017, 44(12): 3007-3014
- Microbiology China, 2017, 44(12): 3007-3014
- DOI: 10.13344/j.microbiol.china.170524
-
文章历史
- 收稿日期: 2017-07-12
- 接受日期: 2017-09-11
- 优先数字出版日期(www.cnki.net): 2017-09-26
密度感应(Quorum sensing,QS)是一种密度依赖性调控机制,其基于细菌间产生的信号分子——自诱导物(Autoinducer,AI)进行交流,信号分子会随着细菌密度的增加而在环境中累积,当达到一定阈值时被细菌感应和摄取,进而对细菌的一系列性状进行调控[1]。其中生物被膜形成、毒力基因表达以及对噬菌体抗性作为病原菌致病力和抵抗力的决定性因素与病原菌防控密切相关,因此了解QS系统的调控机制意义重大。本文将重点综述细菌密度感应系统对细菌致病及耐受相关功能的调控机制,以期为防控病原菌感染提供新的思路与策略。
1 QS系统信号分子的多样性及传导机制的复杂性不同种类细菌的QS系统存在差异,其调控菌群性状依赖的信号分子及其合成、传导机制可能不同。根据信号分子的不同,QS系统主要分为四类:自诱导肽(autoinducing peptides,AIP)介导的革兰氏阳性菌QS系统;酰基高丝氨酸内酯(Acylhomoserine lactones,AHL)类物质介导的革兰氏阴性菌QS系统;自诱导物-2 (Autoinducer 2,AI-2)介导的革兰氏阳性菌及阴性菌共有的LuxS/AI-2型QS系统;能够感受真核生物和原核生物跨“界”信号分子的AI-3型QS系统[2]。
1.1 AIP介导的QS系统信号分子AIP是一类主要由革兰氏阳性菌产生的短肽,随着细菌密度的增长,细菌QS系统合成AIP前体(proAIP),在细胞膜处经过加工、修饰形成具有稳定结构的活性物质后,利用ABC (ATP-binding-cassette)转运系统转运至胞外,并在胞外累积[3]。当AIP浓度超过一定阈值时,会被位于细胞膜上的双组分系统识别并与之结合,进而将磷酸基团传递给胞质相应调节蛋白,使其磷酸化并激活调控功能,调控QS相关基因的表达[4]。这种QS系统操纵子基因的表达受AIP正反馈调控,有利于菌群接受信号后作出快速有效的群体反应[5]。
1.2 AHL介导的QS系统AHL类物质作为细菌种内交流的信号分子,是一种由革兰氏阴性菌QS系统以S-腺苷甲硫氨酸(S-adenosylmethionine,SAM)和酰基载体蛋白(Acyl carrier protein,ACP)为底物在LuxI催化作用下合成的AI分子。AHL在细胞内外的进出属于自由扩散,不需要ABC转运系统。因此,当AHL随细菌浓度的升高抵达阈值后会与胞内受体蛋白LuxR结合,进而LuxR作为转录因子对QS系统基因的表达及细菌其他性状进行调控[6]。目前已在100多种革兰氏阴性菌中发现了LuxI/LuxR的同源蛋白,鉴于酰基侧链的不同,不同细菌分泌的AHL具有一定的特异性,使其能够在一定程度上特异地与同源LuxR结合,形成种内交流的基础。然而AHL分子的主体骨架是由相同的高丝氨酸内酯(Homoserine lactones,HSL)构成[7],因此,某些AHL分子可以作为一种“通用货币”在不同细菌中流通,发挥交互调控作用。
1.3 AI-2介导的QS系统AI-2是一类广泛存在于革兰氏阳性菌和阴性菌并以(4S)-4, 5-二羟基-2, 3-戊二酮[(4S)-4, 5-dihydroxy-2, 3-pentanedione,DPD]为前体的信号分子,在细菌种间信息传递中具有重要作用。与前两种QS系统相比,AI-2介导的QS系统中AI-2信号分子的合成及传导机制相对复杂,AI-2的形成需经过3个酶促反应。SAM作为初始底物在甲基化酶的催化作用下,甲基被转移至其他底物,从而形成毒性副产物S-腺苷高半胱氨酸(S-adenosylhomocysteine,SAH);SAH在S-核苷酶Pfs催化下,腺嘌呤被裂解形成毒性较弱的S-核糖同型半胱氨酸(S-ribosylhomocysteine,SRH);SRH进一步被LuxS蛋白催化水解生成DPD和同型半胱氨酸;DPD在水中不稳定,会自发进行化学键重排形成不同构象的分子,这些分子被统称为AI-2[8]。目前已经发现(2S, 4S)-2-甲基-2, 3, 3, 4-四羟基四氢呋喃(S-THMF)和(2R, 4S)-2-甲基-2, 3, 3, 4-四羟基四氢呋喃(R-THMF)两种构象可以相互转变的AI-2分子,分别可被哈维弧菌(Vibrio harveyi) LuxP型受体和鼠伤寒沙门菌(Salmonella typhimurium) LsrB型受体识别,通过信号传导调控细菌的生物学性状[9-10]。
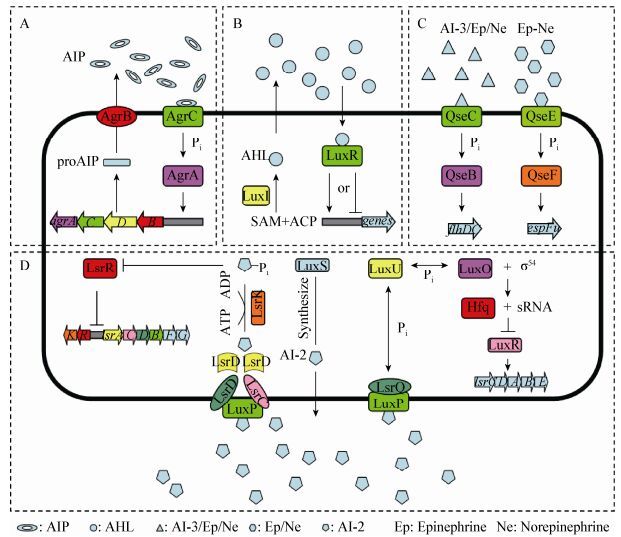
1.4 AI-3型QS系统AI-3型QS系统是以AI-3、肾上腺素和去甲肾上腺素介导对细菌毒力具有调控作用的QS系统。目前AI-3分子的化学结构及产生机制均未得到阐明,但研究发现肾上腺素和去甲肾上腺素能够替代AI-3发挥调控作用。细菌与真核生物不同,细胞膜上没有G蛋白偶联受体,只能通过双组分系统QseBC和QseEF接收传递AI-3型QS系统的信号分子[2, 11]。目前已在至少25种动植物病原菌中发现了QseBC双组分系统,该系统参与鞭毛调控子的转录激活,从而影响细菌的运动性。QseEF双组分系统主要存在于肠道细菌中,能够间接调控毒力效应分子espFu的转录[12-13]。四种QS系统信号传导过程如图 1所示。
|
| 图 1 四种QS系统信号传导过程 Figure 1 Signal transduction processes of QS systems 注:A:AIP介导的QS系统,AIP前体由agrD基因编码,转运至细胞膜时AgrB蛋白对其加工修饰,形成直链或环化的稳定结构并分泌至胞外,当胞外AIP累积到足够浓度后激活AgrCA双组分系统调控基因表达[14]. B:AHL介导的QS系统,LuxI及其同源蛋白负责催化SAM的高半胱氨酸基团与ACP的酰基侧链结合,并通过内酯化形成能够自由扩散的AHL,AHL随细菌浓度的升高抵达阈值后与胞内受体蛋白LuxR结合,进而LuxR作为转录因子对QS系统基因的表达及细菌其他性状进行调控[15]. C:AI-3型QS系统,QseC作为传感激酶,能够结合AI-3型QS系统信号分子,受体蛋白QseB磷酸化,对鞭毛调控子flhDC的转录起到调控作用[16];QseE为传感激酶,QseF为反应调控蛋白能够间接调控毒力效应分子espFu的转录[12]. D:AI-2介导的QS系统,LuxS蛋白负责催化合成AI-2前体DPD,后者自发结构重排形成AI-2,LsrB与AI-2结合后通过由LsrC与LsrD组成的异二聚体跨膜通道,LsrA通过水解ATP为转运AI-2提供能量;AI-2进入胞浆后被磷酸激酶LsrK磷酸化,经过LsrF和LsrG加工后的磷酸化AI-2可以与lsrACDBFGE操纵子的抑制蛋白LsrR结合消除LsrR蛋白的抑制作用,启动lsr操纵子的转录[17];LuxP是一种周质蛋白,作为AI-2的受体与LuxQ蛋白形成跨膜复合结构;当AI-2处于低浓度时,LuxQ蛋白磷酸化,并将磷酸化信号通过LuxU传递至LuxO蛋白,磷酸化的LuxO蛋白协同可变因子σ54激活sRNA转录,sRNA可与伴侣蛋白Hfq形成复合物破坏lux操纵子的转录调控蛋白LuxR的转录,从而抑制lux操纵子的转录[18]. Note: A: AIP mediated QS system. The proAIP, encoded by agrD, is first processed into active AIP in linear or annular structure by AgrB transporter and then releases into extracellular environment. Once the concentration of AIP reaches to the threshold, it activates AgrCA two-component system (TCS) to subsequently modify gene expression[14]. B: AHL mediated QS system. Synthesis of AHL requiring SAM and ACP is catalyzed by LuxI and its homologue, and then diffuses into and out of bacterial cells. Once the AHL has bound to LuxR, LuxR can be activated to regulate gene expression[15]. C: Type AI-3 QS system. As sensor kinases, QesC and QseE can sense type AI-3 signals and then phosphorylate QseB and QseF which are capable of modifying gene expression[12]. D: AI-2 mediated QS system. The DPD is the precursor of AI-2 which is produced by the enzyme LuxS. LsrB can directly bind AI-2 and is required for AI-2 transportation into cells with the assistance of LsrF and LsrG which compose transmembrane channel. LsrA is capable of catalyzing hydrolysis of ATP to provide energy for AI-2 transport. LsrK is a kinase that phosphorylates AI-2 once the AI-2 has entered cytoplasm. Phospho-AI-2 then directly binds the repressor LsrR, thus relieving the repression of the lsrACDBFGE operon by LsrR[17]. Two proteins, LuxP and LuxQ, function together as the sensor for AI-2 and LuxP is a soluble periplasmic protein resembling the ribose and galactose binding proteins. At low cell density, LuxQ acts as a kinase and transfers phosphorylation signal to the shared phosphotransferase protein LuxU that conveys it to the σ54-dependent response regulator LuxO, which activates the expression of small regulatory RNAs (sRNAs). The complexes of these sRNAs and chaperone protein Hfq destabilizes the mRNA of master regulator LuxR, thereby repressing the transcription of the lux operon[18]. |
|
|
随着转录组学和蛋白组学的应用,发现QS系统不仅发挥局部调节功能,更是一类全局性调控系统,参与调控细菌众多生物学性状。其中QS系统可调控细菌抵抗噬菌体感染是新发现,预计也将是未来数年研究的热点[19]。对QS调控机制的研究不仅有利于细菌治疗药物新靶位的筛选,也能为减毒疫苗和针对QS信号分子的新型疫苗的研制提供理论依据。
2.1 QS系统调控毒力基因表达病原菌通常通过某些毒力基因的表达产生对宿主的致病力,如产气荚膜梭菌(Clostridium perfringens)的穿孔素编码基因、大肠杆菌(Escherichia coli)的LEE毒力岛、沙门菌(Salmonella)的Ⅵ型分泌系统相关基因等,这些毒力基因不仅与致病菌的致病力密切相关,而且受不同类型QS系统的调控[19-21]。
产气荚膜梭菌的毒力基因受AIP介导QS系统的显著调控。其毒力基因netB编码一种穿孔毒素NetB,是造成鸡坏死性肠炎(Necrotic enteritis,NE)的重要因素。研究发现AIP前体加工蛋白ArgB能够通过一种扩散性信号分子正向调控鸡坏死性肠炎相关毒素蛋白NetB的表达,从而对机体产生致病力。不仅如此,AIP介导的QS系统还可调控磷脂代谢、黏附等相关基因的表达,这些基因可能与NE有关[3]。
由AHL介导的QS系统同样能够调控毒力基因的表达。早在2000年,Kanamaru等发现SdiA作为LuxR的同源蛋白,能够抑制肠出血性大肠杆菌O157:H7毒力因子EspD和黏附素的表达[22]。胞外信号分子能与SdiA蛋白N端结合改变SdiA构象,使SdiA蛋白C端与esp和eae基因的启动子区结合,抑制毒力基因的转录。2013年,Nguyen等发现肠出血性大肠杆菌O157:H7也能够通过感受结肠炎耶尔森菌释放的AHL分子,降低毒力基因LEE的表达[23]。
Ⅵ型分泌系统(Type Ⅵ secretion system,T6SS)作为病原菌重要的毒力因子,能够通过分泌效应蛋白,发挥阻止巨噬细胞对细菌吞噬、辅助细菌生物被膜形成并增强细菌种间竞争能力等多种生物学功能[24]。在霍乱弧菌(Vibrio cholerae)中,T6SS表达受AI-2介导的QS系统严格调控,Hcp蛋白作为T6SS的主要构成元件之一,LuxS蛋白的缺失会导致Hcp表达量显著下降[25]。
AI-3型QS系统QseBC对毒力基因同样具有调控作用,AI-3对LEE操纵子以及QseBC对鞭毛基因的调控作用也在大肠杆菌中得到证实[16]。
虽然QS系统调控细菌毒力基因表达的报道很多,但具体的分子调控机制却鲜有阐明。QS系统对毒力基因的调控不仅体现在信号分子受体与毒力基因作用从而激活或抑制其表达上,更是基于信号分子介导、多基因参与的复杂调控过程。调控机制虽然是研究的难点,但研究结果将为降低病原菌致病力提供更多可能的作用靶位,应该加大相关领域的攻关力度。
2.2 QS系统调控生物被膜形成生物被膜是一类由固着于生物或非生物表面的微生物及其胞外分泌物构成的复杂膜样结构。与浮游状态的细菌相比,生物被膜态细菌具有较强的耐药性且不易清除。一方面生物被膜能够吸附部分抗菌药物的灭活酶,造成抗菌药物失活;另一方面生物被膜中可能形成对药物不敏感的持留菌;细菌形成的生物被膜还能够显著降低由细菌引起的免疫反应,逃避宿主的免疫[26-27]。生物被膜耐药性强且不易清除的特点使其成为潜在的感染源,并随着宿主免疫力的变化而造成反复感染,使抗感染治疗变得异常困难。QS系统对生物被膜的调控作用使其成为抗菌治疗的新思路。
目前已知多种细菌中QS系统能够调控生物被膜的形成。革兰氏阳性菌QS系统通过双组分系统感应并传导AIP,进而调控基因的表达。研究发现,金黄色葡萄球菌AIP信号分子介导的QS系统能够显著调控其生物被膜形成。QS系统编码释放的AIP信号分子达到阈值后能够通过AgrCA双组分系统激活RNAⅢ非编码小RNA的转录,RNAⅢ通过抑制黏附素基因表达减弱生物被膜的形成能力[28]。
AHL与生物被膜的关系最早被发现于铜绿假单胞菌(Pseudomonas aeruginosa),研究发现AHL合成蛋白LasI或受体蛋白LasR突变株均不能形成成熟的生物被膜,外源加入AHL信号分子后则能恢复LasI突变株生物被膜的形成,随后Schaber等发现AHL阴性铜绿假单胞菌临床分离株不能形成生物被膜[29],证实了AHL分子在生物被膜形成过程中的重要调控作用[30]。虽然在研究AHL调控机制时Davey等认为QS系统通过合成鼠李糖脂维持生物被膜的结构,但目前更深层的分子调控机制仍未完全阐明[31]。
LuxS/AI-2型QS系统同样在多种细菌中参与对生物被膜形成的调控。部分细菌AI-2的调控机制已研究得较为透彻,如霍乱弧菌通过LuxPO途径感受环境中AI-2浓度激活或者抑制QS系统主要调控蛋白HapR的表达,从而对生物被膜的形成进行调控[32];哈维弧菌(Vibrio harveyi) QS系统通过调控Ⅲ型分泌系统影响其生物被膜形成[33];大肠杆菌YncC蛋白能够促进生物被膜形成,并受到MqsR的正向调控,AI-2能够通过调控MqsR的表达影响生物被膜形成能力[34];表皮葡萄球菌(Staphylococcus epidermidis)通过AI-2增强ica操纵子和bhp基因的转录水平促进生物被膜的形成[35]。
QseBC双组分系统常作为级联效应组分发挥调控功能,其不仅在EHEC中通过感受AI-2分子调控毒力因子表达,在伴放线放线杆菌(Actinobacillus actinomycetemcomitans)中也能执行由AI-2介导的生物被膜调控功能[36]。
2.3 QS系统调控噬菌体感染噬菌体是生物圈中数量最庞大的微生物,达到细菌总数10倍之多,对环境中的微生物群体造成了巨大的选择压力。噬菌体通过吸附、基因注射、复制、转录、翻译、基因包装、颗粒组装、子代释放等一系列复杂过程感染并裂解细菌。近年来随着致病菌耐药谱的扩大,噬菌体作为治疗耐药菌感染的一种新方式,无疑具有重大意义[37]。然而面对噬菌体的威胁,细菌进化出多种防御机制来抵抗噬菌体的感染。因此,对细菌抵抗噬菌体感染机制的研究也逐渐成为热点,QS系统对抵抗噬菌体感染功能的调控作用则是近几年研究的新成果,目前发现其主要通过调控细菌的生物被膜形成、噬菌体受体表达、溶原性噬菌体释放以及CRISPR-Cas系统影响噬菌体的感染能力。
噬菌体感染的第一步通过与细菌表面某些受体结合实现对细菌的吸附,噬菌体吸附率的大小直接影响到对细菌的感染程度[38]。前文已经提到QS系统能够调控细菌生物被膜的形成,细菌形成的生物被膜结构紧密,能够部分隐藏噬菌体受体,因此QS系统能够通过调控生物被膜的形成对噬菌体产生一定的抵抗能力[39]。
细菌表面的噬菌体受体同样受到QS系统的调控。Høyland-Kroghsbo等发现,AHL介导的QS系统能够调控大肠杆菌K-12表面噬菌体受体蛋白的表达,进而影响大肠杆菌对噬菌体的易感程度。虽然大肠杆菌的QS系统没有LuxI同源蛋白,不能合成AHL信号分子,但SdiA作为LuxR的同源蛋白能够识别并结合外源的AHL分子,进而调控一系列基因的表达。AHL通过下调噬菌体受体蛋白LamB的表达,降低λ噬菌体的吸附率。在AHL存在时,χ噬菌体对大肠杆菌的吸附率同样显著降低[40]。因此可以推断,在复杂的微生物环境中,大肠杆菌可以通过QS系统感受外界AHL来应对噬菌体的威胁,从而不利于噬菌体治疗方案的执行。在霍乱弧菌中,加入外源AI-2能够下调O抗原合成基因的表达水平,通过噬菌体受体O抗原的减少导致噬菌体对霍乱弧菌吸附率的显著降低[41]。
噬菌体虽然给细菌带来了生存压力,但同时也能为增强细菌致病力做贡献。一些温和噬菌体感染细菌后能够将毒力基因整合至细菌基因组,提高细菌的致病力[42]。Rossmann等研究发现,QS系统与温和噬菌体的释放有关。外源加入高浓度AI-2会导致多重溶原粪肠球菌(Enterococcus faecalis)噬菌体的释放和已形成的生物被膜消失,利用AI-2诱导的噬菌体上清液感染粪肠球菌益生菌,能够导致粪肠球菌益生菌生物被膜形成和致病力的显著提高。因此,在针对QS系统治疗方案的设计中,如何平衡生物被膜的形成和由噬菌体介导的毒力基因的转移是也值得考虑的问题[43]。
CRISPR-Cas系统作为细菌抵抗噬菌体等外源基因入侵的重要方式,显然对维持细菌的正常生理功能非常重要,那么其在细菌整个生命活动过程中是否一直处于激活状态?答案显然是否定的[44]。细菌在正常生理状态下,若CRISPR-Cas持续激活,需要大量消耗细菌微环境中的有限资源,是一种不合理、不经济的抗感染模式[45-47];同时,持续激活的CRISPR-Cas容易导致自身免疫(Auto-immunity)的发生,造成细菌死亡[44]。那么细菌如何激活和有效调控CRISPR-Cas的功能?这是该领域科学家普遍关注但仍未阐明的重要科学问题。本课题组在研究CRISPR-Cas机制时发现,高密度时大肠杆菌对噬菌体抵抗力较低密度时显著增强,同时发现hns基因缺失的大肠杆菌其生物被膜的形成能力显著改变,CRISPR-Cas的功能被有效激活,而生物被膜的形成能力通常与细菌的QS系统密切相关,该结果进一步间接提示大肠杆菌的密度感应系统可能与CRISPR-Cas的激活具有相关性[48-49]。那么该现象是否与QS系统相关?无独有偶,这一推测在2016年底和2017年初被两个研究团队分别在沙雷菌(Serratia)和铜绿假单胞菌中的研究所证实[50-51]。研究发现,QS系统对细菌CRISPR-Cas系统具有调控作用。沙雷菌LuxIR的同源蛋白SmaI和SmaR能够通过调控cas操纵子的表达影响细菌CRISPR的干扰过程。在缺失AHL合成基因smaI后,SmaR作为抑制蛋白能够抑制cas操纵子的转录,在smaI缺失的基础上外源添加AHL分子或缺失smaR基因能够使cas操纵子转录水平恢复[50]。同样,铜绿假单胞菌在高密度状态时也能通过QS系统上调cas基因的表达,增强CRISPR-Cas对外源DNA入侵的抵抗力及对间隔序列spacer的整合能力[51],而且更为关键的是,在QS信号通路缺失或阻断的情况下,CRISPR-Cas的适应性免疫功能会受到显著抑制,进一步说明QS系统在CRISPR-Cas的激活上具有不可或缺的作用。因此,可以推测包括大肠杆菌在内的大多数细菌能够通过QS信号分子交流,在细菌处于高密度易被噬菌体等外源DNA入侵时,激活其CRISPR-Cas适应性免疫系统,实现细菌内部的高效、低成本、精准调控。尽管如此,国内外对QS调控CRISPR-Cas激活的分子基础、调控途径和具体机制均未系统阐明[51]。
3 展望综上所述,革兰氏阳性菌和阴性菌都能利用QS系统来感知周围环境中的信号分子,从而调控整个菌群的行为。随着研究的深入,发现越来越多的生物学性状与QS系统相关,其中致病力和抵抗力尤为引人关注。一些细菌不仅能够利用QS系统促进毒力因子的表达,促进生物被膜的形成产生耐药性,促进携带毒力基因溶原噬菌体的释放再感染导致非致病菌向致病菌的转化,甚至利用QS信号分子提高对噬菌体的抵抗力。与此同时,在耐药基因水平转移频发、耐药病原菌不断出现、抗生素治疗面临挑战的情况下,噬菌体治疗策略成为人们研究的重点。然而噬菌体治疗不仅要面对由生物被膜产生的噬菌体抵抗力,同时还要面对细菌适应性免疫机制——CRISPR-Cas系统。幸运的是,目前针对QS系统的研究成果可能为改善该现状提供了新的思路。虽然QS系统调控病原菌致病力及抵抗力是一个复杂的过程,众多基因涉及其中,然而一旦揭示了QS调控CRISPR-Cas激活的分子机制,不仅在理论上有所突破,而且为筛选、应用免疫抑制剂,降低病原菌的适应性免疫能力,提高治疗性噬菌体的感染效率,防控耐药性病原菌感染提供理论依据,既具有重要的理论意义又具有应用潜力。在阐明QS调控分子机制的基础上,从抑制QS信号分子产生、阻断信号分子传递和抑制信号分子受体等角度,减弱或消除QS系统对细菌致病力和抵抗力的促进作用能够一举两得地提高对病原菌的治疗效果。目前已发现多种动植物、微生物分泌或人工合成的QS抑制物,如人体产生的LL-37肽、铜绿假单胞菌产生的的AHL内酯酶、海藻产生的卤代呋喃酮等均能有效阻断QS信号的传导[52-54]。未来对于QS系统的研究可以从阐明分子机制和QS信号分子阻断药物入手,通过将抗菌药物与噬菌体联合使用形成新型的有效的病原菌防控模式。
| [1] |
Bassler BL, Losick R. Bacterially speaking[J]. Cell, 2006, 125(2): 237-246. DOI:10.1016/j.cell.2006.04.001 |
| [2] |
Kendall MM, Sperandio V. Quorum sensing by enteric pathogens[J]. Current Opinion in Gastroenterology, 2007, 23(1): 10-15. DOI:10.1097/MOG.0b013e3280118289 |
| [3] |
Yu Q, Lepp D, Mehdizadeh GI, et al. The Agr-like quorum sensing system is required for necrotic enteritis pathogenesis in poultry caused by Clostridium perfringens in Poultry[J]. Infection and Immunity, 2017, 85(6): e00975-16. |
| [4] |
Hoch JA. Two-component and phosphorelay signal transduction[J]. Current Opinion in Microbiology, 2000, 3(2): 165-170. DOI:10.1016/S1369-5274(00)00070-9 |
| [5] |
Rutherford ST, Bassler BL. Bacterial Quorum Sensing: Its role in virulence and possibilities for its control[J]. Cold Spring Harbor Perspectives in Medicine, 2012, 2(11): a012427. |
| [6] |
Suchetha A, Jayachandran C, Darshan BM, et al. Quorum sensing-meetings in the microbial world[J]. Journal of Research in Medical and Dental Science, 2015, 3(3): 161-165. DOI:10.5455/jrmds. |
| [7] |
Gui M, Wu RY, Liu L, et al. Effects of quorum quenching by AHL lactonase on AHLs, protease, motility and proteome patterns in Aeromonas veronii LP-11[J]. International Journal of Food Microbiology, 2017, 252: 61-68. DOI:10.1016/j.ijfoodmicro.2017.04.005 |
| [8] |
Federle MJ, Bassler BL. Interspecies communication in bacteria[J]. Journal of Clinical Investigation, 2003, 112(9): 1291-1299. DOI:10.1172/JCI20195 |
| [9] |
Almasoud A, Hettiarachchy N, Rayaprolu S, et al. Inhibitory effects of lactic and malic organic acids on autoinducer type 2 (AI-2) quorum sensing of Escherichia coli O157:H7 and Salmonella Typhimurium[J]. Lwt-Food Science and Technology, 2016, 66: 560-564. DOI:10.1016/j.lwt.2015.11.013 |
| [10] |
Lorenz N, Shin JY, Jung K. Activity, abundance, and localization of quorum sensing receptors in Vibrio harveyi[J]. Frontiers in Microbiology, 2017, 8: 634. |
| [11] |
Weigel WA, Demuth DR. QseBC, a two-component bacterial adrenergic receptor and global regulator of virulence in Enterobacteriaceae and Pasteurellaceae[J]. Molecular Oral Microbiology, 2016, 31(5): 379-397. DOI:10.1111/omi.2016.31.issue-5 |
| [12] |
Reading NC, Torres AG, Kendall MM, et al. A novel two-component signaling system that activates transcription of an enterohemorrhagic Escherichia coli effector involved in remodeling of host actin[J]. Journal of Bacteriology, 2007, 189(6): 2468-2476. DOI:10.1128/JB.01848-06 |
| [13] |
Reading NC, Rasko D, Torres AG, et al. A transcriptome study of the QseEF two-component system and the QseG membrane protein in enterohaemorrhagic Escherichia coli O157:H7[J]. Microbiology, 2010, 156(4): 1167-1175. DOI:10.1099/mic.0.033027-0 |
| [14] |
Novick RP, Geisinger E. Quorum sensing in Staphylococci[J]. Annual Review of Genetics, 2008, 42(1): 541-564. DOI:10.1146/annurev.genet.42.110807.091640 |
| [15] |
Fuqua C, Parsek MR, Greenberg EP. Regulation of gene expression by cell-to-cell communication: Acyl-homoserine lactone quorum sensing[J]. Annual Review of Genetics, 2001, 35(1): 439-468. DOI:10.1146/annurev.genet.35.102401.090913 |
| [16] |
Clarke MB, Sperandio V. Transcriptional regulation of flhDC by QseBC and σ28 (FliA) in enterohaemorrhagic Escherichia coli[J]. Molecular Microbiology, 2005, 57(6): 1734-1749. DOI:10.1111/j.1365-2958.2005.04792.x |
| [17] |
Thijs IM, Zhao H, de Weerdt A, et al. The AI-2-dependent regulator LsrR has a limited regulon in Salmonella Typhimurium[J]. Cell Research, 2010, 20(8): 966-969. DOI:10.1038/cr.2010.104 |
| [18] |
Miller ST, Xavier KB, Campagna SR, et al. Salmonella typhimurium recognizes a chemically distinct form of the bacterial quorum-sensing signal Al-2[J]. Molecular Cell, 2004, 15(5): 677-687. DOI:10.1016/j.molcel.2004.07.020 |
| [19] |
Zaitseva YV, Popova AA, Khmel IA. Quorum sensing regulation in bacteria of the family enterobacteriaceae[J]. Russian Journal of Genetics, 2014, 50(4): 323-340. DOI:10.1134/S1022795414030120 |
| [20] |
Pezoa D, Andrews-Polymenis H, Blondel CJ, et al. The type Ⅵ secretion systems encoded in Salmonella pathogenicity islands SPI-6 and SPI-19 contribute to intestinal colonization of chickens and systemic spread by Salmonella enterica serotype Typhimurium[J]. FEBS Journal, 2012, 279: 227-228. |
| [21] |
Le KY, Otto M. Quorum-sensing regulation in Staphylococci-an overview[J]. Frontiers in Microbiology, 2015, 6: 1174. |
| [22] |
Kanamaru K, Kanamaru K, Tatsuno I, et al. SdiA, an Escherichia coli homologue of quorum-sensing regulators, controls the expression of virulence factors in enterohaemorrhagic Escherichia coli O157:H7[J]. Molecular Microbiology, 2000, 38(4): 805-816. DOI:10.1046/j.1365-2958.2000.02171.x |
| [23] |
Nguyen YN, Sheng HQ, Dakarapu R, et al. The acyl-homoserine lactone synthase YenI from Yersinia enterocolitica modulates virulence gene expression in enterohemorrhagic Escherichia coli O157:H7[J]. Infection and Immunity, 2013, 81(11): 4192-4199. DOI:10.1128/IAI.00889-13 |
| [24] |
Tian YL, Zhao YL, Shi LY, et al. Type Ⅵ secretion systems of Erwinia amylovora contribute to bacterial competition, virulence, and exopolysaccharide production[J]. Phytopathology, 2017, 107(6): 654-661. DOI:10.1094/PHYTO-11-16-0393-R |
| [25] |
Ishikawa T, Rompikuntal PK, Lindmark B, et al. Quorum sensing regulation of the two hcp alleles in Vibrio cholerae O1 strains[J]. PLoS One, 2009, 4(8): e6734. DOI:10.1371/journal.pone.0006734 |
| [26] |
Wang R. The significance of bacterial biofilm formation[J]. National Medical Journal of China, 2004, 84(22): 1915-1916. 王睿. 细菌生物被膜的产生与临床意义[J]. 中华医学杂志, 2004, 84(22): 1915-1916. DOI:10.3760/j:issn:0376-2491.2004.22.020 |
| [27] |
Jiang B, Li S, Hu XM. New progresses of research methods and achievements on persisters[J]. Journal of Medical Postgraduate, 2014, 27(6): 665-668. 姜北, 黎庶, 胡晓梅. 持留菌研究方法的新进展[J]. 医学研究生学报, 2014, 27(6): 665-668. |
| [28] |
Coelho LR, Souza RR, Ferreira FA, et al. agr RNAIII divergently regulates glucose-induced biofilm formation in clinical isolates of Staphylococcus aureus[J]. Microbiology, 2008, 154(11): 3480-3490. DOI:10.1099/mic.0.2007/016014-0 |
| [29] |
Schaber JA, Carty NL, McDonald NA, et al. Analysis of quorum sensing-deficient clinical isolates of Pseudomonas aeruginosa[J]. Journal of Medical Microbiology, 2004, 53(9): 841-853. DOI:10.1099/jmm.0.45617-0 |
| [30] |
Davies DG, Parsek MR, Pearson JP, et al. The involvement of cell-to-cell signals in the development of a bacterial biofilm[J]. Science, 1998, 280(5361): 295-298. DOI:10.1126/science.280.5361.295 |
| [31] |
Davey ME, Caiazza NC, O'Toole GA. Rhamnolipid surfactant production affects biofilm architecture in Pseudomonas aeruginosa PAO1[J]. Journal of Bacteriology, 2003, 185(3): 1027-1036. DOI:10.1128/JB.185.3.1027-1036.2003 |
| [32] |
Hammer BK, Bassler BL. Regulatory small RNAs circumvent the conventional quorum sensing pathway in pandemic Vibrio cholerae[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(27): 11145-11149. DOI:10.1073/pnas.0703860104 |
| [33] |
Henke JM, Bassler BL. Quorum sensing regulates type Ⅲ secretion in Vibrio harveyi and Vibrio parahaemolyticus[J]. Journal of Bacteriology, 2004, 186(12): 3794-3805. DOI:10.1128/JB.186.12.3794-3805.2004 |
| [34] |
Zhang XS, Garcia-Contreras R, Wood TK. Escherichia coli transcription factor YncC (McbR) regulates colanic acid and biofilm formation by repressing expression of periplasmic protein YbiM (McbA)[J]. The ISME Journal, 2008, 2(6): 615-631. DOI:10.1038/ismej.2008.24 |
| [35] |
Xue T, Ni JT, Shang F, et al. Autoinducer-2 increases biofilm formation via an ica-and bhp-dependent manner in Staphylococcus epidermidis RP62A[J]. Microbes and Infection, 2015, 17(5): 345-352. DOI:10.1016/j.micinf.2015.01.003 |
| [36] |
Novak EA, Shao HJ, Daep CA, et al. Autoinducer-2 and QseC control biofilm formation and in vivo virulence of Aggregatibacter actinomycetemcomitans[J]. Infection and Immunity, 2010, 78(7): 2919-2926. DOI:10.1128/IAI.01376-09 |
| [37] |
Wang ZF, Zheng PP, Ji WH, et al. SLPW: A virulent bacteriophage targeting methicillin-resistant Staphylococcus aureus in vitro and in vivo[J]. Frontiers in Microbiology, 2016, 7: 934. |
| [38] |
Azeredo J, Sutherland IW. The use of phages for the removal of infectious biofilms[J]. Current Pharmaceutical Biotechnology, 2008, 9(4): 261-266. DOI:10.2174/138920108785161604 |
| [39] |
Kim M, Ryu S. Characterization of a T5-like coliphage, SPC35, and differential development of resistance to SPC35 in Salmonella enterica serovar Typhimurium and Escherichia coli[J]. Applied and Environmental Microbiology, 2011, 77(6): 2042-2050. DOI:10.1128/AEM.02504-10 |
| [40] |
Høyland-Kroghsbo NM, Mærkedahl RB, Lo Svenningsen S. A quorum-sensing-induced bacteriophage defense mechanism[J]. mBio, 2013, 4(1): e00362-12. |
| [41] |
Hoque MM, Bin Naser I, Bari SMN, et al. Quorum regulated resistance of Vibrio cholerae against environmental bacteriophages[J]. Scientific Reports, 2016, 6(1): 37956. DOI:10.1038/srep37956 |
| [42] |
Cao DM, Ji WH, Fu Q, et al. Escherichia coli nfuA is essential for maintenance of shiga toxin phage Min27 lysogeny under iron-depleted condition[J]. FEMS Microbiology Letters, 2015, 362(19). DOI:10.1093/femsle/fnv149 |
| [43] |
Rossmann FS, Racek T, Wobser D, et al. Phage-mediated dispersal of biofilm and distribution of bacterial virulence genes is induced by quorum sensing[J]. PLoS Pathogens, 2015, 11(2): e1004653. DOI:10.1371/journal.ppat.1004653 |
| [44] |
Semenova E, Severinov K. Come together: CRISPR-Cas immunity senses the quorum[J]. Molecular Cell, 2016, 64(6): 1013-1015. DOI:10.1016/j.molcel.2016.11.037 |
| [45] |
Vale PF, Lafforgue G, Gatchitch F, et al. Costs of CRISPR-Cas-mediated resistance in Streptococcus thermophilus[J]. Proceedings of the Royal Society B-Biological Sciences, 2015, 282(1812): 20151270. DOI:10.1098/rspb.2015.1270 |
| [46] |
Westra ER, Van Houte S, Oyesiku-Blakemore S, et al. Parasite exposure drives selective evolution of constitutive versus inducible defense[J]. Current Biology, 2015, 25(8): 1043-1049. DOI:10.1016/j.cub.2015.01.065 |
| [47] |
Tan DM, Lo Svenningsen S, Middelboe M. Quorum sensing determines the choice of antiphage defense strategy in Vibrio anguillarum[J]. mBio, 2015, 6(3): e00627-15. |
| [48] |
Fu Q, Li SY, Wang ZF, et al. H-NS mutation-mediated CRISPR-Cas activation inhibits phage release and toxin production of Escherichia coli Stx2 phage lysogen[J]. Frontiers in Microbiology, 2017, 8: 652. |
| [49] |
Fu Q, Su ZX, Cheng YQ, et al. Clustered, regularly interspaced short palindromic repeat (CRISPR) diversity and virulence factor distribution in avian Escherichia coli[J]. Research in Microbiology, 2017, 168(2): 147-156. DOI:10.1016/j.resmic.2016.10.002 |
| [50] |
Patterson AG, Jackson SA, Taylor C, et al. Quorum sensing controls adaptive immunity through the regulation of multiple CRISPR-Cas systems[J]. Molecular Cell, 2016, 64(6): 1102-1108. DOI:10.1016/j.molcel.2016.11.012 |
| [51] |
Høyland-Kroghsbo NM, Paczkowski J, Mukherjee S, et al. Quorum sensing controls the Pseudomonas aeruginosa CRISPR-Cas adaptive immune system[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(1): 131-135. DOI:10.1073/pnas.1617415113 |
| [52] |
Overhage J, Campisano A, Bains M, et al. Human host defense peptide LL-37 prevents bacterial biofilm formation[J]. Infection and Immunity, 2008, 76(9): 4176-4182. DOI:10.1128/IAI.00318-08 |
| [53] |
Liu DL, Thomas PW, Momb J, et al. Structure and specificity of a quorum-quenching lactonase (AiiB) from Agrobacterium tumefaciens[J]. Biochemistry, 2007, 46(42): 11789-11799. DOI:10.1021/bi7012849 |
| [54] |
Rabin N, Delago A, Inbal B, et al. Tailor-made LasR agonists modulate quorum sensing in Pseudomonas aeruginosa[J]. Organic & Biomolecular Chemistry, 2013, 11(41): 7155-7163. |
 2017, Vol. 44
2017, Vol. 44