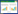扩展功能
文章信息
- 张奇文, 凌晨, 马勋, 杜冬冬, 钱凌霄, 李红欢
- ZHANG Qi-Wen, LING Chen, MA Xun, DU Dong-Dong, QIAN Ling-Xiao, LI Hong-Huan
- 单增李斯特菌新疆分离株lmo0160基因克隆及序列分析
- Cloning and sequence analysis of lmo0160 gene of Listeria monocytogenes strain from Xinjiang
- 微生物学通报, 2017, 44(12): 2905-2913
- Microbiology China, 2017, 44(12): 2905-2913
- DOI: 10.13344/j.microbiol.china.170495
-
文章历史
- 收稿日期: 2017-07-06
- 接受日期: 2017-09-11
- 优先数字出版日期(www.cnki.net): 2017-09-11
李斯特菌属包括单增李斯特菌(Listeria monocytogenes,LM或L. monocytogenes)、伊氏李斯特菌(L. ivanovii)、英诺克李斯特菌(L. innocua)、威尔斯李斯特菌(L. welshimeri)、西尔李斯特菌(L. seeligeri)和格氏李斯特菌(L. grayi) 6种菌[1],在李斯特菌属中,LM是唯一能引起人和动物发病的重要食源性人畜共患病病原菌。20世纪90年代,LM已被世界卫生组织(WHO)列为仅次于大肠杆菌O157、沙门氏菌、志贺氏菌后的第四大重要的食源性致病菌[2]。
LM是革兰氏阳性短杆菌,是一种常见的食源性致病菌,广泛分布于自然界中,有较强的环境适应性,可在低温、高盐、酸碱等不利的环境条件下生长繁殖[3],通过多种途径进入食品及食品加工环境。LM作为一种具有侵袭性的胞内寄生菌,可穿越宿主肠道屏障、胎盘屏障、血脑屏障,入侵宿主机体后可在吞噬细胞和非吞噬细胞内存活并大量增殖。临床上引起肠胃炎、脑膜炎、败血症、流产等症状,尤其对新生儿、妊娠母畜和免疫功能不全者的发病率和死亡率均较高[4]。LM的感染过程主要包括:抵抗宿主体内环境的应激、侵袭、感染及胞间扩散等,这些都与LM的一系列毒力因子表达及调控相关。
LM共表达2 853种蛋白,其中133种属于表面蛋白[5]。LM表面蛋白的数量和类型比其他任何细菌都丰富,这些表面蛋白在LM感染机体过程中起着重要的作用。其中有一类表面蛋白,其前体蛋白C端含有保守基序LPXTG (Leu-Pro-X-Thr-Gly),这些蛋白由分选酶Sort A通过识别LPXTG保守基序,将蛋白共价结合到细胞壁肽聚糖上,呈现在细胞表面,在感染机体过程中发挥重要毒力作用[6]。
对标准株EGD-e全基因组序列分析发现,该菌株含有41个LPXTG保守基序蛋白,但目前仅有部分蛋白的功能被研究。Popowska等[7]对Lmo0327的研究发现,该蛋白具有胞壁质酶水解活性,缺失后不会降低侵染和黏附人胚胎上皮细胞的水平,但在细胞分裂和胞壁质翻转中起到重要作用。Cummins等[8]通过小鼠口腔感染Lmo0842缺失株,明显减少了在小鼠肝脏和脾脏的载菌量,但并未证实Lmo0842缺失后对Caco-2细胞的侵袭具有增强的作用。Pucciarelli等[9]研究发现,Lmo0433 (InlA)与宿主受体E-cadherin特异性结合可介导LM对上皮细胞的黏附、侵入,提前终止InlA蛋白表达可显著降低LM对宿主细胞黏附和侵袭能力,证实了该蛋白结构完整性对黏附侵袭能力及致病力起到关键作用。表面蛋白Lmo0160是预测的41个蛋白之一,其功能目前尚未研究。本研究对LM90SB2菌株lmo0160基因进行PCR扩增、克隆及测序,并对其编码蛋白进行生物信息学分析,为探讨lmo0160基因在LM90SB2致病和生存中的功能奠定基础。
1 材料与方法 1.1 菌株和质粒单增李斯特菌LM90SB2分离自新疆某羊场患李斯特菌病绵羊脑组织,血清型为4b型;大肠杆菌(Escherichia coli) DH5α购自北京全式金生物技术有限公司。
1.2 主要试剂和仪器Trans5K DNA Marker、Pfu Polymerase、10×Pfu PCR buffer (含MgCl2),北京全式金生物技术有限公司;细菌基因组DNA提取试剂盒,天根生化科技(北京)有限公司;琼脂糖凝胶电泳回收试剂盒,北京诺维森生物科技有限公司;dNTP mix、ddH2O、pMD19-T载体宝生物工程(大连)有限公司;BHI培养基,青岛高科园海博生物技术有限公司;LB培养基(g/L):胰蛋白胨10.0,酵母提取物5.0,NaCl 10.0,pH 7.4。
生物安全柜,美国Baker公司;CR22E高速冷冻离心机,日本日立公司;隔水式电热恒温培养箱,北京永光明医疗仪器厂;PCR扩增仪,美国Thermo公司;电泳凝胶成像仪,美国Bio-Rad公司。
1.3 引物设计与合成参考菌株LM F2365 (GenBank登录号为AE017262)公布的基因序列,运用Primer 5.0软件设计lmo0160序列特异性引物,预期扩增片段长度为1 708 bp。lmo0160-F:5′-CGGAATTCATGAAGAAACGCACGACAAT-3′ (含EcoR Ⅰ酶切位点和保护碱基);lmo0160-R:5′-AACTGCAGAAATAGCTACGAGCAGTATTCCC-3′ (含Pst Ⅰ酶切位点和保护碱基)。引物由北京六合华大基因科技有限公司合成。
1.4 菌株培养及基因组DNA提取将实验室保存的LM90SB2菌株接种于BHI平板,37 ℃培养16−18 h,挑取单个菌落接种于BHI液体培养基中37 ℃、180 r/min培养16−18 h,用1.5 mL试管收集菌体,参考细菌基因组DNA提取试剂盒说明书提取LM90SB2全基因组DNA,于−20 ℃保存备用。
1.5 PCR扩增目的条带以LM90SB2全基因组DNA为模板,以lmo0160-F和lmo0160-R为上下游引物扩增目的片段。PCR反应体系(50 μL):10×Pfu PCR buffer (含MgCl2) 5.0 μL,dNTP mix (2.5 mmol/L) 2.0 μL,Pfu Polymerase (5 U/μL) 0.5 μL,上下游引物(10 µmol/L)各1.0 μL,DNA模板1.0 μL、ddH2O补足50 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 30 s,60 ℃ 30 s,72 ℃ 2.5 min,35个循环;72 ℃ 10 min。同时设置空白对照组。取7 μL扩增产物于1%琼脂糖凝胶电泳检测。
1.6 目的基因克隆及测序PCR扩增产物经琼脂糖凝胶DNA回收试剂盒回收后,与pMD19-T载体连接过夜,冷热激法将连接产物转化至大肠杆菌DH5α感受态细胞,涂布于含Ampr (100 μg/mL)的LB固体培养基上,37 ℃培养12−16 h。挑取单菌落通过菌液PCR筛选出阳性克隆菌送北京六合华大基因科技有限公司测序。
1.7 生物信息学分析用DNAMAN软件进行序列比对,用Mega 6.0软件构建不同来源菌株lmo0160基因系统进化树;利用ProtParam软件(http://web.expasy.org/protparam/)分析lmo0160基因序列的基本理化性质;利用SignalP 4.1 Server软件(http://www.cbs.dtu.dk/services/SignalP/)预测蛋白质的信号肽;利用TMHMM软件(http://www.cbs.dtu.dk/services/TMHMM-2.0/)分析蛋白质跨膜结构;利用TargetP 1.1 Server在线软件(http://www.cbs.dtu.dk/services/TargetP/)预测Lmo0160蛋白在细胞内的定位;利用SMART软件(http://smart.embl-heidelberg.de/smart/set_mode.cgi?NORMAL=1)预测蛋白质的结构域;利用SOPMA软件(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)和SWISS-MODEL软件(http://swissmodel.expasy.org/)预测蛋白质二级结构和三级结构。
2 结果与分析 2.1 lmo0160基因PCR扩增及克隆测序利用特异性引物扩增出lmo0160基因序列,经1%琼脂糖凝胶电泳检测得到约1 708 bp片段,目的条带清晰、单一,与预期片段大小一致(图 1)。经测序lmo0160基因序列长为1 708 bp,结果显示获得阳性转化子。在NCBI上对获得的lmo0160基因序列进行分析,该基因开放阅读框为1 428 bp,共编码475个氨基酸。
|
| 图 1 LM90SB2 lmo0160基因PCR扩增结果 Figure 1 Amplification product of lmo0160 gene from LM90SB2 by PCR 注:M:Trans5K DNA Marker;1、2:LM90SB2菌株lmo0160基因扩增产物. Note: M: Trans5K DNA Marker; 1, 2: lmo0160 gene amplification products of LM90SB2. |
|
|
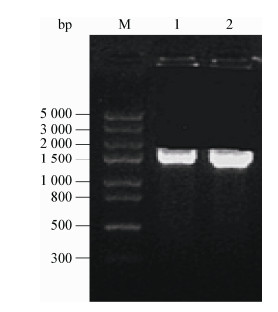
将克隆测序所得LM90SB2的lmo0160核苷酸序列与GenBank上公布的10株不同来源的lmo0160核苷酸序列进行同源性比对。结果显示,LM90SB2的lmo0160核苷酸序列与国际标准株EGD-e株(1/2a型)的相似性为87.2%;与F2365株(4b型,奶酪,美国)、NTSN株(4b型,绵羊脑,中国扬州)、CFSAN008100株(4b型,美国)、CFSAN023463株(4b型,美国)和J2-064株(4b型,美国)相似性均达到99.0%以上;与M7株(4a型,牛奶,中国浙江)相似性为97.2%;与Finland株(1/2a型,美国)、N53-1株(1/2a型,熟火腿,瑞士)和N1546株(1/2a型,鱼,丹麦)相似性均为91.1% (图 2)。
|
| 图 2 不同菌株lmo0160核苷酸和氨基酸序列与参考菌株同源性比对分析 Figure 2 Homology analysis of nucleotide and amino acid sequences of lmo0160 gene in various strains 注:右上角表示lmo0160基因核苷酸同源性,左下角表示lmo0160基因氨基酸同源性. Note: The right corner of lmo0160 nucleotide homology and the left corner of lmo0160 amino acid homology. |
|
|
利用DNAStar软件推导出LM90SB2的lmo0160编码的氨基酸序列,将其与10株不同来源lmo0160编码的氨基酸序列进行同源性比对。结果显示,LM90SB2的lmo0160编码的氨基酸序列与F2365株(4b型,美国)、CFSAN008100株(4b型,美国)、CFSAN023463株(4b型,美国)、J2-064株(4b型,美国)和NTSN株(4b型,中国扬州)相似性为99.2%–99.4%;与M7株(4a型,中国浙江)相似性为96.2%;与国际标准株EGD-e株(1/2a型)、美国Finland株(1/2a型)、瑞士N53-1株(1/2a型)和丹麦N1546株(1/2a型)相似性均为91.8% (图 2)。
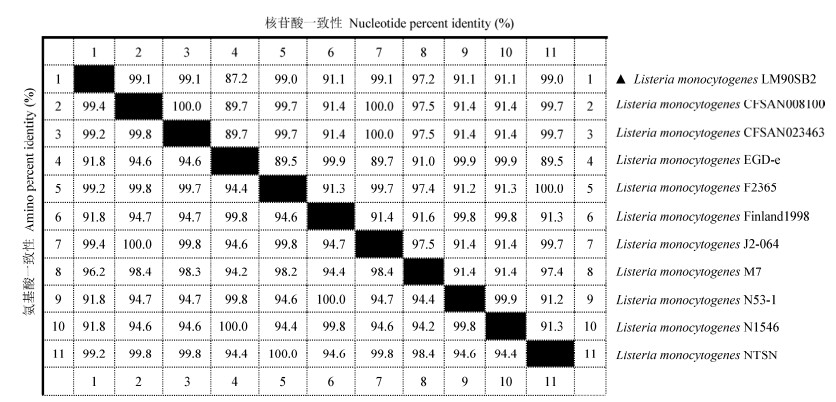
2.4 lmo0160基因序列的系统进化分析为了研究LM90SB2 lmo0160基因的分子进化关系,使用Mega 6.0软件,采用邻接法(Neighbor-Joining,NJ),利用自举分析(Bootstrap,1 000次重复)检验各分支的置信度,构建该基因及GenBank中检索到的10株其他来源基因编码区序列的分子系统进化树。如图 3所示,分子系统进化关系与血清型有密切关系,而与菌株来源关系不大。其中LM90SB2与4b血清型菌株(NTSN、F2365、CFSAN023463、J2-064和CFSAN008100)形成一分支,与中国M7株(4a型)聚类成一个类群,它们亲缘性关系较近;而1/2a血清型的国际标准株EGD-e株、Finland1998株、N1546株和N53-1株形成另一个分支,与LM90SB2株亲缘性关系较远。
|
| 图 3 LM90SB2的lmo0160核苷酸序列的系统进化树分析 Figure 3 Phylogenetic tree analysis of nucleotide sequences of lmo0160 gene of LM90SB2 注:括号中的序号表示GenBank登录号;分支上的数字表示Bootstrap检验值(> 60的Bootstrap值较为可信);标尺表示每单位核苷酸的变化(0.05表示在两个序列中有5%的变化). Note: The numbers in parenthesis represent GenBank accession numbers; The numbers on each branch indicate the bootstrap values (believable if > 60); Scale represents the nucleotide changes in per unit (0.05 means 5% changes were observed between two sequences). |
|
|
利用ExPASY在线软件中ProtParam工具分析LM90SB2的lmo0160基因编码蛋白的理化性质,该蛋白分子质量约为51.83 kD,其分子式为C2307H3603N589O761S2,含有51个强碱性氨基酸(R、H、K)、73个强酸性氨基酸(D、E)、174个疏水性氨基酸(A、I、L、M、F、P、W、V);极性氨基酸总数301个:177个极性不带电氨基酸(N、C、Q、G、S、T、Y)、51个极性带正电氨基酸(R、H、K)、73个极性带负电氨基酸(D、E);理论等电点(PI)为4.61;带负电荷的残基总数为73个,带正电荷的残基总数为45个;不稳定指数为19.89,根据Guruprasad方法表明Lmo0160蛋白为稳定性蛋白;脂溶指数为75.45,总疏水性平均数为−0.578。ProtScale在线工具进一步预测表明,Lmo0160氨基酸序列中疏水最大值为1.978,最小值为−3.000,大部分氨基酸属于亲水性氨基酸,可见该蛋白属于亲水性蛋白(图 4)。

|
| 图 4 LM90SB2的Lmo0160蛋白疏水性分析 Figure 4 Analysis hydrophobicity of Lmo0160 protein of LM90SB2 注:横轴为氨基酸位置,纵轴为疏水性得分;正值表示疏水性,负值表示亲水性. Note: The horizontal axis stands for the position of amino acids in this protein and vertical axis stands for the hydrophohicity. The positive value of vertical axis represent hydrophohicity and the negative value represent hydrophohicity. |
|
|
利用TMHMM 2.0 Server在线服务器分析Lmo0160蛋白跨膜结构域,结果显示Lmo0160蛋白无跨膜螺旋,均处于膜外,不存在跨膜结构。利用SignalP 4.1 Server在线服务器分析发现,该蛋白无信号肽序列。
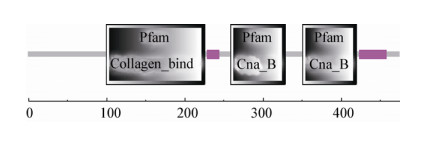
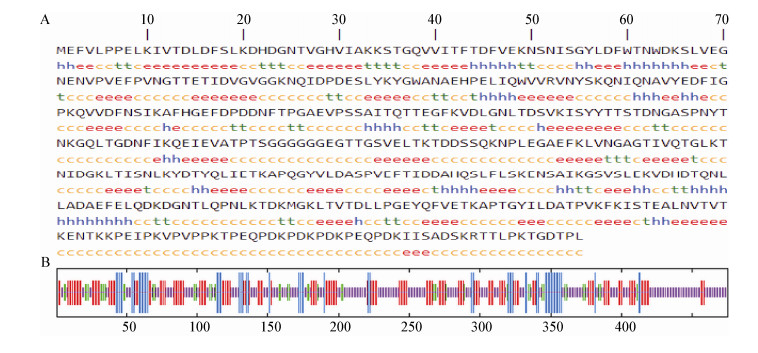
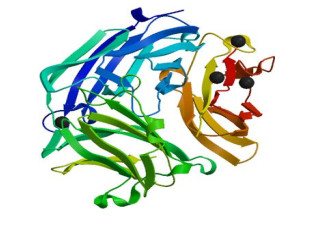
2.7 Lmo0160蛋白结构预测利用ExPASY软件中TargetP程序分析Lmo0160蛋白进行亚细胞定位显示,分泌信号通路位点(SP)占比为0.112,说明该蛋白不属于分泌型蛋白。利用SMART软件对Lmo0160蛋白结构域预测,结果表明,Lmo0160蛋白的100−226位氨基酸为胶原结合域;259−329位和351−420位氨基酸为Cna B结构域(图 5)。利用SOPMA软件预测Lmo0160蛋白的二级结构发现,无规卷曲234个,占49.26%;延伸链139个,占29.26%;α-螺旋58个,占12.21%;β-转角44个,占9.26% (图 6)。SWISS-MODEL分析平台预测该蛋白的三级结构(图 7),进一步验证了二级结构的预测。
|
| 图 5 LM90SB2的Lmo0160蛋白的结构域预测 Figure 5 The predicted structure domain of Lmo0160 protein of LM90SB2 |
|
|
|
| 图 6 LM90SB2 Lmo0160蛋白的二级结构预测 Figure 6 The predicted secondary structure of Lmo0160 protein of LM90SB2 注:A:h:α-螺旋;t:β-转角;c:无规则卷曲;e:延伸链. B:图中线条按照从长到短依次代表α-螺旋、延伸链、β-转角和无规则卷曲. Note: A: h: Alpha helix; t: Beta turn; c: Random coli; e: Beta extended strand. B: The lines in the picture represent alpha helix, beta extended strand, beta turn and random coli respectively by the length. |
|
|
|
| 图 7 Lmo0160蛋白的三级结构预测 Figure 7 The predicted tertiary structure of Lmo0160 protein of LM90SB2 |
|
|
细菌表面蛋白参与细胞各种生理和生化过程的调节,其结构与功能十分复杂。例如参与细菌生长、应激、耐受环境压力胁迫、致病性、黏附与侵袭宿主细胞、免疫防御等重要过程。同样,表面蛋白对LM也至关重要,可使其广泛生存于不同环境中,包括食物和真核细胞胞质[10-12]。EGD-e含有133个表面蛋白,根据不同的锚定系统和潜在结构域,将其表面蛋白分为4种,分别为LPXTG表面蛋白、疏水性尾部蛋白、GW蛋白和脂蛋白[13]。其中LPXTG表面蛋白可通过分选酶Sort A锚定到细菌细胞壁表面,在感染机体过程中发挥重要毒力作用[14]。
对LM参考菌株全基因组进行生物信息学研究,预测LM表面有41个LPXTG基序蛋白。目前,国内外已有研究者对部分LPXTG基序蛋白进行了初步研究,如InlA (Lmo0433)[15]、InlF[16]、InlJ[17-19]、Lmo2085[20]、Vip (lmo0320)[21]、LapB (lmo1666)[22-23]、InlH (lmo0263)[24]、Lmo0327[7]、Lmo0842[25]、Lmo1413[13]、Lmo1290[26]、P60[27]等,但Lmo0130、Lmo0160、Lmo0610、Lmo0842、Lmo0880、Lmo0159、Lmo2714等这些LPXTG基序蛋白功能未见相关报道。本试验采用PCR方法从LM90SB2菌株克隆lmo0160基因,经测序、拼接和比对后获得lmo0160全长序列1 708 bp,开放阅读框为1 428 bp,共编码475个氨基酸。同源性比对分析显示:LM90SB2菌株lmo0160核苷酸序列与4b血清型菌株相似性较高,为99.0%−99.1%,其次与4a血清型菌株相似性为97.2%,与1/2a血清型菌株相似性为87.2%−91.1%;而其编码氨基酸序列相似性均较高,为91.8%−99.4%。系统进化树结果显示,LM90SB2菌株lmo0160基因与4b血清型菌株亲缘关系较近,处在同一分支上。lmo0160基因同源性和进化树结果发现,同一血清型菌株lmo0160基因序列相似性高且聚集在同一进化分支上,提示lmo0160基因可能在不同血清型菌株中有一定变异,利用lmo0160基因构建进化树可将同一血清型聚类。本试验结果与童文彬[28]利用肠道病毒的核苷酸序列构建进化树判断毒株血清型的结果一致。
LM90SB2菌株Lmo0160蛋白的理化性质分析显示,该蛋白是一种偏酸性(PI理论值为4.61)、稳定(不稳定指数为19.89 < 40.00为稳定性蛋白)、亲水性蛋白质。Lmo0160蛋白结构域分析显示,该蛋白无跨膜区域,无信号肽区域,但含有3个结构域;Lmo0160蛋白二级结构预测发现,该蛋白是含有234个无规卷曲(49.26%)、139个延伸链(29.26%)、58个α-螺旋(12.21%)、44个β-转角(9.26%)的混合模型,其中以无规则卷曲为主;Lmo0160蛋白具有多个高亲水性区域,由此推断该蛋白具有B细胞表位形成的结构基础。
金黄色葡萄球菌Cna黏附素由Cna基因编码,该黏附素能够结合胶原蛋白,在细菌侵袭过程中起重要作用[29]。Elasri等[30]研究发现,骨骼中的金黄色葡萄球菌表面蛋白Cna可通过结合宿主的胶原蛋白,黏附于组织(如软骨)表面后侵袭骨骼,最终引起人和动物的关节炎和骨髓炎。Hienz等[31]构建的Cna基因缺失株可减弱结合胶原蛋白的能力,同时在小鼠毒力试验中发现其毒力减弱。Cna基因序列由一个非重复A域和重复B域组成,Cna A域决定Cna的活性,Cna B域由B1−B4重复序列组成,该域既不结合胶原蛋白,又不影响黏附素的活性,但Cna A通过Cna B的介导作用结合胶原蛋白[32-35]。Lmo0160蛋白结构域预测显示,该蛋白含有1个胶原蛋白结合域(第100–226位)和2个Cna B域,分别位于第259−329位和351−420位,推测该蛋白可能参与细菌的黏附过程,在LM90SB2的致病过程中发挥重要作用。这为正确认识Lmo0160蛋白的结构、功能、分类、作用部位等特性提供参考。
本研究首次克隆了lmo0160基因,并利用生物学软件对其结构进行预测,为今后研究该蛋白在LM90SB2菌株的致病作用及其机制提供了理论基础。但要确定该基因的具体功能,还需通过构建lmo0160基因缺失株并进行动物试验和侵袭细胞来进一步验证。
| [1] |
Hain T, Chatterjee SS, Ghai R, et al. Pathogenomics of Listeria spp.[J]. International Journal of Medical Microbiology, 2007, 297(7/8): 541-557. |
| [2] |
Gandhi M, Chikindas ML. Listeria: a foodborne pathogen that knows how to survive[J]. International Journal of Food Microbiology, 2007, 113(1): 1-15. DOI:10.1016/j.ijfoodmicro.2006.07.008 |
| [3] |
Cao SZ, Ma X, Chen S, et al. Construction and expression of eukaryotic expressing vector of ACTG1 from human brain microvascular endothelial cells[J]. Journal of Shihezi University (Natural Science), 2016, 34(6): 713-717. 曹树珠, 马勋, 陈朔, 等. 人脑微血管内皮细胞ACTG1基因真核表达载体的构建及表达[J]. 石河子大学学报:自然科学版, 2016, 34(6): 713-717. |
| [4] |
Chen YF. Construction and partial biological characteristics of Listeria monocytogenes ∆InlAB, ∆InlABC mutant strains[D]. Xinjiang: Master's Thesis of Shihezi University, 2015 (in Chinese) 陈云飞. 单增李斯特菌ΔInlAB、ΔInlABC突变株的构建及部分生物学特性研究[D]. 新疆: 石河子大学硕士学位论文, 2015 http://cdmd.cnki.com.cn/Article/CDMD-10759-1015993638.htm |
| [5] |
Li ZQ. Production of antibodies of high specificity and affinity for constitutively and stably expressed Listeria monocytogenes surface antigens[D]. Guangdong: Master's Thesis of Jinan University, 2012 (in Chinese) 李志清. 单增李斯特菌特异性的膜表面蛋白的抗体的制备[D]. 广东: 暨南大学硕士学位论文, 2012 http://cdmd.cnki.com.cn/Article/CDMD-10559-1012031267.htm |
| [6] |
Mariscotti JF, Quereda JJ, Pucciarelli MG. Contribution of sortase A to the regulation of Listeria monocytogenes LPXTG surface proteins[J]. International Microbiology, 2012, 15(1): 43-51. |
| [7] |
Popowska M, Markiewicz Z. Characterization of Listeria monocytogenes protein Lmo0327 with murein hydrolase activity[J]. Archives of Microbiology, 2006, 186(1): 69-86. DOI:10.1007/s00203-006-0122-8 |
| [8] |
Cummins J, Casey PG, Joyce SA, et al. A mariner transposon-based signature-tagged mutagenesis system for the analysis of oral infection by Listeria monocytogenes[J]. PLoS One, 2013, 8(9): e75437. DOI:10.1371/journal.pone.0075437 |
| [9] |
Pucciarelli MG, Calvo E, Sabet C, et al. Identification of substrates of the Listeria monocytogenes sortases A and B by a non-gel proteomic analysis[J]. Proteomics, 2005, 5(18): 4808-4817. DOI:10.1002/(ISSN)1615-9861 |
| [10] |
Camejo A, Carvalho F, Reis O, et al. The arsenal of virulence factors deployed by Listeria monocytogenes to promote its cell infection cycle[J]. Virulence, 2011, 2(5): 379-394. DOI:10.4161/viru.2.5.17703 |
| [11] |
McGann P, Ivanek R, Wiedmann M, et al. Temperature-dependent expression of Listeria monocytogenes internalin and internalin-like genes suggests functional diversity of these proteins among the Listeriae[J]. Applied and Environmental Microbiology, 2007, 73(9): 2806-2814. DOI:10.1128/AEM.02923-06 |
| [12] |
Jiang JJ. Disruption of inlC2 enhances the internalization of Listeria monocytogenes by epithelial cells[D]. Xinjiang: Doctoral Dissertation of Shihezi University, 2011 (in Chinese) 蒋建军. 缺失inlC2突变株增强单核细胞增多症李斯特菌对上皮细胞的内化作用的研究[D]. 新疆: 石河子大学博士学位论文, 2011 http://cdmd.cnki.com.cn/article/cdmd-10759-1011407236.htm |
| [13] |
Mariscotti JF, Quereda JJ, García-Del Portillo F, et al. The Listeria monocytogenes LPXTG surface protein Lmo1413 is an invasin with capacity to bind mucin[J]. International Journal of Medical Microbiology, 2014, 304(3/4): 393-404. |
| [14] |
Cabanes D, Dehoux P, Dussurget O, et al. Surface proteins and the pathogenic potential of Listeria monocytogenes[J]. Trends in Microbiology, 2002, 10(5): 238-245. DOI:10.1016/S0966-842X(02)02342-9 |
| [15] |
Wang HY, Liu ZX, Shi XH, et al. Research progress of Listeria monocytogenes and itssurface proteins[J]. Inspection and Quarantine Science, 2006, 16(2): 76-80. 王海艳, 刘中学, 石新华, 等. 单增李斯特菌及其表面蛋白的研究进展[J]. 检验检疫科学, 2006, 16(2): 76-80. |
| [16] |
Kirchner M, Higgins DE. Inhibition of ROCK activity allows InlF-mediated invasion and increased virulence of Listeria monocytogenes[J]. Molecular Microbiology, 2008, 68(3): 749-767. DOI:10.1111/j.1365-2958.2008.06188.x |
| [17] |
Bublitz M, Holland C, Sabet C, et al. Crystal structure and standardized geometric analysis of InlJ, a listerial virulence factor and leucine-rich repeat protein with a novel cysteine ladder[J]. Journal of Molecular Biology, 2008, 378(1): 87-96. DOI:10.1016/j.jmb.2008.01.100 |
| [18] |
Sabet C, Lecuit M, Cabanes D, et al. LPXTG protein InlJ, a newly identified internalin involved in Listeria monocytogenes virulence[J]. Infection and Immunity, 2005, 73(10): 6912-6922. DOI:10.1128/IAI.73.10.6912-6922.2005 |
| [19] |
Sabet C, Toledo-Arana A, Personnic N, et al. The Listeria monocytogenes virulence factor InlJ is specifically expressed in vivo and behaves as an adhesin[J]. Infection and Immunity, 2008, 76(4): 1368-1378. DOI:10.1128/IAI.01519-07 |
| [20] |
Samarin S, Romero S, Kocks C, et al. How VASP enhances actin-based motility[J]. Journal of Cell Biology, 2003, 163(1): 131-142. |
| [21] |
Cabanes D, Sousa S, Cebriá A, et al. Gp96 is a receptor for a novel Listeria monocytogenes virulence factor, Vip, a surface protein[J]. The EMBO Journal, 2005, 24(15): 2827-2838. DOI:10.1038/sj.emboj.7600750 |
| [22] |
Reis O, Sousa S, Camejo A, et al. LapB, a novel Listeria monocytogenes LPXTG surface adhesin, required for entry into Eukaryotic cells and virulence[J]. The Journal of Infectious Diseases, 2010, 202(4): 551-562. DOI:10.1086/653016 |
| [23] |
Sievers S, Lilleb k EMS, Jacobsen K, et al. A multicopy sRNA of Listeria monocytogenes regulates expression of the virulence adhesin LapB[J]. Nucleic Acids Research, 2014, 42(14): 9383-9398. DOI:10.1093/nar/gku630 |
| [24] |
Personnic N, Bruck S, Nahori MA, et al. The stress-induced virulence protein InlH controls interleukin-6 production during murine Listeriosis[J]. Infection and Immunity, 2010, 78(5): 1979-1989. DOI:10.1128/IAI.01096-09 |
| [25] |
Cummins J, Casey PG, Joyce SA, et al. A mariner transposon-based signature-tagged mutagenesis system for the analysis of oral infection by Listeria monocytogenes[J]. PLoS One, 2013, 8(9): e75437. DOI:10.1371/journal.pone.0075437 |
| [26] |
Dortet L, Mostowy S, Louaka AS, et al. Recruitment of the major vault protein by InlK: a Listeria monocytogenes strategy to avoid autophagy[J]. PLoS Pathogens, 2011, 7(8): e1002168. DOI:10.1371/journal.ppat.1002168 |
| [27] |
Pilgrim S, Kolb-M urer A, Gentschev I, et al. Deletion of the gene encoding p60 in Listeria monocytogenes leads to abnormal cell division and loss of actin-based motility[J]. Infection and Immunity, 2003, 71(6): 3473-3484. DOI:10.1128/IAI.71.6.3473-3484.2003 |
| [28] |
Tong WB. Correlation between enterovirus nucleotide sequences and serotype[J]. Journal of Preventive Medicine Information, 2007, 23(2): 163-167. 童文彬. 肠道病毒核苷酸序列和血清型的相关性[J]. 预防医学情报杂志, 2007, 23(2): 163-167. |
| [29] |
Patti JM, Allen BL, McGavin MJ, et al. MSCRAMM-mediated adherence of microorganisms to host tissues[J]. Annual Review of Microbiology, 1994, 48: 585-617. DOI:10.1146/annurev.mi.48.100194.003101 |
| [30] |
Elasri MO, Thomas JR, Skinner RA, et al. Staphylococcus aureus collagen adhesin contributes to the pathogenesis of osteomyelitis[J]. Bone, 2002, 30(1): 275-280. DOI:10.1016/S8756-3282(01)00632-9 |
| [31] |
Hienz SA, Schennings T, Heimdahl A, et al. Collagen binding of Staphylococcus aureus is a virulence factor in experimental endocarditis[J]. The Journal of Infectious Diseases, 1996, 174(1): 83-88. DOI:10.1093/infdis/174.1.83 |
| [32] |
Deivanayagam CCS, Rich RL, Carson M, et al. Novel fold and assembly of the repetitive B region of the Staphylococcus aureus collagen-binding surface protein[J]. Structure, 2000, 8(1): 67-78. DOI:10.1016/S0969-2126(00)00081-2 |
| [33] |
Symersky J, Patti JM, Carson M, et al. Structure of the collagen-binding domain from a Staphylococcus aureus adhesin[J]. Nature Structural Biology, 1997, 4(10): 833-838. DOI:10.1038/nsb1097-833 |
| [34] |
Gillaspy AF, Patti JM, Pratt Jr FL, et al. The Staphylococcus aureus collagen adhesin-encoding gene (cna) is within a discrete genetic element[J]. Gene, 1997, 196(1/2): 239-248. |
| [35] |
Rich RL, Demeler B, Ashby K, et al. Domain structure of the Staphylococcus aureus collagen adhesin[J]. Biochemistry, 1998, 37(44): 15423-15433. DOI:10.1021/bi981773r |
 2017, Vol. 44
2017, Vol. 44