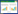扩展功能
文章信息
- 靳换, 李逸, 姜楠, 周磊, 盖新娜, 杨汉春, 郭鑫
- JIN Huanv, LI Yi, JIANG Nan, ZHOU Lei, GE Xin-Na, YANG Han-Chun, GUO Xin
- 与PRRSV nsp11互作的宿主细胞蛋白鉴定及生物信息学分析
- Identification and bioinformatics analysis of host cellular proteins interacting with PRRSV nsp11
- 微生物学通报, 2017, 44(12): 2856-2870
- Microbiology China, 2017, 44(12): 2856-2870
-
文章历史
- 收稿日期: 2017-07-15
- 接受日期: 2017-09-18
- 优先数字出版日期(www.cnki.net): 2017-09-26
猪繁殖与呼吸综合征(Porcine reproductive and respiratory syndrome,PRRS)是由猪繁殖与呼吸综合征病毒(Porcine reproductive and respiratory syndrome virus,PRRSV)引起的猪的一种高度接触性传染病,临床上主要表现为妊娠母猪流产、早产、产死胎和木乃伊胎等繁殖障碍以及各生长阶段猪(特别是仔猪)的呼吸道症状[1-2]。1987年,该病首次在美国北卡罗纳州、爱荷华等州发现,随后该病迅速蔓延,在北美、欧洲和亚洲等地相继暴发和流行,给全球养猪业造成了巨大经济损失[3-5]。1991年,在中国台湾地区首次暴发PRRS[6];1996年郭宝清等首次分离到PRRSV (CH-1a株),从而确证中国大陆存在PRRS[7]。2006年,我国出现高致病性PRRSV (Highly pathogenic PRRSV)[8],造成大批猪死亡,此次疫情严重危害了我国养猪生产。PRRSV的致病机制非常复杂,其中非结构蛋白在调节病毒的复制和致病性以及拮抗宿主抗病毒免疫调控中发挥重要作用[9-12]。PRRSV非结构蛋白nsp11具有特异性切割嘧啶碱基的核糖核酸内切酶活性、去泛素化酶活性,可抑制Ⅰ型干扰素、IRF3和NF-κB激活[13-15]以及抑制细胞因子IL-1β的产生[16]。
鉴定宿主细胞蛋白与病毒蛋白之间的相互作用是研究病毒蛋白的功能以及在复制过程中作用的关键步骤[17-18]。Gene Ontology (简称GO)是生物信息领域中一个极为重要的方法和工具,通过建立一套具有动态形式的控制字集,预测真核基因及蛋白质在细胞内所扮演的角色,从而全面描述生物体中基因和基因产物的属性[19];Clusters of Orthologous Groups of proteins (简称COG)注释的作用主要是通过参考NCBI数据库中已知的蛋白对未知序列进行功能注释;在生物体内,不同蛋白相互协调而行使其生物学功能,基于Pathway的分析有助于更进一步了解其生物学功能。Kyoto Encyclopedia of Genes and Genomes (简称KEGG)是有关Pathway的主要公共数据库[20],通过Pathway分析能确定宿主蛋白质参与的最主要生化代谢途径和信号转导途径。为了探究PRRSV nsp11的生物学功能以及nsp11在病毒复制和致病性过程中所发挥的作用,本研究以nsp11与宿主细胞蛋白的相互作用为切入点,通过筛选和鉴定与nsp11相互作用的宿主细胞蛋白并进行GO注释、COG注释和KEGG代谢通路注释,分析预测这些宿主细胞蛋白在病毒的复制和致病过程中所发挥的作用,为探究nsp11的生物学功能以及PRRSV的致病机制奠定基础。
1 材料与方法 1.1 材料 1.1.1 细胞、质粒和病毒: MARC-145细胞、真核表达质粒pCMV-HA和pCMV-Myc由农业部动物流行病学重点开放实验室(以下简称“本实验室”)保存。PRRSV JXwn06 P8毒株由本实验室分离、鉴定并保存。 1.1.2 主要试剂和仪器: Protein G Sepharose 4 Fast Flow,购自GE Healthcare Life Sciences公司;Alexa-fluor-488标记的山羊抗鼠IgG (H+L) F (ab′) 2、Alexa-fluor-568标记的山羊抗兔IgG (H+L) F (ab′) 2、PierceTM Silver Stain for Mass Spectrometry试剂盒和Q-Exactive质谱仪,购自Thermo Fisher Scientific公司;Anti-HA、Anti-Myc标签单克隆抗体,购自Sigma-Aldrich公司;Anti-IRAK1多克隆抗体,购自proteintech公司;Hoechst No. 33258染色液,购自北京华菁科技有限公司,Aqua Poly/Mount试剂,购自Polysciences公司。FluorChem E化学发光一体机,购自美国proteinsimple公司。 1.2 方法 1.2.1 质粒的构建: 分别以PRRSV基因组cDNA和猪原代肺泡巨噬细胞cDNA为模板,设计引物扩增nsp11和IRAK1基因,将获得的基因片段连接至平末端克隆载体pEASY-Blunt,通过PCR扩增、双酶切、连接的方法,将nsp11和IRAK1基因片段分别插入pCMV-HA和pCMV-Myc载体中,命名为pCMV-HA-nsp11和pCMV-Myc-IRAK1。设计的引物见表 1。| Primer | Sequence (5′→3′) | Size (bp) |
| IRAK1-F | ATGGCCGGGGGGCCAGGCCCG | 2 136 |
| IRAK1-R | TCAGCTCTGAAATTCATCACTC | |
| nsp11-F | GGGTCGAGCTCCCCGCTCC | 669 |
| nsp11-R | TTCAAGTTGAAAATAGGCCG | |
| pCMV-Myc-IRAK1-F | GGAATTCATATGGCCGGGGG | 2 152 |
| pCMV-Myc-IRAK1-R | GGGTACCTCAGCTCTGAAATTC | |
| pCMV-HA-nsp11-F | GCGAATTCGGATGGGGTCGAGCTCCCCGC | 692 |
| pCMV-HA-nsp11-R | GCGCGGCCGCTTCAAGTTGAAAATAGGCCG |
为了确定PRRSV感染宿主细胞后nsp11表达量的变化情况,用PRRSV感染MARC-145细胞,并于感染后12、24、36、48和60 h收取细胞样品,与nsp11单克隆抗体反应并进行Western blot分析,以β-actin作为内参对照。实验结果显示,PRRSV感染后36 h到48 h nsp11的表达量最高(图 1),因此选择36 h作为筛选与nsp11互作宿主细胞蛋白的时间点。
|
| 图 1 感染PRRSV后nsp11表达量的变化 Figure 1 Analysis of the nsp11 expression at the indicated time points following PRRSV infection |
|
|
|
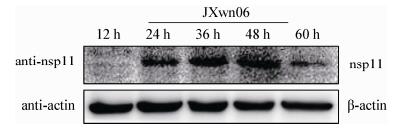
| 图 2 免疫沉淀及银染结果分析 Figure 2 Analysis of the immunoprecipitation and silver staining |
|
|
| Accession No. | Score | Coverage | Proteins | Unique peptides | Peptides | PSMs | AAs | MW (kD) | calc. pI | Name |
| F7EMW7 | 5 274.96 | 59.86 | 4 | 69 | 69 | 428 | 1 014 | 112.8 | 8.98 | PARP1 |
| H9FQM8 | 2 977.35 | 48.38 | 2 | 3 | 34 | 191 | 649 | 71.3 | 4.45 | DBN1 |
| H9Z082 | 2 030.99 | 50.19 | 6 | 49 | 49 | 114 | 1 044 | 119.6 | 9.41 | MYO1C |
| G7NQI2 | 1 954.80 | 55.99 | 3 | 45 | 45 | 133 | 893 | 99.4 | 5.55 | MVP |
| H9ERI1 | 1 405.82 | 49.74 | 5 | 1 | 50 | 93 | 951 | 106.8 | 6.15 | RAI14 |
| G7MV27 | 1 394.09 | 49.29 | 7 | 1 | 50 | 92 | 980 | 109.9 | 6.28 | RAI14 |
| F7B786 | 1 005.64 | 38.36 | 3 | 25 | 32 | 50 | 636 | 65.2 | 8.12 | KRT1 |
| H9ETH2 | 953.97 | 31.39 | 5 | 27 | 27 | 62 | 806 | 89.0 | 5.72 | HNRNPU |
| H9YWZ9 | 935.51 | 45.27 | 9 | 46 | 46 | 69 | 1 078 | 124.9 | 9.20 | MYO1B |
| H9Z5S3 | 799.14 | 40.32 | 1 | 6 | 32 | 52 | 940 | 104.1 | 6.90 | AP2A2 |
| H9FUT3 | 748.97 | 33.88 | 6 | 31 | 31 | 44 | 1 101 | 120.7 | 7.33 | ACLY |
| H9FQM0 | 704.49 | 41.53 | 4 | 5 | 33 | 51 | 939 | 103.9 | 6.93 | AP2A2 |
| F6PL31 | 694.08 | 47.75 | 5 | 23 | 38 | 56 | 955 | 105.3 | 7.66 | AP2A1 |
| F6R5B1 | 644.19 | 36.19 | 9 | 34 | 34 | 44 | 1 003 | 113.5 | 8.73 | MOV10 |
| H9FQN1 | 641.38 | 43.01 | 4 | 21 | 34 | 55 | 937 | 104.5 | 5.38 | AP2B1 |
| G7N721 | 595.73 | 42.73 | 24 | 2 | 31 | 44 | 564 | 59.9 | 8.00 | EGK_03678 |
| I0FH43 | 588.20 | 34.33 | 7 | 32 | 32 | 37 | 1 066 | 116.7 | 5.99 | VCL |
| I2CWY8 | 583.22 | 30.68 | 2 | 1 | 40 | 40 | 712 | 78.3 | 5.42 | IRAK1 |
| H9ERQ9 | 582.39 | 29.34 | 5 | 26 | 26 | 36 | 1 009 | 113.7 | 8.48 | HLTF |
| F7GC25 | 557.38 | 35.66 | 2 | 32 | 33 | 51 | 858 | 95.3 | 6.83 | EEF2 |
| F7HAX4 | 514.06 | 42.82 | 6 | 27 | 27 | 31 | 808 | 91.0 | 5.77 | MCM3 |
| F6WRA1 | 463.16 | 26.59 | 2 | 24 | 24 | 34 | 1 117 | 124.4 | 5.76 | SPECC1L |
| H9EMU1 | 459.83 | 28.62 | 4 | 19 | 19 | 36 | 856 | 95.7 | 6.92 | RNPUL1 |
| H9FQQ0 | 458.39 | 24.96 | 4 | 24 | 24 | 29 | 1 166 | 130.9 | 6.33 | DHX9 |
| H9F6W2 | 451.51 | 35.54 | 7 | 25 | 25 | 37 | 664 | 76.4 | 5.01 | HNRNPUL2 |
| F7APH2 | 411.66 | 42.81 | 8 | 16 | 16 | 27 | 306 | 32.8 | 8.16 | HNRNPD |
| F7FHJ3 | 389.78 | 22.42 | 9 | 6 | 19 | 26 | 919 | 101.2 | 5.11 | AP1B1 |
| H9EPZ5 | 384.87 | 26.06 | 16 | 21 | 21 | 27 | 898 | 95.8 | 8.81 | ILF3 |
| F6TKP6 | 384.82 | 24.09 | 6 | 1 | 19 | 21 | 963 | 109.8 | 6.87 | KIF5B |
| H9G179 | 380.20 | 27.52 | 3 | 18 | 18 | 25 | 814 | 92.3 | 4.96 | EIF3B |
| G7N720 | 362.55 | 34.93 | 15 | 5 | 22 | 27 | 564 | 59.9 | 8.00 | EGK_03677 |
| F7AUV0 | 360.89 | 19.16 | 1 | 16 | 16 | 21 | 971 | 110.3 | 5.77 | CSE1L |
| H9FV03 | 347.02 | 41.69 | 3 | 17 | 17 | 30 | 391 | 42.3 | 10.05 | RBMX |
| G7N1U0 | 344.26 | 24.40 | 6 | 1 | 19 | 21 | 963 | 109.7 | 6.44 | KIF5B |
| F7BMX8 | 342.28 | 33.51 | 2 | 30 | 30 | 35 | 934 | 101.3 | 7.06 | MTHFD1 |
| H9FUD1 | 336.58 | 15.67 | 2 | 2 | 22 | 28 | 1 385 | 158.3 | 6.23 | LMO7 |
| I2CXY9 | 324.37 | 23.48 | 8 | 8 | 16 | 19 | 724 | 83.2 | 5.03 | HSP90AB1 |
| F7H579 | 311.27 | 20.30 | 8 | 12 | 16 | 21 | 857 | 96.6 | 9.16 | HSP90AB1 |
| G7NS66 | 296.65 | 43.28 | 8 | 17 | 17 | 23 | 409 | 43.9 | 8.41 | EGK_20668 |
| H9EQB9 | 278.40 | 13.58 | 3 | 12 | 12 | 13 | 1 038 | 119.4 | 4.82 | IPO7 |
| F6U9A3 | 243.69 | 22.57 | 23 | 4 | 13 | 19 | 483 | 53.6 | 5.59 | KRT8 |
| F6RLH5 | 231.96 | 31.73 | 5 | 14 | 18 | 23 | 646 | 70.9 | 5.52 | HSPA8 |
| H9FNU8 | 230.67 | 30.66 | 4 | 12 | 12 | 14 | 424 | 46.9 | 7.27 | PAICS |
| H9YXH3 | 224.24 | 15.52 | 8 | 20 | 20 | 23 | 1 675 | 191.5 | 5.69 | CLTC |
| I2CXH1 | 221.05 | 26.59 | 5 | 16 | 16 | 22 | 707 | 76.1 | 9.44 | SFPQ |
| F7DBX8 | 218.22 | 9.98 | 4 | 8 | 8 | 8 | 962 | 109.9 | 5.39 | XPOT |
| H9FQK6 | 206.00 | 17.77 | 5 | 16 | 16 | 18 | 968 | 106.7 | 5.62 | AARS |
| F6WWS6 | 201.34 | 13.78 | 2 | 10 | 10 | 12 | 798 | 87.2 | 5.35 | USP10 |
| F6R5W0 | 198.75 | 21.53 | 4 | 6 | 6 | 7 | 339 | 37.8 | 5.62 | ERLIN2 |
| H9Z8D1 | 191.80 | 22.49 | 5 | 3 | 8 | 10 | 449 | 49.3 | 6.30 | HNRNPH2 |
| F7A985 | 187.44 | 14.82 | 2 | 6 | 9 | 10 | 614 | 69.1 | 8.92 | DDX5 |
| H9FVV7 | 185.54 | 30.32 | 13 | 13 | 13 | 16 | 531 | 57.9 | 7.84 | PKM2 |
| F6UEF5 | 177.19 | 19.52 | 1 | 4 | 6 | 7 | 415 | 45.6 | 5.58 | HNRNPF |
| G7ND21 | 174.71 | 18.16 | 4 | 18 | 18 | 21 | 1 140 | 126.9 | 5.26 | DDB1 |
| H9EYZ1 | 173.34 | 32.54 | 6 | 9 | 9 | 12 | 421 | 45.1 | 8.91 | UQCRC2 |
| H9FRU7 | 169.15 | 15.83 | 8 | 14 | 14 | 15 | 1 118 | 121.8 | 8.85 | FAM120A |
| I2CYY2 | 166.79 | 10.41 | 4 | 9 | 9 | 10 | 961 | 106.8 | 6.52 | LONP1 |
| F6V6M4 | 159.38 | 24.10 | 2 | 7 | 7 | 7 | 361 | 37.8 | 5.45 | EIF3F |
| G7NQG9 | 158.91 | 12.71 | 2 | 12 | 12 | 13 | 913 | 105.2 | 5.68 | EIF3C |
| G7MUQ7 | 157.88 | 7.22 | 2 | 5 | 5 | 6 | 790 | 88.2 | 4.96 | CDH6 |
| F7DBK9 | 157.69 | 11.63 | 2 | 9 | 9 | 11 | 843 | 96.0 | 8.22 | FTSJ3 |
| F7GV16 | 157.54 | 22.00 | 14 | 6 | 6 | 11 | 350 | 38.2 | 8.18 | LOC710682 |
| H9FUX2 | 151.96 | 14.25 | 3 | 10 | 10 | 10 | 807 | 88.8 | 9.19 | NOP2 |
| G7N7G0 | 151.83 | 13.97 | 1 | 1 | 9 | 9 | 759 | 85.8 | 6.76 | EGK_03838 |
| G7N725 | 149.16 | 10.37 | 10 | 1 | 6 | 9 | 511 | 55.8 | 8.07 | EGK_03682 |
| H9FUZ0 | 148.08 | 11.94 | 6 | 9 | 9 | 10 | 997 | 109.7 | 5.40 | ATP2A2 |
| F6VFC8 | 146.24 | 16.78 | 14 | 6 | 6 | 10 | 441 | 47.8 | 8.97 | LOC710682 |
| F6XTF7 | 141.90 | 29.27 | 2 | 5 | 5 | 6 | 205 | 22.8 | 6.40 | HSPB1 |
| H9FAB5 | 139.49 | 20.10 | 11 | 6 | 6 | 6 | 393 | 44.7 | 5.47 | EIF4A1 |
| H9ENE8 | 139.24 | 9.10 | 2 | 6 | 6 | 6 | 835 | 93.3 | 5.06 | USP5 |
| H9F675 | 138.51 | 7.26 | 3 | 6 | 6 | 6 | 881 | 100.4 | 7.85 | ERCC4 |
| H9ENQ1 | 134.40 | 7.98 | 4 | 6 | 6 | 6 | 1 053 | 112.9 | 6.25 | ANKRD28 |
| H9FUU5 | 133.80 | 11.33 | 15 | 9 | 9 | 10 | 900 | 104.0 | 6.04 | NEDD4 |
| H9F0X7 | 132.62 | 9.33 | 1 | 10 | 10 | 10 | 1 082 | 129.6 | 6.98 | EIF3A |
| F7BX98 | 132.36 | 20.82 | 1 | 11 | 11 | 13 | 802 | 92.2 | 8.18 | CDC5L |
| F7HRC0 | 131.55 | 8.23 | 3 | 8 | 8 | 8 | 1155 | 129.4 | 8.53 | DHX30 |
| F6QY64 | 130.74 | 12.23 | 3 | 10 | 10 | 10 | 981 | 112.3 | 5.22 | USP15 |
| F6U0V1 | 126.28 | 13.07 | 3 | 12 | 12 | 14 | 979 | 111.4 | 7.75 | DHX36 |
| I0FRR1 | 125.94 | 9.12 | 14 | 8 | 8 | 8 | 1 107 | 116.8 | 6.05 | MAP4 |
| F7DZD6 | 124.01 | 12.66 | 4 | 5 | 5 | 6 | 379 | 43.5 | 9.58 | LYAR |
| H9F109 | 117.37 | 15.00 | 3 | 4 | 4 | 7 | 300 | 32.1 | 8.91 | ZNF787 |
| F7DC34 | 115.88 | 8.96 | 1 | 8 | 8 | 8 | 937 | 102.7 | 7.01 | DDX42 |
| F6ST49 | 114.62 | 20.37 | 8 | 6 | 6 | 7 | 427 | 47.8 | 9.52 | DDB2 |
| F7HPP2 | 111.11 | 26.50 | 2 | 6 | 7 | 8 | 351 | 39.5 | 5.27 | TMOD2 |
| G7N406 | 109.64 | 8.29 | 3 | 8 | 8 | 8 | 941 | 106.8 | 8.18 | PRPF6 |
| F7HE98 | 106.35 | 12.19 | 2 | 4 | 4 | 4 | 320 | 35.6 | 6.13 | EIF3G |
| F7CA88 | 105.97 | 7.68 | 1 | 3 | 3 | 3 | 456 | 49.2 | 4.82 | RNH1 |
| H9FBQ3 | 105.51 | 28.88 | 3 | 5 | 5 | 5 | 277 | 31.4 | 6.95 | PURA |
| G7NKJ4 | 104.56 | 3.34 | 2 | 2 | 2 | 2 | 1 049 | 113.9 | 4.94 | EGK_09609 |
| F6Q1Z2 | 104.47 | 4.28 | 1 | 2 | 5 | 5 | 1 052 | 121.8 | 8.09 | SMARCA5 |
| F6TF39 | 104.31 | 20.44 | 2 | 6 | 7 | 8 | 367 | 40.5 | 8.90 | DRG1 |
| H9F194 | 104.21 | 14.16 | 4 | 2 | 2 | 2 | 226 | 24.6 | 9.98 | SERBP1 |
| G7NJF1 | 103.16 | 8.46 | 5 | 7 | 7 | 8 | 958 | 109.0 | 7.20 | EGK_09369 |
| G7NRD4 | 103.11 | 7.93 | 7 | 5 | 8 | 8 | 996 | 116.8 | 8.65 | EGK_20912 |
| H9EVP0 | 101.23 | 8.84 | 4 | 3 | 3 | 4 | 475 | 54.3 | 8.73 | DHCR7 |
| F7HIW8 | 99.58 | 9.00 | 3 | 4 | 4 | 4 | 489 | 54.1 | 9.63 | SPATS2L |
| G7NGX7 | 97.70 | 23.64 | 3 | 7 | 7 | 7 | 385 | 42.4 | 8.47 | PRPSAP1 |
| H9FYW3 | 97.70 | 8.45 | 7 | 4 | 7 | 8 | 698 | 78.9 | 5.22 | SRPK2 |
| F6VC72 | 97.44 | 12.79 | 1 | 11 | 11 | 11 | 821 | 92.8 | 5.41 | MCM6 |
| G7NPI5 | 96.88 | 23.30 | 4 | 5 | 5 | 7 | 309 | 35.3 | 5.86 | EGK_12445 |
| F6SFZ4 | 96.78 | 13.61 | 6 | 4 | 4 | 6 | 382 | 41.1 | 8.41 | PGK1 |
| H9FRE3 | 95.82 | 12.53 | 3 | 9 | 9 | 9 | 950 | 108.5 | 7.47 | XRN2 |
| H9F7A9 | 94.72 | 1.81 | 5 | 7 | 7 | 7 | 4 086 | 464.6 | 7.24 | PRKDC |
| H9F922 | 94.37 | 4.92 | 2 | 5 | 5 | 5 | 914 | 104.7 | 7.59 | RB1 |
| F6VB85 | 92.78 | 12.07 | 5 | 9 | 9 | 9 | 820 | 95.6 | 9.55 | DDX23 |
| F7GEU6 | 92.48 | 13.24 | 2 | 9 | 9 | 10 | 793 | 88.8 | 5.22 | SF3A1 |
| F7D6M8 | 92.47 | 5.01 | 1 | 4 | 4 | 4 | 1 037 | 119.9 | 5.16 | IPO8 |
| F7GN57 | 91.88 | 9.66 | 3 | 3 | 4 | 4 | 414 | 46.6 | 6.87 | IDH1 |
| F6UDV1 | 90.03 | 10.23 | 3 | 4 | 4 | 4 | 430 | 47.4 | 9.01 | GOT2 |
| H9EYM1 | 88.41 | 25.28 | 3 | 3 | 3 | 4 | 178 | 18.8 | 5.02 | PDCD2L |
| H9EP24 | 85.03 | 4.42 | 6 | 5 | 5 | 5 | 1 087 | 123.8 | 6.34 | XPO7 |
| I0FTY4 | 84.53 | 8.68 | 7 | 8 | 8 | 9 | 1 141 | 127.6 | 9.31 | RFC1 |
| F6ZSK0 | 84.30 | 10.22 | 3 | 6 | 7 | 7 | 890 | 97.2 | 9.88 | RBM15B |
| H9FSW2 | 83.42 | 9.10 | 7 | 3 | 7 | 8 | 857 | 97.2 | 9.16 | EIF2C1 |
| F7GMY9 | 81.88 | 6.23 | 5 | 5 | 5 | 5 | 931 | 103.5 | 8.57 | ADAR |
| G7NLX0 | 80.65 | 17.12 | 6 | 6 | 6 | 6 | 368 | 41.5 | 5.44 | EGK_09938 |
| F7H0C7 | 77.76 | 22.12 | 3 | 7 | 7 | 7 | 339 | 38.6 | 7.75 | ANXA2 |
| G7NLB1 | 76.94 | 21.84 | 2 | 5 | 5 | 8 | 348 | 38.8 | 4.91 | ASNA1 |
| H9FY79 | 76.16 | 6.13 | 10 | 0 | 6 | 7 | 913 | 102.2 | 5.83 | SAFB |
| H9G2A7 | 74.56 | 5.68 | 2 | 1 | 6 | 6 | 933 | 103.9 | 5.87 | SAFB2 |
| F7CKR9 | 73.54 | 19.18 | 3 | 6 | 6 | 6 | 391 | 45.1 | 7.74 | CSNK2A3 |
| H9ZCT4 | 71.04 | 13.48 | 3 | 8 | 8 | 9 | 712 | 83.1 | 6.51 | STAT1 |
| H9F6A4 | 70.63 | 7.26 | 8 | 1 | 4 | 5 | 634 | 69.3 | 5.72 | HSPA1A |
| G7N4I8 | 70.02 | 5.16 | 2 | 4 | 4 | 4 | 892 | 96.8 | 7.68 | EGK_02290 |
| F7DF58 | 68.19 | 1.54 | 2 | 1 | 1 | 1 | 781 | 87.2 | 8.00 | NPHP3 |
| G7N7D7 | 67.54 | 7.68 | 4 | 3 | 3 | 3 | 495 | 52.9 | 5.72 | EGK_03812 |
| H9G140 | 67.40 | 6.40 | 8 | 7 | 7 | 7 | 1 094 | 121.2 | 5.82 | AP3B1 |
| F6YEC2 | 67.11 | 3.19 | 3 | 3 | 3 | 3 | 1 221 | 138.8 | 5.45 | EIF5B |
| F6ZH77 | 66.61 | 8.80 | 4 | 6 | 6 | 6 | 852 | 92.8 | 6.52 | KDM1A |
| H9ZBR2 | 66.22 | 3.09 | 3 | 3 | 3 | 3 | 906 | 105.5 | 7.21 | CWC22 |
| H9ZC54 | 65.72 | 4.38 | 2 | 1 | 4 | 5 | 936 | 106.1 | 5.71 | SAFB |
| H9F7S5 | 65.17 | 7.24 | 5 | 7 | 7 | 7 | 925 | 104.2 | 4.82 | PPP4R1 |
| G7N067 | 65.01 | 2.81 | 6 | 3 | 3 | 3 | 1 067 | 120.8 | 6.64 | EGK_19304 |
| F7ETD0 | 64.37 | 10.49 | 1 | 5 | 5 | 5 | 553 | 59.7 | 9.11 | ATP5A1 |
| I2CV68 | 63.59 | 7.96 | 5 | 7 | 7 | 7 | 1 081 | 118.6 | 5.00 | IPO4 |
| H9FWY9 | 63.22 | 8.00 | 5 | 8 | 8 | 9 | 1 012 | 113.9 | 7.05 | IARS2 |
| F7GH80 | 61.11 | 1.70 | 3 | 1 | 1 | 1 | 825 | 92.2 | 6.87 | DDX20 |
| H9FBN5 | 60.95 | 3.62 | 3 | 3 | 3 | 3 | 995 | 108.8 | 6.81 | MIB1 |
| H9F0N2 | 58.13 | 5.36 | 2 | 1 | 1 | 1 | 261 | 27.6 | 9.83 | ZNF362 |
| H9F138 | 56.72 | 6.83 | 8 | 2 | 5 | 6 | 761 | 86.0 | 9.28 | EIF2C3 |
| F6QE81 | 56.25 | 10.26 | 4 | 3 | 3 | 3 | 341 | 36.0 | 8.65 | HNRNPA2B1 |
| H9FUU1 | 55.76 | 2.63 | 3 | 1 | 1 | 1 | 457 | 51.9 | 7.97 | NDUFS2 |
| F6VI02 | 55.51 | 13.21 | 1 | 4 | 4 | 4 | 318 | 36.8 | 6.46 | UBLCP1 |
| F6X4D3 | 53.95 | 2.22 | 2 | 3 | 3 | 3 | 1 304 | 145.7 | 7.09 | SF3B1 |
| H9FN11 | 52.76 | 3.05 | 1 | 1 | 1 | 1 | 393 | 44.2 | 5.12 | ATG4B |
| H9G2G0 | 52.42 | 2.67 | 7 | 2 | 2 | 2 | 748 | 86.0 | 10.40 | BCLAF1 |
| F7HGD2 | 52.39 | 3.59 | 1 | 1 | 1 | 1 | 390 | 43.0 | 5.26 | ILF2 |
| H9F9C2 | 51.69 | 3.37 | 3 | 1 | 1 | 1 | 415 | 49.0 | 5.15 | RBM25 |
| F7HBM4 | 50.58 | 7.18 | 8 | 3 | 3 | 3 | 390 | 43.3 | 8.06 | PDHA1 |
| H9Z9F1 | 49.80 | 4.81 | 8 | 6 | 6 | 6 | 1 019 | 115.5 | 7.02 | OGDH |
| F6XE81 | 49.26 | 8.37 | 1 | 3 | 3 | 3 | 406 | 46.2 | 5.83 | COPS4 |
| F7H6Z9 | 48.98 | 6.50 | 1 | 7 | 7 | 8 | 1 230 | 136.3 | 5.78 | CAND1 |
| H9FH50 | 48.92 | 4.64 | 5 | 2 | 2 | 2 | 496 | 52.1 | 5.55 | NFKB2 |
| H9FIL0 | 48.38 | 3.26 | 5 | 1 | 2 | 2 | 582 | 67.5 | 5.80 | JMY |
| F6VRQ3 | 48.18 | 4.78 | 4 | 3 | 3 | 3 | 983 | 103.9 | 6.98 | UBAP2L |
| H9ZBD4 | 46.83 | 2.00 | 4 | 1 | 1 | 1 | 699 | 78.0 | 8.38 | ZC3HAV1 |
| H9FLN9 | 46.80 | 2.80 | 1 | 1 | 1 | 1 | 428 | 47.9 | 8.22 | RFX1 |
| H9G1Y1 | 46.19 | 1.31 | 3 | 1 | 1 | 1 | 1 066 | 122.5 | 5.24 | UBE4A |
| H9F1W7 | 44.74 | 9.04 | 3 | 1 | 1 | 1 | 177 | 19.5 | 8.47 | MBOAT7 |
| D5MSD7 | 43.81 | 9.77 | 4 | 2 | 2 | 2 | 215 | 24.7 | 5.94 | Mamu-B |
| F7HML3 | 43.74 | 8.24 | 3 | 3 | 3 | 3 | 437 | 50.1 | 6.74 | EEF1G |
| G7N0C6 | 42.96 | 1.14 | 2 | 1 | 1 | 1 | 1 142 | 125.5 | 8.46 | EGK_19382 |
| H9ZB27 | 42.57 | 3.38 | 3 | 1 | 1 | 1 | 325 | 38.4 | 10.04 | LUC7L |
| A4LAA1 | 42.42 | 2.17 | 4 | 1 | 1 | 1 | 554 | 62.1 | 8.03 | EIF2AK2 |
| F7H310 | 42.37 | 8.16 | 1 | 1 | 1 | 2 | 98 | 11.1 | 5.22 | CSTA |
| F6ZWF5 | 41.77 | 7.52 | 2 | 1 | 1 | 1 | 133 | 14.4 | 10.17 | FAU |
| G7MW18 | 40.75 | 4.56 | 3 | 4 | 4 | 5 | 856 | 95.6 | 9.09 | DDX24 |
| H9FTS8 | 39.31 | 11.11 | 7 | 3 | 3 | 3 | 459 | 50.4 | 5.39 | HNRNPK |
| H9EPG0 | 38.09 | 2.44 | 5 | 1 | 1 | 1 | 532 | 59.6 | 9.16 | HNRNPR |
| H9F226 | 38.09 | 5.90 | 1 | 2 | 2 | 2 | 288 | 32.8 | 8.27 | BZW1 |
| H9EZJ4 | 34.70 | 5.37 | 4 | 4 | 4 | 4 | 726 | 83.0 | 9.33 | GNL2 |
| H9FNM5 | 34.55 | 4.55 | 3 | 2 | 2 | 2 | 396 | 43.0 | 5.17 | TFG |
| Q4G3W3 | 33.36 | 13.33 | 3 | 1 | 1 | 1 | 75 | 8.1 | 8.02 | UQCRC1 |
| G7NEX7 | 33.13 | 6.65 | 3 | 3 | 3 | 3 | 376 | 43.5 | 5.38 | EGK_07498 |
| H9F228 | 31.72 | 5.58 | 2 | 1 | 1 | 1 | 197 | 21.4 | 4.97 | RGP1 |
| H9ENN2 | 30.70 | 0.66 | 2 | 1 | 1 | 1 | 1 976 | 228.9 | 5.54 | MYH10 |
| H9FZB2 | 29.81 | 2.20 | 5 | 2 | 2 | 2 | 727 | 84.9 | 5.55 | UBTF |
| H9YWJ9 | 28.79 | 1.09 | 4 | 1 | 1 | 1 | 916 | 103.8 | 5.55 | USP4 |
| H9F8G7 | 28.46 | 9.91 | 8 | 2 | 2 | 2 | 212 | 24.0 | 7.03 | EEF2 |
| H9FSS7 | 28.14 | 1.97 | 4 | 1 | 1 | 1 | 406 | 44.1 | 9.03 | GTPBP5 |
| F6YRM9 | 27.25 | 3.31 | 7 | 1 | 1 | 1 | 272 | 31.2 | 11.59 | SRSF5 |
| F7F549 | 27.01 | 1.77 | 1 | 1 | 1 | 1 | 396 | 45.5 | 8.84 | VRK1 |
| I2CTF1 | 26.79 | 4.93 | 6 | 2 | 2 | 3 | 568 | 63.6 | 8.40 | SEPT9 |
| H9YYW4 | 26.53 | 1.97 | 1 | 1 | 1 | 1 | 559 | 61.4 | 7.50 | CHAF1B |
| H9F840 | 26.44 | 8.33 | 2 | 2 | 2 | 2 | 348 | 39.5 | 6.39 | EIF3H |
| H9EWH2 | 26.30 | 2.43 | 2 | 1 | 1 | 1 | 329 | 36.7 | 8.25 | SORBS3 |
| H9ESD0 | 26.04 | 6.67 | 2 | 2 | 2 | 2 | 375 | 42.6 | 8.63 | DEK |
| G8F1R1 | 25.56 | 8.98 | 2 | 2 | 2 | 2 | 245 | 27.8 | 9.32 | EGK_21411 |
| G7MJE1 | 25.37 | 8.46 | 3 | 2 | 2 | 2 | 402 | 47.2 | 6.18 | TRIM59 |
| F7HSJ3 | 25.28 | 3.59 | 1 | 1 | 1 | 1 | 418 | 46.9 | 6.93 | AP3M1 |
| F7HPF9 | 25.19 | 6.94 | 3 | 2 | 2 | 2 | 432 | 49.9 | 7.44 | ORC4 |
| F7H3Z6 | 24.78 | 2.89 | 1 | 1 | 1 | 1 | 380 | 42.5 | 8.84 | FEN1 |
| F7DIW5 | 24.65 | 1.95 | 1 | 1 | 1 | 1 | 462 | 51.4 | 7.39 | NFIL3 |
| F6Z1M1 | 24.33 | 3.19 | 3 | 1 | 1 | 1 | 345 | 38.7 | 6.68 | ADK |
| B8Q9H3 | 24.29 | 2.54 | 1 | 1 | 1 | 1 | 354 | 39.8 | 6.15 | Mamu-A |
| H9EYZ5 | 23.57 | 9.30 | 7 | 1 | 1 | 1 | 86 | 9.9 | 6.79 | SRSF8 |
| I0FWG2 | 23.00 | 0.60 | 4 | 1 | 1 | 1 | 1 993 | 220.2 | 8.48 | TRIP12 |
| F6YQX3 | 22.46 | 1.51 | 1 | 1 | 1 | 1 | 795 | 90.8 | 7.46 | DHX15 |
| G7NQG0 | 22.15 | 0.89 | 4 | 1 | 1 | 1 | 1 124 | 128.6 | 6.40 | EGK_12629 |
| H9F8L3 | 20.98 | 2.43 | 3 | 2 | 2 | 2 | 904 | 103.7 | 5.39 | USP11 |
| F7HIU3 | 20.55 | 3.41 | 4 | 3 | 3 | 3 | 792 | 91.2 | 6.47 | WWP1 |
|
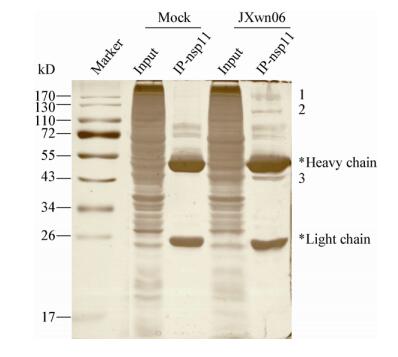
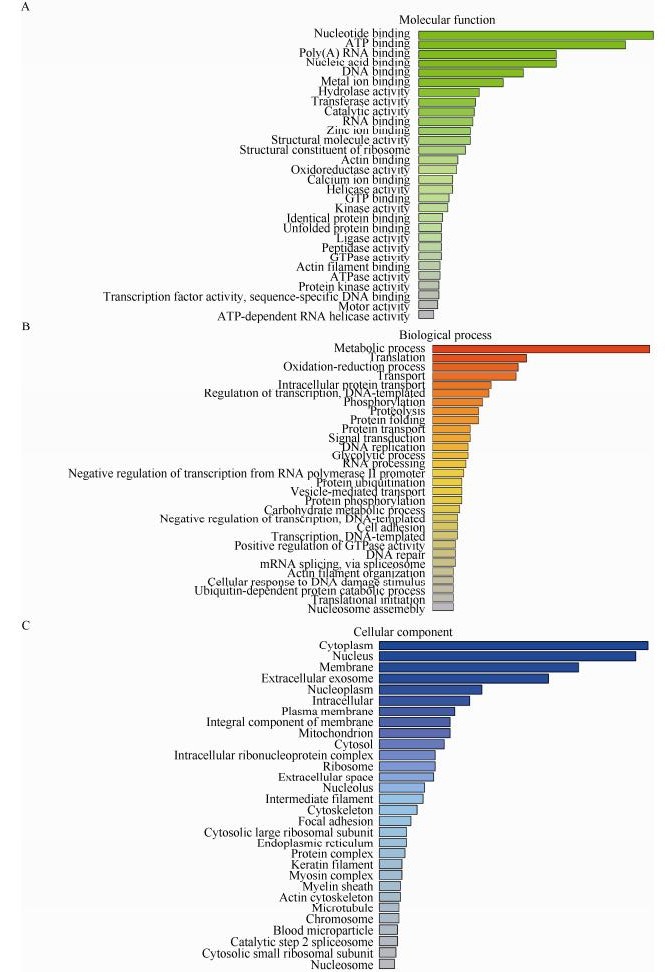
| 图 3 与nsp11互作的宿主细胞蛋白的GO功能分析 Figure 3 GO function analysis of the cellular proteins interacting with nsp11 注:A:分子功能;B:生物过程;C:细胞组成. Note: A: Molecular function; B: Biological process; C: Cellular component. |
|
|
|
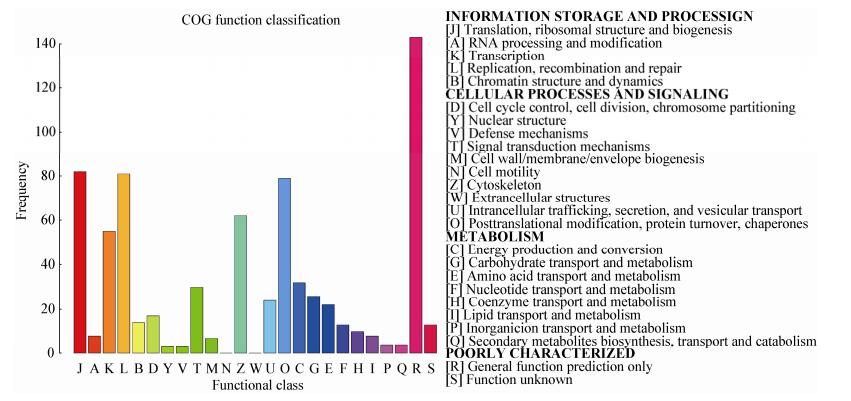
| 图 4 与nsp11互作的宿主细胞蛋白的COG功能分类 Figure 4 COG function classification of the cellular proteins interacting with nsp11 |
|
|
|
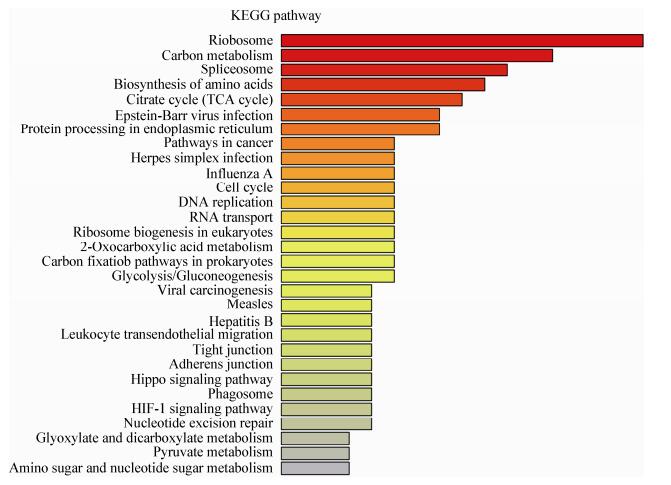
| 图 5 与nsp11互作的宿主细胞蛋白的KEGG代谢通路注释 Figure 5 KEGG pathway annotation of the cellular proteins interacting with nsp11 |
|
|
|
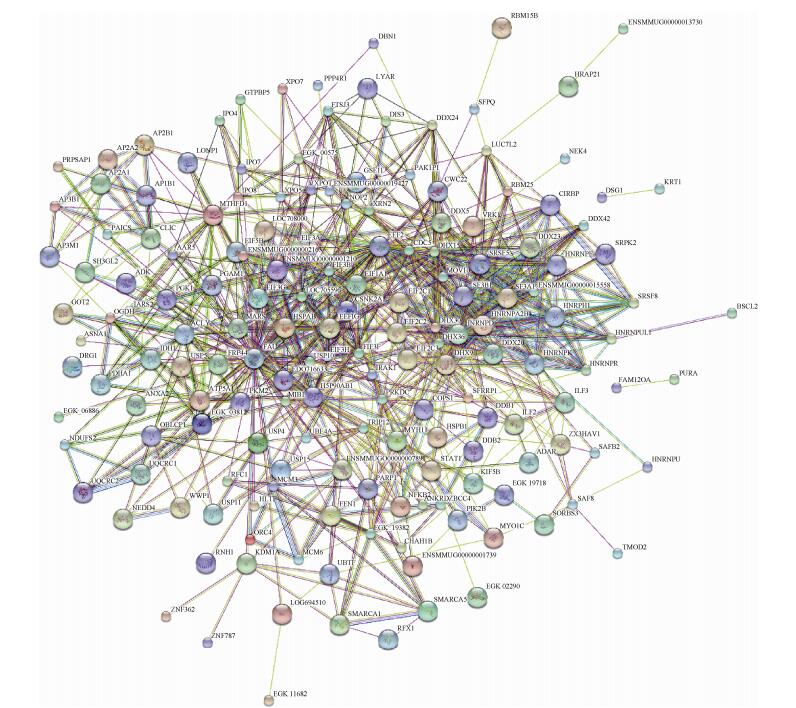
| 图 6 与nsp11互作的宿主细胞蛋白的互作图谱 Figure 6 The interaction network of the cellular proteins interacting with nsp11 |
|
|
|
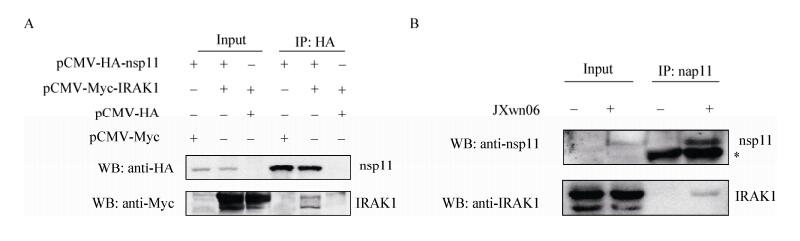
| 图 7 nsp11与IRAK1之间相互作用的验证 Figure 7 Confirmation of the interaction between nsp11 and IRAK1 注:A:在293FT细胞中共转染pCMV-HA-nsp11和pCMV-Myc-IRAK1质粒,验证nsp11和IRAK1之间的相互作用;B:将JXwn06感染于MARC-145细胞,验证nsp11和IRAK1之间的相互作用. Note: A: 293FT cells were co-transfected with pCMV-HA-nsp11 and pCMV-Myc-IRAK1, the interaction between nsp11 and IRAK1 were validated; B: MARC-145 cells were infected with JXwn06, the interaction between nsp11 and IRAK1 were validated. |
|
|
|
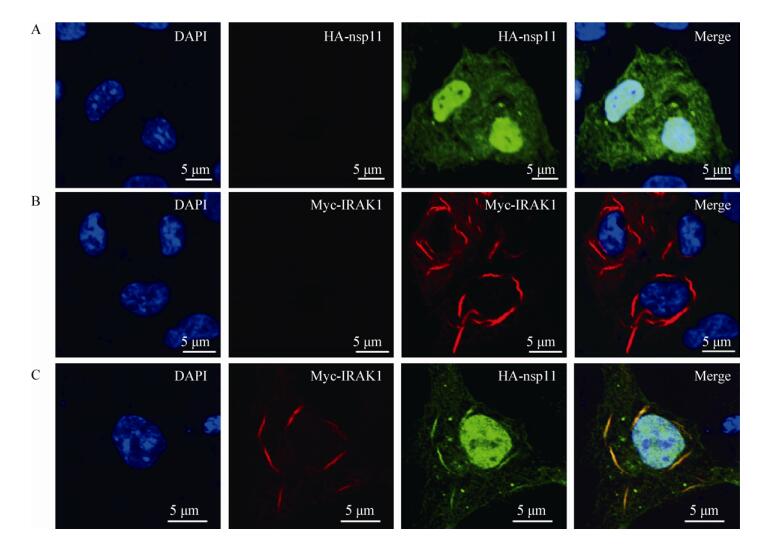
| 图 8 nsp11与IRAK1在细胞中的共定位分析 Figure 8 Co-localization of nsp11 and IRAK1 in MARC-145 cells 注:A:nsp11在MARC-145细胞中的定位情况;B:IRAK1在MARC-145细胞中的定位情况;C:nsp11和IRAK1在MARC-145细胞中的共定位情况. Note: A: Localization of nsp11 in MARC-145 cells; B: Localization of IRAK1 in MARC-145 cells; C: Co-localization of nsp11 and IRAK1 in MARC-145 cells. |
|
|
病毒蛋白可以通过利用与宿主细胞蛋白之间的相互作用而为病毒的复制创造有利的环境,从而引起宿主的严重疾病[23]。研究病毒蛋白与宿主细胞蛋白之间的相互作用可以为解析病毒蛋白的功能以及病毒复制的分子机制奠定基础[24],近年来病毒非结构蛋白与宿主细胞蛋白之间的相互作用以及互作对病毒的复制和致病性的调控成为研究热点。Beura等研究发现PRRSV nsp1β可以与宿主细胞蛋白PCBP1和PCBP2相互作用,PCBP1和PCBP2不仅共定位于病毒的复制复合体上,而且还可以与病毒5' UTR结合来调控病毒RNA的合成[25];Dong等研究证明PRRSV nsp9通过与宿主细胞蛋白pRb的相互作用使pRb被泛素蛋白酶体降解而有利于病毒的复制,nsp9与pRb的互作为病毒的复制提供了有利环境[26];Zhao等研究表明PRRSV nsp9通过与宿主细胞蛋白DDX5的相互作用而改变其亚细胞定位,DDX5从细胞核转移到细胞质中与nsp9进行共定位,作为一种辅助因子正调控病毒的复制[27]。以上研究结果表明,PRRSV的非结构蛋白与宿主细胞蛋白之间的相互作用在调控病毒复制和致病性中扮演着重要角色,而与PRRSV nsp11相互作用的宿主细胞蛋白及互作对PRRSV复制调控和致病性的影响鲜有报道。PRRSV nsp11具有核酸内切酶的活性和去泛素化酶活性[14, 28],对PRRSV的复制起关键作用;此外,nsp11作为干扰素的拮抗剂可以抑制INF-β、IRF3和NF-κB的激活[14-15],为病毒的复制提供有利的环境。PRRSV nsp11在发挥以上功能时需要大量宿主细胞蛋白协助,为了进一步明确nsp11的生物学功能,本研究从nsp11与宿主细胞蛋白的相互作用入手,筛选和鉴定与nsp11相互作用的宿主细胞蛋白并进行生物信息学分析,对于揭示nsp11在病毒复制过程中可能发挥的功能有重要的参考价值。
利用单克隆抗体在病毒感染的宿主细胞中进行免疫沉淀筛选与病毒蛋白相互作用的宿主细胞蛋白,能够更加全面和真实地反映两者的相互作用。本研究利用自行研制的nsp11单克隆抗体进行免疫沉淀结合串联质谱鉴定,筛选得到了201个与nsp11相互作用的宿主细胞蛋白,并对这些蛋白进行了全面的生物信息学分析。分析结果显示,与nsp11互作的宿主细胞蛋白与蛋白质的代谢过程密切相关,由此可以推测,病毒利用了宿主细胞的翻译系统来完成自身蛋白质的合成,进一步证明了nsp11在病毒的复制过程中发挥重要作用;与nsp11互作的宿主细胞蛋白与细胞信号通路的转导有关,为研究PRRSV对宿主的免疫应答的影响提供了新的靶标蛋白和通路;与nsp11互作的宿主细胞蛋白与病原的致病性相关,为研究PRRSV的致病机制提供了参考和新的思路。
为了验证生物信息学分析的可信性,我们利用免疫共沉淀及激光共聚焦实验对筛选出的宿主细胞蛋白IRAK1与PRRSV nsp11的相互作用进行检测,证实PRRSV nsp11可以与内源表达的IRAK1相互作用,且两者共定位于细胞质中。IRAK1是IRAKs家族成员之一,具有多种生物学功能,是一种关键的固有免疫信号调节因子[29],作为细胞溶质激酶、核激酶及接头蛋白,参与调控TLR/IL-1R两个受体家族的信号级联反应,调节TNF-α、Ⅰ型干扰素及AP-1等炎症因子的表达[30-32]。IRAK1在TLR信号通路中发挥正调控的作用,可通过直接调控IRF7的磷酸化而参与调控TLR7/9信号通路介导IFN-α的产生[33];也有研究表明,IRAK1参与调控TLR7信号通路介导IRF5/7的激活[34]。由于nsp11具有拮抗宿主先天性免疫应答反应的作用,而IRAK1又是先天性免疫信号通路中关键的调节因子[29],根据生物信息学分析结果,推测nsp11与IRAK1之间的相互作用可能通过影响IRAK1对其下游信号分子IRF5/7和Ⅰ型干扰素等的调控来影响宿主的先天性免疫反应,从而有利于病毒的复制。
| [1] |
Albina E. Epidemiology of porcine reproductive and respiratory syndrome (PRRS): an overview[J]. Veterinary Microbiology, 1997, 55(1/4): 309-316. |
| [2] |
Keffaber KK. Reproductive failure of unknown etiology[J]. American Association of Swine Practitioners Newsletter, 1989, 1(2): 1-9. |
| [3] |
Wensvoort G, Terpstra C, Pol JMA, et al. Mystery swine disease in the Netherlands: the isolation of Lelystad virus[J]. Veterinary Quarterly, 1991, 13(3): 121-130. DOI:10.1080/01652176.1991.9694296 |
| [4] |
Kuwahara H, Nunoya T, Tajima M, et al. An outbreak of porcine reproductive and respiratory syndrome in Japan[J]. Journal of Veterinary Medical Science, 1994, 56(5): 901-909. DOI:10.1292/jvms.56.901 |
| [5] |
Neumann EJ, Kliebenstein JB, Johnson CD, et al. Assessment of the economic impact of porcine reproductive and respiratory syndrome on swine production in the United States[J]. Journal of the American Veterinary Medical Association, 2005, 227(3): 385-392. DOI:10.2460/javma.2005.227.issue-3 |
| [6] |
Chang CC, Chung WB, Lin MW, et al. Porcine reproductive and respiratory syndrome (PRRS) in Taiwan Ⅰ. Viral isolation[J]. Journal of the Chinese Society Veterinary Science, 1993, 19(4): 268-276. |
| [7] |
Guo BQ, Chen ZS, Liu WX, et al. Isolation and identification of porcine reproductory and respiratory syndrome (PRRS) virus from aborted fetuses suspected of PRRS[J]. Chinese Journal of Animal and Poultry Infectious Diseases, 1996(2): 1-5. 郭宝清, 陈章水, 刘文兴, 等. 从疑似PRRS流产胎儿分离PRRSV的研究[J]. 中国畜禽传染病, 1996(2): 1-5. |
| [8] |
Tian KG, Yu XL, Zhao TZ, et al. Emergence of fatal PRRSV variants: unparalleled outbreaks of atypical PRRS in China and molecular dissection of the unique hallmark[J]. PLoS One, 2007, 2(6): e526. DOI:10.1371/journal.pone.0000526 |
| [9] |
Li Y, Zhou L, Zhang JL, et al. Nsp9 and Nsp10 Contribute to the fatal virulence of highly pathogenic porcine reproductive and respiratory syndrome virus emerging in China[J]. PLoS Pathogens, 2014, 10(7): e1004216. DOI:10.1371/journal.ppat.1004216 |
| [10] |
Du JG, Ge XN, Liu Y, et al. Targeting swine leukocyte antigen class Ⅰ molecules for proteasomal degradation by the nsp1α replicase protein of the Chinese highly pathogenic porcine reproductive and respiratory syndrome virus strain JXwn06[J]. Journal of Virology, 2016, 90(2): 682-693. DOI:10.1128/JVI.02307-15 |
| [11] |
Shi XB, Zhang XZ, Chang YZ, et al. Nonstructural protein 11 (nsp11) of porcine reproductive and respiratory syndrome virus (PRRSV) promotes PRRSV infection in MARC-145 cells[J]. BMC Veterinary Research, 2016, 12: 90. DOI:10.1186/s12917-016-0717-5 |
| [12] |
Rascón-Castelo E, Burgara-Estrella A, Mateu E, et al. Immunological features of the non-structural proteins of porcine reproductive and respiratory syndrome virus[J]. Viruses, 2015, 7(3): 873-886. DOI:10.3390/v7030873 |
| [13] |
Nedialkova DD, Ulferts R, van den Born E, et al. Biochemical characterization of arterivirus nonstructural protein 11 reveals the nidovirus-wide conservation of a replicative endoribonuclease[J]. Journal of Virology, 2009, 83(11): 5671-5682. DOI:10.1128/JVI.00261-09 |
| [14] |
Wang D, Fan JX, Fang LR, et al. The nonstructural protein 11 of porcine reproductive and respiratory syndrome virus inhibits NF-κB signaling by means of its deubiquitinating activity[J]. Molecular Immunology, 2015, 68(2): 357-366. DOI:10.1016/j.molimm.2015.08.011 |
| [15] |
Sun Y, Ke H, Han M, et al. Nonstructural protein 11 of porcine reproductive and respiratory syndrome virus suppresses both MAVS and RIG-Ⅰ expression as one of the mechanisms to antagonize type Ⅰ interferon production[J]. PLoS One, 2016, 11(12): e0168314. DOI:10.1371/journal.pone.0168314 |
| [16] |
Wang C, Shi XB, Zhang XZ, et al. The endoribonuclease activity essential for the nonstructural protein 11 of porcine reproductive and respiratory syndrome virus to inhibit NLRP3 inflammasome-mediated IL-1β induction[J]. DNA and Cell Biology, 2015, 34(12): 728-735. DOI:10.1089/dna.2015.2929 |
| [17] |
Zhang X, Shi HY, Chen JF, et al. Identification of the interaction between vimentin and nucleocapsid protein of transmissible gastroenteritis virus[J]. Virus Research, 2015, 200: 56-63. DOI:10.1016/j.virusres.2014.12.013 |
| [18] |
Brito AF, Pinney JW. Protein-Protein interactions in virus-host systems[J]. Frontiers in Microbiology, 2017, 8: 1557. DOI:10.3389/fmicb.2017.01557 |
| [19] |
Conesa A, G tz S, García-Gómez JM, et al. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research[J]. Bioinformatics, 2005, 21(18): 3674-3676. DOI:10.1093/bioinformatics/bti610 |
| [20] |
Kanehisa M, Araki M, Goto S, et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Research, 2008, 36(Database issue): D480-D484. |
| [21] |
Wang L, Zhou L, Zhang H, et al. Interactome profile of the host cellular proteins and the nonstructural protein 2 of porcine reproductive and respiratory syndrome virus[J]. PLoS One, 2014, 9(6): e99176. DOI:10.1371/journal.pone.0099176 |
| [22] |
Jin H, Zhou L, Ge XN, et al. Cellular DEAD-box RNA helicase 18 (DDX18) promotes the PRRSV replication via interaction with virus nsp2 and nsp10[J]. Virus Research, 2017, 238: 204-212. DOI:10.1016/j.virusres.2017.05.028 |
| [23] |
Khadka S, Vangeloff AD, Zhang CY, et al. A physical interaction network of dengue virus and human proteins[J]. Molecular & Cellular Proteomics, 2011, 10(12): M111.012187. |
| [24] |
Toh QC, Tan TL, Teo WQ, et al. Identification of cellular membrane proteins interacting with hepatitis B surface antigen using yeast split-ubiquitin system[J]. International Journal of Medical Sciences, 2005, 2(3): 114-117. |
| [25] |
Beura LK, Dinh PX, Osorio FA, et al. Cellular poly(C) binding proteins 1 and 2 interact with porcine reproductive and respiratory syndrome virus nonstructural protein 1β and support viral replication[J]. Journal of Virology, 2011, 85(24): 12939-12949. DOI:10.1128/JVI.05177-11 |
| [26] |
Dong JG, Zhang N, Ge XN, et al. The interaction of nonstructural protein 9 with retinoblastoma protein benefits the replication of genotype 2 porcine reproductive and respiratory syndrome virus in vitro[J]. Virology, 2014, 464-465: 432-440. DOI:10.1016/j.virol.2014.07.036 |
| [27] |
Zhao SC, Ge XN, Wang XL, et al. The DEAD-box RNA helicase 5 positively regulates the replication of porcine reproductive and respiratory syndrome virus by interacting with viral Nsp9 in vitro[J]. Virus Research, 2015, 195: 217-224. DOI:10.1016/j.virusres.2014.10.021 |
| [28] |
Zhang MF, Li XR, Deng ZQ, et al. Structural biology of the arterivirus nsp11 endoribonucleases[J]. Journal of Virology, 2016, 91(1): e01309-16. |
| [29] |
Flannery S, Bowie AG. The interleukin-1 receptor-associated kinases: critical regulators of innate immune signalling[J]. Biochemical Pharmacology, 2010, 80(12): 1981-1991. DOI:10.1016/j.bcp.2010.06.020 |
| [30] |
Cai JJ, Zhou J. The research advance of IRAK1 in immune regulation[J]. International Journal of Immunology, 2011, 34(6): 327-331. 蔡娇娇, 周俊. 白介素-1受体相关激酶1在免疫调控中的研究进展[J]. 国际免疫学杂志, 2011, 34(6): 327-331. |
| [31] |
Ho BC, Yu IS, Lu LF, et al. Inhibition of miR-146a prevents enterovirus-induced death by restoring the production of type Ⅰ interferon[J]. Nature Communications, 2014, 5: 3344. |
| [32] |
Rhyasen GW, Bolanos L, Fang J, et al. Targeting IRAK1 as a therapeutic approach for myelodysplastic syndrome[J]. Cancer Cell, 2013, 24(1): 90-104. DOI:10.1016/j.ccr.2013.05.006 |
| [33] |
Uematsu S, Sato S, Yamamoto M, et al. Interleukin-1 receptor-associated kinase-1 plays an essential role for Toll-like receptor (TLR)7-and TLR9-mediated interferon-α induction[J]. The Journal of Experimental Medicine, 2005, 201(6): 915-923. DOI:10.1084/jem.20042372 |
| [34] |
Schoenemeyer A, Barnes BJ, Mancl ME, et al. The interferon regulatory factor, IRF5, is a central mediator of toll-like receptor 7 signaling[J]. Journal of Biological Chemistry, 2005, 280(17): 17005-17012. DOI:10.1074/jbc.M412584200 |
 2017, Vol. 44
2017, Vol. 44