扩展功能
文章信息
- 陈诚, 李强, 黄文丽, 王剑, 伏荣桃, 罗曦, 卢代华
- CHEN Cheng, LI Qiang, HUANG Wen-Li, WANG Jian, FU Rong-Tao, LUO Xi, LU Dai-Hua
- 羊肚菌白霉病发生对土壤真菌群落结构的影响
- Effects of Morchella white mold disease on soil fungal community structure
- 微生物学通报, 2017, 44(11): 2652-2659
- Microbiology China, 2017, 44(11): 2652-2659
- DOI: 10.13344/j.microbiol.china.170030
-
文章历史
- 收稿日期: 2017-01-11
- 接受日期: 2017-04-24
- 优先数字出版日期(www.cnki.net): 2017-05-29
2. 四川省农业科学院生物技术核技术研究所 四川 成都 610066
2. Biotechnology and Nuclear Technology Research Institute, Sichuan Academy of Agricultural Sciences, Chengdu, Sichuan 610066, China
羊肚菌(Morel)属子囊菌亚门Ascomycota盘菌纲Discomycetes盘菌目Pezizales羊肚菌科Morchellaceae羊肚菌属Morchella,是一类名贵的食药两用真菌[1]。羊肚菌含有丰富的人体必需氨基酸、多糖和萜类化合物,具有增强免疫、抗氧化、抗肿瘤、保肝保肾等活性作用[2-6]。随着栽培规模的扩大,病虫害成为限制羊肚菌产业发展的重要因素之一。白霉病因其在田间发生的症状而得名,是在羊肚菌子实体菌柄或菌盖部位表现白色绒毛的症状,由长毛拟青霉(Paecilomyces penicillatus)引发[7],是羊肚菌栽培最常见的病害,严重影响了羊肚菌的产量和品质。因此,研究白霉病发生后土壤的微生物群落结构,可以为该病害的发生机制和生物防治研究提供一定的基础数据。
羊肚菌不适宜连作,连作会造成产量降低,白霉病发病更严重。然而农作物连作障碍和病害高发又与土壤的微生物群落结构有密切的联系[8]。土壤中的微生物包括有益微生物和有害微生物,作物连作导致病害高发的原因主要是有益微生物减少和病原菌的积累[9]。根际是植物生态系统和土壤生态系统交叉形成的特殊区域,根际微生物可通过竞争、拮抗和诱导系统抗性等机制抑制病原菌,也可通过病原菌的积累导致植物死亡[10]。如张丽娟等[11]对大蒜根际土壤微生物群落多样性进行研究表明,根际土壤微生物与大蒜根腐病存在一定的相关性。
随着羊肚菌栽培规模的不断扩大,白霉病对羊肚菌产量和品质的影响日益突出,其发病率由2013年的10%增加到2015年的60%-80%,该病害发生在子实体整个生长时期乃至储藏期,但其发生的机制仍不明确,且尚无对白霉病有效的防治办法[7]。本研究运用高通量测序技术研究未栽培羊肚菌土壤和羊肚菌栽培地白霉病发病子实体和未表现白霉病子实体的根际土壤的真菌群落结构,探讨白霉病发生对栽培地真菌群落结构的影响,为羊肚菌白霉病的防控提供理论指导。
1 材料与方法 1.1 样品采集土壤样品采集于四川省成都市温江区羊马镇羊肚菌栽培基地,选用随机取样的办法,采集白霉病发病子实体和正常生长子实体各12株,按Cui等[12]的方法采集子实体根际土,正常生长的子实体根际土样编号记为Soil-1,染病子实体根际土样编号记为Soil-2,每个处理采集3个样品。以相同环境下未栽培羊肚菌的土壤为对照,对照土样标号记为CK-1。将土样装入无菌EP管,用冰盒保存,立即带回微生物实验室。
1.2 主要试剂和仪器DNA提取试剂盒(E.Z.N.A.® soil DNA Kit),杭州西顿生物科技有限公司;胶回收试剂盒(DP209-02),北京天根生化科技有限公司;引物合成,上海派森诺生物科技有限公司;Q5® High-Fidelity DNA Polymerase (MO491L),北京NEB有限公司。2700型PCR扩增仪,美国ABI公司;DYY-6C电泳仪,北京六一生物科技有限公司;MiSeq PE300测序平台,美国Illumina公司。
1.3 DNA提取、PCR扩增及高通量测序基因组DNA按试剂盒操作说明进行。实验采用18S V4区作为目标DNA序列,扩增引物为0817F (5′-TTAGCATGGAATAATRRAATAGGA-3′)和1196R (5′-TCTGGACCTGGTGAGTTTCC-3′)[13]。PCR反应体系(25μL)为:5×reaction buffer 5 μL,5×GC buffer 5 μL,dNTPs (2.5 mmol/L) 2 μL,正反向引物(10μmol/L)各1μL,DNA模板(20 ng/μL) 2μL,ddH2O 8.75 μL,Q5 DNA聚合酶(2 U/μL) 0.25 μL。PCR反应条件为:95 ℃ 2 min;95℃ 45 s,55℃ 45 s,72℃ 45 s,32个循环;72 ℃ 10 min。PCR扩增完成后用1.0%琼脂糖凝胶电泳检测,电压100 V,电泳时间为20 min。PCR产物检测合格后,将样品送至上海派森诺生物科技有限公司在Illumina MiSeq平台进行高通量测序。
1.4 数据分析采用滑动窗口法对原始数据的双端序列逐一做质量筛查,利用FLASH软件[14]对通过质量初筛的双端序列根据重叠碱基进行配对连接。运用QIIME软件[15]识别疑问序列,通过Mothur软件[16]调用UCHIME检查并剔除嵌合体序列[17]。使用QIIME软件,调用UCLUST这一序列比对工具[18],对获得的高质量序列按97%的序列相似度进行归并和OTU划分,采用Silva数据库对序列进行比对[19]。获得OTU丰度矩阵之后绘制稀疏曲线[20]。采用Mothur软件分析真菌样品的Alpha多样性,包括Chao1指数[21]、ACE指数[22]、Simpson指数[23]和Shannon指数[24]。使用R软件,对丰度前50位的属进行聚类分析并绘制热图。PCA分析采用Ramette的方法[25]。柱状图制作采用Exclle (2007),试验数据采用SPSS (19.0)[26]进行方差分析。
2 结果与分析 2.1 测序结果及真菌多样性分析通过Illumina MiSeq高通量测序,9份样品总共得到393 347条有效序列,经过过滤和去除嵌合体之后得到384 732条优质序列。优质序列的长度在153-447 bp,多集中在320-340 bp。样品的稀释曲线(图 1)显示,随着测序数据量的增大,样品的稀释曲线趋于平坦,说明测序结果基本覆盖了样品中绝大多数真菌的生物学信息,可用于后续分析。
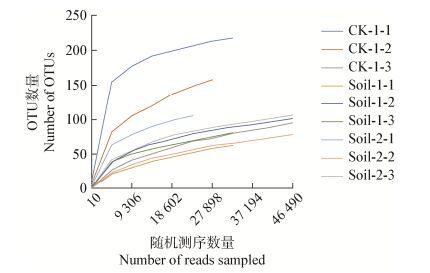
|
| 图 1 各样品测序稀释曲线 Figure 1 Rarefaction curve of the sequenced samples 注:图表示OTU数随着样本测序深度而变化. CK-1为对照土样;Soil-1为正常生长的子实体根际土;Soil-2为白霉病染病子实体根际土.各3个重复,下同. Note: The figure shows that the number of OTUs varies with the number of sequence reads. CK-1: control soil; Soil-1: rhizosphere soil of healthy Morchella; Soil-2: Rhizosphere soil of diseased Morchella. Three replicates of each sample, the same below. |
|
|
样品的Alpha多样性指数结果(表 1)表明,未栽培羊肚菌的真菌丰富度明显高于羊肚菌栽培地土壤真菌,发生白霉病的羊肚菌根际土壤中的真菌OTU数比未表现出白霉病症的羊肚菌的根际土壤中多。未栽培羊肚菌土壤的Chao1指数和ACE指数显著高于羊肚菌根际土,其中有白霉病症的羊肚菌Chao1指数和ACE指数均大于正常生长的羊肚菌根际土。综合考虑了真菌群落的丰富度和均匀度,未栽培羊肚菌土壤的Simpson指数和Shannon指数显著大于羊肚菌生长的土壤,正常生长的羊肚菌根际土的Simpson指数和Shannon指数小于染病的羊肚菌根际土,说明羊肚菌栽培地区土壤真菌多样性降低,相比于正常生长羊肚菌,染病羊肚菌子实体根际土的真菌多样性更高。
| Items | CK-1 | Soil-1 | Soil-2 |
| OTUs | 160.33±36.20a | 86.00±13.23b | 98.67±13.65b |
| Chao1 | 112.00±43.15a | 47.67±8.82b | 57.00±13.65b |
| ACE | 161.91±27.01a | 67.90±12.05b | 71.00±11.39b |
| Simpson | 0.797±0.031a | 0.094±0.045b | 0.189±0.134b |
| Shannon | 3.860±0.362a | 0.437±0.179b | 0.752±0.462b |
| 注:同一行中不同字母表示差异显著(P<0.05). Note: Different letters in the same row indicate significant difference (P<0.05). | |||
根据分类结果,所测样品由5个门的真菌组成,包括子囊菌门、担子菌门、接合菌门、壶菌门和Rozellomycota (图 2)。各样品子囊菌门的种类所占的比例都较大,接合菌门和壶菌门的真菌在未栽培羊肚菌的土壤中所占比例都显著高于羊肚菌根际土,说明羊肚菌栽培后该类群的真菌受到了抑制。Rozellomycota的真菌在羊肚菌子实体根际土的比例比对照土壤的比例大,这可能是羊肚菌的栽培有利于该类群真菌的生长。此外,担子菌门的真菌在羊肚菌染病子实体根际土的比例最大,其次是对照土壤,在正常生长子实体根际土的比例最小,显示羊肚菌的栽培可能抑制了此类真菌,而白霉病发生后,该类群的真菌比例又一定程度地增长。
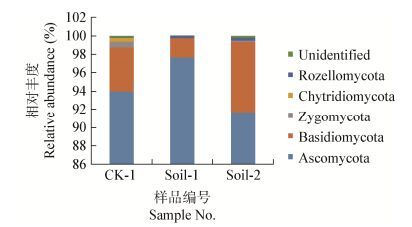
|
| 图 2 门水平上各样品的真菌群落组成 Figure 2 The composition of the samples at the level of the phylum 注:不同颜色代表不同真菌门,纵坐标代表各真菌门所占百分比,在门水平未被注释的真菌种用unidentified表示. Note: Different colors represent different phylum of fungus, the ordinate represents the percentage of each phylum. Sequences could not be classified into any groups were assighed as unidentified. |
|
|
通过UNITE数据库比对,统计各样本在各个分类等级水平的群落组成。选择Soil-1和Soil-2含量大于0.1%且CK土壤中含量大于1%的真菌分类单元为代表类群,其余部分合并为Others (其他),获得各样品的群落组成(图 3)。由图 3可以看出,对照土样的优势类群主要包括Trichoderma、Rhynchosporium、Staphylotrichum、Cyphellophora、Myriangiales和Microascaceae。Soil-1样品中占比最高的类群为Morchella,所占比例达到96.207%,其他优势类群有Trichoderma、Rozellomycota、Myriangiales和Polyporales。Soil-2样品中占比最高的类群也是Morchella,所占比例为89.949%,其他优势类群有Trichoderma、Ascomycota、Rozellomycota、Myriangiales、Mortierella、Polyporales和Ceratobasidiaceae。
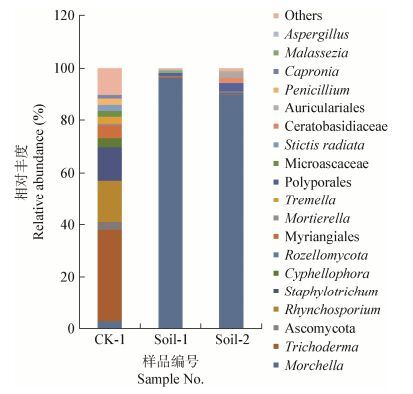
|
| 图 3 各样品的真菌群落组成 Figure 3 The composition of the samples 注:不同颜色代表不同真菌属,若在属水平上未被注释则以上一级分类单元表示,以此类推;纵坐标代表各真菌属所占的百分比. Note: Different colors represent different genus. Sequences could not be classified into genus were given names of the upper level and so on. The ordinate represents the percentage of each genus. |
|
|
通过统计发现,羊肚菌栽培后,有23个类群的真菌丰度降低或消失,包括Onygenales、Myceliophthora、Arthrinium、Stachybotrys、Glomerellaceae、Trichocomaceae、Verticillium、Phaeoacremonium、Devriesia、Clonostachys、Mycothermus、Neoaleurodiscus、Graphium、Lectera、Sphaerulina、Phaeosphaeriaceae、Lulwoana、Chaetomiaceae、Codinaeopsis、Eurotiales、Sordariale、Pseudeurotium和Cordyceps。有11个类群的真菌仅在对照土壤和正常生长的羊肚菌根际子实体中被检测到,在染病子实体根际土中消失,包括Chytridiomycota、Cladophialophora、Thermoascus、Microascus、Periconia、Humicola、Spizellomyces、Cryptococcus、Simocybe、Cylindrocarpon和Thermomyces。此外,有6个类群的真菌仅在白霉病染病子实体根际土中被检测到,包括Halosphaeriaceae、Candida、Arachnomycetaceae、Rhodotorula、Ochroconis和Cordycipitaceae。可见,羊肚菌栽培和白霉病发生对土壤真菌群落结构有较大的影响。
2.3 真菌群落结构聚类分析利用Heatmap图通过颜色梯度及相似程度来反映各处理在各分类阶元水平上真菌群落组成的相似性、多样性及丰富度,其中红色代表在对应样本中丰度较高的属,绿色代表丰度较低的属;纵向是样品的聚类情况,反映各样品在属水平上群落组成的相似性(图 4)。从图 4结果看出,对照土壤和羊肚菌根际土的优势类群有较大差异,染病子实体根际土和未表现病害的羊肚菌根际土的优势类群也有一定差异。对照土样中有2个及以上处理表现出有较高丰度的属有Conocybe、Cyphellophora、Sagenomella、Hypocrea、Ascobolus、Trichoderma、Lecanicillium和Rhizophydium。正常生长的羊肚菌子实体根际土中有2个及以上处理表现较高丰度的属有Morchella、Ascobolus、Chaetomium和Rhizophydium。白霉病发病子实体根际土中有2个及以上处理表现出较高丰度的属有Morchella、Aspergillus、Aureobasidium、Paecilomyces、Conocybe和Pseudaleuria。可见,白霉病发生地土壤的优势真菌类群产生了一定的变化。

|
| 图 4 各样品真菌聚类分析热图 Figure 4 Heatmap of samples clustering 注:横向为各样本的聚类结果,纵向为各分类单元的聚类结果.红色代表在对应样本中丰度较高的属,绿色代表丰度较低的属. Note: The clustering results for each sample were in the horizontal direction and the clustering results for the taxonomic units were in the vertical direction. Red represents the genus with higher abundance in the corresponding sample, and green represents the genus with lower abundance. |
|
|
PCA分析通过线性变换,将原始的高维数据通过线性变换组合,投影到维度较低的空间坐标系(即主成分)中,从而达到降维、简化数据结构的目的,展现样本的自然分布。每个点代表一个样本,不同颜色的点属于不同样本(组),两点之间的距离越近,表明两个样本之间的微生物群落结构相似度越高,差异越小。坐标轴括号中的百分比代表了对应的主成分所能解释的原始数据中差异的比例。从图 5结果可看出,未栽培羊肚菌土壤的真菌群落结构与羊肚菌子实体根际土的真菌群落结果有较大的差异;有2个染病和正常生长的羊肚菌子实体根际土的真菌群落结构存在重叠的现象,说明两者的真菌群落相似性很高,差异很小;同时,各有一个白霉病染病子实体和正常生长的羊肚菌子实体根际真菌群落结构与其他样品有较大差异,说明白霉病发生后,土壤真菌群落结构产生了一定变化。
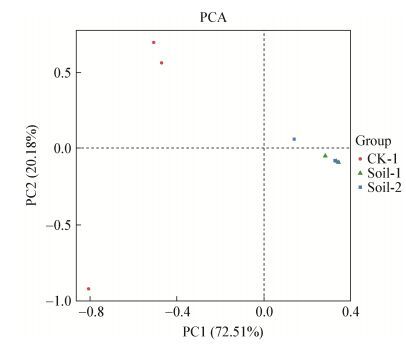
|
| 图 5 各样品真菌的主成分分析 Figure 5 Principal component analysis of fungal diversity of samples |
|
|
可培养的方法观察到的微生物不到微生物群落的1%[27],无法全面反映微生物的真实状况。Illumina MiSeq测序技术作为新一代测序技术,能够直观地反映微生物群落组成,而且具有高通量、价格低、运行周期短的优势,已被广泛应用于微生物群落结构研究[28]。本实验中各样品测序的稀释曲线都趋于平缓,能够真实全面地反映各样品真菌群落结构的组成情况。从土壤真菌多样性指数可看出,未栽培羊肚菌土壤的真菌丰度和多样性显著高于羊肚菌根际土真菌多样性,这与前人的研究结果一致[29],可能与土壤中优势的羊肚菌种群对其它种群的抑制有关。此外,染病子实体根际土真菌丰度和多样性大于正常生长的羊肚菌根际土,这可能是白霉病病原菌的侵袭打破了羊肚菌的优势地位,促进了其他羊肚菌根际真菌的繁殖,也有可能是多种病原真菌在羊肚菌根际聚集扩散,从而导致羊肚菌发病,具体机制需进一步研究。
土壤真菌群落结构分析中,木霉属和多腔菌目的真菌在3种样品中均是优势类群,说明羊肚菌栽培和白霉病发生对其影响较小。被孢霉属和角担菌科的真菌仅在白霉病染病子实体中表现为优势类群,被孢霉属的真菌被广泛用于脂质合成[30],而角担菌科的真菌能造成植物的多种病害[31-32],可能它们加快了羊肚菌白霉病发病的进程。羊肚菌白霉病发生后,有11个类群的真菌消失,有6个类群的真菌仅在染病子实体根际土中被检测到,可能是病原菌入侵后特定类群的真菌受到了抑制而消亡,同时又促进一些类群真菌的生长,原因有待进一步分析。从单一样品的角度分析各处理的群落组成结果可看出,有6个属的真菌在2个以上的重复样品中表现较高的丰度,其中包括拟青霉属的真菌。羊肚菌白霉病是由拟青霉属的长毛拟青霉引发[7],可能白霉病的病原菌是通过根部侵染而导致子实体发病。
主成分分析结果中,未栽培羊肚菌土壤真菌与羊肚菌根际真菌差异很大,有2个染病子实体和正常生长的子实体根际真菌群落结构存在重叠,表明羊肚菌栽培对土壤真菌群落结构具有较大影响,可以塑造土壤真菌的群落,而单个样品的差异可能与白霉病病原菌的入侵有关,接下来将对羊肚菌白霉病的发生、传播规律和病原菌的侵染机制进行深入研究,从而推动对羊肚菌白霉病的防控。
4 结论通过Illumina MiSeq高通量测序,9份样品总共得到393 347条有效序列,经过过滤和去除嵌合体之后得到384 732条优质序列。未栽培羊肚菌土壤真菌OTU数、Chao1指数、ACE指数、Simpson指数和Shannon指数最大,染病子实体根际土次之,正常生长羊肚菌子实体根际土最小;各个处理的真菌群落结构和优势真菌类群有较大差异。可见,羊肚菌栽培后真菌多样性降低,白霉病发生后根际真菌种类增多,土壤真菌群落结构发生变化,优势真菌类群也产生了较大差异。
致谢: 感谢成都市农林科学院曾先富老师和熊维全老师在采样时提供的帮助。| [1] |
Kuo M. Morels[M]. Ann Arbor: University of Michigan Press, 2005, SB353.K86.
|
| [2] |
Cui HL, Chen Y, Wang SS, et al. Isolation, partial characterisation and immunomodulatory activities of polysaccharide from Morchella esculenta[J]. Journal of the Science of Food and Agriculture, 2011, 91(12): 2180-2185. |
| [3] |
Fu LH, Wang YP, Wang JJ, et al. Evaluation of the Antioxidant activity of extracellular polysaccharides from Morchella esculenta[J]. Food & Function, 2013, 4(6): 871-879. |
| [4] |
Hu ML, Chen Y, Wang C, et al. Induction of apoptosis in HepG2 cells by polysaccharide MEP-Ⅱ from the fermentation broth of Morchella esculenta[J]. Biotechnology Letters, 2013, 35(1): 1-10. DOI:10.1007/s10529-012-0917-4 |
| [5] |
Nitha B, Fijesh PV, Janardhanan KK. Hepatoprotective activity of cultured mycelium of Morel mushroom, Morchella esculenta[J]. Experimental and Toxicologic Pathology, 2013, 65(1/2): 105-112. |
| [6] |
Nitha B, Janardhanan KK. Aqueous-ethanolic extract of morel mushroom mycelium Morchella esculenta, protects cisplatin and gentamicin induced nephrotoxicity in mice[J]. Food and Chemical Toxicology, 2008, 46(9): 3193-3199. DOI:10.1016/j.fct.2008.07.007 |
| [7] |
He XL, Peng WH, Miao RY, et al. White mold on cultivated morels caused by Paecilomyces penicillatus[J]. FEMS Microbiology Ecology, 2017, 364(5): fnx037. |
| [8] |
Wang F, Li SG, Xu FH, et al. The research progress on mechanism of continuous cropping obstacle[J]. Soil and Fertilizer Sciences in China, 2013(5): 6-13. 王飞, 李世贵, 徐凤花, 等. 连作障碍发生机制研究进展[J]. 中国土壤与肥料, 2013(5): 6-13. DOI:10.11838/sfsc.20130502 |
| [9] |
Xiong W, Zhao QY, Zhao J, et al. Different continuous cropping spans significantly affect microbial community membership and structure in a vanilla-grown soil as revealed by deep pyrosequencing[J]. Microbial Ecology, 2015, 70(1): 209-218. DOI:10.1007/s00248-014-0516-0 |
| [10] |
Zhou WJ, Lü DG, Qin SJ. Research progress in interaction between plant and rhizosphere microorganism[J]. Journal of Jilin Agricultural University, 2016, 38(3): 253-260. 周文杰, 吕德国, 秦嗣军. 植物与根际微生物相互作用关系研究进展[J]. 吉林农业大学学报, 2016, 38(3): 253-260. |
| [11] |
Zhang LJ, Mao J, Zhang ZD, et al. Study of microbial community diversity in rhizosphere soil from garlic infected by root rot disease[J]. Xinjiang Agricultural Sciences, 2013, 50(11): 2109-2117. 张丽娟, 茆军, 张志东, 等. 新疆大蒜根腐型病害根际土壤微生物群落多样性初探[J]. 新疆农业科学, 2013, 50(11): 2109-2117. |
| [12] |
Cui HY, Yang XY, Lu DX. Isolation and characterization of bacteria from the rhizosphere and bulk soil of Stellera chamaejasme L[J]. Canadian Journal of Microbiology, 2015, 61(3): 171-181. DOI:10.1139/cjm-2014-0543 |
| [13] |
Rousk J, B th E, Brookes PC, et al. Soil bacterial and fungal communities across a pH gradient in an arable soil[J]. The ISME Journal, 2010, 4(10): 1340-1351. DOI:10.1038/ismej.2010.58 |
| [14] |
Magoč T, Salzberg SL. FLASH: fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [15] |
Caporaso JG, Kuczynski J, Stombaugh J, et al. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [16] |
Schloss PD, Westcott SL, Ryabin T, et al. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. DOI:10.1128/AEM.01541-09 |
| [17] |
Edgar RC, Haas BJ, Clemente JC, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381 |
| [18] |
Edgar RC. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [19] |
Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41(D1): D590-D596. |
| [20] |
Heck KL, van Belle G, Simberloff D. Explicit calculation of the rarefaction diversity measurement and the determination of sufficient sample size[J]. Ecology, 1975, 56(6): 1459-1461. DOI:10.2307/1934716 |
| [21] |
Chao A. Nonparametric estimation of the number of classes in a population[J]. Scandinavian Journal of Statistics, 1984, 11(4): 265-270. |
| [22] |
Chao A, Yang MCK. Stopping rules and estimation for recapture debugging with unequal failure rates[J]. Biometrika, 1993, 80(1): 193-201. DOI:10.1093/biomet/80.1.193 |
| [23] |
Simpson EH. Measurement of Diversity[J]. Nature, 1949, 163: 688. DOI:10.1038/163688a0 |
| [24] |
Shannon CE. A mathematical theory of communication[J]. Bell Labs Technical Journal, 1948, 27(3): 379-423. DOI:10.1002/bltj.1948.27.issue-3 |
| [25] |
Ramette A. Multivariate analyses in microbial ecology[J]. FEMS Microbiology Ecology, 2007, 62(2): 142-160. DOI:10.1111/fem.2007.62.issue-2 |
| [26] |
Park E, Cho M, Ki CS. Correct use of repeated measures analysis of variance[J]. The Korean Journal of Laboratory Medicine, 2009, 29(1): 1-9. DOI:10.3343/kjlm.2009.29.1.1 |
| [27] |
Wang SG, Hou YL. Application of phospholipid fatty acid method in soil microbial analysis[J]. Microbiology China, 2004, 31(1): 114-117. 王曙光, 侯彦林. 磷脂脂肪酸方法在土壤微生物分析中的应用[J]. 微生物学通报, 2004, 31(1): 114-117. |
| [28] |
Yang YY, Wang Z, He T, et al. Sediment bacterial communities associated with anaerobic biodegradation of Bisphenol A[J]. Microbial Ecology, 2015, 70(1): 97-104. DOI:10.1007/s00248-014-0551-x |
| [29] |
Shen H, Wang H, Zhao YC, et al. Analysis of fungal communities present in Morchella growth soil using denaturing gradient gel electrophoresis[J]. Acta Edulis Fungi, 2009, 16(2): 6-9. 沈洪, 汪虹, 赵永昌, 等. 采用变性梯度凝胶电泳研究羊肚菌土壤真菌群落结构[J]. 食用菌学报, 2009, 16(2): 6-9. |
| [30] |
Münchberg U, Wagner L, Rohrer C, et al. Quantitative assessment of the degree of lipid unsaturation in intact Mortierella by Raman microspectroscopy[J]. Analytical and Bioanalytical Chemistry, 2015, 407(12): 3303-3311. DOI:10.1007/s00216-015-8544-2 |
| [31] |
Asad-Uz-Zaman M, Bhuiyan MR, Khan MAI, et al. Integrated options for the management of black root rot of strawberry caused by Rhizoctonia solani Kuhn[J]. Comptes Rendus Biologies, 2015, 338(2): 112-120. DOI:10.1016/j.crvi.2014.11.006 |
| [32] |
Wang HH, Meng J, Peng XX, et al. Rice WRKY4 acts as a transcriptional activator mediating defense responses toward Rhizoctonia solani, the causing agent of rice sheath blight[J]. Plant Molecular Biology, 2015, 89(1/2): 157-171. |
 2017, Vol. 44
2017, Vol. 44




