扩展功能
文章信息
- 刘紫嫣, 马桥, 由胜男, 张旭旺, 张西迪, 殷庆鑫, 李炫莹, 厉舒祯, 张照婧, 曲媛媛
- LIU Zi-Yan, MA Qiao, YOU Sheng-Nan, ZHANG Xu-Wang, ZHANG Xi-Di, YIN Qing-Xin, LI Xuan-Ying, LI Shu-Zhen, ZHANG Zhao-Jing, QU Yuan-Yuan
- Burkholderia sp. IDO3中靛蓝合成基因的克隆表达及其合成特性
- Cloning, expression and application of an indigo-producing gene from Burkholderia sp. IDO3
- 微生物学通报, 2017, 44(11): 2634-2643
- Microbiology China, 2017, 44(11): 2634-2643
- DOI: 10.13344/j.microbiol.china.170063
-
文章历史
- 收稿日期: 2017-01-22
- 接受日期: 2017-05-18
- 优先数字出版日期(www.cnki.net): 2017-06-27
2. 大连理工大学食品与环境学院 工业生态与环境工程教育部重点实验室 辽宁 盘锦 124000
2. Key Laboratory of Industrial Ecology and Environmental Engineering (Ministry of Education), School of Food and Environmental Science and Technology, Dalian University of Technology, Panjin, Liaoning 124000, China
靛蓝是一种古老的染料,被称为染料之王,广泛应用于印染、食品和医药等行业[1]。目前市场上的靛蓝以化学合成为主,合成产量高、稳定性好,但是所用原料有毒,生产过程能耗高且会产生大量染料废水,危害环境。随着社会环保意识的提高,以植物提取靛蓝为代表的天然绿色染料重新受到人们的青睐。除了植物靛蓝,微生物也能利用体内的相关基因合成靛蓝类色素,微生物反应条件温和、环境友好,是近年来靛蓝合成研究的热点[1-4]。
靛蓝的微生物合成始于1983年,研究人员利用大肠杆菌异源表达萘双加氧酶实现了吲哚到靛蓝的合成[5]。后续研究表明其他加氧酶,如苯酚羟化酶、苯乙烯单加氧酶、联苯双加氧酶、P450单加氧酶等都具有催化吲哚合成靛蓝的能力[2, 6-8],这些酶主要都是以其他芳烃为天然底物的芳烃加氧酶。芳烃加氧酶主要通过两种途径催化吲哚合成靛蓝,双加氧酶能够同时在吲哚环C-2和C-3位加羟基生成顺式吲哚2, 3-二氢二醇,随后脱水生成吲哚酚,吲哚酚本身不稳定,在空气中会自发聚合生成靛蓝[5, 7]。然而单加氧酶能够直接在吲哚环C-3位加羟基生成吲哚酚,继而生成靛蓝[6, 8]。微生物合成靛蓝已开展了较多研究,已报道了多种酶资源能够转化吲哚合成靛蓝,但是不同酶的合成能力具有一定差异,并且很多加氧酶合成靛蓝过程中会生成其他副产物。靛玉红是靛蓝合成中最常见的副产物,吲哚氧化产物吲哚酚会继续被氧化生成靛红,吲哚酚和靛红反应会生成靛玉红,有些加氧酶还会催化吲哚生成少量2-羟基吲哚,该物质也能和吲哚酚反应生成靛玉红[6, 9-10]。因此,开发新颖且能够专一合成靛蓝的酶资源将有助于推动生物合成靛蓝的研究和工业化应用进程。
1 材料与方法 1.1 实验材料 1.1.1 菌株和质粒: Burkholderia sp. IDO3为本实验前期从活性污泥中分离筛选得到的吲哚降解菌[11],其16S rRNA基因序列的GenBank登录号为KP895480,在中国微生物菌株保藏中心的保藏编号为CGMCC 10457。表达宿主大肠杆菌Escherichia coli BL21(DE3)和表达载体pET-28a(+)由实验室保存。 1.1.2 培养基: LB培养基(g/L):NaCl 10.0,蛋白胨10.0,酵母粉5.0。卡那霉素工作浓度为50.0 mg/L。 1.1.3 主要试剂和仪器: 吲哚、色氨酸,国药集团化学试剂有限公司;异丙基-β-D-硫代半乳糖苷(IPTG),上海阿拉丁生化科技股份有限公司;硫酸卡那霉素,大连美仑生物技术有限公司;蛋白胨、酵母粉,英国Oxoid公司;其余试剂均为国产分析纯;2×Taq DNA聚合酶和2×Pfu Master Mix高保真DNA聚合酶,南京诺唯赞生物科技有限公司;胶回收试剂盒,上海捷瑞生物工程有限公司;限制性内切酶(Nco I,Xho I)、T4 DNA连接酶,美国NEB公司。Metash UV-9000双光束紫外可见分光光度计,上海元析仪器有限公司;离心机,德国Eppendorf公司;Primaide高效液相色谱仪,日本HITACHI公司;Agilent G6224A型液相色谱/飞行时间/质谱联用仪(LC/TOF/MS),美国Agilent公司。 1.2 实验方法 1.2.1 靛蓝合成基因的克隆和表达: 用于扩增靛蓝合成基因indAB的引物序列为:F:5′-CATG CCATGGTTGAGAAAGATCGGAATCGT-3′ (下划线部分为Nco I酶切位点);R:5′-CGATCTCGAG TCAGCGTTCCTGTCGGTTCG-3′ (下划线部分为Xho I酶切位点)。利用菌株IDO3基因组DNA为模板,采用2×Pfu Master Mix高保真DNA聚合酶对靛蓝合成基因indAB进行PCR扩增。PCR反应体系(100 μL)为:菌株IDO3基因组2 μL,引物F和R (10 μmol/L)各4 μL,2×Pfu Master Mix 50 μL,无菌水40 μL。PCR反应条件为:94 ℃ 5 min;95 ℃ 30 s,58 ℃ 30 s,72 ℃ 2 min,30 个循环;72 ℃ 5 min。利用DNA Gel Extraction Kit对扩增产物进行切胶回收和纯化。随后利用限制性内切酶Nco I和Xho I对pET-28a(+)及indAB纯化产物进行双酶切,酶切条件为37℃、6 h。酶切产物经过纯化后,使用T4连接酶将基因片段与载体于16℃连接过夜。连接产物热激转化导入E. coli BL21(DE3)感受态细胞中,孵育后涂布LB平板。随机挑取转化子培养,采用菌液PCR进行验证,阳性克隆进一步进行测序验证以确定没有碱基突变。正确构建的基因工程菌命名为E. coli IND_AB。
1.2.2 SDS-PAGE电泳分析: 菌株IND_AB于30℃、培养过夜作为种液,种液放于4℃保存。转接种液至含卡那霉素的LB培养基中,37℃、150 r/min培养。当菌体OD600达到0.4-0.6时,一瓶加入终浓度为0.5 mmol/L的IPTG诱导表达,另外一瓶不添加IPTG。16℃诱导20 h后,收集菌体、煮沸,SDS-PAGE电泳分析表达产物。以含有空白pET-28a(+)质粒的E. coli BL21 (DE3)菌株作为对照。 1.2.3 靛蓝合成实验及产物分析: 靛蓝合成实验采用LB培养基,反应体系为30 mL,菌株IND_AB以1% (体积比)的接种量接种于灭菌后培养基中,于30℃、培养24 h后检测靛蓝产量。靛蓝定量方法如下:将反应液于12 000 r/min离心3 min,沉淀用二甲基亚砜(DMSO)悬浮,混合液于12 000 r/min再次离心3 min,蓝色上清液采用紫外-可见分光光度计检测618 nm下的吸光度,根据吸光度和标准曲线计算靛蓝产量。标准曲线测定如下:利用DMSO配制各种不同浓度的靛蓝标准液并于618 nm测定吸光度,拟合吸光值和靛蓝浓度绘制标准曲线,其方程为A=0.066 2 Cindigo+0.003,A为吸光值,Cindigo为靛蓝浓度,方程R2为0.999。将DMSO萃取蓝色上清液用0.45 μm的滤膜过滤,分别进行高效液相色谱(HPLC)以及液相色谱/飞行时间/质谱联用(LC/TOF/MS)检测。液相色谱检测条件为:色谱柱Hypersil ODS2 (5 μm,250 mm× 4.6 mm),进样量10μL,流速1 mL/min,检测波长280 nm,流动相为甲醇和水,采用梯度洗脱,梯度为0-20 min,60%-70%甲醇(体积比)。质谱采用电喷雾电离源(ESI),负模式进行扫描。
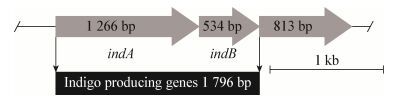
1.2.4 靛蓝合成的条件优化: 以24 h的靛蓝产量为考察目标,采用单因素法对菌株IND_AB合成靛蓝过程中的重要影响因素进行条件优化。首先考察环境因素对靛蓝产量的影响,包括摇床转速和培养温度,摇床转速选择0-250 r/min,培养温度选择20、25、30、37、45℃;随后优化培养基组分,NaCl浓度范围1.0-30.0 g/L,酵母粉1.0-20.0 g/L,蛋白胨1.0-15.0 g/L;最后考察外加吲哚或色氨酸对靛蓝产量的影响,吲哚浓度范围0-200.0 mg/L,色氨酸浓度范围选定为0-1.0 g/L。在上述最优条件下培养菌株IND_AB,定时取样检测靛蓝产量,绘制靛蓝合成曲线。 2 结果与分析 2.1 靛蓝合成基因的克隆表达和重组菌的构建实验室前期采用克隆文库技术将菌株Burkholderia sp. IDO3的基因组进行随机酶切,回收酶切片段并将其与处理好的pUC19质粒连接,重组质粒在大肠杆菌中表达。利用靛蓝显色法从克隆文库中筛选阳性克隆,获取一个在平板上显蓝色的克隆。培养该重组菌,提取质粒并送测序,测序结果显示插入序列全长为3 035 bp,基因预测结果表明该片段含有3个基因片段(图 1),长度分别为1 266、534和813 bp。序列提交Genbank数据库获得登录号为KX611609。
|
| 图 1 菌株IDO3的文库片段基因结构分析 Figure 1 Gene organization of the DNA fragment from strain IDO3 genomic clone library |
|
|
利用BLAST将获得的片段与NCBI中的数据库进行比对分析,结果如表 1所示。长度为1 266 bp的基因较为新颖,与它相似性高的序列均为未知蛋白,相似性为40%左右的多个蛋白注释为氧化还原酶的加氧酶组分,进一步与UniProtKB/Swiss-Prot数据库中的蛋白比对显示它与苯乙烯单加氧酶的大组分相似性为28%和29%,表明该基因可能是加氧酶基因,命名为indA。对长度为534 bp的基因进行比对分析,发现它与多个还原酶具有较高的相似性,其中与苯乙烯单加氧酶的小组分相似性为36%,表明该基因可能是氧化还原酶的还原酶组分,命名为indB。综上,我们推测indA和indB为靛蓝合成基因的加氧酶组分和还原酶组分。
| 蛋白描述 Protein description |
相似性 Similarity (%) |
菌株 Strains |
|
| IndA | Oxygenase (APT36898) | 100 | Burkholderia sp. IDO3 |
| Hypothetical protein (WP_043185613) | 94 | Burkholderia cepacia | |
| Hypothetical protein (WP_017224118) | 71 | Cupriavidus basilensis | |
| Flavin oxidoreductase (WP_019656472) | 44 | Variovorax paradoxus | |
| Oxygenase (WP_031505929) | 42 | Streptomyces megasporus | |
| Oxygense subunit protein (WP_009461224) | 42 | Alcaligenes | |
| Styrene monooxygenase large component (O50214) | 29 | Pseudomonas sp. VLB120 | |
| Styrene monooxygenase large component (O06834) | 28 | Pseudomonas fluorescens ST | |
| IndB | Flavin reductase (APT36899) | 100 | Burkholderia sp. IDO3 |
| Hypotherical protein (WP_043185899) | 92 | Burkholderia cepacia | |
| Monooxygenase component (WP_015435487) | 55 | Azoarcus sp. KH32C | |
| Flavin reductase (WP_043742128) | 52 | Thauera sp. SWB20 | |
| Flavin oxidoreductase (WP_019656472) | 55 | Variovorax paradoxus | |
| FMN reductase (NADH) NtaB (P54990) | 51 | Aminobacter aminovorans | |
| Styrene monooxygenase small component (O06835) | 36 | Pseudomonas fluorescens ST |
以菌株Burkholderia sp. IDO3的基因组DNA为模板进行PCR扩增,得到长度约为1 800 bp的基因片段indAB,如图 2a所示。该片段连接到载体pET-28a(+)并转化到菌株E. coli BL21(DE3)中,得到基因工程菌E. coli IND_AB。为验证该基因成功表达,利用SDS-PAGE电泳分析了重组菌和对照组的蛋白表达,结果如图 2b所示。与对照组相比,实验组在~43 kD和~19 kD处有两条特异表达条带,其分子量大小与蛋白IndA和IndB预测大小基本一致,证明靛蓝合成基因indAB在E. coli BL21(DE3)中得到表达。此外对诱导后重组菌粗酶液的上清和沉淀进行电泳分析,结果显示IndA在上清和沉淀中均有条带,表明IndA部分可溶,但IndB条带不明显,仅在全细胞和沉淀中有微弱条带。

|
| 图 2 indAB基因扩增产物(A)和表达产物SDS-PAGE分析(B) Figure 2 Gel analysis of indAB amplification products (A) and SDS-PAGE analysis (B) of indAB expression 注:A:M:DNA marker;1:PCR扩增产物. B:M:蛋白Marker;1:含空质粒pET-28a(+)的E. coli BL21(DE3);2:未诱导的重组菌IND_AB;3:IPTG诱导的重组菌IND_AB;4:诱导重组菌粗酶的上清液;5:诱导重组菌粗酶的沉淀. Note: A: M: DNA marker; 1: PCR products of gene indAB. B: M: protein marker; 1: E. coli BL21(DE3) with empty pET-28a(+) plasmid; 2: strain IND_AB without IPTG induction; 3: strain IND_AB with IPTG induction; 4: supernatant of induced IND_AB crude enzyme mixture; 5: precipitation of the induced IND_AB crude enzyme mixture. |
|
|
在培养重组菌时,发现温度为30 ℃时菌株IND_AB在LB培养基生长过程中可直接产生蓝色物质(图 3),而含有空载体pET-28a(+)的大肠杆菌没有蓝色物质生成,表明该物质的产生是IndAB的作用结果。随后对菌株IND_AB合成的蓝色产物进行分析。

|
| 图 3 含空载体的大肠杆菌(A)和重组菌IND_AB (B)在LB培养基中的生长照片 Figure 3 LB growth media images of E. coli strain with empty pET-28a(+) (A) and recombinant strain IND_AB (B) |
|
|
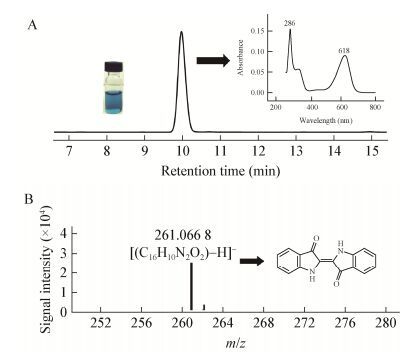
HPLC结果显示,该蓝色产物与标准靛蓝样品具有相同的保留时间,均在9.9 min出峰,其紫外-可见吸收图谱也与靛蓝标准品一致,最大吸收峰在286 nm和618 nm (图 4A),因此推测该物质为靛蓝。之后利用LC/TOF/MS对产物进行进一步分析,结果如图 4B所示。该吸收峰对应质谱中分子离子峰[M–H]-的m/z为261.066 8,与标准靛蓝样品一致。综上,菌株IND_AB在LB培养基中合成的蓝色物质为靛蓝。靛玉红是靛蓝合成中常见的副产物,然而本研究中并没有检测到靛玉红的产生,说明菌株IND_AB合成的靛蓝色素纯度很高。
|
| 图 4 重组菌IND_AB合成蓝色产物的液相分析(A)和质谱分析(B) Figure 4 HPLC (A) and mass spectral (B) analyses of the catalytic products by strain IND_AB |
|
|
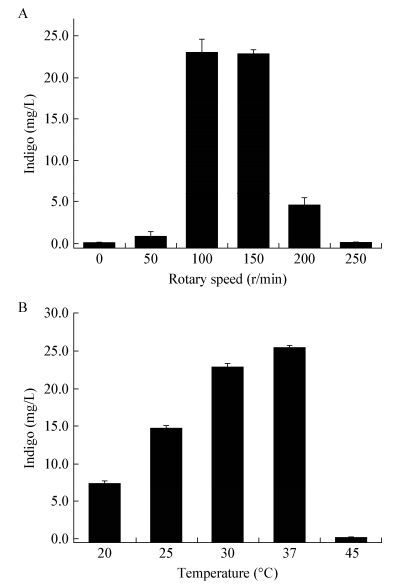
摇床转速直接影响反应体系的传质和氧气的传递,而文献[12]报道氧气的参与在微生物转化吲哚合成靛蓝过程中起到重要作用。由图 5A可知,菌株IND_AB在摇床转速为100 r/min和150 r/min时靛蓝产量最大;转速小于100 r/min或大于150 r/min时,靛蓝产量显著降低。转速的提高使得反应体系中氧气充足,且菌株与底物能够充分接触,从而能够明显提高靛蓝产量。但当转速过高(>200 r/min)时,靛蓝产量有所下降,可能是由于剧烈振荡影响了酶和反应体系的稳定性。
|
| 图 5 转速(A)和温度(B)对靛蓝产量的影响 Figure 5 Effects of rotary speed (A) and temperature (B) on the production of indigo |
|
|
反应温度对靛蓝合成过程中酶的催化活性有显著影响[13-14]。由图 5B可知,当反应温度为37℃时,菌株IND_AB合成的靛蓝产量最大;随着温度的降低,靛蓝产量逐渐降低;而温度升高至45℃时,靛蓝产量几乎为零。37℃为多数大肠杆菌的最适生长温度,此时菌体的酶活性较高,较低或较高的温度都会对菌株的生长以及酶活产生一定的抑制作用,从而影响靛蓝产量。
综上,选取37℃和150 r/min为最佳培养温度和转速。未优化前,菌株IND_AB是在30℃和150 r/min下培养,此时靛蓝产量约22.9 mg/L,优化后菌株IND_AB能够利用LB培养基合成靛蓝25.4 mg/L。
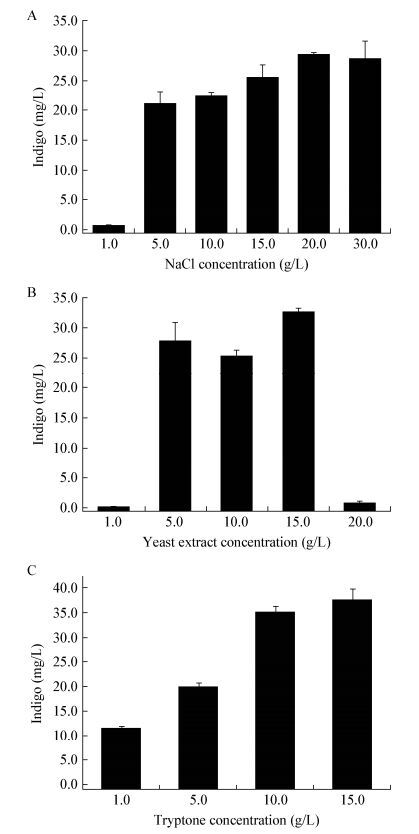
2.4 培养基组分对靛蓝合成的影响本研究中LB培养基既是菌株IND_AB的生长原料,又是其合成靛蓝的底物来源。NaCl能提供Na+以平衡细胞内外渗透压,酵母粉和蛋白胨能够为细胞提供生长所需的维生素、矿物质、微量元素和其他营养元素。依次考察LB培养基各组分对靛蓝合成的影响,结果如图 6所示。首先固定酵母粉和蛋白胨浓度为5.0 g/L和10.0 g/L,当NaCl浓度范围为15.0-30.0 g/L时,靛蓝产量趋于稳定,约为25-30 mg/L;固定NaCl、蛋白胨和酵母粉浓度分别为20.0、10.0和15.0 g/L时,靛蓝产量达到最大值,为32.7 mg/L;固定NaCl和酵母粉浓度分别为20.0 g/L和15.0 g/L,而蛋白胨浓度范围在10.0-15.0 g/L时,靛蓝产量能够达到35.1 mg/L。因此,综合考虑靛蓝合成量与培养基各组分用量,得到NaCl、酵母粉和蛋白胨最佳浓度分别为20.0、15.0和10.0 g/L,得到的靛蓝产量(35.1 mg/L)为初始值(22.9 mg/L)的1.53倍。然而与原始LB培养基相比,各组分用量增多导致成本增加,靛蓝的合成量增加却有限,因此需要探索其他方法来提高靛蓝产量。
|
| 图 6 NaCl (A)、酵母粉(B)以及蛋白胨(C)浓度对靛蓝产量的影响 Figure 6 Effects of NaCl (A), yeast extract (B), and tryptone (C) concentrations on the production of indigo |
|
|
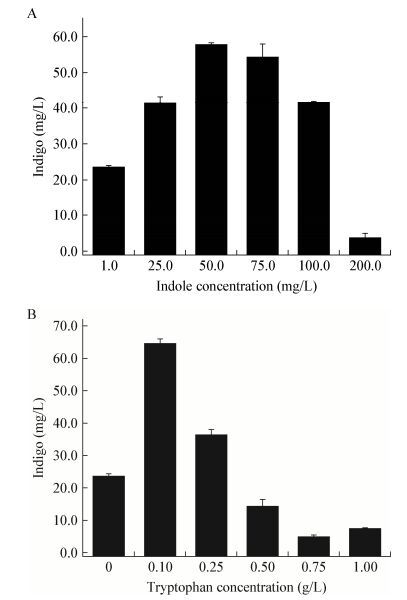
菌株IND_AB合成靛蓝的本质是将培养基中的色氨酸转化为吲哚[14],随后靛蓝合成酶氧化吲哚合成靛蓝,因此添加底物吲哚或者色氨酸会直接影响靛蓝的合成。在原始LB培养基组分基础上添加不同浓度吲哚,接种培养24 h后的靛蓝产量如图 7A所示,在一定范围内,靛蓝产量随着吲哚浓度的升高而升高。吲哚浓度为50.0 mg/L时,靛蓝产量可达57.7 mg/L;继续提高吲哚浓度靛蓝产量反而降低。这可能是由于吲哚具有一定的毒性[15-16],浓度较高时抑制了相关酶的活性。添加色氨酸也达到相似的效果(图 7B),当色氨酸浓度为0.1 g/L时,靛蓝产量可提高至64.4 mg/L。
|
| 图 7 外加吲哚(A)和色氨酸(B)对靛蓝产量的影响 Figure 7 Effects of indole (A) and tryptophan (B) on the production of indigo |
|
|
上述优化实验表明,外源添加吲哚或色氨酸是一种有效提高靛蓝产量的方式,因此考察菌株IND_AB在此条件下合成靛蓝的曲线。图 8A显示的是菌株IND_AB在外加50.0 mg/L吲哚的LB培养基中合成靛蓝的过程。在反应前6 h内没有靛蓝产生,6-15 h之间靛蓝的产量呈线性增加,随后产量增长趋于缓慢,18 h达到平衡,靛蓝浓度基本维持在58 mg/L左右。图 8B显示的是菌株IND_AB在外加0.1 g/L色氨酸的LB培养基中靛蓝合成的过程,其趋势和外加吲哚实验组基本一致,同样在18 h左右达到平衡期。但靛蓝产量比添加吲哚的更高,最终产量为64 mg/L左右。

|
| 图 8 菌株IND_AB在外加50 mg/L吲哚(A)和0.1 g/L色氨酸(B)的LB培养基中合成靛蓝曲线 Figure 8 Time course of indigo production from LB medium by adding 50 mg/L indole (A) and 0.1 g/L tryptophan (B) |
|
|
微生物合成靛蓝的研究至今已有三十多年的历史,目前报道的可用于靛蓝合成的酶有萘双加氧酶、苯酚羟化酶、苯乙烯单加氧酶、联苯双加氧酶、P450单加氧酶等[2, 6-8, 17]。虽然这些酶都能转化吲哚合成靛蓝,但是它们的天然底物并非吲哚,因此利用以吲哚为天然底物的靛蓝合成酶合成靛蓝可能具有一定的优势。本研究中利用靛蓝筛选法从野生菌株Burkholderia sp. IDO3的基因克隆文库中获得一段序列,它与已报道的靛蓝合成酶如萘双加氧酶等几乎没有相似性,表明IndAB是一个新颖的靛蓝合成酶,关于其酶学特性需要进一步研究。
产物分析表明重组菌IND_AB在LB培养基中能够直接合成靛蓝。LB培养基中并不存在吲哚,但是酵母粉和蛋白胨中都含有一定量的色氨酸,大肠杆菌自身含有色氨酸酶能够催化色氨酸转化为吲哚[14],为靛蓝合成提供底物。随后吲哚在靛蓝合成酶的作用下会生成3-羟基吲哚,即吲哚酚。吲哚酚在空气中不稳定,会自发聚合生成靛蓝。基于此,我们提出菌株IND_AB在LB培养基中合成靛蓝的途径,如图 9所示。靛玉红是靛蓝合成过程中常见的副产物,也是阻碍生物靛蓝工业化应用的一个重要原因,已经报道的萘双加氧酶、P450酶、苯酚羟化酶等都会产生靛玉红[9, 18-20]。本研究中尚未检测到到靛玉红的存在,表明靛蓝合成菌IND_AB合成的靛蓝纯度较高,具有实际应用的潜力。

|
| 图 9 菌株IND_AB合成靛蓝途径分析 Figure 9 Proposed indigo formation pathway by strain IND_AB |
|
|
条件优化实验表明,在培养基中添加吲哚或色氨酸是提高靛蓝产量的有效途径。添加50.0 mg/L吲哚的LB培养基中菌株IND_AB合成靛蓝产量可达57.7 mg/L,比初始条件下合成的靛蓝产量提高了152.0%;添加0.1 g/L色氨酸的LB培养基中合成靛蓝产量可达64.4 mg/L,产量提高了181.2%。在前期报道中,萘双加氧酶是研究最为透彻的靛蓝合成酶,携带来源于菌株Pseudomonas putida PgG7萘双加氧酶的重组大肠杆菌能在LB培养基中合成约25 mg/L的靛蓝[5];之后通过对萘双加氧酶和色氨酸操纵子进行改造,重组大肠杆菌能从葡萄糖出发合成135 mg/L靛蓝[21];而本课题组前期构建的源于Comamonas sp. MQ中萘双加氧酶的大肠杆菌最高能够合成205 mg/L靛蓝[17]。目前靛蓝产量最高的报道是韩国的Si Wouk Kim研究小组,他们从Methylophaga sp. strain SK1中外源表达了一段黄素单加氧酶,得到的重组大肠杆菌可产生160 mg/L靛蓝[13],后来从Methylophaga aminisulfidivorans MPT中克隆得到一段黄素单加氧酶基因,通过基因序列优化和培养优化,重组菌最高合成920 mg/L靛蓝[14]。其他合成靛蓝的报道中,Doukyu等将Acinetobacter sp. ST-550中的苯酚羟化酶基因导入到大肠杆菌中表达,重组菌在水-有机溶剂两相体系中能合成52 mg/L靛蓝[22-23];已报道的甲苯单加氧酶合成产物复杂,其中靛蓝的产量只有3-10 mg/L左右[20, 24];本课题组之前构建的联苯双加氧酶能合成约44 mg/L靛蓝[7];Kwon等从Rhodococcus sp. T104中扩增了一段靛蓝合成基因ipoA,在大肠杆菌中外源表达后可产靛蓝30.9 mg/L,突变氨基酸位点后,可达到50.7 mg/L左右[25];Lim等在森林土壤宏基因组筛选到一个新颖的靛蓝合成基因,将其构建到pJEC54质粒中并导入大肠杆菌,能够获得34 mg/L靛蓝[26];最近,Cheng等将苯乙烯单加氧酶基因在假单胞菌中过量表达,24 h内可以合成52.1 mg/L靛蓝[2]。综上分析,可以发现菌株IND_AB合成靛蓝的产量与大部分加氧酶合成能力相当,后续可进一步利用分子改造等手段提高加氧酶的活性,以期提高靛蓝产量。
| [1] |
Ma Q, Qu YY, Zhang XW, et al. Recent advances in microbial synthesis of indigo[J]. Chinese Journal of Applied and Environmental Biology, 2012, 18(2): 344-350. 马桥, 曲媛媛, 张旭旺, 等. 靛蓝的微生物合成研究新进展[J]. 应用与环境生物学报, 2012, 18(2): 344-350. |
| [2] |
Cheng L, Yin S, Chen M, et al. Enhancing indigo production by over-expression of the styrene monooxygenase in Pseudomonas putida[J]. Current Microbiology, 2016, 73(2): 248-254. DOI:10.1007/s00284-016-1055-3 |
| [3] |
Zhang XW, Qu YY, Ma Q, et al. Illumina MiSeq sequencing reveals diverse microbial communities of activated sludge systems stimulated by different aromatics for indigo biosynthesis from indole[J]. PLoS One, 10(4): e0125732
|
| [4] |
Qu YY, Liu ZY, Shen WL, et al. Genome sequence of an indigoid-producing strain, Pseudomonas sp. PI1[J]. Genome Announcements, 2015, 3(3): e00622-15. |
| [5] |
Ensley BD, Ratzkin BJ, Osslund TD, et al. Expression of naphthalene oxidation genes in Escherichia coli results in the biosynthesis of indigo[J]. Science, 1983, 222(4620): 167-169. DOI:10.1126/science.6353574 |
| [6] |
Qu YY, Shi SN, Zhou H, et al. Characterization of a novel phenol hydroxylase in indoles biotranformation from a strain Arthrobacter sp. W1[J]. PLoS One, 2012, 7(9): e44313. DOI:10.1371/journal.pone.0044313 |
| [7] |
Qu YY, Xu BW, Zhang XW, et al. Biotransformation of indole by whole cells of recombinant biphenyl dioxygenase and biphenyl-2, 3-dihydrodiol-2, 3-dehydrogenase[J]. Biochemical Engineering Journal, 2013, 72: 54-60. DOI:10.1016/j.bej.2012.12.021 |
| [8] |
Gillam EMJ, Aguinaldo AMA, Notley LM, et al. Formation of indigo by recombinant mammalian cytochrome P450[J]. Biochemical and Biophysical Research Communications, 1999, 265(2): 469-472. DOI:10.1006/bbrc.1999.1702 |
| [9] |
Gillam EMJ, Notley LM, Cai HL, et al. Oxidation of indole by cytochrome P450 enzymes[J]. Biochemistry, 2000, 39(45): 13817-13824. DOI:10.1021/bi001229u |
| [10] |
Singh A, Chauhan NS, Thulasiram HV, et al. Identification of two flavin monooxygenases from an effluent treatment plant sludge metagenomic library[J]. Bioresource Technology, 2010, 101(21): 8481-8484. DOI:10.1016/j.biortech.2010.06.025 |
| [11] |
Ma Q, Qu YY, Zhang XW, et al. Systematic investigation and microbial community profile of indole degradation processes in two aerobic activated sludge systems[J]. Scientific Reports, 2015, 5: 17674. |
| [12] |
Han GH, Bang SE, Babu BK, et al. Bio-indigo production in two different fermentation systems using recombinant Escherichia coli cells harboring a flavin-containing monooxygenase gene (fmo)[J]. Process Biochemistry, 2011, 46(3): 788-791. DOI:10.1016/j.procbio.2010.10.015 |
| [13] |
Choi HS, Kim JK, Cho EH, et al. A novel flavin-containing monooxygenase from Methylophaga sp. strain SK1 and its indigo synthesis in Escherichia coli[J]. Biochemical and Biophysical Research Communications, 2003, 306(4): 930-936. DOI:10.1016/S0006-291X(03)01087-8 |
| [14] |
Han GH, Shin HJ, Kim SW. Optimization of bio-indigo production by recombinant E. coli harboring fmo gene[J]. Enzyme and Microbial Technology, 2008, 42(7): 617-623. DOI:10.1016/j.enzmictec.2008.02.004 |
| [15] |
Kim J, Hong H, Heo A, et al. Indole toxicity involves the inhibition of adenosine triphosphate production and protein folding in Pseudomonas putida[J]. FEMS Microbiology Letters, 2013, 343(1): 89-99. DOI:10.1111/femsle.2013.343.issue-1 |
| [16] |
Han Y, Sun MM, Wang JP, et al. Progress on indole: an intercellular signal molecule in microbial communities[J]. Microbiology China, 2015, 42(4): 736-748. 韩茵, 孙苗苗, 王建平, 等. 吲哚作为细菌细胞间信号分子的研究进展[J]. 微生物学通报, 2015, 42(4): 736-748. |
| [17] |
Zhang XW, Qu YY, Ma Q, et al. Cloning and expression of naphthalene dioxygenase genes from Comamonas sp. MQ for indigoids production[J]. Process Biochemistry, 2013, 48(4): 581-587. DOI:10.1016/j.procbio.2013.02.008 |
| [18] |
Han GH, Gim GH, Kim W, et al. Enhanced indirubin production in recombinant Escherichia coli harboring a flavin-containing monooxygenase gene by cysteine supplementation[J]. Journal of Biotechnology, 2014, 164(2): 179-187. |
| [19] |
Berry A, Dodge TC, Pepsin M, et al. Application of metabolic engineering to improve both the production and use of biotech indigo[J]. Journal of Industrial Microbiology and Biotechnology, 2002, 28(3): 127-133. DOI:10.1038/sj.jim.7000228 |
| [20] |
Rui LY, Reardon KF, Wood TK. Protein engineering of toluene ortho-monooxygenase of Burkholderia cepacia G4 for regiospecific hydroxylation of indole to form various indigoid compounds[J]. Applied Microbiology and Biotechnology, 2005, 66(4): 422-429. DOI:10.1007/s00253-004-1698-z |
| [21] |
Murdock D, Ensley BD, Serdar C, et al. Construction of metabolic operons catalyzing the de novo biosynthesis of indigo in Escherichia coli[J]. Nature Biotechnology, 1993, 11(3): 381-386. DOI:10.1038/nbt0393-381 |
| [22] |
Doukyu N, Toyoda K, Aono R. Indigo production by Escherichia coli carrying the phenol hydroxylase gene from Acinetobacter sp. strain ST-550 in a water-organic solvent two-phase system[J]. Applied Microbiology and Biotechnology, 2003, 60(6): 720-725. DOI:10.1007/s00253-002-1187-1 |
| [23] |
Ishikawa M, Shigemori K, Hori K. Application of the adhesive bacterionanofiber AtaA to a novel microbial immobilization method for the production of indigo as a model chemical[J]. Biotechnology and Bioengineering, 2014, 111(1): 16-24. DOI:10.1002/bit.25012 |
| [24] |
McClay K, Boss C, Keresztes I, et al. Mutations of toluene-4-monooxygenase that alter regiospecificity of indole oxidation and lead to production of novel indigoid pigments[J]. Applied and Environmental Microbiology, 2005, 71(9): 5476-5483. DOI:10.1128/AEM.71.9.5476-5483.2005 |
| [25] |
Kwon NR, Chae JC, Choi KY, et al. Identification of functionally important amino acids in a novel indigo-producing oxygenase from Rhodococcus sp. strain T104[J]. Applied Microbiology and Biotechnology, 2008, 79(3): 417-422. DOI:10.1007/s00253-008-1445-y |
| [26] |
Lim HK, Chung EJ, Kim JC, et al. Characterization of a forest soil metagenome clone that confers indirubin and indigo production on Escherichia coli[J]. Applied and Environmental Microbiology, 2005, 71(12): 7768-7777. DOI:10.1128/AEM.71.12.7768-7777.2005 |
 2017, Vol. 44
2017, Vol. 44




