扩展功能
文章信息
- 彭倩楠, 霍冬雪, 徐传标, 胡淇淞, 张家超
- PENG Qian-Nan, HUO Dong-Xue, XU Chuan-Biao, HU Qi-Song, ZHANG Jia-Chao
- 黎族人肠道微生物群落结构特征及其与饮食关联性
- Intestinal microbiota in Li cohort and its correlation with their diets
- 微生物学通报, 2017, 44(11): 2624-2633
- Microbiology China, 2017, 44(11): 2624-2633
- DOI: 10.13344/j.microbiol.china.170106
-
文章历史
- 收稿日期: 2017-02-10
- 接受日期: 2017-05-31
- 优先数字出版日期(www.cnki.net): 2017-06-26
人体肠道中栖息着约30个属500多种动态细菌,这些细菌的动态变化在维持人体健康方面起着不可替代的作用,它们与人体形成了密不可分的互惠共生关系[1]。研究发现,肠道微生物在不同结构和组成上会影响宿主的营养物质加工、能量代谢平衡[2]、免疫发育和成熟[3]及其它多种重要的生理活动。前期研究表明,宿主基因型[4]、性别[5]、年龄、身体质量指数、地理环境[5]、饮食结构[6]以及生活方式等均可影响肠道菌群的构成。
在众多影响因素中,宿主基因型和饮食的影响最为显著。不同民族肠道菌群的构成差异显著,如西方人肠道菌群中含量最多的是拟杆菌属(Bacteroides)[7],韩国人肠道菌群中含量最多的是栖粪杆菌属(Faecalibacterium)[8],蒙古人肠道菌群中含量最多的是普氏菌属(Prevotella)[9],俄罗斯人肠道菌群中普遍缺乏Prevotella和Bacteroides,但有些样本双歧杆菌(Bifidobacterium)和乳杆菌(Lactobacillus)占绝对优势,还存在很多古细菌[10]。虽然不同人群肠道菌群差异较大,但其自身肠道菌群均能使宿主保持良好的健康状况。长期的饮食习惯同样影响着肠道菌群结构的形成[11],前期研究表明,饮食结构以蛋白质和脂肪为主时,其肠道的主要菌属为Bacteroides;饮食结构以糖类为主时,其肠道主要菌属为Prevotella[12];而当摄入较高的膳食纤维时,肠道菌属则以Bifidobacterium含量较高[13]。
目前关于世界各地区和各民族人群肠道菌群多样性的报道已有很多,但迄今为止并没有关于黎族人肠道菌群的相关研究报道。黎族是海南省的主要少数民族,黎族人作为海南最早的居民,主要聚居在中国海南省中南部,其历史可追溯到新石器时代,在数千年的历史发展中逐步形成了本民族特有的风俗和饮食习惯。黎族人饮食较为清淡,饮食结构类似于地中海饮食,主要以丰富的植物性食物为主,如葫芦瓜、萝卜和小白菜等,食物加工简单,烹调方式为水煮;在动物性食物方面,主要以海洋性食品为主,如田螺、鱼等;椰子、芒果、木瓜、香蕉等是其食用的主要水果。从营养的角度来看,其饮食结构中膳食纤维含量较高。
本研究基于16S rRNA基因V3–V4可变区的高通量测序技术,对采集自海南省白沙黎族自治县的黎族志愿者粪便微生物进行研究,并对志愿者的饮食进行详细记录,旨在揭示黎族人肠道菌群结构及其与饮食的关联性。通过对黎族人肠道菌群构成和饮食关联性的研究,可有效反映出肠道微生物在不同地域和民族之间的差异,这有利于进一步了解我国少数民族的健康情况,同时也为基于肠道菌群为靶点的人体肠道微生态平衡调控及宿主健康管理提供基础数据。
1 材料与方法 1.1 材料 1.1.1 样品来源: 从海南省白沙黎族自治县的牙叉镇和白沙镇共征集22名农村志愿者,采集其晨便。其中男性12名,女性10名,志愿者年龄在23-46岁之间,平均年龄33.5岁;BMI指数17.42-28.67,平均BMI指数21.33,所有志愿者均已基于口头询问和户口证件的结果确定其典型黎族身份,且身体健康,无肠道疾病史,近3个月内未服用抗生素类药物。同时,详细记录了22名志愿者的饮食情况。 1.1.2 主要试剂: 51504-粪便微生物宏基因组DNA提取试剂盒,德国QIAGEN公司;粪便/土壤宏基因组DNA提取试剂盒,美国MOBIO公司;Agilent DNA 1000试剂盒,Agilent Technologies公司;琼脂糖凝胶回收试剂盒及质粒小提试剂盒、Taq DNA聚合酶、DNA Marker,大连宝生物工程有限公司;TE缓冲液(pH 8.0)、5×TBE电泳缓冲液液、10% SDS、20 mg/mL蛋白酶K、酚/氯仿/异戊醇(25:24:1,体积比)、氯仿/异戊醇(24:1,体积比)、0.5 mol/L EDTA、10 mol/L CTAB、5 mol/L NaCl、1%的琼脂糖凝胶、预冷异丙醇、3 mol/L醋酸钠,试验所用缓冲液和常用试剂均参照供应商所提供实验手册配制。 1.1.3 主要仪器: DYY-12电泳仪,北京六一仪器厂;CDS-800型UVP凝胶成像分析系统,Sagecreation公司;5804台式高速冷冻离心机、微量加样器,Eppendorf公司;HHSI-NI电热恒温水浴槽,北京长安科学仪器厂;ND-2000型微量紫外分光光度计,基因有限公司;THZ-C恒温振荡器,太仓实验设备厂;ZHWY-200D恒温培养振荡器,上海智城分析仪器制造有限公司;PL303/01电子天平,Mettler Toledo公司;MLS-3751L型全自动高压蒸汽灭菌器,Panasonic仪器公司;DHP-9272型电热恒温培养箱,上海一恒科学仪器有限公司;HC-2518型台式高速离心机,安徽中科中佳科学仪器有限公司;SW-CJ-2FD型双人单面净化工作台,苏州净化设备有限公司;Agilent 2100生物分析仪,Agilent Technologies公司。 1.2 试验方法 1.2.1 粪便样品采集及保存: 用一次性采样勺采集约10 g志愿者的新鲜晨便样品于50 mL离心管中,向离心管中加入约2-3倍体积的保护液充分混匀,用封口膜封好采样管管口,立即将其置于液氮中,并置于-80 ℃保存。 1.2.2 粪便中细菌宏基因组DNA的提取: 经过前期试验条件摸索,采用Bead-Beating加试剂盒的方法提取。质地较稀的样品选用德国QIAGEN粪便基因组DNA提取试剂盒,而较为粘稠或较硬的样品则选用美国MOBIO粪便基因组DNA提取试剂盒提取。用琼脂糖凝胶电泳检测提取后的DNA的完整性和纯度,然后用微量紫外分光光度计检测其浓度,将符合试验要求的DNA置-80 ℃保存备用。 1.2.3 细菌16S rRNA基因V3–V4可变区扩増: 用细菌16S rRNA基因V3–V4可变区通用PCR扩增引物,扩增引物为:338F (5′-ACTCCTACGGGAGG CAGCA-3′)和806R (5′-GGACTACHVGGGTWTCT AAT-3′)。以提取的DNA作为模板,用上述引物进行扩增。PCR反应体系为:10×PCR buffer 5 μL,dNTP mix (2.5 mmol/µL) 4 μL,正反向引物(10 μmol/L)各1.5 μL,模板DNA 1.5 μL,Taq DNA聚合酶(5 U/µL) 0.5 μL,ddH2O补足至50 μL。PCR反应条件为:95 ℃ 4 min;95 ℃ 1 min,55 ℃ 45 s,72 ℃ 1 min,20-25次循环;72 ℃ 7 min,4 ℃终止反应。 1.2.4 PCR产物纯化、平衡及测序: 对扩增的PCR产物采用柱纯化的方式进行纯化,并采用Agilent DNA 1000试剂盒和Agilent 2100生物分析仪定量PCR产物。将22份样品的宏基因组DNA定量到100 nmol/L的浓度后,分别加载到Illumina Miseq高通量测序平台进行测序。 1.2.5 测序结果的生物信息学处理: 筛选高质量DNA序列后,使用QIIME分析平台对所提取的高质序列在去除前后引物和标签后进行生物信息学分析。先使用PyNAST校准排齐序列,再使用100%相似性进行Uclust归并建立无重复的V3–V4序列集。在100%相似性归并的基础上进行了97%相似性的归并,建立了分类操作单元(Operational taxonomic units,OTU)。所得OTU序列用ChimeraSlayer去除其可能存在的嵌合体序列后,用Ribosomal database project (RDP) classifier对剩余序列进行同源性比对和种属分类学鉴定。将剩余的代表序列插入FastTree软件生成所有OTU代表序列的系统发育进化树,并在此基础上进行α和β多样性计算。可通过α多样性中的香农指数(Shannon-Wiener index)和超1指数(Chao1 index),分别对样品菌群构成的丰度和多样性进行评价。在基于UniFrac Distance上对各样品之间进行加权(Weighted)和非加权(Unweighted)的主坐标分析(Principal coordinate analysis,PCoA),同时采用基于UniFrac Distance的非加权组平均法(Unweighted pair-group method with arithmetic mean,UPGMA)进行样品聚类。 1.2.6 数据的统计分析及图表绘制: 使用Mann-Whitney或Kruskal-Wallis tests对黎族人肠道菌属的相对含量进行显著性分析,使用多元方差分析(Multivaraiate analysis of variance,MANOVA)对黎族人肠道菌群群落结构的差异性进行显著性分析,使用Spearman Rank进行肠道核心菌群与营养元素的相关性分析。应用R语言Pheatmap包进行聚类分析和热图构建,应用Ade4包进行菌群与饮食的相关性分析。试验设计原始测序数据上传NCBI网站,数据项目编号是:PRJNA356130。 2 结果与分析 2.1 志愿者信息及样品测序情况概述通过高通量测序和序列提取质控,22份测序样品共产生251 719条高质序列,平均每个样品获取高质序列数超过11 000条(range=7 300-15 099),可以满足后续分析需求(一般认为对于人类肠道微生物宏基因组的研究,细菌16S rRNA基因可变区测序条带数平均大于8 000条即可满足后续多样性分析要求)。按97%的相似度共划分出15 172个OTU进行后续分析,平均每个样品OTU为690个(Range=403-1 187)。志愿者信息及样品测序情况概述见表 1。
| 样品 Sample |
性别 Sex |
年龄 Age |
身体质量指数 BMI |
条带 Band |
操作分类单元 OTU |
| LN1 | 男 | 36 | 22.66 | 11 344 | 752 |
| LN2 | 女 | 44 | 21.33 | 10 908 | 511 |
| LN3 | 男 | 29 | 28.67 | 12 107 | 623 |
| LN4 | 女 | 23 | 17.48 | 9 782 | 645 |
| LN5 | 男 | 29 | 19.23 | 11 078 | 464 |
| LN6 | 男 | 32 | 19.95 | 9 750 | 599 |
| LN7 | 男 | 29 | 17.19 | 10 412 | 518 |
| LN8 | 女 | 42 | 21.63 | 11 913 | 610 |
| LN9 | 男 | 39 | 24.77 | 15 099 | 830 |
| LN10 | 女 | 26 | 21.46 | 14 247 | 629 |
| LN11 | 男 | 29 | 21.19 | 11 374 | 750 |
| LN12 | 女 | 38 | 23.56 | 10 198 | 951 |
| LN13 | 男 | 44 | 21.99 | 8 989 | 1 062 |
| LN14 | 女 | 36 | 23.18 | 14 917 | 993 |
| LN15 | 女 | 42 | 20.96 | 12 254 | 752 |
| LN16 | 男 | 33 | 20.03 | 9 931 | 737 |
| LN17 | 女 | 24 | 23.31 | 9 753 | 403 |
| LN18 | 女 | 37 | 17.42 | 7 300 | 546 |
| LN19 | 男 | 38 | 24.24 | 11 973 | 606 |
| LN20 | 女 | 23 | 19.53 | 12 771 | 1 187 |
| LN21 | 男 | 26 | 18.99 | 12 827 | 436 |
| LN22 | 男 | 38 | 20.45 | 12 792 | 568 |
在门水平上,通过对筛选出的高质序列进行RDP和BLAST同源性序列比对发现,健康黎族志愿者粪便中含量最高的两个细菌门为拟杆菌门(Bacteroidetes,58.96%)和硬壁菌门(Firmicutes,37.77%),总含量达到序列总数的96.73%。此外,变形菌门(Proteobacteria,1.20%)、软壁菌门(Tenericutes,0.70%)、放线菌门(Actinobacteria,0.47%)、迷踪菌门(Elusimicrobia,0.18%)和黏胶球形菌门(Lentisphaerae,0.11%)含量依次递减。
在属水平上,健康黎族志愿者粪便中含量最高的是Prevotella,平均含量达到菌群总量的49.38%。此外,在黎族人粪便中隶属于硬壁菌门且相对含量大于1.0%的细菌属为:栖粪杆菌属(Faecalibacterium,4.56%)、真细菌(Eubacterium,3.23%)、颤杆菌属(Oscillibacter,2.83%)、巨型球菌属(Megasphaera,1.78%)、瘤胃球菌属(Ruminococcus,1.54%)、小杆菌属(Dialister,1.49%)、链型杆菌属(Catenibacterium,1.42%)和罗氏菌属(Roseburia,1.02%),含量依次递减。属于拟杆菌门且相对含量大于1.0%的细菌属为Prevotella (49.38%)和Bacteroides (4.00%),含量依次递减。从以上结果可知,黎族人肠道细菌的优势细菌门为Firmicutes和Bacteroidetes,优势细菌属为Prevotella、Faecalibacterium和Bacteroides。
应用斯皮尔曼等级级数计算黎族人肠道核心菌属的相关系数,并根据菌属的相对含量应用重心聚类方法(centroid linkage)进行聚类分析,分析结果如图 1所示。在核心菌属中,罕见小球菌属(Subdoligranulum)与Oscillibacter呈明显正相关,相关系数为0.54。柯林斯菌属(Collinsella)与多尔氏菌属(Dorea)呈明显正相关,相关系数为0.46。Collinsella与Roseburia呈明显负相关,相关系数为-0.58。

|
| 图 1 黎族人肠道菌群构成及核心微生物群落特征 Figure 1 The composition of gut microbiota and community characters of core gut microbiota of Li cohort |
|
|
22名志愿者肠道中优势菌群的分布情况如图 2所示,结合表 1志愿者的身体质量指数BMI可以看出,大部分志愿者体质偏瘦,相应肠道菌群中以Prevotella为主要菌群,而Bacteroides菌群偏少。然而LN3、LN9和LN19志愿者的身体质量指数BMI值偏大,尤其是LN3志愿者的身体质量指数BMI值超过28,相应的LN3、LN9和LN19志愿者肠道内Bacteroides菌群数量高于其他志愿者。

|
| 图 2 各志愿者肠道优势菌属含量分布 Figure 2 The distribution of intestinal dominant bacteria genera in each volunteer |
|
|
基于加权和非加权(weighted和unweighted) Unifrac Distance的主坐标分析(principal coordinate analysis,PCoA)对样品进行分析,与已有文献中关于中国其他民族人群肠道菌群的研究结果[14]进行对比,展示出黎族人与中国其他民族人群肠道菌群的相互关系。由图 3A、B可见,黎族人肠道中的细菌属与其他民族人群差异显著。在属水平上(表 2),Catenibacterium、Prevotella和Megasphaera等在黎族人肠道中含量较高的细菌属在其他民族人群肠道中含量较低或者没有,巨单胞菌属(Megamonas)、考拉杆菌属(Phascolarctobacterium)和布劳特氏菌属(Blautia)等在其他民族人群肠道中含量较高的细菌属在黎族人肠道中含量较低或者没有。
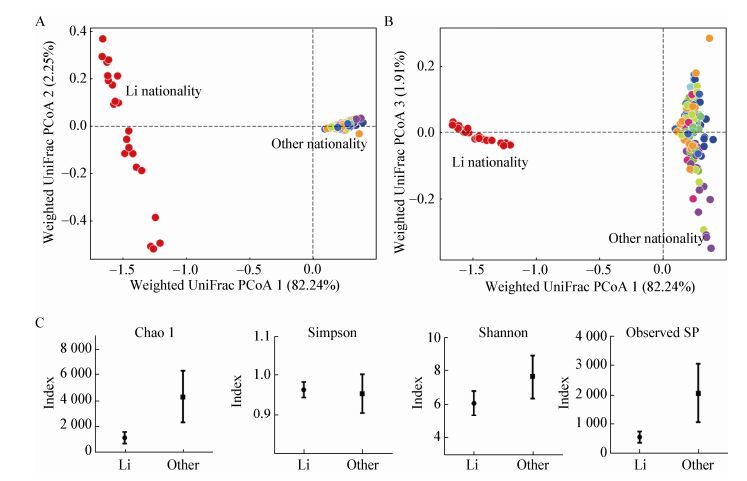
|
| 图 3 黎族与其他民族志愿者肠道菌群α和β多样性比较 Figure 3 The α diversity and β diversity of intestinal microbiota among Li cohort and other national volunteers |
|
|
| 菌属 Bacterial genus |
白族 Bai |
藏族 Tibetan |
维族 Wei |
蒙族 Mongol |
壮族 Zhuang |
汉族 Han |
黎族 Li |
P值 P-value |
| Catenibacterium | 0.02 | 0.01 | 0.02 | 0.00 | 0.00 | 0.00 | 1.42 | 7.54E-19 |
| Methanosphaera | 0.15 | 0.00 | 0.78 | 0.06 | 0.02 | 0.16 | 0.00 | 3.85E-17 |
| Bifidobacterium | 0.77 | 0.71 | 4.67 | 4.53 | 0.52 | 1.49 | 0.00 | 4.29E-15 |
| Methanobrevibacter | 0.19 | 1.53 | 5.10 | 1.55 | 0.60 | 0.87 | 0.00 | 1.02E-12 |
| Megamonas | 12.70 | 3.46 | 5.79 | 1.24 | 8.67 | 9.99 | 0.00 | 1.41E-12 |
| Prevotella | 4.73 | 18.48 | 1.40 | 0.36 | 6.02 | 2.58 | 47.27 | 6.47E-12 |
| Gemmiger | 0.36 | 0.24 | 0.78 | 0.37 | 0.34 | 0.76 | 0.00 | 8.21E-12 |
| Trabulsiella | 0.52 | 0.51 | 3.41 | 2.25 | 2.18 | 2.05 | 0.00 | 8.95E-12 |
| Blautia | 4.77 | 4.22 | 3.70 | 4.88 | 5.83 | 6.93 | 0.22 | 2.54E-11 |
| Oscillibacter | 0.03 | 0.39 | 0.08 | 0.08 | 0.07 | 0.05 | 2.83 | 3.43E-11 |
| Enterobacter | 0.29 | 0.04 | 0.31 | 0.29 | 0.37 | 0.89 | 0.00 | 4.09E-11 |
| Sutterella | 0.09 | 0.23 | 0.05 | 0.44 | 0.05 | 0.03 | 0.58 | 1.69E-09 |
| Megasphaera | 0.17 | 0.14 | 2.07 | 0.07 | 0.01 | 0.65 | 1.78 | 1.47E-08 |
| Mitsuokella | 0.00 | 0.00 | 0.41 | 0.00 | 0.00 | 0.02 | 0.29 | 4.90E-08 |
| Parasuttereila | 0.37 | 0.07 | 0.57 | 0.81 | 0.41 | 0.35 | 0.09 | 3.24E-07 |
| Butyricicoccus | 0.12 | 0.05 | 0.10 | 0.19 | 0.20 | 0.19 | 0.04 | 5.57E-07 |
| Phascolarctobacterium | 8.30 | 13.47 | 13.25 | 21.49 | 16.63 | 8.17 | 0.60 | 6.18E-07 |
| Dialister | 0.06 | 0.06 | 0.59 | 0.01 | 0.01 | 0.07 | 1.49 | 2.24E-06 |
| Akkermansia | 0.02 | 2.59 | 0.18 | 0.76 | 0.15 | 0.23 | 0.06 | 3.08E-06 |
| Succinivibrio | 0.05 | 0.54 | 1.25 | 0.00 | 0.00 | 0.01 | 0.20 | 1.96E-05 |
| Clostridium | 2.49 | 2.38 | 1.97 | 2.46 | 2.22 | 3.12 | 0.95 | 0.000 1 |
| Lachnospira | 0.33 | 0.09 | 0.04 | 0.10 | 0.12 | 0.19 | 0.03 | 0.000 2 |
| Streptococcus | 0.19 | 0.07 | 0.76 | 0.23 | 1.07 | 0.34 | 0.77 | 0.000 3 |
| Dorea | 1.48 | 1.21 | 1.08 | 1.01 | 1.53 | 1.71 | 0.56 | 0.000 7 |
| Lactobacillus | 0.90 | 0.34 | 0.62 | 0.72 | 0.24 | 0.34 | 0.46 | 0.001 0 |
| Ruminococcus | 2.45 | 3.43 | 1.63 | 2.13 | 3.66 | 3.02 | 1.54 | 0.001 5 |
| Coprococcus | 0.65 | 1.09 | 0.52 | 1.29 | 0.99 | 1.14 | 0.68 | 0.001 8 |
| Parabacteroides | 0.17 | 0.26 | 0.09 | 0.34 | 0.26 | 0.30 | 0.88 | 0.001 9 |
| Collinsella | 0.29 | 0.70 | 1.10 | 0.82 | 0.61 | 0.66 | 0.29 | 0.012 6 |
| Bacteroides | 4.83 | 7.04 | 5.34 | 10.40 | 3.05 | 3.63 | 6.31 | 0.024 7 |
通过计算样品中香农指数、辛普森指数(Simpson index)、超1指数和获得菌种数量值(Observerd species)并取平均值,可得到黎族志愿者肠道菌群的α多样性,反映出样品中微生物多样性和物种丰富度。如图 3C所示,获得的香农指数、超1指数和获得菌种数量值显著低于中国其他民族人群肠道菌群相应多样性指数,说明黎族人肠道菌群α多样性显著低于中国其他民族。
2.4 饮食特征描述及核心微生物与饮食的相关性通过对22名黎族志愿者饮食摄入情况的详细记录,推算出黎族人日常饮食营养物质表(表 3)。分析可知,黎族人日常饮食中膳食纤维的摄入量明显高于全国平均水平、蛋白质和脂肪的摄入量明显低于全国平均水平[15],尤其是脂肪的摄入更少。这与黎族人日常饮食中以植物性食物为主、动物性食物摄入较少的饮食结构相一致。
| 营养成分 Nutrient content |
日均摄入量 Daily intake |
| Protein (g) | 64.91 |
| Fat (g) | 34.16 |
| Carbohydrate (g) | 206.13 |
| Fiber (g) | 20.25 |
| VA (μg) | 156.79 |
| VC (mg) | 52.85 |
| VB1 (mg) | 1.03 |
| VB2 (mg) | 0.34 |
| Niacin (mg) | 7.59 |
| Mg (mg) | 248.31 |
| Ca (mg) | 241.31 |
| Fe (mg) | 14.49 |
| Zn (mg) | 7.77 |
| Cu (mg) | 0.47 |
| Mn (mg) | 0.93 |
| K (mg) | 1 460.46 |
| P (mg) | 484.71 |
| Se (μg) | 38.06 |
通过对肠道核心微生物与营养物质相关性分析发现(图 4),普氏粪杆菌(Faecalibacterium prausnitzii)与膳食纤维、Cu、Mg和Mn呈显著正相关,与脂肪和VB2呈显著负相关,说明饮食中膳食纤维、Cu、Mg和Mn含量较高的人群肠道菌群中Faecalibacterium prausnitzii数量较多,而饮食中脂肪和VB2含量较高的人群肠道菌群中Faecalibacterium prausnitzii数量则较少。罗氏乳杆菌(Lactobacillus rogosae)与膳食纤维、Zn和Fe呈显著正相关,与烟酸呈显著负相关,说明饮食中膳食纤维、Zn和Fe含量较高的人群肠道菌群中Lactobacillus rogosae数量较多,而饮食中烟酸含量较高的人群肠道菌群中Lactobacillus rogosae数量则较少。
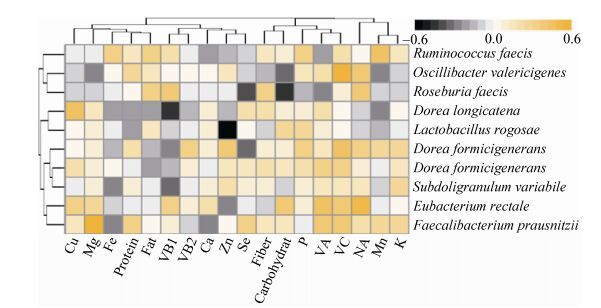
|
| 图 4 肠道核心微生物与营养物质相关性分析 Figure 4 The correlation of core gut microbiota and nutrients |
|
|
黎族人肠道菌群的优势细菌门为拟杆菌门和硬壁菌门,其含量分别为58.96%和37.77%,这与欧洲人[16]、韩国人[17]和蒙古人[18]的肠道优势菌群一致。在属水平上,普氏菌属在黎族人肠道菌群中所占比例最大,达到了总含量的49.38%。经过查阅文献发现,Prevotella (尤其是Prevotella ruminicola)在肠道中主要参与碳水化合物及植物蛋白质的分解代谢,分解后的主要产物有乙酸和氢气等物质[19],而拟杆菌属在肠道中主要参与动物蛋白、氨基酸和脂肪的代谢。Wu等[20]的研究证实长期的饮食结构与肠道内Bacteroides和Prevotella的含量有着密切关系,特别是长期摄入蛋白质和动物脂肪会使Bacteroides含量提高,而长期以碳水化合物为主的膳食结构则会使其肠道内Prevotella富集。结合采样过程中所记录的黎族志愿者的饮食情况,我们发现黎族人的饮食结构类似于地中海饮食结构,日常饮食中以素食为主,喜好清淡。因此,Prevotella成为黎族志愿者肠道中的优势菌属也是食物调控的结果。Liu等[9]的研究表明,处于内陆干旱、半干旱型气候环境中以红肉和乳制品为主要膳食的蒙古人肠道内含量最高的是Prevotella,该菌群产生短链脂肪酸发挥抗炎症作用,使膳食纤维摄入比例很低的蒙古人仍然可以保持健康。Schnorr等[21]的研究发现,适应稀树大草原环境生活的哈扎人肠道菌群多样性非常丰富,含有高水平的螺旋体菌属(Treponema)和低水平的Bifidobacterium,且哈扎人男女的肠道菌群结构显著不同,这可能与其男女的饮食结构不同有直接关系,哈扎人中男性主要狩猎和采集蜂蜜,女性则收集块茎等植物食物。由此可知,在生活环境、基因等其他因素相同的情况下,饮食是塑造肠道菌群结构的主要因素。
基于微生物群落α和β多样性的分析结果,我们证实了黎族人的肠道菌群与中国其他民族人群的肠道菌群差异显著,差异显著的菌属为Catenibacterium、Prevotella、Megasphaera、Megamonas、Phascolarctobacterium和Blautia等,而且黎族人肠道菌群差异比中国其他民族人群的肠道菌群差异要小。早在2012年,Yatsunenko等[22]通过对531名分布于南美洲委内瑞拉亚马逊州、非洲南部马拉威共和国和北美洲美国城市人的肠道菌群结构进行研究,认为不同地区人群的肠道菌群的构成及其功能差异显著。郑艺等[23]和黄卫强等[24]分别对壮族人和白族人肠道菌群的多样性进行了研究,证明不同民族成年人肠道菌群聚类现象明显。黎族人的饮食结构是导致其与中国其他民族人群肠道菌群差异显著的主要原因,其肠道菌群中参与碳水化合物以及植物蛋白质分解代谢的菌属比较多,如Prevotella等。此外,本研究所采用的是基于16S rRNA基因V3–V4可变区的高通量测序技术对黎族人的肠道菌群进行研究,而此前研究中国其他民族人群肠道菌群多样性的文献中多采用PCR和DGGE技术,研究方法的不同可能也会对研究结果产生一定的影响。
4 结论基于16S rRNA基因V3–V4可变区的高通量测序技术分析黎族人肠道菌群的多样性,结果发现黎族人肠道菌群中以Firmicutes和Bacteroidetes为优势菌门,Prevotella为优势菌属。结合黎族志愿者的饮食情况,可发现黎族人日常饮食以素食为主,脂肪摄入较少,这与黎族人肠道菌群中Prevotella为最优势菌属结论一致。对黎族人肠道菌群多样性的研究,实质上是探讨基因型和饮食结构对肠道菌群的影响。基于微生物群落α和β多样性的分析结果证实,黎族人肠道菌群与中国其他民族人群肠道菌群呈现出显著差异且明显低于其他民族,差异显著菌属为Catenibacterium、Prevotella和Megasphaera等。通过对黎族人肠道菌群构成和饮食关联性的研究,可有效反映出肠道微生物在不同地域和民族之间的差异,这有利于进一步了解我国少数民族的健康情况,同时也为基于肠道菌群为靶点的人体肠道微生态平衡调控及宿主健康管理提供基础数据。
| [1] |
Hooper LV, Gordon JI. Commensal host-bacterial relationships in the gut[J]. Science, 2001, 292(5519): 1115-1118. DOI:10.1126/science.1058709 |
| [2] |
Matsumoto M, Kibe R, Ooga T, et al. Impact of intestinal microbiota on intestinal luminal metabolome[J]. Scientific Reports, 2012, 2: 233. DOI:10.1038/srep00233 |
| [3] |
Mazmanian SK, Liu CH, Tzianabos AO, et al. An immunomodulatory molecule of symbiotic bacteria directs maturation of the host immune system[J]. Cell, 2005, 122(1): 107-118. DOI:10.1016/j.cell.2005.05.007 |
| [4] |
Zoetendal EG, Akkermans ADL, Akkermans WM, et al. The host genotype affects the bacterial community in the human gastronintestinal tract[J]. Microbial Ecology in Health and Disease, 2001, 13(3): 129-134. DOI:10.1080/089106001750462669 |
| [5] |
Mueller S, Saunier K, Hanisch C, et al. Differences in fecal microbiota in different European study populations in relation to age, gender, and country: a cross-sectional study[J]. Applied and Environmental Microbiology, 2006, 72(2): 1027-1033. DOI:10.1128/AEM.72.2.1027-1033.2006 |
| [6] |
Ley RE, Hamady M, Lozupone C, et al. Evolution of mammals and their gut microbes[J]. Science, 2008, 320(5883): 1647-1651. DOI:10.1126/science.1155725 |
| [7] |
Arumugam M, Raes J, Pelletier E, et al. Enterotypes of the human gut microbiome[J]. Nature, 2011, 473(7346): 174-180. DOI:10.1038/nature09944 |
| [8] |
Nam YD, Jung MJ, Roh SW, et al. Comparative analysis of Korean human gut microbiota by barcoded pyrosequencing[J]. PLoS One, 2011, 6(7): e22109. DOI:10.1371/journal.pone.0022109 |
| [9] |
Liu WJ, Zhang JC, Wu CY, et al. Unique features of ethnic Mongolian gut microbiome revealed by metagenomic analysis[J]. Scientific Reports, 2016, 6: 34826. DOI:10.1038/srep34826 |
| [10] |
Tyakht AV, Kostryukova ES, popenko AS, et al. Human gut microbiota community structures in urban and rural populations in Russia[J]. Nature Communications, 2013, 4: 2469. |
| [11] |
Finegold SM, Attebery HR, Sutter VL. Effect of diet on human fecal flora: comparison of Japanese and American diets[J]. The American Journal of Clinical Nutrition, 1974, 27(12): 1456-1469. |
| [12] |
Zhai QX, Tian FW, Wang G, et al. Progress in research on the role of intestinal microbiota in human health[J]. Food Science, 2013, 34(15): 337-341. 翟齐啸, 田丰伟, 王刚, 等. 肠道微生物与人体健康的研究进展[J]. 食品科学, 2013, 34(15): 337-341. DOI:10.7506/spkx1002-6630-201315069 |
| [13] |
Connolly ML, Lovegrove JA, Tuohy KM. In vitro evaluation of the microbiota modulation abilities of different sized whole oat grain flakes[J]. Anaerobe, 2010, 16(5): 483-488. DOI:10.1016/j.anaerobe.2010.07.001 |
| [14] |
Zhang JC, Guo Z, Xue ZS, et al. A phylo-functional core of gut microbiota in healthy young chinese cohorts across lifestyles, geography and ethnicities[J]. The ISEM Journal, 2015, 9(9): 1979-1990. DOI:10.1038/ismej.2015.11 |
| [15] |
Fan YO, Liu AL, He YN, et al. Assessment of nutrient adequacy of adult residents in China[J]. Acta Nutrimenta Sinica, 2012, 34(1): 15-19. 范轶欧, 刘爱玲, 何宇纳, 等. 中国成年居民营养素摄入状况的评价[J]. 营养学报, 2012, 34(1): 15-19. |
| [16] |
Qin JJ, Li RQ, Raes J, et al. A human gut microbial gene catalogue established by metagenomic sequencing[J]. Nature, 2010, 464(7285): 59-65. DOI:10.1038/nature08821 |
| [17] |
Nam YD, Jung MJ, Roh SW, et al. Comparative analysis of Korean human gut microbiota by barcoded pyrosequencing[J]. PLoS One, 2011, 6(7): 22109. DOI:10.1371/journal.pone.0022109 |
| [18] |
Zhang J, Zheng Y, Guo Z, et al. The diversity of intestinal microbiota of Mongolians living in Inner Mongolia, China[J]. Benefical Microbes, 2013, 4(4): 319-328. DOI:10.3920/BM2013.0028 |
| [19] |
Maslowski KM, Vieira AT, Ng A, et al. Regulation of inflammatory responses by gut microbiota and chemoattractant receptor GPR43[J]. Nature, 2009, 461(7268): 1282-1286. DOI:10.1038/nature08530 |
| [20] |
Wu GD, Chen J, Hoffmann C, et al. Linking long-term dietary patterns with gut microbial enterotypes[J]. Science, 2011, 334(6052): 105-108. DOI:10.1126/science.1208344 |
| [21] |
Schnorr SL, Candela M, Rampelli S, et al. Gut microbiome of the Hadza hunter-gatherers[J]. Nature Communications, 2014, 5: 3654. |
| [22] |
Yatsunenko T, Rey FE, Manary MJ, et al. Human gut microbiome viewed across age and geography[J]. Nature, 2012, 486(7402): 222-227. |
| [23] |
Zheng Y, Zhang JC, Qiao JM, et al. The diversity of gut Microbiota of Zhuang ethnic group living in urban and rural[J]. Journal of Chinese Institute of Food Science and Technology, 2016, 16(1): 226-236. 郑艺, 张家超, 乔健敏, 等. 壮族人群肠道菌群多样性分析[J]. 中国食品学报, 2016, 16(1): 226-236. |
| [24] |
Huang WQ, Zhang JC, Zheng Y, et al. The diversity of gut microbiota of Bai ethnic group[J]. Microbiology China, 2015, 42(3): 504-515. 黄卫强, 张家超, 郑艺, 等. 白族成年人肠道菌群多样性研究[J]. 微生物学通报, 2015, 42(3): 504-515. |
 2017, Vol. 44
2017, Vol. 44




