扩展功能
文章信息
- 沈媚, 陈冰清, 于瑞嵩, 朱于敏, 李震
- SHEN Mei, CHEN Bing-Qing, YU Rui-Song, ZHU Yu-Min, LI Zhen
- 冠状病毒S蛋白及其受体的结构和功能
- Structure and function of coronaviral S proteins and their receptors
- 微生物学通报, 2017, 44(10): 2452-2462
- Microbiology China, 2017, 44(10): 2452-2462
- DOI: 10.13344/j.microbiol.china.170256
-
文章历史
- 收稿日期: 2017-03-29
- 接受日期: 2017-05-27
- 优先数字出版日期(www.cnki.net): 2017-06-26
2. 上海市农业科学院畜牧兽医研究所 上海 201106;
3. 上海市农业遗传育种重点实验室 上海 201106
2. Institute of Animal Science and Veterinary Medicine, Shanghai Academy of Agricultural Sciences, Shanghai 201106, China;
3. Shanghai key laboratory of agricultural genetics and breeding, Shanghai 201106, China
冠状病毒感染人、鼠、猪、鸟等脊椎动物,具有胃肠道、呼吸道、肝脏和神经系统的嗜性。近年来,冠状病毒感染人和动物导致疾病流行的事件屡有发生,如2003年SARS、2010年PEDV和2012年MERS的暴发,引起世界各国对冠状病毒的高度重视[1-6]。
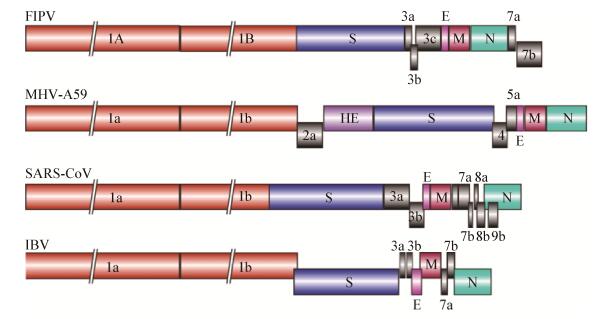
冠状病毒属于尼多病毒目冠状病毒科,病毒粒子呈球形或不规则形,有囊膜,大小为80-120 nm。其基因组为单股、正链RNA,基因组的5'端带有帽子结构,其后包含6-10个开放阅读框(Open reading frames,ORFs)。占据基因组2/3的第一个阅读框编码复制酶,基因组的后1/3主要编码结构蛋白,一般包括纤突蛋白(spike,S)、小包膜蛋白(envelope,E)、囊膜蛋白(membrance,M)、核蛋白(nucleocapsid,N)(图 1)。E蛋白和M蛋白主要参与病毒的装配过程,N蛋白包裹基因组形成核蛋白复合体,S蛋白则主要通过与宿主细胞受体结合介导病毒的入侵并决定病毒组织或宿主嗜性。
根据基因组的结构特点,目前将冠状病毒分为4个属,即α、β、γ和δ。α属冠状病毒包括PEDV、人冠状病毒NL63 (Human coroanvirus,HCoV-NL63)、犬冠状病毒(Canine coroanvirus,CCoV)以及传染性胃肠炎病毒(Transmissible gastroenteritis virus,TGEV)等;β属冠状病毒包括SARS、MERS、鼠肝炎病毒(Murine hepatitis virus,MHV)等;γ属冠状病毒包括鸡传染性支气管炎病毒(Avian infectious bronchitis,IBV);δ属冠状病毒包括鹦鹉冠状病毒(Parrot coronavirus,PaCoV)、猪δ冠状病毒(Porcine deltacoronavirus,PDCoV)等。不同属间甚至同一属内不同种的冠状病毒的附属蛋白基因的数量并不相同(图 1),它们编码的附属蛋白也往往并不是病毒装配所必需的[7]。
自从SARS暴发以后,冠状病毒的分子生物学及其入侵机制等方面的研究取得了长足进展。鉴于冠状病毒S蛋白及其相关受体的研究对病毒防控和抗病毒药物研发的重要意义,本文就冠状病毒S蛋白功能、S蛋白裂解活化方式和时空特点、S蛋白受体结合功能域的最新划分、冠状病毒受体种类以及病毒与受体结合方式的进化特征等方面的研究做简要阐述,以期为相关科研人员提供参考。
1 冠状病毒S蛋白冠状病毒S蛋白由1 160-1 400个氨基酸组成,包含21-35个N-糖基化位点。S蛋白以三聚体的形式在病毒表面形成特殊的花冠结构,冠状病毒因此而得名。S蛋白在宿主细胞蛋白酶的作用下被裂解为S1和S2两部分,S1主要功能是与宿主细胞表面受体结合,S2亚基介导病毒-细胞以及细胞-细胞膜融合。相比S2区域氨基酸序列的相对保守,S1的变异性则大得多,即便是同一种病毒的不同毒株也有一定差异。虽然理论上S1和S2之间存在蛋白酶的裂解位点,但不同种属冠状病毒S蛋白是否发生裂解以及裂解时间和裂解方式相差很大。越来越多的研究证明冠状病毒S蛋白的裂解活化对病毒入侵能力以及毒力起关键作用[9]。
1.1 S蛋白的裂解S蛋白的裂解大致分为两种情况,一种是S蛋白在病毒组装时发生裂解,即在病毒组装过程中,S蛋白在弗林蛋白酶(Furin)作用下S1/S2之间发生裂解,裂解过程发生于宿主细胞的高尔基体上;另一种是S蛋白在病毒侵染时发生裂解,即S蛋白在病毒组装过程中未发生裂解,其S蛋白的裂解和活化依赖于侵染过程中的蛋白酶类。
部分β属冠状病毒和所有γ属冠状病毒的S蛋白属于上面的第一种情况,即在病毒组装时发生裂解,如大部分MHV,因其S蛋白在病毒组装前就处于活化状态,不仅可以诱导病毒-细胞膜融合使病毒直接进入细胞,还会造成细胞-细胞膜融合从而出现合胞体现象[10]。
α属冠状病毒以及β属冠状病毒的S蛋白属于上面的第二种情况,即在病毒侵染时发生裂解,S蛋白在侵染过程中的裂解可以直接被侵染细胞内的蛋白酶类进行裂解,如α属人冠状病毒229E (HCoV-229E)的S蛋白在病毒侵染细胞后,被细胞内的组织蛋白酶L (Cathepsin L,CPL)或其它蛋白酶裂解活化[11];SARS-CoV同样依赖于侵染细胞内的CPL,但S蛋白的裂解需要与受体ACE2结合引起S蛋白构象改变后才能完成[12]。MHV-Ⅱ的S蛋白在病毒组装时不能被弗林蛋白酶裂解,其裂解也发生在S蛋白与受体结合以后,由靶细胞内的组织蛋白酶完成[13]。S蛋白在侵染过程中裂解的另一种方式是被环境中的蛋白酶类裂解,如α属冠状病毒中PEDV野生型毒株的S蛋白不能被靶细胞表面或靶细胞内的蛋白酶裂解活化,其S蛋白的裂解需要环境中的蛋白酶类,如胰酶(Trypsin),这可能与PEDV适应猪肠道的胰酶等蛋白酶丰富的感染环境有关。
1.2 PEDV S蛋白裂解与胰酶依赖性目前,PEDV的细胞培养大多需要在培养基中添加胰酶。已有研究证明胰酶对S蛋白的裂解发生在PEDV结合受体后,而且S蛋白的裂解有利于病毒粒子的释放[14]。细胞培养成功的PEDV毒株(如CV777) S蛋白在外源胰酶作用下发生裂解活化,可诱导膜融合,其形成的病变具有典型的合胞体结构。也有的PEDV毒株(如DR13)经过长期细胞培养传代后,其细胞培养不需要添加胰酶,因为其S蛋白未发生裂解,所以不能诱导膜融合,所形成的病变无典型的合胞体结构。因此对PEDV胰酶依赖分子机理的研究不仅有助于了解该病毒的生物学特性,也有利于改进疫苗的生产工艺。
为解决PEDV体外培养依赖胰酶的问题,Li等通过在野生型PEDV毒株预测的融合肽N端突变一个氨基酸的方法引入一个弗林蛋白酶作用位点,获得的重组毒株PEDV-SFCS的细胞培养不再依赖胰酶复制,但重组病毒的复制能力非常低,这也说明单独弗林蛋白酶作用位点的引入不能彻底改变PEDV细胞培养的胰酶依赖性问题[15]。我们实验室前期的PEDV反向遗传学研究也发现,弗林蛋白酶作用位点的引入并不能在不添加胰酶的条件下成功拯救重组PEDV毒株,但在胰酶存在的情况下携带弗林蛋白酶作用位点的重组毒株的复制能力有所提高(未发表数据)。在另一项研究中,我们也初步证明PEDV细胞培养酶的依赖可能与S蛋白600-900氨基酸范围的数个功能域有关(未发表数据)。
1.3 S蛋白裂解与冠状病毒入侵冠状病毒是有囊膜的病毒,病毒通过其表面糖蛋白与宿主细胞上的受体结合,然后通过病毒囊膜与细胞膜融合进入细胞。融合可以发生在细胞质膜如MHV-Ⅳ (MHV-JHMV)[16],也可以先通过受体介导的内吞作用形成内体(Endosome),然后在细胞内发生病毒膜与内体膜融合,如SARS、HCoV-229E。通过内体途径膜融合的机制分为单纯酸性诱导的膜融合以及酸性环境兼蛋白酶共同诱导的膜融合两种类型。Ⅱ型MHV属前种类型,仅酸性环境就可以诱导S蛋白发生构象改变导致病毒膜与内体膜融合[17]。SARS-CoV属后种类型,内体中的CPL可被酸性环境活化,然后此酶裂解活化S蛋白引发病毒膜与内体膜融合[11]。关于PEDV是通过什么途径进入细胞的目前尚不清楚,但根据同属的冠状病毒判断有可能是通过内体途径,但在补加胰酶的情况下也可能是通过与细胞质膜融合直接进入细胞,具体的进入模式还需更多实验证明。
不能直接进入细胞的病毒,当环境条件改变时,也可能会改变行为。如SARS-CoV病毒侵染早期,病人肺部上皮细胞会分泌弹性蛋白酶(Elstase,或称胰肽酶-E),在这种蛋白酶的作用下,SARS-CoV可以直接进入宿主细胞[18-19]。显然,病毒直接通过与细胞质膜融合进入细胞效率更高,SARS的发病机理可能与此有关。除此之外,MHV-Ⅱ和HCoV-229E也有此现象。近期有研究认为冠状病毒都应该是利用网格蛋白(Clathrin)介导的细胞内吞结合内体途径,不同种病毒的不同之处在于S蛋白是在早期内体中被裂解还是在溶酶体内裂解,而且他们认为其裂解一般不发生在病毒组装时期,而应该在侵染靶细胞过程中完成[20]。总之,关于不同冠状病毒S蛋白裂解与进入细胞方式还有诸多不同观点,许多现象也是百思不得其解,今后需要更多创新性的探索研究阐明相关机理。部分冠状病毒S蛋白的裂解活化方式及进入宿主细胞途径总结在表 1中。
| 病毒种类 Strains of viruses | 进入细胞途径 Way of cell enter | S蛋白活化时期及活化因子 Time and trigger for S protein cleavage | 是否产生合胞体 With cyncytia or not after infection |
| HCoV-229E | Via endosome | In assembly cell, CPL | No |
| FIPV | Via endosome | After binding to target cell, CPB | No |
| PEDV | Via endosome? | After binding to target cell, low pH, CPL? | No |
| SARS-CoV | Via endosome | After binding to target cell, low pH, CPL | No |
| MHV-Ⅱ | Via endosome | After binding to target cell, low pH | No |
| MHV-Ⅳ (JHMV) | Fusion with plasma membrane | In assembly cell, furin | Yes |
| MERS-CoV | Fusion with plasma membrane | In assembly cell, furin | Yes |
| IBV | Via endosome | In assembly cell, low pH, furin | Yes |
| 注:CPB为组织蛋白酶B. Note: CPB is Cathepsin B. | |||
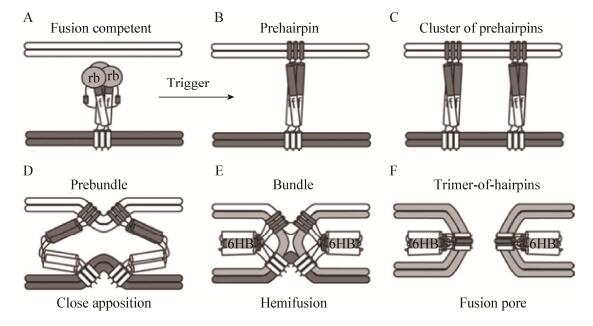
为方便理解,可把S蛋白参与的膜融合过程分解成图 2所示的6个过程[21]。首先,S蛋白存在于完整的病毒表面,处于亚稳态的融合准备状态;随后,S蛋白受体结合域(Receptor binding domain,rb)与细胞表面受体结合,在中性pH (在细胞表面融合的情况)或者内体区域低pH (通过内体融合的情况)的诱发作用下(Fusion triggering)(图 2A)转换为发夹前体状态(Prehaipin intermediate),由非极性的融合肽(Fusion peptide,FP)嵌入细胞膜(图 2B)。冠状病毒天然的融合蛋白的膜外区和嵌入细胞膜的发夹前体中间体都是同源三聚体。融合过程中下一步便是几个发夹前体在融合位点聚集(图 2C),紧接着发生融合蛋白的折回,最终融合亚单位转变成紧密的杆状三聚体发夹(图 2D-F),此时的蛋白构象处能量最稳定状态,因此融合蛋白构象变化有助于克服膜融合中的能量障碍。在融合蛋白折回过程中,嵌入细胞膜的FP与病毒膜上的跨膜域靠近,促进病毒膜与细胞膜的逐渐接近并密切接触、发生半融合,直至形成小融合孔,最终形成大融合孔,允许包裹病毒遗传物质的核衣壳通过并进入细胞质开始复制。
|
| 图 2 Ⅰ型融合蛋白的三聚体发夹膜融合模型[22] Figure 2 The common trimer-of-hairpins pathway of membrane fusion by S protein[22] 注:S蛋白引导膜融合过程中不需其他病毒膜蛋白参与,其包含受体结合亚单位(rb)和融合亚单位(f).图中细胞和病毒的膜分别在图的上方和下方.图A中有rb,其余的图中没有显示,已有研究表明该亚单位在随后的蛋白结构变化中移出原来位置,这样对f亚单位的钳制作用消除,有利于融合继续进行.在Ⅰ型融合蛋白中6螺旋束(6 helix bundles,6HBs)以束捆(bundle)和三聚体发夹的形式出现[22]. Note: The depicted S protein is one that does not require any other viral surface proteins for fusion, it contains both a receptor binding subunit (labeled rb) and a fusion subunit (labeled f). The target and viral membranes are, respectively, at the top and bottom of the images. The receptor binding subunit (rb) is not shown beyond image A as its location at the later stages is not known; in all cases studied, however, the rb subunit of this type of class Ⅰ fusion protein must move out of the way, thus unclamping the fusion subunit in the metastable fusion competent state and allowing fusion to proceed. For class Ⅰ fusion proteins six helix bundles (6HBs) are seen in their bundle and trimer-of-hairpins forms[22]. |
|
|
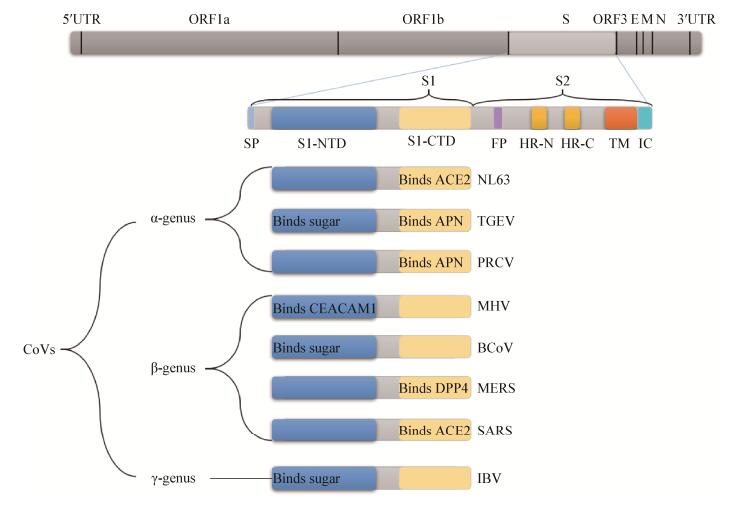
目前研究倾向于把冠状病毒S蛋白S1亚单位划分成2个结构域——N末端结构域(S1-NTD)和C末端结构域(S1-CTD)(图 2),2个功能域都能作为受体结合域(Receptor binding domain,RBD)。一般认为,S1-NTD结合糖类受体,S1-CTD结合蛋白类受体。但MHV结构功能域结合受体的模式似乎相反,其S1-NTD结合蛋白类癌胚抗原相关细胞黏附分子1受体(Carcinoembryonic antigen-cell adhension molecule,CEACAM1)。经细胞多次传代适应的MHV毒株可以利用硫酸肝素作为受体,但是结合硫酸肝素受体的功能由S1-CTD及一部分S2联合作用实现[23]。不同冠状病毒S蛋白上功能域的不一致可能与病毒的进化过程有关[24-25]。目前的研究发现,除δ属冠状病毒外,其他3个主要属的两个功能域至少可以识别两种多糖类受体和5种蛋白类受体(见“2冠状病毒S蛋白相关受体”),呈现出复杂的受体结合模式,参考Li等研究,我们绘制了冠状病毒S蛋白与受体结合模式图[26](图 3)。
|
| 图 3 冠状病毒基因组及通过S蛋白结合受体模式 Figure 3 Schematic of coronavirus genome and its receptor binding pattern by S protein 注:灰色:冠状病毒整段基因组;浅灰色:S基因;浅蓝色:信号肽(SP);深蓝色:S1基因N末端功能域(S1-NTD);浅黄色:S1基因C末端功能域(S1-CTD);紫色:融合肽(FP);深黄色:七肽重复序列(HR-N和HR-N);橘黄色:跨膜域(TM);青色:胞内结构(IC). Note: Gray: Genomes of the coronavirus; Light gray: S genes; Llight blue: Signal peptide (SP); Dark blue: N-terminal functional domain of the S1 gene (S1-NTD); Light yellow: C-terminal functional domain of the S1 gene (S1-CTD); Purple part: Fusion peptide (FP); Dark yellow: Heptad repeat (HR-N and HR-N); Orange: Transmembrane domain (TM); Cyan: Intracellular structure (IC). |
|
|
冠状病毒的嗜性决定于S蛋白和细胞受体的配对关系。病毒通过与宿主细胞的特异性受体结合,进入易感宿主细胞内完成入侵。目前,冠状病毒的部分细胞受体已经得到确定,按照受体的化学性质可以分为蛋白多肽类受体和糖类受体,蛋白多肽类占多数,如CEACAM1、氨基肽酶N (Amino peptidase N,APN)、血管紧张素转化酶2 (Angiotensin-converting enzyme 2,ACE2)、丝氨酸蛋白酶(Dipeptidyl peptidase 4,DPP4)等;多糖类受体如硫酸肝素(Heparin sulfate)、唾液酸(Sialic acid)等(表 2)。一种病毒可能利用几种不同的受体,如SARS-CoV、MHV;一种受体也可能被几种病毒利用,如APN、唾液酸等。
| 受体类型Receptor type | 属Genus | 种Species | |
| Protein or ploypeptide | APN (CD13) | Alpha-coronavirus | FCoV |
| CCoV | |||
| TGEV | |||
| HCoV-229E | |||
| PEDV | |||
| CEACAM1 | Beta-coronavirus | MHV-Ⅱ | |
| ACE2 | Alpha-coronavirus Beta-coronavirus | HCoV-NL63 SARS | |
| DPP4 | Beta-coronavirus | MERS | |
| Agglutinins | Alpha-coronavirus | FCoV FIPV | |
| Beta-coronavirus | SARS | ||
| Gamma-coronavirus | IBV | ||
| Polysaccharide | Heparin sulfate | Beta-coronavirus | MHV |
| Sialic acid | Alpha-coronavirus | FIPV | |
| Beta-coronavirus Alpha-coronavirus | BCV HCoV-NL63 | ||
| Gamma-coronavirus | IBV | ||
在α属冠状病毒中,HCoV-229E、TGEV、Ⅱ型猫冠状病毒(Feline coronavirus,FCoV)以及CCoV都可以利用各自宿主的APN作为受体,而且与APN结合的都是S蛋白的S1-CTD功能域(图 2)[28]。不同宿主的APN存在种属差异性,但上述病毒都可以与猫APN (fAPN)结合,因此研究者们推测此属的病毒是由同一株病毒进化而来,并且其初始宿主是猫[29]。
2.1.3 ACE2: ACE2是近年来新发现的一种金属蛋白酶,属于Ⅰ型跨膜糖蛋白,具有羧肽酶活性,在调节心、肾功能以及控制血压中起关键作用。人类ACE2的细胞外区域由2个亚基组成,其中锌金属肽酶区域可以进一步分成2个亚域(Ⅰ和Ⅱ),形成一个长而深的裂缝,环绕裂缝顶端的隆起线带有很高的负电荷,这些负电荷与S蛋白上面RBD的正电荷互补,从而与S蛋白结合[30-31]。SARS-CoV的S蛋白能够结合Vero E6细胞表面的ACE2,可溶性形式的ACE2能够阻止SARS-CoV对Vero E6的入侵,由此可以推断SARS-CoV的受体是ACE2;此外ACE2对SARS-CoV的复制有促进作用[32]。HCoV-NL63的受体也是ACE2[33];通过比较SARS-CoV和HCoV-NL63二者RBD与ACE2结合位置发现,两种病毒的RBD都围绕着ACE2的K353残基,这说明SARS-CoV和HCoV-NL63都倾向于结合受体蛋白上一个特定的区域。
2.1.4 DPP4: 2013年,DPP4作为MERS-CoV的受体首次被发现[34]。DPP4又称CD26,为Ⅱ型跨膜糖蛋白,含有776个氨基酸,在细胞膜表面呈二聚体,是继ACE2和APN后发现的第三种冠状病毒的外肽酶类受体。机体组织中的DPP4通常通过裂解激素和趋化因子的末端肽键而改变其活性,由此改变机体或细胞的代谢过程,但冠状病毒以DPP4作为受体应该不是利用其外肽酶的作用,而是因为其广泛分布于上皮组织和内皮组织内部,这些组织往往是冠状病毒入侵的主要场所。 2.1.5 凝集素类(Agglutinins): 冠状病毒还可以利用细胞表面不同种类的分子(如凝集素)作为受体进入细胞,如SARS-CoV、IBV和FCoV感染细胞时有钙依赖性凝集素(C型凝集素)的参与。树突状细胞特异的细胞间粘连分子-3-结合的非整合素(Dendritic cell-specific intercellular adhesion molecule-3-grabbing non-integrin,DC-SIGN)是巨噬细胞和树突状细胞表达的C型凝集素,也称CD209,其作用是识别常见的病毒及细菌的高度甘露糖糖基化模式。冠状病毒的S蛋白也是高度糖基化的,因此可以同宿主凝集素DC/L-SIGN结合。与DC-SIGN类似,L-SIGN也是一种C型凝集素,通常表达于肝脏或肺部内皮细胞,有研究认为L-SIGN是SARS-CoV和HCoV-229E的受体[35]。猫传染性腹膜炎病毒(Feline infectious peritonitis virus,FIPV)属于入侵免疫系统的冠状病毒,通过感染单核细胞和巨噬细胞导致全身蔓延。但非易感细胞可以通过表达外源性DC-SIGN变为易感,而且Ⅰ型和Ⅱ型FIPV都能够以DC-SIGN作为fAPN的共同受体或替代受体[36]。DC-SIGN和L-SIGN都能促进非敏感细胞对IBV的易感性,并且是以唾液酸非依赖的方式[37]。
2.2 多糖类受体唾液酸和硫酸肝素是冠状病毒常用的多糖类受体。唾液酸又名N-乙酰神经氨酸,是九碳单糖衍生物,广泛存在于动物组织中,其中脑部的含量最高。硫酸肝素属线性的多糖类物质,通常与细胞表面或细胞外的基质蛋白相结合构成糖蛋白发挥生物学功能。冠状病毒S蛋白与多糖结合的生物学意义越来越受关注。
α属冠状病毒FIPV的S蛋白可以获得结合硫酸肝素的活性,后文2.4中将详细叙述。β属冠状病毒S蛋白有结合唾液酸的活性,特别是BCV和HCoV-OC43[38]。MHV经细胞多次传代后,其S蛋白发生少量氨基酸突变,可结合细胞表面的硫酸肝素作为受体[39]。除MHV以外,冠状病毒S蛋白结合糖类主要靠S1-NTD功能域。有研究证明TGEV除以APN为受体外,还可结合唾液酸,此特性有助于病毒进一步结合肠绒毛刷状缘上的粘液素样糖蛋白[40],而且通过突变体的研究证明TGEV结合唾液酸就是依赖S1-NTD[41]。属于γ属冠状病毒的IBV Beaudette株通过鸡胚内多次传代后获得了与硫酸肝素等糖类结合的活性,这种特性可能与该病毒株的嗜性范围扩大有关[42]。研究发现IBV M41株结合糖类的特性与该株病毒S蛋白的S1-NTD功能域有关[43]。
2.3 PEDV受体近年来,随着PEDV疫情的暴发,关于其受体的研究受到广泛关注。前期的研究认为pAPN (猪APN)是PEDV感染的唯一受体,Li等将pAPN转染至PEDV的非易感性犬肾细胞(Madin darby canine kidney,MDCK)发现,该细胞能够表达pAPN并能被PEDV感染,因此推断pAPN是PEDV的功能性受体;通过免疫荧光和中和试验还发现pAPN具有介导PEDV复制的能力[44],pAPN参与PEDV感染的关键氨基酸区域也得以定位[45]。PEDV可以识别pAPN和hAPN,但TGEV与hAPN的结合能力则低很多;而且PEDV可以感染来源于猪、人、猴以及蝙蝠的细胞,据此有研究者提出蝙蝠可能是PEDV进化的源头宿主[28]。
然而,随着PEDV受体研究的深入,pAPN作为PEDV的功能性受体开始受到质疑。有研究表明PEDV不能感染猪睾丸细胞(Swine testis,ST细胞),也不能感染转染后成功表达pAPN的人宫颈癌细胞(Hela-pAPN)和CPK细胞(CPK-pAPN),但TGEV却可以感染这三种细胞(已经证实确定pAPN是TGEV的受体)。在CPK细胞中过量表达pAPN也只能小幅度地增加PEDV的增殖,但在CPK-pAPN细胞中加入氨肽酶抑制剂可以一定程度上抑制PEDV的增殖。这些结果表明pAPN不是PEDV的功能性受体,但可以促进PEDV的感染[46-47]。众所周知,PEDV可以感染非洲绿猴肾细胞(Verda reno,Vero),但Vero细胞并不表达pAPN,这些结果也提示PEDV可能通过不同受体感染宿主细胞[48]。
上述研究初步证明pAPN不是PEDV的唯一功能性受体,甚至可能不是主要的功能受体。作为TGEV的近亲,PEDV S1-CTD能够与pAPN结合,S1-NTD也可以结合糖,糖受体可作为PEDV的共同受体;而且与经典毒株相比,流行强毒株显示更强的糖结合活性,说明强毒株通过S蛋白变异获得了更强的与糖类受体相结合的能力,此能力有助于病毒更有效地侵染宿主[49]。
2.4 S蛋白突变与冠状病毒嗜性改变冠状病毒可以通过S蛋白的突变或与其他毒株重组获得与新的宿主受体结合的能力,从而发生组织或宿主嗜性的改变。冠状病毒受体特异性的改变决定了病毒的进化方向和跨种间的传播。
TGEV主要感染小肠的上皮细胞。20世纪80年代中期,一个TGEV的弱毒株——猪呼吸道冠状病毒(PRCoV)在比利时被分离得到,这株病毒可以感染呼吸道细胞,研究证明TGEV噬性变异是因为S蛋白基因缺失造成的[50]。原来不能细胞培养的FIPV-UCD经细胞培养适应后可以结合细胞膜表面硫酸肝素作为受体,该细胞适应的病毒株命名为FIPV-UCD1。其亲代毒株FIPV-UCD含有弗林蛋白酶裂解位点(RRXRR),此酶可将S蛋白裂解为S1和S2。然而FIPV-UCD1株经细胞适应后该位点突变为RRXRG,不能被弗林蛋白酶裂解;但该位点和附近区域构成可以与硫酸肝素结合的功能区,因硫酸肝素分布更加广泛,因此经传代突变的FIPV的宿主范围也更加广泛[38]。
3 总结与展望人和动物冠状病毒疫情的频繁暴发促发了世界范围内针对该病原的大量研究。当前对冠状病毒的研究主要集中在PEDV、MERS以及SARS等。大量实验数据证明冠状病毒S蛋白不仅可以结合宿主细胞受体,还可以诱导病毒-细胞膜融合,在病毒入侵过程中发挥着至关重要的作用。对冠状病毒S蛋白及其受体的结构与功能的认识不仅可以了解病毒入侵和致病机制,还可以根据不同受体特点研制对应的阻断药物;S蛋白决定病毒细胞嗜性的特点也可用于反向遗传操作重组系统进行病毒的筛选。本文主要针对冠状病毒S蛋白结构功能域、不同种属冠状病毒S蛋白裂解活化方式和时空特点、S蛋白受体结合功能域的最新划分、冠状病毒受体种类以及病毒与受体结合方式的进化特征等方面做出简要阐述。但总体来说对于S蛋白与受体功能的研究尚处于起步阶段,有许多问题有待解决和回答:一些冠状病毒(如PEDV)的入侵方式及主要功能受体究竟是什么?不同属冠状病毒S蛋白的裂解特点及诱导膜融合是否有各自的规律性?病毒组织嗜性与致病性改变同S蛋白结构的关系(如FIPV)等。这些方面还有待进一步的深入研究,这些研究成果不仅会更加深入揭示S蛋白与受体的结构功能关系,也会给人类和动物健康带来福利。
| [1] |
Sun RQ, Cai RJ, Chen YQ, et al. Outbreak of porcine epidemic diarrhea in suckling piglets, China[J]. Emerging Infectious Diseases, 2012, 18(1): 161-163. DOI:10.3201/eid1801.111259 |
| [2] |
Stevenson GW, Hoang H, Schwartz KJ, et al. Emergence of porcine epidemic diarrhea virus in the United States: clinical signs, lesions, and viral genomic sequences[J]. Journal of Veterinary Diagnostic Investigation, 2013, 25(5): 649-654. DOI:10.1177/1040638713501675 |
| [3] |
Huang YW, Dickerman AW, Pi eyro P, et al. Origin, evolution, and genotyping of emergent porcine epidemic diarrhea virus strains in the United States[J]. mBio, 2013, 4(5): e00737-13. |
| [4] |
Ksiazek TG, Erdman D, Goldsmith CS, et al. A novel coronavirus associated with severe acute respiratory syndrome[J]. New England Journal of Medicine, 2003, 348(20): 1953-1966. DOI:10.1056/NEJMoa030781 |
| [5] |
Peiris JS, Guan Y, Poon LL. Severe acute respiratory syndrome (SARS)[J]. Journal of Clinical Virology the Official Publication of the Pan American Society for Clinical Virology, 2004, 10(S12): S88. |
| [6] |
Zaki AM, van Boheemen S, Bestebroer TM, et al. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia[J]. New England Journal of Medicine, 2012, 367(19): 1814-1820. DOI:10.1056/NEJMoa1211721 |
| [7] |
Zhang JQ, Guy JS, Snijder EJ, et al. Genomic characterization of equine coronavirus[J]. Virology, 2008, 369(1): 92-104. |
| [8] |
Belouzard S, Millet JK, Licitra BN, et al. Mechanisms of coronavirus cell entry mediated by the viral spike protein[J]. Viruses, 2012, 4(12): 1011-1033. DOI:10.3390/v4061011 |
| [9] |
Millet JK, Whittaker GR. Host cell proteases: critical determinants of coronavirus tropism and pathogenesis[J]. Virus Research, 2015, 202: 120-134. DOI:10.1016/j.virusres.2014.11.021 |
| [10] |
de Haan CAM, Stadler K, Godeke GJ, et al. Cleavage inhibition of the murine coronavirus spike protein by a furin-like enzyme affects cell-cell but not virus-cell fusion[J]. Journal of Virology, 2004, 78(11): 6048-6054. DOI:10.1128/JVI.78.11.6048-6054.2004 |
| [11] |
Kawase M, Shirato K, Matsuyama S, et al. Protease-mediated entry via the endosome of human coronavirus 229E[J]. Journal of Virology, 2009, 83(2): 712-721. DOI:10.1128/JVI.01933-08 |
| [12] |
Simmons G, Gosalia DN, Rennekamp AJ, et al. Inhibitors of cathepsin L prevent severe acute respiratory syndrome coronavirus entry[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(33): 11876-11881. DOI:10.1073/pnas.0505577102 |
| [13] |
Qiu ZZ, Hingley ST, Simmons G, et al. Endosomal proteolysis by cathepsins is necessary for murine coronavirus mouse hepatitis virus type 2 spike-mediated entry[J]. Journal of Virology, 2006, 80(12): 5768-5776. DOI:10.1128/JVI.00442-06 |
| [14] |
Wicht O, Li WT, Willems L, et al. Proteolytic activation of the porcine epidemic diarrhea coronavirus spike fusion protein by trypsin in cell culture[J]. Journal of Virology, 2014, 88(14): 7952-7961. DOI:10.1128/JVI.00297-14 |
| [15] |
Li WT, Wicht O, Kuppeveld FJ, et al. A single point mutation creating a furin cleavage site in the spike protein renders porcine epidemic diarrhea coronavirus trypsin independent for cell entry and fusion[J]. Journal of Virology, 2015, 89(15): 8077-8081. DOI:10.1128/JVI.00356-15 |
| [16] |
Gallagher TM, Escarmis C, Buchmeier MJ. Alteration of the pH dependence of coronavirus-induced cell fusion: effect of mutations in the spike glycoprotein[J]. Journal of Virology, 1991, 65(4): 1916-1928. |
| [17] |
Chernomordik LVM, Kozlov MM. Membrane hemifusion: crossing a chasm in two leaps[J]. Cell, 2005, 123(3): 375-382. DOI:10.1016/j.cell.2005.10.015 |
| [18] |
Watanabe R, Matsuyama S, Shirato K, et al. Entry from the cell surface of severe acute respiratory syndrome coronavirus with cleaved S protein as revealed by pseudotype virus bearing cleaved S protein[J]. Journal of Virology, 2008, 82(23): 11985-11991. DOI:10.1128/JVI.01412-08 |
| [19] |
Matsuyama S, Ujike M, Morikawa S, et al. Protease-mediated enhancement of severe acute respiratory syndrome coronavirus infection[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(35): 12543-12547. DOI:10.1073/pnas.0503203102 |
| [20] |
Burkard C, Verheije MH, Wicht O, et al. Coronavirus cell entry occurs through the endo-/lysosomal pathway in a proteolysis-dependent manner[J]. PLoS Pathogens, 2014, 10(11): e1004502. DOI:10.1371/journal.ppat.1004502 |
| [21] |
Cohen FS, Melikyan GB. The energetics of membrane fusion from binding, through hemifusion, pore formation, and pore enlargement[J]. The Journal of Membrane Biology, 2004, 199(1): 1-14. DOI:10.1007/s00232-004-0669-8 |
| [22] |
White JM, Delos SE, Brecher M. Structures and mechanisms of viral membrane fusion proteins: multiple variations on a common theme[J]. Critical Reviews in Biochemistry and Molecular Biology, 2008, 43(3): 189-219. DOI:10.1080/10409230802058320 |
| [23] |
Li F, Li WH, Farzan M, et al. Structure of SARS coronavirus spike receptor-binding domain complexed with receptor[J]. Science, 2005, 309(5742): 1864-1868. DOI:10.1126/science.1116480 |
| [24] |
de Haan CAM, Li Z, Te Lintelo E, et al. Murine coronavirus with an extended host range uses heparan sulfate as an entry receptor[J]. Journal of Virology, 2005, 79(22): 14451-14456. DOI:10.1128/JVI.79.22.14451-14456.2005 |
| [25] |
de Haan CAM, Te Lintelo E, Li Z, et al. Cooperative involvement of the S1 and S2 subunits of the murine coronavirus spike protein in receptor binding and extended host range[J]. Journal of Virology, 2006, 80(22): 10909-10918. DOI:10.1128/JVI.00950-06 |
| [26] |
Li F. Receptor recognition mechanisms of coronaviruses: a decade of structural studies[J]. Journal of Virology, 2015, 89(4): 1954-1964. DOI:10.1128/JVI.02615-14 |
| [27] |
Konishi S. Taguchi Methods: Signal-to-Noise Ratio for Quality Evaluation[M]. ASI Press, Japanese Standards Association, 1991
|
| [28] |
Liu C, Tang J, Ma YM, et al. Receptor usage and cell entry of porcine epidemic diarrhea coronavirus[J]. Journal of Virology, 2015, 89(11): 6121-6125. DOI:10.1128/JVI.00430-15 |
| [29] |
Weiss SR, Navas-Martin S. Coronavirus pathogenesis and the emerging pathogen severe acute respiratory syndrome coronavirus[J]. Microbiology and Molecular Biology Reviews, 2005, 69(4): 635-664. DOI:10.1128/MMBR.69.4.635-664.2005 |
| [30] |
Towler P, Staker B, Prasad SG, et al. ACE2 X-ray structures reveal a large hinge-bending motion important for inhibitor binding and catalysis[J]. Journal of Biological Chemistry, 2004, 279(17): 17996-18007. DOI:10.1074/jbc.M311191200 |
| [31] |
Prabakaran P, Xiao XD, Dimitrov DS. A model of the ACE2 structure and function as a SARS-CoV receptor[J]. Biochemical and Biophysical Research Communications, 2004, 314(1): 235-241. DOI:10.1016/j.bbrc.2003.12.081 |
| [32] |
Li WH, Moore MJ, Vasilieva N, et al. Angiotensin-converting enzyme 2 is a functional receptor for the SARS coronavirus[J]. Nature, 2003, 426(6965): 450-454. DOI:10.1038/nature02145 |
| [33] |
Hofmann H, Pyrc K, van der Hoek L, et al. Human coronavirus NL63 employs the severe acute respiratory syndrome coronavirus receptor for cellular entry[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(22): 7988-7893. DOI:10.1073/pnas.0409465102 |
| [34] |
Raj VS, Mou HH, Smits SL, et al. Dipeptidyl peptidase 4 is a functional receptor for the emerging human coronavirus-EMC[J]. Nature, 2013, 495(7440): 251-254. DOI:10.1038/nature12005 |
| [35] |
Jeffers SA, Tusell SM, Gillim-Ross L, et al. CD209L (L-SIGN) is a receptor for severe acute respiratory syndrome coronavirus[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(44): 15748-15753. DOI:10.1073/pnas.0403812101 |
| [36] |
Zhang YT, Buckles E, Whittaker GR. Expression of the C-type lectins DC-SIGN or L-SIGN alters host cell susceptibility for the avian coronavirus, infectious bronchitis virus[J]. Veterinary Microbiology, 2012, 157(3/4): 285-293. |
| [37] |
Schwegmann-Wessels C, Zimmer G, Schr der B, et al. Binding of transmissible gastroenteritis coronavirus to brush border membrane sialoglycoproteins[J]. Journal of Virology, 2003, 77(21): 11846-11848. DOI:10.1128/JVI.77.21.11846-11848.2003 |
| [38] |
Paltrinieri S, Rossi G, Meregalli A, et al. Sialic acid and sialyltransferase activity in serum and tissues of dogs with mammary tumors[J]. Veterinary Pathology, 2012, 49(4): 669-681. DOI:10.1177/0300985811402842 |
| [39] |
de Haan CAM, Haijema BJ, Schellen P, et al. Cleavage of group 1 coronavirus spike proteins: how furin cleavage is traded off against heparan sulfate binding upon cell culture adaptation[J]. Journal of Virology, 2008, 82(12): 6078-6083. DOI:10.1128/JVI.00074-08 |
| [40] |
Krempl C, Ballesteros ML, Zimmer G, et al. Characterization of the sialic acid binding activity of transmissible gastroenteritis coronavirus by analysis of haemagglutination-deficient mutants[J]. Journal of General Virology, 2000, 81(2): 489-496. DOI:10.1099/0022-1317-81-2-489 |
| [41] |
Krempl C, Schultze B, Laude H, et al. Point mutations in the S protein connect the sialic acid binding activity with the enteropathogenicity of transmissible gastroenteritis coronavirus[J]. Journal of Virology, 1997, 71(4): 3285-3287. |
| [42] |
Madu IG, Chu VC, Lee H, et al. Heparan sulfate is a selective attachment factor for the avian coronavirus infectious bronchitis virus beaudette[J]. Avian Diseases, 2007, 51(5): 45-51. |
| [43] |
Promkuntod N, van Eijndhoven REW, de Vrieze G, et al. Mapping of the receptor-binding domain and amino acids critical for attachment in the spike protein of avian coronavirus infectious bronchitis virus[J]. Virology, 2014, 448: 26-32. DOI:10.1016/j.virol.2013.09.018 |
| [44] |
Li BX, Ge JW, Li YJ. Porcine aminopeptidase N is a functional receptor for the PEDV coronavirus[J]. Chinese Journal of Virology, 2007, 365(1): 166-172. |
| [45] |
Shan ZF, Yin JY, Wang ZY, et al. Identification of the functional domain of the porcine epidemic diarrhoea virus receptor[J]. Journal of General Virology, 2015, 96(9): 2656-2660. DOI:10.1099/vir.0.000211 |
| [46] |
Nam E, Lee C. Contribution of the porcine aminopeptidase N (CD13) receptor density to porcine epidemic diarrhea virus infection[J]. Veterinary Microbiology, 2010, 144(1/2): 41-50. |
| [47] |
Shirato K, Maejima M, Islam MT, et al. Porcine aminopeptidase N is not a cellular receptor of porcine epidemic diarrhea virus, but promotes its infectivity via aminopeptidase activity[J]. Journal of General Virology, 2016, 97(10): 2528-2539. DOI:10.1099/jgv.0.000563 |
| [48] |
Oh JS, Song DS, Park BK. Identification of a putative cellular receptor 150 kDa polypeptide for porcine epidemic diarrhea virus in porcine enterocytes[J]. Journal of Veterinary Science, 2003, 4(4): 269-275. |
| [49] |
Feng D, Gang Y, Liu QQ, et al. Identification and comparison of receptor binding characteristics of the spike protein of two porcine epidemic diarrhea virus strains[J]. Viruses, 2016, 8(3): 55. DOI:10.3390/v8030055 |
| [50] |
Rasschaert D, Duarte M, Laude H. Porcine respiratory coronavirus differs from transmissible gastroenteritis virus by a few genomic deletions[J]. Journal of General Virology, 1990, 71(11): 2599-2607. DOI:10.1099/0022-1317-71-11-2599 |
 2017, Vol. 44
2017, Vol. 44