扩展功能
文章信息
- 鲁梅, 黄庆华, 崔宁, 许传田
- LU Mei, HUANG Qing-Hua, CUI Ning, XU Chuan-Tian
- 传染性支气管炎病毒N蛋白CTL表位与BF2*15鸡MHC Ⅰ基序的相互作用
- Interactions of the major histocompatibility complex class Ⅰ BF2*15 and cytotoxic T lymphocyte epitopes from nucleoprotein of avian infectious bronchitis virus
- 微生物学通报, 2017, 44(10): 2407-2414
- Microbiology China, 2017, 44(10): 2407-2414
- DOI: 10.13344/j.microbiol.china.170417
-
文章历史
- 收稿日期: 2017-06-10
- 接受日期: 2017-08-15
- 优先数字出版日期(www.cnki.net): 2017-08-16
2. 山东潍坊工程职业学院 山东 青州 262500
2. Weifang Engineering Vocational College, Qingzhou, Shandong 262500, China
表位疫苗是近年来发展起来的新型疫苗,其免疫效力在动物实验中得到验证,表位疫苗主要选取保护性抗原基因中决定抗原特异性的簇团。Zhou等研究表明,用转基因植物表达的IBV Sl蛋白免疫后,雏鸡可以对致病性IBV产生血清中和抗体,其脾脏可在体外分泌IL-2并发生T淋巴细胞增殖反应[1]。通过迟发型超敏反应(delayed typehyper sensitivity,DTH)研究进一步发现,S1蛋白的亚单位免疫可诱导产生强烈的DTH反应,这充分证实了细胞介导免疫在IBV感染后的免疫保护中起着重要作用[2-3]。
T细胞识别的MHC复合体是细胞免疫的先决条件,通过识别MHC Ⅰ类分子与表位肽复合物的CTL能够杀死感染病毒的细胞并能清除病毒[4-6]。因此,识别CTL抗原表位对于制造疫苗和理解病毒感染清除的免疫机制至关重要[7]。近年来在病原蛋白序列方面发展了基于特定的MHC Ⅰ蛋白结构,预测CTL抗原表位的一系列计算方法[8-10],以及可以识别在特殊抗原表位发生高于预期频率氨基酸的稳定性矩阵算法或人工神经网络(ANNAs)[11-15]。Liu等报道了鸡MHC Ⅰ (BF2*15)四聚体[16],表明了IBV 52株传染性支气管炎病毒N蛋白细胞表位(WRRQARYK)能与B15单倍体结合,并能诱导免疫应答[17],但是该研究并没有对该细胞表位和MHC Ⅰ结合的作用机理做进一步解释。
本研究是在已经鉴定IBV N蛋白的细胞表位基础之上,进一步用同源建模、分子动力学和分子对接的计算方法,通过探究IBV N蛋白T细胞表位多肽和BF2*15之间的关系,并最终确定BF2*15结合序列,并且用流式细胞术证明IBV N蛋白的细胞表位(WRRQARYK)能在体外诱导活化的鸡淋巴细胞产生CTL表位。
1 材料与方法 1.1 BF2*15和Chβ-2M序列从GenBank上获取BF2*15和Chβ-2M编码区序列(登录号:L28958,M84767)。此外,蛋白序列特征来源于UniProtKB的信息(登录号:Q9GIP6,P21611)。Chβ-2M是组成主要组织相容性复合体(MHC) Ⅰ类分子复合体的轻链分子,对于MHC Ⅰ类分子在细胞表面稳定表达和有效递呈抗原肽必不可少。
1.2 同源建模、分子动力模拟和蛋白对接首先通过BLAST,从PDB数据库(protein data bank)中寻找与目标序列相似的模板结构。使用Modeler 9v6程序包,选择序列相似性比较高的一个蛋白结构作为模板进行同源模建。然后利用NAMD 2.7程序包,在CHARMM力场下完成分子动力模拟[18],模拟前,蛋白/多肽需要先溶解在一个立方体水盒子中,并确保水盒边界距离蛋白的最小距离不小于10 Å[19],在模拟中使用周期性边界条件。非键作用力的截断值设为10 Å。蛋白对接由ZDOCK软件包完成,通过刚性对接的方式,模拟两个蛋白或者一条多肽与一个蛋白之间所有可能的相互作用模式,然后对每个作用模型打分,根据分值来评估最合理的相互作用模式。
1.3 BF2*15与WRRQARYK八肽相互作用关系分析WRRQARYK八肽来自传染性支气管炎病毒(IBV) N蛋白,八肽由Discovery Studio Visualizer的Build and Edit Protein模块直接产生,并通过分子动力学模拟软件对结构进行优化。蛋白对接使用ZDOCK软件包完成,经过蛋白对接后,共获得了2 000个不同的构象。选择ZDOCK Score打分高于14分的103个构象,使用RDOCK进一步优化。经过RDOCK优化后,选择了8个属于不同聚类的蛋白构象,并对RDOCK Score打分比较高的构象进行下一步的分析,通过分析确定八肽与BF2*15的结合序列。“WRRQARYK”、无关多肽NPaa89–97由生工生物工程(上海)股份有限公司合成。
1.4 CD8+ T淋巴细胞的增殖试验20只6周龄SPF鸡平均分成2组,实验组通过滴鼻免疫IBV 52疫苗,另一组滴鼻等量的PBS缓冲液作为空白对照。免疫后3周,无菌取实验组鸡的脾脏,置于平皿中,用Hanks洗涤3次,加入2 mL RPMI-1640培养液。粗剪成小块,用无菌的注射器芯管轻轻碾碎,200目筛网过滤到25 mL试管内。1 000 r/min离心3 min,弃上清,再用Hanks清洗3次,每次800 r/min离心3 min。将细胞悬于含10%胎牛血清的1640培养液中。取细胞悬液,小心加入到等体积的细胞分离液面上。收集淋巴细胞层放入含细胞洗涤液的试管中,充分混匀后,1 200 r/min离心20 min,弃上清留沉淀。将细胞重悬,重复洗涤2次即得淋巴细胞。取9 μL重悬细胞液,加入1 μL的0.4%台盼蓝染色液,染色3−5 min,计数活细胞后,用RPMI-1640完全培养液调整细胞浓度为1×106个/mL。将接种于24孔板,每孔1 mL,分别加入WRRQARYK合成肽刺激物及不相关多肽NPaa89-97,终浓度为100 μg/mL。同时设置正常的脾淋巴细胞为空白对照组。37 ℃、5% CO2培养3 d。取出培3 d后的脾脏淋巴细胞,将1×106个/mL的细胞放入1.5 mL的EP管中,3 000 r/min离心5 min,弃上清,加入100 μL PBS悬浮细胞。加入封闭抗体mouse-IgG (1×106/μL),室温15 min。加入荧光抗体Mouse Anti-Chicken CD8-PE (0.2 μg/106 cell),4 ℃,避光,30 min。加入350 μL PBS轻轻混匀,3 000 r/min离心5 min,弃上清。重复洗涤2次,最后将细胞重悬于500 μL PBS中,用流式细胞仪进行检测。
2 结果与分析 2.1 BF2*15 MHC Ⅰ结构预测目前已经获得鸡的MHC Ⅰ结构有12个,分别来自B2、B4、B14和B21[20-22]。本研究使用2个BF2*21结构(3BEV和3BEW)作为模板来构建BF2*15构型。B21和B15序列相似性和一致性分别为95.4%和93.2%。3BEV和3BEW的分辨率都比较高,分别为2.1 Å和2.6 Å,因此采用3BEV和3BEW作为模板进行同源模建是合理的。构建的BF2*15结构与鸡的其它MHC Ⅰ蛋白十分相似(图 1A)。接下来利用分子动力学模拟,利用NAMD软件包,在显式水模型下,对构建完成的MHC Ⅰ分子进行分子动力学模拟。模拟了3条轨迹,每条轨迹30 ns。对模拟轨迹中RMSD的变化和能量变化进行了分析,如图 1B和图 1C所示。最终得到的最低能量结构通过拉氏构象图分析确认了其结构是合理的(图 2)。BF2*15与3BEV和3BEW的均方根偏差(RMSD)分别为0.684 Å和0.649 Å (图 1B),说明该结构与其他单倍型MHC Ⅰ的结构是非常相似的。筛选出具有最低势能的BF2*15结构构象(图 1D)。

|
| 图 1 BF2*15 MHC Ⅰ最低能量的结构选择 Figure 1 The lowest energy structure choice of BF2*15 A:B15、B2 (4CVX)、B4 (4E0R)、B14 (4CW1)和B21 (3BEV)结构对比,B15以绿色ribbon显示,B2、B4、B14和B21分别以蓝色、橙色、黄色和红色ribbon显示;B:BF2*15 MHC结构分子模拟轨迹的RMSD分析,3条轨迹分别以黑色、红色和绿色标出;C:BF2*15 MHC Ⅰ结构分子模拟轨迹的势能分析,3条轨迹分别以黑色、红色和绿色标出;D:BF2*15最低能量构象,绿色卡通和紫色卡通分别表示MHC Ⅰ分子和β-2M分子. Note: A: The comparison of constructed B15, B2 (4CVX), B4 (4E0R), B14 (4CW1) and B21 (3BEV); All these proteins are shown as ribbon, and are colored green (B15), blue (B2), orange (B4), yellow (B14) and red (B21). B: The RMSD analysis of BF2*15 simulation trajectories, the RMSD analysis of BF2*15 simulation trajectories, and colored with black (trajectory 1), red (trajectory 2) and green (trajectory 3), respectively. C: The potential analysis of BF2*15 simulation trajectories, colored with black (trajectory 1), red (trajectory 2) and green (trajectory 3), respectively. D: The top view of lowest potential energy conformation of BF2*15, green and purple cartoon indicate BF2*15 and β-2M, respectively. |
|
|

|
| 图 2 BF2*15最低能量构象的拉氏构象图分析 Figure 2 The Ramachandran plot analysis of the BF2*15 lowest potential energy conformation |
|
|
首先利用Discovery Studio Visualizer构建未经折叠的IBV八肽结构,该结构呈直线形。然后我们通过分子动力学模拟对八肽结构进行充分的优化,以探索其在溶液中的折叠构象。分子动力学模拟进行了4条轨迹,每条轨迹的模拟时间为50 ns。在所有的模拟轨迹中,选择势能最低的八肽结构作为溶液中的折叠构象进行分子对接(图 3)。结构分析显示,由于存在4个正电残基,同时缺少负电残基,IBV多肽整体是带正电荷。此外,氨基端的游离氨基也对正电势有贡献。Lys8的游离羧基与Arg3的侧链相互作用,中和部分正电荷,产生八肽的环状构象。

|
| 图 3 最低势能的IBV八肽结构 Figure 3 The lowest potential energy conformation of IBV octapeptide 注:最低势能的IBV八肽结构由带颜色的条棍(A)和静电势面(B)表示.绿色、红色、蓝色和白色条棍分别表示碳原子、氧原子、氮原子和氢原子.蓝色表示正电性比较强区域,红色表示负电性比较强的区域. Note:The lowest potential energy conformation of IBV octapeptide, shown by colored sticks (A) and electrostatic potential surface (B). Green, red, blue and white sticks indicate carbon, oxygen, nitrogen and hydrogen atoms, respectively. Blue and red surface indicate positive charge and negative charged electrostatic potential, respectively. |
|
|
利用ZDOCK把BF2*15和IBV N蛋白多肽进行对接,在对接结果中选出ZDOCK评分高的8个构象进一步分析相互作用的可能构象(图 4)。经过模拟后,只有1个构象在116 ns模拟中始终能够紧密结合在MHC Ⅰ的沟槽中,且逐渐发生构象变化,最终形成一个伸展的构象,这表明八肽通过这个构象与BF2*15紧密结合(图 5)。在相互作用构象中,Arg2和Arg6定向到结合部位并分别与Asp24/Asp73和Asp73/Asp113形成盐桥。此外,Trp1的游离氨基与BF2*15的Glu62形成静电相互作用,主链则与Tyr7侧链氧形成氢键。最后,八肽中的Arg3、Ala5以及Gln4与BF2*15的Tyr149、Tyr97以及Thr64分别形成复杂的氢键网络,这些氢键大大稳定了八肽和MHC Ⅰ间的相互作用。

|
| 图 4 BF2*15与WRRQARYK八肽可能的相互作用模式示意图 Figure 4 Possible interactions between BF2*15 and IBV octapeptide 注:白色表面展示的是BF2*15分子,而对接后的多肽以不同颜色的卡通展示. Note: BF2*15 was displayed as with surface, and IBV octapeptide are shown as cartoon by different colors. |
|
|
分析发现最重要的相互作用力可能来自P2和P6两个位置的Arg,这两个Arg均指向沟槽内部,且都通过侧链与MHC Ⅰ形成了两个静电相互作用力。而其它氨基酸,要么没有形成稳定的相互作用,比如八肽的Tyr7,要么是以主链原子与MHC Ⅰ相互作用,比如八肽的Trp1、Gln4、Ala5。八肽的Lys8虽然通过侧链与Asp148形成了静电相互作用,但是这一静电作用力位于沟槽外侧,暴露在水溶液中,因此稳定性不如Arg2和Arg6在沟槽内部与MHC Ⅰ形成的静电相互作用力。Arg3的侧链氨基、Ala5主链羰基以及BF2*15的Tyr149侧链羟基形成了氢键网络,这一氢键网络对于多肽的结合也是有很大贡献的,但是Arg3的侧链指向沟槽的侧面,并非锚定氨基酸,因此重要性要低于Arg2和Arg6,由此,我们推测B15鸡MHC Ⅰ结合序列是“x-Arg-x-x-x-Arg-x-x”,其中P3位置也比较倾向于正电氨基酸,如Arg或者Lys。
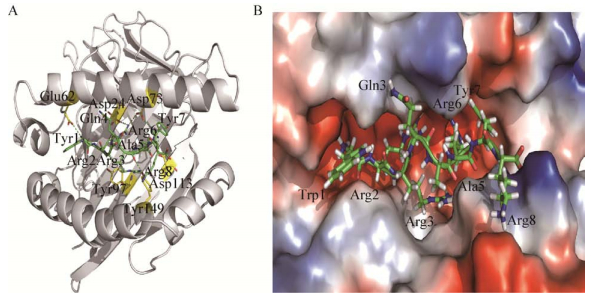
|
| 图 5 最低势能构象的BF2*15-IBV八肽复合物 Figure 5 The lowest potential energy conformation of BF2*15-IBV octapeptide complex 注:BF2*15与WRRQARYK八肽经过分子动力学模拟之后的最低势能结构卡通图(A)和表面图(B)。A:白色卡通表示BF2*15,绿色卡通表示WRRQARYK八肽;B:蓝色和红色分别表示正电性和负电性比较强的区域. WRRQARYK八肽以及参与相互作用的关键氨基酸以彩色条棍表示,绿色、红色、蓝色和白色条棍分别表示碳原子、氧原子、氮原子和氢原子. Note: The lowest potential energy conformation of BF2*15-IBV octapeptide complex, shown as electrostatic potential surface (B) and sticks as well as cartoons (A). A: Green and white cartoon indicate WRRQARYK and BF2*15, respectively. B: Blue and red surface indicate postive charge and negative charged electrostatic potential, respectively. Green, red, blue and white sticks indicate carbon, oxygen, nitrogen and hydrogen atoms, respectively. |
|
|
用Mouse Anti-Chicken CD8-PE对淋巴细胞标记,经流式细胞仪分群计数:发现经多肽WRRQARYK刺激后CD8+ T细胞有33.7%的增殖,不相关多肽NPaa89-97刺激的CD8+ T细胞略有增殖(2.3%),空白对照CD8+ T细胞增殖为0 (图 6)。这表明多肽WRRQARYK能有效刺激活化CD8+T淋巴细胞增殖。

|
| 图 6 不同肽刺激后CD8+ T细胞增值流式检测结果 Figure 6 CD8+ T cell proliferation after stimulation 注:A:正常脾细胞,作为对照;B:用WRRQARYK刺激后的脾细胞;C:用不相关多肽NPaa89–97刺激的脾细胞. Note: A: the control was normal spleen lymphocytes; B: the group was spleen lymphocytes stimulated with WRRQARYK; C: the group was spleen lymphocytes stimulated with NPaa89–97. |
|
|
传染性支气管炎病毒(IBV)是威胁养禽行业的主要病原之一,各种日龄的鸡均易感[23]。IBV不断出现新的血清型和基因型[24],而且同血清型或毒株间缺乏交叉保护或保护性差[25],因此IBV仍然流行不断,给养殖行业造成了巨大的经济损失。因此,研制更加高效、安全的IB疫苗已成为兽医科研的重点。研究表明细胞免疫可能在清除病毒方面起着重要作用[26]。因此,研发抗病毒的T细胞免疫疫苗更加值得关注。
研发T细胞免疫疫苗首先要解决的问题是鉴定正确的T细胞表位多肽,在本研究中,我们尝试利用蛋白对接配合分子动力学模拟的手段来预测BF2*15与WRRQARYK八肽的相互作用关系;然后通过分析BF2*15沟槽的化学特征、结构特征以及与抗原表位肽的结合模式,来推测其所识别的抗原表位肽motif。由于实验手段的限制,利用现有的实验条件,除了直接解析蛋白结构之外,比较难以确定BF2*15与WRRQARYK八肽的相互作用关系。然而结构的解析需要消耗大量的人力、财力和时间。
本研究利用分子建模、蛋白对接和MD模拟等技术描述了IBV八肽(WRRQARYK)与B15鸡MHC Ⅰ蛋白之间的相互作用过程,从蛋白分子结构水平上鉴定了IBV N蛋白的T细胞表位八肽(WRRQARYK)是鸡BF2*15单倍体的一个潜在的CTL表位,并且预测了B15鸡MHC Ⅰ的一条潜在的结合序列“x-Arg-x-x-x-Arg”,当然这个基序列需要通过进一步的分子生物学实验来验证,本研究仅仅是提出了一种假设。值得注意的是,这个基序与先前Wallny等[21]研究的基序有所不同,这说明BF2*15可以结合不同的基序。
淋巴细胞增殖试验结果显示:多肽(WRRQARYK)刺激活化T细胞后,CD8+ T细胞增殖33.7%,表明多肽(WRRQARYK)能够诱导CTL反应,是IBV N蛋白潜在的T细胞表位。
本研究为快速预测其它家禽病毒的CTL表位提供了一条思路,该研究结果也有助于对家禽病毒免疫机制更深层次的理解。
| [1] |
Zhou JY, Cheng LQ, Zheng XJ, et al. Generation of the transgenic potato expressing full-length spike protein of infectious bronchitis virus[J]. Journal of Biotechnology, 2004, 111(2): 121-130. DOI:10.1016/j.jbiotec.2004.03.012 |
| [2] |
Chen BY, Itakura C. Cytopathology of chick renal epithelial cells experimentally infected with avian infectious bronchitis virus[J]. Avian Pathology, 1996, 25(4): 675-690. DOI:10.1080/03079459608419174 |
| [3] |
Tian ZC, Sun YK, Wang YF, et al. The immunological efficacies of recombinant fowlpox virus expressing the S1 gene of LX4 strain of infectious bronchitis virus in specific-pathogen-free (SPF) chickens[J]. Acta Veterinaria et Zootechnica Sinica, 2006, 37(6): 580-586. 田占成, 孙永科, 王云峰, 等. 表达鸡传染性支气管炎病毒S1基因重组鸡痘病毒对SPF鸡的免疫保护作用[J]. 畜牧兽医学报, 2006, 37(6): 580-586. |
| [4] |
Travers P, Walport M, Shlomchik M, et al. Immunobiology: The Immune System in Health and Disease[M]. 5th Edition. New York: Taylor & Francis, Inc., 2001
|
| [5] |
Sette A, Peters B. Immune epitope mapping in the post-genomic era: lessons for vaccine development[J]. Current Opinion in Immunology, 2007, 19(1): 106-110. DOI:10.1016/j.coi.2006.11.002 |
| [6] |
Sette A, Fleri W, Peters B, et al. A roadmap for the immunomics of category A-C pathogens[J]. Immunity, 2005, 22(2): 155-161. DOI:10.1016/j.immuni.2005.01.009 |
| [7] |
Bui HH, Sidney J, Dinh K, et al. Predicting population coverage of T-cell epitope-based diagnostics and vaccines[J]. BMC Bioinformatics, 2006, 7: 153. DOI:10.1186/1471-2105-7-153 |
| [8] |
Rammensee H, Bachmann J, Emmerich NPN, et al. SYFPEITHI: database for MHC ligands and peptide motifs[J]. Immunogenetics, 1999, 50(3/4): 213-219. |
| [9] |
Parker KC, Bednarek MA, Coligan JE. Scheme for ranking potential HLA-A2 binding peptides based on independent binding of individual peptide side-chains[J]. The Journal of Immunology, 1994, 152(1): 163-175. |
| [10] |
Zhang GL, Srinivasan KN, Veeramani A, et al. PREDBALB/c: a system for the prediction of peptide binding to H2d molecules, a haplotype of the BALB/c mouse[J]. Nucleic Acids Research, 2005, 33(S2): W180-W183. |
| [11] |
Nielsen M, Lundegaard C, Worning P, et al. Improved prediction of MHC class Ⅰ and class Ⅱ epitopes using a novel Gibbs sampling approach[J]. Bioinformatics, 2004, 20(9): 1388-1397. DOI:10.1093/bioinformatics/bth100 |
| [12] |
Bui HH, Sidney J, Peters B, et al. Automated generation and evaluation of specific MHC binding predictive tools: ARB matrix applications[J]. Immunogenetics, 2005, 57(5): 304-314. DOI:10.1007/s00251-005-0798-y |
| [13] |
Peters B, Sette A. Generating quantitative models describing the sequence specificity of biological processes with the stabilized matrix method[J]. BMC Bioinformatics, 2005, 6: 132. DOI:10.1186/1471-2105-6-132 |
| [14] |
Larsen MV, Lundegaard C, Lamberth K, et al. An integrative approach to CTL epitope prediction: a combined algorithm integrating MHC class Ⅰ binding, TAP transport efficiency, and proteasomal cleavage predictions[J]. European Journal of Immunology, 2005, 35(8): 2295-2303. DOI:10.1002/(ISSN)1521-4141 |
| [15] |
Nielsen M, Lundegaard C, Worning P, et al. Reliable prediction of T-cell epitopes using neural networks with novel sequence representations[J]. Protein Science, 2003, 12(5): 1007-1017. DOI:10.1110/(ISSN)1469-896X |
| [16] |
Liu GL, Wang Q, Tong TH, et al. Construction and functional test of a chicken MHC-Ⅰ (BF2*15)/peptide tetramer[J]. Veterinary Immunology and Immunopathology, 2008, 122(1/2): 1-7. |
| [17] |
Boots AM, Kusters JG, van Noort JM, et al. Localization of a T-cell epitope within the nucleocapsid protein of avian coronavirus[J]. Immunology, 1991, 74(1): 8-13. |
| [18] |
Brooks BR, Brooks CL, Mackerell AD, et al. CHARMM: the biomolecular simulation program[J]. Journal of Computational Chemistry, 2009, 30(10): 1545-1614. DOI:10.1002/jcc.v30:10 |
| [19] |
Vreven T, Pierce BG, Hwang H, et al. Performance of ZDOCK in CAPRI rounds 20-26[J]. Proteins, 2013, 81(12): 2175-2182. DOI:10.1002/prot.v81.12 |
| [20] |
Kaufman J, Völk H, Wallny HJ. A "minimal essential Mhc" and an "unrecognized Mhc": two extremes in selection for polymorphism[J]. Immunological Reviews, 1995, 143(1): 63-88. DOI:10.1111/imr.1995.143.issue-1 |
| [21] |
Wallny HJ, Avila D, Hunt LG, et al. Peptide motifs of the single dominantly expressed class Ⅰ molecule explain the striking MHC-determined response to Rous sarcoma virus in chickens[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(5): 1434-1439. DOI:10.1073/pnas.0507386103 |
| [22] |
Chappell PE, Meziane EK, Harrison M, et al. Expression levels of MHC class Ⅰ molecules are inversely correlated with promiscuity of peptide binding[J]. eLife, 2015, 4: e05345. |
| [23] |
Bijlenqa G, Cook JKA, Gelb J, et al. Development and bronchitis virus from the Netherlands as a vaccine: a review use of the H strain of avian infectious[J]. Avian Pathology, 2004, 33(6): 550-557. DOI:10.1080/03079450400013154 |
| [24] |
Bonnefoy L, Bouquet JF, Picault JP, et al. Characterization of IBV variant strain PL 84084 isolated in France[A]//Laude H, Vautherot JF, eds. Coronaviruses[M]. Advances in Experimental Medicine and Biology. Boston, MA: Springer
|
| [25] |
Casais R, Dove B, Cavanagh D, et al. Recombinant avian infectious bronchitis virus expressing a heterologous spike gene demonstrates that the spike protein is a determinant of cell tropism[J]. Journal of Virology, 2003, 77(16): 9084-9089. DOI:10.1128/JVI.77.16.9084-9089.2003 |
| [26] |
Thomas PG, Keating R, Hulse-Post DJ, et al. Cell-mediated protection in influenza infection[J]. Emerging Infectious Diseases, 2006, 12(1): 48-54. DOI:10.3201/eid1201.051237 |
 2017, Vol. 44
2017, Vol. 44




