扩展功能
文章信息
- 陈夏林, 李由然, 顾正华, 丁重阳, 张梁, 石贵阳
- CHEN Xia-Lin, LI You-Ran, GU Zheng-Hua, DING Zhong-Yang, ZHANG Liang, SHI Gui-Yang
- 两种L-天冬氨酸α-脱羧酶的表达与酶学性质分析
- Expression and characterization of two L-aspartate alpha-decarboxylases
- 微生物学通报, 2017, 44(10): 2337-2344
- Microbiology China, 2017, 44(10): 2337-2344
- DOI: 10.13344/j.microbiol.china.170009
-
文章历史
- 收稿日期: 2017-01-04
- 接受日期: 2017-03-22
- 优先数字出版日期(www.cnki.net): 2017-03-24
2. 江南大学生物工程学院 江苏 无锡 214122
2. School of Biotechnology, Jiangnan University, Wuxi, Jiangsu 214122, China
L-天冬氨酸α-脱羧酶(L-aspartate-α-decarboxylase, EC4.1.1.11, ADC, PanD), 又称L-天冬氨酸1-脱羧酶, 可催化L-天冬氨酸脱掉α羧基生成β-丙氨酸[1-3]。β-丙氨酸是生物体内合成泛酸的重要前体物质, 是自然界中唯一存在的β型氨基酸, 是一种非蛋白氨基酸。β-丙氨酸及其衍生物广泛应用于医药、美容、食品、饲料及化工等领域, 市场需求量呈日渐上升趋势[4]。相较于国内外目前采用的化学合成法[5-6], 利用PanD生物转化L-天冬氨酸生产β-丙氨酸具有工艺简单、纯化方便、绿色无污染的特点, 具有十分明显的经济和社会效益[7]。
PanD主要存在于细菌、古细菌、放线菌等低等生物中, 可分为丙酮酰基团依赖型和磷酸吡哆醛依赖型两类。与后者相比, 前者具有明显的优势, 其催化能力依赖自加工形成的丙酮酰基团, 无需外源辅助因子, 且催化特异性高, 因此更具工业应用前景。PanD合成时, 最初转录翻译成无活性的原酶, 随后在Gly24-Ser25处发生裂解, 水解生成N端带有丙酮酰基团的α亚基和C端带有羟基的β亚基, 该丙酮酰基团即为酶催化的关键位点[8-9]。
酶转化反应中最关键的是生物催化剂(酶)的开发, 微生物的多样性为生物转化提供了更广阔的发展空间。NCBI数据库中已报道的panD基因有761种, 但目前的研究主要集中在大肠杆菌(Escherichia coli)[10]、结核杆菌(Mycobacterium tuberculosis)[11]、幽门螺杆菌(Helicobacter pylori)[12]、谷氨酸棒杆菌(Corynebacterium glutamicum)[13]、钝齿棒杆菌(Corynebacterium crenatum)[14]和枯草芽孢杆菌(Bacillus subtilis)[7], 而实际适合开发酶制剂的只有后3种。其中PanDB.s的酶活最高为8.4 U/mg, PanDC.g酶学性质的相关报道最多, 最适反应温度为55 ℃, 最适pH为6.0, 在低于37 ℃, pH 4.0−7.0稳定性较好。现有的PanD在催化过程中都存在一定程度的底物依赖性失活现象[10, 15], 这是限制其工业应用的关键性因素。已有的研究中, 尝试通过定点突变[16]、固定化作用[10]改变酶的性能, 但未见显著成果。因此挖掘新的panD基因来源并研究其特性对酶法合成β-丙氨酸具有重要的研究意义和工业价值。本文选择单核增生李斯特菌(Listeria monocytogenes)和杰氏棒杆菌(Corynebacterium jeikeium)来源的panD基因为研究对象, 首次实现了这两个基因的异源表达, 并对其酶学性质及底物抑制情况进行了比较分析, 为生物转化生产β-丙氨酸的工业应用提供了科学依据。
1 材料与方法 1.1 材料 1.1.1 目的基因、菌种及质粒: 研究中所用panD基因由苏州金唯智生物科技有限公司合成; E. coli BL21(DE3)、E. coli BL21(DE3)-panDC.g (携带有C. glutamicum来源的panD基因)和质粒pET-28a(+)均为本实验室保藏。 1.1.2 主要试剂、仪器和培养基: 限制性内切酶Nde Ⅰ、Hind Ⅲ、标准分子量蛋白购自Fermentas公司; T4 DNA连接酶、DNA marker购自TaKaRa公司; 卡那霉素购自生工生物工程(上海)股份有限公司; 质粒小量提取试剂盒、胶回收试剂盒购自北京博大泰克生物基因技术有限责任公司; Sepharose His trap HP购自美国GenScript公司; 其他试剂为国产分析纯。核酸电泳仪、Bio-Rad 525BR蛋白电泳仪购自美国Bio-Rad公司; 蛋白纯化仪AKTA Avant 25购自GE Healthcare公司; 高效液相色谱仪Agilent 1260购自美国Agilent公司。LB和TB培养基见参考文献[13]。 1.2 方法 1.2.1 重组菌E. coli DE3/pET28a(+)-panD的构建: 从NCBI上查阅并筛选获得L. monocytogenes和C. jeikeium来源的panD序列, 由苏州金唯智生物科技有限公司合成。基因序列按照E. coli密码子偏好性进行优化, 5′端添加CATATG (引入酶切位点Nde Ⅰ), 3′端去除终止密码子后加AATTGC (引入酶切位点Hind Ⅲ), 在蛋白N端和C端加上His-tag。基因合成后连接载体pUC57-simple, 获得pUC57-panD。提取pUC57-panD质粒, 用Nde Ⅰ和Hind Ⅲ双酶切, 经琼脂糖凝胶电泳鉴定、回收、纯化后, 用T4 DNA连接酶连接到同样经过Nde Ⅰ和Hind Ⅲ双酶切的pET28a(+)载体上, 构建重组表达载体pET28a(+)-panD, 将重组质粒转化到E. coli BL21(DE3)感受态细胞中, 选取卡那平板上阳性转化子提取质粒酶切验证, 得到重组菌E. coli BL21(DE3)/pET28a(+)-panD, 并保存于甘油管中。 1.2.2 重组蛋白的诱导表达: 从甘油管中接10 μL菌体至20 mL含50 mg/L卡那霉素的LB培养基中, 37 ℃、200 r/min培养8 h, 取2 mL菌液转接至50 mL含50 mg/L卡那霉素的TB培养基中, 37 ℃、200 r/min培养, 2 h后加入25 μl异丙基-β-D-硫代半乳糖苷(IPTG, 1 mol/L), 20 ℃、200 r/min培养16 h。发酵结束后收集菌体, 超声破碎, 通过Tricine-SDS-PAGE分析鉴定重组蛋白。Tricine-SDS-PAGE分析方法参见文献[17]。 1.2.3 重组蛋白的纯化: 用结合缓冲液A (20 mmol/L Na2HPO4·12H2O、20 mmol/L NaH2PO4·2H2O、500 mmol/L NaCl、100 mmol/L咪唑, pH 7.4)洗涤菌体2次, 加入5 mL结合缓冲液A, 振荡混匀后测OD600, 稀释OD至10后破碎, 破1 s停2 s, 破碎时间5 min, 破碎液于4 ℃、12 000 r/min离心20 min, 取上清, 即为粗酶液。Sepharose His trap HP用结合缓冲液A平衡后上样, 再平衡, 洗脱缓冲液B (20 mmol/L Na2HPO4·12H2O、20 mmol/L NaH2PO4·2H2O、500 mmol/L NaCl、500 mmol/L咪唑, pH 7.4)洗脱, 收集的洗脱液为纯酶液, 用Tricine-SDS-PAGE分析。采用Bradford法测定蛋白质浓度。 1.2.4 酶活的测定: 酶活定义:在37 ℃, pH 7.0的条件下, 每分钟转化产生1 μmol β-丙氨酸所需要的酶量为一个酶活单位U (μmol/min)。酶活的测定方法: 2.5 mL转化体系中, 将0.5 mg的酶与磷酸缓冲液(20 mmol/L Na2HPO4·12H2O, 20 mmol/L NaH2PO4·2H2O, pH 7.0)混合, 37 ℃预热20 min, 加入同样预热过的终浓度100 mmol/L的L-天冬氨酸(pH 7.0, NaOH溶解)进行转化反应, 反应20 min, 煮沸20 min灭活, 1 000 r/min离心20 min, 取上清保存, 用邻苯二甲醛(OPA)衍生β-丙氨酸, 使用HPLC检测衍生后β-丙氨酸含量[18]。
1.2.5 重组酶最适温度及热稳定性测定: 将重组酶分别置于30、37、45、55、60、65、70、80 ℃下反应20 min, 测定酶活, 以确定最适反应温度。将酶分别在以上温度放置12 h, 然后在37 ℃条件下测定酶活, 比较酶在不同温度条件下的稳定性。 1.2.6 重组酶最适pH及pH稳定性: 将0.5 mg重组酶分别加入20 mmol/L pH 4.0、5.0、6.0的柠檬酸-柠檬酸钠缓冲液、20 mmol/L pH 6.5、7.0、7.5的磷酸氢二钠-磷酸二氢钠缓冲液及20 mmol/L pH 8.0、9.0的Tris-盐酸缓冲液中, 再加入166 μL的L-天冬氨酸(200 g/L, pH 7.0, NaOH溶解), 37 ℃反应20 min, 测酶活, 以确定最适反应pH。将酶分别置于以上缓冲液中, 37 ℃放置12 h, 然后测酶活, 比较酶在不同pH条件下的稳定性。 1.2.7 底物L-天冬氨酸对PanD的抑制作用: 实验组取5 mg酶(PanD)加入到含有100 g/L的L-天冬氨酸和20 mmol/L的磷酸缓冲液(pH 7.0)的5 mL反应体系中, 37 ℃反应1 h, 用Ni柱回收酶, 取回收后的酶测酶活, 对照组底物浓度为0 g/L。 1.2.8 重组酶的转化实验: 用pH 7.0磷酸缓冲液配制终浓度为100 mmol/L的L-天冬氨酸底物50 mL, 加入10 mg的重组酶, 37 ℃、200 r/min反应24 h, 测β-丙氨酸转化率。 2 结果与分析 2.1 PanD氨基酸序列分析及密码子优化研究中的PanDC.j和PanDL.m氨基酸序列和目前报道较多的PanD的氨基酸序列进行比对, 从图 1可以看出, 这几个氨基酸序列相似程度较低, 仅有59.91%, 其中严格保守的残基有Lys9、His11、Arg12、Thr16、Ala18、Leu20、Tyr22、Gly24、Ser25、Ile28、Asp29、Ile46、Asn51、Gly52、Arg54、Thr57、Tyr58、Ile60、Ile69、Asn71、Gly72、Ala73、Ala74、Ala75、Gly81、Asp82、Val84、Ile85和Asn111, 高度保守位点较多。这些酶的自加工位点均为Gly24-Ser25键, 有研究表明Arg3、Arg54、Thr57、Tyr58可能与PanD自加工和催化活性有关[19], Arg54可能与PanD的底物特异性有关, Lys9可能对底物的α羧基的去质子化起到保护作用。PanD的来源不同, 其PanD氨基酸序列差异也较大, 这可能是导致其酶活与酶学性质差异的原因。

|
| 图 1 8种不同来源的PanD氨基酸序列对比 Figure 1 Amino acid sequence alignment of 8 PanDs 注: 8种PanD的GenBank登录号: M. tuberculosis (NP_218118.1)、C. jeikeium (WP_011273004.1)、C. glutamicum (NP_599388.1)、H. pylori (NP_206836.1)、B. subtilis (NP_390122.1)、L. monocytogenes (NP_465424.1)、S. enterica (NP_459185.1)、E. coli (NP_414673.1);严格保守残基用黑色框出, 自加工位点用三角标出. Note: Reference sequence of 8 PanDs: M. tuberculosis (NP_218118.1), C. jeikeium (WP_011273004.1), C. glutamicum (NP_599388.1), H. pylori (NP_206836.1), B. subtilis (NP_390122.1), L. monocytogenes (NP_465424.1), S. enterica (NP_459185.1), E. coli (NP_414673.1); Strictly conserved residues are boxed in black and the self-processing site is marked by a triangle. |
|
|
根据E. coli所偏爱的密码子对panDL.m和panDC.j基因进行密码子优化, (G+C)%含量分别由45.7%和49.3%变成54.18%和54.67%, 氨基酸未发生改变。人工合成的序列经克隆连接载体pUC57-simple, 获得pUC57-panD。
2.2 重组菌的构建将pUC57-panD质粒用Nde Ⅰ和Hind Ⅲ双酶切, 经琼脂糖凝胶电泳鉴定、回收、纯化后获得panD序列, 长度均在400 bp左右, 与理论值一致。将目的基因序列连接到pET28a(+) T7启动子下游, 获得重组质粒pET28a(+)-panD, 将上述重组质粒转化到E. coli BL21(DE3)感受态细胞中, 获得重组菌E. coli BL21(DE3)/pET28a(+)-panD。双酶切验证, 均得到了400 bp左右的目的条带(图 2), 说明外源基因已经成功连接到表达载体上。
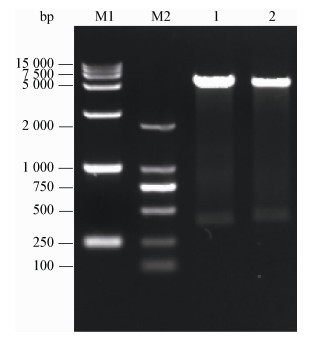
|
| 图 2 重组载体pET28a(+)-panD酶切验证 Figure 2 The enzymatic digestion of pET28a(+)-panD 注: M1: DL15000 DNA marker; M2: DL2000 DNA marker; 1−2: Nde Ⅰ和Hind Ⅲ双酶切重组载体pET28a(+)-panDL.m和pET28a(+)-panDC.j. Note: M1: DL15000 DNA marker; M2: DL2000 DNA marker; 1−2: Recombinant plasmid pET28a(+)-panDL.mand pET28a(+)-panDC.j digested by Nde Ⅰ and Hind Ⅲ. |
|
|
重组菌E. coli BL21(DE3)/pET28a(+)-panD经IPTG诱导16 h后, 超声破碎, 离心后取上清, 经Tricine-SDS-PAGE电泳检验, 图 3显示, 重组菌均成功表达了异源的PanD, 条带分析结果显示, 表达量均达到60%以上。这两组重组酶的自剪切类型与PanDC.g相同, 均不需要孵育, 可自加工裂解成α亚基与β亚基。破碎得到的上清液, 用His Trap HP镍柱纯化。Tricine-SDS-PAGE电泳验证结果如图 3所示, 均获得了较纯的目的蛋白, 可用于进一步研究。重组酶纯酶PanDL.m和PanDC.j在常规的酶活测定条件下(37 ℃, pH 7.0), 比酶活分别为8.9 U/mg和11.8 U/mg。
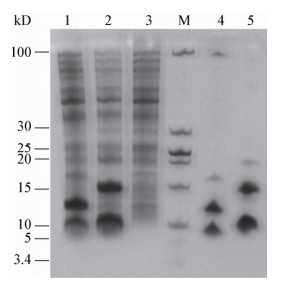
|
| 图 3 重组菌E. coli BL21(DE3)/pET28a(+)-panD表达产物的Tricine-SDS-PAGE电泳分析 Figure 3 The Tricine-SDS-PAGE analysis of expression product in recombinant strain 注: M:标准蛋白Marker; 1−3:重组菌BL21(DE3)/pET28a(+)-panDL.m、BL21(DE3)/pET28a(+)-panDC.j和BL21(DE3)/pET28a(+)细胞破碎上清; 4, 5:纯化后的重组蛋白PanDL.m和PanDC.j. Note: M: Standard protein marker; 1−3: The centrifugated supernatant of the recombinant BL21(DE3)/pET28a(+)-panDL.m, BL21(DE3)/pET28a(+)-panDC.j and BL21(DE3)/pET28a(+); 4, 5: Purified PanDL.m and PanDC.j. |
|
|
温度对两种PanD的酶活影响如图 4A所示, 两者都在60 ℃时达到最高酶活, 分别为10.9 U/mg和14.5 U/mg。随着温度的升高, PanD酶活均呈现先升高后降低的趋势。各温度下PanDC.j酶活均比PanDL.m高, 对温度变化也更敏感。从重组酶的热稳定性实验结果图 4B可以看出, 酶在设定的温度条件下放置12 h, 酶活均有一定程度的损失, 温度越高酶活损失越快。50–65 ℃范围内, PanDL.m较PanDC.j稳定, 当温度达到80 ℃, 两者的酶活基本完全丧失。
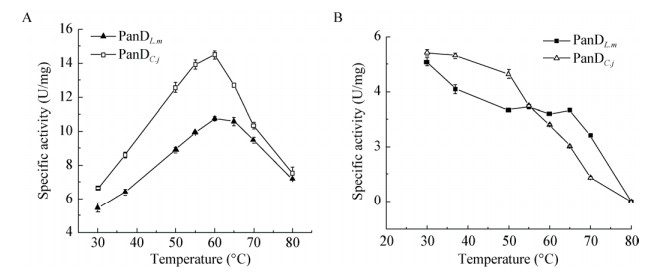
|
| 图 4 温度对酶活力的影响(A)及酶的热稳定性(B) Figure 4 Effect of reaction temperature on PanD activity (A) and thermal stability of the enzyme (B) |
|
|
PanDL.m和PanDC.j的最适pH和pH稳定性实验结果如图 5A和图 5B所示, 随着pH的增加, 两者的酶活均呈现先增后降的趋势, 都在酸性条件下比较稳定。PanDL.m适合在中性或酸性环境下作用, 最适pH为7.0, 当pH大于7.5时, 酶活迅速降低。PanDC.j可以在较宽的pH范围条件下作用, 最适pH为pH 6.0, 此时稳定性也最好, 酶活仅损失21%。

|
| 图 5 pH对酶活力的影响(A)和酶的pH稳定性(B) Figure 5 Effect of reaction pH on PanD activity (A) and pH stability of the enzyme (B) |
|
|
丙酮酰依赖型PanD在有底物L-天冬氨酸存在时, 催化位点丙酮酰基团与磷酸吡哆醛辅因子作用相似, 可与底物共价结合形成席夫碱[2], 促进底物脱羧, 最终产生β-丙氨酸, 但此过程中酶会发生自杀式的转氨作用, 发生底物依赖性失活现象[10, 15]。本研究中PanD酶与底物反应后, 通过Ni柱再次纯化回收PanD酶, 并去除L-天冬氨酸和反应产生的β-丙氨酸。对比PanDL.m、PanDC.j和目前研究较多的PanDC.g与底物反应前后酶活(图 6), 发现酶与底物反应后皆呈现出不同程度的失活现象, 分别损失了11%、43%和16%, 其中底物对PanDL.m的抑制作用最弱。
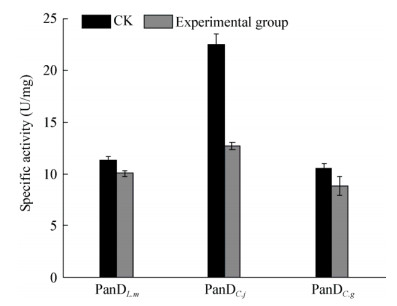
|
| 图 6 底物对酶活力的影响 Figure 6 Effect of substrate on PanDs activity |
|
|
转化反应过程中每隔1 h取样, 测定底物的转化率, 结果如图 7所示。不同目的基因来源的PanD对L-天冬氨酸的转化结果不同。反应前4 h, 底物的转化率均迅速增长, 但随后底物转化率的增长变得缓慢, 这可能与底物对酶的不可逆抑制作用有关。反应24 h时, PanDL.m、PanDC.j和PanDC.g三者对底物的转化率分别为67.7%、82.2%和58.0%, PanDL.m和PanDC.j对底物的转化率明显高于PanDC.g, 值得进一步研究应用于工业生产。
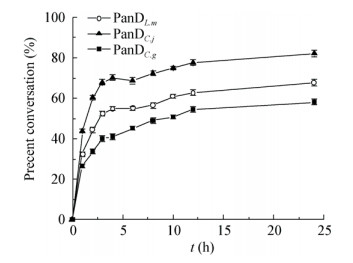
|
| 图 7 L-天冬氨酸的转化率 Figure 7 The conversion ratio of L-Asp |
|
|
PanD作为泛酸合成途径中的重要调控酶, 可作为一些病原微生物的药物靶细胞, 如H. pylori和M. tuberculosis, 是一种潜在的抗菌剂[2, 20]。在工业领域, PanD主要用于特异性催化L-天冬氨酸生成β-丙氨酸, 可将从多肽藻青素获得的大量L-天冬氨酸转化成更高价值的β-丙氨酸, 还可用于手性拆分制备D-天冬氨酸和β-丙氨酸[10, 21]。目前有关PanD的研究主要集中于少数几种来源的PanD酶的结构解析和自剪切机理方面, 很少关注PanD的酶学性质, 而更多来源的PanD的酶学特性尚不清楚, 有待研究者的开发。
本研究选择L. monocytogenes和C. jeikeium来源的PanD在E. coli中表达, 并对酶学性质进行了初步分析。PanDL.m和PanDC.j在普通测定条件下, 酶活分别达到了8.9 U/mg和11.8 U/mg, 高于现有报道PanD酶活[7]。两种酶催化反应适宜的温度及pH范围与现有的C. glutamicum来源的PanD无明显不同, 对底物的转化率分别为67.7%和82.2%, 明显高于PanDC.g, 为工业化应用奠定了基础。在实验过程中, 我们发现这两种重组酶的热稳定性并不是很好, 其中PanDC.j对温度尤为敏感, 后期可能需要优化反应条件与反应体系, 或从分子水平上对其进行改造, 提高酶反应的稳定性。同时, 我们也发现PanDL.m和PanDC.j与其他丙酮酰依赖型PanD相同, 在转化过程中也存在不同程度的底物依赖性失活现象。虽然PanDL.m酶活损失程度相对较低, 有助于提升β-Ala生物转化的效率, 但是目前这种不可逆的失活现象的机理研究并不透彻, 需要深入的研究, 从根本上解决PanD的底物依赖性失活问题, 这对于PanD转化L-天冬氨酸生产β-丙氨酸具有重要的意义。
| [1] |
Gopalan G, Chopra S, Ranganathan A, et al. Crystal structure of uncleaved L-aspartate-α-decarboxylase from Mycobacterium tuberculosis[J]. Proteins, 2006, 65(4): 796-802. DOI:10.1002/prot.21126 |
| [2] |
Lee BI, Suh SW. Crystal structure of the Schiff base intermediate prior to decarboxylation in the catalytic cycle of aspartate α-decarboxylase[J]. Journal of Molecular Biology, 2004, 340(1): 1-7. DOI:10.1016/j.jmb.2004.04.049 |
| [3] |
Cronaa JE Jr. β-Alanine synthesis in Escherichia coli[J]. Journal of Bacteriology, 1980, 141(3): 1291-1297. |
| [4] |
Luo JX, Xue JP, Shen YC. Synthesis and application of β-Alanine[J]. Amino Acids & Biotic Resources, 2005, 27(1): 52-55. 罗积杏, 薛建萍, 沈寅初. β-氨基丙酸的合成与应用[J]. 氨基酸和生物资源, 2005, 27(1): 52-55. |
| [5] |
Huang XM, Zhang ZB, Hong M, et al. Study on solation and purification of beta-alanine by biosythesis[J]. Amino Acids & Biotic Resources, 2008, 30(3): 46-50. 黄秀敏, 张正波, 洪敏, 等. 生物法合成β-丙氨酸的分离纯化工艺研究[J]. 氨基酸和生物资源, 2008, 30(3): 46-50. |
| [6] |
Zhang ZB, Gao LJ, Qiu JP. Determination of β-alanine in biocatalysis process[J]. Bulletin of Science and Technology, 2008, 24(6): 779-791. 张正波, 高丽娟, 裘娟萍. 生物转化体系中β-丙氨酸含量测定方法建立与优化[J]. 科技通报, 2008, 24(6): 779-791. |
| [7] |
Deng SY, Zhang JL, Cai Z, et al. Characterization of L-aspartate-α-decarboxylase from Bacillus subtilis[J]. Chinese Journal of Biotechnology, 2015, 31(8): 1184-1193. 邓思颖, 张君丽, 蔡真, 等. 枯草芽胞杆菌L-天冬氨酸α脱羧酶的酶学性质[J]. 生物工程学报, 2015, 31(8): 1184-1193. |
| [8] |
Monteiro DC, Patel V, Bartlett CP, et al. The structure of the PanD/PanZ protein complex reveals negative feedback regulation of pantothenate biosynthesis by coenzyme A[J]. Chemistry & Biology, 2015, 22(4): 492-503. |
| [9] |
Fouad WM, Rathinasabapathi B. Expression of bacterial L-aspartate-α-decarboxylase in tobacco increases β-alanine and pantothenate levels and improves thermotolerance[J]. Plant Molecular Biology, 2006, 60(4): 495-505. DOI:10.1007/s11103-005-4844-9 |
| [10] |
K nst PM, Franssen MCR, Scottel EL, et al. A study on the applicability of l-aspartate α-decarboxylase in the biobased production of nitrogen containing chemicals[J]. Green Chemistry, 2009, 11(10): 1646-1652. DOI:10.1039/b902731a |
| [11] |
Chen T, Xu SY, Feng Y. Inducing Conditions of Recombined L-aspartate α-decarboxylase in Fermentor[J]. Journal of Jinling Instutute of Technologoy, 2016, 32(3): 80-83. 陈涛, 徐世永, 冯炎. 结核杆菌L-天冬氨酸α-脱羧酶诱导表达条件研究[J]. 金陵科技学研学报, 2016, 32(3): 80-83. |
| [12] |
Kwon AR, Lee BI, Han BW, et al. Crystallization and preliminary X-ray crystallographic analysis of aspartate 1-decarboxylase from Helicobacter pylori[J]. Acta Crystallographica Section D: Biological Crystallography, 2002, 58(5): 861-863. DOI:10.1107/S0907444902004833 |
| [13] |
Zhao LZ, Zhang L, Shi GY. Expression of L-aspartate α-decarboxylase from Corynebacterium glutamicum in Escherichia coli and its application in enzymatic synthesis of β-alanine[J]. Microbiology China, 2013, 40(12): 2161-2170. 赵连真, 张梁, 石贵阳. 谷氨酸棒杆菌L-天冬氨酸α-脱羧酶在大肠杆菌中的表达及酶转化生产β-丙氨酸[J]. 微生物学通报, 2013, 40(12): 2161-2170. |
| [14] |
Hong M. Study on enzymatic production of β-alanine[D]. Hangzhou: Master's Thesis of Zhejiang University of Technology, 2010 (in Chinese) 洪敏. 酶法生产β-丙氨酸的研究[D]. 杭州: 浙江工业大学硕士学位论文, 2010 |
| [15] |
Anton DL, Kutny R. Mechanism of substrate inactivation of Escherichia coli S-adenosylmethionine decarboxylase[J]. Biochemistry, 1987, 26(20): 6444-6447. DOI:10.1021/bi00394a022 |
| [16] |
Shen Y. Molecular modification of L-aspartate-α-decarboxylase from Corynebacterium glutamicum by site-directed mutagenesis[D]. Wuxi: Master's Thesis of Jiangnan University, 2014 (in Chinese) 沈艳. 谷氨酸棒杆菌L-天冬氨酸α-脱羧酶催化性能的定点突变分子改造[D]. 无锡: 江南大学硕士学位论文, 2014 http://www.cnki.com.cn/Article/CJFDTOTAL-SWHZ201511008.htm |
| [17] |
Sch gger H. Tricine-SDS-PAGE[J]. Nature Protocols, 2006, 1(1): 16-22. DOI:10.1038/nprot.2006.4 |
| [18] |
Shen Y, Zhao LZ, Li YR, et al. Synthesis of β-alanine from L-aspartate using L-aspartate-α-decarboxylase from Corynebacterium glutamicum[J]. Biotechnology Letters, 2014, 36(8): 1681-1686. DOI:10.1007/s10529-014-1527-0 |
| [19] |
Cui WJ, Shi ZX, Fang YQ, et al. Significance of Arg3, Arg54, and Tyr58 of L-aspartate α-decarboxylase from Corynebacterium glutamicum in the process of self-cleavage[J]. Biotechnology Letters, 2014, 36(1): 121-126. DOI:10.1007/s10529-013-1337-9 |
| [20] |
Chopra S, Pai H, Ranganathan A. Expression, purification, and biochemical characterization of Mycobacterium tuberculosis aspartate decarboxylase, PanD[J]. Protein Expression and Purification, 2002, 25(3): 533-540. DOI:10.1016/S1046-5928(02)00039-6 |
| [21] |
Gao LJ, Qiu JP. Research advances in L-aspartate decarboxylase[J]. Industrial Microbiology, 2007, 37(5): 54-59. 高丽娟, 裘娟萍. L-天冬氨酸脱羧酶研究进展[J]. 工业微生物, 2007, 37(5): 54-59. |
 2017, Vol. 44
2017, Vol. 44




