扩展功能
文章信息
- 范森, 段梦洁, 刘亚兰, 林晓珊, 张毅
- FAN Sen, DUAN Meng-Jie, LIU Ya-Lan, LIN Xiao-Shan, ZHANG Yi
- 聚乳酸材料在不同土壤环境中生物降解的菌群结构分析
- Microbial diversity and community structure in biodegradation of poly(lactic) acid (PLA) in different soil environments
- 微生物学通报, 2017, 44(10): 2321-2329
- Microbiology China, 2017, 44(10): 2321-2329
- DOI: 10.13344/j.microbiol.china.160921
-
文章历史
- 收稿日期: 2016-12-14
- 接受日期: 2017-03-03
- 优先数字出版日期(www.cnki.net): 2017-03-03
塑料污染是当今世界面临的一个严重的环境问题,研究者们认为,使用可生物降解塑料是解决塑料污染的可行方案之一[1]。聚乳酸(PLA)是目前应用最为广泛的一种可生物降解材料,可通过玉米、木薯、马铃薯和甘蔗等可再生资源发酵生产[2],在微生物环境中可以被完全水解为CO2和H2O[3]。有研究表明,在自然环境中,聚乳酸的降解可大致分为两个过程:简单水解和酶催化降解。简单水解是水分子攻击聚乳酸分子中的酯键,使其分解为羧酸和醇的反应,主要受水解环境的温度、湿度、酸度以及聚合物本身的性质等因素影响;而聚乳酸的酶催化降解是指聚乳酸分子先水解为低聚物,然后相关微生物进入其组织物内,在微生物产生的特定酶的作用下,被分解为二氧化碳和水[4],此过程与天然聚合物的降解不同,属于间接方式降解。
聚乳酸材料有光降解、氧化降解、土壤降解3种降解方式,在一般情况下,光降解不能完全降解聚乳酸材料,氧化降解往往会造成额外污染,因此土壤降解是最为有效的降解方法[5]。Weng等通过控制不同的发酵条件研究聚乳酸的降解作用[6];Rudnik和Briassoulis比较了聚乳酸材料在堆肥环境下和自然环境下的降解效果,结果表明,聚乳酸材料在堆肥环境和自然环境下都可以被有效降解[7]。然而聚乳酸材料在自然环境下的降解速率非常缓慢,如何提高它的降解速率就成为了当前研究的热点之一。
聚乳酸在土壤中的降解已经有了一些研究[3, 7-8],对于聚乳酸的水解方面,研究主要集中在材料的类型、掩埋的方式及温度、湿度、pH等发酵条件的控制上。在降解聚乳酸的微生物方面,研究者们最早发现对聚乳酸有降解作用的微生物是放线菌,它具有高效降解聚酯的作用[8-9]。Pranamuda等分离了第一株可降解聚乳酸的放线菌HT-32,属于拟无枝酸菌属(Amycolatopsis)[10],其他可降解聚乳酸的放线菌分别属于假诺卡氏菌属(Pseudonocardia)、糖丝菌属(Saccharothrix)、伦茨氏菌属(Lentzea)、拟孢囊菌属(Kibdelosporangium)[1]和马都拉放线菌属(Actinomadura)[11]。随着相关研究的深入,越来越多的微生物被发现具有降解聚乳酸的作用,如史氏芽孢杆菌、镰刀酶念珠菌、青霉菌、腐殖菌等。
聚乳酸的降解是个复杂的过程,需要在多种酶的协同作用下进行,单一菌株对聚乳酸的降解效果并不理想[9]。另外,在自然环境中只有不到1%的微生物可以被培养,一些不可培养的微生物也可能和聚乳酸的降解有关。因此,从整个环境微生物种群结构方面进行研究,可以更全面地了解微生物对聚乳酸材料的降解作用,但目前对于这方面的研究还比较匮乏。
本文选取了3种不同的土壤,在相同的处理条件下比较它们对聚乳酸材料的降解能力,并利用高通量测序技术对这3种土壤中细菌群落组成和结构进行分析,结合它们对聚乳酸材料的降解能力,通过比较它们种群结构之间的差异,找到环境中能够降解聚乳酸材料的优势菌群,为有效降解聚乳酸材料微生物资源的开发提供理论依据。
1 材料与方法 1.1 聚乳酸材料聚乳酸材料来自于华南理工大学材料科学与工程学院。材料中含有96%的L-乳酸异构体和4%的D-乳酸异构体,材料的密度为2.3 g/cm3,熔点为145−170℃,玻璃化转变温度为55−58 ℃。
1.2 土壤采集3种土壤于2015年6月分别采自广东省广州市番禺区内沼泽地、芒果林地、稻田15−20 cm土壤,并用2 mm筛子处理土壤,去除与实验无关的杂物,放在4 ℃保存。
1.3 主要试剂和仪器Soil total DNA提取试剂盒,美国OMEGA公司;Premix Taq,上海捷瑞生物工程有限公司;无机试剂,天津永大化学试剂公司。扫描电镜,德国ZEISS公司;电子强力机,美国INSTRON公司;MiSeq,美国Illumina公司。
1.4 聚乳酸材料在土壤中的生物降解聚乳酸材料剪成250 mm×50 mm大小,分别掩埋在装有3种土壤的纸箱中,掩埋深度大约7 cm,并保持在温度25、湿度60%条件下进行降解实验。整个降解过程共4个月,每月取一次样,每次3个平行样。所取样品用蒸馏水冲洗4遍,室温下干燥后4 ℃保存,以备分析测试[12]。
1.5 材料降解效果检测 1.5.1 样品表面形态变化观察: 使用扫描电镜对降解前后的聚乳酸材料进行电镜观察。加速电压20 kV,测试之前样品表面经过真空喷金处理,以消除电荷积累。 1.5.2 机械性能测试实验: 利用电子强力机,执行标准为GB/T3923.1-1997,控制测试房间温度20±2 ℃、湿度为65%的条件下进行拉伸性能测试,测试时样品夹距150 mm,拉伸速度20 mm/min,测量不同降解时间段取样样品的断裂强力和伸长率变化[13],每个取样时间段(每月一次)、每类样品各测3个平行样取平均值。 1.5.3 无机碳含量测试: 根据EN ISO 14852-2004的方法[9],搭建装置如图 1所示,气泵流量控制在60−100 mL/min,每个三角瓶中装有200 mL的液体,反应器中分别装有5 g 3种土壤和聚乳酸粉末。前两个三角瓶中的NaOH (0.05 mol/L)溶液用于吸收气泵吹出气体中的CO2,第三个三角瓶中的Ba(OH)2(0.012 5 mol/L)溶液用于确定吹出气体中的CO2是否除尽,后面3个瓶子中的Ba(OH)2(0.012 5 mol/L)溶液用于吸收反应过程中样品分解后释放的CO2。用标准0.05 mol/L的NaOH溶液矫正配制的0.05 mol/L的HCl溶液,再用酸碱滴定的方法测定样品分解后释放的CO2量。设置灭菌土壤对照组和无聚乳酸材料对照组,分别用来消除PLA自然水解和微生物自然生长所释放的CO2量。
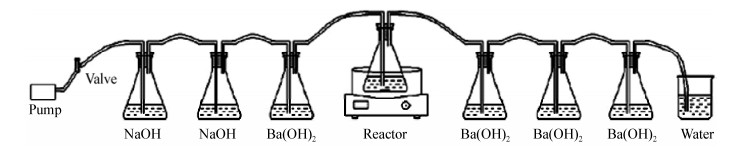
|
| 图 1 生物降解试验CO2释放量测试装置 Figure 1 Devices detecting the amount of CO2 evolved by PLA in the degradation process |
|
|
分别取0.5 g 3种土壤,使用DNA试剂盒提取土壤总DNA。用0.8%的琼脂糖凝胶电泳检查DNA的纯度和浓度,胶回收纯化DNA,并使用无菌水稀释样品至1 ng/μL。
1.7 PCR扩增及上机测序PCR扩增采用16S rRNA基因的V3-V4区域作为目标DNA序列,以通用引物338F (5′-ACTCCT ACGGGAGGCAGCA-3′)和506R (5′-GGACTACHV GGGTWTCTAAT-3′)对16s rRNA基因的V3-V4进行扩增。PCR反应体系:模板DNA 1 μL,上下游引物(10 μmol/L)各1 μL,ddH2O 10.5 μL,Premix Taq 12.5 μL。反应条件:94 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,25个循环;72 ℃ 10 min。扩增后的PCR产物经琼脂糖电泳检测呈阳性后,使用荧光计TBS-380对PCR产物进行均一化混匀,构建MiSeq文库,然后用Illumina MiSeq上机测序。该测序工作由广州基迪奥生物科技有限公司完成。
1.8 数据分析处理 1.8.1 OTU (Operating taxonomic unit)分类: 为获得更加精确的生物学信息,利用Mothur软件包对Tag序列进行了去冗余处理,从中挑选出Unique tag序列。为了更好地获得样品中物种的多样性信息,先对Tags进行OTU聚类,利用Mothur计算0.03距离下(97%的相似度)的OTU数量,用Mothur软件构建稀释性曲线[14-15]。 1.8.2 Alpha多样性分析: 基于OTU的结果,利用QIIME软件计算样品Chao1指数和Simpson指数[15]。其中群落丰富度用Chao1指数描述,其值越高表明群落物种的丰富度越高;而样品的多样性用Simpson指数描述,其值越大表明样品群落多样性越低。Chao1指数和Simpson指数的计算公式为:
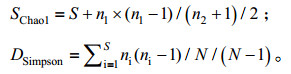
|
式中,SChao1是Chao1指数,DSimpson是Simpson指数,S是得到的OTU数量,n1是只有一条序列的OTU数目,n2是只有2条序列的OTU数目,ni是含有i条序列的OTU数目。N是微生物群落中总个体的数量[16-17]。
1.8.3 Beta多样性分析: 利用QIIME软件计算样品Beta多样性距离矩阵,并根据Beta多样性距离矩阵进行层次聚类分析,根据OTU的表达谱计算样品间距离,并对样品进行聚类分析,以判断在OTU水平上各样品的相似性。距离计算方法为Kld_jsd[18],并用R语言绘制物种分布堆叠图。 2 结果与分析 2.1 聚乳酸材料在3种土壤中的降解效果 2.1.1 扫描电镜下聚乳酸材料表面形态的变化: 用电子显微镜对降解前后的聚乳酸材料进行观察,电镜图如图 2所示。图 2A、B、C、D分别为未降解前样品及分别经过沼泽地土壤、芒果林地土壤、稻田土壤降解4个月后的样品。从图 2中可以明显看出,聚乳酸材料在降解前纤维表面很光滑,无断裂现象,经过沼泽地土壤降解后样品纤维表面被严重破坏,单根纤维裂痕很明显;经过芒果林地土壤降解后样品纤维表面有轻微腐蚀现象,但无明显腐蚀断裂;而经过稻田土壤降解后的样品整体感觉完好,纤维并没有明显断裂情况。由此可得出结论:材料在3种土壤中的降解效果为沼泽地土壤 > 芒果林地土壤 > 稻田土壤。

|
| 图 2 聚乳酸材料降解前后SEM照片 Figure 2 SEM images of PLA before and after degradation |
|
|
| 降解土壤 Soil samples |
降解时间 Degradation time (months) |
断裂强度 Tensile strength (MPa) |
断裂拉伸率 Elongation at break (%) |
| 沼泽土壤Swamp soil | 0 | 76.30±4.77 | 5.32±0.45 |
| 1 | 57.70±3.44 | 5.33±0.32 | |
| 2 | 50.30±3.70 | 7.94±1.09 | |
| 3 | 46.40±2.54 | 7.60±1.70 | |
| 4 | 43.90±2.90 | 4.67±1.14 | |
| 芒果林地土壤Orchard soil | 0 | 76.30±4.77 | 5.32±0.45 |
| 1 | 60.60±3.89 | 7.26±0.71 | |
| 2 | 53.30±3.41 | 5.82±0.71 | |
| 3 | 49.40±2.00 | 4.80±1.00 | |
| 4 | 46.60±2.54 | 4.45±0.54 | |
| 稻田土壤Farmland soil | 0 | 76.30±4.77 | 5.32±0.45 |
| 1 | 67.20±3.23 | 5.73±0.73 | |
| 2 | 61.50±3.05 | 5.96±0.68 | |
| 3 | 58.40±3.12 | 4.65±0.92 | |
| 4 | 55.30±3.16 | 4.81±0.58 |

|
ThCO2:样品理论CO2含量;M:实验所用降解材料的质量;Xc:实验材料的含碳量,25.93%;m:CO2释放量;CA:HCl溶液的准确浓度;CB:滴定前Ba(OH)2溶液的准确浓度;VB:试验开始时Ba(OH)2溶液的体积;VBt:滴定前在t时间Ba(OH)2溶液的体积;VBZ:中和滴定时消耗的Ba(OH)2溶液体积;VA:中和滴定时所用HCl体积。
实验结果如图 3所示。因为灭菌土壤对照组和无PLA材料对照组释放的CO2量很少,可忽略不计。聚乳酸材料在沼泽地土壤、芒果林地土壤和稻田土壤中的降解率分别为13.7%、10.6%和4.5%。沼泽地土壤对聚乳酸材料的降解率分别是芒果林地土壤和稻田土壤的1.29和3.10倍。
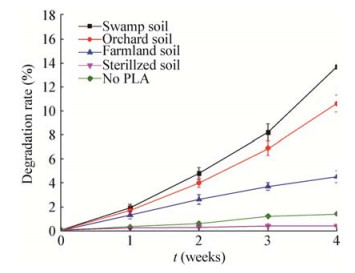
|
| 图 3 聚乳酸材料在3种土壤中的降解率 Figure 3 Degradation of PLA after incubation in three kinds of soil |
|
|
综合扫描电镜、断裂拉伸强度和CO2释放量的测试结果可以得出结论:(1)聚乳酸材料可以在土壤条件下自然降解;(2)聚乳酸材料在沼泽地土壤中降解效果最好,芒果林地土壤次之,在稻田土壤中降解效果最差;(3)聚乳酸材料在沼泽地土壤和芒果林地土壤中的降解效果接近,并远胜于稻田土壤。
2.2 3种土壤的群落多样性通过对V3-V4区测序,沼泽、林地、稻田3种土壤样品分别得到原始序列76 553、74 094和59 461条,经过去冗余处理,3种土壤样品分别得到74 566、71 442和57 613个有效序列,分别测得11 110、11 236和8 848个OTU (表 1)。
对样品间Alpha多样性分析,采用Alpha多样性指标中的Chao1指数和Simpsonz指数来评价样品的丰富度和多样性。结果如表 2所示,沼泽地土壤和林地土壤的物种多样性和丰富度高于稻田土壤。
| Soil samples | Tag | Unique tag | OTUs | Chao1 | Simpson |
| Swamp soil | 76 553 | 74 566 | 11 110 | 20 844.8 | 0.001 837 |
| Orchard soil | 74 094 | 71 442 | 11 236 | 20 147.7 | 0.002 408 |
| Farmland soil | 59 461 | 57 613 | 8 848 | 15 511.9 | 0.003 180 |
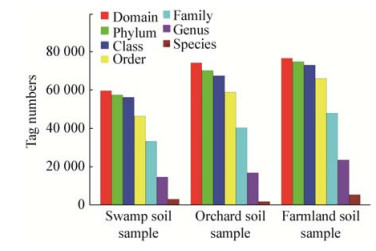
利用RDP classifier对各样品中OTU依次进行门(Phylum)、纲(Class)、目(Order)、科(Family)、属(Genus)分类分析,进一步挖掘样品中种群性群落组成。3种土壤样品共涉及细菌域的酸杆菌门(Acidobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、绿弯菌门(Chloroflexi)、厚壁菌门(Firmicutes)、芽单胞菌门(Gemmatimonadetes)、硝化螺旋菌门(Nitrospira)、变形菌门(Proteobacteria)、WS3 (Candidate division类之一,这一类微生物为原核微生物,没有发现可培养的代表菌株,但已通过16S rRNA基因宏基因组分析确定其存在分支)等9个主要菌门及多个少量细菌门和未知菌群。
如图 4所示,3种土壤样品中微生物群落结构组成情况相似,但各种微生物所占比例有着明显差异。变形菌门(Proteobacteria)在3种土壤中均占主要优势,在沼泽地土壤、芒果林地土壤和稻田土壤中分别占微生物群落的45%、38%、36%。拟杆菌门(Bacteroidetes)是沼泽地土壤的第二大优势菌门,占比接近20%,但在芒果林地土壤和稻田土壤中占比很低,尤其是在稻田土壤中,仅有3%。酸杆菌门(Acidobacteria)和厚壁菌门(Firmicutes)是芒果林地土壤的第二和第三大菌门。酸杆菌门(Acidobacteria)和放线菌门(Actinobacteria)是稻田土壤的第二和第三大菌门。硝化螺旋菌门(Nitrospira)和WS3在3种土壤中的占比都最少。

|
| 图 4 Phylum水平各样本菌群分布图 Figure 4 Relative abundance of Phylum in microbial communities from samples |
|
|
在进行分类分析时,随着注释等级的降低(如从属到种),能够注释到下一等级tag序列数总是呈下降趋势,因此需要找到最佳物种分类水平,即分类等级尽可能低和注释上的Tag数量尽可能多的水平。为了确定最佳的物种分类水平,对各个分类水平上的Tag序列数进行统计。如图 5所示,3个样品均有多于54.49%的Tag序列能够注释到“科”的水平,而能够注释到“属”水平的Tag序列低于22.67%,这种情况下选择“科”作为3个样品的最佳分类水平。
|
| 图 5 样品各分类水平中Tag数统计 Figure 5 Statistics of tags number at each classification level |
|
|
在科的水平上,各样品中共涉及115个科,表达丰度占总样本2%以上的科共有16个。如图 6所示,样品间优势科分布存在极大的差异性。黄杆菌科(Flavobacteriaceae)、丛毛单胞菌科(Comamonadaceae)和噬纤维菌科(Cytophagaceae)在沼泽地土壤中占比最多,它们分别属于拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)和放线菌门(Actinobacteria)。前两个门类为沼泽土壤的优势门类,而在放线菌门中,大量不同种属的放线菌已被证明具有降解聚乳酸的作用。林地土壤和稻田土壤中各科微生物占比比较平均,没有特别优势的科类。在3种样品中,未分类的科均为最主要菌群,分别占36%、46%和42%,表明还有大量的未知微生物资源有待开发。

|
| 图 6 Famliy水平各样本菌群分布图 Figure 6 Relative abundance of Famliy in microbial communities from samples |
|
|
结合土壤对材料的降解作用,沼泽地土壤 > 芒果林地土壤 > > 稻田土壤的结果,可以得出结论:(1)土壤菌群多样性和丰富度越高,对聚乳酸材料的降解作用越好。(2)变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes)在对聚乳酸材料的降解中起到了更主要的作用,这两个门类中可能存在着数量更多、降解能力更好的微生物,尤其是黄杆菌科(Flavobacteriaceae)和丛毛单胞菌科(Comamonadaceae)。这一结果不仅找到了环境中能降解聚乳酸材料的优势菌群,而且为降解聚乳酸材料微生物资源的发掘提供了理论依据。
3 结论与讨论随着聚乳酸可生物降解材料的广泛应用,建立可快速降解聚乳酸材料的体系对扩大聚乳酸材料的工业应用具有重要意义。只有尽可能多地获取与聚乳酸生物降解和循环利用有关的信息,才能使聚乳酸的降解更加快速、安全、无污染,从而真正实现环境友好化[19]。近年来有很多研究者从特定的环境中分离出降解聚乳酸材料的菌株进行研究,如南开大学的宋存江课题组对分离出的200多株细菌进行聚乳酸材料降解实验,发现聚乳酸的降解是个复杂的过程,需要在多种酶的协同作用下进行,单一菌株对材料的降解效果并不理想[9]。因为在自然环境中,只有不到1%的微生物可以被培养,一些不可培养的微生物也可能和聚乳酸的降解有关。因此,本文从环境微生物的整体水平进行研究,通过对聚乳酸材料在3种土壤中自然降解后的表面形态变化、机械性能变化、二氧化碳释放量的比较,证明聚乳酸材料可以在土壤条件下自然降解,而且在沼泽地土壤中降解效果最好,芒果林地土壤次之,在稻田土壤中降解效果最差。利用高通量测序技术对沼泽地、芒果林地、稻田3种土壤的微生物群落结构及多样性进行分析。结果表明土壤菌群多样性和丰富度越高,对聚乳酸材料的降解作用越好。
此前,研究者们通过克隆文库和DNA测序的方法鉴定了一些对聚乳酸材料有降解作用的细菌,克隆出的基因序列属于多个门类,主要集中在放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、绿弯菌门(Chloroflexi)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)[20-21]。本文对3种土壤进行高通量测序,沼泽地、芒果林地、稻田3种土壤的样品分别测得11 110、11 236和8 848个OTU,共涉及细菌域的9个主要门和16个主要科。结合土壤的降解效果,变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes)是降解聚乳酸材料的优势菌群。在科水平上,黄杆菌科(Flavobacteriaceae)、丛毛单胞菌科(Comamonadaceae)和噬纤维菌科(Cytophagaceae)的微生物对聚乳酸材料的降解最有潜力。在一些研究者的报道中,Brevundimonas和Acidisphaera作为聚乳酸材料的降解菌株属于变形菌门(Proteobacteria)[22],Cytophaga属于拟杆菌门(Bacteroidetes),可以降解纤维素[23]。这些报道与我们的研究结果一致,也证实在这两个门中存在着大量有待开发的可降解聚乳酸材料的微生物。
Sun等指出,16S rRNA基因在原核生物内往往同时存在多个拷贝,而且拷贝之间的基因序列并不完全一致,因此基于16S rRNA基因的菌群多样性分析会引起一定程度的高估[24]。为了降低这一高估的影响,本文选取了高估程度较低的V3-V4区(约5%)作为高通量测序的目的片段。尽管这一高估不可避免,使3种土壤测得的菌群多样性比真实情况略高,但对本研究的结论影响不大。
变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes)作为能够降解聚乳酸材料的优势菌群,如果把分离出的几种或几十种此门类的细菌进行不同比例的混合,也许可以大大提高材料的降解速率。此外,研究变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes)微生物的生长习性,在自然土壤中处理聚乳酸材料时,控制有利于这两个门类细菌生长的条件,也可以大大提高材料的降解速率。总之,本研究为有效降解聚乳酸材料微生物资源的开发提供了理论依据。
| [1] |
Jarerat A, Pranamuda H, Tokiwa Y. Poly(L-lactide)-degrading activity in various actinomycetes[J]. Macromolecular Bioscience, 2002, 2(9): 420-428. DOI:10.1002/mabi.200290001 |
| [2] |
John RP, Nampoothiri KM, Pandey A. Fermentative production of lactic acid from biomass: an overview on process developments and future perspectives[J]. Applied Microbiology and Biotechnology, 2007, 74(3): 524-534. DOI:10.1007/s00253-006-0779-6 |
| [3] |
Tokiwa Y, Jarerat A. Biodegradation of poly(l-lactide)[J]. Biotechnology Letters, 2004, 26(10): 771-777. DOI:10.1023/B:BILE.0000025927.31028.e3 |
| [4] |
Copinet A, Legin-Copinet E, Erre D. Compostability of co-extruded starch/poly(lactic acid) polymeric material degradation in an activated inert solid medium[J]. Materials, 2009, 2(3): 749-764. DOI:10.3390/ma2030749 |
| [5] |
Kim J, Seo SM, Lee SG, et al. Nematicidal activity of plant essential oils and components from coriander (Coriandrum sativum), Oriental sweetgum (Liquidambar orientalis), and valerian (Valeriana wallichii) essential oils against pine wood nematode (Bursaphelenchus xylophilus)[J]. Journal of Agricultural and Food Chemistry, 2008, 56(16): 7316-7320. DOI:10.1021/jf800780f |
| [6] |
Weng YX, Jin YJ, Meng QY, et al. Biodegradation behavior of poly(butylene adipate-co-terephthalate) (PBAT), poly(lactic acid) (PLA), and their blend under soil conditions[J]. Polymer Testing, 2013, 32(5): 918-926. DOI:10.1016/j.polymertesting.2013.05.001 |
| [7] |
Rudnik E, Briassoulis D. Comparative biodegradation in soil behaviour of two biodegradable polymers based on renewable resources[J]. Journal of Polymers and the Environment, 2011, 19(1): 18-39. DOI:10.1007/s10924-010-0243-7 |
| [8] |
Apinya T, Sombatsompop N, Prapagdee B. Selection of a Pseudonocardia sp. RM423 that accelerates the biodegradation of poly(lactic) acid in submerged cultures and in soil microcosms[J]. International Biodeterioration & Biodegradation, 2015, 99: 23-30. |
| [9] |
Guo WB, Tao J, Yang C, et al. The rapid evaluation of material biodegradability using an improved ISO 14852 method with a microbial community[J]. Polymer Testing, 2010, 29(7): 832-839. DOI:10.1016/j.polymertesting.2010.07.004 |
| [10] |
Pranamuda H, Tokiwa Y, Tanaka H. Polylactide degradation by an Amycolatopsis sp.[J]. Applied and Environmental Microbiology, 1997, 63(4): 1637-1640. |
| [11] |
Sukhumaporn S, Shinji T, Vichien K. Development of fermentation process for PLA-degrading enzyme production by a new thermophilic Actinomadura sp. T16-1[J]. Biotechnology and Bioprocess Engineering, 2009, 14(3): 302-306. DOI:10.1007/s12257-008-0207-0 |
| [12] |
Karamanlioglu M, Houlden A, Robson GD, et al. Isolation and characterisation of fungal communities associated with degradation and growth on the surface of poly(lactic) acid (PLA) in soil and compost[J]. International Biodeterioration & Biodegradation, 2014, 95: 301-310. |
| [13] |
Luan C, You CF. A novel experimental investigation into sintered neck tensile strength of ash at high temperatures[J]. Powder Technology, 2015, 269: 379-384. DOI:10.1016/j.powtec.2014.09.031 |
| [14] |
Jin L, Gao XM, Du JH, et al. Peat bacterial diversity and community structure in Gahai Lake wetlandin Gan'nan[J]. Microbiology China, 2016, 43(11): 2396-2404. 靳亮, 高学梅, 杜建华, 等. 尕海湖湿地泥炭细菌多样性分析[J]. 微生物学通报, 2016, 43(11): 2396-2404. |
| [15] |
Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [16] |
Huang ZT, Song XF, Li X, et al. Analysis of microbial diversity of submerged biofilters in recirculating aquaculture system (RAS) for grouper (Epinehelus moara) based on high-throughput DNA sequencing[J]. Transactions of the Chinese Society of Agricultural Engineering, 2016, 32(S1): 242-247. 黄志涛, 宋协法, 李勋, 等. 基于高通量测序的石斑鱼循环水养殖生物滤池微生物群落分析[J]. 农业工程学报, 2016, 32(S1): 242-247. |
| [17] |
Zhang ZD, Gu MY, Wang W, et al. Analysis of bacterial community in radiation polluted soils by high-throughput sequencing[J]. Microbiology China, 2016, 43(6): 1218-1226. 张志东, 顾美英, 王玮, 等. 基于高通量测序的辐射污染区细菌群落特征分析[J]. 微生物学通报, 2016, 43(6): 1218-1226. |
| [18] |
Sun H, Terhonen E, Koskinen K, et al. Bacterial diversity and community structure along different peat soils in boreal forest[J]. Applied Soil Ecology, 2014, 74: 37-45. DOI:10.1016/j.apsoil.2013.09.010 |
| [19] |
Lin J, Zhou JW, Kang Z, et al. Isolation, identification of poly lactic acid degrading microorganisms and optimization of the degradation process[J]. Microbiology China, 2013, 40(9): 1560-1569. 林娟, 周景文, 康振, 等. 聚乳酸降解菌株筛选鉴定及降解过程优化[J]. 微生物学通报, 2013, 40(9): 1560-1569. |
| [20] |
Sangwan P, Wu DY. New insights into polylactide biodegradation from molecular ecological techniques[J]. Macromolecular Bioscience, 2008, 8(4): 304-315. DOI:10.1002/(ISSN)1616-5195 |
| [21] |
Sangwan P, Way C, Wu DY. New insight into biodegradation of polylactide (PLA)/Clay nanocomposites using molecular ecological techniques[J]. Macromolecular Bioscience, 2009, 9(7): 677-686. DOI:10.1002/mabi.v9:7 |
| [22] |
Marsh TL. Culture-independent microbial community analysis with terminal restriction fragment length polymorphism[J]. Methods in Enzymology, 2005, 397: 308-329. DOI:10.1016/S0076-6879(05)97018-3 |
| [23] |
Longieras A, Tanchette JB, Erre D, et al. Compostability of Poly(lactide): Degradation in an inert solid medium[J]. Journal of Polymers and the Environment, 2007, 15(3): 200-206. DOI:10.1007/s10924-007-0061-8 |
| [24] |
Sun DL, Jiang X, Wu QLL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79(19): 5962-5969. DOI:10.1128/AEM.01282-13 |
 2017, Vol. 44
2017, Vol. 44




