扩展功能
文章信息
- 莫婷, 刘马峰, 程安春
- MO Ting, LIU Ma-Feng, CHENG An-Chun
- 部分革兰氏阴性菌脂多糖运输蛋白LptD的结构及功能研究进展
- Structure and function of LptD in some Gram-negative bacteria
- 微生物学通报, 2017, 44(1): 217-224
- Microbiology China, 2017, 44(1): 217-224
- DOI: 10.13344/j.microbiol.china.160413
-
文章历史
- 收稿日期: 2016-05-23
- 接受日期: 2016-08-25
- 优先数字出版日期(www.cnki.net): 2016-09-20
2. 四川农业大学动物医学院 禽病防治中心 四川 成都 611130;
3. 动物疫病与人类健康四川省重点实验室 四川 成都 611130
2. Avian Disease Research Center, College of Veterinary Medicine, Sichuan Agricultural University, Chengdu, Sichuan 611130, China;
3. Key Laboratory of Animal Disease and Human Health of Sichuan Province, Chengdu, Sichuan 611130, China
脂多糖(Lipopolysaccharide)是革兰氏阴性菌外膜的主要组成成分,由核心多糖、O抗原链和类脂A三部分构成[1],其不仅在维持细菌外膜的稳定性方面具有重要作用,且脂多糖作为革兰氏阴性菌独有的一种强有力的免疫激活剂,可与宿主的Toll样受体4 (TLR4)和脂质结合蛋白MD2一起组成TLR4-MD2异源复合体,并激活宿主的炎症反应信号通路,所以脂多糖也是导致炎症和人体天然免疫反应的主要诱因[2-3]。脂多糖也被称为内毒素[4],在细菌生长过程中会形成细菌的固有保护屏障,保护一些由脂质介导的疏水性抗生素的入侵过程,并且也可以对同样有疏水性质的有机溶剂起到有效的耐受作用。自100多年前对脂多糖的研究开始以来,经过科学家们的探索,现有研究对细菌脂多糖的胞质内合成途径已较为深入[5],并由Ruiz等在20世纪初首次明确,脂多糖从胞质到外膜的输出过程和外膜上的组装过程,需要7个脂多糖运输蛋白(Lipopolysaccharide transport proteins,LptA-G)协同完成[6],这使得脂多糖的研究进入了新的阶段。随后有多篇文章通过研究证明定位于外膜上的LptD-LptE膜蛋白复合物参与了脂多糖运输的最后一步--输出脂多糖跨过外膜并将脂多糖插入外膜的外部小叶(External leaflet)中[7-9],并共同组成了经典的“桶与塞子”结构,直接影响了由脂多糖参与的多个生物学过程,包括细菌耐有机溶剂的性质、抵抗有脂质介导的疏水性药物的耐受性[10]及细胞膜的正确装配等性质[11]。
LptD (Lipopolysaccharide transport proteins D)这个命名首次出现是在脑炎奈瑟氏菌(Neisseria meningitidis)中,表示其为脂多糖运输蛋白[12]。在此(2004年)之前20年左右的研究中,LptD被认为影响了细菌膜的通透性而被称作Imp (Increased membrane permeability),或参与了有机溶剂耐受性而被称为OstA (Organic solvent tolerance A)[13-15]。后经研究学者们更深入的研究后发现LptD在细菌的整个生物进程中最重要的作用是其对脂多糖的运输功能[16-17],并开始从分子水平、晶体结构上进行了进一步研究。2014年,Qiao等首次将对LptD晶体结构的观察精确到了2.4 Å,使得对LptD分子水平上的研究技术有了质的飞跃[7]。目前对LptD蛋白结构及其功能的研究已有大量报道,但对脂多糖如何输出并装配到外膜的精确机制仍不很清楚,需要进一步研究[18]。因此本文就LptD蛋白的结构、功能研究进展及其装配过程做详细综述,为该通路上其它蛋白的研究和脂多糖装配至外膜的精确机制研究奠定基础。
1 脂多糖运输系统(Lpt)简介脂多糖正常转运至外膜发挥屏障作用和其它重要功能,需要Lpt系统7个不同的脂多糖运输蛋白(LptABCDEFG)协同完成。其中Lpt系统在细胞内膜转运上涉及3个蛋白LptB、LptF和LptG,且这3个蛋白共同构成一个ABC转运体复合物(ATP-binding cassette transporter complex)。其中的NBDs (Nucleotidebinding domains,核苷酸结合区) lptB在大肠杆菌的基因组中位于全基因组的72.0 min处,编码一个ABC蛋白,结构上呈完全折叠构型,具有NBDs蛋白典型的“L”构型,而且包含高度保守的Q-loop (Q回路),用于提供Mg2+和ABC转运蛋白特殊的结合基序,并利用该回路在其暴露的结合面形成一个凹槽[19],使LptB能顺利地与其所在的ABC转运体复合物中的跨膜区TMDs (Transmembrane domains,由LptF、LptG构成)构建一个针对LPS的信号传导通路;另有文献报道lptB邻近的基因缺乏编码内膜亚单位,也是该复合物(LptB2FG)形成的诱因之一[20-21]。跨膜区TMDs的组成成分LptF和LptG的研究较少,早前通常认为是革兰氏阴性菌必需的内膜蛋白,被命名为YjgP和YjgQ,直到Ruiz等在2008年对这两个蛋白进行生物信息学分析后,发现其与脂多糖的运输及一个特殊的ABC转运复合物形成密切相关后才将这两个蛋白正式更名为LptF (YjgP)和LptG (YjgQ)[22]。Ruiz等经研究指出LptF和LptG都是必需基因,且无法用一般缺失方法进行基因敲除,只能通过构建一个含有可诱导的反向LptFG操纵子的pBAD质粒干扰株来验证其在LPS运输中起到的作用;在成功构建该干扰株之后,他们发现这两个基因的干扰会引起LPS在内膜上的累积,并影响LptB的正常合成和装配,Daley等的研究中也得出了相似的结论[22-23],这进一步说明LptBFG确实作为一个ABC复合物锚定在内膜上,并参与了LPS的内膜输出过程。
Lpt系统除以上内膜蛋白外,还包括两个周质运输蛋白(LptA、LptC)及一对外膜蛋白复合物(LptD/E),其中LptA蛋白具有一个比较新颖的结构,包含了16个连续且反向平行的β链,并经整合成了一个独特的轻微扭转的β折叠结构[24],其与Lpt通路上和它其有相互作用的LptC和LptD蛋白的N端结构都具有显著相似性(图 1),但经过序列比对发现lptA和lptC并不具有高序列相似性,且已有研究表明LptC的C端结构域与LptA是相连的,这些发现是否预示着这两个蛋白的功能是否相似还未可知[25]。LptE则是一个形似“三明治”形状的脂蛋白,其有2个α螺旋和4个交叠的β折叠而成[7](图 1),尽管在不同的细菌中LptE并非保守基因,但其蛋白结构在多种革兰氏阴性菌中都极为相似,且在LptD/E的复合物中也具有显著的结构相似性[26]。

|
| 图 1 脂多糖运输蛋白(Lipopolysaccharide transport proteins) LptACDE的结构示意图 Figure 1 Schematic of the structure of lipopolysaccharide transport proteins LptACDE |
|
|
Lpt (Lipopolysaccharide transport)系统包括以上7个贯通细胞内膜至外膜的蛋白,且缺一不可,任意一个蛋白的缺失均会影响脂多糖的正常输出过程,进而影响脂多糖参与的革兰氏阴性菌致病性、有机溶剂耐受性、疏水性药物耐受性等过程[20, 24]。由于技术难度的限制,目前对LPS的内膜至外膜运输过程中的机制研究还较为浅显,但自2014年Qiao等成功获取LptD/E晶体以来[7],近两年对LptD蛋白的研究已较为深入,因此下文将对这个被科学家们称为脂多糖“命门”的蛋白LptD的研究进展做进一步介绍。
2 LptD的结构与装配 2.1 LptD的蛋白结构编码LptD蛋白的基因大小在多种革兰氏阴性菌中相差不大,片段大小均在2 000-3 000 bp左右[27-28]。其序列在不同的菌株中同源性较高。即使在亲缘关系较远的大肠杆菌(Escherichia coli)和鸭疫里默氏杆菌(Riemerella anatipestifer)中,同源性也有35.77%。这很有可能暗示LptD在不同的菌株中发挥相同的功能。研究显示,在大肠杆菌中,lptD基因位于大肠杆菌遗传图谱的约1.2 min处,位于ksgA基因下游、ara基因的上游,并与SurA的基因存在共转录现象[11]。在现有的研究中,Sampson等对大肠杆菌lptD突变株进行了遗传学分析,并认为lptD在大肠杆菌中是一个必需基因[13]。类似的,我们也试图敲除黄杆菌属的鸭疫里默氏杆菌的lptD基因,但至今并未成功,这很可能暗示lptD在鸭疫里默氏杆菌中也是必需基因。
起初科学家们对LptD的结构研究只能指出其为β桶状蛋白,并将其定位于外膜上[12, 29]。随着研究的深入,发现LptD广泛分布于外膜并与细胞外膜形成有关,且其具有一个高度保守的C端结构,使其能成功锚定在外膜上,也因此在多种细菌中LptD都具有相同或相似的生物学功能[30]。现有研究对LptD的描述已经精确到了2.4 Å,对其二级结构也有了较为深入的研究。在Qiao等[7]的研究中,他们指出LptD与大多数外膜蛋白一样都属于β桶状蛋白,但LptD的跨膜区是迄今所发现有最多β链所组成的(共26条),其也是尺寸最大的单体β桶状膜蛋白。它之所以在脂多糖的转运中起关键性作用,是因为LptD的26条β链中,第1条和第2条各存在的一个脯氨酸使其不能形成规则的二级结构,从而削弱了这两条β链和相邻β链之间的相互作用。因此,在LptD的第1至第26条β链间创造了一个宽度为16 Å的脂多糖出口,其大小可以允许脂多糖从细胞间质进入,并在其中进行侧向运输,在中途经过LptE的方向调整作用,LPS最终成功从LptD的C端结构域垂直输出,并装配到外膜表面上。Dong等也在同年(2014年)用X-ray技术成功解析了LptD的蛋白结构,进一步验证LptD是由26条反向平行的β折叠链构成的,并明确其为一个70 Å (1 Å=10-1 nm)长、50 Å宽、50 Å高的典型的跨膜型β桶状蛋白[31]。在LptD的整体蛋白结构上,Chng等也用β-巯基乙醇和SDS-Page的实验手段对LptD进行了蛋白分析,并发现LptD的N端和C端结构域通过两个不连续二硫键来维持稳态和功能的正常发挥[32]。
2.2 LptD的规范性装配LptD本身是较为典型的β桶状蛋白[30],所以LptD通常以β桶状蛋白的主要运输途径--SurA途径进行转运,在该途径中LptD先经由细胞内合成,然后由内膜输出系统(Sec系统)输出后进入细胞间质内,由间质分子伴侣SurA转运到细胞外膜附近[33]。在先前的研究中已证实大肠杆菌中SurA和LptD存在共转录现象,这进一步说明了SurA确实参与了LptD的正确装配[34]。Vertommen等通过构建surA的基因缺失株与野生株对比,发现LptD的含量有显著减少但依旧存在,于是作者大胆猜想认为SurA途径是LptD周质运输的主要途径但非唯一途径[33],该猜想也在两年后的报道中得到证实--当缺失SurA之后,LptD将会通过备用途径Skp/DegP通路完成细胞周质运输过程[35],但其具体机制尚不明确。在经过间质内运输后,未经折叠的LptD会被呈递给硫基氧化酶DsbA,届时LptD将作为DsbA的基质而存在,在该过程中DsbA对LptD共进行两次氧化,第一次氧化过程在细胞间质中完成--DsbA先与LptD结构域N端的C31半胱氨酸残基临时结合,再促进LptD的C31和C173半胱氨酸残基间形成一个临时的不连续二硫键。然后这个LptD中间体(未折叠结构)被BamA识别,并通过外膜β桶状蛋白的折叠和锚定Bam系统(包括BamABCDE)合成完整的LptD蛋白,LptD与LptE (有Lol系统完成装配)一起形成外膜LPS运输复合物LptD/E[36] (图 2)。第二次氧化过程则发生在LptD/E复合物形成后,这时LptD上C31和C173间的临时二硫键断开,N端上的C173残基与C725残基末端一起构成稳定的不连续二硫键,并在形成该二硫键后与DsbA再次临时结合,同时由二硫化物异构酶DsbC来纠正DsbA引进的外来二硫化物,使得在LptD蛋白的C31与C724间形成另一个稳定的不连续二硫键[27, 32],以维持LptDN端和C端结构域间的区域稳定,Chng等通过构建LptDD330-352和LptDD529-538的点突变株证实破坏这两个二硫键会导致LptD蛋白的形成缺陷[32]。
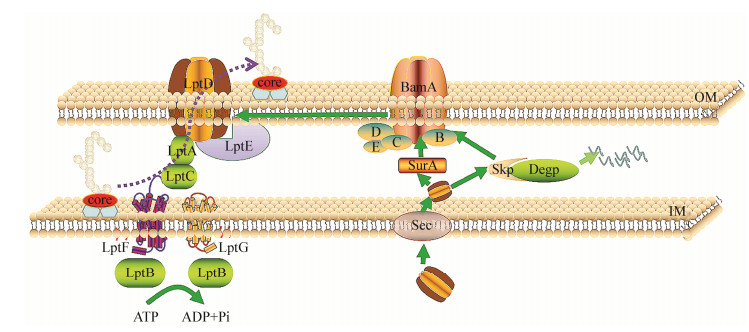
|
| 图 2 LptD的装配及Lpt系统运输LPS的过程模型 Figure 2 Schematic of the assembly mechanism of LptD and LPS |
|
|
正如上文所述,LPS的跨膜过程需要需要7个脂多糖运输蛋白(Lipopolysaccharide transport proteins,LptA-G)协同完成。现在的普遍观点认为Lpt系统的动力来自于各蛋白之间的相互作用--首先LPS经过膜内的一系列生化作用进行合成[37],然后由细胞内膜上的ABC转运体MabA将已完成翻转、合成好的LPS呈递给另一个ABC复合转运体LptB2FG[30, 38],这个复合物将LPS从内膜胞质面排出至内膜的细胞间质面,进而通过LptC-LptA的类桥梁结构,并将各自的表面与脂多糖的Lipid A一端接触,由于其本身携带有大量的负电荷,与邻近的分子间具有静电排斥力,所以其能沿LptA-LptC顺利地进行单向移动,再通过LptA与LptD间氨基端相似的水溶性周质结构[39],完成脂多糖从LptA到LptD运输过程,但Lpt系统的细胞间质内运输机制到目前为止还尚不清楚[40]。当LPS接触到LptD的N端结构后便会从LptD蛋白的β1-β26间的约16 Å宽的脂多糖出口进入LptD的内部,并在输出至LptD的C端结构时经LptE进行方向的调整,使其垂直于外膜,并沿着LptD的β1-β26进行侧向移动,然后由外膜上带正电的Mg2+帮助LPS完成在LptD蛋白中的侧向移动过程[18],最终成功装配到外膜上。
3.2 LptD的其他功能 3.2.1 LptD帮助细胞膜正确装配的功能: 研究表明在大肠杆菌中,LptD不仅参与细胞被膜的生成,还在细胞外膜上形成了一个包含高分子二硫键的复合物。Braun等通过构建imp (lptD)干扰株证实在大肠杆菌中lptD基因的表达降低可以引起膜蛋白错误定位、蛋白外渗、被膜密度升高等一些非正常生物特征的产生[11]。同样的情况在脑膜炎双球菌中也存在[12],这说明lptD的缺失或表达降低都会使脑膜炎奈瑟氏菌(Neisseria meningitides)细胞膜的生成受到影响。近年来,随着对LptD研究的逐渐深入,Weski等研究发现LptD的表达降低会导致新合成的构成膜上脂质和蛋白的密度降低,这暗示LptD可能与外膜上某些蛋白异构酶和脂质的合成有关[41],但具体机制尚不明确。 3.2.2 LptD参与细菌有机溶剂耐受性的功能: 有机溶剂对于所有的生物体而言都是有剧毒的,因为它们可以作用于生物细胞膜,从而影响细胞膜的完整性,使细胞正常功能被破坏,主要是膜通透性和细胞能量转换器的缺失;同时有机溶剂也会侵害细胞的代谢系统,造成生长抑制,最终死亡[42]。大多数革兰氏阴性菌具有许多革兰氏阳性菌所没有的有机溶剂耐受性,能在某些有毒的化合物中生长,这是因为革兰氏阴性菌多了一层外膜结构[43],膜上的一些蛋白对部分有机溶剂和疏水性抗生素的跨膜过程影响尤为明显。其中lptD就是这样的一个基因,由于其自身编码的蛋白具有改变膜通透性的功能,参与了革兰氏阴性菌有机溶剂耐受过程及细菌耐药过程,因此被命名为OstA (Organic solvent tolerance A)。 LptD/OstA在多种革兰氏阴性菌中都发挥了增强其有机溶剂耐受性的作用。大肠杆菌中,该基因的转录起始位点影响其对有机溶剂的耐受性水平[14]。Ohtsu等以大肠杆菌的JA300和OST4251作为研究对象(Aono等,1994年构建成功)的研究结果表明,JA300和OST4251对毒性较弱的有机溶剂二苯醚均有一定的抵抗作用,但在面对毒性较强的正己烷时,OST4251却表现为敏感性,而JA300仍有较强的耐受性。这是因为OST4251菌株在其lptD启动子上游有两个序列--IS2和IS5插入,其中IS2的插入,导致了lptD启动子的活性下降,进行Western blotting也可发现,在OST4251中LptD蛋白的含量明显少于JA300。作者推测参与大肠杆菌JA300株正己烷耐受性的决定性基因是OstA,果然在将OstA插入质粒构成重组质粒转入OST4251株后,发现后者被赋予了有机溶剂耐受性,说明OstA确实参与了大肠杆菌的有机溶剂耐受过程[34]。该发现最早出现于常见的大肠杆菌野生株K-12中[13]。另外,在我们研究与大肠杆菌亲缘关系较远的鸭疫里默氏杆菌时,发现其对疏水性抗菌药物戊二醛具有一定的抵抗作用,这暗示有某种物质参与其中,我们猜测这可能与LptD有关;在用荧光定量手段对戊二醛处理组(用感应浓度0.05%处理)和未处理组进行检测后发现,处理组的LptD表达量上调3倍,暗示其可能参与了鸭疫里默氏杆菌对戊二醛的耐受作用;我们进一步利用穿梭质粒在鸭疫里默氏杆菌中过表达LptD,发现该过表达株对戊二醛的抵抗作用强于野生株,这说明LptD在鸭疫里默氏杆菌对戊二醛的耐受作用中确实起到作用,其具体机制还需要进一步研究。除此之外,LptD还参与了幽门螺旋杆菌[44]、绿脓杆菌[45]等革兰氏阴性菌的有机溶剂耐受过程。 3.2.3 LptD参与细菌抗生素耐受的功能: 许多抗生素都要渗透细胞被膜,然后在细胞内的生物过程中发挥作用。早在1998年,科学家们就发现革兰氏阴性菌拥有独特的外膜结构,所以抗生素更难进入细胞内发挥作用[45]。当根据抗生素的水溶性进行分类后(分为亲水性和疏水性),抗生素通常是由两个途径穿过细胞外膜:亲水性抗生素一般通过孔蛋白透过外膜;疏水性抗生素则通过脂质介导的通路渗入磷脂双分子层而进入细胞,脂多糖在这个过程中常常作为细菌的固有屏障而发挥耐药作用[46]。LptD位于外膜,直接影响了LPS的输出情况,所以对抗生素的耐受性起着重要作用,相关研究表明这很可能是因为LPS中唯一的疏水基团类脂A的运输受阻造成。类脂A是细菌外膜层的重要组成部分,LPS的O抗原链和核心寡糖也必需通过类脂A与外膜的相互作用而粘附于细菌外膜[47]。但LptD在类脂A的运输过程中是否有特殊影响还尚不明确,所以透彻分析其功能对解决现在日益严重的抗生素耐药问题具有重要意义。Srinivas等就通过对自己建立的Protegrin I库中的抗菌素进行筛选,找到了一种针对假单胞菌的LptD蛋白的多肽,该研究指出,这类多肽药物能通过摧毁绿脓杆菌(Pseudomonas aeruginosa)的LptD来杀灭该菌,从而解决其多重耐药问题,这暗示利用抗生素来特异性定位基因的构想,未来很可能在抵抗不同类型的革兰氏阴性菌感染上发挥重要作用[48]。 4 展望在先前的研究中,LptD常被认为与革兰氏阴性菌的有机溶剂耐受性和抗生素耐药性相关,同样我们也发现鸭疫里默氏杆菌的LptD与其有机溶剂耐受性相关。总结早前与近年来的研究资料,研究者们发现LptD具有以上功能的关键很可能是因为其参与了脂多糖的输出,因此影响了由脂质介导的化合物的耐受性;另一方面,我们发现LptD的过表达可以明显地促进鸭疫里默氏杆菌的生长,但单独过表达其N端或C端并无此效果,这暗示LptD也许是作为信号因子而发挥作用。至今LptD促进细菌增殖的研究还尚未被报道,若其在鸭疫里默氏杆菌中确实作为生长信号因子存在,那么如何利用LptD切断该信号通路抑制细菌增殖将至关重要。另外,若明确LptD在耐有机溶剂和耐抗生素入侵的功能都源于其在脂多糖运输方面起的作用,那么就可以针对LptD设计特异性定位的药物来破坏脂多糖运输通路,由此解决抗生素耐药问题,现已有通过破坏细菌的脂多糖运输来解决超级细菌耐药问题的有关报道[48],这进一步说明对LptD的深入研究可帮助研究者更好地研发新型抗生素,并且更好地揭示脂多糖的正确装配机制。
| [1] | Li H, Liao TT, Debowski AW, et al. Lipopolysaccharide structure and biosynthesis in Helicobacter pylori[J]. Helicobacter 2016. DOI:10.1111/hel.12301 |
| [2] | Bryant CE, Spring DR, Gangloff M, et al. The molecular basis of the host response to lipopolysaccharide[J]. Nature Reviews Microbiology 2010, 8(1) : 8–14. |
| [3] | Meseguer V, Alpizar YA, Luis E, et al. TRPA1 channels mediate acute neurogenic inflammation and pain produced by bacterial endotoxins[J]. Nature Communications 2014, 5 : 3125. |
| [4] | Roh E, Lee HS, Kwak JA, et al. MD-2 as the target of nonlipid chalcone in the inhibition of endotoxin LPS-induced TLR4 activity[J]. Journal of Infectious Diseases 2011, 203(7) : 1012–1020. DOI:10.1093/infdis/jiq155 |
| [5] | Simpson BW, May JM, Sherman DJ, et al. Lipopolysaccharide transport to the cell surface: biosynthesis and extraction from the inner membrane[J]. Philosophical Transactions of the Royal Society Series B: Biological Sciences 2015, 370(1679) : 20150029. DOI:10.1098/rstb.2015.0029 |
| [6] | Ruiz N, Kahne D, Silhavy TJ. Transport of lipopolysaccharide across the cell envelope: the long road of discovery[J]. Nature Reviews Microbiology 2009, 7(9) : 677–683. DOI:10.1038/nrmicro2184 |
| [7] | Qiao S, Luo QS, Zhao Y, et al. Structural basis for lipopolysaccharide insertion in the bacterial outer membrane[J]. Nature 2014, 511(7507) : 108–111. DOI:10.1038/nature13484 |
| [8] | Freinkman E, Okuda S, Ruiz N, et al. Regulated assembly of the transenvelope protein complex required for lipopolysaccharide export[J]. Biochemistry 2012, 51(24) : 4800–4806. DOI:10.1021/bi300592c |
| [9] | Chng SS, Ruiz N, Chimalakonda G, et al. Characterization of the two-protein complex in Escherichia coliresponsible for lipopolysaccharide assembly at the outer membrane[J]. Proceedings of the National Academy of Sciences of the United States of America 2010, 107(12) : 5363–5368. DOI:10.1073/pnas.0912872107 |
| [10] | Delcour AH. Outer membrane permeability and antibiotic resistance[J]. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics 2009, 1794(5) : 808–816. DOI:10.1016/j.bbapap.2008.11.005 |
| [11] | Braun M, Silhavy TJ. Imp/OstA is required for cell envelope biogenesis in Escherichia coli[J]. Molecular Microbiology 2002, 45(5) : 1289–1302. DOI:10.1046/j.1365-2958.2002.03091.x |
| [12] | Bos MP, Tefsen B, Geurtsen J, et al. Identification of an outer membrane protein required for the transport of lipopolysaccharide to the bacterial cell surface[J]. Proceedings of the National Academy of Sciences of the United States of America 2004, 101(25) : 9417–9422. DOI:10.1073/pnas.0402340101 |
| [13] | Sampson BA, Misra R, Benson SA. Identification and characterization of a new gene of Escherichia coli K-12 involved in outer membrane permeability[J]. Genetics 1989, 122(3) : 491–501. |
| [14] | Abe S, Okutsu T, Nakajima H, et al. n-Hexane sensitivity of Escherichia coli due to low expression of imp/ostA encoding an 87 kDa minor protein associated with the outer membrane[J]. Microbiology 2003, 149(5) : 1265–1273. DOI:10.1099/mic.0.25927-0 |
| [15] | Chiu HC, Lin TL, Yang JC, et al. Synergistic effect of imp/ostA and msbA in hydrophobic drug resistance of Helicobacter pylori[J]. BMC Microbiology 2009, 9 : 136. DOI:10.1186/1471-2180-9-136 |
| [16] | Silhavy TJ, Kahne D, Walker S. The bacterial cell envelope[J]. Cold Spring Harbor Perspectives in Biology 2010, 2(5) : a000414. |
| [17] | Freinkman E, Chng SS, Kahne D. The complex that inserts lipopolysaccharide into the bacterial outer membrane forms a two-protein plug-and-barrel[J]. Proceedings of the National Academy of Sciences of the United States of America 2011, 108(6) : 2486–2491. DOI:10.1073/pnas.1015617108 |
| [18] | Putker F, Bos MP, Tommassen J. Transport of lipopolysaccharide to the Gram-negative bacterial cell surface[J]. FEMS Microbiology Reviews 2015, 39(6) : 985–1002. DOI:10.1093/femsre/fuv026 |
| [19] | Sherman DJ, Lazarus MB, Murphy L, et al. Decoupling catalytic activity from biological function of the ATPase that powers lipopolysaccharide transport[J]. Proceedings of the National Academy of Sciences of the United States of America 2014, 111(13) : 4982–4987. DOI:10.1073/pnas.1323516111 |
| [20] | Narita SI, Tokuda H. Biochemical characterization of an ABC transporter LptBFGC complex required for the outer membrane sorting of lipopolysaccharides[J]. FEBS Letters 2009, 583(13) : 2160–2164. DOI:10.1016/j.febslet.2009.05.051 |
| [21] | Wang ZS, Xiang QJ, Zhu XF, et al. Structural and functional studies of conserved nucleotide-binding protein LptB in lipopolysaccharide transport[J]. Biochemical and Biophysical Research Communications 2014, 452(3) : 443–449. DOI:10.1016/j.bbrc.2014.08.094 |
| [22] | Ruiz N, Gronenberg LS, Kahne D, et al. Identification of two inner-membrane proteins required for the transport of lipopolysaccharide to the outer membrane of Escherichia coli[J]. Proceedings of the National Academy of Sciences of the United States of America 2008, 105(14) : 5537–5542. DOI:10.1073/pnas.0801196105 |
| [23] | Daley DO, Rapp M, Granseth E, et al. Global topology analysis of the Escherichia coli inner membrane proteome[J]. Science 2010, 308(5726) : 1321–1323. |
| [24] | May JM, Sherman DJ, Simpson BW, et al. Lipopolysaccharide transport to the cell surface: periplasmic transport and assembly into the outer membrane[J]. Philosophical Transactions of the Royal Society B: Biological Sciences 2015, 370(1679) : 20150027. DOI:10.1098/rstb.2015.0027 |
| [25] | Sperandeo P, Villa R, Martorana AM, et al. New insights into the Lpt machinery for lipopolysaccharide transport to the cell surface: LptA-LptC interaction and LptA stability as sensors of a properly assembled transenvelope complex[J]. Journal of Bacteriology 2011, 193(5) : 1042–1053. DOI:10.1128/JB.01037-10 |
| [26] | Malojčić G, Andres D, Grabowicz M, et al. LptE binds to and alters the physical state of LPS to catalyze its assembly at the cell surface[J]. Proceedings of the National Academy of Sciences of the United States of America 2014, 111(26) : 9467–9472. DOI:10.1073/pnas.1402746111 |
| [27] | Denoncin K, Vertommen D, Paek E, et al. The protein-disulfide isomerase DsbC cooperates with SurA and DsbA in the assembly of the essential β-barrel protein LptD[J]. Journal of Biological Chemistry 2010, 285(38) : 29425–29433. DOI:10.1074/jbc.M110.119321 |
| [28] | Schmidt J, Patora-Komisarska K, Moehle K, et al. Structural studies of β-hairpin peptidomimetic antibiotics that target LptD in Pseudomonas sp.[J]. Bioorganic & Medicinal Chemistry 2013, 21(18) : 5806–5810. |
| [29] | Sperandeo P, Lau FK, Carpentieri A, et al. Functional analysis of the protein machinery required for transport of lipopolysaccharide to the outer membrane of Escherichia coli[J]. Journal of Bacteriology 2008, 190(13) : 4460–4469. DOI:10.1128/JB.00270-08 |
| [30] | Yuan B, Cheng AC, Wang MS. Polysaccharide export outer membrane proteins in Gram-negative bacteria[J]. Future Microbiology 2013, 8(4) : 525–535. DOI:10.2217/fmb.13.13 |
| [31] | Dong HH, Xiang QJ, Gu YH, et al. Structural basis for outer membrane lipopolysaccharide insertion[J]. Nature 2014, 511(7507) : 52–56. DOI:10.1038/nature13464 |
| [32] | Chng SS, Xue MY, Garner RA, et al. Disulfide rearrangement triggered by translocon assembly controls lipopolysaccharide export[J]. Science 2012, 337(6102) : 1665–1668. DOI:10.1126/science.1227215 |
| [33] | Vertommen D, Ruiz N, Leverrier P, et al. Characterization of the role of the Escherichia coli periplasmic chaperone SurA using differential proteomics[J]. Proteomics 2009, 9(9) : 2432–2443. DOI:10.1002/pmic.v9:9 |
| [34] | Ohtsu I, Kakuda N, Tsukagoshi N, et al. Transcriptional analysis of the ostA/imp gene involved in organic solvent sensitivity in Escherichia coli[J]. Bioscience, Biotechnology, and Biochemistry 2004, 68(2) : 458–461. DOI:10.1271/bbb.68.458 |
| [35] | Volokhina EB, Grijpstra J, Stork M, et al. Role of the periplasmic chaperones Skp, SurA, and DegQ in outer membrane protein biogenesis in Neisseria meningitidis[J]. Journal of Bacteriology 2011, 193(7) : 1612–1621. DOI:10.1128/JB.00532-10 |
| [36] | Chimalakonda G, Ruiz N, Chng SS, et al. Lipoprotein LptE is required for the assembly of LptD by the β-barrel assembly machine in the outer membrane of Escherichia coli[J]. Proceedings of the National Academy of Sciences of the United States of America 2011, 108(6) : 2492–2497. DOI:10.1073/pnas.1019089108 |
| [37] | Raetz CRH, Whitfield C. Lipopolysaccharide endotoxins[J]. Annual Review of Biochemistry 2002, 71(1) : 635–700. DOI:10.1146/annurev.biochem.71.110601.135414 |
| [38] | Woebking B, Reuter G, Shilling RA, et al. Drug-lipid a interactions on the Escherichia coli ABC transporter MsbA[J]. Journal of Bacteriology 2005, 187(18) : 6363–6369. DOI:10.1128/JB.187.18.6363-6369.2005 |
| [39] | Schwalm J, Mahoney TF, Soltes GR, et al. Role for Skp in LptD assembly in Escherichia coli[J]. Journal of Bacteriology 2013, 195(16) : 3734–3742. DOI:10.1128/JB.00431-13 |
| [40] | Tran AX, Dong CJ, Whitfield C. Structure and functional analysis of LptC, a conserved membrane protein involved in the lipopolysaccharide export pathway in Escherichia coli[J]. Journal of Biological Chemistry 2010, 285(43) : 33529–33539. DOI:10.1074/jbc.M110.144709 |
| [41] | Weski J, Ehrmann M. Genetic analysis of 15 protein folding factors and proteases of the Escherichia coli cell envelope[J]. Journal of Bacteriology 2012, 194(12) : 3225–3233. DOI:10.1128/JB.00221-12 |
| [42] | Torres S, Pandey A, Castro GR. Organic solvent adaptation of Gram positive bacteria: applications and biotechnological potentials[J]. Biotechnology Advances 2011, 29(4) : 442–452. DOI:10.1016/j.biotechadv.2011.04.002 |
| [43] | Rajagopal S, Sudarsan N, Nickerson KW. Sodium dodecyl sulfate hypersensitivity of clpP and clpB mutants of Escherichia coli[J]. Applied and Environmental Microbiology 2002, 68(8) : 4117–4121. DOI:10.1128/AEM.68.8.4117-4121.2002 |
| [44] | Chiu HC, Lin TL, Wang JT. Identification and characterization of an organic solvent tolerance gene in Helicobacter pylori[J]. Helicobacter 2007, 12(1) : 74–81. |
| [45] | Li XZ, Zhang L, Poole K. Role of the multidrug efflux systems of Pseudomonas aeruginosa in organic solvent tolerance[J]. Journal of Bacteriology 1998, 180(11) : 2987–2991. |
| [46] | Pagès JM, James CE, Winterhalter M. The porin and the permeating antibiotic: a selective diffusion barrier in Gram-negative bacteria[J]. Nature Reviews Microbiology 2008, 6(12) : 893–903. DOI:10.1038/nrmicro1994 |
| [47] | Raetz CRH, Reynolds CM, Trent MS, et al. Lipid a modification systems in gram-negative bacteria[J]. Annual Review of Biochemistry 2007, 76(1) : 295–329. DOI:10.1146/annurev.biochem.76.010307.145803 |
| [48] | Srinivas N, Jetter P, Ueberbacher BJ, et al. Peptidomimetic antibiotics target outer-membrane biogenesis in Pseudomonas aeruginosa[J]. Science 2010, 327(5968) : 1010–1013. DOI:10.1126/science.1182749 |
 2017, Vol. 44
2017, Vol. 44