扩展功能
文章信息
- 徐巧林, 吴文良, 赵桂慎, 朱燕云, 郭岩彬
- XU Qiao-Lin, WU Wen-Liang, ZHAO Gui-Shen, ZHU Yan-Yun, GUO Yan-Bin
- 微生物硒代谢机制研究进展
- Selenium metabolism in microorganisms
- 微生物学通报, 2017, 44(1): 207-216
- Microbiology China, 2017, 44(1): 207-216
- DOI: 10.13344/j.microbiol.china.160380
-
文章历史
- 收稿日期: 2016-05-12
- 接受日期: 2016-09-28
- 优先数字出版日期(www.cnki.net): 2016-09-30
硒元素(Se)由瑞典化学之父Berzelius于1817年在硫酸矿尾中发现[1],而关于硒的最早记录可以追溯到13世纪马可波罗在游记中所提到的马蹄病[2]。硒是人和动物生命代谢所必需的微量元素,也是25种人体含硒蛋白和酶的重要组成成分[3-4]。在自然界中,硒主要以四种价态存在:硒酸盐SeO42-(+6)、亚硒酸盐SeO32-(+4)、单质硒Se0(0)和硒化物Se2-(-2)[5]。其中,硒氧化物(包括SeO42-和SeO32-)都具有高度水溶性、生物累积性和较强的毒性[6],不仅会对机体造成伤害,还会引起严重的环境污染。
微生物在硒的地球化学循环中发挥着重要的作用,而且在硒污染修复方面具有广阔的应用前景。研究发现许多微生物可以耐受高浓度的SeO42-和SeO32-,并将其转化为不溶性低毒的红色纳米硒(selenium nanoparticles,SeNPs)[7-10],或同化还原为硒蛋白,或甲基化为具有高挥发性的硒甲基硒化物[11-13],有些微生物还可以将低价态的硒氧化为高价态的硒[14-18]。因此,微生物对硒形态转化是有效解决硒引起的环境和健康问题的有效手段之一。
1 硒循环硒的地球化学循环是一个复杂的过程[19]。硒不仅有多种价态:Se (-2)、Se (0)、Se (+4)、Se (+6)[20],而且有不同的物理(固态、液态、气态)、化学(有机、无机)形态;还有5种稳定的同位素:74Se、76Se、77Se、78Se和80Se,其中78Se和80Se是最常见的[21]。
不同种类的硒在环境中的分布也不同,这取决于氧化还原条件。硒酸盐(SeO42-)和亚硒酸盐(SeO32-)主要存在于好氧环境,两者具有高度的可溶性,毒性也很高[22-23]。单质硒(Se0)大多存在于厌氧环境,由于在自然环境中的溶解度低,一般认为是不可利用的[24],但是胶状单质纳米硒在自然环境中是可以转运的,也可以被水生生物所利用[25]。
在还原条件下,单质硒可以进一步还原为硒化物,硒化物可以结合金属形成金属硒化物,通过沉积循环成为矿石和沉积物;硒化物也可以结合有机物形成有机硒化物,如硒代蛋氨酸和硒代半胱氨酸,这些物质在生物体内进一步参与合成硒蛋白;硒化物的另外一种存在形态是甲基化的硒,如二甲基硒化物(DMSe)和二甲基联硒化物(DMDSe),这些硒化物又可以以挥发态的形式返回到空气中[26]。另外,硒化物还可以形成挥发性强和毒性高的硒化氢(H2Se),它是H2S的类似物。
在自然界中,物理、化学过程都可以调控硒的转化,但越来越多的证据表明微生物在硒的循环中扮演了关键角色,它可以通过多种方式代谢硒[25]。因此,微生物调控硒转化的生物过程研究也越来越受到关注。
2 微生物硒代谢机制微生物对硒的代谢主要包括硒的转运、还原、氧化、同化、甲基化等。总体而言,微生物硒代谢的研究以硒的同化和还原机制较为清楚,在真菌和细菌(包括古生菌)中硒的同化机制均已阐明;在革兰氏阴性菌和阳性菌中也都发现了专一性的硒酸盐还原酶,但目前尚未发现专一性的亚硒酸盐还原酶或其他亚硒酸盐还原关键基因。硒的转运与硫酸盐或磷酸盐转运系统有关,同时还会受到温度、pH、碳源、氮源等因素的影响。硒的甲基化也发现了一些关键基因和酶;而对于硒氧化的微生物了解甚少,截止目前还缺乏对微生物硒氧化机制的系统研究。
2.1 硒的转运硒元素与硫元素化学性质相似,微生物对硒的转运与硫具有类似的转运机制。Sirko等发现在大肠杆菌(Escherichia coli)中硒酸盐通过硫酸盐ABC转运通透酶系统(CysAWTP)进入细胞[27]。但硫酸盐通透酶不是亚硒酸盐进入细胞的唯一通道,研究发现抑制硫酸盐通透酶的表达,并不会完全抑制亚硒酸盐吸收[12]。目前还发现GutS、SmoK和DedA等蛋白也参与亚硒酸盐向细胞内的转运[28-30]。
在真菌对硒的吸收转运研究中,酵母的研究较为清楚。在酵母细胞中,硒酸盐或亚硒酸盐的转运不仅受温度、pH、碳源种类的影响,而且与磷(P)、硫(S)两种元素有关。在酿酒酵母(Saccharomyces cerevisiae)中发现硫酸盐转运蛋白(Sul1p和Sul2p)和硫酸盐通透酶(Sul1和Sul2)都与亚硒酸盐的转运有关,其机制与大肠杆菌(E. coli)相似[31]。Lazard等发现在酵母细胞的培养基中,分别加入高浓度或低浓度磷酸盐,亚硒酸盐的转运则分别由2个高亲和性的(Pho84p,Pho89p)和3个低亲和性的(Pho87p,Pho90p,Pho91p)磷酸盐转运蛋白控制[32]。McDermott等发现酵母细胞中的一元羧酸同向转运体(Jen1p)也与亚硒酸盐的转运有关[33]。
微生物对硒的吸收转运往往会受到多重因素的影响,除了微生物本身的遗传差异外,还与其培养条件和环境干扰有关联。
2.2 硒的还原近十年的研究表明,可以进行硒还原的微生物种类繁多,已报道的至少有15个属的细菌,它们属于变形菌门(Proteobacteria)和厚壁菌门(Firmicutes)[34];但是对硒矿中硒还原细菌的群落分析时发现,硒还原细菌不仅属于变形菌门(Proteobacteria)和厚壁菌门(Firmicutes),还包括放线菌门(Actinobacteria)[35];这些微生物在生理学特性上具有较大差异,呈现丰富的多样性。
2.2.1 硒酸盐的还原: 很多微生物在无氧条件下均可以利用SeO42-作为最终电子受体进行呼吸作用,将SeO42-还原为SeO32-,最终还原为不溶性单质硒(Se0),这两个过程可以用两个公式表示[36]:| $\text{SeO}_{\text{4}}^{\text{2-}}\text{+2}{{\text{e}}^{-}}\text{+2}{{\text{H}}^{+}}\to \text{SeO}_{\text{3}}^{\text{2-}}\text{+}{{\text{H}}_{\text{2}}}\text{O}$ | (1) |
| $\text{SeO}_{3}^{\text{2-}}\text{+4}{{\text{e}}^{-}}\text{+6}{{\text{H}}^{+}}\to \text{S}{{\text{e}}^{\text{0}}}\text{+3}{{\text{H}}_{\text{2}}}\text{O}$ | (2) |
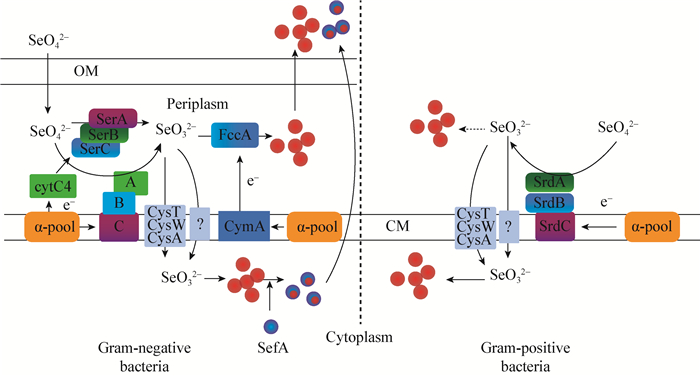
|
| 图 1 微生物对硒氧化物的还原模式图[41] Figure 1 Schematic diagram showing the microbial reduction of selenium oxyanions[41] 注:CM:细胞质膜;OM:外膜; SefA:硒因子A蛋白;SerABC:硒酸盐还原酶;SrdBCA:硒酸盐还原酶;cytC4:细胞色素C4;CymA:c-型细胞色素;FccA:延胡索酸还原酶;CysTWA:硫酸盐通透酶;Q-pool:醌池. Note: CM: Cytoplasmic membrane; OM: Outer membrane; SefA: Selenium factor A protein; SerABC: Selenate reductase; SrdBCA: Selenate reductase; cytC4: Cytochrome C4; CymA: C-type cytochrome; FccA: Fumarate reductase; CysTWA: Sulphate permease; Q-pool: Quinone pool. |
|
|

|
| 图 2 亚硒酸盐在细菌(A)[50]和酵母(B)细胞中的还原途径[54] Figure 2 Pathway of reduction of selenium in bacteria (A)[50] and yeast cells (B)[54] 注:GSH:谷胱甘肽;GR:胱甘肽还原酶;TR:硫氧还蛋白还原酶;a:ATP-硫酸化酶;b:APSe-激酶;c:PAPSe-还原酶;d:硫酸盐还原酶. Note: GSH: Glutathione; GR: Glutathione reductase; TR: Thioredoxin reductase; a: ATP Sulfurylase; b: APSe kinase; c: PAPSe reductase; d: Sulfate reductase. |
|
|
细菌、真菌和酵母均可以进行硒的生物甲基化,这些微生物在含有硒酸盐或亚硒酸盐的培养基中生长时会产生一种大蒜气味,这种气味就是由甲基化的硒产生。常见的甲基硒化物为二甲基硒化物(DMSe)和二甲基联硒化物(DMDSe),两者中的硒均为完全还原价态(Se2-)[5]。
近几年的研究表明,可以进行硒的甲基化的细菌种类繁多,包括假单胞菌属(Pseudomonas sp.)、气单胞菌属(Aeromonas sp.)、棒状杆菌属(Corynebacterium sp.)、脱硫弧菌属(Desulfovibrio sp.)、荚膜红细菌(R. capsulatus)、深红红螺菌(R. rubrum)以及弗氏柠檬酸杆菌(Citrobacter freundii KS8)、梭状芽胞杆菌(Clostridium collagenovorans)等;真菌主要包括头孢霉属(Cephalosporium sp.)、镰刀菌属(Fusarium sp.)、青霉菌属(Penicillium sp.)以及链状支顶孢菌(Acremonium falciforme)、互隔交链孢菌(Alternaria alternata)、桔青霉(Penicillium citrinum)、短柄帚霉(Scopulariopsis brevicaulis)等;古细菌主要有甲烷杆菌(Methanobacterium formicicum)和巴氏甲烷八叠球菌(Methanosarcina barkeri)[70]。
在原核生物中,硒的甲基化需要甲基转移酶的参与[71]。Ranjard等在假单胞杆菌(Pseudomonas)中发现了两种不同的甲基转移酶:一种是细菌的巯基嘌呤甲基转移酶(bTPMT),另一种是烯二炔类抗生素(calichaemicin)甲基转移酶的同源物,均可将亚硒酸盐和硒代半胱氨酸转化为二甲基硒醚和二甲基联硒化物。后续的研究又发现甲基转移酶基团--MmtA也参与了硒甲基化,并且在许多细菌中可以找到它的同源基因[72-73]。纤细红螺菌(Rhodocyclus tenuis)和深红红螺菌(R. rubrum)可以在光能自养的过程中,分别利用亚硒酸盐和硒酸盐产生甲基硒[13]。谷胱甘肽与亚硒酸盐反应生成的GS-Se-SG,还可以进一步与S-腺苷甲硫氨酸(SAM)反应生成甲烷硒醇和二甲基硒醚[74]。另外,硒代半胱氨酸也可以与谷胱甘肽反应还原为硒化氢[75]。此外,大肠杆菌(E. coli)中的氧硫族解毒蛋白TehB也与硒的甲基化有关[76],丁香假单胞菌(Pseudomonas syringae)的三甲基嘌呤甲基化酶基因(tmp)与硒酸盐、亚硒酸盐和硒代半胱氨酸的甲基化都有关[77]。
2.4 硒氨基酸和硒蛋白的合成硒在微生物细胞中可以被同化为硒氨基酸,包括硒代蛋氨酸(Se-Met)、硒代胱氨酸(SeCys2)和硒代半胱氨酸(Sec,U),并进一步参与硒蛋白的合成。在硒代蛋氨酸、硒代胱氨酸和硒代半胱氨酸中硒与氨基酸通过共价键结合。硒代半胱氨酸是第21种氨基酸[78],也是唯一不能循环利用再合成硒蛋白的氨基酸,在硒代半胱氨酸裂解酶(Lyase)的作用下被完全降解转化为还原态的硒[79],其合成主要与sel A、sel B、sel C、sel D四个基因的产物直接相关[5]。
SelC是一个特异性的硒代半胱氨酸tRNASec,在RNA翻译时,tRNASec首先被氨酰化,此过程由seryl-tRNA合成酶(SerRS)催化完成,形成的Ser-tRNASec为硒代半胱氨酸的形成提供了基本的碳骨架[77]。在大肠杆菌中(图 3A),硒代磷酸盐合成酶(SelD)可以将还原态的硒([Se])转化为硒代磷酸盐(Se-P)。Ser-tRNASec在SelA (硒代半胱氨酸合成酶)作用下进一步转化为Sec-tRNASec[80],此过程的活性硒由硒代磷酸盐(Se-P)提供。而在古细菌(例如Methanosarcina maripaludis)中(图 3B),Ser-tRNASec的合成由O-phosphoseryl-tRNASec激酶(PSTK)完成,活性硒由SPS激活的Se-P提供[81]。硒代半胱氨酸延长因子(SelB)通过mRNA的特殊二级结构元素(SECIS)识别密码子UGA来编码硒代半胱氨酸[82-83]。在酵母细胞中,Sec-tRNASec复合体在Secp43因子的作用下被转运到核糖体中,在SBP2等结合蛋白以及eEFSec延长因子的作用下,进一步完成硒代半胱氨酸的合成[80]。

|
| 图 3 硒代半胱氨酸生物合成及插入多肽链机制[79] Figure 3 Mechanism of Selenocysteine and peptides biosynthesis[79] 注:A:E. coli;B:M. maripaludis. 3′-UTR:3′-非编码区;PSTK:Seryl-tRNAsec激酶;[Se]:还原态硒形态;SelA:硒代半胱氨酸合酶;SelB:硒半胱氨酸专用延长因子;SelD、SPS:硒代磷酸盐合成酶;SepSecS:O-phosphoseryl-Trna;Sec:硒代半胱氨酸合酶;Ser:丝氨酸;SerRS:Seryl-tRNA合成酶;Se-P:硒代磷酸盐;虚线代表未知机理. Note: A: E. coli; B: M. maripaludis. 3′-UTR:3′-untranslated region; PSTK: seryl-tRNAsec kinase; [Se]: reduced Se-species; SelA: selenocysteine synthase; SelB: selenocysteine-specific elongation factor; SelD/SPS: Selenophosphate synthetase; SepSecS: O-phosphoseryl-tRNA; Sec: selenocysteine synthase; Ser: serine; SerRS: seryl-tRNA synthetase; Se-P: seleno (mono) phosphate; The dashed arrow indicates an unknown recoding mechanism. |
|
|
另外,在古细菌和真核生物中,硒代半胱氨酸插入序列(SECIS)位于mRNA的3′端-非编码区,而原核生物中的SECIS位于mRNA的开放阅读区(3′-UTR) UGA密码子的上游。
2.5 硒的氧化Lipman和Waksman于1923年首次报道了硒氧化细菌,他们在新鲜土壤中加入单质硒,发现单质硒被氧化,土壤酸度增加,当把这些土壤加入以单质硒作为唯一能量来源的培养基中,发现培养基中有棒状细菌的生长[14],在此之后,有关硒氧化的微生物却少有报道。因此,在1964年Shirft刚提出硒的微生物地球化学循环时,关于微生物对硒的氧化作用存在较大分歧[84]。
后来,Torma等于1972年发现了可以将铜硒化物中硒(Se2-)氧化为元素硒(Se0)的T. ferrooxidan菌[15]。Sarathchandra和Watkinson也报道了巨大芽胞杆菌(Bacillus megatherium)可以将单质硒氧化成亚硒酸盐或硒酸盐,主要产物为亚硒酸盐[16]。1998年,Dowdle和Oremland将土壤污泥高压蒸气灭菌或者添加代谢抑制剂(如:福尔马林、抗生素、叠氮化物、2, 4-二硝基苯酚)后,发现亚硒酸盐和硒酸盐的产量减少,但是当加入醋酸盐、葡萄糖或硫化物则提高单质硒的氧化,说明参与硒氧化的包括化能异养细菌和化能自养的硫杆菌[17]。几乎在同时,Losi和Frankenberger也报道了氧化单质硒的化能自养细菌。但仍然缺乏这些细菌的全基因组序列和硒氧化规律、相关基因的研究[18]。直到2016年,Wang等才报道了一株硒氧化细菌的全基因序列,Caulobacter ibrioides T5M6为革兰氏阴性细菌,分离于湖北恩施根围土壤样品,该细菌可以将化学单质硒(Se0)和含硒矿物(硒混合物)转化为亚硒酸盐(SeO32-)[85]。这些研究都证明微生物可以进行硒的氧化作用,也说明了微生物对硒的生物地球化学循环的重要作用。
有研究显示硒的氧化过程比还原过程慢3-4个数量级[17],这可能是阻碍硒氧化微生物研究的一个重要原因。目前,几乎没有关于古细菌和真菌对硒氧化的报道,也没有关于硒氧化细菌的系统报道,同时缺乏硒氧化分子机制的研究。
3 小结与展望本文从微生物对硒的转运、还原、甲基化、同化及氧化五个方面综述了微生物对硒的生物地球化学循环的重要作用。近年来,虽然硒的研究逐渐成为人们研究的热点,但仍然有很多问题有待解决,未来需要对以下几个方面做进一步研究:
(1)微生物对硒的全球地化循环的贡献,特别是在水圈、大气圈、土壤圈、岩石圈和生物圈:硒的全球地化循环中到底是生物过程占主导还是非生物过程占主导?微生物对全球硒的分布和硒在环境中的迁移有怎样的影响?微生物对硒的全球地化循环的贡献率是多少?
(2)对硒的转运研究发现,在细菌和真菌中,硒的转运与硫的转运机制相似,在古菌中是否也存在专一性的硒转运酶系统?对硒的甲基化研究中,已经在原核生物中找到了硒甲基化的关键基因和酶,但在真核生物中硒的甲基化机理尚不清楚,其是否与原核生物具有相似性,又或是具有其独特性?对硒的氧化研究相对最少,后续不仅要丰富硒氧化微生物资源及全基因组序列,还需进一步研究硒的氧化规律及其机制。
(3)微生物在自然界中的自解毒机理:在自然界中微生物将硒酸盐或亚硒酸盐还原为单质硒,毒性下降,是否与其自解毒机制有关?是否是其长期进化适应环境的结果?其微观和宏观生态机理又是什么?
(4)单质硒合成机制:亚硒酸盐被还原为单质硒后,单质硒是如何聚集为纳米硒颗粒的?如果纳米硒在细胞内聚集完成,又是如何被转运到细胞外的?
| [1] | Berzelius JJ, Lettre de M, Berzelius a M. Berthollet surdeux metaux nouveaux[J]. Annales de Chimie et de Physique 1817, 7 : 199–207. |
| [2] | Oldfield JE. Historical perspectives on selenium[J]. Nutrition Today 2001, 36(2) : 100. DOI:10.1097/00017285-200103000-00017 |
| [3] | Rayman MP. The importance of selenium to human health[J]. Lancet 2006, 356(9225) : 233–241. |
| [4] | Zhang JS, Spallholz JE. Toxicity of selenium compounds and nanoselenium particles[A]//General, Applied and Systems Toxicology[M]. New York: John Wiley & Sons, 2011: 787-802 |
| [5] | Stolz JF, Basu P, Santini JM, et al. Arsenic and selenium in microbial metabolism[J]. The Annual Review of Microbiology 2006, 60 : 107–130. DOI:10.1146/annurev.micro.60.080805.142053 |
| [6] | Stolz JF, Oremland RS. Bacteria respiration of arsenic and selenium[J]. FEMS Microbiology Reviews 1999, 23(5) : 615–627. DOI:10.1111/j.1574-6976.1999.tb00416.x |
| [7] | Ike M, Takahash K, Fujita T, et al. Selenate reduction by bacteria isolated from aquatic environment free from selenium contamination[J]. Water Research 2000, 34(11) : 3019–3025. DOI:10.1016/S0043-1354(00)00041-5 |
| [8] | Tomei FA, Barton LL, Lemanski CL, et al. Transformation of selenate and selenite to elemental selenium by Desulfovibrio desulfuricans[J]. Journal of Industrial Microbiology 1995, 14(3/4) : 329–336. |
| [9] | Li BZ, Liu N, Li YQ, et al. Reduction of selenite to red elemental selenium by Rhodopseudomonas palustris strain N[J]. PLoS One 2014, 9(4) : e95955. DOI:10.1371/journal.pone.0095955 |
| [10] | Lampis S, Zonaro E, Bertolini C, et al. Delayed formation of zero-valent selenium nanoparticles by Bacillus mycoides SeITE01 as a consequence of selenite reduction under aerobic conditions[J]. Microbial Cell Factories 2014, 13 : 35. DOI:10.1186/1475-2859-13-35 |
| [11] | Zehr JP, Oremland RS. Reduction of selenate to selenide by sulfate-reducing bacteria: experiments with cell suspensions and estuarine sediments[J]. Applied and Environmental Microbiology 1987, 53(6) : 1365–1369. |
| [12] | Turner RJ, Weiner JH, Taylor DE. Selenium metabolism in Escherichia coli[J]. BioMetals 1998, 11(3) : 223–227. DOI:10.1023/A:1009290213301 |
| [13] | McCarty S, Chasteen T, Marshall M, et al. Phototrophic bacteria produce volatile, methylated sulfur and selenium compounds[J]. FEMS Microbiology Letters 1993, 112(1) : 93–98. DOI:10.1111/fml.1993.112.issue-1 |
| [14] | Lipman JG, Waksman SA. The oxidation of selenium by a new group of autotrophic microorganisms[J]. Science 1923, 57(1463) : 60. DOI:10.1126/science.57.1463.60 |
| [15] | Torma AE, Habashi F. Oxidation of copper (Ⅱ) selenide by Thiobacillus ferrooxidans[J]. Canadian Journal of Microbiology 1972, 18(11) : 1780–1781. DOI:10.1139/m72-278 |
| [16] | Sarathchandra SU, Watkinson JH. Oxidation of elemental selenium to selenite by Bacillus megaterium[J]. Science 1981, 211(4482) : 600–601. DOI:10.1126/science.6779378 |
| [17] | Dowdle PR, Oremland RS. Microbial oxidation of elemental selenium in soil slurries and bacterial cultures[J]. Environmental Science & Technology 1998, 32(23) : 3749–3755. |
| [18] | Losi ME, Frankenberger WT Jr. Microbial oxidation and solubilization of precipitated elemental selenium in soil[J]. Journal of Environmental Quality 1998, 27(4) : 836–843. |
| [19] | Winkel LHE, Annette JC, Lenz M, et al. Environmental selenium research: from microscopic processes to global understanding[J]. Environmental Science & Technology 2012, 46(2) : 571–579. |
| [20] | Fernández-Martínez A, Charlet L. Selenium environmental cycling and bioavailability: a structural chemist point of view[J]. Reviews in Environmental Science and Bio/Technology 2009, 8(1) : 81–110. DOI:10.1007/s11157-009-9145-3 |
| [21] | Boyd R. Selenium stories[J]. Nature Chemistry 2011, 3(7) : 570. DOI:10.1038/nchem.1076 |
| [22] | Barceloux DG. Selenium[J]. Clinical Toxicology 1999, 37(2) : 147–172. |
| [23] | Wu L. Review of 15 years of research on ecotoxicology and remediation of land contaminated by agricultural drainage sediment rich in selenium[J]. Ecotoxicology and Environmental Safety 2004, 57(3) : 257–269. DOI:10.1016/S0147-6513(03)00064-2 |
| [24] | Minaev VS, Timoshenkov SP, Kalugin VV. Structural and phase transformations in condensed selenium[J]. Journal of Optoelectronics and Advanced Materials 2005, 7(4) : 1717–1741. |
| [25] | Nancharaiah YV, Lens PNL. Ecology and Biotechnology of Selenium-Respiring Bacteria[J]. Microbiology and Molecular Biology Reviews 2015, 79(1) : 61–80. DOI:10.1128/MMBR.00037-14 |
| [26] | Markus L, Lens PNL. The essential toxin: the changing perception of selenium in environmental sciences[J]. Science of the Total Environment 2009, 407(12) : 3620–3633. DOI:10.1016/j.scitotenv.2008.07.056 |
| [27] | Sirko A, Hryniewicz M, Hulanicka D, et al. Sulfate and thiosulfate transport in Escherichia coli K-12: nucleotide sequence and expression of the cys TWAM gene cluster[J]. Journal of Bacteriology 1990, 172(6) : 3351–3357. DOI:10.1128/jb.172.6.3351-3357.1990 |
| [28] | Guzzo J, Dubow MS. A novel selenite-and tellurite-inducible gene in Escherichia coli[J]. Applied and Environmental Microbiology 2000, 66(11) : 4972–4978. DOI:10.1128/AEM.66.11.4972-4978.2000 |
| [29] | Bebien M, Chauvin JP, Adriano JM, et al. Effect of selenite on growth and protein synthesis in the phototrophic bacteria Rhodobacter sphaeroides[J]. Applied and Environmental Microbiology 2001, 67(10) : 4440–4447. DOI:10.1128/AEM.67.10.4440-4447.2001 |
| [30] | Ledgham F, Quest B, Vallaeys T, et al. A probable link between the DedA protein and resistance to selenite[J]. Research in Microbiology 2005, 156(3) : 367–374. DOI:10.1016/j.resmic.2004.11.003 |
| [31] | Mapelli V, Hillestr m PR, Patil K, et al. The interplay between sulphur and selenium metabolism influences the intracellular redox balance in Saccharomyces cerevisiae[J]. FEMS Yeast Research 2012, 12(1) : 20–32. DOI:10.1111/fyr.2012.12.issue-1 |
| [32] | Lazard M, Blanquet S, Fisicaro P, et al. Uptake of selenite by Saccharomyces cerevisiae involves the high and low affinity orthophosphate transporters[J]. The Journal of Biological Chemistry 2010, 285(42) : 32029–32037. DOI:10.1074/jbc.M110.139865 |
| [33] | McDermott JR, Rosen BP, Liu ZJ. Jen1p: a high affinity selenite transporter in yeast[J]. Molecular Biology of the Cell 2010, 21(22) : 3934–3941. DOI:10.1091/mbc.E10-06-0513 |
| [34] | Zheng SX, Su J, Wang R, et al. Metabolism of selenium in microorganisms[J]. Journal of Huazhong Agricultural University 2013, 32(5) : 1–8. (in Chinese) 郑世学, 粟静, 王瑞, 等. 硒是双刃剑? --谈微生物中的硒代谢[J]. 华中农业大学学报 2013, 32(5) : 1–8. |
| [35] | Wang HY. Isolation and identification of selenium-tolerance bacteria[D]. Beijing: Master's Thesis of China Agricultural University, 2015 (in Chinese) 王浩阳.耐硒细菌的筛选与鉴定[D].北京:中国农业大学硕士学位论文, 2015 |
| [36] | Debieuxa CM, Dridgea EJ, Mueller CM, et al. A bacterial process for selenium nanosphere assembly[J]. Proceedings of the National Academy of Sciences of the United States of America 2011, 108(33) : 13480–13485. DOI:10.1073/pnas.1105959108 |
| [37] | Macy JM, Rech S, Auling G, et al. Thauera selenatis gen. nov., sp. nov., a member of the beta subclass of proteobacteria with a novel type of anaerobic respiration[J]. International Journal of Systematic and Evolutionary Microbiology 1993, 43(1) : 135–142. |
| [38] | Butler CS, Debieux CM, Dridge EJ, et al. Biomineralization of selenium by the selenate-respiring bacterium Thauera selenatis[J]. Biochemical Society Transactions 2012, 40(6) : 1239–1243. DOI:10.1042/BST20120087 |
| [39] | Schr der I, Rech S, Krafft T, et al. Purification and characterization of the selenate reductase from Thauera selenatis[J]. The Journal of Biological Chemistry 1997, 272(38) : 23765–23768. DOI:10.1074/jbc.272.38.23765 |
| [40] | Krafft T, Bowen A, Theis F, et al. Cloning and sequencing of the genes encoding the periplasmic-cytochrome B -containing selenate reductase of Thauera selenatis[J]. DNA Sequence 2000, 10(6) : 365–377. DOI:10.3109/10425170009015604 |
| [41] | Yarlagadda VN, Piet NLL. Selenium biomineralization for biotechnological applications[J]. Trends in Biotechnology 2015, 33(6) : 323–330. DOI:10.1016/j.tibtech.2015.03.004 |
| [42] | Ridley H, Watts CA, Richardson DJ, et al. Resolution of distinct membrane-bound enzymes from Enterobacter cloacae SLD1a-1 that are responsible for selective reduction of nitrate and selenate oxyanions[J]. Applied and Environmental Microbiology 2006, 72(8) : 5173–5180. DOI:10.1128/AEM.00568-06 |
| [43] | Yee N, Ma J, Dalia A, et al. Se (Ⅵ) Reduction and the precipitation of se (0) by the facultative bacterium Enterobacter cloacae SLD1a-1 are regulated by FNR[J]. Applied and Environmental Microbiology 2007, 73(6) : 1914–1920. DOI:10.1128/AEM.02542-06 |
| [44] | Fujita M, Ike M, Nishimoto S, et al. Isolation and characterization of a novel selenate-reducing bacterium, Bacillus sp. SF-1[J]. Journal of Fermentation and Bioengineering 1997, 83(6) : 517–522. DOI:10.1016/S0922-338X(97)81130-0 |
| [45] | Yamamura S, Yamashita M, Fujimoto N, et al. Bacillus selenatarsenatis sp. nov., a selenateand arsenate-reducing bacterium isolated from the effluent drain of a glass-manufacturing plant[J]. International Journal of Systematic and Evolutionary Microbiology 2007, 57(5) : 1060–1064. DOI:10.1099/ijs.0.64667-0 |
| [46] | Losi ME, Frankenberger WT. Reduction of selenium oxyanions by Enterobacter cloacae SLD1a-1: isolation and growth of the bacterium and its expulsion of selenium nanoparticles[J]. Applied and Environmental Microbiology 1997, 63(8) : 3079–3084. |
| [47] | Kuroda M, Yamashita M, Miwa E, et al. Molecular cloning and characterization of the srdBCA operon, encoding the respiratory selenate reductase complex, from the selenate-reducing bacterium Bacillus selenatarsenatis SF-1[J]. Journal of Bacteriology 2011, 193(9) : 2141–2148. DOI:10.1128/JB.01197-10 |
| [48] | Avazéri C, Pommier J, Blasco F, et al. Reduction of oxyanions by photosynthetic bacteria and Escherichia coli : role of the nitrate reductase in the reduction of tellurite and selenate[A]//Proceedings of the 10th International Photosynthesis Congress[C]. Montpellier, France: IPC, 1995: 423-426 |
| [49] | Hockin SL, Gadd GM. Linked redox precipitation of sulfur and selenium under anaerobic conditions by sulfate-reducing bacterium biofilms[J]. Applied and Environmental Microbiology 2003, 69(12) : 7063–7072. DOI:10.1128/AEM.69.12.7063-7072.2003 |
| [50] | Kessi J, Hanselmann KW. Similarities between the abiotic reduction of selenite with glutathione and the dissimilatory reaction mediated by Rhodospirillum rubrum and Escherichia coli[J]. The Journal of Biological Chemistry 2004, 24(49) : 50662–50669. |
| [51] | Kessi J. Enzymic systems proposed to be involved in the dissimilatory reduction of selenite in the purple non-sulfur bacteria Rhodospirillum rubrum and Rhodobacter capsulatus[J]. Microbiology 2006, 152(3) : 731–743. DOI:10.1099/mic.0.28240-0 |
| [52] | Li DB, Cheng YY, Wu C, et al. Selenite reduction by Shewanella oneidensis MR-1 is mediated by fumarate reductase in periplasm[J]. Scientific Reports 2014, 4 : 3735. |
| [53] | Newton GL, Fahey RC. Glutathione in prokaryotes[A]//Vina J. Glutathione: Metabolism and Physiological Functions[M]. Boca Raton, Florida, USA: The Chemical Rubber Company Press, 1989: 69-77 |
| [54] | Kieliszek M, Blażejak S, Gientka I, et al. Accumulation and metabolism of selenium by yeast cells[J]. Applied Microbiology and Biotechnology 2015, 99(13) : 5373–5382. DOI:10.1007/s00253-015-6650-x |
| [55] | Hoefig CS, Renko K, K hrle J, et al. Comparison of different selenocompounds with respect to nutritional value vs. toxicity using liver cells in culture[J]. The Journal of Nutritional Biochemistry 2011, 22(10) : 945–955. DOI:10.1016/j.jnutbio.2010.08.006 |
| [56] | Mapelli V, Hillestr m PR, Kápolna E, et al. Metabolic and bioprocess engineering for production of selenized yeast with increased content of seleno-methylselenocysteine[J]. Metabolic Engineering 2011, 13(3) : 282–293. DOI:10.1016/j.ymben.2011.03.001 |
| [57] | Kitajima T, Chiba Y. Selenomethionine metabolism and its toxicity in yeast[J]. Biomolecular Concepts 2013, 4(6) : 611–616. |
| [58] | Hunter WJ, Kuykendall LD. Reduction of selenite to elemental red selenium by Rhizobium sp. strain B1[J]. Current Microbiology 2007, 55(4) : 344–349. DOI:10.1007/s00284-007-0202-2 |
| [59] | Hunter WJ, Manter DK. Bio-reduction of selenite to elemental red selenium by Tetrathiobacter kashmirensis[J]. Current Microbiology 2008, 57(1) : 83–88. DOI:10.1007/s00284-008-9160-6 |
| [60] | Hunter WJ, Manter DK. Reduction of selenite to elemental red selenium by Pseudomonas sp. strain CA5[J]. Current Microbiology 2009, 58(5) : 493–498. DOI:10.1007/s00284-009-9358-2 |
| [61] | Hunter WJ. A Rhizobium selenitireducens protein showing selenite reductase activity[J]. Current Microbiologyl 2014, 68(3) : 311–316. DOI:10.1007/s00284-013-0474-7 |
| [62] | deMoll-Decker H, Macy JM. The periplasmic nitrite reductase of Thauera selenatis may catalyze the reduction of selenite to elemental selenium[J]. Archives of Microbiology 1993, 160(3) : 241–247. |
| [63] | Yanke LJ, Bryant RD, Laishley EJ. Hydrogenase I of Clostridium pasteurianum functions as a novel selenite reductase[J]. Anaerobe 1995, 1(1) : 61–67. DOI:10.1016/S1075-9964(95)80457-9 |
| [64] | Li JX, Wu WL, Guo YB. Review on nano selenium produced by bacteria[J]. Journal of Agricultural Resources and Environment 2014, 31(6) : 551–554. (in Chinese) 李吉祥, 吴文良, 郭岩彬. 细菌生物合成纳米硒的研究进展[J]. 农业资源与环境学报 2014, 31(6) : 551–554. |
| [65] | Zhu YY, Ren BY, Guo YB, et al. Biosynthesis of selenium nanoparticles and their interaction in Rahnella aquatilis HX2[J]. Applied Microbiology and Biotechnology, in press |
| [66] | Ren BY. Transformation of different selenium and bacteria-mediated plant of absorption in Rahnella aquatilis HX2[D]. Beijing: Master's Thesis of China Agricultural University, 2014 (in Chinese) 任宝玉.植物促生菌Rahnella aquatilis HX2对不同硒源的转化以及玉米硒吸收的影响[D].北京:中国农业大学硕士学位论文, 2014 |
| [67] | Guo YB, Wu WL, Li JX, et al. A method of preparing biological selenium nanoparticles by Rahnella aquatilis HX2: China, 201510047365.4[P]. 2015-07-15 (in Chinese) 郭岩彬, 吴文良, 李吉祥, 等.一种利用水生拉恩氏菌制备生物纳米硒的方法:中国, 201510047365.4[P]. 2015-07-15 |
| [68] | Gurinder K, Mohammad I, Mandeep SB. Biomineralization of fine selenium crystalline rods and amorphous spheres[J]. The Journal of Physical Chemistry 2009, 113(31) : 13670–13676. |
| [69] | Lenz M, Kolvenbach B, Gygax B, et al. Shedding light on selenium biomineralization: proteins associated with bionanominerals[J]. Applied and Environmental Microbiology 2011, 77(13) : 4676–4680. DOI:10.1128/AEM.01713-10 |
| [70] | Chasteen TG, Bentley R. Biomethylation of selenium and tellurium: microorganisms and plants[J]. Chemical Reviews 2003, 103(1) : 1–26. DOI:10.1021/cr010210+ |
| [71] | Heider J, B ck A. Selenium metabolism in microorganisms[J]. Advances in Microbial Physiology 1993, 35 : 71–109. DOI:10.1016/S0065-2911(08)60097-1 |
| [72] | Ranjard L, Nazaret S, Cournoyer B. Freshwater bacteria can methylate selenium through the thiopurine methyltransferase pathway[J]. Applied and Environmental Microbiology 2003, 69(7) : 3784–3790. DOI:10.1128/AEM.69.7.3784-3790.2003 |
| [73] | Ranjard L, Combaret CP, Sabine FB, et al. Characterization of a novel selenium methyltransferase from freshwater bacteria showing strong similarities with the calicheamicin methyltransferase[J]. Biochimica et Biophysica Acta 2004, 1679(1) : 80–85. DOI:10.1016/j.bbaexp.2004.05.001 |
| [74] | Doran JW. Microorganisms and the biological cycling of selenium[A]//Marshall KC. Advances in Microbial Ecology[M]. New York: Springer, 1982, 6: 1-32 |
| [75] | Sayato Y, Nakamuro K, Hasegawa T. Selenium methylation and toxicity mechanism of selenocysteine[J]. Yakugaku Zasshi 1997, 117(10/11) : 665–672. |
| [76] | Choudhury HG, Cameron AD, Iwata S, et al. Structure and mechanism of the chalcogen-detoxifying protein TehB from Escherichia coli[J]. Biochemical Journal 2011, 435(1) : 85–91. DOI:10.1042/BJ20102014 |
| [77] | Ranjard L, Prigent-Combaret C, Nazaret S, et al. Methylation of inorganic and organic selenium by the bacterial thiopurine methyltransferase[J]. Journal of Bacteriology 2002, 184(11) : 3146–3149. DOI:10.1128/JB.184.11.3146-3149.2002 |
| [78] | Stadtman TC. Selenocysteine[J]. Annual Review of Biochemistry 1996, 65 : 83–100. DOI:10.1146/annurev.bi.65.070196.000503 |
| [79] | Hatfield DL, Berry MJ, Gladyshev VN. Selenium: It's Molecular Biology and Role in Human Health[M].3rd ed. Berlin: Springer, 2012 . |
| [80] | Xu XM, Mix H, Carlson BA, et al. Evidence for direct roles of two additional factors, SECp43 and soluble liver antigen, in the selenoprotein synthesis machinery[J]. The Journal of Biological Chemistry 2005, 280(50) : 41568–41575. DOI:10.1074/jbc.M506696200 |
| [81] | Thanbichler M, B ck A. Functional analysis of prokaryotic SELB proteins[J]. Biofactors 2001, 14(1/4) : 53–59. |
| [82] | Raymana MP. The use of high-selenium yeast to raise selenium status: how does it measure up?[J]. British Journal of Nutrition 2004, 92(4) : 557–573. DOI:10.1079/BJN20041251 |
| [83] | Papp LV, Lu J, Holmgren A, et al. From selenium to selenoproteins: synthesis, identity, and their role in human health[J]. Antioxidants and Redox Signalling 2007, 9(7) : 775–806. DOI:10.1089/ars.2007.1528 |
| [84] | Shirft DRA. A selenium cycle in nature?[J]. Nature 1964, 201(4926) : 1304–1305. DOI:10.1038/2011304a0 |
| [85] | Wang YH, Qin Y, Witold K, et al. Genome sequence of selenium-solubilizing bacterium Caulobacter vibrioides T5M6[J]. Genome Announcements 2016, 4(1) : e01721–15. |
 2017, Vol. 44
2017, Vol. 44