扩展功能
文章信息
- 葛元洁, 陈实
- GE Yuan-Jie, CHEN Shi
- 专论与综述
- Single molecule real time sequencing and its applications in microbial epigenetics-a review
- 微生物学通报, 2017, 44(1): 186-199
- Microbiology China, 2017, 44(1): 186-199
- DOI: 10.13344/j.microbiol.china.160229
-
文章历史
- 收稿日期: 2016-03-21
- 接受日期: 2016-05-09
- 优先数字出版日期(www.cnki.net): 2016-05-24
生命的基本遗传信息存在于核酸序列中,四种脱氧核苷酸的排列顺序决定了影响生命活动的RNA和蛋白质的序列。表观遗传是在不改变DNA核苷酸序列的条件下,调节特定基因的时空表达,使基因表达发生可遗传的改变。表观遗传学是调节不同化学基团共价结合到DNA、染色体、组蛋白及其他相关蛋白过程的分子生化机制的集合。微生物中的表观遗传信息主要是通过DNA修饰(如DNA的甲基化)传达的,它们广泛存在于微生物中,并对微生物的生命进程产生重大影响。在一些模式细菌如大肠杆菌(Escherichia coli)、新月柄杆菌(Caulobacter crescentus)等中,DNA甲基化的重要性已经被确定,由Dam和Ccrm甲基转移酶催化产生的腺嘌呤甲基化在染色体的复制、DNA修复和基因表达的控制中尤为关键。微生物中不同类型的DNA修饰及调控修饰酶的本质和功能都有待于发现和研究。
过去几十年测序平台的革新和发展极大地推动着基因组测序的脚步,而对于DNA修饰的测序分析却迟迟没有起色,使得表观遗传学的研究远远落后。为了获得DNA修饰在基因组中的分布信息,以更深入地研究表观遗传学对生命活动的影响,要求测序技术不断提升,实现对DNA修饰的检测,并提高测序分辨率,逐步实现在总体水平、区域水平、特异性链水平、单个核苷酸水平的修饰检测。单分子实时测序技术的发展使得从DNA上同时获得基因组序列信息和单核苷酸水平的表观遗传信息成为可能,也为微生物表观遗传的进一步探索做好了准备。
1 第三代测序技术的兴起DNA作为生物遗传信息的载体,其序列的获取对于探索生命奥秘至关重要。DNA测序技术无疑已经成为生物学研究中最常用的技术手段。自20世纪70年代中期DNA测序技术出现以来的40年,DNA测序技术取得了飞跃式的发展,同时也推动着生命科学领域及其相关交叉学科领域的极速发展。
1977年,Sanger等的双脱氧核苷酸末端终止测序法和Maxam、Gilbert的化学降解测序法开启了解密生命信息的大门,在这两种方法的基础上发展起来的荧光自动化测序技术、毛细管电泳测序技术、杂交测序技术等都属于第一代测序技术。第一代测序技术在最初的基因组谱图测序工作中起到了关键作用,小到噬菌体基因组,大到人类基因组计划,第一代测序技术凭借其超过1 000 bp的读长及高达99.999%的原始数据准确率仍在目前的市场应用中占有一定的比例。但其测序通量较低、速度较慢、时间较长、成本较高已经无法满足大规模的测序任务。
以高通量、低成本为特征的第二代测序技术应运而生。它的原理主要是将DNA片段固定在固体表面,通过PCR扩增,进行边合成边测序。其代表主要有Roche公司以焦磷酸测序为原理的454测序技术、Illumina公司以合成测序为原理的Solexa测序技术、ABI公司以连接酶技术开发的SOLiD测序平台。通量高、速度快、价格低的第二代测序技术很快得到了广泛应用。然而,第二代测序技术也有不可忽略的弊端,其较短的读长给序列的组装拼接带来困难,另外,由于第二代测序依赖PCR扩增,G+C含量异常的基因组测序结果将受到影响。
第三代测序技术以单分子测序为特点已逐渐崭露头角。它在纳米级的反应空间内对DNA进行单分子水平的边合成边测序。该方法有着更快的数据读取速度,测序读长可达到20 kb,不再需要PCR扩增,进一步简化测序步骤,降低测序成本,提高测序通量和数据产出。单分子测序通过检测原始状态的样本获取更直接真实的遗传信息,实现了RNA和核酸修饰的直接测序。第三代测序技术以其无与伦比的优势取得了越来越多的关注和应用。已出现的第三代测序技术主要有Helicos Biosciences公司研发的单分子测序(True single molecular sequencing,tSMSTM)技术及单分子测序仪(HeliScope Single Molecular Sequencer),Pacific Biosciences公司研发的单分子实时(Single molecule realtime,SMRT)测序技术平台,Visi GenBiotechnologies公司研发的荧光能量共振转移(Fluorescence resonance energy transfer,FRET)测序技术,Oxoford Nanopore Technologies公司研发的纳米孔单分子测序(Single-molecule nanopore DNA sequencing)技术。目前,当属Pacific Biosciences公司运用SMRT单分子实时测序技术的PacBio RS系列测序仪在商业化中有较好的应用。
在检测DNA修饰方面,一代测序虽然可以检测到一部分被修饰的碱基,但它并没有在这方面得到广泛应用;而二代测序由于其依赖的DNA扩增会抹掉DNA修饰信息,需要对样本进行预处理来区别修饰和未被修饰的核苷酸再进行测序,如利用亚硫酸氢盐将胞嘧啶转化为尿嘧啶而甲基胞嘧啶不变从而检测出甲基胞嘧啶,处理过程繁琐且方法、效率有限,造成DNA修饰的检测在二代测序中的难度;三代测序中的单分子实时测序技术,无需样本预处理,通过测序过程中DNA聚合酶动力学信息的变化,直接获得DNA修饰信息。三代测序凭借其简单直接获取修饰信息的能力将在表观遗传学研究领域发挥重大作用。
2 单分子实时测序技术 2.1 SMRT测序原理2009年,Pacific Biosciences公司推出了SMRT测序技术。SMRT测序的基本流程是首先将待测DNA打断成一定长度的片段,并进行DNA损伤修复及末端修复,再在DNA片段两端接上发夹结构的接头使DNA能够进行环化测序,纯化后将DNA样本上样到PacBio RS系列测序仪的SMRT cells中进行测序[1]。
实现单分子实时测序有赖于3个关键核心技术:
(1)磷酸-荧光基团连接的核苷酸:带有不同荧光标记的脱氧核苷酸在参与DNA合成中被检测到荧光信号。该核苷酸的荧光基团连接在磷酸基团(图 1a)上,与以往测序中使用的连接在碱基上的核苷酸不同(图 1b),在DNA聚合酶的作用下生成磷酸二酯键,该荧光标签被切除,不会保留在延伸的DNA链中对后续的DNA合成产生空间位阻影响DNA酶的活性,也不会增加背景荧光,合成和天然DNA相同的DNA链。
(2)零模波导孔(Zero-Mode waveguides,ZMWs):测序是在厚度为100 nm的金属薄膜SMRT cells上进行的,每个SMRT cell中有150 000个ZMWs (图 2a)。ZMW是直径为10 nm−50 nm的纳米微孔,由于其短于激光的单个波长,激光无法穿过小孔而发生衍射,只照亮ZMW底部形成一个很小的检测区域,DNA聚合酶和DNA模板复合物就被固定在这个区域内,并且有约三分之一的ZMWs中只有一个DNA聚合酶,且在后续数据分析中没有DNA链或含有两条及以上DNA链的ZMWs数据会被过滤掉,从而实现单分子测序;另外,在这个空间有限的纳米结构中,游离在DNA链周围的核苷酸也十分有限,因此形成的背景荧光信号也十分稳定(图 2b)。
(3)实时检测:带有不同荧光标签的核苷酸进行布朗运动快速地进出ZMW,一旦某个核苷酸与模板DNA碱基互补配对就会被DNA聚合酶捕捉形成磷酸二酯键延伸DNA,这个过程时间较长,荧光基团被激光激发产生一定强度的荧光,特定的荧光信号会维持一段时间,成千上万个ZMWs中DNA合成反应平行进行,过程中产生的荧光信号通过共聚焦荧光显微镜被实时监测收集(图 3a),即无数个ZMWs中核苷酸掺入DNA链产生的荧光脉冲被实时记录下来(图 3b),再通过生物信息学分析得出相应位置上的DNA序列[1-2]。
所有生物的基因蓝图都包含在他们的基因组中。A、T、G、C四种基本核苷酸碱基的顺序决定了调节细胞功能的RNA和蛋白序列。附加的表观遗传信息是通过这四种碱基的化学修饰传达的,它们在所有生物中都存在并且产生着意义重大的生物学影响。微生物的表观基因组是动态的,在生长过程中微生物对外部刺激产生反应,大规模地控制基因交换、转录及基因组的稳定性,从而促进其对环境条件变化的适应。表观基因组由核苷酸上的小分子修饰(如甲基化)和两个核苷酸之间的原子修饰,如磷硫酰化(Phosphorothioation,PT)修饰[8]组成。这些修饰也会改变蛋白-DNA结合,从而改变DNA的生化特征,直接影响其表型。DNA修饰最初是在噬菌体的遗传研究中发现的[9]。此发现引领着DNA修饰对外来DNA影响的研究,也预示着研究微生物DNA修饰的重要性。原核生物中大量调节DNA甲基化的酶和不同类型DNA修饰的本质、范围及影响都有待于研究。
3.1 微生物中的DNA甲基化修饰目前,微生物中被鉴定的最广泛存在的DNA修饰就是DNA的甲基化修饰,甲基化修饰也是被研究得最全面的DNA修饰[9-11]。DNA甲基化是由3个家族的DNA甲基转移酶(MTases)催化,共价地将甲基加到腺嘌呤或胞嘧啶上,通常这个过程发生在特定序列中。其中两种酶作用在环外的氨基上,产生甲基腺嘌呤(6-methyladenine,6mA)和4-甲基胞嘧啶(4-methylcytosine,4mC),这两种DNA甲基化在哺乳动物DNA中不存在或极少存在。另外,微生物编码了一类与真核生物同源的MTases,它能够催化5-甲基胞嘧啶(5-methylcytosine,5mC)的形成。在不同的菌种中,MTases的数量和修饰序列基序(Motif)有很大的不同,即便是在关系密切的生物体和酶中。在3 300多个完成全基因组测序的细菌中94%的菌体内发现了可能的MTases,基因组分析也显示一些形式的DNA甲基化在几乎所有的细菌中都存在。然而,绝大多数MTases精确的序列靶标和生物学作用仍属未知。
3.1.1 甲基化与限制-修饰(Restriction-modification,RM)系统: 在探究噬菌体感染实验中,RM系统被第一次描述出来[9]。在超过3 600个细菌菌株中发现了43 650个RM酶。基因组分析显示很多细菌的MTases在限制性核酸内切酶(Restriction enzymes,REs)附近编码,这表明它们是RM系统的一部分。RM系统需要一个DNA识别位点、一个MTase和一个RE。目前已发现4种RM系统,Ⅰ、Ⅱ、Ⅲ型RM系统识别甲基化修饰的DNA[12-14],Ⅳ型RM系统识别PT修饰的DNA和甲基化修饰的DNA[15]。 MTases在RM系统识别区域内特异的Motif中选择性地进行甲基化修饰产生4mC、5mC和6mA。RM系统作为一个保护细菌抵御外源DNA的基础细菌免疫系统,成对的MTases和REs识别相同的目标序列,甲基化能够保护目标位点不被REs剪切;而外来DNA (如噬菌体、接合因子、外源质粒等)没有保护性的甲基化,很容易被REs剪切消化。RM系统被广泛证实能够保护细胞抵御外来DNA的入侵。另外,通过操控RM系统产生具有R+M+和R-M+表型的活细胞,而R+M-表型是致死的,这表明没有DNA甲基化,REs会消化自己的DNA,导致细胞死亡。 近期的研究发现RM系统也可能对调节天然DNA序列的表达起着重要作用。在一些菌种中,与RM系统关联的MTases相转变能够调节基因的表达,尽管它们的作用机理还没有被研究清楚。这样的相转变使得单菌株能够通过多重独特的表型来适应变化多样的生态环境。RM系统的组分也被鉴定为可以广泛影响包括细胞周期、毒性、基因表达等在内的其他生命机制。 3.1.2 孤儿甲基转移酶: 为了逃脱宿主的RM作用,噬菌体在感染过程中往往会引入其他的MTases。通过可动因子介导的水平基因转移等多种机制,这些MTases在噬菌体感染后可能在宿主中保留下来,产生没有REs与之关联的孤儿MTases[10]。孤儿MTases不仅增加了细菌MTases的多样性,还在不可预知的方向影响着DNA的甲基化修饰,导致个别菌株变异成具有特殊特征的菌株,很多MTases有多种目标识别区域,使不同的表型中含有多样的甲基化Motifs。 孤儿MTases通过调节基因的表达来改变表型,提高细菌存活率[10]。在各种肠道细菌中都存在的一种产生6mA的MTase--Dam,它是典型的孤儿MTases,对染色体复制的起始、DNA修复、基因表达和一些病原菌的毒性都有调节作用。大多数Dam的调节作用都是由于DNA甲基化影响了蛋白与它们靶标位点的结合。同样,另一个产生6mA的孤儿MTases--CcrM,通过控制细胞周期的时间和细胞分化,对新月柄杆菌(Caulobacter crescentus)的复制和生存能力产生影响。然而,即使是这些突出的例子也没有进行过全基因组甲基化位点及其影响的综合分析,绝大多数孤儿MTases (包括类似于真核生物中5mC的MTases)的生物学意义还有待于探究。 3.2 微生物中的磷硫酰化修饰除了甲基化以外的其他形式的DNA修饰如磷硫酰化修饰的生物学意义也有待于发掘。
磷硫酰化(PT)修饰是近期发现的一种新型DNA修饰类型,它是磷酸二酯键上的非桥连氧原子被硫原子取代产生的一种修饰。最早是Zhou等[16]在变铅青链霉菌(Streptomyces lividans)基因组DNA的凝胶电泳中发现了降解现象,后续研究表明这种降解现象是由于含有PT修饰的DNA在Tris过酸衍生物的作用下发生的氧化切割反应[17-18]。Wang等[19]通过液质联用的方法鉴定出PT修饰的结构,硫原子取代了两个脱氧核糖核苷酸之间磷酸骨架上的非桥连氧原子形成硫代磷酸二酯键,并且PT修饰是以RP空间构象存在的(图 4)。PT修饰的发现和解构不仅证明了DNA中不只有碳、氢、氧、氮、磷这5种元素,还有硫元素的存在,也是首次发现DNA中除了有核酸碱基上的修饰,还有DNA骨架上的修饰。目前,在微生物中发现的PT修饰连接的双核苷酸包括d (APSA)、d (APSC)、d (CPSA)、d (CPSC)、d (GPSA)、d (GPSG)、d (GPST)、d (TPSA)、d (TPSC)[8, 19-20]。Ou等[21]发现dnd基因簇负责PT修饰,并且通过电泳的基因组拖尾实验得知PT修饰广泛而非普遍存在于细菌中。Wang等[8]利用液相-串联质谱的方法建立了原核基因组中PT修饰的定量方法,并且在不同种类的细菌中检测并定量了PT修饰。
虽然PT修饰已经被发现了一段时间,但是它在细菌中的生物学功能尚属未知。Xie等[22]发现立体定向的硫连接对于DNA是一种抗氧化剂。当沙门氏菌(Salmonella)用H2O2处理时,PT修饰会增多抵抗氧化剂的作用。Howard等[23]在从美国临床医学分离出的脓肿分枝杆菌(Mycobacterium abscessus)中研究PT修饰。他们发现dnd是一个可移动的基因岛,推测PT修饰与人类疾病相关,但是仍然缺乏PT修饰与病原菌毒性直接相关的证据。Xu等[24]发现含有PT修饰的肠道沙门氏菌(Salmonella enterica serovar Cerro 87)对来源于大肠杆菌(Escherichia coli)的DNA有限制作用,但能通过PT修饰保护自己的DNA,通过基因克隆及突变分析证实了PT修饰能够阻止限制作用,发现了一个新型RM系统,也确认了PT的RM系统除了需要负责PT修饰的基因簇,还需要其下游由3个基因组成的具有限制作用的基因簇。尽管这些发现很振奋人心,并且为研究PT修饰的生物学功能奠定了基础,但是它们仍无法快速精确地定位PT修饰的位点来更深入研究其生物学意义。
4 表观遗传学的检测方法表观基因组参与并影响着大量的生命活动,检测DNA修饰信息是研究表观遗传学及探索其生物学意义的关键环节。很多技术被发展用于量化被修饰的核苷酸数量,提供不同分辨率水平(总体的、区域的、位点特异的)的表观遗传信息。
4.1 用分析化学的方法检测DNA修饰被修饰的核苷酸可以通过色谱和质谱(Mass spectrometry,MS)等非测序的分析化学方法进行检测,且MS分析始终是发现新型修饰的唯一方法[25]。在MS分析前,需要将提取纯化后的DNA进行化学消化或剪切。提取并纯化后的DNA,经热、化学、核酸酶和磷酸酶的水解作用消化释放单个核苷或核酸碱基。检测单个核苷比直接检测DNA寡核苷酸更灵敏、精确[8, 25]。产生的DNA单体可以通过液-液萃取、固相萃取、免疫亲和、液相色谱(Liquid chromatography,LC)等方法富集[22]。水解过程中超滤也是一个备选的去除蛋白和试剂的纯化方法[8]。
很多DNA表观遗传修饰和加合物的存在是由于压力引起的损伤,合适的分析设备的选择很值得考量。如果可以获得足量的DNA [低频率(1/100 000 000−1/1 000)的DNA修饰信号检测需要大量的样本DNA[8, 25]],LC分离技术可被用于新型修饰的筛选。核苷酸可以通过飞行时间质谱(Time-of-flight mass spectrometry,TOF-MS)的实时直接分析(Direct analysis in real time,DART)[26]检测得到。电子耦合等离子体质谱(Inductively coupled plasma mass spectrometry,ICP-MS)也可以定量痕迹量级的修饰DNA的金属、准金属、非金属[27]。通常,气相色谱(Gas chromatography,GC)或LC与MS联用进行DNA修饰的检测,由于GC需要对DNA单体进行衍生提高其挥发性,LC的应用更广泛。选择离子监测模式的单级四极杆MS和三重四级杆设备可以用于灵敏地量化已知修饰碱基。三重四级杆LC-MS联用仪被用于灵敏地定量细菌中16种PT修饰[8]。离子阱质谱也可以被用于量化修饰,这个方法还适用于获得新型DNA修饰的结构信息。对于未知的加合物,由精确的质谱数据确定的分子式和由MS/MS的碎片信息获得的结构分析是十分重要的。新一代轨道阱和四级-TOF设备可以用于这些分析。振动光谱也被用于鉴定DNA修饰,微拉曼光谱可以检测人类总DNA甲基化水平的变化[28],这个技术可以直接检测分析未经消化的DNA,保证DNA不被破坏,目前也已被用于细菌样本的检测。
4.2 用测序的方法检测DNA修饰分析化学的方法虽然可以定量DNA中修饰的含量,也可以鉴别修饰类型,但是这些方法无法在特异性链、特定序列及单个核苷酸水平上反馈全基因组的修饰位点信息。测序平台的不断发展推动着基因组测序步伐的迅速加快。而对于被修饰碱基的检测仍存在着较大的技术挑战,DNA修饰的测定还没有成为常规测序分析的一部分,使得对表观遗传学的认识与基因组之间存在着很大差距。在DNA特异性链和单个核苷酸水平上检测DNA修饰的高通量测序方法的缺失严重阻碍了对这些修饰生物学意义的综合分析。
4.2.1 传统测序方法对DNA修饰的检测: 利用测序技术测定DNA修饰需要得知其在基因组的位点及修饰类型。在直接测序之前,先利用分析化学的方法确定化学基团。DNA修饰位点测序分析的3个主要内容是:(1)在完全消化DNA后检测到精确的碱基;(2)检测到修饰基团;(3)检测其在基因组中的位置。将DNA测序技术和质谱方法结合起来即可实现修饰在基因组中的检测和定位。 尽管Sanger测序的方法也可能检测到一些被修饰的碱基,但是这项技术并没有被广泛应用于这方面。第二代高通量DNA测序技术在测序过程中需要对DNA模板进行扩增(DNA的扩增会抹去表观遗传的标识,因此检测被修饰的核苷酸只能用天然DNA作为测序模板),因此不能被用于天然DNA中修饰碱基的直接检测。很多方法被发展用于DNA甲基化的测序,这些方法基于亚硫酸氢盐的转化、限制酶的消化、亲和富集三种预处理技术中的一种。经过亚硫酸氢盐的处理,DNA样本中未被甲基化的胞嘧啶去氨基后转化为尿嘧啶,而甲基化的胞嘧啶不受到影响。这样在很多测序平台中就可以区分开被修饰和未被修饰的胞嘧啶了,亚硫酸氢盐测序是检测DNA甲基化状态的一种常用方法,再结合生物信息学分析得到甲基化的修饰位点[29-32]。然而,亚硫酸氢盐处理样本DNA的准备工作是相当繁琐的,并且这个技术无法区分5mC和5-羟甲基胞嘧啶(5-Hydroxymethylcytosine,5hmC)[33],虽然目前这个方法的发展已经可以避开对5hmC的检测[34-35]。亲和纯化技术配合微阵列或DNA测序技术可以很有效地检测被修饰碱基,但是这些特异富集修饰的方法无法实现对修饰位点的精确定位,还要求基因组DNA中含有较高含量的修饰。而且,对于在微生物基因组中最常见的6mA的测序分析仍然存在很大困难。 4.2.2 SMRT测序技术对DNA修饰的检测: 随着新一代测序技术的出现,微生物基因组测序和表观基因组检测都得到了极大的发展,实现了对已知和新型DNA修饰在特异性链和单个核苷酸水平上的高通量测序分析。这些技术也提高了我们研究这些修饰在微生物中生物学意义的能力,为更多大规模测序任务做好了准备,也将加速修饰密度、位点、链型及其酶的发现和探索。测序技术和数据处理技术的进步,对研究已知和发现新型DNA修饰、作用于表观基因组的基因、催化这些DNA修饰的酶及蛋白网络有很大帮助。 新一代测序技术中的SMRT测序技术实现了同时获得基因组及单个核苷酸水平的表观遗传信息[7]。基于对DNA合成反应的动力学分析,SMRT测序技术已经可以在获得DNA基本序列数据的同时实现对修饰核苷酸(如6mA、4mC、5mC等)的检测。目前,在微生物中对于甲基化和非甲基化位点的定位是可行的。这个技术进步为实现对微生物中无处不在的、多样的DNA甲基转移酶对基因表达、染色体结构、染色体复制等基本生命活动的影响及机理的综合评价做好了准备。SMRT测序也可以用于检测受损DNA,发现新型DNA修饰。 由Pacific Biosciences公司开发的SMRT测序技术应用在很多微生物中,产出了大量的基因组序列和甲基化组信息[36-40]。SMRT测序技术正如它的首字母缩略词所示,在单分子水平实时监测DNA的合成。在SMRT测序技术中[33, 41],纳米结构零模波导孔被用于分离单个phi29衍生的DNA聚合酶[2],每个DNA聚合酶捕捉一个环形模板DNA单分子,并利用带有不同荧光标记的四种核苷酸进行DNA合成。成千上万个DNA合成反应平行进行,核苷酸掺入DNA链时产生的荧光脉冲被实时监测收集,即得到基本的序列数据。监测DNA合成会产生两个重要参数,脉冲宽度(Pulse width,PW,核苷酸在聚合酶活性部位的保留时间)和脉冲间隔时间(Interpulse duration,IPD,核苷酸结合状态之间的时间间隔),反映了DNA合成动力学的信息。存在修饰位点的模板DNA的这两个参数与没有修饰的模板不同,可用于鉴别修饰位点存在与否[7, 33, 41]。 Flusberg等[7]用合成的DNA模板证实了具有独特修饰的脱氧核苷酸在测序中有着特殊的动力学特征变化(Kinetic variation,KV),即IPD和PW的变化。因为在DNA合成过程中,当DNA聚合酶在模板DNA上遇到被修饰位点(如6mA、5mC、5hmC、4mC等),合成速度就会下降(图 5A、B),速度降低的程度主要由碱基修饰的类型决定,不同的DNA修饰有着各自特有的KV。尤其是6mA和4mC有着强烈、可靠的动力学特征;而可能是由于5mC的甲基没有直接参与碱基配对,5mC对于合成动力学的影响并不是很显著[42]。Clark等[43]也鉴别出一些受损碱基的动力学变化,如8-氧桥鸟嘌呤、8-氧桥腺嘌呤、O6-甲基鸟嘌呤、1-甲基腺嘌呤、O4-胸腺嘧啶、5-羟基胞嘧啶、5-羟基尿嘧啶、5-羟甲基尿嘧啶或胸腺嘧啶二聚体。Cao等[44]利用SMRT测序方法实现了对PT修饰的检测。通过比较天然DNA模板(DNA修饰)和扩增DNA模板(无DNA修饰)每个位点的IPD来量化修饰碱基对合成动力学的影响,并且通过筛出两个样本IPD比值不同于基线的位点来识别出修饰位点[35](图 5C、D)。
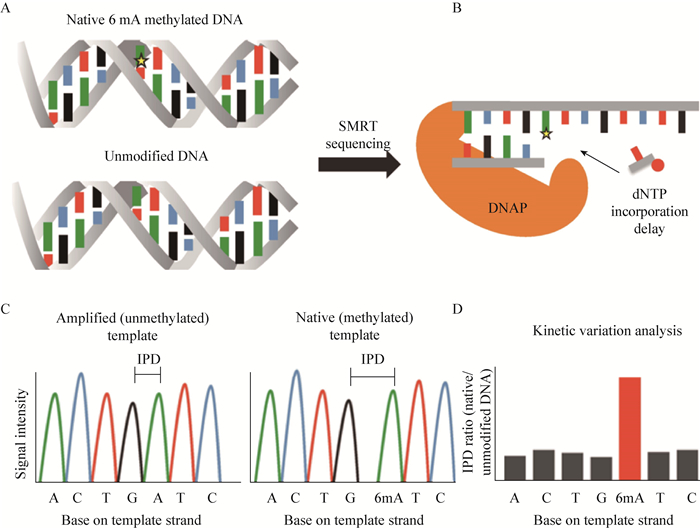
|
| 图 5 SMRT测序检测DNA修饰的原理(以6mA为例) Figure 5 Principle of detection of DNA modification with SMRT sequencing (taking 6mA for instance) 注:A:天然DNA模板(有DNA修饰)和扩增DNA模板(无DNA修饰);B:在DNA修饰位点对面掺入核苷酸前,DNA聚合酶会发生停顿;C:DNA修饰位点的IPD会延长;D:天然DNA模板和扩增DNA模板的IPD比值在DNA修饰位点会高于基线水平[41]. Note: A: Native DNA template (modified) and amplified DNA template (unmodified); B: Pause of DNA polymerase before dNTP inserting opposite the site of DNA modification; C: Extended IPD at the site of DNA modification; D: Higher IPD ratio between native and amplified DNA template at the site of DNA modification than baseline levels[41]. |
|
|
SMRT测序技术的发展使得从天然微生物DNA上同时获得基因组和表观遗传信息成为可能。SMRT测序技术结合先进的分析化学技术也使新型修饰及酶活性的发现得以实现。因此,我们已经开始步入了一个在微生物王国中探索DNA修饰的时代。
随着Flusberg等[7]和Clark等[42]确定了DNA甲基化及损伤的DNA碱基[43]等DNA修饰在SMRT测序中检测的可操作性,以及为了提高检测DNA修饰的灵敏性,增强DNA修饰位点信号的技术发展,如由于5mC在SMRT测序中信号较弱,Tet1酶[45]和来源于尾刺耐格里原虫(Naegleria gruberi)的类似于Tet的5mC加氧酶(TET-like C-5m oxygenase,NgTET1)[46]可以氧化5mC,使其在测序中动力学信号增强,在微生物基因组中系统检测DNA修饰成为可能,很多利用SMRT测序技术探索微生物全基因组表观遗传学的研究成果不断涌现。
DNA甲基化是微生物中最常见的DNA修饰,也是被研究得最广泛和最深入的DNA修饰。SMRT测序为DNA甲基化研究提供了新的平台,也将为DNA甲基化的探索带来新的突破。利用SMRT测序技术,通过动力学信号的变化及生物信息学分析,已经在大肠杆菌(Escherichia coli)[38, 42, 47-48]、硫还原泥土杆菌(Geobacter metallireducens)[40, 47]、需盐色盐杆菌(Chromohalobacter salexigens)[40, 47]、弧菌(Vibrio breoganii)[40]、蜡样芽胞杆菌(Bacillus cereus)[40]、空肠弯曲杆菌(Campylobacter jejuni)[40, 47, 49]、生殖支原体(Mycoplasma genitalium)[39]、肺炎支原体(Mycoplasma pneumoniae)[39]、金属还原的希瓦氏菌(Metal-Reducing Bacterium Shewanella oneidensis)[37]、幽门螺杆菌(Helicobacter pylori)[36, 47, 50-51]、肠道沙门氏菌(Salmonella enterica subsp. enterica Serovars)[52]、脑膜炎双球菌(Neisseria meningitides)[53-54]、乳酸杆菌(Lactobacillus)[55]、粪肠球菌(Enterococcus faecalis)[56]、新月柄杆菌(Caulobacter crescentus)[47]、盐水球菌(Salinicoccus halodurans)[57]、螺杆菌(Helicobacter typhlonius)[58]、嗜水气单胞菌(Aeromonas hydrophila)[59]等细菌及富盐菌(Haloferax volcanii)[60]等古菌中确定了DNA甲基化的存在,分析出甲基化的全基因组位点、修饰频率、链特异性、修饰Motif、甲基化模式等甲基化组信息。通过与突变株SMRT测序结果的比对鉴定出有活性的MTases[40]、MTases的特异识别序列、所属RM系统类型。获得DNA甲基化除了保护自身DNA防御外来DNA以外的生物学意义:通过甲基化位点的分布推断出它们可以调节基因表达[36, 39, 47]、改变转录本[38, 47]、扩增基因[38]、影响细胞周期[39]、调节基因组复制[37]、致病毒性[48]等对生命进程的影响。亲缘关系相近的菌株所有的甲基化模式也会有很大区别[47, 55],同一株菌在不同培养条件下也有着不同的甲基化模式[37]。可见,不同的甲基化模式影响着不同的表型,甲基化模式的转变也是对环境变化的一种适应。通过SMRT测序技术检测微生物的全基因组甲基化,不仅可以确认用其他方法研究DNA甲基化的成果,还可以发现很多新型的MTases种类、修饰Motif、甲基化功能等来扩充甲基化模式的多样性。
Blow等[61]利用SMRT测序技术测定230种不同的细菌、古菌的全基因组甲基化(6mA、4mC、5mC)。研究表明DNA甲基化修饰广泛存在于微生物中,其中93%的菌都被检测到有甲基化。研究提供了每种微生物的单碱基水平的甲基化信息,共鉴定出与这些微生物中活性MTases特异性相一致的834个不同的甲基化Motif。Ⅰ型RM系统(于距结合位点较远的地方剪切DNA)、ⅡG型和Ⅲ型RM系统(很难完全剪切DNA)在分析限制消化模式的传统方法中很难得到研究进展,SMRT测序给这些类型RM系统的研究带来了福音。他们分析了264个来自于这些RM系统的MTases特异性,结果显示它们有着较目前已知的两倍还多的620个特异性DNA结合位点,且其中有85%是目前独一无二的。而对于Ⅱ型RM修饰系统(在结合位点或离结合位点很近的地方发生剪切),他们几乎没有发现新的Ⅱ型MTases特异性,这个表明先前对于Ⅱ型RM修饰系统特异性的研究已经涵盖了自然界中存在的绝大多数Ⅱ型MTases的特异性。研究也对大量存在的不属于RM系统的DNA甲基化进行了分析,他们在48%的被测菌(覆盖了研究中75%类群的菌)中确认了100多个新的孤儿Ⅱ型MTases活性和序列特异性,表明了原核生物中广泛存在的DNA甲基化并不局限于RM系统中。通常,孤儿Ⅱ型MTases在靶标位点是不完全甲基化模式。测序结果显示,与孤儿MTases相关的未被甲基化位点通常在基因上游的非编码序列中,因此它们可能具有调节功能。此外,他们还发现孤儿MTases和与它们相关的甲基化模式在亲缘相近的生物中都是保守的。研究在螺旋菌(Spirochaetae)和放线菌(Actinobacteria)中鉴定出了可能具有基因调控作用的新型保守MTases,在嗜盐古菌(Haloarchaea)中鉴定出可能具有调节DNA复制功能的保守MTases,这些都为无RM修饰系统的DNA甲基化具有基因组调控功能提供了证据。研究展现了微生物中DNA甲基化的广泛性和多样性,极大地扩充了微生物MTases特异性的已知文库,研究结果也显示DNA甲基化在微生物中有着重要的功能,这些大量的新型RM系统及相关甲基化组的数据将为未来对这个领域的探索提供很有价值的资源。SMRT测序技术在未来DNA甲基化的突破性研究中将起到重要作用。
PT修饰是近期发现的一种新的DNA修饰类型,它也广泛存在于细菌中[8, 21],但是目前PT修饰的功能尚未研究透彻。SMRT测序技术在PT修饰研究中的应用将为揭示PT修饰在微生物中的生物学意义提供很大帮助。Cao等[44]利用SMRT测序技术及碘诱导在PT位点发生断裂的深度测序技术检测了大肠杆菌Escherichia coli B7A (带有修饰基因dndB-E和限制基因dndF-H,具有PT修饰的RM功能,质谱结果显示106个核苷酸中分别有370±11个GPSA和398±17个GPST修饰)和弧菌Vibrio cyclitrophicus FF75 (没有限制基因dndF-H,不具有RM功能,质谱结果显示每106个核苷酸中有2 600±22个CPSC修饰)两株菌中的全基因组PT修饰。经过测序和生物信息学、统计学分析,在B7A中检测到发生在GPSAAC和GPSTTC序列中的PT修饰,且PT修饰含量与LC-MS/MS定量的d (GPSA)和d (GPST)结果一致;双核苷酸d (GPSA)和d (GPST)所在的GAAC和GTTC Motif互补,加上测序结果明确显示GAAC/GTTC序列的两条链上都存在着PT修饰,证明了B7A中的PT修饰是双链的DNA修饰;而在基因组中的40 701个GAAC/GTTC Motif中仅12%的位点被修饰,在不同细胞中被修饰的位点也不同,但针对这部分位点并没有发现它们被PT修饰选择的明确原因;由PT修饰在基因组中是部分修饰的现象也可以推断出,没有PT修饰的Motif位点不会被RE作用而发生断裂;这些结果显示,与PT修饰相关的RM系统是一个在基因组中只有部分修饰的新型RM系统,不同于已知的Ⅰ、Ⅱ、Ⅲ、Ⅳ型RM系统。FF75中的PT修饰发生在单链上,Motif为CPSCA,修饰含量与LC/MS-MS中d (CPSC)的定量结果一致;与B7A相同,FF75也是部分修饰,在基因组160 541个Motif中只有14%的位点被修饰,这些PT修饰的位点也是动态的;基于没有与dndF-H同源的限制基因、单链修饰、部分修饰这些特征,可以推测出PT修饰在FF75中发挥着非RM功能的其他生物学作用,如可能具有表观遗传调控基因表达的功能等。这个研究提供了第一个高分辨率(单个核苷酸水平)的PT修饰基因组谱图,证实了SMRT测序技术可以成为研究PT修饰的有效手段,也意外地发现了PT修饰与众不同的特性,对未来PT修饰的研究起到了奠基和引领的作用。
微生物中与基因组修饰相关的很多问题用SMRT测序技术都更容易处理。综合SMRT测序技术和分析化学、生物信息学、转录组、代谢组等其他生物学技术可以逐步阐明微生物表观遗传系统的复杂性及这些修饰的生物学作用。随着测序成本的降低,微生物基因组实用性的不断提高及相关技术不断进步,生物学领域会有更多新的发现。
6 SMRT测序技术在表观遗传学中应用的未来展望微生物表观基因组研究的快速发展开启了重新定义表观遗传学及研究其在生命周期中作用的新纪元。能够提供碱基修饰的准确检测、基因组中修饰位点、修饰密度等表观遗传信息的DNA测序技术为研究表观遗传功能提供了根据和线索。随着SMRT测序技术的改进和具有能产出表观遗传数据等类似功能的测序技术的发展,未来可以实现对微生物基因组中所有碱基修饰的全面分析。随着SMRT测序通量的提高,在高等真核生物中进行类似的研究也将成为可能。目前,SMRT技术可以被用于收集线粒体和具有较小基因组的简单真核生物中的表观遗传信息,并且在其中一些基因组中已经发现含有不寻常的、具有生物学意义的化学修饰(如在动基体目原生动物中发现J碱基[62])。
新兴的表观遗传学技术将会引导更多具有重要生物学意义的碱基修饰的发现。发现新的DNA修饰及其功能需要新一代测序技术与其他高通量组学研究相结合提供随着微生物行为改变而发生的分子变化信息。开发能够整合基因组序列、表观基因组、转录组、代谢组等数据的生物信息学技术对未来功能基因组研究是十分重要的。提供基因组信息的新一代测序技术及其他组学资源的结合将使微生物表观遗传学领域的研究发生重大突破,对表观遗传修饰调控基因组信息的表达(调节蛋白结合DNA,转录起始、延长、持续合成能力和染色体结构的影响)进行更深刻的洞察,对未编码在基因组中的包括突变率、增长率、代谢变化、DNA修复、毒性、环境持久性、宿主适应性、动物疾病传染性等微生物生命周期动态变化的分子机制了解更加深入。另外,表观遗传信息也有助于宏基因组中单个基因组的组装。最后,表观遗传信息还可以应用在生物学以外的领域,比如DNA修饰随着环境变化而改变,表观遗传数据可以在医学鉴定分析中起到非常重要的作用。未来,新兴技术相结合的多组学综合研究在表观遗传学领域的挑战和机会并存。
| [1] | http://www.pacificbiosciences.com/. |
| [2] | Eid J, Fehr A, Gray J, et al. Real-time DNA sequencing from single polymerase molecules[J]. Science 2009, 323(5910) : 133–138. DOI:10.1126/science.1162986 |
| [3] | English AC, Richards S, Han Y, et al. Mind the gap: upgrading genomes with Pacific Biosciences RS long-read sequencing technology[J]. PLoS One 2012, 7(11) : e47768. DOI:10.1371/journal.pone.0047768 |
| [4] | Heiner C, Wang S, Ashby M, et al. Greater than 10 kb read lengths routine when sequencing with Pacific Biosciences' XL release[J]. Journal of Biomolecular Techniques 2013, 24(Suppl) : S43. |
| [5] | Mosher JJ, Bowman B, Bernberg EL, et al. Improved performance of the PacBio SMRT technology for 16S rDNA sequencing[J]. Journal of Microbiological Methods 2014, 104 : 59–60. DOI:10.1016/j.mimet.2014.06.012 |
| [6] | Shin SC, Ahn DH, Kim SJ, et al. Advantages of single-molecule real-time sequencing in high-GC content genomes[J]. PLoS One 2013, 8(7) : e68824. DOI:10.1371/journal.pone.0068824 |
| [7] | Flusberg BA, Webster DR, Lee JH, et al. Direct detection of DNA methylation during single-molecule, real-time sequencing[J]. Nature Methods 2010, 7(6) : 461–465. DOI:10.1038/nmeth.1459 |
| [8] | Wang LR, Chen S, Vergin KL, et al. DNA phosphorothioation is widespread and quantized in bacterial genomes[J]. Proceedings of the National Academy of Sciences of the United States of America 2011, 108(7) : 2963–2968. DOI:10.1073/pnas.1017261108 |
| [9] | Loenen WAM, Dryden DTF, Raleigh EA, et al. Highlights of the DNA cutters: a short history of the restriction enzymes[J]. Nucleic Acids Research 2014, 42(1) : 3–19. DOI:10.1093/nar/gkt990 |
| [10] | Murphy J, Mahony J, Ainsworth S, et al. Bacteriophage orphan DNA methyltransferases: insights from their bacterial origin, function, and occurrence[J]. Applied and Environmental Microbiology 2013, 79(24) : 7547–7555. DOI:10.1128/AEM.02229-13 |
| [11] | Vasu K, Nagaraja V. Diverse functions of restriction-modification systems in addition to cellular defense[J]. Microbiology and Molecular Biology Reviews 2013, 77(1) : 53–72. DOI:10.1128/MMBR.00044-12 |
| [12] | Youell J, Firman K. Mechanistic insight into Type I restriction endonucleases[J]. Frontiers in Bioscience 2011, 17(6) : 2122–2139. |
| [13] | Pingoud A, Fuxreiter M, Pingoud V, et al. Type II restriction endonucleases: structure and mechanism[J]. Cellular and Molecular Life Sciences 2005, 62(6) : 685–707. DOI:10.1007/s00018-004-4513-1 |
| [14] | Rao DN, Dryden DTF, Bheemanaik S. Type III restriction-modification enzymes: a historical perspective[J]. Nucleic Acids Research 2014, 42(1) : 45–55. DOI:10.1093/nar/gkt616 |
| [15] | Liu G, Ou HY, Wang T, et al. Cleavage of phosphorothioated DNA and methylated DNA by the type Ⅳ restriction endonuclease ScoMcrA[J]. PLoS Genetics 2010, 6(12) : e1001253. DOI:10.1371/journal.pgen.1001253 |
| [16] | Zhou XF, Deng ZX, Firmin JL, et al. Site-specific degradation of Streptomyces lividans DNA during electrophoresis in buffers contaminated with ferrous iron[J]. Nucleic Acids Research 1988, 16(10) : 4341–4352. DOI:10.1093/nar/16.10.4341 |
| [17] | Zhou XF, He XY, Liang JD, et al. A novel DNA modification by sulphur[J]. Molecular Microbiology 2005, 57(5) : 1428–1438. DOI:10.1111/j.1365-2958.2005.04764.x |
| [18] | Ray T, Mills A, Dyson P. Tris-dependent oxidative DNA strand scission during electrophoresis[J]. Electrophoresis 1995, 16(1) : 888–894. DOI:10.1002/(ISSN)1522-2683 |
| [19] | Wang LR, Chen S, Xu TG, et al. Phosphorothioation of DNA in bacteria by dnd genes[J]. Nature Chemical Biology 2007, 3(11) : 709–710. DOI:10.1038/nchembio.2007.39 |
| [20] | Chen S, Wang LR, Deng ZX. Twenty years hunting for sulfur in DNA[J]. Protein & Cell 2010, 1(1) : 14–21. |
| [21] | Ou HY, He XY, Shao YC, et al. dnd DB: a database focused on phosphorothioation of the DNA backbone[J]. PLoS One 2009, 4(4) : e5132. DOI:10.1371/journal.pone.0005132 |
| [22] | Xie XQ, Liang JD, Pu TN, et al. Phosphorothioate DNA as an antioxidant in bacteria[J]. Nucleic Acids Research 2012, 40(18) : 9115–9124. DOI:10.1093/nar/gks650 |
| [23] | Howard ST, Newman KL, McNulty S, et al. Insertion site and distribution of a genomic island conferring DNA phosphorothioation in the Mycobacterium abscessus complex[J]. Microbiology 2013, 159(11) : 2323–2332. |
| [24] | Xu TG, Yao F, Zhou XF, et al. A novel host-specific restriction system associated with DNA backbone S-modification in Salmonella[J]. Nucleic Acids Research 2010, 38(20) : 7133–7141. DOI:10.1093/nar/gkq610 |
| [25] | Tretyakova N, Villalta PW, Kotapati S. Mass spectrometry of structurally modified DNA[J]. Chemical Reviews 2013, 113(4) : 2395–2436. DOI:10.1021/cr300391r |
| [26] | Curtis M, Minier MA, Chitranshi P, et al. Direct analysis in real time (DART) mass spectrometry of nucleotides and nucleosides: elucidation of a novel fragment [C5H5O]+ and its in-source adducts[J]. Journal of the American Society for Mass Spectrometry 2010, 21(8) : 1371–1381. DOI:10.1016/j.jasms.2010.03.046 |
| [27] | Wrobel K, Wrobel K, Caruso JA. Epigenetics: an important challenge for ICP-MS in metallomics studies[J]. Analytical and Bioanalytical Chemistry 2009, 393(2) : 481–486. DOI:10.1007/s00216-008-2472-3 |
| [28] | Chapsky A, Melamed O, Galron D, et al. Detection of polymorphism in the methlyenetetrahydrofolate reductase gene by Raman spectroscopy[J]. Journal of Raman Spectroscopy 2012, 43(8) : 1083–1088. DOI:10.1002/jrs.v43.8 |
| [29] | Benoukraf T, Wongphayak S, Hadi LHA, et al. GBSA: a comprehensive software for analysing whole genome bisulfite sequencing data[J]. Nucleic Acids Research 2013, 41(4) : e55. DOI:10.1093/nar/gks1281 |
| [30] | Booth MJ, Ost TWB, Beraldi D, et al. Oxidative bisulfite sequencing of 5-methylcytosine and 5-hydroxymethylcytosine[J]. Nature Protocols 2013, 8(10) : 1841–1851. DOI:10.1038/nprot.2013.115 |
| [31] | Hebestreit K, Dugas M, Klein HU. Detection of significantly differentially methylated regions in targeted bisulfite sequencing data[J]. Bioinformatics 2013, 29(13) : 1647–1653. DOI:10.1093/bioinformatics/btt263 |
| [32] | Suzuki M, Greally JM. Genome-wide DNA methylation analysis using massively parallel sequencing technologies[J]. Seminars in Hematology 2013, 50(1) : 70–77. DOI:10.1053/j.seminhematol.2013.01.002 |
| [33] | Korlach J, Turner SW. Going beyond five bases in DNA sequencing[J]. Current Opinion in Structural Biology 2012, 22(3) : 251–261. DOI:10.1016/j.sbi.2012.04.002 |
| [34] | Booth MJ, Branco MR, Ficz G, et al. Quantitative sequencing of 5-methylcytosine and 5-hydroxymethylcytosine at single-base resolution[J]. Science 2012, 336(6083) : 934–937. DOI:10.1126/science.1220671 |
| [35] | Yu M, Hon GC, Szulwach KE, et al. Base-resolution analysis of 5-hydroxymethylcytosine in the mammalian genome[J]. Cell 2012, 149(6) : 1368–1380. DOI:10.1016/j.cell.2012.04.027 |
| [36] | Krebes J, Morgan RD, Bunk B, et al. The complex methylome of the human gastric pathogen Helicobacter pylori[J]. Nucleic Acids Research 2014, 42(4) : 2415–2432. DOI:10.1093/nar/gkt1201 |
| [37] | Bendall ML, Luong K, Wetmore KM, et al. Exploring the roles of DNA methylation in the metal-reducing bacterium Shewanella oneidensis MR-1[J]. Journal of Bacteriology 2013, 195(21) : 4966–4974. DOI:10.1128/JB.00935-13 |
| [38] | Fang G, Munera D, Friedman DI, et al. Genome-wide mapping of methylated adenine residues in pathogenic Escherichia coli using single-molecule real-time sequencing[J]. Nature Biotechnology 2012, 30(12) : 1232–1239. DOI:10.1038/nbt.2432 |
| [39] | Lluch-Senar M, Luong K, Lloréns-Rico V, et al. Comprehensive methylome characterization of Mycoplasma genitalium and Mycoplasma pneumoniae at single-base resolution[J]. PLoS Genetics 2013, 9(1) : e1003191. DOI:10.1371/journal.pgen.1003191 |
| [40] | Murray IA, Clark TA, Morgan RD, et al. The methylomes of six bacteria[J]. Nucleic Acids Research 2012, 40(22) : 11450–11462. DOI:10.1093/nar/gks891 |
| [41] | Davis BM, Chao MC, Waldor MK. Entering the era of bacterial epigenomics with single molecule real time DNA sequencing[J]. Current Opinion in Microbiology 2013, 16(2) : 192–198. DOI:10.1016/j.mib.2013.01.011 |
| [42] | Clark TA, Murray IA, Morgan RD, et al. Characterization of DNA methyltransferase specificities using single-molecule, real-time DNA sequencing[J]. Nucleic Acids Research 2012, 40(4) : e29. DOI:10.1093/nar/gkr1146 |
| [43] | Clark TA, Spittle KE, Turner SW, et al. Direct detection and sequencing of damaged DNA bases[J]. Genome Integrity 2011, 2 : 10. DOI:10.1186/2041-9414-2-10 |
| [44] | Cao B, Chen C, DeMott MS, et al. Genomic mapping of phosphorothioates reveals partial modification of short consensus sequences[J]. Nature Communications 2014, 5 : 3951. |
| [45] | Clark TA, Lu X, Luong K, et al. Enhanced 5-methylcytosine detection in single-molecule, real-time sequencing via Tet1 oxidation[J]. BMC Biology 2013, 11 : 4. DOI:10.1186/1741-7007-11-4 |
| [46] | Pais JE, Dai N, Tamanaha E, et al. Biochemical characterization of a Naegleria TET-like oxygenase and its application in single molecule sequencing of 5-methylcytosine[J]. Proceedings of the National Academy of Sciences of the United States of America 2015, 112(14) : 4316–4321. DOI:10.1073/pnas.1417939112 |
| [47] | Furuta Y, Namba-Fukuyo H, Shibata TF, et al. Methylome diversification through changes in DNA methyltransferase sequence specificity[J]. PLoS Genetics 2014, 10(4) : e1004272. DOI:10.1371/journal.pgen.1004272 |
| [48] | Mou KT, Muppirala UK, Severin AJ, et al. A comparative analysis of methylome profiles of Campylobacter jejuni sheep abortion isolate and gastroenteric strains using PacBio data[J]. Frontiers in Microbiology 2015, 5 : 782. |
| [49] | Forde BM, Phan MD, Gawthorne JA, et al. Lineage-specific methyltransferases define the methylome of the globally disseminated Escherichia coli ST131 clone[J]. mBio 2015, 6(6) : e01602-15. DOI:10.1128/mBio.01602-15 |
| [50] | Beaulaurier J, Zhang XS, Zhu SJ, et al. Single molecule-level detection and long read-based phasing of epigenetic variations in bacterial methylomes[J]. Nature Communications 2015, 6 : 7438. DOI:10.1038/ncomms8438 |
| [51] | Lee WC, Anton BP, Wang S, et al. The complete methylome of Helicobacter pyloriUM032[J]. BMC Genomics 2015, 16 : 424. DOI:10.1186/s12864-015-1585-2 |
| [52] | Pirone-Davies C, Hoffmann M, Roberts RJ, et al. Genome-wide methylation patterns in Salmonella enterica subsp. enterica serovars[J]. PLoS One 2015, 10(4) : e0123639. DOI:10.1371/journal.pone.0123639 |
| [53] | Seib KL, Jen FEC, Tan A, et al. Specificity of the ModA11, ModA12 and ModD1 epigenetic regulator N6-adenine DNA methyltransferases of Neisseria meningitidis[J]. Nucleic Acids Research 2015, 43(8) : 4150–4162. DOI:10.1093/nar/gkv219 |
| [54] | Sater MRA, Lamelas A, Wang GL. DNA methylation assessed by SMRT sequencing is linked to mutations in Neisseria meningitides isolates[J]. PLoS One 2015, 10(12) : e0144612. DOI:10.1371/journal.pone.0144612 |
| [55] | Zhang WY, Sun ZH, Menghe B, et al. Short communication: Single molecule, real-time sequencing technology revealed species-and strain-specific methylation patterns of 2 Lactobacillusstrains[J]. Journal of Dairy Science 2015, 98(5) : 3020–3024. DOI:10.3168/jds.2014-9272 |
| [56] | Huo WW, Adams HM, Zhang MQ, et al. Genome modification in Enterococcus faecalis OG1RF assessed by bisulfite sequencing and single-molecule real-time sequencing[J]. Journal of Bacteriology 2015, 197(11) : 1939–1951. DOI:10.1128/JB.00130-15 |
| [57] | Jiang K, Xue YF, Ma YH. Complete genome sequence of Salinicoccus halodurans H3B36, isolated from the Qaidam Basin in China[J]. Standards in Genomic Sciences 2015, 10 : 116. DOI:10.1186/s40793-015-0108-8 |
| [58] | Frank J, Dingemanse C, Schmitz AM, et al. The complete genome sequence of the murine pathobiont Helicobacter typhlonius[J]. Frontiers in Microbiology 2016, 6 : 1549. |
| [59] | Lim YL, Roberts RJ, Ee R, et al. Complete genome sequence and methylome analysis of Aeromonas hydrophila strain YL17, isolated from a compost pile[J]. Genome Announcements 2016, 4(2) : e00060-16. DOI:10.1128/genomeA.00060-16 |
| [60] | Ouellette M, Jackson L, Chimileski S, et al. Genome-wide DNA methylation analysis of Haloferax volcanii H26 and identification of DNA methyltransferase related PD-(D/E) XK nuclease family protein HVO_A0006[J]. Frontiers in Microbiology 2015, 6 : 251. |
| [61] | Blow MJ, Clark TA, Daum CG, et al. The epigenomic landscape of prokaryotes[J]. PLoS Genetics 2016, 12(2) : e1005854. DOI:10.1371/journal.pgen.1005854 |
| [62] | van Luenen HGAM, Farris C, Jan S, et al. Glucosylated hydroxymethyluracil, DNA base J, prevents transcriptional readthrough in Leishmania[J]. Cell 2012, 150(5) : 909–921. DOI:10.1016/j.cell.2012.07.030 |
 2017, Vol. 44
2017, Vol. 44