扩展功能
文章信息
- 熊彬彬, 曾虎, 刘云坤, 孙宇辉
- XIONG Bin-Bin, ZENG Hu, LIU Yun-Kun, SUN Yu-Hui
- CRISPR-Cas9驱动的基因编辑新纪元
- New era of gene editing driven by CRISPR-Cas9
- 微生物学通报, 2017, 44(1): 178-185
- Microbiology China, 2017, 44(1): 178-185
- DOI: 10.13344/j.microbiol.china.160128
-
文章历史
- 收稿日期: 2016-02-02
- 接受日期: 2016-03-18
- 优先数字出版日期(www.cnki.net): 2016-04-13
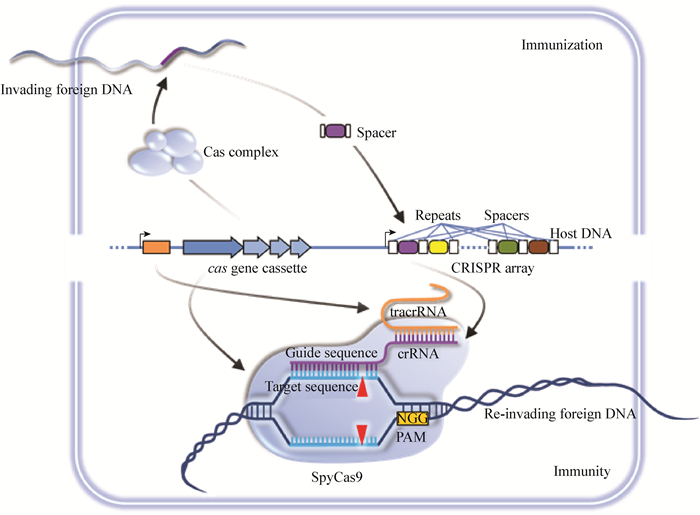
在自然界生物长期的进化过程中,细菌和古细菌演化出了一种独特的适应性免疫系统。在该系统中,一种称之为Cas的限制性内切酶能在短链RNA的引导下,靶向降解外来入侵的DNA或RNA[1-4],人们将这一系统命名为CRISPR-Cas,即成簇规律间隔的短回文重复序列及其相关基因(Clustered regularly interspaced short palindromic repeat sequences and CRISPR-associated genes,CRISPR-Cas)。回顾以往,这一成簇规律间隔的短回文重复序列于1987年首先在Escherichia coli染色体上被发现[5],然而其蕴藏的生物学意义似乎并没有立即被人们所了解。之后的研究显示,CRISPR其实在约40%的细菌和90%的古细菌中都普遍存在[6]。直到2005年,CRISPR与抗病毒免疫之间的联系才逐渐被人们认识到[7-9]。2007年,对Steptococcus thermophilus中CRISPR的研究揭示出了间隔序列对抗病毒免疫的重要作用[10]。2008年,CRISPR RNA (crRNA)在整个免疫过程中的作用也被发现[11]。2011年,Cas9蛋白在Ⅱ型CRISPR免疫系统中的功能被首次揭示[12]。2012年,人们认识到CRISPR-Cas9实质上是一种由RNA引导的核酸内切酶[13]。2013年,CRISPR-Cas9开始被作为一种基因编辑技术而被广泛使用[14-16]。在CRISPR-Cas9技术被开发之前,主要有两种应用于基因编辑的人工核酸内切酶系统,即锌指核酸内切酶(Zinc finger endonuclease,ZFNs)系统以及类转录激活因子效应物核酸酶(Transcription activator-like effector nuclease,TALENs)系统。ZFNs由锌指DNA结合域和相应限制性内切酶切割结构域组成,从而可识别特定DNA结合位点并进行切割,其优点是定点整合效率高,但因其构建难度大、费用高的缺点,使其应用受到很大的限制[17];TALENs由TALE蛋白和核酸内切酶FokI组成,TALE蛋白可识别两条反向的DNA位点,FokI配对后二聚体化切割DNA,TALENs技术相比ZFNs技术构建更为简单,细胞毒性较低,但构建成本依然偏高,其应用也有局限性[18]。而CRISPR-Cas9技术相较而言设计更加简单、操作更便捷、基因编辑效率更高,因此该技术被开发后在各个领域都迅速得到了广泛应用。随着Cas9晶体结构研究工作的开展,CRISPR-Cas9的作用机制也被逐渐阐释[19-23]。人们已经认识到在相继与单链引导RNA (sgRNA)及靶标DNA序列结合的过程中,Cas9蛋白将发生构象重排。这使得Cas9蛋白的两个具有切割活性的结构域(RuvC和HNH)之间形成一个可与RNA:DNA互补配对体以及与RNA双链体结合的通道。其中Cas9蛋白上的与原间隔区临近基序(Protospacer adjacent motif,PAM)相互作用的结构域和sgRNA上的核心序列(Seed sequence)共同作用,识别PAM位点,导致靠近PAM位点的DNA双链解链。sgRNA上的核心序列与靶标DNA序列发生碱基互补配对,使Cas9蛋白进入近活性构象。而当sgRNA上的引导序列与靶标DNA序列全部配对完成时,Cas9蛋白即进入完全活性状态,RuvC和HNH结构域分别切割DNA两条单链,造成双链DNA断裂(图 1)。根据Cas蛋白类型和CRISPR构成特点,目前已经发现至少存在三种类型的CRISPR-Cas:Ⅰ、Ⅱ和Ⅲ型[4]。其中,Ⅰ和Ⅲ型CRISPR-Cas只需要一条crRNA来引导Cas蛋白与靶标位点的结合与切割;而Ⅱ型CRISPR-Cas系统还需要反式激活CRISPR RNA (Trans-activating CRISPR RNA,tracrRNA)的辅助[24]。Ⅰ和Ⅱ型CRISPR-Cas系统可以降解外源DNA,而Ⅲ型CRISPR-Cas系统不仅可以降解外源DNA,还可以降解外源RNA。另外,Ⅰ和Ⅲ型CRISPR-Cas系统介导的外源核酸的降解需要多种Cas蛋白共同参与[11, 25],而Ⅱ型CRISPR-Cas系统则只需要一个单一的Cas9蛋白[12],它的这一特点,使得Ⅱ型Cas9迅速在生物学领域得到了广泛的应用。
CRISPR-Cas自发现以来,虽然时间并不算长,但其所迸发出来的巨大潜力却是人们所始料未及的。如今这一简便、高效的技术已风靡于生物学各重要研究领域,并被广泛应用于多种生物和细胞的基因编辑、基因调节和基因修饰中。本文重点综述了近年来CRISPR-Cas9系统在基因编辑、基因调节以及作为体外工具酶和特异性等方面的若干前沿进展。
1 CRISPR-Cas9基因编辑技术机理与体内应用在Ⅱ型CRISPR-Cas系统中,RNA双链体tracrRNA:crRNA中的靶向序列与DNA靶标序列碱基互补配对,引导CRISPR相关蛋白Cas9对DNA进行特异性切割,导致DNA双链断裂[13, 26](图 1)。RNA双链体tracrRNA:crRNA可被融合为单链引导sgRNA而仍保有其功能。sgRNA的5′端序列与靶标DNA互补配对,3′端的双链RNA茎环结构绑定Cas9蛋白。因此通过改变sgRNA的引导序列,即可引导Cas9蛋白靶向切割任意的在3′端存在PAM位点的DNA序列[8]。在不存在外源同源重组模板的情况下,DNA双链断裂将激发生物体内的非同源末端连接修复(Non-homologous end-joining,NHEJ),在DNA断裂处引入小片段的插入或缺失,导致移码突变。当有外源同源重组模板存在时,DNA双链断裂可引发同源重组修复(Homology-directed repair,HDR),实现对断裂位点依照同源片段进行精确修复。由CRISPR-Cas系统切割DNA所引发的这些修复给人们设计符合各类愿望的基因编辑创造了巨大的空间,这一基因编辑方法如今已成功应用于包括水稻、小麦、疟原虫、果蝇、斑马鱼以及哺乳动物等真核生物中。例如,Shan等通过CRISPR-Cas系统完成了对水稻和小麦的定点基因修饰,成功使水稻基因组上的4个基因失去功能,并敲除了小麦基因组上的一个基因使小麦获得了白粉病抗性,表明此系统可高效地完成植物体内基因编辑工作[27]。Ghorbal等用CRISPR-Cas系统对疟原虫进行了体内染色组定点敲除以及完成了高效率的单核苷酸替换工作,为人类抗击疟疾提供了一种对疟原虫基因更加高效准确的敲除方法[28]。Ren等通过选用不同的启动子以及调整其与sgRNA的拼接方式,成功构建了一套可对果蝇进行体内高效基因编辑的系统[29]。Hwang等在斑马鱼胚胎中成功利用CRISPR-Cas系统在体内完成了基因定点修饰,证明此系统可为脊索动物研究提供合适的模式生物[30]。Cong等基于CRISPR-Cas系统分别完成了在小鼠细胞和人类细胞内对基因组定点位置进行精确切割并引入相应突变的工作[14]。Niu等通过CRISPR-Cas系统成功对孪生食蟹猴进行了精确的基因编辑,且因为灵长类动物与人类基因具有高度的同源性,此工作对一些相关疾病动物模型的构建具有重要意义[31]。不仅在真核生物中,此系统在原核生物,如大肠杆菌以及微生物药物重要来源的链霉菌[32-34]等体内基因功能研究方面也显示出优于其它现有技术之处。Jiang等利用CRISPR-Cas系统在原核生物中对基因组的切割具有致死性这一特点,实现了对肺炎链球菌和大肠杆菌的无筛选标记的基因组编辑[35]。作者课题组利用来自Streptococcus pyogenes并经密码子优化后的Cas9与胞嘧啶脱氨酶CodA反向筛选系统联用,建立了一种快速高效的链霉菌基因组编辑和筛选技术,并以模式链霉菌Streptomyces coelicolor M145中放线紫红素聚酮链延长因子基因actI-ORF2为例,运用这一基因编辑筛选系统成功实现了对靶标基因快速高效的的同框敲除。只要具备成熟的遗传操作系统,均可尝试使用此系统来进行基因敲除工作。该系统显著提高了无标记突变株的获得频率并大大缩短了筛选所需的时间,为利用合成生物学发掘来自链霉菌巨大的天然药物合成潜力提供了重要的工具[36]。
|
| 图 1 CRISPR-Cas免疫系统示意图(以Ⅱ型SpyCas9为例) Figure 1 CRISPR-Cas immune system (using SpyCas9 from type II as example) |
|
|
相对于NHEJ这种较为随机的双链DNA断裂修复方式,HDR可在DNA靶标位点定向引入目标外源DNA序列,进行更为可控和精确的修复。因此,研究者们期望HDR的效率更高。但是在脊椎动物细胞中,HDR的效率一般远低于NHEJ的效率[37-39]。所以,通过基因沉默或是加入抑制剂等手段抑制细胞中参与NHEJ修复的关键蛋白,如Ku70、Ku80、Ligase Ⅳ等,可显著增强HDR的效率[40-41],从而实现预期的基因编辑。
2 基于活性改造的CRISPR-Cas9系统在基因编辑和调节方面的应用Ⅱ型CRISPR-Cas系统除了主要应用于体内基因编辑之外,利用Cas9在sgRNA的引导下能够特异性地结合到靶标DNA的特点,将Cas9中负责切割双链DNA的结构域HNH和RuvC都进行失活,即得到没有切割活性而只保留有DNA识别和结合活性的dCas9。根据dCas9的这一特点,可将其应用于基因调节。在不同sgRNA的引导下,无切割活性的dCas9蛋白能定向结合在目标基因的不同区域,根据结合的位置可阻遏RNA聚合酶与转录模板的结合、转录的延伸或是转录因子的结合,最终达到在转录水平上抑制基因表达的作用。这一方法类似于RNA干扰(RNA interference,RNAi),因此被称为CRISPRi (CRISPR interference)。相比于CRISPR-Cas9介导的突变,CRISPRi重要的特点之一是它不仅具有较高的特异性[26],还可同时抑制多个基因的表达,而且这种抑制作用是可逆的[42-45]。
此外,还可将一些具有特定调节功能的结构域如VP64、KRAB与dCas9共价融合,这些dCas9的嵌合体可在sgRNA的引导下,结合到相应的靶标DNA位点,激活或抑制目标基因的转录[44]。而通过在dCas9或sgRNA上融合一连串调节蛋白或RNA适配子,借助CRISPR-Cas9系统对DNA的定位识别功能,将调节蛋白绑定到目标区域附近,可灵活地对距离sgRNA靶标序列不同间距的位点进行不同强度的调节和修饰[46-48]。这些可编程的调节技术对于合成生物学和代谢工程学等领域具有极其重要的价值和意义。
利用对DNA靶向结合的特性,dCas9也可以用来对染色体上特定的基因进行定位。例如将dCas9与绿色荧光蛋白(Green fluorescent protein,GFP)融合,产生的dCas9-GFP融合体可以用来对活细胞中的DNA靶点进行示踪[49]。这一技术的应用给人们研究染色体构象和动力学提供了新的视角。
另据报道,dCas9在人为提供的含PAM的寡核苷酸(PAM-presenting oligonucleotides, PAMmer)存在情况下,也可与单链RNA靶标结合,且该靶标RNA不需要有PAM的存在[50]。这预示着在不久的将来,人们也许可以利用dCas9对细胞内的RNA转录本进行程序化的编辑,从而进一步扩大CRISPR-Cas系统的应用空间。
无论是Cas9介导的基因编辑还是dCas9介导的基因调节,都可以方便地被用来阻断特定基因的表达,既可以对单一基因的功能进行研究,也可以用于在细胞内进行染色体规模的基因功能考察。在人类细胞和小鼠细胞中,已有报道利用Cas9和慢病毒sgRNA文库的介导来对基因组规模的基因敲除进行筛选[51-54]。dCas9和相应的sgRNA文库组合,通过上调或者下调基因的表达水平,可以在全基因组水平上进行高通量筛选[46-47]。相对于Cas9介导的高通量筛选,dCas9介导的转录沉默可能对某些非编码RNA的研究更适用。而dCas9与转录激活因子介导的转录激活在功能获得性筛选方面,则具有Cas9介导的高通量筛选及其他技术所不能替代的地位。
3 CRISPR-Cas9系统作为体外工具酶的应用自CRISPR-Cas9系统在体内研究得到广泛应用以来,因Cas9蛋白靶向的特异性以及操作的简易性等优势,使其也拥有了作为体外工具酶的巨大潜力。利用Cas9蛋白与引导RNA相结合,识别并切割特定DNA序列的特点,Cas9系统可成功与体外重组系统进行联用,直接从染色体上精确克隆片段DNA[55]。Cas9系统也被证明可以成功与体内重组系统联用进行大片段DNA的精确克隆[56]。除此之外,Cas9系统还被用于与体外重组系统联用进行无缝克隆[57]。其原理均为使用Cas9蛋白和引导RNA切割特定序列的目标DNA,暴露出两端特异序列的同源臂,该同源臂可以与经设计带有与之匹配的同源臂的载体在体内或体外完成同源重组,从而进行大片段的组装。
Cas9系统在体内或体外与同源重组系统等技术的联用,除了在传统克隆技术难以胜任的一些领域展现出其独特魅力之外,它还被迅速扩展到常规的分子克隆实验之中,为人们提供了更多的技术和策略选择。例如利用来自Streptococcus thermophilus的Cas9 (StCas9),根据它可以精确编程并切割双链DNA产生平端的双链DNA的特性,从而可以方便地与PCR扩增的DNA片段酶连产生重组质粒,由此完成StCas9介导的DNA编辑[58]。对于更为通用的来自Streptococcus pyogenes的Cas9 (SpCas9),其作用特点与StCas9基本一致,但是却具有3′→5′外切酶活性,容易导致经SpCas9切割后的DNA末端产生少量碱基缺失,从而影响后续精确的克隆。针对SpCas9的这一缺陷,利用T4 DNA聚合酶对经由SpCas9切割后的DNA末端进行补齐,可以明显缓解这种非平末端带来的问题,由此提高后续克隆和基因编辑的精确度[59]。
4 CRISPR-Cas9系统在靶向特异性方面的研究进展CRISPR-Cas9作为一种高效的体内和体外编辑系统,其优越性已经得到充分的体现,然而,该系统也不可避免地存在其自身的弱点,即该系统的靶向特异性一直受到研究者们的密切关注。为了提高系统特异性,降低脱靶率(Off-target),一系列方法被开发出来。这些方法可以大致分为对Cas9蛋白的改造和对sgRNA的改造两大类。
对Cas9蛋白的改造来提高CRISPR-Cas9系统的特异性,可以通过失活Cas9中切割与sgRNA引导序列非互补链的结构域RuvC,将Cas9变成了只能切割DNA单链的切刻酶Cas9n[16, 29, 60]。然后在一对相隔一定间距的sgRNA的引导下,Cas9n在两个靶标DNA位点发生切刻。因为由单一sgRNA引发的脱靶切刻能被精确修复而不造成非特异性突变,因此,成对sgRNA引导的Cas9n能显著提高基因编辑的特异性而降低了脱靶率。
通过对Cas9蛋白的改造来提高CRISPR-Cas9系统的特异性,还可以将dCas9与ForkI融合,在一对相隔一定间距的sgRNA的引导下,dCas9-ForkI融合体便以类似ZFNs和TALENs的方式引入突变,即dCas9-ForkI在目标突变区域上下游结合DNA,形成ForkI二聚体,由该ForkI二聚体切割DNA双链,引入突变[61-62]。由于一条sgRNA引导的dCas9-ForkI脱靶结合无法切割DNA,所以成对sgRNA引导的dCas9-ForkI也可提高基因编辑的特异性。但是无论是Cas9n还是dCas9-ForkI,目标突变区域的上下游必需同时包含可供Cas9识别的序列,这使得它们靶标序列选择的灵活性受到了一定的限制。针对这一不足,人们将一个可编程的DNA结合结构域(Programmable DNA-binding domain,pDBD)与Cas9融合,这样可同时降低Cas9本身与DNA结合的亲和性,产生Cas9-pDBD嵌合体。由于DNA识别的特异性与亲和性可由pDBD调节,因此Cas9-pDBD嵌合体在基因编辑的特异性和靶标位点的选择性方面都得到了明显的提高[63]。
通过使用5′端截短的sgRNA也可以提高CRISPR-Cas9系统基因编辑的特异性[64]。这是因为靠近sgRNA 5′端的引导序列对于sgRNA的活性不是必需的,反而有可能与潜在的脱靶位点形成错配,造成脱靶切割。所以,使用5′端截短的sgRNA也可提高CRISPR-Cas9系统的特异性。
另外,还有通过将Cas9的mRNA、sgRNA以及包含用于同源重组的质粒混合体直接注射到待编辑细胞中来提高筛选效率。其原理是由于有些细胞难以通过常规转染的方式导入外源DNA片段,然而通过体外直接注射的方式可以直接将充足的所需基因编辑元件导入细胞体内,从而克服了转染的障碍,达到基因高效编辑的目的。该方法的另一个重要目的是由于这样避免了Cas9在体内的持续表达,从而减少了Cas9发挥作用的时间,由此减少脱靶的产生[65]。该方法已在隐杆秀丽线虫、多功能干细胞、斑马鱼等模式生物中得到成功使用[66-68]。
5 总结与展望近几年来,无论是在机理研究还是在应用方面,CRISPR-Cas都表现出了日新月异的迅猛发展势头。在DNA测序技术快速发展的今天,数量更多的菌株全基因组测序得以完成。通过生物信息学分析,人们在更多的生物基因组中寻找到了CRISPR-Cas和类似家族的身影,特别是在一些关系到人类健康安全的致病菌中也不例外[69-71],通过这些致病菌中CRISPR-Cas系统的探索,将可能对病原微生物在分类、毒性与耐药性方面的研究有所启示,在寻找制服与人类朝夕相处的病原微生物方面提供新的思路和策略。另外,随着Cas9晶体结构的解析,CRISPR-Cas9作用机制已经被深入阐述,通过对Cas9蛋白的功能定向改造,使其在靶标位点选择的灵活性和切割的特异性方面都得到极大的改善[72-73]。通过对不同微生物来源的Cas9蛋白的了解,研究者也发现这些新颖的家族成员的特点并非千篇一律,如能识别更长PAM位点、“体型”更小巧的来自于Staphylococcus aureus的Cas9。相对于来自Streptococcus pyogenes的SpyCas9,这种新型的Cas9蛋白的靶标选择范围扩大了2-4倍,且切割的特异性更高。CRISPR-Cas9以其简便、高效、灵活的特性,已经在生物学各领域取得了令人瞩目的成就,而其生物学潜能更有待于进一步开发。相信在不久的将来,CRISPR-Cas9将如同PCR和DNA测序等技术一样,成为具有划时代意义的另一生物学技术重要里程碑。
| [1] | Bhaya D, Davison M, Barrangou R. CRISPR-Cas systems in bacteria and archaea: versatile small RNAs for adaptive defense and regulation[J]. Annual Review of Genetics 2011, 45(1) : 273–297. DOI:10.1146/annurev-genet-110410-132430 |
| [2] | Deveau H, Garneau JE, Moineau S. CRISPR/Cas system and its role in phage-bacteria interactions[J]. Annual Review of Microbiology 2010, 64(1) : 475–493. DOI:10.1146/annurev.micro.112408.134123 |
| [3] | Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea[J]. Science 2010, 327(5962) : 167–170. DOI:10.1126/science.1179555 |
| [4] | Makarova KS, Haft DH, Barrangou R, et al. Evolution and classification of the CRISPR-Cas systems[J]. Nature Reviews Microbiology 2011, 9(6) : 467–477. DOI:10.1038/nrmicro2577 |
| [5] | Ishino Y, Shinagawa H, Makino K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product[J]. Journal of Bacteriology 1987, 169(12) : 5429–5433. DOI:10.1128/jb.169.12.5429-5433.1987 |
| [6] | Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J]. BMC Bioinformatics 2007, 8(1) : 172. DOI:10.1186/1471-2105-8-172 |
| [7] | Bolotin A, Quinquis B, Sorokin A, et al. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin[J]. Microbiology 2005, 151(8) : 2551–2561. DOI:10.1099/mic.0.28048-0 |
| [8] | Mojica FJM, Díez-Villase or C, García-Martínez J, et al. Short motif sequences determine the targets of the prokaryotic CRISPR defence system[J]. Microbiology 2009, 155(3) : 733–740. DOI:10.1099/mic.0.023960-0 |
| [9] | Pourcel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies[J]. Microbiology 2005, 151(3) : 653–663. DOI:10.1099/mic.0.27437-0 |
| [10] | Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science 2007, 315(5819) : 1709–1712. DOI:10.1126/science.1138140 |
| [11] | Brouns SJJ, Jore MM, Lundgren M, et al. Small CRISPR RNAs guide antiviral defense in prokaryotes[J]. Science 2008, 321(5891) : 960–964. DOI:10.1126/science.1159689 |
| [12] | Sapranauskas R, Gasiunas G, Fremaux C, et al. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli[J]. Nucleic Acids Research 2011, 39(21) : 9275–9282. DOI:10.1093/nar/gkr606 |
| [13] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science 2012, 337(6096) : 816–821. DOI:10.1126/science.1225829 |
| [14] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science 2013, 339(6121) : 819–823. DOI:10.1126/science.1231143 |
| [15] | Jinek M, East A, Cheng A, et al. RNA-programmed genome editing in human cells[J]. eLife 2013, 2 : e00471. |
| [16] | Mali P, Aach J, Stranges PB, et al. CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering[J]. Nature Biotechnology 2013, 31(9) : 833–838. DOI:10.1038/nbt.2675 |
| [17] | Miller JC, Holmes MC, Wang JB, et al. An improved zinc-finger nuclease architecture for highly specific genome editing[J]. Nature Biotechnology 2007, 25(7) : 778–785. DOI:10.1038/nbt1319 |
| [18] | Miller JC, Tan SY, Qiao GJ, et al. A TALE nuclease architecture for efficient genome editing[J]. Nature Biotechnology 2011, 29(2) : 143–148. DOI:10.1038/nbt.1755 |
| [19] | Nishimasu H, Cong L, Yan WX, et al. Crystal structure of Staphylococcus aureus Cas9[J]. Cell 2015, 162(5) : 1113–1126. DOI:10.1016/j.cell.2015.08.007 |
| [20] | Nishimasu H, Ran FA, Hsu PD, et al. Crystal structure of Cas9 in complex with guide RNA and target DNA[J]. Cell 2014, 156(5) : 935–949. DOI:10.1016/j.cell.2014.02.001 |
| [21] | Anders C, Niewoehner O, Duerst A, et al. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease[J]. Nature 2014, 513(7519) : 569–573. DOI:10.1038/nature13579 |
| [22] | Jinek M, Jiang FG, Taylor DW, et al. Structures of Cas9 endonucleases reveal RNA-mediated conformational activation[J]. Science 2014, 343(6176) : 1247997. DOI:10.1126/science.1247997 |
| [23] | Jiang FG, Zhou KH, Ma LL, et al. A Cas9-guide RNA complex preorganized for target DNA recognition[J]. Science 2015, 348(6242) : 1477–1481. DOI:10.1126/science.aab1452 |
| [24] | Deltcheva E, Chylinski K, Sharma CM, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ[J]. Nature 2011, 471(7340) : 602–607. DOI:10.1038/nature09886 |
| [25] | Hale CR, Zhao P, Olson S, et al. RNA-guided RNA cleavage by a CRISPR RNA-Cas protein complex[J]. Cell 2009, 139(5) : 945–956. DOI:10.1016/j.cell.2009.07.040 |
| [26] | Gasiunas G, Barrangou R, Horvath P, et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America 2012, 109(39) : E2579–E2586. DOI:10.1073/pnas.1208507109 |
| [27] | Shan QW, Wang YP, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system[J]. Nature Biotechnology 2013, 31(8) : 686–688. DOI:10.1038/nbt.2650 |
| [28] | Ghorbal M, Gorman M, Macpherson CR, et al. Genome editing in the human malaria parasite Plasmodium falciparum using the CRISPR-Cas9 system[J]. Nature Biotechnology 2014, 32(8) : 819–821. DOI:10.1038/nbt.2925 |
| [29] | Ren XJ, Sun J, Housden BE, et al. Optimized gene editing technology for Drosophila melanogasterusing germ line-specific Cas9[J]. Proceedings of the National Academy of Sciences of the United States of America 2013, 110(47) : 19012–19017. DOI:10.1073/pnas.1318481110 |
| [30] | Hwang WY, Fu YF, Reyon D, et al. Efficient genome editing in zebrafish using a CRISPR-Cas system[J]. Nature Biotechnology 2013, 31(3) : 227–229. DOI:10.1038/nbt.2501 |
| [31] | Niu YY, Shen B, Cui YQ, et al. Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos[J]. Cell 2014, 156(4) : 836–843. DOI:10.1016/j.cell.2014.01.027 |
| [32] | Cobb RE, Wang YJ, Zhao HM. High-efficiency multiplex genome editing of Streptomyces species using an engineered CRISPR/Cas system[J]. ACS Synthetic Biology 2015, 4(6) : 723–728. |
| [33] | Huang H, Zheng GS, Jiang WH, et al. One-step high-efficiency CRISPR/Cas9-mediated genome editing in Streptomyces[J]. Acta Biochimica et Biophysica Sinica 2015, 47(4) : 231–243. DOI:10.1093/abbs/gmv007 |
| [34] | Tong YJ, Charusanti P, Zhang LX, et al. CRISPR-Cas9 based engineering of actinomycetal genomes[J]. ACS Synthetic Biology 2015, 4(9) : 1020–1029. DOI:10.1021/acssynbio.5b00038 |
| [35] | Jiang WY, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nature Biotechnology 2013, 31(3) : 233–239. DOI:10.1038/nbt.2508 |
| [36] | Zeng H, Wen SS, Xu W, et al. Highly efficient editing of the actinorhodin polyketide chain length factor gene in Streptomyces coelicolor M145 using CRISPR/Cas9-CodA (sm) combined system[J]. Applied Microbiologyand Biotechnology 2015, 99(24) : 10575–10585. DOI:10.1007/s00253-015-6931-4 |
| [37] | Yang H, Wang HY, Shivalila CS, et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering[J]. Cell 2013, 154(6) : 1370–1379. DOI:10.1016/j.cell.2013.08.022 |
| [38] | Wang HY, Yang H, Shivalila CS, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering[J]. Cell 2013, 153(4) : 910–918. DOI:10.1016/j.cell.2013.04.025 |
| [39] | Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9[J]. Science 2013, 339(6121) : 823–826. DOI:10.1126/science.1232033 |
| [40] | Chu VT, Weber T, Wefers B, et al. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells[J]. Nature Biotechnology 2015, 33(5) : 543–548. DOI:10.1038/nbt.3198 |
| [41] | Maruyama T, Dougan SK, Truttmann MC, et al. Increasing the efficiency of precise genome editing with CRISPR-Cas9 by inhibition of nonhomologous end joining[J]. Nature Biotechnology 2015, 33(5) : 538–542. DOI:10.1038/nbt.3190 |
| [42] | Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platformfor sequence-specific control of gene expression[J]. Cell 2013, 152(5) : 1173–1183. DOI:10.1016/j.cell.2013.02.022 |
| [43] | Zhao YC, Dai Z, Liang Y, et al. Sequence-specific inhibition of microRNA via CRISPR/CRISPRi system[J]. Scientific Reports 2014, 4 : 3943. |
| [44] | Gilbert LA, Larson MH, Morsut L, et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes[J]. Cell 2013, 154(2) : 442–451. DOI:10.1016/j.cell.2013.06.044 |
| [45] | Bikard D, Jiang WY, Samai P, et al. Programmable repression and activation of bacterial gene expression using an engineered CRISPR-Cas system[J]. Nucleic Acids Research 2013, 41(15) : 7429–7437. DOI:10.1093/nar/gkt520 |
| [46] | Gilbert LA, Horlbeck MA, Adamson B, et al. Genome-scale CRISPR-mediated control of gene repression and activation[J]. Cell 2014, 159(3) : 647–661. DOI:10.1016/j.cell.2014.09.029 |
| [47] | Konermann S, Brigham MD, Trevino AE, et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex[J]. Nature 2015, 517(7536) : 583–588. |
| [48] | Zalatan JG, Lee ME, Almeida R, et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds[J]. Cell 2015, 160(1/2) : 339–350. |
| [49] | Chen BH, Gilbert LA, Cimini BA, et al. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system[J]. Cell 2013, 155(7) : 1479–1491. DOI:10.1016/j.cell.2013.12.001 |
| [50] | O'Connell MR, Oakes BL, Sternberg SH, et al. Programmable RNA recognition and cleavage by CRISPR/Cas9[J]. Nature 2014, 516(7530) : 263–266. DOI:10.1038/nature13769 |
| [51] | Koike-Yusa H, Li YL, Tan EP, et al. Genome-wide recessive genetic screening in mammalian cells with alentiviral CRISPR-guide RNA library[J]. Nature Biotechnology 2014, 32(3) : 267–273. |
| [52] | Shalem O, Sanjana NE, Hartenian E, et al. Genome-scale CRISPR-Cas9 knockout screening in human cells[J]. Science 2014, 343(6166) : 84–87. DOI:10.1126/science.1247005 |
| [53] | Wang T, Wei JJ, Sabatini DM, et al. Genetic screens in human cells using the CRISPR-Cas9 system[J]. Science 2014, 343(6166) : 80–84. DOI:10.1126/science.1246981 |
| [54] | Zhou YX, Zhu SY, Cai CZ, et al. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells[J]. Nature 2014, 509(7501) : 487–491. DOI:10.1038/nature13166 |
| [55] | Jiang WJ, Zhao XJ, Gabrieli T, et al. Cas9-Assisted Targeting of CHromosome segments CATCH enables one-step targeted cloning of large gene clusters[J]. Nature Communications 2015, 6 : 8101. DOI:10.1038/ncomms9101 |
| [56] | Lee NCO, Larionov V, Kouprina N. Highly efficient CRISPR/Cas9-mediated TAR cloning of genes and chromosomal loci from complex genomes in yeast[J]. Nucleic Acids Research 2015, 43(8) : e55. DOI:10.1093/nar/gkv112 |
| [57] | Wang JW, Wang A, Li KY, et al. CRISPR/Cas9 nuclease cleavage combined with Gibson assembly for seamless cloning[J]. BioTechniques 2015, 58(4) : 161–170. |
| [58] | Karvelis T, Gasiunas G, Siksnys V. Programmable DNA cleavage in vitro by Cas9[J]. Biochemical Society Transactions 2013, 41(6) : 1401–1406. DOI:10.1042/BST20130164 |
| [59] | Liu YK, Tao WX, Wen SS, et al. In vitro CRISPR/Cas9 system for efficient targeted DNA editing[J]. mBio 2015, 6(6) : e01714–e01715. |
| [60] | Shen B, Zhang WS, Zhang J, et al. Efficient genome modification by CRISPR-Cas9 nickase with minimal off-target effects[J]. Nature Methods 2014, 11(4) : 399–402. DOI:10.1038/nmeth.2857 |
| [61] | Guilinger JP, Thompson DB, Liu DR. Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification[J]. Nature Biotechnology 2014, 32(6) : 577–582. DOI:10.1038/nbt.2909 |
| [62] | Tsai SQ, Wyvekens N, Khayter C, et al. Dimeric CRISPR RNA-guided FokI nucleases for highly specific genome editing[J]. Nature Biotechnology 2014, 32(6) : 569–576. DOI:10.1038/nbt.2908 |
| [63] | Bolukbasi MF, Gupta A, Oikemus S, et al. DNA-binding-domain fusions enhance the targeting range and precision of Cas9[J]. Nature Methods 2015, 12(12) : 1150–1156. DOI:10.1038/nmeth.3624 |
| [64] | Fu YF, Sander JD, Reyon D, et al. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs[J]. Nature Biotechnology 2014, 32(3) : 279–284. DOI:10.1038/nbt.2808 |
| [65] | Aida T, Chiyo K, Usami T, et al. Cloning-free CRISPR/Cas system facilitates functional cassette knock-in in mice[J]. Genome Biology 2015, 16(1) : 87. DOI:10.1186/s13059-015-0653-x |
| [66] | Paix A, Folkmann A, Rasoloson D, et al. High efficiency, homology-directed genome editingin Caenorhabditis elegansusing CRISPR-Cas9 ribonucleoprotein complexes[J]. Genetics 2015, 201(1) : 47–54. DOI:10.1534/genetics.115.179382 |
| [67] | Seah YFS, El Farran CA, Warrier T, et al. Induced pluripotency and gene editing in disease modelling: perspectives and challenges[J]. International Journal of Molecular Sciences 2015, 16(12) : 28614–28634. DOI:10.3390/ijms161226119 |
| [68] | Kotani H, Taimatsu K, Ohga R, et al. Efficient multiple genome modifications induced by the crRNAs, tracrRNA and Cas9 protein complex in zebrafish[J]. PLoS One 2015, 10(5) : e0128319. DOI:10.1371/journal.pone.0128319 |
| [69] | Burley KM, Sedgley CM. CRISPR-Cas, a prokaryotic adaptive immune system, in endodontic, oral, and multidrug-resistant hospital-acquired Enterococcus faecalis[J]. Journal of Endodontics 2012, 38(11) : 1511–1515. DOI:10.1016/j.joen.2012.07.004 |
| [70] | Gao ZQ, Wang DS, Feng EL, et al. Clustered regularly interspaced short palindromic repeats (CRISPR) site in Bacillus anthracis[J]. Acta Microbiologica Sinica 2014, 54(11) : 1362–1368. (in Chinese) 高志奇, 王东澍, 冯尔玲, 等. 炭疽芽胞杆菌中CRISPR位点[J]. 微生物学报 2014, 54(11) : 1362–1368. |
| [71] | Guo XJ, Wang YF, Duan GC, et al. Distribution of CRISPR/Cas system in Shigella clinical strains and its relationship with virulence genes[J]. Microbiology China 2015, 42(3) : 543–549. (in Chinese) 郭向娇, 王颖芳, 段广才, 等. 临床分离志贺菌中CRISPR/Cas系统的分布及其与毒力基因的关系[J]. 微生物学通报 2015, 42(3) : 543–549. |
| [72] | Kleinstiver BP, Prew MS, Tsai SQ, et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities[J]. Nature 2015, 523(7561) : 481–485. DOI:10.1038/nature14592 |
| [73] | Kleinstiver BP, Prew MS, Tsai SQ, et al. Broadening the targeting range of Staphylococcus aureusCRISPR-Cas9 by modifying PAM recognition[J]. Nature Biotechnology 2015, 33(12) : 1293–1298. DOI:10.1038/nbt.3404 |
 2017, Vol. 44
2017, Vol. 44