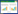扩展功能
文章信息
- 于新惠, 张颖, 王文静, 罗勤
- YU Xin-Hui, ZHANG Ying, WANG Wen-Jing, LUO Qin
- rmlB基因在单核细胞增生李斯特菌耐药性、生物被膜形成和毒力方面的作用
- Contribution of rmlB in envelope-acting antibiotic resistance, biofilm formation and virulence in Listeria monocytogenes
- 微生物学通报, 2017, 44(1): 161-171
- Microbiology China, 2017, 44(1): 161-171
- DOI: 10.13344/j.microbiol.china.160121
-
文章历史
- 收稿日期: 2016-01-31
- 接受日期: 2016-04-14
- 优先数字出版日期(www.cnki.net): 2016-05-04
单核细胞增生李斯特菌(Listeria monocytogenes,以下简称Lm)是一种短小的革兰氏阳性无芽胞兼性厌氧杆菌,属于李斯特菌属,是人畜共患传染病李斯特菌病(Listeriosis)的主要病原菌,能引起人和动物脑膜炎、败血症、流产和单核细胞增多等症状[1]。Lm在自然界分布广泛,并能在多数固体表面(如食品及食品加工运输和储藏的接触面,甚至非食品加工接触面)形成高度组织化和系统化的生物被膜(Biofilm)结构,从而具有更强的抗逆性,常规杀菌方法不能有效杀灭它们,严重威胁食品卫生安全[2]。虽然到目前为止Lm对绝大多数常见的抗生素都是敏感的,临床上也通常使用氨苄西林、利福平、盘尼西林或者与庆大霉素结合治疗李斯特菌病,但越来越多的文献报道显示Lm耐药性已逐渐增强[3-4]。因此深入研究Lm对抗生素的耐受性机制不仅具有重要的基础科研意义,而且具有潜在的临床应用价值。
细胞壁是细菌赖以生存的重要结构基础。细菌细胞壁的结构和成分与机体对细菌的特异性反应,以及自然因素对细菌的选择性作用(抗生素、噬菌体等)等密切相关[5]。L-鼠李糖(Rhamnose)是一种6-脱氧己糖,广泛存在于许多病原菌的细胞壁及荚膜中。合成该糖的4种关键酶分别由rmlA、rmlB、rmlC、rmlD四个基因所形成的操纵子编码,其核苷酸组成在不同细菌中较为保守[6]。由于人体内不存在L-鼠李糖,针对L-鼠李糖合成中相关酶系的药物可能具有较好的选择毒性,因此,以dTDP-L-鼠李糖的合成途径作为潜在的药物靶点是近几年病原菌的研究热点[6]。Lm中也存在编码鼠李糖合成途径所需酶的基因,但其对于细菌的生存和致病力方面的研究报道并不多见。本研究通过同源重组的方法构建了Lm中L-鼠李糖合成过程关键酶dTDP-D-葡萄糖-4, 6-脱氢酶的编码基因rmlB的缺失突变株,并对其生物学特征、生物被膜形成能力、抗生素耐受性和毒力基因的转录表达等进行分析,证实了rmlB基因在Lm生物被膜的形成、耐受作用位点位于细胞壁和细胞膜的抗生素以及细菌毒力等方面具有重要作用。
1 材料与方法 1.1 实验材料 1.1.1 菌株、质粒及引物: 单核细胞增生李斯特菌野生菌株EGDe (血清型1/2a;全基因组序列已知[7])和温度敏感型穿梭整合质粒pLSV101为德国维尔茨堡大学微生物系Werner Goebel教授馈赠。引物(表 1)由天一辉远生物科技有限公司合成。| 引物 Primers |
序列 Sequences (5′→3′) |
| P1: rmlB-A-BamHI-F | TCTGGATCCGTGGAAGCGGGTGT |
| P2: rmlB-A-R | CCAATAAAACCTGCCCCACCTGTTACTAATAAAT |
| P3: rmlB-B-F | GGCAGGTTTTATTGGGATTTACGTTATGCTATTGACCCT |
| P4: rmlB-B-EcoRI-R | CCAGAATTCTTTACTTCCCAGT |
| P5: rmlB -ck-F | TATTTCTGTGGAAGCGGGTGT |
| P6: rmlB -ck-R | CTTTGCGCGTGATTTTAGTGG |
| P7: rpoB-F | TGCCATTTATGCCAGAC |
| P8: rpoB-R | TTCTTCCACTGTGCTCC |
| P9: prfA-F | TTAGCGAGAACGGGACCAT |
| P10: prfA-R | TGCGATGCCACTTGAATAT |
| P11: plcA-F | ACGATGAGCTATAACGGAGAC |
| P12: plcA-R | GCCGAATTTGCGGAGT |
| P13: plcB-F | GCAAATGCCTGTTGTGATG |
| P14: plcB-R | TATTGGCGTGCATAGGTTG |
| P15: actA-F | GGCGAAAGAGTCACTTGC |
| P16: actA-R | GTTGGAGGCGGTGGAAAT |
| P17: mpl-F | TGCTCCAGAGGCCACTACAT |
| P18: mpl-R | CTGATTGCCCTTCGTATTCC |
| P19: hly-F | TGTAAACTTCGGCGCAATC |
| P20: hly-R | TAAGCAATGGGAACTCCTG |
| P21: lmo0441-F | CGCCGAAGACATTGAG |
| P22: lmo0441-R | AGTAATCCCGAGCACC |
| P23: lmo2229-F | CTCCAAGTGCGTATGACC |
| P24: lmo2229-R | TCCACCAGTAGCTGGGTC |
| P25: lmo0540-F | ATCACAACCTGGGAAATG |
| P26: lmo0540-R | CCATCCGGTCACAACTC |
| P27: lmo1438-F | CTACGCAATGGGCTCTG |
| P28: lmo1438-R | TTTGTTCACCTGGGAGG |
| P29: lmo2754-F | CATGCGAATGGTGGAG |
| P30: lmo2754-R | TTACCAACTTCCGTGCC |
| P31: lmo1892-F | GCAATGTTGTTTCCGTCC |
| P32: lmo1892-R | TCTCCAGGATTACCGCC |
| P33: lmo2039-F | GTCCTTCCTTCAACCCTG |
| P34: lmo2039-R | ATCGTGCCGTCACTTGC |
采用SOEing PCR方法(即PCR-mediated recombination method[8])构建缺失菌株:以EGDe基因组DNA为模板用引物P1和P2、P3和P4分别扩增rmlB基因ORF上下游同源臂A (452 bp)和B (406 bp)片段;PCR产物经纯化后作为模板,以P1和P4为引物进行再一次PCR,从而获得含有BamH Ⅰ和EcoR I酶切位点的AB融合片段rmlB (A+B) (987 bp)。PCR反应体系:EGDe基因组2 μL,Prime STAR Max 25 μL,上下游引物(10 μmol/L)各1 μL,超纯水补足至50 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 45 s,60 ℃ 45 s,72 ℃ 45 s,30个循环;72 ℃ 10 min。
用BamH Ⅰ和EcoR Ⅰ对融合片段rmlB(A+B)和质粒pLSV101进行双酶切,将酶切并纯化后的融合片段rmlB(A+B)连接到质粒pLSV101上,构建重组穿梭质粒pLSV101-rmlB(A+B),然后将其电转入EGDe感受态细胞中,按照张强等的方法[8]进行同源重组和筛选rmlB基因缺失的突变子EGDe∆rmlB。所得突变子经PCR (以rmlB基因上下游引物对P5和P6 PCR扩增EGDe∆rmlB中的rmlB基因片段,以EGDe基因组为对照来验证rmlB基因的缺失)和测序验证。
1.3 细菌鞭毛运动性试验采用半固体琼脂穿刺法观察细菌鞭毛的运动性[9]:将待测菌株培养至OD600达0.8−1.0,用直的接种针挑取菌液后穿刺接种于半固体BHI培养基中,24鞭毛蛋白的表达受到温度严格调控,30以上鞭毛蛋白不表达)静置培养1−2周,每天观察。如果细菌由穿刺线向四周云雾状扩散,则判定该细菌有运动性。
1.4 细菌抗生素敏感性实验 1.4.1 最低抑菌浓度法(Minimal inhibitory concentration, MIC): 将待测菌株培养至对数期(OD600达0.5),在96孔微孔板中加入含有两倍倍比稀释抗生素的BHI培养基,取适量菌液加入96孔微孔板中使其终浓度约5×105CFU/mL。同时设置2种对照:(1) 培养物对照,接种同数量的菌液,不加抗生素;(2) 培养基对照,不接种菌液,不加抗生素。将微孔板置于37 ℃,静置培养18−22 h后取出在酶标仪(BioTek,Synergy 2)下测定其OD600值,以在小孔内完全抑制细菌生长的最低药物浓度为MIC,每次试验重复3次[10]。 1.4.2 K-B纸片琼脂扩散法(Disc agar diffusion method): 将待测菌株培养至对数期(OD600达0.5)稀释至5×106CFU/mL后,取固定量菌液0.35 mL均匀涂布于BHI平板[11]。将含有待测抗生素的药敏纸片(Oxoid)均匀贴在涂好菌的平板表面,37 ℃培养22−24 h后,测量纸片周围没有细菌生长的完全抑制区直径,统计数据,每种抗生素重复3次。 1.4.3 抗生素耐受试验(The tolerance assay): 将待测菌株培养至稳定期(OD600达2.5),1:50接种于新鲜的含有32 mg/L Penicillin G[12-13]的BHI培养基中,37振荡培养至相应时间后(0、0.3、3、6、9、12、24 h),取菌液稀释至适当倍数后涂布于BHI平板,37培养约22 h后,计数平板中菌落数,绘制细菌存活曲线,每次试验重复3次。 1.5 微孔板法检测菌株的生长和生物被膜的形成具体方法参照冯飞飞等[14]和Djordjevic等[15]的报道进行:将待测菌株培养至对数期早期(OD600达0.2)稀释后加入96孔微孔板。37静置培养至相应时间(1、2、3、5、7 d)后,先用酶标仪测定其生长情况(OD600),然后弃去各孔培养基,经草酸铵结晶紫染色,95%乙醇溶液脱色后,用酶标仪测定其OD595值。将脱色并干燥后的生物被膜直接放在倒置显微镜下(重光COIC XDS-1B)观察,选取最佳视野拍照。所获得实验数据使用Origin 6.1和SPSS进行分析处理。
1.6 细菌溶血活性检测取1 mL无菌脱纤维绵羊血,2 600 r/min离心5 min,弃上清。用生理盐水洗涤血细胞沉淀,清洗3次。用50倍体积生理盐水重悬血细胞,并以每支1 mL分装于1.5 mL离心管中。将待测菌培养至OD600达1.0左右,取待测菌液10、20、30、50、100 μL不同量的菌液加入含有血细胞的离心管中,37 ℃静置培养30 min后,2 600 r/min离心5 min,收集上清,在分光光度计上检测543 nm处吸光度值。以只含有血细胞的上清作为阴性对照,加入了等量无菌水的上清作为阳性对照。试验重复3次。
1.7 RT-PCR检测基因的转录表达水平将离心收集的细菌在液氮中充分地研磨成粉,之后采用Trizol法提取细菌总RNA[16]。按照反转录试剂盒(TaKaRa)操作说明将总RNA反转录成cDNA。应用PrimerPremier 5.0软件设计目的基因引物(见表 1),以cDNA为模板,PCR扩增目的基因(以管家基因rpoB为内参基因)。试验重复3次。PCR反应体系:cDNA 1.6 μL,2×EsTaqMaster Mix 10 μL,上下游引物(10 μmol/L)各0.8 μL,超纯水补足至20 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 30 s,62 ℃ to 52 ℃ 30 s,72 ℃ 30 s,20个循环;95 ℃ 30 s,52 ℃ 30 s,72 ℃ 30 s,10个循环;72 ℃ 10 min。用Quantity One软件分析RT-PCR凝胶电泳图灰度值。所有数据采用SPSS17.0统计软件进行统计学分析并作图。
2 结果与分析 2.1 EGDe∆rmlB缺失突变株的构建及其生物学特性 2.1.1 EGDe∆rmlB缺失突变株的构建: 如图 1所示:用rmlB基因两端引物P5和P6进行PCR扩增,以EGDe∆rmlB为模板所得片段比EGDe为模板短约790 bp,其长度与预期结果一致。表明EGDe∆rmlB成功缺失了rmlB基因,同时DNA测序结果也进一步证明EGDe∆rmlB构建成功。
|
| 图 1 PCR验证EGDe∆rmlB中rmlB基因的缺失 Figure 1 Verification of the deletion of rmlB in EGDe∆rmlB by PCR Note: M: marker; 1: pLSV101-rmlB(A+B); 2: EGDe; 3: EGDe∆rmlB. |
|
|
|
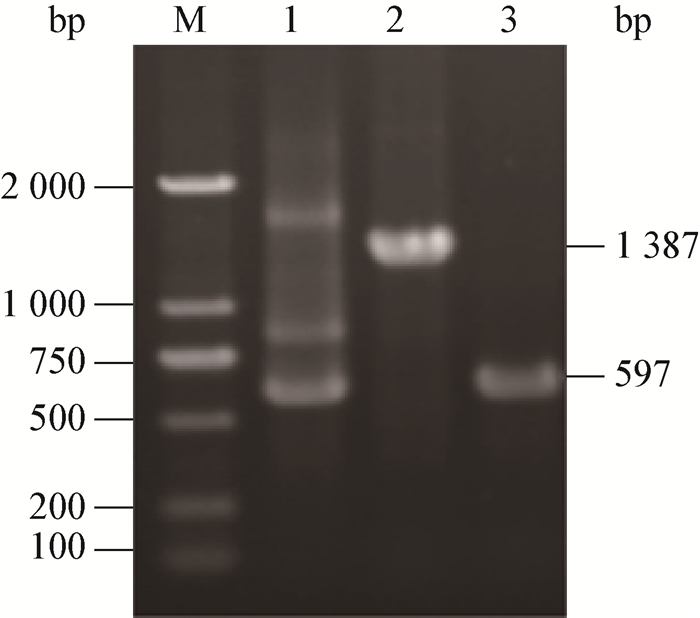
| 图 2 EGDe和EGDe∆rmlB的生长、运动性及细菌形态的比较 Figure 2 Comparison of the growth, motility and bacterial morphology of EGDe and EGDe∆rmlB |
|
|
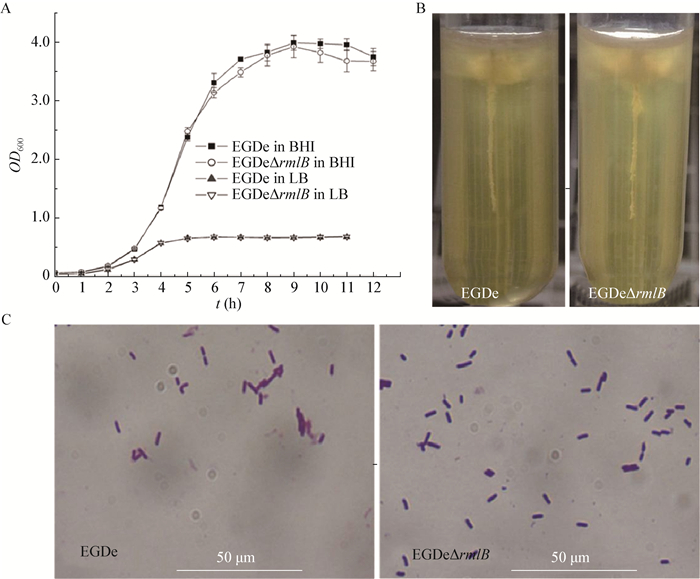
如图 3A所示:在1、2、3、5、7 d的培养过程中,野生菌株和EGDe∆rmlB在各时间段的生长量无明显差异;与野生菌株EGDe相比,EGDe∆rmlB形成的生物被膜量显著降低,在1、2、3、5、7 d分别下降了27.10%、37.52%、46.42%、37.97%和38.85% (图 3B)。

|
| 图 3 EGDe和EGDe∆rmlB生物被膜形成能力的比较 Figure 3 Biofilm formation by EGDe and EGDe∆rmlB in BHI at 37 注:A:两种菌株分别培养1、2、3、5、7 d后的生长量;B:两种菌株分别培养1、2、3、5、7 d后生物被膜的形成量;C:显微镜下,两种菌株形成的生物被膜结构(7 d,40×);**:在同一培养时间段内,与野生型菌株EGDe相比,突变株EGDe∆rmlB的生物被膜形成量具有极显著性差异(P≤0.01). Note: A: The biomass of the two strains after 1, 2, 3, 5, 7 d of cultivation; B: Biofilm quantities for the two strains measured by microtiter plate assay after 1, 2, 3, 5, 7 d of incubation; C: Biofilm formed in the seventh day by the two strains observed under a 40× objective; **: Significant differences of the biomass of biofilm between the mutant strain EGDe∆rmlB and the wild type strain EGDe during the same culture time (P≤0.01). |
|
|
显微镜下两种菌株形成的生物被膜结构显示(图 3C):野生型菌株EGDe形成的链网状生物被膜结构致密,孔隙很小;而EGDe∆rmlB虽然能形成完整的链网状生物被膜,但其结构疏散、孔隙较大。该结果与上述微孔板定量检测结果相符合,表明rmlB基因的缺失降低了Lm生物被膜形成的能力。
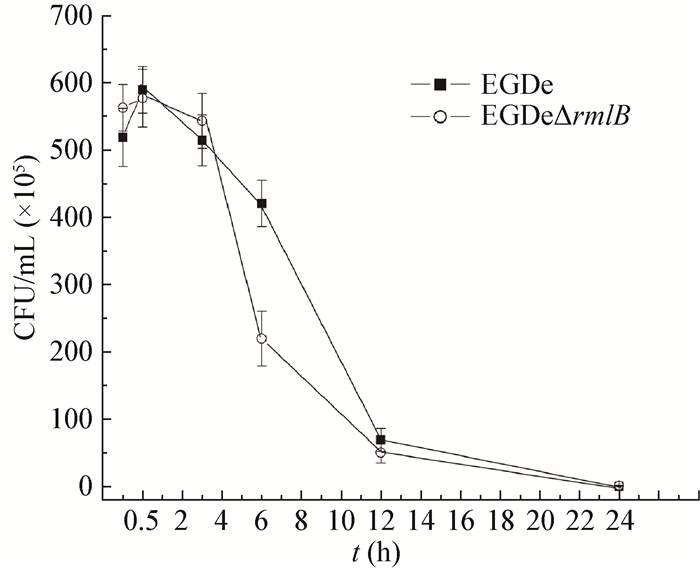
2.3 rmlB基因缺失对单核细胞增生李斯特菌耐药性的影响 2.3.1 比较EGDe和EGDe∆rmlB对抗生素的敏感性: 如表 2所示,与EGDe相比,EGDe∆rmlB对盘尼西林(Penicillin G)、氨苄西林(Ampicillin)和红霉素(Erythromycin)的MIC值以及对利福平(Rifampicin)的抑菌圈直径大小没有显著差异(P≥0.05),但对杆菌肽(Bacitracin)的MIC值显著下降,抑菌圈直径显著增加(P≤0.01)。同时,EGDe∆rmlB对头孢菌素类(头孢呋辛Cefuroxime、头孢噻肟Cefotaxime、头孢吡肟Cefepime)的抑菌圈直径明显增大(P≤0.01)。由于杆菌肽、头孢菌素、盘尼西林和氨苄西林都是常用的作用位点在细菌细胞壁和细胞膜的抗生素,而红霉素和利福平则是作用于细菌蛋白质合成的抗生素,因此,上述结果暗示rmlB基因的缺失可能专一性地影响细菌对作用位点在细胞壁和细胞膜的抗生素的敏感性。由于EGDe∆rmlB对盘尼西林和氨苄西林的MIC与野生株EGDe相比没有差异,因此,我们以盘尼西林为例,进一步比较了EGDe与EGDe∆rmlB对高浓度盘尼西林的耐受性。如图 4所示:在含有致死浓度盘尼西林(32 mg/L)的BHI培养基中,与EGDe相比,EGDe∆rmlB在3 h后出现明显迟滞,活菌数快速下降,在6 h时活菌数降低最为显著;之后,EGDe活菌数也快速下降,12 h后两菌死亡速率逐渐趋同。以上结果表明,尽管EGDe∆rmlB与野生株EGDe相比对盘尼西林的MIC没有差异,但是缺失rmlB后,细菌对高浓度盘尼西林的耐受性显著降低。| 抗生素 Antibiotic |
EGDe | EGDe-rmlB | |||
| 抑菌圈直径 Zone of inhibition (mm)±sa |
MIC (mg/L) | 抑菌圈直径 Zone of inhibition (mm)±s |
MIC (mg/L) | ||
| Ampicillin | 28.39±0.39 | 0.130±0.050 | 28.35±0.21 | 0.120±0.070 | |
| Penicillin G | 26.48±0.09 | 0.224±0.089 | 27.06±0.34 | 0.224±0.089 | |
| Erythromycin | 28.57±0.46 | 0.160 | 28.39±0.21 | 0.160 | |
| Bacitracin | 16.41±0.20 | 88.000±10.950 | 16.86±0.03** | 72.000±8.360** | |
| Cefuroxim | 17.24±0.49 | 20.54±0.41** | |||
| Cefotaxime | 16.92±0.14 | 19.39±0.67** | |||
| Cefepime | 15.71±0.45 | 19.19±0.39** | |||
| Rifampicin | 28.05±0.42 | 28.01±0.31 | |||
| 注:a:结果为3次实验的平均值;**:野生菌株与缺失菌株之间有显著性差异(P≤0.01);s:标准偏差. Note: a: Values are averages of triplicate experiments; *: Significant differences between the mutant strain and the wild-type (P≤0.01); s: Standard deviation. |
|||||
|
| 图 4 EGDe和EGDe∆rmlB在含有32 mg/L penicillin G的液体培养基中的存活曲线 Figure 4 Survival of EGDe and EGDe∆rmlB in broth supplemented with 32 mg/L penicillin G |
|
|
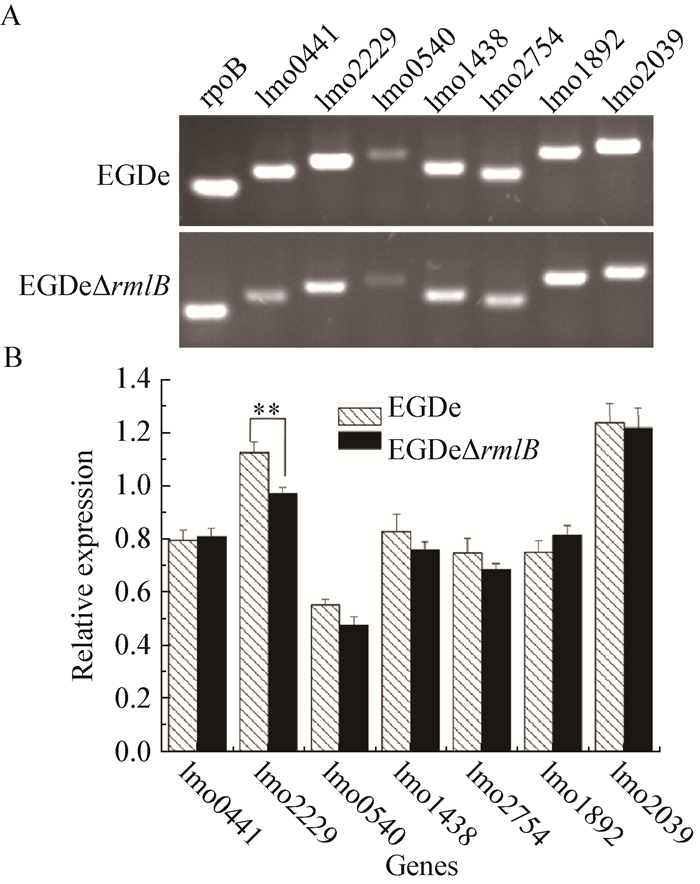
|
| 图 5 青霉素结合蛋白在EGDe和EGDe∆rmlB中的转录表达 Figure 5 The relative expression of the PBPs in the EGDe and EGDe∆rmlB 注:A:青霉素结合蛋白在EGDe和EGDe∆rmlB中转录表达的典型RT-PCR电泳图;B:用Quantity One软件分析3次RT-PCR凝胶电泳图灰度值的结果(**:P≤0.01). Note: A: The typical RT-PCR electrophoregram of PBPs in EGDe and EGDe∆rmlB; B: Quantification of the transcripts intensities performed by Quantity One software (**: P≤0.01). |
|
|

|
| 图 6 主要毒力基因在EGDe和EGDe∆rmlB中的转录表达 Figure 6 The relative transcription expression of the major virulence genes in the EGDe and EGDe∆rmlB |
|
|

|
| 图 7 EGDe和EGDe∆rmlB的溶血活性 Figure 7 Hemolytic activity of EGDe and EGDe∆rmlB |
|
|
L-鼠李糖是广泛存在于许多病原菌的细胞壁及荚膜中的六碳糖。细胞壁和荚膜对于细菌的存活至关重要。在感染阶段,细菌通过细胞壁和荚膜与宿主细胞相互作用。L-鼠李糖合成途径在革兰氏阳性菌和革兰氏阴性菌中都是普遍存在且高度保守的。迄今为止,在人体内还没有发现L-鼠李糖及其合成相关基因。因此,以dTDP-L-鼠李糖的合成途径作为潜在的药物靶点是近几年病原菌的研究热点[6]。然而,研究发现,敲除保守的L-鼠李糖合成途径中的关键基因后,不同细菌的表征并不完全相同。在结核分枝杆菌(Mycobacterium smegmatis)中,敲除rmlB和(或) rmlC后,细菌不再生长[20];在变异链球菌(Streptococcus mutans)中,敲除操纵子中任何一个基因都将抑制细菌细胞壁多糖的合成,导致细菌毒性的降低[21];但是在大肠杆菌(Escherichia coli O2:K1)中,缺失rmlB基因后细菌的生长并不受任何影响,突变菌株中的O抗原仍然含有L-鼠李糖,作者认为该菌株中可能存在RmlB的同工酶或者L-鼠李糖的其它合成途径[22]。
Lm中也存在L-鼠李糖合成途径rmlACBD操纵子的基因位点[23]。当该操纵子缺失后,细菌壁磷壁酸(Wall teichoic acids)中就缺少L-鼠李糖成分,说明在Lm中rmlACBD操纵子对于L-鼠李糖的合成是必不可少的[24]。但Lm的13个血清型里只有血清型1/2a、1/2b、1/2c、3c和7菌株中具有该基因位点,而且在3c和7菌株中因rmlACBD操纵子具有碱基突变而不能合成L-鼠李糖,因此只有血清型1/2a、1/2b、1/2c菌株的壁磷壁酸的主要成分是L-鼠李糖[23]。由此我们推测,尽管壁磷壁酸是革兰氏阳性菌细胞壁的特殊组分,在细菌形态、分裂和耐药性等方面起着重要作用[25],但是,L-鼠李糖合成并不是Lm形态和生长所必需的细胞功能途径。事实上,当我们构建rmlB基因缺失菌株(EGDe∆rmlB)后,发现EGDe∆rmlB的生长及表型都没有明显的变化,该结果与预期高度一致。同时,由于血清型1/2a、1/2b等菌株常在食品中检出,是除血清型4b外导致李斯特菌病的主要血清型[26],因此,EGDe∆rmlB生物被膜形成能力显著下降,暗示鼠李糖的合成尽管对于细菌生长和形态没有重要意义,但可能对细菌适应千变万化的食品以及食品加工运输和储藏环境具有重要作用。
Lm对于单环β-内酰胺类(monobactams)及β-内酰胺类抗生素具有天然的抵抗力。例如甲氧西林及广谱头孢菌素类(包括头孢噻肟和头孢他啶)。而李斯特菌的这一特性具有重要的临床意义,因为医院在治疗不明病原菌所引起的发热时首先会选用头孢菌素类抗生素进行治疗[27],因此研究李斯特菌耐药性产生的分子机制具有重要意义。我们的实验证明,当缺失合成鼠李糖的关键基因rmlB后,细菌对作用于细胞壁和细胞膜的抗生素(如杆菌肽和头孢菌素等)的耐药性显著降低,其可能机制与lmo2229 (Lm青霉素结合蛋白PBP4的编码基因)表达量降低相关。研究表明,lmo2229基因突变后可导致Lm对β-内酰胺类抗生素敏感性增加[4]。同时,在肺炎链球菌中,Smith等[28]和Zhao等[29]已证明lmo2229同系物PBP2A编码基因序列的变异可降低与β-内酰胺类抗生素的亲和性,从而极大地提高细菌的耐药性和致病力。以上研究说明,lmo2229基因对于Lm抵抗β-内酰胺类抗生素具有重要作用,但其与鼠李糖合成关键酶的相互作用尚未见文献报道,这将会是我们今后研究的重点。
最近,Carvalho等[24]发现:缺失EGDe的rmlACBD操纵子后,细菌的毒力下降,感染小鼠的肝脏及脾脏中细菌数降低。我们的实验也证明缺失rmlB后,Lm主要毒力因子溶血素LLO的编码基因hly的转录表达及细菌溶血活性显著降低,但是包括毒力基因转录调控蛋白PrfA在内的其它主要毒力因子的表达都没有受到明显影响。该结果一方面验证了Lm壁磷壁酸中的L-鼠李糖对于细菌的致病性具有重要作用,另一方面也暗示其致病机制与hly基因的转录表达相关,因此,阐明其作用途径和方式也成为了我们今后研究的方向。
综上所述,我们的研究结果表明,rmlB基因在Lm生物被膜形成和耐受作用位点在细胞壁和细胞膜的抗生素及细菌毒力方面具有重要作用。
| [1] | Dussurget O, Bierne H, Cossart P. The bacterial pathogen Listeria monocytogenes and the interferon family: type I, type Ⅱ and type Ⅲ interferons[J]. Frontiers in Cellular and Infection Microbiology 2014, 4 : 50. |
| [2] | Law JWF, Mutalib NSA, Chan KG, et al. An insight into the isolation, enumeration, and molecular detection of Listeria monocytogenes in food[J]. Frontiers in Microbiology 2015, 6 : 1227. |
| [3] | Hof H. Listeriosis: therapeutic options[J]. FEMS Immunology and Medical Microbiology 2003, 35(3) : 203–205. DOI:10.1016/S0928-8244(02)00466-2 |
| [4] | Guinane CM, Cotter PD, Ross RP, et al. Contribution of penicillin-binding protein homologs to antibiotic resistance, cell morphology, and virulence of Listeria monocytogenes EGDe[J]. Antimicrobial Agents and Chemotherapy 2006, 50(8) : 2824–2828. DOI:10.1128/AAC.00167-06 |
| [5] | Chapot-Chartier MP, Kulakauskas S. Cell wall structure and function in lactic acid bacteria[J]. Microbial Cell Factories 2014, 13(S1) : S9. |
| [6] | Giraud MF, Naismith JH. The rhamnose pathway[J]. Current Opinion in Structural Biology 2000, 10(6) : 687–696. DOI:10.1016/S0959-440X(00)00145-7 |
| [7] | Glaser P, Frangeul L, Buchrieser C, et al. Comparative genomics of Listeria species[J]. Science 2001, 294(5543) : 849–852. |
| [8] | Zhang Q, Feng YY, Deng LF, et al. SigB plays a major role in Listeria monocytogenes tolerance to bile stress[J]. International Journal of Food Microbiology 2011, 145(1) : 238–243. DOI:10.1016/j.ijfoodmicro.2010.12.028 |
| [9] | Zhu XF. Modern Experimental Technique of Microbiology[M]. Hangzhou: Zhejiang University Press, 2011: 44 . (in Chinese) 朱旭芬. 现代微生物学实验技术[M]. 杭州: 浙江大学出版社, 2011: 44 . |
| [10] | Langfield RD, Scarano FJ, Heitzman ME, et al. Use of a modified microplate bioassay method to investigate antibacterial activity in the Peruvian medicinal plant Peperomiagalioides[J]. Journal of Ethnopharmacology 2004, 94(2/3) : 279–281. |
| [11] | Yu XZ. The specification of the bacterial amount in K-B method susceptibility testing[J]. Journal of Sichuan Continuing Education College of Medical Sciences 2002, 21(3) : 177–178. (in Chinese) 余修中. K-B法药敏试验中菌液量的规范[J]. 四川省卫生管理干部学院学报 2002, 21(3) : 177–178. |
| [12] | Krawczyk-Balska A, Korsak D, Popowska M. The surface protein Lmo1941 with LysM domain influences cell wall structure and susceptibility of Listeria monocytogenes to cephalosporins[J]. FEMS Microbiology Letters 2014, 357(2) : 175–183. |
| [13] | Begley M, Hill C, Ross RP. Tolerance of Listeria monocytogenes to cell envelope-acting antimicrobial agents is dependent on SigB[J]. Applied and Environmental Microbiology 2006, 72(3) : 2231–2234. DOI:10.1128/AEM.72.3.2231-2234.2006 |
| [14] | Feng FF, Zhang Q, Wang L, et al. The virulence regulator PrfA plays a significant role in the Listeria monocytogenes biofilm formation[J]. Microbiology China 2011, 38(9) : 1450–1457. (in Chinese) 冯飞飞, 张强, 王莉, 等. 毒力基因调控蛋白PrfA促进单核细胞增生李斯特菌生物被膜的形成[J]. 微生物学通报 2011, 38(9) : 1450–1457. |
| [15] | Djordjevic D, Wiedmann M, MclandsboroughLA. Microtiter plate assay for assessment of Listeria monocytogenes biofilm formation[J]. Applied and Environmental Microbiology 2002, 68(6) : 2950–2958. DOI:10.1128/AEM.68.6.2950-2958.2002 |
| [16] | Luo Q, Shang JL, Feng XQ, et al. PrfA led to reduced biofilm formation and contributed to altered gene expression patterns in biofilm-forming Listeria monocytogenes[J]. Current Microbiology 2013, 67(3) : 372–378. DOI:10.1007/s00284-013-0377-7 |
| [17] | Xiong YL, Fan XJ. The research development of penicillin-binding protein[J]. World Notes on Antibiotics 2004, 25(5) : 193–197. (in Chinese) 熊亚莉, 范昕建. 青霉素结合蛋白研究进展[J]. 国外医药抗生素分册 2004, 25(5) : 193–197. |
| [18] | Li J, Yang H, Huyan T, et al. Penicillin binding proteins of bacteria[J]. Chemistry of Life 2013, 33(4) : 418–426. (in Chinese) 李冀, 杨慧, 呼延霆, 等. 细菌青霉素结合蛋白[J]. 生命的化学 2013, 33(4) : 418–426. |
| [19] | Luo Q, Zhang XL, Li B, et al. Regulation of PrfA-dependent virulence genes expression in Listeria monocytogenes[J]. Microbiology China 2008, 35(2) : 275–280. (in Chinese) 罗勤, 张晓莉, 李兵, 等. 单核细胞增生李斯特菌PrfA蛋白转录调控毒力基因表达的分子机制[J]. 微生物学通报 2008, 35(2) : 275–280. |
| [20] | Li W, Xin Y, McNeil MR, et al. rmlB and rmlC genes are essential for growth of mycobacteria[J]. Biochemical and Biophysical Research Communications 2006, 342(1) : 170–178. DOI:10.1016/j.bbrc.2006.01.130 |
| [21] | Yamashita Y, Tomihisa K, Nakano Y, et al. Recombination between gtfB and gtfC is required for survival of a dTDP-rhamnose synthesis-deficient mutant of Streptococcus mutans in the presence of sucrose[J]. Infection and Immunity 1999, 67(7) : 3693–3697. |
| [22] | Ding WP, Luo YM, Ai GM, et al. Function of rmlB in the pathogenic Escherichia coli 44277 (O2: K1)[J]. Acta Microbiologica Sinica 2011, 51(9) : 1168–1176. (in Chinese) 丁卫平, 罗元明, 艾国民, 等. 大肠杆菌44277(O2:K1)中rmlB基因功能[J]. 微生物学报 2011, 51(9) : 1168–1176. |
| [23] | Doumith M, Cazalet C, Simoes N, et al. New aspects regarding evolution and virulence of Listeria monocytogenes revealed by comparative genomics and DNA arrays[J]. Infection and Immunity 2004, 72(2) : 1072–1083. DOI:10.1128/IAI.72.2.1072-1083.2004 |
| [24] | Carvalho F, Atilano ML, Pombinho R, et al. L-Rhamnosylation of Listeria monocytogenes wall teichoic acids promotes resistance to antimicrobial peptides by delaying interaction with the membrane[J]. PLos Pathogens 2015, 11(5). |
| [25] | Brown S, Santa Maria JP, Walker S. Wall teichoic acids of gram-positivebacteria[J]. Annual Review of Microbiology 2013, 67 : 313–336. DOI:10.1146/annurev-micro-092412-155620 |
| [26] | Nightingale KK, Windham K, Wiedmann M. Evolution and molecular phylogeny of Listeria monocytogenes isolated from human and animal listeriosis cases and foods[J]. Journal of Bacteriology 2005, 187(16) : 5537–5551. DOI:10.1128/JB.187.16.5537-5551.2005 |
| [27] | Krawczyk-Balska A, Markiewicz Z. The intrinsic cephalospor in resistome of Listeria monocytogenesin the context of stress response, gene regulation, pathogenesis and therapeutics[J]. Journal of Applied Microbiology 2016, 120(2) : 251–265. DOI:10.1111/jam.12989 |
| [28] | Smith AM, Feldman C, Massidda O, et al. Altered PBP 2A and its role in the development of penicillin, cefotaxime, and ceftriaxone resistance in a clinical isolate of Streptococcus pneumoniae[J]. Antimicrobial Agents and Chemotherapy 2005, 49(5) : 2002–2007. DOI:10.1128/AAC.49.5.2002-2007.2005 |
| [29] | Zhao GS, Meier TI, Hoskins J, et al. Identification and characterization of the penicillin-binding protein 2a of Streptococcus pneumoniae and its possible role in resistance to β-lactam antibiotics[J]. Antimicrobial Agents and Chemotherapy 2000, 44(6) : 1745–1748. DOI:10.1128/AAC.44.6.1745-1748.2000 |
 2017, Vol. 44
2017, Vol. 44