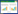扩展功能
文章信息
- 徐玉松, 高土玲, 于昊, 邓子新, 贺新义
- XU Yu-Song, GAO Tu-Ling, YU Hao, DENG Zi-Xin, HE Xin-Yi
- 亚硝基胍消除链霉菌FR-008的线性质粒
- Curing linear plasmids by nitrosoguanidine in Streptomyces sp. FR-008
- 微生物学通报, 2017, 44(1): 141-149
- Microbiology China, 2017, 44(1): 141-149
- DOI: 10.13344/j.microbiol.china.160339
-
文章历史
- 收稿日期: 2016-04-27
- 接受日期: 2016-09-07
- 优先数字出版日期(www.cnki.net): 2016-10-09
2010年美国生物学家Gibson等[1]在Science杂志发表了由化学合成的基因组控制的细胞的文章,对最小基因组的研究引起了广泛的关注。最小基因组(Minimal genome)是指在最适宜的条件下,仅仅拥有维持细胞生长繁殖所必需的最少数目的基因。在最小基因组中,敲除任何基因后细胞将无法生存。在研究最小基因组的探索过程中,主要有两种研究思路:一种是“自下而上”地从头合成基因组,例如Smith等[2]和Gibson等[3]小组成功报道人工设计和化学合成目前最小基因组;另一种是“自上而下”地逐步敲除冗余基因获取最小基因组,例如Kolisnychenko等[4]敲除了包括前噬菌体、转座子和未知功能基因片段,使基因组减小了8.1%。由于必需基因精确预测和基因组装的高难度,所以目前对于最小基因组的研究探索中以“自上而下”地逐步敲除冗余基因获取最小基因组为代表的基因组简化(Reduced-genome)是该领域研究的热点之一。
基因组简化是指基因组被适度删除,复杂程度相对减小,并削弱某些非必需基因的代谢影响,从而获得代谢途径相对简单,且对能量的利用效率明显提高的简化细胞。其作为理想的宿主细胞,在生物合成和生产微生物药物中得以广泛的应用。基因组简化的技术策略通常采用染色体大片段缺失。该操作技术主要包括两种:以Red同源重组和Cre/LoxP重组为代表的定点敲除和利用转座子[5]或者是T-DNA随机插入或切离引发的重组实现DNA片段的随机缺失。Komatsu等[6]利用Cre/LoxP重组系统对阿维链霉菌进行删减,获得基因组减少1.4 Mb的突变株。Murakami等[7]通过PCR介导染色体分裂技术,获得了染色体片段缺失531.5 kb的酿酒酵母突变株。Pósfai等[8]通过大片段基因敲除成功将Escherichia coli K-12基因组减少了15%。除了染色体大片段缺失外,染色体外的质粒消除也是基因组简化研究中的一个重要方向。对于质粒消除的方法并不统一,主要包括物理方法、化学方法和分子生物学方法。如Heery等[9]第一次报道了用高压电穿孔方法成功消除大肠杆菌DH1的内源质粒pUG2;Patrignani等[10]利用SDS结合高温成功消除5株乳杆菌属的内源质粒;Imre等[11]报道了基于转座子两步法消除肠炎沙门氏菌毒性质粒的方法。除列举之外还有如利用中药[12]、吖啶类[13]与EB[14]等诱变剂和冻融法[15]以及质粒不相容[16]等方法。
线性质粒普遍存在于链霉菌中[17],一般比环形质粒大,有的可以高达1.8 Mb[18],而现在已知的抗生素70%以上是由链霉菌产生的,所以质粒消除遗传育种在链霉菌中具有一定的应用潜力。但是,链霉菌线性质粒拷贝数低,非常稳定,常见的质粒消除方法对于环形质粒比较有效,据我们所知,以前还没有使用NTG消除链霉菌线性质粒用于遗传育种的报道。
本研究选用链霉菌FR-008不仅是因为FR-008具有简便的遗传操作,而且其有生长迅速、发酵条件宽松、抗生素产量高等诸多优势[19-20],同时肖湘等[21]报道链霉菌FR-008具有两条明显的线性质粒(pHZ227和pHZ228),Wang等[22]将FR-008的砷抗性基因簇定位在大线性质粒pHZ227上。FR-008基因组测序显示pHZ227大小为142 kb (又名pSSFR1,登录号:CP009803.1),pHZ228大小为24 kb (又名pSSFR2,登录号:CP009804.1)。本研究首次使用NTG诱变来消除FR-008的线性质粒,其丢失频率为4%,并获得了两个杀念菌素产量提高的突变株,是一种有效的遗传育种方法。
1 材料与方法 1.1 实验材料 1.1.1 菌株: 链霉菌FR-008(Streptomycessp. FR-008)、清酒酵母(Saccharomyces sake)与变铅青链霉菌(Streptomyces lividans)由上海交通大学分子微生物学实验室保存。 1.1.2 培养基[23]: 链霉菌FR-008和变铅青链霉菌固体培养基为SFM,链霉菌FR-008液体培养基为TSBY,清酒酵母固体培养基为PDA,清酒酵母液体培养基为YEME[1]。 1.1.3 试剂和仪器: 细胞悬浮液:蔗糖0.3 mol/L,EDTA (pH 8.0) 25 mmol/L,Tris (pH 8.0) 25 mmol/L。蛋白酶K缓冲液:EDTA 93.0 g,Tris碱0.605 g,十二烷基肌氨酸钠5.0 g,蒸馏水定容至500 mL (pH 9.5)。TBE缓冲液:浓贮存液5×:Tris碱54.0 g,硼酸27.5 g,0.5 mol/L EDTA (pH 8.0) 20 mL;使用液(0.5×):0.045 mol/L Tris−硼酸,0.002 mol/L EDTA。TE缓冲液:10 mmol/L Tris-HCl,1 mmol/L EDTA (pH 8.0)。Tris-Maleic acid (Tris-马来酸)溶液:Tris-碱与马来酸的终浓度各为0.25 mol/L (pH 8.0)。1.5%低熔点琼脂糖(Sigma公司):1.5 g低熔点琼脂糖溶于100 mL TE缓冲液中。1%琼脂糖(Sigma公司):10 g琼脂糖溶于1 000 mL TBE缓冲液中。NaAsO2、NTG,购于上海试一化学试剂有限公司。 脉冲场凝胶电泳仪(CHEF-DR Ⅲ system),伯乐生命医学产品(上海)有限公司;生化培养箱(SPX-250B-Z),上海博迅实业有限公司医疗设备厂。 1.2 实验方法 1.2.1 链霉菌FR-008孢子悬浮液制备: 链霉菌FR-008在SFM培养基于30培养4 d后,用无菌棉签轻轻地将孢子刮下悬浮在TES溶液中,振荡混匀,用孢子过滤器过滤,收集滤液,离心去上清并用无菌水将孢子悬浮。 1.2.2 NTG诱变链霉菌FR-008孢子悬浮液: 将孢子悬浮液分别加上1 ml不同浓度的NTG溶液(0、1.6、2.0 g/L),在30 ℃恒温箱中反应12 h后,用无菌水洗涤孢子3遍以终止NTG的诱变作用。将诱变后的孢子悬浮液稀释成不同的浓度梯度涂布于SFM平板上,30 ℃恒温箱培养3 d后观察结果。计算致死率并将诱变后存活的诱变株保种于20%甘油的菌种管并置于−20 ℃。 1.2.3 砷抗性筛选丧失抗砷能力突变株: 链霉菌FR-008的大线性质粒pSSFR1含有砷抗性基因[22],所以本研究通过砷敏感筛选丧失抗砷能力的突变株。本研究直接将250 μmol/L亚砷酸钠溶液加到20 mL SFM培养基中,混合均匀,以不加砷盐的SFM培养基作对照,其背面画格一一对应标记并接种诱变株,30培养1−2 d后观察结果。 1.2.4 筛选线性质粒消除突变株: 突变株不抗砷并不能完全说明大线性质粒pSSFR1的消除,也可能是抗砷基因发生了突变导致功能丧失。PFGE可以直接观测质粒的消除情况,找到突变株砷敏感的原因。PFGE技术是基于不同长度的DNA长链大分子在电场中所经历的构型变化不同,通过变换电场方向的变化及间隔时间,从而达到分离大片段DNA的目的。操作过程如下[24]:(1) Plug的制备:将收集到的菌体用细胞悬浮液清洗2遍,然后按下列比例混合:菌体悬浮液500 μL,l.5%低熔点琼脂糖500 μL。迅速混匀并加入制胶模孔中,置于4 ℃冰箱中使菌体和琼脂糖凝固形成Plug;(2) Plug的处理:将Plug转入溶菌酶溶液中(终浓度2 g/L),37 ℃水浴8 h后,去除溶菌酶溶液,用TE缓冲液清洗至少3遍,再用终浓度为1%的SDS 50 ℃水浴2 h,用TE缓冲液清洗至少3遍,再用蛋白酶K缓冲液清洗一遍后加入蛋白酶K溶液(终浓度为1 g/L),50 ℃水浴16 h;(3) Plug的电泳:将清洗后的Plug整齐置于电泳梳子上,固定制胶槽,缓慢倒入熔化后的琼脂糖凝胶(浓度为1%,用0.5×TBE配制),待凝胶凝固后拔除梳子,将凝胶连同制胶板一同放入电泳槽进行脉冲场凝胶电泳。本研究数据设置如下:电压:6 V/cm;起始时间:1 s;最终时间:50 s;温度:14 ℃;电泳时间:20 h;电场夹角:120°。 1.2.5 链霉菌FR-008特异性引物设计: 为验证突变株来源于链霉菌FR-008,设计了3条特异性引物。引物合成由上海杰李生物技术有限公司完成。primer1-F:5′-TCTTCTTCGTCACCGGGACGAT-3′;primer1-R:5′-ATGTCGTTGAGGTCCCAGCCGT-3′;primer2-F:5′-AAATCTAGAGAGCCCGCATTCAC GCACTTTCG-3′;primer2-R:5′-AAAGGATCCTTCC AGTCCTCGCTGTCGGTGTTGTT-3′;primer3-F:5′-AAAGAATTCTACCTCTGGTTCGTCCTCGCCTCCC-3′;primer3-R:5′-AAAAAGCTTCCGATGCT GCCGTCGTCGTTGAT-3′。 1.2.6 线性质粒消除突变株生物活性测定: FR-008具有较好的抗真菌活性[25],本研究选择清酒酵母(Saccharomyces sake)作为生测的指示菌。首先在PDA平板上划线分离清酒酵母单克隆,接种至YEME (10.3%蔗糖)培养基中,30 ℃、220 r/min培养约24 h,离心收集菌体,用LB培养基洗涤一次,熔化PDA培养基并冷却至50 ℃左右,每20 mL培养基中加入20 μL新鲜指示菌,立即混匀后倒入培养皿,待其凝固。从FR-008野生型和突变株的菌坪上(30 ℃培养5−7 d)接种于SFM固体培养基平板上,用蓝枪头将培养物打成一圆形菌块。在含有指示菌平板的合适的位置上摆好待测的菌块,30 ℃培养1−2 d后观察抑菌圈的大小。 1.2.7 HPLC检测各线性质粒消除突变株的抗生素产量: 将各突变株及链霉菌FR-008野生型等量孢子接种到40 mL TSBY液体培养基的摇瓶(250 mL)中,30 ℃培养2 d制备种子发酵液。再将等量种子液接种到40 mL YEME (10.3%蔗糖)液体培养基的摇瓶(250 mL)中,30 ℃、220 r/min培养3−4 d得到发酵液。各突变株取等湿重的菌体,加入甲醇溶液重复萃取2次,收集萃取液,并用0.25 μm有机相滤膜过滤后,用于HPLC分析。HPLC使用的是安捷伦公司的Agilent 1100 series LC/MSD trap system。色谱柱为Agilent SB-C18 (5 μm,4.6 mmx250 mm)反向柱,流动相为HPLC级乙腈:5.5 mmol/L NH4AC (pH 4.5)=50׃50 (体积比),流速为0.3 mL/min,检测波长为380 nm,柱温为25 ℃。 2 结果与分析 2.1 NTG诱变链霉菌FR-008孢子悬浮液采用不同浓度的NTG处理(NTG溶液与孢子悬浮液共培养),无菌水处理之后(终止NTG的诱变作用)十倍稀释法均匀平铺于SFM固体培养基,培养3 d得到以下结果(图 1)。
|
| 图 1 经不同浓度的NTG处理存活的诱变株 Figure 1 Survival rates for mutants treated with different concentration of NTG 注:A:未经处理的FR-008孢子悬浮液,稀释107倍存活105个孢子;B:经1.6 g/L NTG处理后稀释106倍存活40个孢子;C:经2 g/L NTG处理后稀释105存活17个孢子. Note: A: 105 colonies of wild type strains grew (107-fold dilution); B: 40 colonies of mutants survived after 1.6 g/L NTG treatment (106-fold dilution); C: 17 colonies of mutants survived after 2 g/L NTG treatment (105-fold dilution). |
|
|
实验结果表明链霉菌FR-008孢子悬浮液对于NTG比较敏感,能够达到比较高的致死率而且随着NTG浓度的增加致死率上升。1.6 g/L和2 g/L的NTG诱变处理后,致死率分别为96.2%和99.8%。本研究采用2 g/L的浓度用于线性质粒的消除。
2.2 砷抗性筛选的结果收集经2 g/L NTG处理后的103个诱变株。由于链霉菌FR-008的大线性质粒pSSFR1上具有抗砷的基因,因此将103个诱变株30 ℃培养在含有有砷(3价)的SFM固体培养基上3 d。观察是否能够生长。理论上在含有有砷(3价)的SFM固体培养基上不能生长的诱变株有可能其大线性质粒pSSFR1发生消除。实验结果如图 2所示。

|
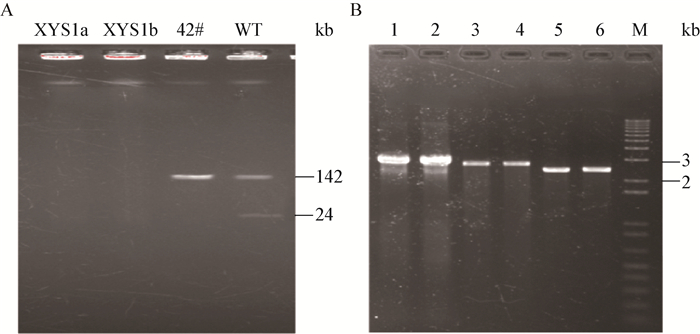
| 图 2 砷抗性筛选亚砷酸钠敏感突变株及复证 Figure 2 Screening of arsenite sensitive mutants and validation 注:A:突变株及对照菌株在不含砷的SFM固体培养基上培养;B:突变株及对照菌株在含250 μmol/L亚砷酸钠的SFM固体培养基上培养. lividans代表变铅青链霉菌(抗砷);WT代表野生型链霉菌FR-008(抗砷). Note: A: Mutants and control strains on SFM; B: Strains grown on SFM supplied with 250 μmol/L arsenite. lividans: Streptomyces lividans(arsenite resistance); WT: Streptomyces sp. FR-008 wild type (arsenite resistance). |
|
|
将经2 g/L NTG处理后的103个诱变株依次接种含250 μmol/L亚砷酸钠溶液的SFM固体培养基上。从103个突变株中筛选到了10#、59#、115# 3个砷敏感的突变株,暗示其对应的pSSFR1大线性质粒(142 kb,砷抗性基因簇)可能被消除了。为消除实验偶然误差,对砷敏感的突变株进行了复证,图 2左边是SFM培养基上均可以生长,图 2右边是SFM培养基上添加250 μmol/L亚砷酸钠溶液,10#、59#、115#不能生长,其中以对砷有抗性的FR-008和S. lividans作为阳性对照。
2.3 PFGE检测的结果为了检测这些砷敏感的突变株是否丢失了大线性质粒,以及小线性质粒丢失的情况,对这103个突变株进行了PFGE检测。结果见图 3。

|
| 图 3 PFGE检测各突变株线性质粒丢失情况 Figure 3 Detection of the presence or loss of linear plasmids in the mutants by PFGE 注:野生型和各个突变株都选取了3个独立的克隆来进行PFGE检测.大线性质粒142 kb,小线性质粒24 kb.脉冲场凝胶电泳的条件为:转换时间为1−50 s;电压:6 V/cm;电泳时间:20 h;凝胶浓度为1%;缓冲液为0.5×TBE. Note: three independent colonies for each strain were selected to detect the linear plasmids. Total DNA in the plug were separated on a 1% agarose gel with a 1−50 s pulse for 20 h at 6 V/cm in 0.5×TBE buffer. |
|
|
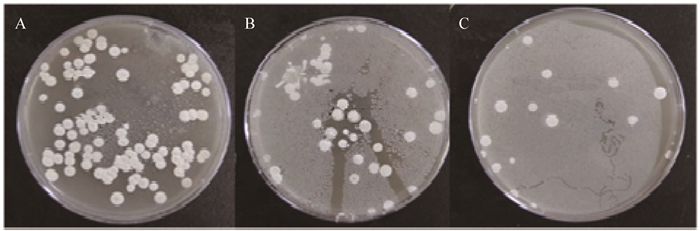
PFGE检测结果显示:10#、59#和115#经NTG诱变后丢失了142 kb的大线性质粒pSSFR1,但是该3株突变株中24 kb的线性质粒pSSFR2仍然保留。42#突变株丢失了pSSFR2但是保留了pSSFR1。综合起来,在致死率为99.8%的情况下,NTG消除线性质粒的效率约为4%,因而是一种有效的线性质粒消除剂。为了证实两个线性质粒是否可能同时被消除,NTG继续处理42#突变株,获得了大小线性质粒均丢失的双突变株XYS1 (图 4A)。我们使用了3套链霉菌FR-008特异性的PCR引物(泳道1、2所用的引物扩增2 871 bp,泳道3、4所用的引物扩增2 654 bp,泳道5、6所用的引物扩增2 368 bp),证明XYS1的确是衍生于42#的突变株(图 4B)。
|
| 图 4 NTG诱变同时消除两条线性质粒及验证 Figure 4 Conformation of XYS1 derived from Streptomyces sp. FR-008 注:A:PFGE检测WT、42#、XYS1的线性质粒,XYS1a和XYS1b是42#突变株经过第二轮NTG诱变后获得对亚砷酸钠敏感的两个克隆;B:PCR验证XYS1是Streptomyces sp. FR-008的衍生菌株,而非污染其它细菌. M为1 kb DNA分子量标记;1、3、5为FR-008的基因组DNA为模板;2、4、6为XYS1的基因组DNA为模板;1、2使用Primer1_F & R;3、4使用Primer2_F & R;5、6使用Primer3_F & R. Note: A: PFGE detection of linear plasmids in Wild Type, Mutant 42# and XYS1 that is mutant 42#-derived mutant sensitive to arsenite; B: XYS1 is confirmed to be a Streptomyces sp. FR-008 derived strain by PCR. M: 1 kb DNA marker; 1, 3, 5: PCR product using total DNA of FR-008 wild type as template; 2, 4, 6: PCR product using total DNA of XYS1 as template. 1, 2: Primer1_F & R; 3, 4: Primer2_F & R; 5, 6: Primer3_F & R. |
|
|
链霉菌FR-008能够产生杀念菌素,该类化合物(杀念菌素具有多个组分,有效组分是Candicidin Ⅲ)具有抗真菌活性,通常利用酵母来检测其产抗生素的能力。本研究要比较各质粒消除突变株同野生型的差异,因此采用酿酒酵母作为生测菌来比较各个菌株的产素能力。
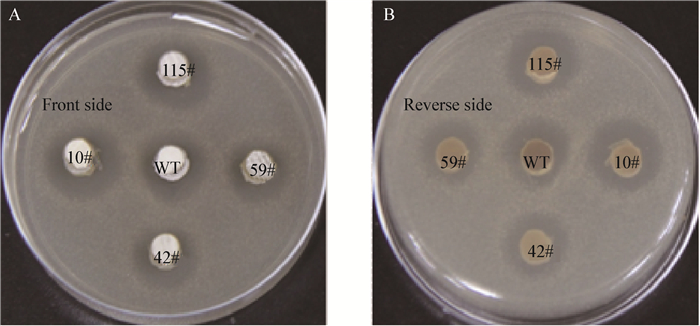
图 5结果表明各线性质粒消除突变株仍然能够合成抗真菌的杀念菌素,对指示菌清酒酵母具有一定的抑菌效果。其中,10#和115#突变株相比较野生型其抑菌圈较大,暗示产生杀念菌素的能力比野生型强。而42#和59#与野生型相比,其抑菌圈似乎要小些。
|
| 图 5 生测实验比较各菌株产杀念菌素的能力 Figure 5 Bioassay to compare candicidin yield of four mutant strains and the wild type 注:在SFM上生长了5 d链霉菌打菌块,并转移到预混有指示菌清酒酵母的PDA平板上.抑菌圈大小指示了各菌株产生杀念菌素的能力. Note: The plugs was taken out from SFM agar where Streptomyces strains have grown for 5 days, and transferred to the PDA agar that is premixed with the indicator Saccharomyces sake. The size of clear halo indicate the candicidin productivity for each strain. |
|
|
测定了这些突变株及野生型液体发酵(TSBY培养基)产杀念菌素的产量,利用HPLC检测了这些突变株发酵产物中的杀念菌素有效组分Ⅲ的产量。结果以相对值(野生型为标准值1)显示如下(图 6)。

|
| 图 6 HPLC比较各菌株杀念菌素有效组分Ⅲ的产量 Figure 6 The yield of candicidin component Ⅲ for each strain was analyzed by HPLC 注:野生型菌株杀念菌素组分Ⅲ的产量在柱状图中标示为1,另外4个柱状图的高度分别代表菌株10#、42#、59#和115#相对于野生型产杀念菌素组分Ⅲ的倍数. Note: The yield of candicidin component Ⅲ for wild type strain is represented by 1 whereas in other four mutants it is represented by the fold to that of WT. |
|
|
图 6数据表明和链霉菌FR-008野生型一样,各个被消除线性质粒的突变株依然能够产生杀念菌素,但对产量影响却不尽相同。总体结果与生测一致:两株消除了pSSFR1线性大质粒的10#和115#突变株相比较链霉菌FR-008野生型,杀念菌素有效组分Ⅲ的产量分别提高了40%和30%。然而42#和59#相比较链霉菌FR-008野生型,抗生素的产量反而下降了。
3 讨论链霉菌FR-008隶属于白色链霉菌亚种,它可以产生多烯类抗真菌抗生素杀念菌素,加上FR-008菌株野生型生长迅速、产孢快、接合转移效率高,因而有潜力发展成为一个异源表达宿主。质粒消除作为一种有效的微生物遗传育种手段,已经有许多早期的应用报道[26-27],一般是加热和SDS多轮处理,但是一般应用于环形质粒的消除,比较繁琐。链霉菌中的线性质粒比较大,和环形质粒不同的是:它的拷贝数低,较为稳定而不易消除,大小通常都在20 kb−1.8 Mb之间。由于质粒上通常携带非必需基因,因而大线性质粒的消除在基因组简化方面具有较好的应用潜力。NTG作为诱变剂,主要引起碱基的转换,至今尚未有研究者用它来消除链霉菌的线性质粒。FR-008包含两个线性质粒pSSFR1和pSSFR2,前者包括了抗砷基因簇,这为利用NTG诱变消除质粒的检测提供了很好的条件,只需测试砷的敏感性,就能轻易筛到砷敏感的菌株(可能的大线性质粒消除突变株)。
本研究中当NTG致死率达到99.8%以上时,线性质粒消除率约为4%,远高于链霉菌自然突变的频率。对于线性质粒之所以能够被NTG消除,猜测可能是因为和它的质粒结构有关系,链霉菌线性质粒的末端通常有端粒结构[28],NTG处理可能影响了端粒的功能而使复制过程受阻,从而被消除。结合抗砷筛选和PFGE证实FR-008野生型两个线性质粒的存在,同时表明抗砷基因确实是位于大线性质粒pSSFR1上。这种研究模式可以推广,即为选择质粒上不同于染色体某个特性作为质粒消除与否的证据。可以人为将某个抗性基因克隆到质粒上作为一种标记,运用于质粒功能的研究。根据质粒消除的结果,不管是pSSFR1还是pSSFR2,两者均不含有宿主生长代谢所必需的基因。而Ye等[29]发现不能将极端嗜盐古生菌质粒pHH205消除。同时Nindita等[30]发现可以分别单独消除Streptomyces rochei中pSLA2-L (211 kb)和pSLA2-M (113 kb),但同时消除两者会使线性染色体环化。说明不同种属的菌株质粒上携带基因组不同的信息。质粒与染色体既可以“共生”也可以“寄生”。这可能是由于进化过程不同而造成的差异。
根据生测和HPLC的结果,获得了2株相对链霉菌FR-008野生型更高产量抗生素的优势突变株。链霉菌FR-008大线性质粒除了砷抗性基因簇、质粒转移复制相关蛋白、CRISPR相关蛋白之外,编码了两个可能的调节蛋白,一个注释为LuxR家族的调节蛋白(基因编号:SFR_7012,218个氨基酸残基),另外一个是可能的调节蛋白(377个氨基酸残基)。然而杀念菌素合成基因簇中4个LuxR家族调节蛋白[31-32]FscRⅠ、FscRⅡ、FscRⅢ和FscRⅣ多属于正调节。同时59#菌株的有效组分Ⅲ的产量相对于野生型下降。因此推测:大线性质粒消除突变株中杀念菌素有效组分Ⅲ的提高可能和质粒上编码的基因没有关系,猜测是因为NTG作用质粒外其他部分引起的突变造成的。当然这些假设还需实验证实。对于被消除线性质粒的突变株,其基因组大小相应减少一部分,是否会增强某个部分的代谢通路,该突变株是否可以作为生产某一代谢通路产物的生产菌株,并在此突变株的基础上改造,使其更有利于某一代谢通路产物的积累。
| [1] | Gibson DG, Glass JI, Lartigue C, et al. Creation of a bacterial cell controlled by a chemically synthesized genome[J]. Science 2010, 329(5987) : 52–56. DOI:10.1126/science.1190719 |
| [2] | Smith HO, Hutchison CA Ⅲ, Pfannkoch C, et al. Generating a synthetic genome by whole genome assembly: φX174 bacteriophage from synthetic oligonucleotides[J]. Proceedings of the National Academy of Sciences of the United States of America 2003, 100(26) : 15440–15445. DOI:10.1073/pnas.2237126100 |
| [3] | Gibson DG, Benders GA, Andrews-Pfannkoch C, et al. Complete chemical synthesis, assembly, and cloning of a Mycoplasma genitalium genome[J]. Science 2008, 319(5867) : 1215–1220. DOI:10.1126/science.1151721 |
| [4] | Kolisnychenko V, Plunkett G Ⅲ, Herring CD, et al. Engineering a reduced Escherichia coli genome[J]. Genome Research 2002, 12(4) : 640–647. DOI:10.1101/gr.217202 |
| [5] | Hutchison CA Ⅲ, Peterson SN, Gill SR, et al. Global transposon mutagenesis and a minimal Mycoplasma genome[J]. Science 1999, 286(5447) : 2165–2169. DOI:10.1126/science.286.5447.2165 |
| [6] | Komatsu M, Uchiyama T, Ömura S, et al. Genome-minimized Streptomyces host for the heterologous expression of secondary metabolism[J]. Proceedings of the National Academy of Sciences of the United States of America 2010, 107(6) : 2646–2651. DOI:10.1073/pnas.0914833107 |
| [7] | Murakami K, Tao E, Ito Y, et al. Large scale deletions in the Saccharomyces cerevisiae genome create strains with altered regulation of carbon metabolism[J]. Applied Microbiology and Biotechnology 2007, 75(3) : 589–597. DOI:10.1007/s00253-007-0859-2 |
| [8] | Pósfai G, Plunkett G Ⅲ, Fehér T, et al. Emergent properties of reduced-genome Escherichia coli[J]. Science 2006, 312(5776) : 1044–1046. DOI:10.1126/science.1126439 |
| [9] | Heery DM, Powell R, Gannon F, et al. Curing of a plasmid from E. coli using high-voltage electroporation[J]. Nucleic Acids Research 1989, 17(23) : 10131. DOI:10.1093/nar/17.23.10131 |
| [10] | Patrignani F, Lanciotti R, Mathara JM, et al. Potential of functional strains, isolated from traditional Maasai milk, as starters for the production of fermented milks[J]. International Journal of Food Microbiology 2006, 107(1) : 1–11. DOI:10.1016/j.ijfoodmicro.2005.08.004 |
| [11] | Imre A, Olasz F, Kiss J, et al. A novel transposon-based method for elimination of large bacterial plasmids[J]. Plasmid 2006, 55(3) : 235–241. DOI:10.1016/j.plasmid.2005.11.006 |
| [12] | Chen Q, Xie KP, Yun BY, et al. Inhibition and plasmid elimination effect of cortex Phellodendron chinensis and other Chinese herbal medicines on MRSA[J]. Journal of Microbiology 2013, 33(3) : 54–57. (in Chinese) 陈晴, 谢鲲鹏, 云宝仪, 等. 黄柏等中草药对MRSA的抑菌作用及其对质粒的消除作用[J]. 微生物学杂志 2013, 33(3) : 54–57. |
| [13] | Brzychczy-Wloch M, Borszewska-Kornacka M, Gulczynska E, et al. Prevalence of antibiotic resistance in multi-drug resistant coagulase-negative staphylococci isolated from invasive infection in very low birth weight neonates in two Polish NICUs[J]. Annals of Clinical Microbiology and Antimicrobials 2013, 12 : 41. DOI:10.1186/1476-0711-12-41 |
| [14] | Yeldho D, Rebello S, Jisha MS. Plasmid-mediated biodegradation of the anionic surfactant sodium dodecyl sulphate, by Pseudomonas aeruginosa S7[J]. Bulletin of Environmental Contamination and Toxicology 2011, 86(1) : 110–113. DOI:10.1007/s00128-010-0162-2 |
| [15] | Zhong YJ, Wu XY, Liu HJ, et al. Plasmid elimination method and conditions of Burkholderia cepacia T1828[J]. Microbiology China 2011, 38(6) : 847–852. (in Chinese) 钟义军, 吴晓玉, 刘好桔, 等. 洋葱伯克霍尔德菌T1828质粒消除方法及条件[J]. 微生物学通报 2011, 38(6) : 847–852. |
| [16] | Wang DS, Gao ZQ, Wang HG, et al. Curing both virulent mega-plasmids from Bacillus anthracis wild-type strain A16 simultaneously using plasmid incompatibility[J]. Journal of Microbiology and Biotechnology 2015, 25(10) : 1614–1620. DOI:10.4014/jmb.1503.03083 |
| [17] | Kinashi H, Shimaji M, Sakai A. Giant linear plasmids in Streptomyces which code for antibiotic biosynthesis genes[J]. Nature 1987, 328(6129) : 454–456. DOI:10.1038/328454a0 |
| [18] | Ou HY. Bioinformatics tools and databases focused on genomic islands and secondary metabolite biosynthesis of Streptomyces[J]. Microbiology China 2013, 40(10) : 1909–1919. (in Chinese) 欧竑宇. 链霉菌基因组岛和次生代谢物合成相关的生物信息学工具及数据库[J]. 微生物学通报 2013, 40(10) : 1909–1919. |
| [19] | Chen S, Huang X, Zhou XF, et al. Organizational and mutational analysis of a complete FR-008/candicidin gene cluster encoding a structurally related polyene complex[J]. Chemistry & Biology 2003, 10(11) : 1065–1076. |
| [20] | Hu ZH, Bao K, Zhou XF, et al. Repeated polyketide synthase modules involved in the biosynthesis of a heptaene macrolide by Streptomyces sp. FR-008[J]. Molecular Microbiology 1994, 14(1) : 163–172. DOI:10.1111/mmi.1994.14.issue-1 |
| [21] | Xiao X, Zhou XF, Deng ZX. Linear plasmid-a new research highlight in fundamental biology for Streptomyces[J]. World Notes on Antibiotics 1999, 20(2) : 49–53. (in Chinese) 肖湘, 周秀芬, 邓子新. 线性质粒--链霉菌基础生物学研究的一个新热点[J]. 国外医药:抗生素分册 1999, 20(2) : 49–53. |
| [22] | Wang LR, Chen S, Xiao X, et al. arsRBOCT arsenic resistance system encoded by linear plasmid pHZ227 in Streptomyces sp. strain FR-008[J]. Applied and Environmental Microbiology 2006, 72(5) : 3738–3742. DOI:10.1128/AEM.72.5.3738-3742.2006 |
| [23] | Kieser T, Bibb M J, Chater K F, et al. Practical Streptomyces Genetics[M]. Norwich: The John Innes Foundation, 2000 . |
| [24] | Schwartz DC, Cantor CR. Separation of yeast chromosome-sized DNAs by pulsed field gradient gel electrophoresis[J]. Cell 1984, 37(1) : 67–75. DOI:10.1016/0092-8674(84)90301-5 |
| [25] | Zhou Y, Li J, Zhu J, et al. Incomplete β-ketone processing as a mechanism for polyene structural variation in the FR-008/candicidin complex[J]. Chemistry & Biology 2008, 15(6) : 629–638. |
| [26] | Feng J, Zhang W, Song CJ. Progress in endogenous plasmid curing of bacteria-a review[J]. Acta Microbiologica Sinica 2013, 53(11) : 1142–1148. (in Chinese) 冯俊, 张伟, 宋存江. 细菌内源质粒消除研究进展[J]. 微生物学报 2013, 53(11) : 1142–1148. |
| [27] | Hu GY. Interaetion among indigenous plasmids of Mesorhizobium huakuii strains and its effect on symbiotic nitregen fixation[D]. Wuhan: Doctoral Dissertation of Huazhong Agricultural University, 2006 (in Chinese) 胡国元.华癸中生根瘤菌内源质粒间的相互作用及其对共生固氮的影响[D].武汉:华中农业大学博士学位论文, 2006 http://cn.bing.com/academic/profile?id=0b328948aa93613028927ab175c4b192&encoded=0&v=paper_preview&mkt=zh-cn |
| [28] | Bao K, Cohen SN. Terminal proteins essential for the replication of linear plasmids and chromosomes in Streptomyces[J]. Genes & Development 2001, 15(12) : 1518–1527. |
| [29] | Ye XC, Ou JH, Ni LN, et al. Characterization of a novel plasmid from extremely halophilic Archaea: nucleotide sequence and function analysis[J]. FEMS Microbiology Letters 2003, 221(1) : 53–57. DOI:10.1016/S0378-1097(03)00175-7 |
| [30] | Nindita Y, Cao ZS, Yang YJ, et al. The tap-tpg gene pair on the linear plasmid functions to maintain a linear topology of the chromosome in Streptomyces rochei[J]. Molecular Microbiology 2015, 95(5) : 846–858. DOI:10.1111/mmi.2015.95.issue-5 |
| [31] | Zhang PP, Zhao ZL, Li H, et al. Production of the antibiotic FR-008/candicidin in Streptomyces sp. FR-008 is co-regulated by two regulators, FscRI and FscRIV, from different transcription factor families[J]. Microbiology 2015, 161(3) : 539–552. DOI:10.1099/mic.0.000033 |
| [32] | Mo SJ, Yoon YJ. Interspecies complementation of the LuxR family pathway-specific regulator involved in macrolide biosynthesis[J]. Journal of Microbiology and Biotechnology 2016, 26(1) : 66–71. DOI:10.4014/jmb.1510.10085 |
 2017, Vol. 44
2017, Vol. 44