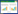扩展功能
文章信息
- 牟慧艳, 刘扬, 王应东, 刘宝爱, 魏建军, 张会图
- MOU Hui-Yan, LIU Yang, WANG Ying-Dong, LIU Bao-Ai, WEI Jian-Jun, ZHANG Hui-Tu
- 恩拉霉素生产菌株的遗传改造
- Genetic modification of an enramycin producing strain
- 微生物学通报, 2017, 44(1): 126-132
- Microbiology China, 2017, 44(1): 126-132
- DOI: 10.13344/j.microbiol.china.160068
-
文章历史
- 收稿日期: 2016-01-19
- 接受日期: 2016-05-06
- 优先数字出版日期(www.cnki.net): 2016-05-24
2. 天津市新星兽药厂 天津 300402
2. Tianjin Xinxing Veterinary Pharmaceutical Factory, Tianjin 300402, China
恩拉霉素(Enramycin)又名恩来霉素、恩霉素、安来霉素、持久霉素,是由日本研究人员在土壤中分离得到的一株杀真菌素链霉菌经过发酵获得的次级代谢产物[1-2]。它是一类含有17个氨基酸的脂肽类抗生素,对革兰氏阳性菌,特别是禽畜肠道内有害梭状芽孢杆菌具有较强的抑制作用[3];饲料中添加适量的恩拉霉素不仅可以预防常见的动物消化道疾病,改善动物肠道内的群落平衡,还可以提高饲料的利用率,促进动物增重。同时,恩拉霉素具有较高的稳定性、低毒性、低药物残留、长期使用后不易产生耐药性等特点,因此被许多国家作为抗生素生长剂使用[4]。目前,工业上常用杀真菌素链霉菌S. fungicidicus作为恩拉霉素生产菌株,但由于该菌株发酵周期长、产素水平低等原因[5],严重阻碍了企业的大规模生产。因此,采用现代技术选育高产的恩拉霉素生产菌株,对于降低恩拉霉素的生产成本具有重要的意义。
常规的选育方法如物理、化学等诱变剂进行随机诱变,无需了解其遗传背景,至今依然被广泛使用,但存在耗时长、效率低等缺点;而基因工程、代谢工程等新一代育种方法虽然效率高,目标明确,但需掌握复杂的遗传信息、代谢调控机制,因而限制了其广泛使用。因此寻找简单、高效、稳定的微生物遗传改造的方法尤为重要。2004年,日本国家食品研究所Ochi等首次提出了核糖体工程概念,指出对微生物的核糖体或RNA聚合酶进行修饰改造,能够直接或间接地激活与微生物次级代谢产物合成有关基因的表达,从而提高次级代谢产物的产量甚至获得新的活性物质[6]。该技术操作简单、可行性高,可以广泛地应用到抗生素高产菌株的选育中。大量研究发现,位于30S小亚基上的核糖体S12蛋白(rpsL基因编码)影响着微生物次级代谢产物的合成,若核糖体S12蛋白的结构发生改变,将导致次级代谢产物相关基因被激活或过量表达。1997年,Hesketh和Ochi等将rpsL基因的K88E突变引入野生型天蓝色链霉菌A3(2)中,使该菌株放线紫红素产量得到了显著提高[7];2000年,Okamoto-Hosoya等筛选到rpsL基因的突变体P91S,其放线紫红素产量是野生型天蓝色链霉菌A3(2)的7−10倍[8]。目前,对S12蛋白编码基因(rpsL)的突变主要集中在S12的循环结构中的两个保守区:区域Ⅰ (TPKKPNS,氨基酸残基)和区域Ⅱ (RVKDLPGVR,氨基酸残基)。对区域Ⅱ中第88位赖氨酸周围的位点进行突变,获得了抗生素高产突变菌株;而对区域Ⅰ中,第43、44位的赖氨酸突变的研究则相对较少。
本文利用定点突变技术,将杀真菌素链霉菌中核糖体S12蛋白(rpsL基因编码)的氨基酸序列上第43位的赖氨酸(Lys)分别替换了天冬酰胺(Asn)和精氨酸(Arg),获得了恩拉霉素产量明显提高、产孢周期明显缩短的突变菌株。该研究旨在为工业化发酵生产恩拉霉素提供优良的高产菌株。
1 材料与方法 1.1 实验材料 1.1.1 菌株、质粒和引物: 本实验所用的菌株与质粒如表 1所示。PCR引物如表 2所示,其中加粗并标下划线的部分为酶切位点及突变位点。| 菌株及质粒 Strains and plasmids |
特征 Characterizations |
来源 Sources |
| Escherichia coli JM109 | Host strain | Laboratory stock |
| E. coli ET12567 | pUZ8002,Kmr,Cmlr | Laboratory stock |
| S. fungicidicus F1 | Wild type | Laboratory stock |
| S. fungicidicusL-M1 | Lys (43) of gene rpsL to Asn | This study |
| S. fungicidicusL-M2 | Lys (43) of gene rpsL to Arg | This study |
| pUCm-T | Cloning vector,Ampr | Sangon Biotech |
| pKC1139 | Shuttle plasmid of Streptomyces and E. coli,Aprar,repts | Laboratory stock |
| 引物 Primers |
引物序列 Primer sequence (5′→3′) |
扩增片段的大小 Size (bp) |
| rpsLF1 | GTGCCTACGATCCAGCAGCT | 370 |
| rpsLR1 | TTACTTCTCCTTCTTGGCGC | 370 |
| rpsLF2 | GCCGAGTTCGGCTTCTTCG | 1 400 |
| rpsLR2 | ACAACCTGCAGGAGCACTCC | 1 400 |
| S12F1 | CCGAATTCTGAACGGCAAGGCGGTCGC | 1 450 |
| S12R1 | GCAAGCTTGCAGGTCAAGTGAAGTGGTA | 1 450 |
| MrpsLF1 | ACCACCCCGAACAAGCCGAA | 778 |
| MrpsLR1 | TTCGGCTTGTTCGGGGTGGT | 671 |
| MrpsLF2 | ACCACCCCGCGGAAGCCGAA | 778 |
| MrpsLR2 | TTCGGCTTCCGCGGGGTGGT | 671 |
| Apra-F1 | AGGTAGCTGTATGGGGTGTTCC | 820 |
| Apra-R1 | CATGTGCGCCGATATAAACAT | 820 |
通过重叠PCR,获得了突变的rpsL基因M1和M2,测序得到了突变基因的完整序列,利用vector NTI将核苷酸序列翻译成氨基酸序列,与原序列进行比对,结果见图 1。

|
| 图 1 原rpsL基因与突变基因M1和M2氨基酸序列比对 Figure 1 Amino acid sequence alignment of rpsLand M1 and M2 |
|
|
由图 1可知,与对照组相比,突变基因M1和M2编码的氨基酸序列的第43位点的赖氨酸分别被替换为天冬酰胺和精氨酸,符合预期设计。
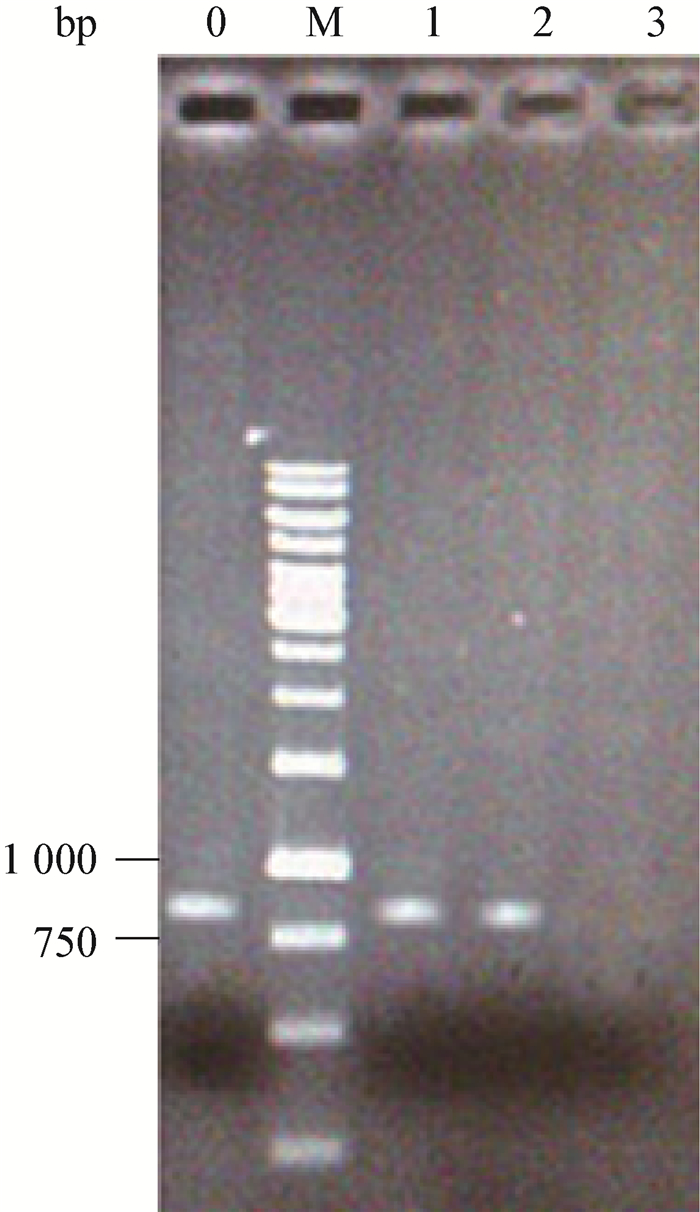
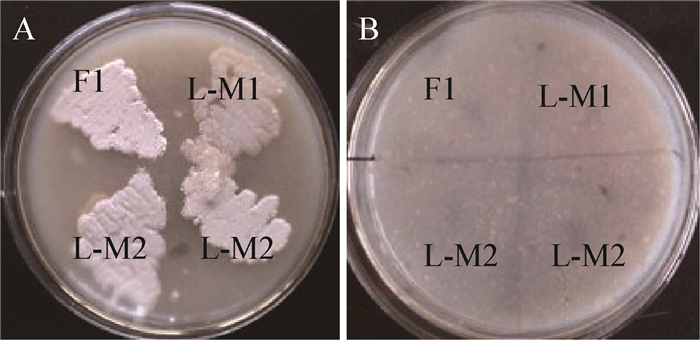
2.2 突变菌株的构建与筛选通过接合转移,成功将突变质粒导入了杀真菌素链霉菌孢子中,以Apra-F1/Apra-R1 (表 2)为引物,进行孢子PCR,对筛选到的突变菌株进行验证,结果如图 2所示。第一次同源重组:由于pKC1139属于温度敏感型质粒,当培养温度升高至40 ℃后,处于游离状态的质粒会自然丢失。利用这一特点,将阳性接合子转接到含有安普霉素抗性的MS培养基中,40 ℃培养,传代2−3次,使游离质粒彻底丢失,筛选在含有安普霉素抗性平板中仍能正常生长的接合子,即游离质粒成功整合到染色体DNA上的接合子;第二次同源重组:将上述接合子转接到无抗的MS培养基中,28 ℃培养,在没有选择压力的情况下,连续传代3−4次后,影印到不含安普霉素抗性和含有安普霉素抗性的平板中,28 ℃培养,挑取在不含抗性的平板中生长而在含有安普霉素的平板中不能生长的接合子(图 3),此时得到的接合子中有一部分可能已经成功发生第二次同源重组。
|
| 图 2 安普霉素抗性基因孢子PCR产物电泳图 Figure 2 Agarose gel electrophoresis of spore PCR production of Aprar 注:M:10 kb marker;0:pKC1139对照;1:M1型阳性接合子;2:M2型阳性接合子;3:野生型杀真菌素链霉菌对照. Note: M: 10 kb marker; 0: pKC1139 control; 1: type M1 positive trans-conjugant; 2: type M2 positive trans-conjugant; 3: S. fungicidicus F1 control. |
|
|
|
| 图 3 接合转化子转接划线平板 Figure 3 The switching streak plate of conjugation transformant 注:A:不含安普霉素的MS培养基;B:含有安普霉素的MS培养基. Note: A: MS medium without apramycin; B: MS medium with apramycin. |
|
|
提取接合子的基因组,利用PCR技术,以rpsLF1/R1为引物,扩增核糖体S12的rpsL基因,送出测序,对测序结果进行比对分析,比对结果如图 1所示,说明突变体构建成功。最终,M1型突变共获得3株接合子;M2型突变共获得4株接合子。
2.3 突变菌株产孢能力的考察将野生型菌株F1、M1及M2型突变菌株转接到同一个MS固体平板上,在同样的培养条件下,野生型菌株需要5−7 d才能产生孢子,而突变菌株在3 d后就可以产生大量的孢子。从图 4中可以看出突变菌株的产孢周期明显早于野生型菌株。

|
| 图 4 突变菌株与野生菌株产孢能力的考察 Figure 4 The sporulation ability of modification strain and wild strain |
|
|
对上述实验获得的3株M1型突变接合子和4株M2型突变接合子进行多批次的摇瓶发酵实验,同时以野生型杀真菌素链霉菌为对照。摇瓶发酵后,经HPLC分析显示:M1型突变菌株的恩拉霉素产量最高可达到1 334 U/mL,与野生型菌株相比,提高了11.9% (表 3);而M2型突变菌株的恩拉霉素产量最高可达到1 456 U/mL,与野生型菌株相比,提高了22.1% (表 3)。同一突变型的不同接合子的恩拉霉素产量存在差异,在误差允许的范围内,可以忽略不计。实验结果表明,将核糖体S12蛋白的编码基因rpsL的第43位赖氨酸进行突变,可以达到提高恩拉霉素的产量目的,尤其是将43位的赖氨酸突变成精氨酸时,产量提高幅度更大,可达到20%以上。
| 菌株编号 Strain number |
发酵时间 Fermentation time (d) |
恩拉霉素产量 Enramycin yield (U/mL) |
提高率 Increase rate (%) |
| F1 | 10 | 1192±23 | 0 |
| L-M1-1 | 10 | 1318±26 | 10.6 |
| L-M1-2 | 10 | 1334±29 | 11.9 |
| L-M1-3 | 10 | 1329±21 | 11.5 |
| L-M2-1 | 10 | 1456±34 | 22.1 |
| L-M2-2 | 10 | 1442±25 | 20.9 |
| L-M2-3 | 10 | 1435±28 | 20.4 |
| L-M2-4 | 10 | 1423±26 | 19.4 |
对突变菌株L-M1-2及L-M2-1连续的传代培养,以4 ℃保存的生长良好的第一代菌株为对照,结果表明,突变菌株L-M1-2第1−5代的相对发酵单位分别为100%、99.3%、98.2%、100.3%、99.1%;突变菌株L-M2-1第1−5代的相对发酵单位分别是100%、97.4%、99.5%、99.3%、101.2%。表明这两株高产突变菌株传4代以内,对恩拉霉素的发酵水平没有较为明显的影响,说明这两株突变菌株具有良好的遗传稳定性。
3 结论利用核糖体工程技术,对核糖体蛋白的结构进行修饰、改造,造成核糖体功能改变,可间接调控次级代谢产物的合成,从而获得突变的高产菌株。大量研究案例表明,利用该技术所进行的实验研究具有一定的可行性和实用性,在工业微生物育种中具有良好的应用前景。本文利用核糖体工程技术,对恩拉霉素产生菌S. fungicidicus F1中编码核糖体S12蛋白的基因rpsL进行定点改造,将该蛋白中的第43位的赖氨酸(Lys)替换为天冬酰胺(Asn)和精氨酸(Arg),以安普霉素作为抗性遗传筛选标记,得到突变菌株L-M1和L-M2,与野生菌株F1相比较,突变菌株恩拉霉素产量分别可提高11.9%和22.1%,并且遗传稳定性较好;突变后的菌株生长特性也发生了改变,能在较短时间内产生大量的孢子,大大缩短链霉菌的培养周期及恩拉霉素的生产周期,更适用于工厂大规模生产。同时,本实验结果也为核糖体蛋白其他位点的突变提供了可行性。
| [1] | Higashide E, Hatano K, Shibata M, et al. Enduracidin, a new antibiotic. I. Streptomyces fungicidicus No. B5477, an enduracidin producing organism[J]. The Journal of Antibiotics 1968, 21(2) : 126–137. DOI:10.7164/antibiotics.21.126 |
| [2] | McCafferty DG, Cudic P, Frankel BA, et al. Chemistry and biology of the ramoplanin family of peptide antibiotics[J]. Biopolymers 2002, 66(4) : 261–284. DOI:10.1002/(ISSN)1097-0282 |
| [3] | Yin XH, Zabriskie TM. The enduracidin biosynthetic gene cluster from Streptomyces fungicidicus[J]. Microbiology 2006, 152(Pt10) : 2969–2983. |
| [4] | Pedroso AA, Menten JF, Lambais MR, et al. Intestinal bacterial community and growth performance of chickens fed diets containing antibiotics[J]. Poultry Science 2006, 85(4) : 747–752. DOI:10.1093/ps/85.4.747 |
| [5] | Wang JL, Xu MY, Shi TG, et al. UV mutagenesis for high-yield enramycin producing strains[J]. Industrial Microbiology 2015, 45(5) : 23–28. (in Chinese) 王建玲, 许铭玉, 石天歌, 等. 紫外诱变选育恩拉霉素高产菌株[J]. 工业微生物 2015, 45(5) : 23–28. |
| [6] | Ochi K, Okamoto S, Tozawa Y, et al. Ribosome engineering and secondary metabolite production[J]. Advances in Applied Microbiology 2004, 56 : 155–184. DOI:10.1016/S0065-2164(04)56005-7 |
| [7] | Hesketh A, Ochi K. A novel method for improving Streptomyces coelicolor A3(2) for production of actinorhodin by introduction of rpsL (encoding ribosomal protein S12) mutations conferring resistance to streptomycin[J]. The Journal of Antibiotics 1997, 50(6) : 532–535. DOI:10.7164/antibiotics.50.532 |
| [8] | Okamoto-Hosoya Y, Sato TA, Ochi K. Resistance to paromomycin is conferred by rpsL mutations, accompanied by an enhanced antibiotic production in Streptomyces coelicolor A3(2)[J]. The Journal of Antibiotics 2000, 53(12) : 1424–1427. DOI:10.7164/antibiotics.53.1424 |
| [9] | Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual[M]. 3rd Edition. New York: Cold Spring Harbor Laboratory Press, 2002 . |
| [10] | Kieser T, Bibb MJ, Buttner MJ, et al. Practical Streptomyces Genetics[M]. Norwich: The John Innes Foundation, 2000 . |
| [11] | Fang ZK, Hong WR, Yan LB, et al. Construction of the conjugal transfer system of Streptomyces aureofaciens[J]. Journal of Fujian Agriculture and Forestry University (Natural Science Edition) 2011, 40(5) : 521–524. (in Chinese) 方志锴, 洪文荣, 严凌斌, 等. 金色链霉菌接合转移体系的构建[J]. 福建农林大学学报:自然科学版 2011, 40(5) : 521–524. |
 2017, Vol. 44
2017, Vol. 44