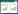扩展功能
文章信息
- 陆利, 曹保华, 王义强
- LU Li, CAO Bao-Hua, WANG Yi-Qiang
- 大肠杆菌CFT073中Curli系统重要基因csgF的克隆、表达、纯化和结构分析
- Expression, purification and structural analysis of csgF gene of curli systems from Escherichia coli CFT073
- 微生物学通报, 2016, 43(9): 2063-2071
- Microbiology China, 2016, 43(9): 2063-2071
- DOI: 10.13344/j.microbiol.china.150752
-
文章历史
- 收稿日期: 2015-10-07
- 接受日期: 2016-02-03
- 优先数字出版日期(www.cnki.net): 2016-03-10
2. 中南林业科技大学 湖南省经济林培育与利用协同创新中心 湖南 长沙 410004 ;
3. 中国科学院生物物理研究所 北京 100101
2. Hunan Provincial Cooperative Innovation Center of Non-Wood Forest Cultivation and Utilization, Central South University of Forestry and Technology, Changsha, Hunan 410004, China ;
3. Institute of Biophysics, Chinese Academy of Sciences, Beijing 100101, China
淀粉样蛋白纤维(Amyloids)是一类错误折叠引发疾病的蛋白聚集体。近年来人们在细菌、真菌、蛛形纲动物、鱼和哺乳动物中发现了功能淀粉样蛋白纤维[1-6]。Curli是一种典型的功能淀粉样纤维,它是大肠杆菌(Escherichia coli)和沙门氏菌(Salmonella spp.)中表达产生的一类附着在细胞基质外的纤维[7]。在生物膜形成过程中,Curli是细胞外基质重要的组成部分,且具有调节细胞表面的吸附能力、细胞间相互作用和稳定三维结构、促使成熟的生物膜形成等重要功能[8-10]。此外,Curli也是细菌抵抗恶劣的外界环境(干旱和抗生素)毒害的 一个重要的保护屏障[11-12]。
Curli除在细菌生理学方面有重要作用,也是研究淀粉样蛋白形成机制的重要模型。Curli是由富含β-折叠的高度有序蛋白质在胞外聚集形成的,4 nm−7 nm宽且长无分支的一类具有功能的淀粉样蛋白[13]。Curli与一系列淀粉样疾病相关,如阿尔兹海默病(Alzheimer disease,AD)、帕金森病(Parkinson’s disease,PD)、2型糖尿病(Type-2 diabetes)和肾功能衰竭等[14]。Curli是由两种亚基组成:CsgA和CsgB,且每种亚基都含有5个重复的谷氨酸和天冬氨模体,均表现出淀粉样蛋白的生化特征[15-16]。CsgA聚合物在体外处于无序状态,自我折叠途径堆积形成纤维,冷静等[17]以融合表达的方法获得了MBP-CsgA融合蛋白。而CsgA在细菌外表面有效正确的组装是在Curli亚基参与调控下完成。CsgG寡聚脂蛋白稳定地镶嵌在细胞外膜上,负责输送Curli途径中最大亚基CsgA和最小亚基CsgB到细胞外[18-19]。黄亿华课题组最近解析出的CsgG蛋白晶体结构表明其以九聚体形式镶嵌在外膜上,形成一个非选择性的离子通道[20],CsgA和CsgB一旦分泌到细胞外膜外侧,CsgB作为成核剂诱发CsgA聚合,通过Type-VIII分泌途径形成细胞外基质纤维[21]。CsgE和CsgF是Curli分泌途径中两个重要的分子伴侣,周质空间的CsgE直接与CsgA相互作用防止其在周质空间过早聚集[22],暴露在细胞外膜上的CsgF参与CsgB调控CsgA的聚合[23]。除此之外,CsgD转录调控因子调控操纵子csgBAC的表达[24],CsgC最新研究表明其具有抑制CsgA在体内过早聚集的特性[25]。Curli的有效组装过程均需CsgE和CsgF两种亚基的参与,且发现它们可能与外膜上的CsgG蛋白有相互作用,但目前对CsgF在Curli生物合成途径中的具体作用尚不清楚。
本研究通过在大肠杆菌中克隆、表达大肠杆菌CFT073重组CsgF蛋白,探索稳定CsgF蛋白的条件,最终获得稳定的、高纯度的CsgF蛋白,研究其与CsgG蛋白的相互作用,并对其结构进行初步分析。这为进一步研究Curli生物合成机制提供重要的理论根据,同时也为研究开发治疗淀粉样疾病的药物和筛选重要的药物靶点提供参考。
1 材料与方法 1.1 菌种、质粒、试剂和仪器Escherichia coli CFT073干粉,购自美国菌种保藏中心(ATCC)。E. coli DH5α、E. coli BL21(DE3),购自TaKaRa公司;T4 DNA连接酶、限制性内切酶,购自New England Biolabs;氨苄青霉素(Ampicillin)、卡那霉素(Kanamycin)以及PCR引物,购自上海生工生物工程有限公司;细菌基因组DNA提取试剂盒、琼脂糖凝胶DNA回收试剂盒、质粒提取试剂盒,购自天根生化科技(北京)有限公司;表达载体pET-28a(+)、pQLink-N,购自Novagen公司;DNA Marker、蛋白质Marker、上样缓冲液,购自博奥森生物技术有限公司;高保真DNA聚合酶、dNTPs体系,购自Genstar公司;PVDF膜,购自Invitrogen公司;其他试剂均为国产分析纯。PCR扩增仪、凝胶成像分析系统、电泳仪,Bio-Rad公司;高速离心机,美国贝克曼库尔特有限公司;AKTA蛋白纯化系统,美国通用电气;超声波细胞破碎仪,宁波市双嘉仪器有限公司。
1.2 重组质粒的构建从NCBI (http:www.ncbi.nlm.nih.gov)网站获取大肠杆菌CFT073中csgF基因的核酸序列(AE014075),E. coliCFT073菌的基因组DNA以细菌基因组DNA提取试剂盒提取,1%的琼脂糖凝胶电泳检测。根据序列分析和二级结构预测,按图 1所示原理,设计的引物见表 1,以E. coliCFT073的基因组DNA为模板,克隆出csgF基因的不同核酸片段,1%的琼脂糖凝胶电泳检测PCR产物。PCR扩增体系为:gDNA 1 μL,10xPCR buffer 5 μL,2.5 mmol/L MgCl2 5 μL,2.5 mmol/L dNTPs 5 μL,5 U/μL高保真的DNA聚合酶0.5 μL,10 μmol/L上游引物和下游引物各1 μL,ddH2O补齐至50 μL。PCR扩增条件:95 ℃ 10 min;95 ℃ 30 s,62 ℃ 30 s,68 ℃ 1 min (延伸时间根据1 kb/min计算),30个循环;72 ℃ 10 min;4 ℃保存。Nde I/Hind III双酶切PCR产物和pET-28a(+)质粒,酶切产物用琼脂糖凝胶回收试剂盒回收,T4连接酶连接,室温连接1 h转化到E. coli DH5α感受态细胞中,涂布卡那霉素(Kanamycin)抗性平板进行培养,挑取单菌落依次进行质粒的提取、酶切和测序,测序由北京睿博生物科技有限公司完成。
|
| 图 1 CsgF蛋白二级结构预测和重组质粒构建原理 Figure 1 The predicted secondary structure of CsgF and sketch map of recombinant plasmids 注:A:psipred (http://bioinf.cs.ucl.ac.uk/psipred/)预测的CsgF蛋白质的二级结构图,其中红色的圆柱代表α-螺旋结构,青绿色的箭头代表β-转角,黑色的实线代表无序卷曲;B:根据预测的二级结构设计出构建的重组质粒模式图. Note: A: The map of secondary structural CsgF was predicted by psipred data,Red columns represent alpha helix structure,Turquoise arrows represent β-corner,The black solid lines represent unordered curled up; B: Schematic diagram of recombinant plasmids. |
|
|
| 引物 Primers | 序列 Sequences (5′→3′) |
| pET28a-csgF-c-6His_F | GGAATTCCATATGGGAACCATGACTTTCCAGTTCCG |
| pET28a-csgF-c-6His_R | CGCCAAGCTTAAAATCGGTTGAGTTATTTTGTAAACC |
| pET28a-csgF(nsp)-c-6His_F | GGAATTCCATATGGGAACCATGACTTTCCAGTTCCG |
| pET28a-csgF(nsp)-c-6His_R | CGCCAAGCTTAAAATCGGTTGAGTTATTTTGTAAACC |
| pET28a-csgF(nsp)-N-6His_F | GGAATTCCATATGCACCATCACCATCACCATGGAACCATGACTTTCCAGTTCCG |
| pET28a-csgF(nsp)-N-6His_R | CGCCAAGCTTTTAAAAATCGGTTGAGTTATTTTGTAAACC |
| pET28a-csgF(20-129)-N-6His_F | GGAATTCCATATGCACCATCACCATCACCATGGAACCATGACTTTCCAGTTCCG |
| pET28a-csgF(20-129)-N-6His_R | CGCCAAGCTTTTATTGGACCTACCAGCTCCA |
| 注:nsp:没有信号肽. Note: nsp: no signal peptide. | |
将构建好的重组质粒转化到E. coli BL21中,挑取单菌落接种于5 mL卡那霉素(50 mg/L)抗性的LB液体培养基[26]中,活化培养过夜,再按1׃1 000的比例接种到1 L卡那霉素抗性的LB培养液中,37 ℃、220 r/min培养。0.1 mmol/L的IPTG、18 ℃、220 r/min诱导18−20 h。4 ℃、4 000 r/min离心 30 min收集菌体,菌体沉淀溶解于30 mL Buffer A重悬,Buffer A:20 mmol/L Tri-HCl pH 7.0、 150 mmol/L NaCl、5% Glycerol;超声波破碎细菌0 ℃,30 W,超声工作1 s,间隔5 s,共超声5 min,4 ℃、18 000 r/min离心1 h,取上清液和沉淀溶解于上样缓存液中,在15% SDS-PAGE电泳分析。
1.4 重组蛋白Western blotting鉴定取10 μL纯化后的重组蛋白进行SDS-PAGE电泳,凝胶在TTBS中平衡20 min后,转印40 min至PVDF膜上;取出PVDF膜放入含1 g/L BSA的TTBS中1 h;将抗6His标签的单抗(1:3 000稀释)浸泡膜并4 ℃过夜,以缓冲液TTBS洗涤膜3次,每次10 min,加入HRP 标记的羊抗小鼠抗 体(1:3 000稀释),置37 ℃孵育1 h;以缓冲液TTBS洗涤膜3次,每次10 min,将PVDF膜放入显色底物溶液,DAB显色10−30 min,用ddH2O洗膜,终止反应,观察。
1.5 脂溶性CsgF蛋白的纯化将诱导表达的菌液离心收集,超声波细胞破碎,离心弃除上清,离心沉淀溶解于Buffer B: 25 mmol/L Tris-HCl pH 7.0、150 mmol/L NaCl、 5% Glycerol、1% N-lauroylsarcosiner (LDAO),匀浆,4 ℃搅拌孵育1 h。蛋白液4 ℃、18 000 r/min离心 1 h,收集上清液,加到预冷的Ni-resin中;4 ℃搅拌孵育1 h装柱;10倍柱体积的咪唑梯度洗脱杂蛋白,5倍柱体积的300 mmol/L咪唑洗脱,获得目的蛋白CsgF-C-6His,用Millipore超滤浓缩管(MWCO:15 kD)浓缩至500 μL,上样于装有Superouse6 10/300 GL的AKTA蛋白纯化系统进行分析,分子筛Buffer D:25 mmol/L Tris-HCl pH 7.0、150 mmol/L NaCl、5% Glycerol、0.1% LDAO,结果蛋白不稳定。随后探索稳定蛋白的条件:改变甘油浓度10%,从DDM、DM、OG中筛选合适的去污剂,改变Buffer的pH值为5.0−9.0。
1.6 水溶性CsgF蛋白的纯化将诱导表达的菌液离心收集,超声波细胞破碎,离心弃除沉淀,收集上清液,加到预处理、预冷2 mL Ni-resin中,4 ℃搅拌孵育1 h后装柱; 10倍柱体积的咪唑梯度洗脱杂蛋白,5倍柱体积的 300 mmol/L咪唑洗脱,获得目的蛋白CsgF(nsp)-C-6His、CsgF(nsp)-N-6His。用Millipore超滤浓缩管(MWCO:15 kD)将目的蛋白浓缩至 500 μL,上样装有Superouse6 10/300 GL的AKTA蛋白纯化系统进行分析,Buffer A:20 mmol/L Tri-HCl pH 7.0、150 mmol/L NaCl、5% Glycerol,结果表明蛋白质还是不稳定。随后开展优化去污剂和Buffer的条件。最终CsgF截短体能稳定存在于Buffer E:50 mmol/L 醋酸钠pH 5.0、150 mmol/L NaCl、5% Glycerol。
1.7 CsgG蛋白的表达纯化csgG基因克隆到pQLink-N载体上进行表达,将构建好的重组质粒pQLink-CsgG转化到E. coliBL21中,挑取单菌落接种于Amp抗性1 L的LB液体培养基中,37 ℃、220 r/min下培养OD值至0.6−0.8,0.1 mmol/L IPTG、18 ℃、220 r/min诱导表达18−20 h。4 ℃、4 000 r/min离心30 min收集菌体。用Buffer A重悬菌体,超声波破碎细胞,4 ℃、18 000 r/min 离心1 h,弃上清,沉淀溶解于Buffer F:25 mmol/L Tris-HCl、150 mmol/L NaCl、 5% Glycerol、1% Sarkosyl;4 ℃搅拌孵育1 h;再4 ℃、18 000 r/min离心1 h,弃上清,沉淀溶解于Buffer D;4 ℃搅拌孵育1 h;再4 ℃、18 000 r/min离心1 h,保留上清液,取样10% SDS-PAGE电泳检测。
1.8 CsgF与CsgG的Pull down实验将预先纯化的CsgF-N-6His、CsgF-C-6His、CsgF-(20−129)-N-6His融合蛋白用透析法去除Buffer E中咪唑,用BCA protein分析试剂盒测定浓度,加到Buffer G预平衡、预冷的2 mL钴螯合Resin中,Buffer G:50 mmol/L醋酸钠pH 5.0、150 mmol/L NaCl、5% Glycerol、40 mmol/L Imidazole;4 ℃孵育1 h,装柱;Buffer G洗脱未结合的杂蛋白(每次10 mL,洗5次),将固定化CsgF的钴Resin转移至预冷的小锥形瓶中(4 ℃);同时用AKTA蛋白纯化系统将CsgG蛋白中的Buffer交换成Buffer H (50 mmol/L醋酸钠pH 5.0、150 mmol/L NaCl、 5% Glycerol、0.1% LDAO ),Millipore超滤浓缩管(MWCO:30 kD)浓缩至2 mL;再平衡CsgG蛋白液中咪唑终浓度为40 mmol/L,将CsgG蛋白液与CsgF蛋白液4 ℃温柔搅拌孵育1 h,装柱,用Wash buffer (50 mmol/L醋酸钠pH 5.0、150 mmol/L NaCl、5% Glycerol、0.1% LDAO、40 mmol/L Imidazole)洗脱,未结合蛋白;Elution buffer:50 mmol/L醋酸钠pH 5.0、150 mmol/L NaCl、5% Glycerol、0 .1% LDAO、300 mmol/L Imidazole洗脱,获得目的蛋白。目标蛋白用Millipore超滤浓缩管(MWCO:30 kD)浓缩至500 μL,上样于装有Superouse6 10/300 GL的AKTA蛋白纯化系统进行分析,收集峰尖几管蛋白样品,制样用15% SDS-PAGE电泳检测。
1.9 CsgF蛋白的结构分析csgF基因的核酸序列和蛋白序列从NCBI (http://www.ncbi.nlm.nih.gov)上获取,采用in silico方法进行分析。信号肽和亚细胞定位分别从Uniprot (http://www.uniprot.org/)和TMHMM (http://www.cds.dtu.dk/services/TMHMM-2.)进行预测。CsgF蛋白质的二级结构通过蛋白质二级结构预测平台预测(http://bioinf.cs.ucl.ac.uk/psipred/)。CsgF蛋白与其他蛋白相互作用通过STRING数据库进行预测(http://string-db.org)。三级结构使用SWISS-MODEL服务平台基于同源性进行建模。
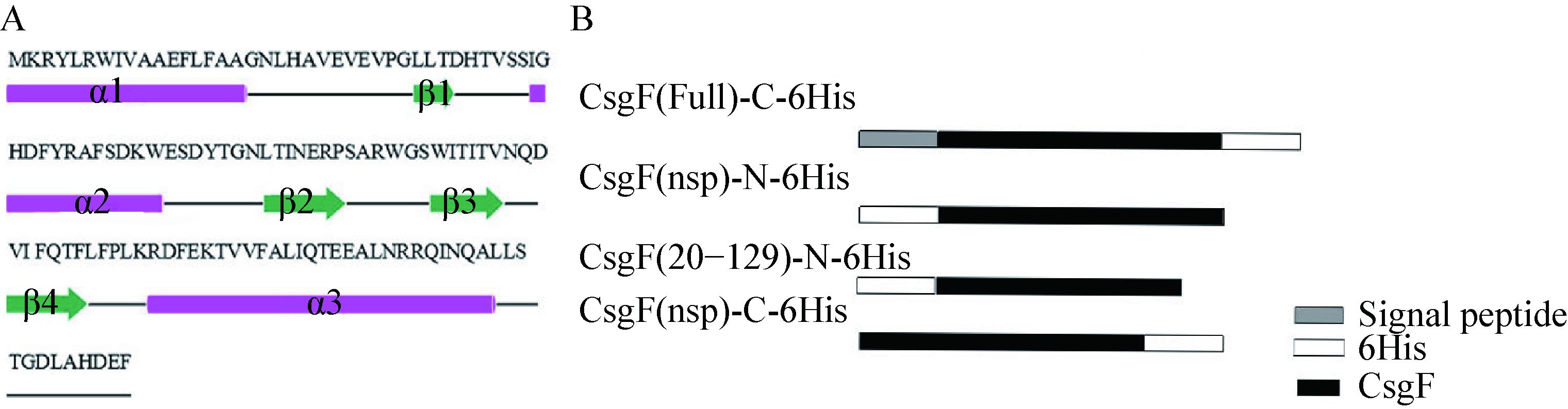
2 结果与分析 2.1 csgF基因的克隆PCR技术克隆出csgF基因不同长度的核酸片段(图 2),大约417 bp的DNA片段,含有139个完整的开发阅读框(ORF),编码139个氨基酸,分子量约为15 kD。csgF基因的不同片段克隆到pET-28a(+)表达载体中,得图 1中4种重组质粒,Nde I/Hind III双酶切鉴定正确,测序结果也表明:重组质粒中插入序列完全正确。
|
| 图 2 目的片段的PCR扩增电泳图 Figure 2 Agarose gel electrophoresis of target PCR products 注:M:DNA marker DL10000;1:CsgF的C端带6个His的扩增片段;2:CsgF无信号肽C端带6个His的扩增片段;3:CsgF无信号肽N端带6个His的扩增片段;4:CsgF截短体(20−129) N端带6个His的扩增片段. Note: M: DNA marker DL10000; 1: CsgF(Full)-C-6His; 2: CsgF(nsp)-C-6His; 3: CsgF(nsp)-N-6His; 4: CsgF(20−129)- N-6His. |
|
|
pET28a-csgF(Full)-c-6His、pET28a-csgF(nsp)- C-6His、pET28a-csgF(nsp)-N-6His、pET28a-csgF (20−129)-N-6His四种重组质粒分别转化到E. coliBL21(DE3)中,异源表达产CsgF-6His标签的融合蛋白。终浓度为0.1 mmol/L IPTG 18 ℃低温诱导CsgF蛋白表达,镍柱亲和层析纯化,15% SDS-PAGE电泳分析结果如图 3所示,结果表明目的蛋白CsgF不管是在膜上还是在胞质中均能表达。

|
| 图 3 重组CsgF蛋白纯化SDS-PAGE电泳检测 Figure 3 SDS-PAGE analysis of CsgF purified through Ni-chelating affinity chromatography 注:M:标准蛋白分子量标记;1:流穿液;2:25 mmol/L咪唑的洗脱液;3:40 mmol/L咪唑的洗脱液;4:50 mmol/L咪唑的洗脱液;5:300 mmol/L咪唑洗脱液. Note: M: Standard protein marker; 1: Flow through liquid; 2: Buffer A contain of 25 mmol/L imidazole; 3: Buffer A contain of 40 mmol/L imidazole; 4: Buffer A contain of 50 mmol/L imidazole; 5: Buffer A contain of 300 mmol/L imidazole. |
|
|
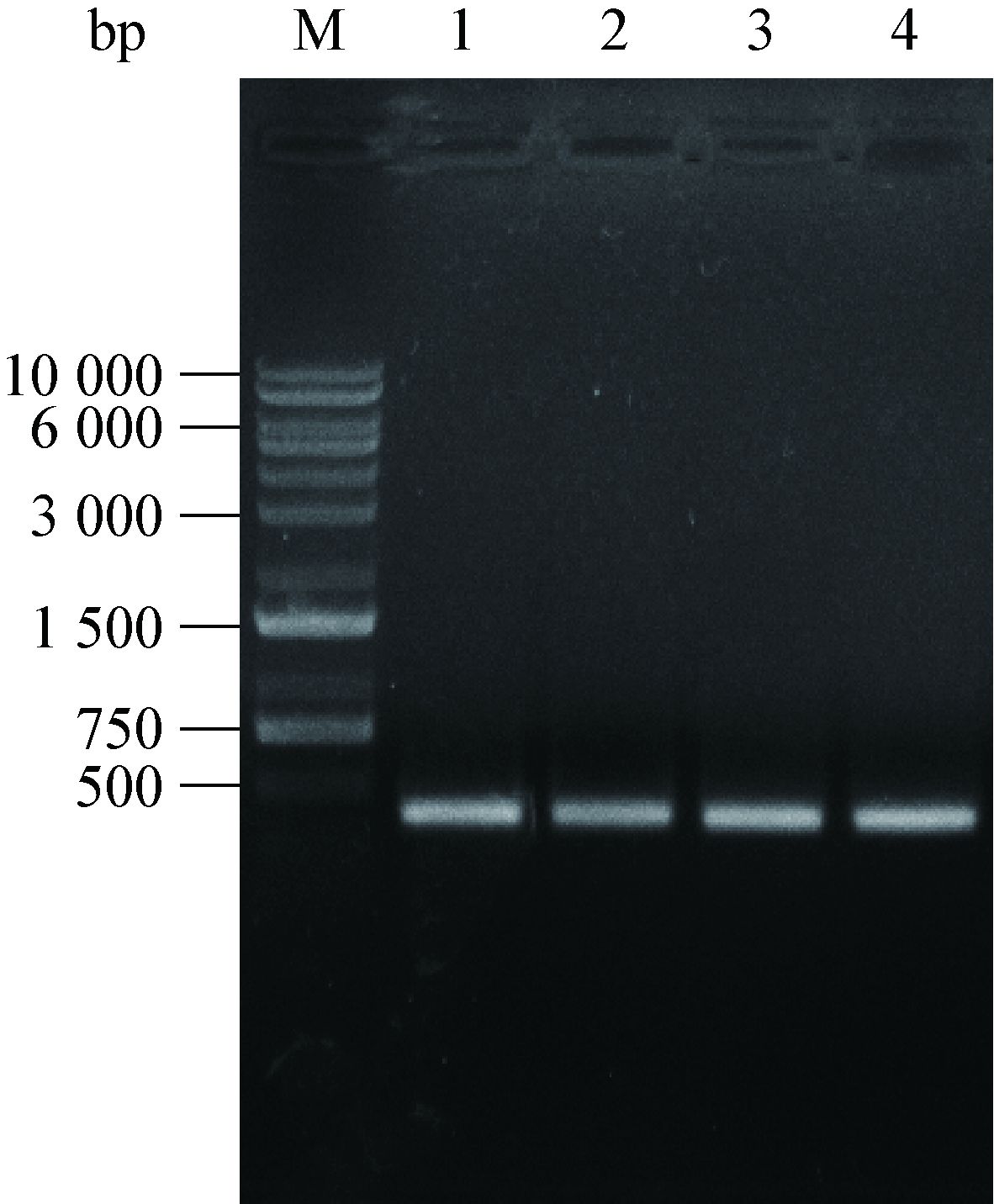
为获得低盐高浓度稳定的CsgF蛋白,需对镍柱亲和层析纯化后的CsgF蛋白用凝胶排阻层析(Gel exclusion chromatography)进行纯化。用Millipore超滤浓缩管(MWCO:30 kD)在4 ℃、 3 000 r/min下浓缩过程中,观察到白色沉淀产生,离心取上清500 μL上样于装有Superose6 10/300GL的AKTA蛋白纯化系统进行分析,结果未观察到目的蛋白吸收峰。优化Buffer条件:pH梯度5.0−9.0;去污剂DDM、LDAO、OG、NG及盐浓度 150−500 mmol/L NaCl和醋酸钠Buffer等,AKTA蛋白纯化系统检测分析及15% SDS-PAGE电泳结果如图 4所示,结果表明只有CsgF (20−129)截短体蛋白能稳定存在于Buffer E (50 mmol/L醋酸钠pH 5.0、150 mmol/L NaCl、5% Glycerol)。
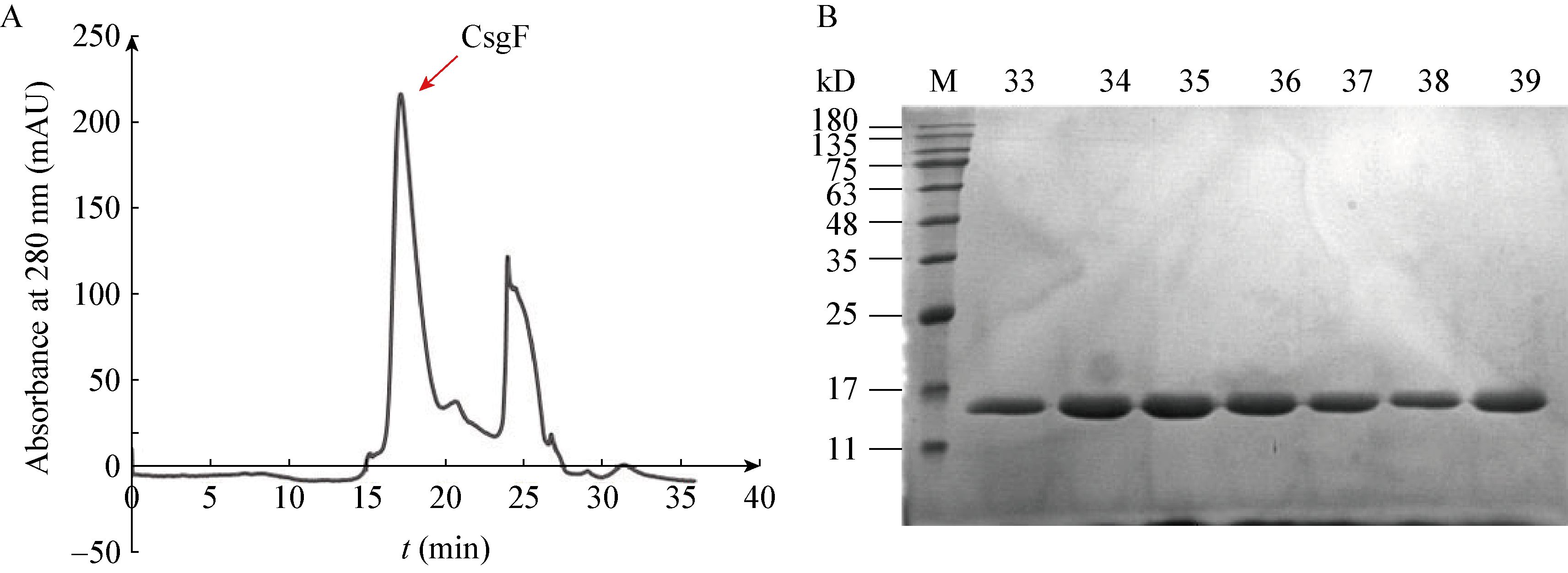
|
| 图 4 CsgF (20−129)截短体的稳定性分析 Figure 4 Stability analysis of CsgF (20−129) truncated body 注:A:CsgF (20−129)截短体在醋酸钠buffer下稳定存在的峰图;B:样品(33−39) 15% SDS-PAGE电泳检测;M:标准蛋白分子量标记. Note: A: The peak of CsgF (20−129) truncated body represent stability under sodium acetate buffer; B: The samples from the peak point location (33−39) detected by 15% SDS-PAGE electrophoresis; M: Standard protein marker. |
|
|
为研究CsgF与CsgG的互作关系,需重组表达纯化CsgG蛋白。CsgG蛋白表达纯化结果如图 5所示,300 mmol/L咪唑洗脱液可以将CsgG蛋白洗脱下来,并且获得了比较纯的CsgG蛋白。

|
| 图 5 重组CsgG蛋白纯化SDS-PAGE电泳检测 Figure 5 SDS-PAGE analysis of CsgG purified through Ni-chelating affinity chromatography 注:M:标准蛋白分子量标记;1:流穿液;2:25 mmol/L咪唑的洗脱液;3:40 mmol/L咪唑的洗脱液;4:50 mmol/L咪唑的洗脱液;5:300 mmol/L咪唑洗脱液. Note: M: Standard protein marker; 1: Flow through liquid; 2: Buffer A contain 25 mmol/L imidazole; 3: Buffer A contain 40 mmol/L imidazole; 4: Buffer A contain 50 mmol/L imidazole; 5: Buffer A contain 300 mmol/L imidazole. |
|
|
CsgF是一类锚定在外膜外侧的一类膜蛋白,黄亿华课题组[20]发现CsgG镶嵌在外膜上以九聚体的形式形成了一个跨膜通道,这个跨膜通道有一个中央空腔和前庭组成,CsgG周质侧与周质空间形成的区域被九聚体的水溶蛋白CsgE盖住,并选择性的调控CsgA蛋白的进出。而CsgF也可能以九聚体的形式与CsgG的外膜外侧部分相互作用。为验证其相互作用,采用His-pull down实验方法验证,分别采用带His标签的CsgF蛋白与无His标签CsgG的Pull down和带His标签的CsgG蛋白与无His标签CsgF的Pull down,Pull down实验结果表明:带His标签的CsgG能把无His标签的CsgF蛋白 Pull down,而带His标签的CsgF蛋白却不能把无His标签的CsgG蛋白Pull down。为进一步观察CsgG与CsgF复合物的分子状态,将CsgG-C-6His-CsgF复合物的蛋白液浓缩,上样于装有Superouse6 10/300GL的AKTA蛋白纯化系统进行分析,观察发现出峰的保留时间前移,并收集峰尖位置的蛋白样品制样用15% SDS-PAGE电泳分析,电泳分析的结果见图 6,结果表明28−35管的泳道均出现两条带,且两条带的位置与CsgG、CsgF大小相一致。为进一步分析其结合比例,采用Gel-Pro analyzer 4.0软件对CsgG和CsgF蛋白灰度定量分析,如表 2所示,CsgG与CsgF结合的比例暂无法确定,上述结果表明CsgG与CsgF存在相互作用。

|
| 图 6 CsgF与CsgG的Pull down实验电泳检测 Figure 6 SDS-PAGE analysis of experimental result of CsgF pull down CsgG 注:M:标准蛋白分子量标记. Note: M: Standard protein marker. |
|
|
| 泳道 Lane | 淀粉样蛋白G CsgG (ng) | 淀粉样蛋白F CsgF (ng) |
| 1 | 224.59 | 60.02 |
| 2 | 321.09 | 96.66 |
| 3 | 358.89 | 135.44 |
| 4 | 304.76 | 139.81 |
| 5 | 338.43 | 178.32 |
| 6 | 323.14 | 170.83 |
| 7 | 240.41 | 236.44 |
| 8 | 131.88 | 313.45 |
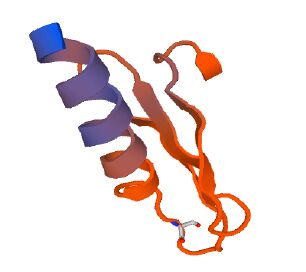
CsgF蛋白是由138个氨基酸组成,理论分子质量为15.05 kD,理论pI为5.59,CsgF在1−19个氨基酸为信号肽序列,并且定位在细胞外膜外侧(图 7)。二级结构预测59.8%是α-螺旋,10.3%是β-折叠,29.9%是无规则卷曲,基于CsgF同源性构建三维结构模型(图 8)。

|
| 图 7 CsgF亚细胞定位和跨膜区预测 Figure 7 Location of subcellular and prediction of transmembrane of CsgF protein |
|
|
|
| 图 8 CsgF蛋白的三维结构模型 Figure 8 the three-dimensional structural model of CsgF protein |
|
|
CsgF是一种定位在细胞外膜上的外膜蛋白。在Curli分泌系统中,CsgF负责将从CsgG孔道转运过来的CsgA呈递给Curli成核中心蛋白CsgB,然后CsgA组装形成Curli纤维。本文研究E. coli CFT073菌中的csgF基因,通过表达载体pET-28a(+)克隆表达。首先是表达脂溶性的CsgF蛋白(其原始定位于细胞外膜上),以去污剂将CsgF蛋白从外膜上提取出来,通过亲和层析和AKTA蛋白纯化系统纯化,结果CsgF蛋白吸收峰特别低,并且以高聚体的形式存在。当将CsgF蛋白在胞质中表达,分别构建除去信号肽序列的N端和C端带6个His的CsgF融合蛋白,将重组质粒转到E. coli BL21(DE3)中。37 ℃诱导表达时,CsgF蛋白主要以包涵体的形式存在。为获得水溶性的CsgF蛋白,通过降低诱导表达温度至18 ℃,结果获得高表达水溶性的CsgF蛋白,亲和层析和AKTA蛋白纯化系统纯化,结果发现与脂溶性的CsgF一样,吸收峰特别低且电泳未观察到CsgF蛋白带,说明CsgF在该Buffer条件下不稳定。尝试在不同温度(4 ℃和16 ℃)、不同的pH值范围(5.0−9.0)、添加1倍CMC浓度的不同去污剂(LDAO、DDM、OG、C8E4)、盐离子浓度(150、300、500 mmol/L NaCl)、Tris-HCl buffer和醋酸钠buffer的条件,结果均未能获得稳定的CsgF蛋白。最后,根据预测的二级结构结果,截掉C端柔性较大的部分,重新构建了CsgF (20−129)截短体,最后在醋酸钠Buffer E中获得了稳定的CsgF (20−129)截短体蛋白。
根据STRING数据库预测表明CsgF与CsgG、CsgE、CsgD存在互作关系,为验证CsgG与CsgF是否存在互作关系,同时也为了进一步揭示CsgF在Curli分泌途径中的功能,采用Pull down实验研究两种蛋白的互作关系。分别设计两种Pull down实验方案:一种是带His标签的CsgF与无His标签的CsgG蛋白Pull down;另一种是带His标签的CsgG与无His标签的CsgF蛋白Pull down。当Buffer E中的CsgF蛋白与Buffer D中的CsgG蛋白进行Pull down实验,未观察到CsgG与CsgF蛋白有互作。CsgG存在于有去污剂(LDAO)的Buffer D中,LDAO可能在CsgG外表面形成胶束团隔离其与CsgF蛋白的互作。改进Pull down实验条件,将CsgG蛋白的buffer交换成醋酸钠buffer,再进行Pull down实验,观察到同一泳道出现两条带,而阴性对照和阳性对照却均只有一条带,表明CsgG与CsgF蛋白之间确实存在互作关系。为进一步研究其结合强度及比例,通过AKTA蛋白纯化系统将CsgG/CsgF蛋白复合物浓缩上样,结果进一步表明 CsgG与CsgF蛋白存在互作关系。由于影响其结合比例的因素比较多,如Buffer的pH值、离子浓度,蛋白灰度定量分析结果暂时无法判断其结合比例。此外,CsgF蛋白本身不能稳定存在于醋酸钠buffer中,而在与CsgG进行Pull down实验中,发现CsgG能稳定CsgF蛋白在醋酸钠Buffer中存在。这一现象进一步说明CsgF与CsgG蛋白存在互作关系。同时Matthews也预测CsgF能与CsgG蛋白以某种特定的比例结合形成一个稳定高效的转运机器,完成CsgA蛋白的转运及组装[27]。本研究将着重研究CsgF与CsgG、CsgB互作位点,根据CsgF预测的三维结构,筛选一些关键位点开展定点突变实验,为开发新型的治疗淀粉样蛋白变性病筛选药物靶点提供重要的理论根据。
| [1] | Chapman MR, Robinson LS, Pinkner JS, et al. Role of Escherichia coli curli operons in directing amyloid fiber formation[J]. Science , 2002, 295 (5556) : 851–855. DOI:10.1126/science.1067484 |
| [2] | Elliot MA, Karoonuthaisiri N, Huang JQ, et al. The chaplins: a family of hydrophobic cell-surface proteins involved in aerial mycelium formation in Streptomyces coelicolor[J]. Genes & Development , 2003, 17 (14) : 1727–1740. |
| [3] | True HL, Lindquist SL. A yeast prion provides a mechanism for genetic variation and phenotypic diversity[J]. Nature , 2000, 407 (6803) : 477–483. DOI:10.1038/35035005 |
| [4] | Kenney JM, Knight D, Wise MJ, et al. Amyloidogenic nature of spider silk[J]. European Journal of Biochemistry , 2002, 269 (16) : 4159–4163. DOI:10.1046/j.1432-1033.2002.03112.x |
| [5] | Podrabsky JE, Carpenter JF, Hand SC. Survival of water stress in annual fish embryos: dehydration avoidance and egg envelope amyloid fibers[J]. American Journal of Physiology. Regulatory, Integrative and Comparative Physiology , 2001, 280 (1) : R123–R131. |
| [6] | Fowler DM, Koulov AV, Alory-Jost C, et al. Functional amyloid formation within mammalian tissue[J]. PLoS Biology , 2006, 4 (1) : e6. |
| [7] | Collinson SK, Emӧdy L, Müller KH, et al. Purification and characterization of thin, aggregative fimbriae from Salmonella enteritidis[J]. Journal of Bacteriology , 1991, 173 (15) : 4773–4781. |
| [8] | Austin JW, Sanders G, Kay WW, et al. Thin aggregative fimbriae enhance Salmonella enteritidis biofilm formation[J]. FEMS Microbiology Letters , 1998, 162 (2) : 295–301. DOI:10.1111/fml.1998.162.issue-2 |
| [9] | Vidal O, Longin R, Prigent-Combaret C, et al. Isolation of an Escherichia coli K-12 mutant strain able to form biofilms on inert surfaces: involvement of a new ompR allele that increases curli expression[J]. Journal of Bacteriology , 1998, 180 (9) : 2442–2449. |
| [10] | Kikuchi T, Mizunoe Y, Takade A, et al. Curli fibers are required for development of biofilm architecture in Escherichia coli K-12 and enhance bacterial adherence to human uroepithelial cells[J]. Microbiology and Immunology , 2005, 49 (9) : 875–884. DOI:10.1111/mim.2005.49.issue-9 |
| [11] | Ryu JH, Beuchat LR. Biofilm formation by Escherichia coli O157:H7 on stainless steel: effect of exopolysaccharide and curli production on its resistance to chlorine[J]. Applied and Environmental Microbiology , 2005, 71 (1) : 247–254. DOI:10.1128/AEM.71.1.247-254.2005 |
| [12] | White AP, Gibson DL, Kim W, et al. Thin aggregative fimbriae and cellulose enhance long-term survival and persistence of Salmonella[J]. Journal of Bacteriology , 2006, 188 (9) : 3219–3227. DOI:10.1128/JB.188.9.3219-3227.2006 |
| [13] | Shewmaker F, McGlinchey RP, Thurber KR, et al. The functional curli amyloid is not based on in-register parallel β-sheet structure[J]. The Journal of Biological Chemistry , 2009, 284 (37) : 25065–25076. DOI:10.1074/jbc.M109.007054 |
| [14] | Moreno-Gonzalez I, Soto C. Misfolded protein aggregates: Mechanisms, structures and potential for disease transmission[J]. Seminars in Cell & Developmental Biology , 2011, 22 (5) : 482–487. |
| [15] | Hammer ND, Schmidt JC, Chapman MR. The curli nucleator protein, CsgB, contains an amyloidogenic domain that directs CsgA polymerization[J]. Proceedings of the National Academy of Sciences of the United States of America , 2007, 104 (30) : 12494–12499. DOI:10.1073/pnas.0703310104 |
| [16] | Collinson SK, Parker JMR, Hodges RS, et al. Structural predictions of AgfA, the insoluble fimbrial subunit of Salmonella thin aggregative fimbriae[J]. Journal of Molecular Biology , 1999, 290 (3) : 741–756. DOI:10.1006/jmbi.1999.2882 |
| [17] | Leng J, Zheng Y, Zeng X, et al. Expression and purification of MBP-CsgAⅢ fusion protein[J]. Immunological Journal , 2006, 22 (4) : 390–392. (in chinese) 冷静, 郑禹, 曾霞, 等. MBP-CsgAⅢ融合蛋白的表达和纯化[J]. 免疫学杂志 , 2006, 22 (4) : 390–392. |
| [18] | Robinson LS, Ashman EM, Hultgren SJ, et al. Secretion of curli fibre subunits is mediated by the outer membrane-localized CsgG protein[J]. Molecular Microbiology , 2006, 59 (3) : 870–881. DOI:10.1111/mmi.2006.59.issue-3 |
| [19] | Loferer H, Hammar M, Normark S. Availability of the fibre subunit CsgA and the nucleator protein CsgB during assembly of fibronectin-binding curli is limited by the intracellular concentration of the novel lipoprotein CsgG[J]. Molecular Microbiology , 1997, 26 (1) : 11–23. DOI:10.1046/j.1365-2958.1997.5231883.x |
| [20] | Cao BH, Zhao Y, Kou YJ, et al. Structure of the nonameric bacterial amyloid secretion channel[J]. Proceedings of the National Academy of Sciences of the United States of America , 2014, 111 (50) : E5439–E5444. DOI:10.1073/pnas.1411942111 |
| [21] | Bian Z, Normark S. Nucleator function of CsgB for the assembly of adhesive surface organelles in Escherichia coli[J]. The EMBO Journal , 1997, 16 (19) : 5827–5836. DOI:10.1093/emboj/16.19.5827 |
| [22] | Nenninger AA, Robinson LS, Hammer ND, et al. CsgE is a curli secretion specificity factor that prevents amyloid fibre aggregation[J]. Molecular Microbiology , 2011, 81 (2) : 486–499. DOI:10.1111/mmi.2011.81.issue-2 |
| [23] | Nenninger AA, Robinson LS, Hultgren SJ. Localized and efficient curli nucleation requires the chaperone-like amyloid assembly protein CsgF[J]. Proceedings of the National Academy of Sciences of the United States of America , 2009, 106 (3) : 900–905. DOI:10.1073/pnas.0812143106 |
| [24] | Hammar M, Arnqvist A, Bian Z, et al. Expression of two csg operons is required for production of fibronectin-and congo red-binding curli polymers in Escherichia coli k-12[J]. Molecular Microbiology , 1995, 18 (4) : 661–670. DOI:10.1111/mmi.1995.18.issue-4 |
| [25] | Evans ML, Chorell E, Taylor JD, et al. The bacterial curli system possesses a potent and selective inhibitor of amyloid formation[J]. Molecular Cell , 2015, 57 (3) : 445–455. DOI:10.1016/j.molcel.2014.12.025 |
| [26] | Anderson EH. Growth requirements of virus-resistant mutants of Escherichia coli strain “B”[J]. Proceedings of the National Academy of Sciences of the United States of America , 1946, 32 (5) : 120–128. DOI:10.1073/pnas.32.5.120 |
| [27] | Taylor JD, Matthews SJ. New insight into the molecular control of bacterial functional amyloids[J]. Frontiers in Cellular and Infection Microbiology , 2015, 5 : 33. |
 2016, Vol. 43
2016, Vol. 43