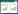扩展功能
文章信息
- 邢少贞, 张湘莉兰, 舒鹏, 孙强, 裴广倩, 米志强, 安小平, 王景林, 赵宝华, 童贻刚
- XING Shao-zhen, ZHANG Xiang-li-lan, SHU Peng, SUN Qiang, PEI Guang-qian, MI Zhi-qiang, AN Xiao-ping, WANG Jing-lin, ZHAO Bao-hua, TONG Yi-gang
- 粪肠球菌噬菌体vB_E.faecalis_IME196的生物学特性及其全基因组分析
- Characterization and complete genome sequence analysis of Enterococcus faecalis bacteriophage vB_E.faecalis_IME196
- 微生物学通报, 2016, 43(9): 2040-2048
- Microbiology China, 2016, 43(9): 2040-2048
- DOI: 10.13344/j.microbiol.china.151053
-
文章历史
- 收稿日期: 2015-12-22
- 接受日期: 2016-03-29
- 优先数字出版日期(www.cnki.net): 2016-04-08
2. 军事医学科学院微生物流行病研究所 北京 100071
2. Beijing Institute of Microbiology and Epidemiology, Beijing 100071, China
肠道微生态系统中寄居着大量菌群,其中既有益生菌、中间型菌群,也有致病菌[1]。益生肠球菌作为一类重要的益生菌,已普遍用于食品的加工[2]。目前,粪肠球菌是最常用的益生肠球菌,主要应用于食品发酵的工艺中,许多食物中均添加了粪肠球菌,例如香肠、奶酪、橄榄油等[3]。粪肠球菌(Enterococcus faecalis)属于肠球菌属[4]。当粪肠球菌异位寄生时可引起败血症、尿路感染、心内膜炎和化脓性腹部感染等疾病,尤其随着抗生素的不断更新换代,耐药肠球菌感染疾病病例逐渐增加[5],万古霉素曾经被认为是对付耐药菌株的最后一道防线,但在1986年出现了首例耐万古霉素肠球菌(VRE),随着肠球菌的耐药性愈加扩大,对人类的健康造成严重威胁,也向临床治疗耐药粪肠球菌提出了极大的挑战[6]。
噬菌体是一类能感染细菌等微生物的病毒。类似于感染动物和植物的病毒,噬菌体必需依靠宿主菌才能进行复制和繁殖。噬菌体是细菌的天然杀手,广泛存在于自然界中,随着细菌耐药性问题日益严重,新抗生素研发的速度远低于耐药菌产生的速度[7]。作为可替代抗生素成为新型抗菌剂的噬菌体,引起了国内外研究者们的重视。噬菌体的治疗具有独特优势:特异性强,只针对相应的病原菌,而不会破坏正常菌群;噬菌体的作用机制与抗生素完全不同,治疗效果不受细菌耐药性影响;噬菌体的指数增殖能力是噬菌体治疗的显著优势;副作用少;不易对噬菌体产生抗性等[8]。
除此之外,通过高通量测序得知全基因组序列,预测基因功能,这些研究有利于噬菌体治疗应用,对噬菌体的进化分析也将有助于对其生命规律进行深入了解。
1 材料与方法 1.1 材料10株粪肠球菌(Enterococcus faecalis)和10株屎肠球菌(Enterococcus faecium)均来自珠海迪尔公司收集医院临床分离女性阴道分泌物或者粪便。经生化鉴定和16S rRNA基因测序鉴定,由本实验室保存。4株金黄色葡萄球菌(Staphylococcus aureus)、2株大肠杆菌(Escherichia coli)来自本实验室细菌库所保存样品。BHI (Brain heart infusion)培养基,BD医疗器械有限公司;酚:氯仿:异戊醇液(25:24:1,体积比),北京索莱宝科技有限公司;聚乙二醇8000 (PEG8000),AMRESCO公司;蛋白酶K、RNase、DNase,Fermentas公司。
1.2 噬菌体的分离取未经处理的307医院污水,10 000 r/min离心10 min,取上清,用0.22 μm的滤器过滤污水,将上述过滤液100 μL加入到5 mL对数期指示菌粪肠球菌中。于37 ℃、220 r/min振荡培养过夜,然后10 000 r/min离心10 min,收集上清,用微孔滤膜(孔径0.22 μm)过滤处理,所得滤液即为噬菌体原液。
1.3 噬菌体的纯化与浓缩在有噬菌斑形成的双层BHI培养基平板[4]上挑取形态大小一致、单个独立噬菌斑,加入到5 mL的OD600值为0.4−0.6的宿主菌菌液内,37 ℃、 220 r/min摇床培养6 h左右,10 000 r/min离心 1 min,取上清液体,加入至500 mL的OD600值为0.4−0.6的宿主菌菌液内,37 ℃、220 r/min摇床培养12 h左右,然后用PEG沉淀法浓缩噬菌体[4]。
1.4 透射电子显微镜下观察噬菌体形态及大小将浓缩噬菌体液体取出50 μL,加50 μL 0.5%的戊二醛和4%的多聚甲醛,充分混合固定,取出30 μL滴入覆盖有Formvar膜的铜网上,大约静置30 min,用滤纸吸收多余液体,然后用2%的磷钨酸染色,静置10 min,室温下自然干燥后,在Philips TECNAI-10型的透射电镜下观察并记录噬菌体的形态和大小。
1.5 噬菌体最佳感染复数测定参照张文惠等[4]的方法,略有改动。
感染复数(multiplicity of infection,MOI),是感染时噬菌体与细菌的数量比值,也就是平均每个细菌所感染噬菌体的数量。而最佳感染复数,是可使噬菌体获得最佳生长状态时的感染复数。具体测定方法为:按MOI分别为0.01、0.1、1、10、100混合噬菌体悬液和宿主菌悬液,加入新鲜的BHI培养基补足体积至8 mL,于37 ℃、220 r/min振荡培养5 h;10 000 r/min离心1 min,分别用0.22 μm滤器过滤;梯度稀释后,双层平板法测定噬菌体的滴度,其中噬菌体滴度最高的MOI即为最佳感染复数。
1.6 噬菌体一步生长曲线测定参照张文惠等[4]的方法,略有改动。
将粪肠球菌2007培养至OD600值为0.4−0.6,取适量菌液倍比稀释后涂板,确定在此状态下每毫升菌液所含细菌个数;将粪肠球菌2007接种至适量体积的新鲜BHI培养基,培养至对数期后离心并重悬于BHI培养基,使其浓度达到108 CFU/mL,以感染复数MOI为0.01加入噬菌体IME196;置于37 ℃振荡培养;自加入噬菌体开始计时,分别在5、10、20、25、30、40、50、60、90和120 min时间点取样,测定样品滴度;以感染时间为横坐标,以各时间点滴度为纵坐标绘制IME196的一步生长曲线。
1.7 噬菌体裂解谱测定测定噬菌体对受试菌是否有裂解作用是通过点板试验来测试,将受试菌(10株粪肠球菌、10株屎肠球菌、4株金黄色葡萄球菌和2株大肠杆菌)培养至OD600为0.6左右,取出500 μL铺双层平板,室温静置5 min,将1.5 μL噬菌体原液滴于双层平板上,室温静置20 min,待噬菌体原液被吸收后倒置于37 ℃温箱中10 h后观察有无噬菌斑产生。
1.8 提取噬菌体IME196基因组取600 μL已纯化的浓缩噬菌体液体,参照张文惠等[4]蛋白酶K/SDS法提取核酸,有改动。
加入DNaseⅠ和RNase A至终浓度1 mg/L,37 ℃过夜处理,80 ℃灭活15 min,使上述酶失活。然后加入0.5 mol/L EDTA (终浓度0.02 mol/L),20 g/L蛋白酶K (终浓度50 mg/L),10% SDS (终浓度0.5%),56 ℃水浴1 h。加入等体积酚抽提核酸,10 000 r/min离心5 min,转移上层水相到新的1.5 mL离心管中。向上述离心管中加入等体积的酚-氯仿-异戊醇(25:24:1,体积比),温和混匀后 10 000 r/min离心10 min,以去除蛋白、糖类等物质污染。取上述离心管中水相到一个新的离心管中,并加入等体积异戊醇,−20 ℃放置4 h后 12 000 r/min离心15 min,收集沉淀。用75%的冰乙醇洗涤上述DNA沉淀,12 000 r/min离心 20 min,室温干燥后用去离子水重悬浮上述DNA沉淀。
1.9 噬菌体IME196的全基因组测序构建大约290 bp的测序文库利用Life technologies公司的Ion Torrent测序仪PGM完成噬菌体全基因组的测序。主要流程如下:基因组DNA进行超声打断,末端补平,加上特异接头,纯化筛选DNA即为构建的测序文库;将测序文库片段克隆至特定磁珠,通过emulsion PCR扩增测序文库。
1.10 噬菌体IME196全基因组序列分析噬菌体IME196全基因组序列利用软件Newbler 2.9进行序列拼接,全基因组使用 RAST (http://rast.nmpdr.org/)和BASys (Bacterial Annotation System)在线基因功能注释,序列相似性比对分析使用BLAST在线工具(http://blast.ncbi.nlm.nih.gov/)进一步通过NCBI Microbial Genome Submission Check验证注释序列是否有问题,将无误基因组注释序列向NCBI提交,获取序列号。利用CGView软件构建(http://wishart.biology.ualberta.ca/cgview/)噬菌体全基因组图谱。
通过Ion Torrent高通量测序产生58 M数据,平均读长为290 bp,拼接出全长为38 895 bp能够环化的噬菌体基因组。
2 结果与分析 2.1 噬菌体IME196形态及大小粪肠球菌2007分离于临床女性阴道分泌物,抗生素耐药性见表 1。所铺双层平板培养5 h后结果如图 1A所示,噬菌体边缘清晰,噬菌斑透亮,稀释10−8的平板上长出36个噬菌斑,纯化后的噬菌体颗粒经负染后在透射电子显微镜下(图 1B)观察,结果显示该噬菌体呈蝌蚪状,头部直径约为58 nm,尾部长约230 nm,该噬菌体被命名为vB_E. faecalis_IME196,属长尾噬菌体科(Siphoviridae)。
| 抗菌药物 Antimicrobial drugs | MIC (mg/L) | 敏感度 Sensitivity | 抗菌药物 Antimicrobial drugs | MIC (mg/L) | 敏感度 Sensitivity |
| 复方新诺明 Cotrimoxazole | ≤20 | S | 奎奴普丁 Quinupristin | ≤4 | R |
| 左氧氟沙星 Levofloxacin | 0.5 | S | 头孢曲松 Ceftriaxone | ≤1 | S |
| 哌拉西林 Piperacillin | ≤4 | S | 头孢唑啉 Cefazolin | ≥64 | R |
| 高链霉素 High streptomycin | 4 | R | 呋喃妥因 Nitrofurantoin | ≤16 | S |
| 头孢他啶 Ceftazidime | ≤1 | S | 头孢吡肟 Cefepime | ≤1 | S |
| 哌拉西林 Ampicillin | ≤4 | S | 庆大霉素 Gentamicin | ≤1 | S |
| 美罗培南 Meropenem | ≤0.25 | S | 四环素 tetracycline | ≥64 | R |
| 注:S:敏感;I:中介度;R:耐药. Note: S: Sensitivity; I: Betweeness; R: Drug resistance. | |||||
|
| 图 1 噬菌体IME196形成的噬菌斑(A)及透射电镜下噬菌体IME196的形态(B) (10 000×) Figure 1 The plaque formed by IME196 (A) and the morphology of phage IME196 (B) (TEM 10 000×) |
|
|
如表 2所示,按照比例加入噬菌体和宿主菌培养5 h,分别测各管的噬菌体滴度。从表 2中看出当MOI=0.01时噬菌体滴度最高,该结果表明噬菌体的最佳感染复数为0.01。
| 编号 Number | 细菌浓度 Bacteria (CFU/mL) | 噬菌体滴度 Phage (PFU/mL) | 感染复数 MOI | 6 h滴度 6 h Titer (PFU/mL) | |
| 1 | 108 | 109 | 0.01 | 4.8x109 | |
| 2 | 108 | 108 | 0.1 | 1.0x109 | |
| 3 | 108 | 107 | 1 | 2.2x108 | |
| 4 | 108 | 106 | 10 | 3.1x108 | |
| 5 | 108 | 105 | 100 | 3.1x107 |
通过研究噬菌体的生长规律来测定噬菌体的裂解能力,由图 2可知,前30 min为噬菌体的潜伏期,暴发期为60 min,噬菌体的裂解量约为噬菌体滴度/细菌的浓度=1.5×109/3×107=50 PFU。

|
| 图 2 噬菌体IME196的一步生长曲线 Figure 2 one-step growth curve of phage IME196 |
|
|
噬菌体IME196只对3株粪肠球菌有裂解能力。IME196对其他粪肠球菌、屎肠球菌、金黄色葡萄球菌和大肠杆菌无裂解能力。
2.5 噬菌体IME196全基因组高通量测序和组装及高频序列分析约100 μg所提取的噬菌体基因组DNA样品用于构建测序文库,测序约产生686 350条平均长度为287 bp的高质量测序片段(Reads),选取约 14 657条平均长度约268 bp的Reads用于基因组序列组装,参与组装碱基约892 810个,平均覆盖倍数为23 (892 810÷38 895≈23)倍。噬菌体IME196基因组组装结果产生长约39 kb的单一重叠群(contig),即测序数据分析可知该噬菌体基因组是环状的。环化后完整基因组长度为38 895 bp。
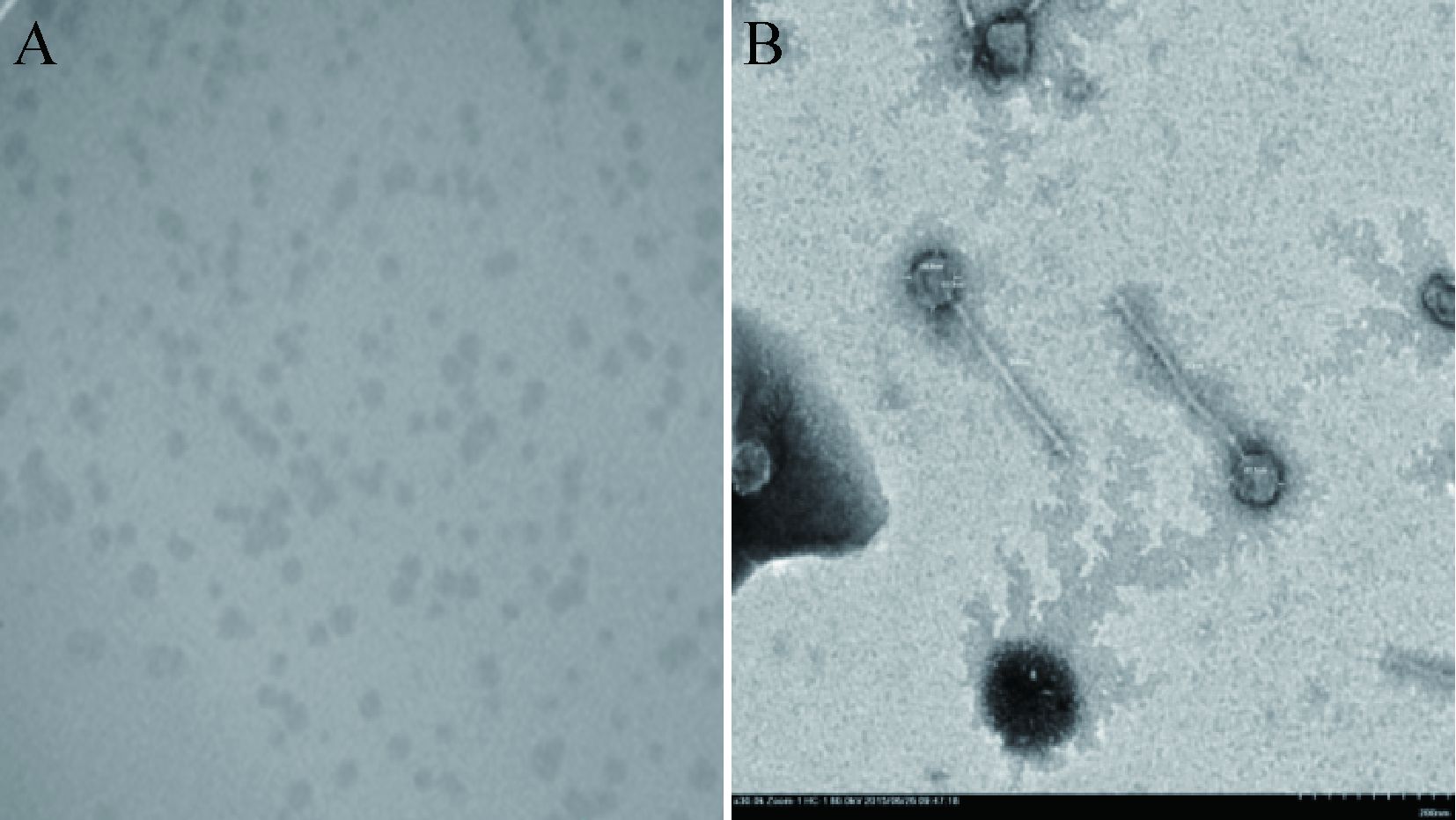
噬菌体末端序列通常在其基因组复制、包装等方面有重要作用。序列分析发现,噬菌体高通量测序中高频出现的序列就是噬菌体末端。根据文献[9]建立的病毒基因组末端结构分析方法,通过把所有原始高通量测序的Reads映射到(Mapping)组装好的基因组上,统计分析所有Reads的出现次数,并根据出现次数从高到低排序。大部分高通量测序的Reads重复次数小于200,出现两个频率特别高的Reads。如图 3所示,只有两个序列的频率极高,分别是序列1 (5′-ATTGTGTTGAGTAAG AATTGGAGGGGGGGCAAAAATTTTT-3′)和序列2 (5′-CTTTCGCTTAAACGAATTTCTAAAAAC GAAAGTTGAAAAC-3′)。所以IME196有固定的基因组末端序列,复制时的开环及末端切割是同一位点(图 4)。
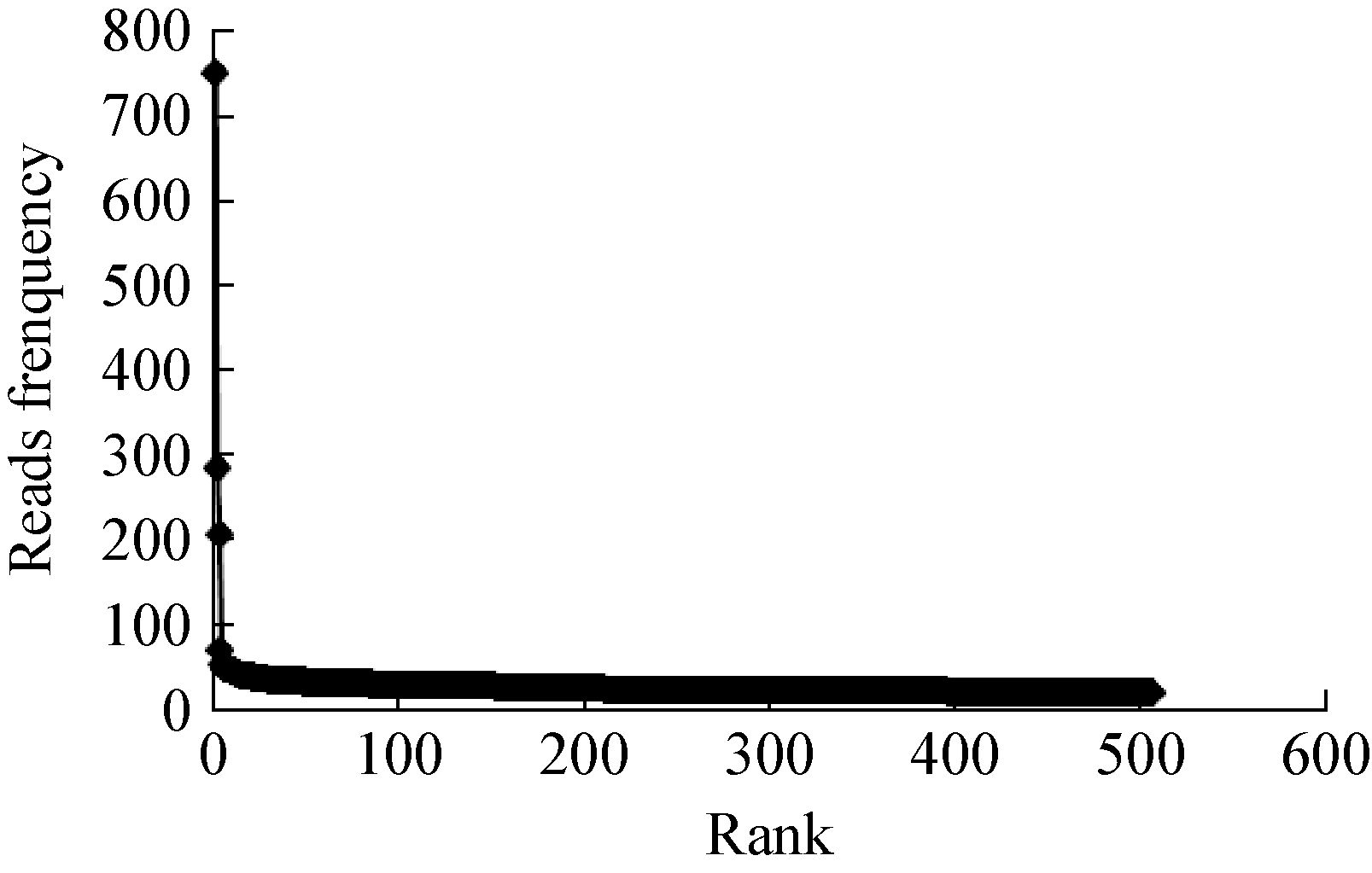
|
| 图 3 噬菌体IME196末端的Reads发生频率 Figure 3 Frequencies of sequence reads representing the genomic termini of IME196 注:X轴表示Reads重复次数取值范围;Y轴表示Reads条数的统计. Note: X axis is the span of Reads frequency; Y axis is the statistic of the number of Reads. |
|
|

|
| 图 4 噬菌体IME196的末端分析 Figure 4 the genomic termini of phage IME196 |
|
|
噬菌体IME196基因组全长38 895 bp,该基因组是一条环状双链DNA分子,G+C含量为33.9%。噬菌体vB_E. faecalis_IME196全基因组核酸序列已经提交GenBank,登录号为KT932701。BLASTn比对表明IME196与粪肠球菌噬菌体EfaCPT1 (40 kb)的相似性最高,其次是粪肠球菌噬菌体IME-EF4 (40 kb),比对覆盖率为92%。核酸序列GenBank登录号分别为JX193904和KF733017。通过全基因组RAST在线初步注释和NCBI的blastp分析,结果显示,该基因组共有57个开放阅读框(ORFs),其中11个ORFs为假想蛋白(hypothetical protein)序列,46个ORFs的功能已知,如表 3所示。ORF1 (噬菌体末端酶小亚基)和ORF2 (噬菌体末端酶大亚基)在噬菌体包装时起重要作用。ORF20 (噬菌体孔蛋白)编码的蛋白质能使细菌细胞膜形成空洞,这有利于噬菌体基因组核酸注入到受体细菌,同时该蛋白还具有连接噬菌体头部与尾部的功能。通常上述3个ORF在基因组上临近。结构蛋白ORF6 (主要衣壳蛋白)是噬菌体主要衣壳蛋白,与包装蛋白邻近。ORF7−ORF16编码噬菌体尾部相关蛋白,这个基因簇在噬菌体感染、吸附宿主菌时发挥重要作用。
| ORF | Top BLAST hita | IDENTITYb (%) (aa) | E value | Accession No. |
| 1 | terminase small subunit [Enterococcus phage EFAP-1] | 98 (154/157) | 2.00E−108 | YP_002727893.1 |
| 2 | terminase large subunit [Enterococcus phage IME-EF4] | 98 (562/572) | 0 | YP_009004370.1 |
| 3 | hypothetical protein [Enterococcus phage IME_EF3] | 93 (50/54) | 2.00E−23 | YP_009008897.1 |
| 4 | putative portal protein [Enterococcus phage EfaCPT1] | 98 (377/383) | 0 | YP_009103820.1 |
| 5 | head maturation protease [Enterococcus phage IME_EF3] | 99 (186/187) | 3.00E−131 | YP_009008895.1 |
| 6 | HK97 family major capsid protein [Enterococcus phage AUEF3] | 89 (370/418) | 0 | AHN83328.1 |
| 7 | major tail protein [Enterococcus phage IME-EF4] | 100 (66/66) | 2.00E−37 | YP_009004365.1 |
| 8 | head-tail joining protein [Enterococcus phage EFRM31] | 99 (97/98) | 2.00E−63 | YP_004306647.1 |
| 9 | head-tail adaptor protein [Enterococcus phage EFRM31] | 48 (282/585) | 1.00E−73 | YP_004306648.1 |
| 10 | head-tail joining protein [Enterococcus phage EFRM31] | 98 (132/135) | 4.00E−93 | YP_004306649.1 |
| 11 | head-tail joining protein [Enterococcus phage EFRM31] | 98 (118/121) | 9.00E−83 | YP_004306650.1 |
| 12 | major tail protein [Enterococcus phage EFRM31]] | 99 (187/188) | 2.00E−129 | YP_004306651.1 |
| 13 | hypothetical protein EFAP1_gp09 [Enterococcus phage EFAP-1] | 99 (102/103) | 3.00E−66 | YP_002727881.1 |
| 14 | Putative tail tape measure chaperone frameshift protein [Enterococcus phage efacpt1] | 90 (35/39) | 4.00E−16 | YP_009103829.1 |
| 15 | Putative tail tape measure protein [Enterococcus phage efacpt1] | 86 (1 253/1 456) | 0 | YP_009103831.1 |
| 16 | Tail family protein [Enterococcus phage AUEF3] | 94 (653/693) | 0 | AHN83270.1 |
| 17 | Minor structural N-terminal region domain protein [Enterococcus phage AUEF3] | 92 (619/676) | 0 | AHN83271.1 |
| 18 | Hypothetical protein efacpt1_gp17 [Enterococcus phage efacpt1] | 93 (88/95) | 6.00E−59 | YP_009103834.1 |
| 19 | Hypothetical protein IME-EF4_44 [Enterococcus phage IME-EF4] | 94 (76/81) | 5.00E−37 | YP_009004355.1 |
| 20 | Holin class II [Enterococcus phage IME_EF3] | 99 (77/78) | 9.00E−47 | YP_009008947.1 |
| 21 | Lysin,N-acetylmuramoyl-L-alanine amidase [Enterococcus phage IME_EF3] | 98 (320/328) | 0 | YP_009008946.1 |
| 22 | Putative glutaredoxin [Enterococcus phage efacpt1] | 87 (65/75) | 4.00E−39 | YP_009103838.1 |
| 23 | HNH endonuclease family protein [Enterococcus phage EFRM31] | 50 (75/150) | 1.00E−31 | YP_004306639.1 |
| 24 | DNA polymerase [Enterococcus phage IME-EF4] | 96 (735/763) | 0 | YP_009004351.1 |
| 25 | Hypothetical protein CO47_0108 [Enterococcus phage AUEF3] | 99 (219/220) | 9.00E−156 | AHN83279.1 |
| 26 | Hypothetical protein IME-EF4_36 [Enterococcus phage IME-EF4] | 54 (45/83) | 2.00E−20 | YP_009004347.1 |
| 27 | Hypothetical protein [Enterococcus phage IME_EF3] | 83 (38/46) | 2.00E−17 | YP_009008938.1 |
| 28 | Hypothetical protein CO47_0111 [Enterococcus phage AUEF3] | 98 (259/265) | 0 | AHN83282.1 |
| 29 | Hypothetical protein IME-EF4_34 [Enterococcus phage IME-EF4] | 100 (62/62) | 9.00E−36 | YP_009004345.1 |
| 30 | Beta-lactamase superfamily domain protein [Enterococcus phage AUEF3] | 97 (250/258) | 0 | AHN83284.1 |
| 31 | Putative HNH homing endonuclease-like protein [Enterococcus phage IME_EF3] | 98 (156/159) | 1.00E−110 | YP_009008933.1 |
| 32 | Hypothetical protein [Enterococcus phage IME_EF3] | 99 (71/72) | 6.00E−180 | YP_009008932.1 |
| 33 | Hypothetical protein IME-EF4_30 [Enterococcus phage IME-EF4] | 98 (165/168) | 3.00E−118 | YP_009004341.1 |
| 34 | Hypothetical protein IME-EF4_29 [Enterococcus phage IME-EF4] | 94 (64/68) | 1.00E−140 | YP_009004340.1 |
| 35 | Putative prim-pol domain protein [Enterococcus phage efacpt1] | 81 (235/290) | 9.00E−164 | YP_009103851.1 |
| 36 | Hypothetical protein IME-EF4_26 [Enterococcus phage IME-EF4] | 98 (61/62) | 8.00E−35 | YP_009004337.1 |
| 37 | Hypothetical protein IME-EF4_25 [Enterococcus phage IME-EF4] | 100 (65/65) | 4.00E−39 | YP_009004336.1 |
| 38 | DNA helicase [Enterococcus phage IME_EF3] | 88 (383/434) | 0 | YP_009008926.1 |
| 39 | HNH homing endonuclease [Enterococcus phage IME_EF3] | 67 (108/162) | 5.00E−67 | YP_009008925.1 |
| 40 | Hypothetical protein [Enterococcus phage IME_EF3] | 91 (112/123) | 2.00E−60 | YP_009008924.1 |
| 41 | Hypothetical protein [Enterococcus phage IME_EF3] | 83 (54/65) | 1.00E−28 | YP_009008923.1 |
| 42 | Hypothetical protein IME-EF4_21 [Enterococcus phage IME-EF4] | 98 (50/51) | 3.00E−24 | YP_009004332.1 |
| 43 | Hypothetical protein CO47_0128 [Enterococcus phage AUEF3] | 88 (79/90) | 1.00E−47 | AHN83299.1 |
| 44 | Hypothetical protein [Enterococcus phage IME_EF3] | 90 (56/62) | 6.00E−31 | YP_009008919.1 |
| 45 | Putative primase [Enterococcus phage efacpt1] | 99 (522/526) | 0 | YP_009103865.1 |
| 46 | Hypothetical protein [Enterococcus phage IME_EF3] | 85 (55/65) | 2.00E−30 | YP_009008915.1 |
| 47 | Hypothetical protein [Enterococcus phage IME_EF3] | 92 (68/74) | 6.00E−40 | YP_009008910.1 |
| 48 | Hypothetical protein EFLK1_ORF093 [Enterococcus phage EFLK1] | 54 (25/46) | 2.00E−10 | AKC05052.1 |
| 49 | No hits | |||
| 50 | No hits | |||
| 51 | No hits | |||
| 52 | Hypothetical protein efacpt1_gp55 [Enterococcus phage efacpt1] | 56 (77/131) | 3.00E−33 | YP_009103872.1 |
| 53 | APC amino acid-polyamine-organocation transporter [Enterococcus phage EFRM31] | 96 (55/57) | 3.00E−31 | YP_004306633.1 |
| 54 | No hits | |||
| 55 | Daunorubicin resistance transmembrane protein [Enterococcus phage EFRM31] | 94 (101/108) | 2.00E−68 | YP_004306631.1 |
| 56 | No hits | |||
| 57 | No hits | |||
| 注:a:最相近基因组的微生物物种的基因和名称. No hits:没有比对到相近的氨基酸序列. b:BLASTp搜索到比对百分比. Note: a: The most closely related genes and names of the organisms. No hits: indicates no significant homology detected for a particular amino acid sequence. b: Percent identity for top hits in BLASTp searches. | ||||
噬菌体DNA复制及调控相关基因主要包括:ORF24 (DNA聚合酶)、ORF45 (DNA引物酶)、ORF187 (DNA拓扑异构酶)、ORF251 (单链DNA结合蛋白)、ORF38 (DNA解旋酶)和ORF184 (转录调控因子)等。
ORF21 (噬菌体裂解酶)和ORF20 (穿孔蛋白)是噬菌体裂解相关基因。ORF20编码穿孔蛋白,使细菌细胞膜穿孔。该噬菌体可能是通过同时表达上述两种蛋白来介导宿主细胞裂解的。
使用在线的CGview制作噬菌体全基因组模式图,如图 5所示:共4圈,由外到内依次为位于最外圈蓝色代表IME196正链所编码的CDS,其次是里圈蓝色代表负链所编码的CDS;黑色代表G+C含量,向外表示大于全基因组平均G+C含量,向内表示低于平均G+C含量;最里面的绿色和紫色圈分别代表G+C偏嗜性G-C/G+C,绿色代表 大于0,紫色代表小于0。

|
| 图 5 IME196全基因组图谱 Figure 5 Genome map of IME196 |
|
|
噬菌体是细菌的天然杀手,有严格的宿主特异性和副作用较小等特点,这使得噬菌体个体化治疗成为具有潜力的研究方向之一[10],这种新的治疗方案未来将极有可能成为治疗人类细菌感染的有效武器。面对日益严峻的细菌耐药性问题,噬菌体疗法备受人们的关注。例如,为了解决抗生素耐药问题,美国在20世纪90年代兴起一些噬菌体研究公司[9]。由于噬菌体具有严格的宿主特异性,以及细菌与噬菌体相互作用进化过程中会对噬菌体产生耐受,单一噬菌体制剂将不能满足治疗要求。所以,噬菌体混合制剂鸡尾酒疗法及个性化噬菌体治疗方法可能是未来噬菌体治疗的研究方向之一。但并不是所有的噬菌体都具有用于治疗的价值,有些噬菌体可能携带耐药基因甚至毒力基因,可能会使患者陷入更大的感染威胁。因此,需要对分离到的噬菌体进行全基因组测序,这样不仅能够知道该噬菌体的基本生物学特性及全基因组结构,更为噬菌体的实际应用提供保障。
综上所述,本实验从307医院污水处理站的污水中分离出一株噬菌体,命名为vB_E. faecalis_IME196,通过透射电镜观察,按照噬菌体分类学中的分类标准,判断该噬菌体属于长尾噬菌体科(Siphoviridae),进一步进行生物学特性研究发现该噬菌体最佳感染复数为0.01,一步生长曲线显示IME196的潜伏期为30 min,暴发期为 60 min,暴发量为50 PFU,裂解量大,加上噬菌体IME196可裂解多株粪肠球菌,不能裂解屎肠球菌和金黄色葡萄球菌,既具有一定的广谱性,又具有宿主专一性,所以适合用于噬菌体治疗的进一步研究。
此外,本文利用Ion Torrent PGM高通量测序仪进行全基因组测序,生物信息学分析得到全长基因组序列;通过查找其末端高频序列,证明了该噬菌体基因组的完整性和滚环复制时末端切割点同一性;通过预测该噬菌体ORF功能,为以后研究噬菌体的基因功能奠定基础,也为未来使用该噬菌体治疗粪肠球菌异位感染提供了安全保障。噬菌体IME196裂解酶实验与动物实验值得深入研究。
| [1] | Bonilla N, Santiago T, Marcos P, et al. Enterophages, a group of phages infecting Enterococcus faecalis, and their potential as alternate indicators of human faecal contamination[J]. Water Science & Technology , 2010, 61 (2) : 293–300. |
| [2] | Dejea C, Wick E, Sears CL. Bacterial oncogenesis in the colon[J]. Future Microbiology , 2013, 8 (4) : 445–460. DOI:10.2217/fmb.13.17 |
| [3] | Lavilla-Lerma L, Pérez-Pulido R, Martínez-Bueno M, et al. Characterization of functional, safety, and gut survival related characteristics of Lactobacillus strains isolated from farmhouse goat's milk cheeses[J]. International Journal of Food Microbiology , 2013, 163 (2/3) : 136–145. |
| [4] | Zhang WH, An XP, Fan H, et al. Isolation and characterization of an Enterococcus faecalis phage[J]. Letters in Biotechnology , 2013, 24 (4) : 484–487. (in chinese) 张文惠, 安小平, 范航, 等. 一株粪肠球菌噬菌体的分离及其生物学特性研究[J]. 生物技术通讯 , 2013, 24 (4) : 484–487. |
| [5] | Takakusagi Y, Kobayashi S, Sugawara F, et al. Camptothecin binds to a synthetic peptide identified by a T7 phage display screen[J]. Bioorganic & Medicinal Chemistry Letters , 2005, 15 (21) : 4850–4853. |
| [6] | Funatsu T, Taniyama T, Tajima T, et al. Rapid and sensitive detection method of a bacterium by using a GFP reporter phage[J]. Microbiology and Immunology , 2002, 46 (6) : 365–369. DOI:10.1111/mim.2002.46.issue-6 |
| [7] | Wang S, Tong YG. Recent advance in bacteriophage therapy[J]. Microbiology China , 2009, 36 (7) : 1019–1024. (in chinese) 王盛, 童贻刚. 噬菌体治疗研究进展[J]. 微生物学通报 , 2009, 36 (7) : 1019–1024. |
| [8] | Li SS, Fan H, An XP, et al. Utility of high throughput sequencing technology in analyzing the terminal sequence of caudovirales bacteriophage genome[J]. Chinese Journal of Virology , 2013, 29 (1) : 39–42. (in chinese) 李莎莎, 范航, 安小平, 等. 采用高通量测序技术分析尾病毒目噬菌体基因组末端序列特点[J]. 病毒学报 , 2013, 29 (1) : 39–42. |
| [9] | Abedon ST, Kuhl SJ, Blasdel BG, et al. Phage treatment of human infections[J]. Bacteriophage , 2011, 1 (2) : 66–85. DOI:10.4161/bact.1.2.15845 |
| [10] | Li XY, Ding P, Han CY. Isolation of a novel Enterococcus faecalis phage IME-EF3 and its potential therapeutic advantages[J]. Biomedical Engineering and Clinical Medicine , 2014, 18 (2) : 113–118. (in chinese) 李晓玉, 丁鹏, 韩传银, 等. 粪肠球菌噬菌体IME-EF3的分离及其治疗优势的发掘[J]. 生物医学工程与临床 , 2014, 18 (2) : 113–118. |
 2016, Vol. 43
2016, Vol. 43