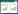扩展功能
文章信息
- 刘鹏娟, 乔小改, 李萍, 黄剑波, 卓秀萍, 方和俊, 邓益超, 徐志文, 朱玲
- LIU Peng-juan, QIAO Xiao-gai, LI Ping, HUANG Jian-bo, ZHUO Xiu-ping, FANG He-jun, DENG Yi-chao, XU Zhi-wen, ZHU Ling
- 猪伪狂犬病毒Fa株侵染对PK-15细microRNAs表达谱的影响
- Differential expression of microRNAs in PK-15 cells infected with Pseudorabies virus-Fa strain
- 微生物学通报, 2016, 43(9): 2010-2018
- Microbiology China, 2016, 43(9): 2010-2018
- DOI: 10.13344/j.microbiol.china.150829
-
文章历史
- 收稿日期: 2015-10-27
- 接受日期: 2016-01-14
- 优先数字出版日期(www.cnki.net): 2016-01-22
2. 动物疫病与人类健康四川省重点实验室 四川成都 611130
2. Key Laboratory of Animal Diseases and Human Health of Sichuan Province,Chengdu,Sichuan 611130,China
伪狂犬病是由α-疱疹病毒甲亚科的伪狂犬病毒(Pseudorabies virus,PRV)引起,可导致母猪流产或产弱胎、死胎、木乃伊胎,导致公猪睾丸炎等,是危害我国养猪业的重大疾病之一。自1813年报道发生以来,目前伪狂犬病已呈世界性分布。我国虽长期采取广泛的免疫、净化措施,但该病仍呈局部流行,种猪淘汰率较高[1]。2011年底,由于抗原变异等原因,一些免疫后猪场暴发严重的伪狂犬病并迅速蔓延到全国大部分猪场,给养殖业带来巨大损失[2]。
microRNA (miRNA)是长度约为22 nt的生物内源性非编码单链小RNA,广泛存在于动植物及病毒等多种有机体中[3],由具有茎环结构的长约70个碱基的单链前体RNA剪切而成。成熟的miRNA小分子通常与细胞内效应蛋白Argonaute等结合组成RNA诱导的沉默复合体(RISC),之后与靶mRNAs 3′端非翻译区(Untranslated region,UTR)、5′UTR或翻译区的碱基特异性互补配对,从而抑制靶mRNA翻译(部分配对)或直接降解mRNA (完全配对),最终下调靶基因的表达水平,发挥靶基因的转录后表达调控功能[4]。在植物中,多数miRNAs与其靶mRNAs的3′UTR完全互补配对,导致切割与降解;在动物中则多数为不完全配对,从而抑制翻译[5-6]。通常一个miRNA可调控多个靶mRNAs,多个miRNAs也可调控同一个靶mRNA。研究显示,miRNA通过与靶mRNA的作用,参与调控细胞的增殖、分化、凋亡、代谢和肿瘤转移等生物学过程[7-9]。
大量研究表明,病毒与宿主都可利用miRNAs调节自身及对方转录物,从而实现免疫逃避和自我保护。病毒侵染可导致宿主miRNAs表达谱发生变化,为其生存创造有利条件,同时宿主也可利用miRNAs来实现免疫清除。目前关于miRNAs水平PRV与宿主的相互作用的研究较少,Huang等[10]通过miRNA芯片杂交筛选获得PRV感染PK-15细胞后差异表达的miR-21,并以小鼠肝癌细胞Hepal-6为模型,经双荧光素酶报告基因结合ELISA实验证明干扰素-γ诱导蛋白10 (IFN-γ inducible protein 10,IP10)是miR-21的靶基因;Anselmo 等[11]经高通量测序研究PRV感染猪树突状细胞后宿主与病毒miRNAs的表达情况,对差异表达miRNAs进行靶基因预测和Pathway通路分析,表明病毒和宿主miRNAs调控对PRV的潜伏感染均起重要作用;Mahjoub等[12]研究表明,当PRV病毒缺失位于潜伏期相关转录位点(Latency-associated-t ranscript locus,LAT)的9个miRNAs后,会影响宿主三叉神经节对病毒潜伏感染的反应。
本研究拟阐明PRV-Fa株侵染后PK-15细胞miRNAs表达谱并对其进行深入分析,为进一步揭示miRNAs在PRV感染过程中的作用及筛选抗PRV候选miRNAs奠定基础。
1 材料与方法 1.1 细胞和病毒猪肾传代细胞(PK-15),由四川农业大学动物医学院动物生物技术中心提供;伪狂犬病毒Fa株,购自中国兽医药品监察所。
1.2 主要试剂和仪器Trizol Reagent,Life Technologies公司;2xSYBR Green PCR master mix,Arraystar公司;miRNA mimics及其对照mir-NC,上海吉玛公司;Xfect™ MicroRNA Transfection Reagent Protocol-At-A-Glance,成都天泰生物技术有限公司。NanoDrop ND-1000分光光度计,Thermo Scientific公司;Illumina HiSeq 2000 instrument,Illumina公司;Agilent 2100Bioanalyzer,Agilent Technologies公司;ViiATM7实时荧光定量PCR仪,Applied Biosystems公司。
1.3 细胞样品RNA的提取及鉴定将长势良好的PK-15细胞传代至3个感染组细胞瓶和3个未感染组(对照组)细胞瓶中,置培养箱中培养(37 ℃,5% CO2)。待单细胞层长至约85%,感染组细胞接种PRV-Fa (MOI为1)并于接毒后24 h收毒,非感染组细胞不接毒且在相同时间点收取。依照细胞RNA抽提方法分别提取RNA[13],并将每组的3个重复样品混合。用NanoDrop ND-1000对提取RNA进行浓度和纯度鉴定,A260/A280比值范围为1.8-2.1时则为合格。
1.4 总RNA的高通量测序将合格RNA产品送上海康成生物工程有限公司,采用Illumina高通量测序技术进行深度测序。
1.5 生物信息学分析高通量测序原始序列存在许多没用信息,经以下分析获得有效、高质量的小RNA序列:(1) 去除低质量Reads;(2) 去除没有3′接头序列Reads;(3) 去除5′接头有污染的Reads;(4) 去除没有插入片段的Reads;(5) 去除包含poly A的Reads;(6) 去除长度≤15 nt和数量≤2的Reads。将剩余序列分别与Rfam数据库(http://rfam.xfam.org/)、Repbase数据库(http://www.girinst.org/repbase/)和NCBI数据库(http://www.ncbi.nlm.nih.gov/)比对,去除可能存在的mRNA、rRNA、tRNA、snRNA、snoRNA及重复序列。
1.6 差异miRNAs表达谱的构建将两组样品miRNAs表达量分别采用Transcripts per million (TMP)法标准化为同一量级(单一miRNA拷贝数×106/总miRNAs拷贝数)。为排除miRNA表达量为0的计算干扰,将每个miRNA的拷贝数加10再计算,最终以Fold Change (感染/非感染)≥2或≤0.5为标准,建立miRNAs差异表达谱。
1.7 miRNAs荧光定量验证为验证高通量测序结果的准确性,随机选取10个差异表达的miRNAs (5个上调和5个下调)。利用Primer 5.0软件设计miRNAs特异性反转录引物及荧光定量PCR引物(表 1),并送英骏生物技术有限公司合成。
| 引物 Primers | 序列 Sequence (5′→3′) |
| ssc-miR-10a-5p-forward | TACCCTGTAGATCCGAATTTGT |
| ssc-miR-16-forward | TAGCAGCACGTAAATATTGGCG |
| ssc-miR-221-3p-forward | AGCTACATTGTCTGCTGGGTTT |
| ssc-miR-30d-forward | TGTAAACATCCCCGACTGGAAGCT |
| ssc-miR-378-forward | ACTGGACTTGGAGTCAGAAGGC |
| ssc-miR-146a-5p-forward | TGAGAACTGAATTCCATGGGTT |
| ssc-miR-152-forward | TGGAGAGAAAGGCAGTTCCTGA |
| ssc-miR-21-forward | TAGCTTATCAGACTGATGTTGA |
| ssc-miR-29a-forward | CTAGCACCATCTGAAATCGGTTA |
| ssc-miR-novel-chr13_ 10861-forward | TTCAAGTAACCCAGGATAGGCT |
| miR-reverse | GTCGGTGTCGTGGAGTCG |
| U6-RT Primer | CGCTTCACGAATTTGCGTGTCAT |
| U6-forward | TCGCTTTGGCAGCACCTAT |
| U6-reverse | AATATGGAACGCTTCGCAAA |
| miR-RT Primer | GTCGGTGTCGTGGAGTCGTTTGCAATT GCACTGGATTTTTTTTTTTTTTTTTTV |
| Note: V=A,G,C. | |
将高通量测序所用细胞样品依细胞RNA抽提方法提取RNA[13],并保证其纯度与质量。对RNA模板依次进行加Poly A尾和RT反应,获得cDNA保存于-20 ℃备用。
荧光定量反应体系:2×SYBR Green PCR master mix 5 μL,上游引物(10 μmol/L) 0.5 μL,下游引物(10 μmol/L) 0.5 μL,cDNA 2 μL,RNase Free H2O 2 μL。反应条件:95 ℃ 10 min;95 ℃ 15 s,60 ℃ 60 s,40个循环,且每个样品重复3次,每个循环后检测体系中荧光信号。U6基因作为内参,采用2-DDCt法计算各miRNA的相对表达量,每组数据中以对照组中miRNA的表达量为标准进行相对表达分析。Ct值为反应的实时荧光强度达到设定的阈值时所需要的扩增循环数,即荧光定量PCR分析仪的直接读数。
1.8 差异表达miRNAs靶基因预测整合TargetScan (http://www.targetscan.org/)和MiRanda (http://microorna.sanger.ac.uk/sequencs/) 两个数据库的算法,对差异表达的miRNAs进行靶基因预测分析,取这两个软件预测的交集作为差异表达miRNAs的靶基因。采用Gene ontology (GO)对靶基因的功能从细胞组成(Cellular component)、生物过程(Biological process)和分子功能(Molecular function) 3个方面进行分析。
2 结果与分析 2.1 细胞样品RNA的提取及鉴定提取感染和未感染PK-15细胞组总RNA,经NanoDrop ND-1000平台测定纯度及浓度,结果显示感染组A260/A280值为1.92,非感染组A260/A280值为1.97,均符合高通量测序所用样品要求。
2.2 总RNA的高通量测序PRV-Fa株感染和未感染PK-15细胞经高通量测序所得原始数据,经一系列去污染去低质量处理,分别获得干净序列(Clean reads) 586 608条和 3 987 148条,占各自测序总数的91.27%和85.42%。对所有clean reads进行长度分布统计,发现2个文库中miRNAs长度均主要分布在 21-24 nt,符合Dicer酶剪切后产物长度。
2.3 病毒感染前后PK-15细胞的miRNAs表达谱在PRV-Fa感染及未感染PK-15细胞样本中分别检测到384个和405个miRNAs。两组样本除了分别包含miRBase19.0数据库公布的已知猪源成熟miRNAs 218个和225个外,还分别鉴定出166个和180个新的成熟猪源miRNAs,但其大部分表达量极低。经比对分析,PRV-Fa感染相对未感染细胞差异表达的miRNAs共127个,其中60个表达水平上调(44个已知的,16个新鉴定的),67个表达下调(47个已知的,20个新鉴定的),表达下调比上调多7个(3个已知的,4个新鉴定的)。在表达上调的miRNAs中,上调5倍以上的有24个,上调10倍以上的有10个,其中ssc-miR-10b上调幅度最大(61倍)且表达水平也最高;在表达下调的miRNAs中,下调10倍以上的有29个,下调25倍以上的有13个,其中ssc-miR-424-5p下调幅度最大为100倍(表 2)。比对可知,PRV-Fa侵染PK-15细胞后,大部分差异miRNAs表现为表达下调。
| microRNAs | 长度 Length (nt) | 样品归一化表达量 Normalized read counts | Fold change | 类型 Type | 靶基因个数 Numbers of target genes | |
| 感染组 Infected | 未感染组 Control | |||||
| ssc-miR-10b | 22 | 248 415 | 4 009 | 61.81 | Up | 738 |
| ssc-miR-192 | 21 | 20 198 | 363 | 54.18 | Up | 718 |
| ssc-miR-146b | 21 | 1 124 | 12 | 51.55 | Up | 1 139 |
| ssc-miR-novel-chr2_20448 | 24 | 423 | 4 | 30.93 | Up | 2 280 |
| ssc-miR-155-5p | 22 | 7 223 | 340 | 20.67 | Up | 1 146 |
| ssc-miR-424-3p | 21 | 181 | 0 | 19.10 | Up | 597 |
| ssc-miR-novel-chrX_40705 | 22 | 3 151 | 235 | 12.90 | Up | 811 |
| ssc-miR-34a | 22 | 1 519 | 119 | 11.85 | Up | 1 661 |
| ssc-miR-novel-chr9_38959 | 22 | 668 | 52 | 10.94 | Up | 1 663 |
| ssc-miR-221-3p | 22 | 2 954 | 274 | 10.44 | Up | 811 |
| ssc-miR-92b-3p | 22 | 5 005 | 594 | 8.30 | Up | 1 757 |
| ssc-miR-126-3p | 22 | 249 | 22 | 8.09 | Up | 88 |
| ssc-miR-486 | 22 | 418 | 44 | 7.93 | Up | 726 |
| ssc-miR-30a-5p | 22 | 134 532 | 17 235 | 7.80 | Up | 1 147 |
| ssc-miR-7 | 24 | 12 266 | 1 594 | 7.65 | Up | 1 619 |
| ssc-miR-450c-5p | 22 | 197 | 18 | 7.39 | Up | 427 |
| ssc-miR-novel-chr6_31692 | 22 | 121 | 8 | 7.28 | Up | 2 019 |
| ssc-miR-17-5p | 23 | 6 309 | 929 | 6.73 | Up | 1 443 |
| ssc-miR-27b-3p | 21 | 8 316 | 1 390 | 5.95 | Up | 1 146 |
| ssc-miR-92a | 22 | 30 702 | 5 186 | 5.91 | Up | 691 |
| ssc-miR-16 | 22 | 32 269 | 5 779 | 5.58 | Up | 1 763 |
| ssc-miR-novel-chr10_5602 | 25 | 186 | 26 | 5.44 | Up | 1 597 |
| ssc-miR-27a | 21 | 7 081 | 1 314 | 5.36 | Up | 1 146 |
| ssc-miR-182 | 22 | 22 335 | 4 271 | 5.23 | Up | 1 446 |
| ssc-miR-424-5p | 22 | 29 | 2 998 | 0.01 | Down | 1 817 |
| ssc-miR-novel-chr9_39041 | 23 | 38 | 3 840 | 0.01 | Down | 1 512 |
| ssc-miR-novel-chr5_29857 | 19 | 26 | 2 081 | 0.02 | Down | 1 711 |
| ssc-miR-194a | 21 | 24 | 1 562 | 0.02 | Down | 842 |
| ssc-miR-542-3p | 22 | 155 | 7 368 | 0.02 | Down | 1 349 |
| ssc-miR-novel-chrX_41190 | 21 | 2 | 491 | 0.02 | Down | 335 |
| ssc-miR-novel-chr5_29676 | 23 | 0 | 392 | 0.02 | Down | 1 066 |
| ssc-miR-152 | 21 | 384 | 14 934 | 0.03 | Down | 1 161 |
| ssc-miR-542-5p | 21 | 2 | 371 | 0.03 | Down | 732 |
| ssc-miR-195 | 21 | 17 | 781 | 0.03 | Down | 1 786 |
| ssc-miR-135 | 23 | 8 | 516 | 0.03 | Down | 1 602 |
| ssc-miR-185 | 22 | 66 | 1 843 | 0.04 | Down | 2 285 |
| ssc-miR-499-5p | 21 | 26 | 760 | 0.05 | Down | 974 |
| ssc-miR-24-3p | 22 | 3 462 | 70 574 | 0.05 | Down | 1 988 |
| ssc-miR-361-5p | 22 | 70 | 1 465 | 0.05 | Down | 867 |
| ssc-miR-29a | 23 | 608 | 11 368 | 0.05 | Down | 1 079 |
| ssc-miR-29c | 22 | 28 | 689 | 0.05 | Down | 1 120 |
| ssc-miR-19a | 23 | 390 | 7 185 | 0.06 | Down | 1 436 |
| ssc-miR-19b | 23 | 2 781 | 48 991 | 0.06 | Down | 1 299 |
| ssc-miR-193a-5p | 22 | 4 | 227 | 0.06 | Down | 1 142 |
| ssc-miR-142-3p | 22 | 20 | 487 | 0.06 | Down | 887 |
| ssc-miR-21 | 22 | 33 829 | 563 808 | 0.06 | Down | 789 |
| ssc-miR-143-3p | 21 | 55 | 912 | 0.07 | Down | 1 069 |
| ssc-miR-145-5p | 24 | 0 | 129 | 0.07 | Down | 1 475 |
| ssc-miR-183 | 23 | 897 | 12 061 | 0.08 | Down | 1 087 |
| ssc-miR-novel-chr6_30729 | 22 | 292 | 3 650 | 0.08 | Down | 1 083 |
为验证高通量测序结果的准确性,随机选取10个差异表达的miRNAs进行相对荧光定量PCR分析,计算感染与非感染细胞组中各miRNA差异表达倍数。结果显示,感染组细胞ssc-miR-221-3p、ssc-miR-16、ssc-miR-30d、ssc-miR-10a-5p和ssc-miR-378的表达水平相对非感染组表现为上调,而ssc-miR-152、ssc-miR-29a、ssc-miR-146a-5p、ssc-miR-21和ssc-miR-novel-chr13_10861相对非感染组的表达水平下调,与高通量测序分析结果一致(图 1)。
|
| 图 1 RT-qPCR验证miRNAs表达水平 Figure 1 Validation of miRNAs expression by RT-qPCR |
|
|
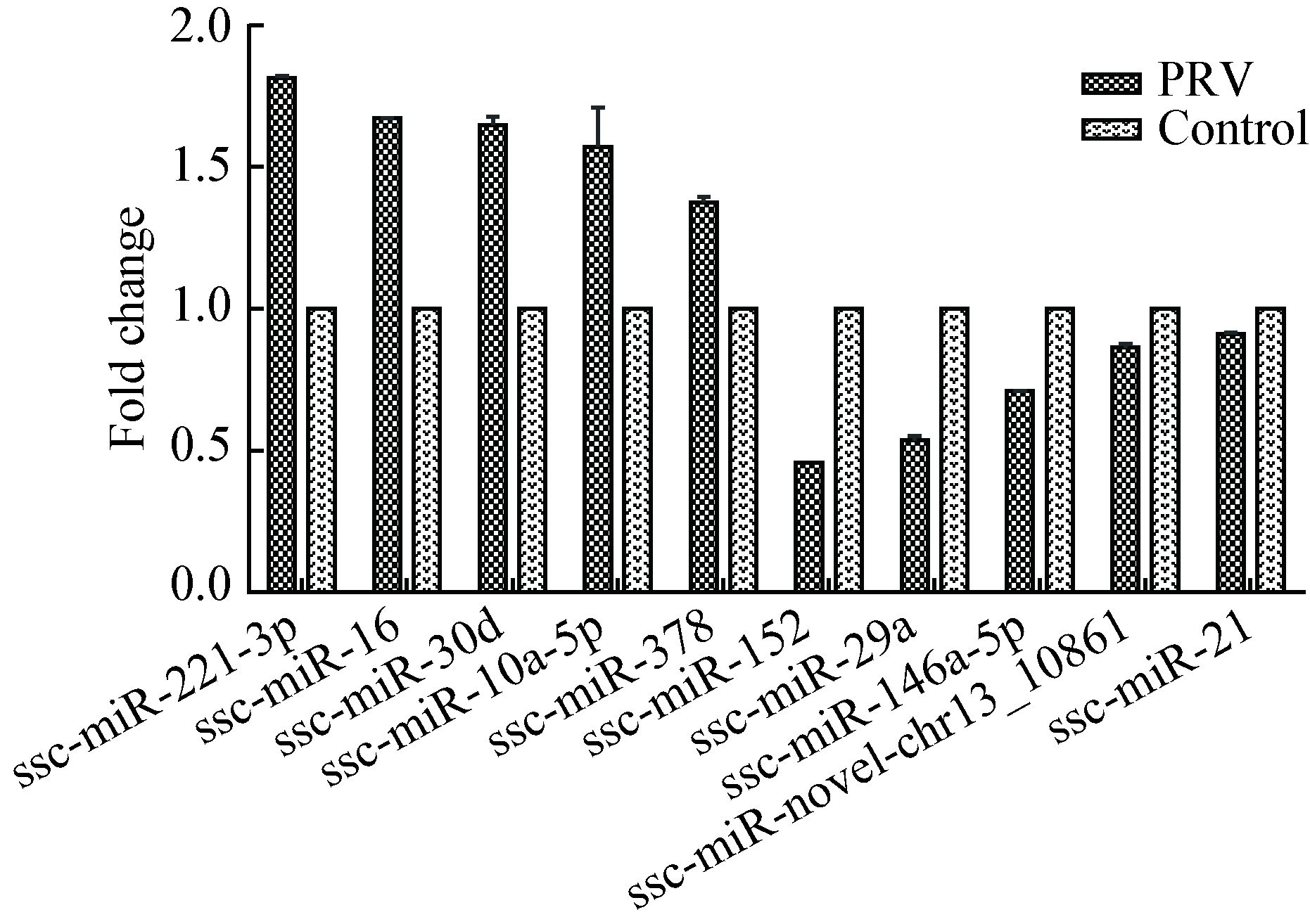
整合TargetScan和MiRanda两个数据库,对显著上调与下调的miRNAs进行靶基因预测,取其预测交集作为miRNAs的靶基因,统计获得各差异表达miRNA的靶基因个数(表 2)。选取9个差异表达miRNAs (上调的ssc-miR-10b、ssc-miR-16、ssc-miR-182、ssc-miR-30a-5p、ssc-miR-30d和下调的ssc-miR-21、ssc-miR-24-3p、ssc-miR-novel-chr6_30729、ssc-miR-novel-chr9_39041)和每个miRNA预测的所有靶基因中综合得分前3%的靶基因作为研究对象,按照四边形节点代表miRNA,圆形节点代表对应的靶基因,箭头代表靶向关系,箭头越多则靶向的节点越大的方式,构建了miRNA-Target调控网络(图 2)。
|
| 图 2 PRV-Fa感染的miRNA-Target转录调控网络 Figure 2 miRNA-Target network of PRV-Fa infected Note: A: ssc-miR-novel-chr6_30729; B: ssc-miR-21; C: ssc-miR-10b; D: ssc-miR-novel-chr9_39041; E: ssc-miR-30d; F: ssc-miR-30a-5p; G: ssc-miR-24-3p; H: ssc-miR-16; I: ssc-miR-182. |
|
|
此外,ssc-miR-30a-5p与ssc-miR-30d拥有较多共同调控的靶基因(表 3),且与其他miRNAs及靶基因形成联系密切的复杂网络,处于核心位置,影响调控网络的稳定性。进一步对ssc-miR-30a-5p与ssc-miR-30d共有靶基因进行GO分析可知,其部分富集在各GO功能上的主要靶基因可相互协同行使一定功能,其中包括在细胞内各种代谢过程中起调节作用的BM25、POLR3G、KLF10、CCNE2、GLCE、LRRK2、RUNX2、ZNF197、CHD1等,也包括细胞定位相关的JPH4等及免疫相关的ITGA4。在整个调控网络中发现有6个靶基因可同时被几个不同的miRNAs调控(表 4)。
| 典型miRNAs Typical miRNAs | 共有靶基因 Common target genes |
| ssc-miR-30a-5p 与ssc-miR-30d | SCN2A,ANKRA2,LHX8,KLHL20,GABRB1,ITGA4,RCC1,RBM25,POLR3G,GALNT7,DESI2,KLF10,CCNE2,IRAK1BP1,RAPGEF4,ZBTB11,FAM199X,GLCE,PDE7A,LRRK2,SOX9,SALL4,SRSF7,RUNX2,SERPINI1,CEP85L,VAT1L,UBE2J1,ZNF197,NSA2,NR4A2,FAM49A,JPH4,CHD1,OTUD6B |
| 典型miRNAs Typical miRNAs | 共有靶基因 Common target genes |
| CHD1 | ssc-miR-novel-chr9_39041,ssc-miR-30d,ssc-miR-30a-5p,ssc-miR-novel-chr6_30729 |
| FAM91A1 | ssc-miR-16,ssc-miR-30d,ssc-miR-30a-5p |
| LRRK2,RCC1 | ssc-miR-30d,ssc-miR-30a-5p,ssc-miR-24-3p |
| IRAK1BP,CEP85L | ssc-miR-30d,ssc-miR-30a-5p,ssc-miR-novel-chr9_39041 |
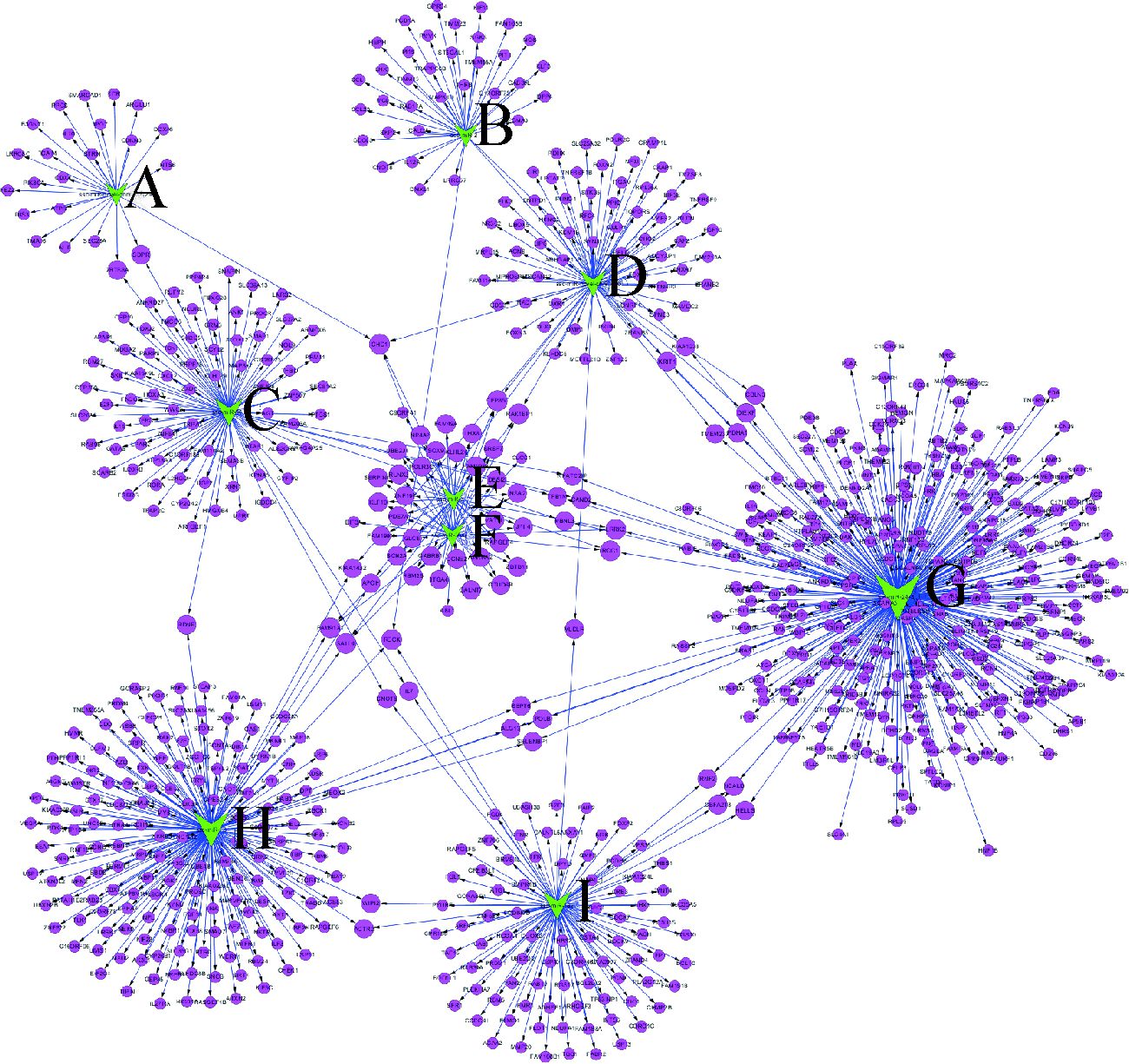
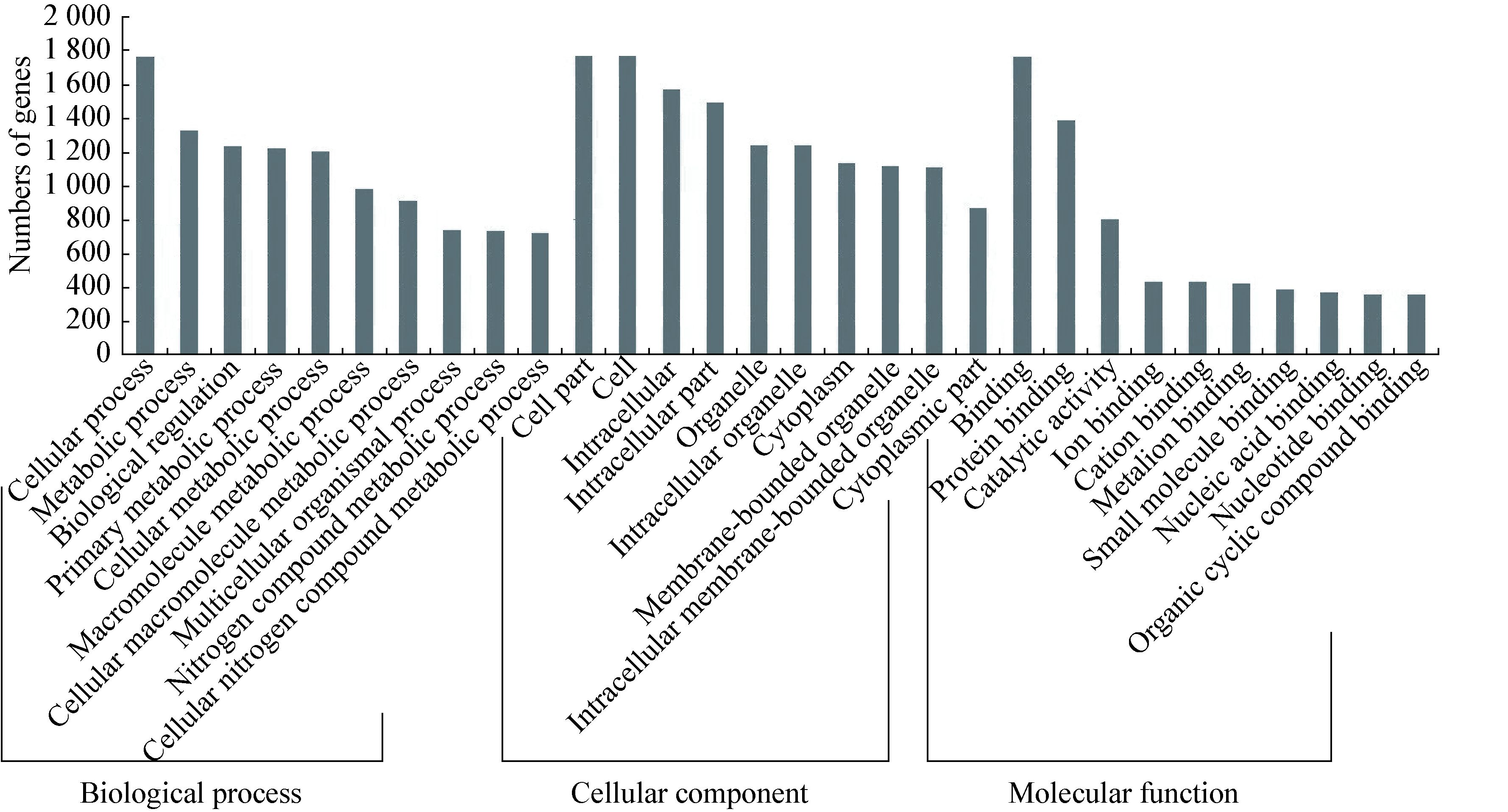
在PRV-Fa感染后PK-15细胞测序所得miRNAs中,检测到5个miRNAs基因序列可比对到PRV (NC-006151.1)基因组中具有典型miRNAs式的发卡结构上,它们均定位于大片段潜伏期转录物(Large latency transcript,LLT),按基因组顺序分别命名为PRV-miR-LLT1、PRV-miR-LLT2、PRV-miR-LLT3、PRV-miR-LLT4、PRV-miR-LLT5。这5个miRNAs表达水平普遍非常低,但也存在显著差异(表 5)。通过软件预测其靶基因,共有1 916个宿主基因被这5个miRNAs靶标,GO分析表明其参与细胞各种生命活动调节(图 3)。但严格预测只发现一个PRV基因(即早期蛋白EPO)被PRV-miR-LLT2与PRV-miR-LLT4靶向,其余3个均未预测到针对病毒自身的靶基因。
| 病毒 miRNAs | 序列 Sequence (5′→3′) | 样品的归一化表达量 Normalized read counts |
| PRV-miR-LLT1 | TCTCACCCCTGGGTCCGTCGC | 809 |
| PRV-miR-LLT2 | CTCATCCCGTCAGACCTGCGCC | 842 |
| PRV-miR-LLT3 | CGTACCGACCCGCCTACCAGGCA | 37 |
| PRV-miR-LLT4 | ATGAGTGGATGGATGGAGGCGA | 307 |
| PRV-miR-LLT5 | ACCCGCGGATGGCGAGGATG | 10 |
|
| 图 3 病毒编码miRNAs预测靶基因的GO注释 Figure 3 GO annotation on the target genes of miRNAs encoded by PRV-Fa |
|
|
PRV的潜伏感染是其难以根除的重要原因,对养猪业的健康发展危害严重。miRNAs在免疫系统功能调节中发挥重要作用,包括免疫细胞的发育、分化和功能,炎症介质的释放及调控细胞内的信号通路等。因此,鉴定PRV感染过程中与宿主免疫反应相关的候选miRNAs极为重要。
目前越来越多免疫相关miRNAs被发现,包括miR-10、miR-101、miR-106a、miR-126、miR-142-3p、miR-146、miR-150、miR-155、miR-181a、miR-196b、miR-21、miR-221、miR-223、miR-326、miR-34、miR-424、miR-9、miR-98、miR-17-92家族和let-7家族等[14-15]。本实验经高通量测序,在PRV-Fa感染及未感染PK-15细胞中均检测到大量猪源成熟的miRNAs,其中免疫相关的差异miRNAs有10个表达上调(ssc-miR-10b、ssc-miR-146b、ssc-miR-155-5p、ssc-miR-34a、ssc-miR-221-3p、ssc-miR-126-3p、ssc-miR-17-5p、ssc-miR-92a、ssc-miR-20a、ssc-let-7i),9个下调(ssc-miR-424-5p、ssc-miR-19a、ssc-miR-19b、ssc-miR-142-3p、ssc-miR-18a、ssc-miR-146a-5p、ssc-let-7d-5p、ssc-let-7f、sssc-miR-21),表明PRV-Fa可能影响这些miRNAs的正常表达,从而造成持续感染。
经对比分析,PRV-Fa感染PK-15细胞后有34% miRNAs表达有显著变化,表明病毒侵染过程中对细胞编码miRNAs产生了较大影响。在感染与未感染细胞中,ssc-miR-21的表达水平均非常高。研究报道miR-21在不同细胞类型及感染病毒状态下都有很高表达量,这可能与miR-21参与细胞周期中的细胞分化有关[11]。此外,miR-21参与前B淋巴细胞瘤的形成,既显著促进细胞增殖,又可抑制细胞凋亡[16]。PRV-Fa感染细胞后,ssc-miR-21的表达出现显著下调,说明病毒的感染可能通过复杂的调控网络引起细胞凋亡。
细胞miRNAs除了直接靶向病毒基因组,还可通过细胞因子引发免疫反应。例如,本实验差异表达最显著的miR-10b能靶向同源异形盒基因D10并抑制其转录表达,进而调控下游转移蛋白发挥其作用[17];过表达的miR-18a能促进T细胞Th1型细胞分化,Th1型细胞具有很强的IFN-γ分泌功能,有利于宿主抗肿瘤免疫应答[18];miR-16通过靶向细胞因子IL-8、IL-6等调节NF-κB活性[19];miR-145-5p通过靶向SOCS7调节IFN-β的表 达[20];miR-499-5p能靶向FOXO4和PDCD4从而加速细胞转移[21]。miR-146a可抑制有抗病毒复制作用的干扰素IFN-β的产生[22],从而达到抗细胞免疫作用。PRV-Fa株感染PK-15细胞后miR-146a表达下调,推测细胞可能通过干扰素对病毒复制产生抑制作用。
本研究筛选PRV-Fa感染组9个差异显著的miRNAs,并构建miRNA-Target调控网络图。结果显示,单个miRNA可同时调控多种靶基因,单个靶基因也可同时受多个miRNAs调控,miRNAs和靶基因都处于一个复杂的调控网络中。其中ssc-miR-30a-5p与ssc-miR-30d几乎共有所有靶基因,推测可能是由于它们同源性极高且种子序列完全相同。种子序列位于miRNA 5′端2-8个核苷酸,是靶标结合的最关键位点[23]。对ssc-miR-30a-5p 与ssc-miR-30共有靶基因进行GO功能分析,发现它们广泛参与细胞代谢、定位和免疫反应等。miR-30a-5p有抑制细胞增殖促进其凋亡的作用[24],而ssc-miR-30a-5p与ssc-miR-30d在PRV-Fa感染后均表现为显著上调,提示miR-30在病毒侵染过程中可能发挥负调控作用。此外,二者也各自拥有自己独特的靶基因,可与其他miRNAs协同作用于某些靶基因功能团,行使调控作用:如ssc-miR-30d、ssc-miR-30a-5p和ssc-miR-24-3p共同调控LRRK2 (蛋白激酶)、RCC1 (染色体浓缩调控蛋白1),GO分析显示它们主要参与细胞代谢。在miRNA-Target网络图中,调控靶基因最多的是ssc-miR-24-3p。miR-24在不同物种间是高度保守的,可通过靶基因促进细胞扩散和抑制细胞凋亡[25]。ssc-miR-24-3p在未感染病毒的PK-15细胞中表达水平非常高,位居所有miRNAs表达量前五,但在感染PRV-Fa后表达水平显著下降,提示ssc-miR-24-3p下调可能是由细胞调控,通过促进自身凋亡来抑制病毒增殖。
通过与PRV病毒基因组对比,本研究鉴定出 5个新的病毒编码的miRNAs均定位于LLT区。预测其针对猪的靶基因有1 916个,GO分析发现其参与多种复杂的细胞调控过程。而预测针对病毒自身的靶基因却只发现一个即早期蛋白基因EPO,且仅被PRV-miR-LLT2与PRV-miR-LLT4靶向。研究表明[26],EPO基因是伪狂犬病毒的早期基因,可激活IE180的转录,从而抑制LLT的神经特异性启动子LAPl,与病毒的潜伏感染相关,但其在PRV中的定位及功能尚不清楚。病毒miRNAs主要通过调控宿主细胞凋亡,细胞周期和免疫应答来促进自身的存活,但是否与病毒逃避免疫、促进潜伏感染有关,需进一步研究。
综上所述,本研究深入分析了猪伪狂犬病毒Fa株侵染对PK-15细胞microRNAs表达谱的影响,并检出大量免疫相关miRNAs,证实了其在PRV感染过程中的重要作用。有助于深入了解miRNAs在宿主-病原体互作关系中的作用,为筛选抗PRV候选miRNAs和从miRNAs角度提出防控伪狂犬病新策略提供参考。
| [1] | Verpoest S, Cay AB, de Regge N. Molecular characterization of Belgian pseudorabies virus isolates from domestic swine and wild boar[J]. Veterinary Microbiology , 2014, 172 (1/2) : 72–77. |
| [2] | Wu R, Bai CY, Sun JZ, et al. Emergence of virulent pseudorabies virus infection in Northern China[J]. Journal of Veterinary Science , 2013, 14 (3) : 363–365. DOI:10.4142/jvs.2013.14.3.363 |
| [3] | Lim LP, Glasner ME, Yekta S, et al. Vertebrate microRNA genes[J]. Science , 2003, 299 (5612) : 1540. DOI:10.1126/science.1080372 |
| [4] | Kim VN. MicroRNA biogenesis: coordinated cropping and dicing[J]. Nature Reviews Molecular Cell Biology , 2005, 6 (5) : 376–385. DOI:10.1038/nrm1644 |
| [5] | Jones-Rhoades MW, Bartel DP, Bartel B. MicroRNAs and their regulatory roles in plants[J]. Annual Review of Plant Biology , 2006, 57 : 19–53. DOI:10.1146/annurev.arplant.57.032905.105218 |
| [6] | Nilsen TW. Mechanisms of microRNA-mediated gene regulation in animal cells[J]. Trends in Genetics , 2007, 23 (5) : 243–249. DOI:10.1016/j.tig.2007.02.011 |
| [7] | Cheng AM, Byrom MW, Shelton J, et al. Antisense inhibition of human miRNAs and indications for an involvement of miRNA in cell growth and apoptosis[J]. Nucleic Acids Research , 2005, 33 (4) : 1290–1297. DOI:10.1093/nar/gki200 |
| [8] | Tavazoie SF, Alarcón C, Oskarsson T, et al. Endogenous human microRNAs that suppress breast cancer metastasis[J]. Nature , 2008, 451 (7175) : 147–152. DOI:10.1038/nature06487 |
| [9] | Ha TY. The role of microRNAs in regulatory T cells and in the immune response[J]. Immune Network , 2011, 11 (1) : 11–41. DOI:10.4110/in.2011.11.1.11 |
| [10] | Huang J, Ma G, Fu L, et al. Pseudorabies viral replication is inhibited by a novel target of miR-21[J]. Virology , 2014, 456 : 319–328. |
| [11] | Anselmo A, Flori L, Jaffrezic F, et al. Co-expression of host and viral microRNAs in porcine dendritic cells infected by the pseudorabies virus[J]. PLoS One , 2011, 6 (3) : e17374. DOI:10.1371/journal.pone.0017374 |
| [12] | Mahjoub N, Dhorne-Pollet S, Fuchs W, et al. A 2.5-kilobase deletion containing a cluster of nine microRNAs in the latency-associated-transcript locus of the pseudorabies virus affects the host response of porcine trigeminal ganglia during established latency[J]. Journal of virology , 2015, 89 (1) : 428–442. DOI:10.1128/JVI.02181-14 |
| [13] | Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual[M]. Beijing: Science Press, 2005: 98 -102. (in chinese) 萨姆布鲁克J, 拉塞尔DW. 分子克隆实验指南[M]. 北京: 科学出版社, 2005: 98 -102. |
| [14] | Ou JT, Meng QG, Li Y, et al. Identification and comparative analysis of the Eriocheir sinensis microRNA transcriptome response to Spiroplasma eriocheiris infection using a deep sequencing approach[J]. Fish & Shellfish Immunology , 2012, 32 (2) : 345–352. |
| [15] | Zhu QY, Liu Q, Chen JX, et al. MicroRNA-101 targets MAPK phosphatase-1 to regulate the activation of MAPKs in macrophages[J]. The Journal of Immunology , 2010, 185 (12) : 7435–7442. DOI:10.4049/jimmunol.1000798 |
| [16] | Medina PP, Nolde M, Slack FJ. OncomiR addiction in an in vivo model of microRNA-21-induced pre-B-cell lymphoma[J]. Nature , 2010, 467 (7311) : 86–90. DOI:10.1038/nature09284 |
| [17] | Liu Z, Zhu J, Cao H, et al. miR-10b promotes cell invasion through RhoC-AKT signaling pathway by targeting HOXD10 in gastric cancer[J]. International journal of oncology , 2012, 40 (5) : 1553–1560. |
| [18] | Trenkmann M, Brock M, Gay RE, et al. Tumor necrosis factor α-induced microrna-18a activates rheumatoid arthritis synovial fibroblasts through a feedback loop in NF-κB signaling[J]. Arthritis & Rheumatism , 2013, 65 (4) : 916–927. |
| [19] | Zhou R, Li XQ, Hu GK, et al. miR-16 targets transcriptional corepressor SMRT and modulates NF-kappaB-regulated transactivation of interleukin-8 gene[J]. PLoS One , 2012, 7 (1) : e30772. DOI:10.1371/journal.pone.0030772 |
| [20] | Noguchi S, Yamada N, Kumazaki M, et al. Socs7,a target gene of microRNA-145,regulates interferon-β induction through STAT3 nuclear translocation in bladder cancer cells[J]. Cell Death & Disease , 2013, 4 (2) : e482. |
| [21] | Liu XQ, Zhang ZY, Sun L, et al. microRNA-499-5p promotes cellular invasion and tumor metastasis in colorectal cancer by targeting FOXO4 and PDCD4[J]. Carcinogenesis , 2011, 32 (12) : 1798–1805. DOI:10.1093/carcin/bgr213 |
| [22] | Wu SY, He L, Li YY, et al. miR-146a facilitates replication of dengue virus by dampening interferon induction by targeting TRAF6[J]. Journal of Infection , 2013, 67 (4) : 329–341. DOI:10.1016/j.jinf.2013.05.003 |
| [23] | Ambros V, Bartel B, Bartel DP, et al. A uniform system for microRNA annotation[J]. RNA , 2003, 9 (3) : 277–279. DOI:10.1261/rna.2183803 |
| [24] | Dai H, Kang B, Zuo DY, et al. Effect of miR-30a-5p on the proliferation,apoptosis,invasion and migration of SMCC-7721 human hepatocellular carcinoma cells[J]. Chinese Journal of Hepatology , 2014, 22 (12) : 915–920. (in chinese) 代航, 康冰, 左的于, 等. MicroRNA-30a-5p对肝癌细胞株 SMMC-7721增殖凋亡及侵袭转移的影响[J]. 中华肝脏病杂志 , 2014, 22 (12) : 915–920. |
| [25] | Qin WM, Shi Y, Zhao BT, et al. miR-24 regulates apoptosis by targeting the open reading frame (ORF) region of FAF1 in cancer cells[J]. PLoS One , 2010, 5 (2) : e9429. DOI:10.1371/journal.pone.0009429 |
| [26] | Ou CJ, Wong ML, Huang C, et al. Suppression of promoter activity of the LAT gene by IE180 of pseudorabies virus[J]. Virus Genes , 2002, 25 (3) : 227–239. DOI:10.1023/A:1020959521745 |
 2016, Vol. 43
2016, Vol. 43