扩展功能
文章信息
- 夏军, 郑明刚, 王玲, 孙承君, 郑立, 祝建波
- XIA Jun, ZHENG Ming-Gang, WANG Ling, SUN Cheng-Jun, ZHENG Li, ZHU Jian-Bo
- 运用CRISPR/Cas系统敲除大肠杆菌磷酸烯醇式丙酮酸羧化酶基因及其对脂肪酸代谢的影响
- Knocking out phosphoenolpyruvate carboxylase gene by CRISPR/Cas and its influence on fatty acid metabolism in Escherichia coli
- 微生物学通报, 2016, 48(8): 1864-1871
- Microbiology China, 2016, 48(8): 1864-1871
- DOI: 10.13344/j.microbiol.china.150732
-
文章历史
- 收稿日期: 2015-09-28
- 接受日期: 2016-01-11
- 优先数字出版日期(www.cnki.net): 2016-01-11
2. 国家海洋局第一海洋研究所 海洋生态重点实验室 山东 青岛 266061 ;
3. 青岛大学化学化工与环境学院 山东 青岛 266071
2. The First Institute of Oceanography, State Oceanic Administration of China, Qingdao, Shandong 266061, China ;
3. College of Chemical Engineering, Qingdao University, Qingdao, Shandong 266071, China
CRISPR (Clustered regularly interspaced short palindromic repeats)/Cas系统是近年来新兴的一个基因组定点编辑技术[1-2]。该技术有三大类型(Ⅰ、Ⅱ、Ⅲ)[3],而Ⅱ型CRISPR/Cas系统因仅需要1个Cas 9蛋白作为切割DNA双链的工具而获得研究者们的青睐。目前该技术已成功应用到大肠杆菌[4]、斑马鱼[5]、拟南芥[6]、酿酒酵母[7]等物种中。CRISPR/Cas系统发挥基因编辑的作用建立在以下几方面:(1)由CRISPR转录来的trancrRNA与crRNA形成一个tracrRNA-crRNA复合体,接着该复合体与Cas9蛋白结合,并在crRNA的引导下识别靶位点;(2)而对目的片段的切割是由crRNA上长度为20 bp的向导序列(guiding sequence)与目的DNA上的特定序列互补来引导Cas9蛋白对目的基因的切割;(3)除此之外,在所识别的目的DNA上还必需有一个3碱基(NGG)组成的PAM (Protospacer adjacent motif)区域,识别区域紧跟PAM的5′-端。
λ-Red重组酶由Exo、Beta和Gam等3种蛋白质组成,分别由exo、bet、gam等3个基因编码。这3种蛋白对于dsDNA的重组是必需的。首先,Gam蛋白通过大肠杆菌RecBCD和SbcCD核酸酶抑制线型dsDNA的降解,然后Exo从5′端到3′端的方向降解dsDNA,产生带3′末端的双链DNA,最后Beta蛋白结合在由Exo产生的3′突出端,防止外源DNA片段的降解,同时介导同源重组片段的退火延伸[8]。
大肠杆菌作为一种原核模式菌,因其遗传背景简单,容易培养,在基因改造以及获得脂肪酸工程菌的研究中得到很好的应用[9-13]。ppc基因是与蛋白质合成相关的基因,其编码的磷酸烯醇式丙酮酸羧化酶(PEPC)可以催化丙酮酸合成草酰乙酸,进而生成天冬氨酸进入蛋白质代谢,而丙酮酸生成乙酰辅酶A后在乙酰辅酶A羧化酶作用下进入脂肪酸合成途径,丙酮酸是两者共同的底物。我们拟通过部分和全部敲除ppc基因,阻断丙酮酸流向蛋白质合成的方向,来促使其流向脂肪酸合成方向[14-15]。另外,在大肠杆菌脂肪酸降解途径中,脂肪酸是被酯酰CoA合成酶FadD激活之后开始进入这一途径,阻断脂肪酸代谢途径有助于大肠杆菌对脂肪酸的积累。
本研究是运用CRISPR/Cas偶联λ-Red重组酶的系统,以一个二元载体pTargetF/pCas系统来敲除ppc和fadD基因,可以获得在基因组上无缺刻的基因编辑菌株,然后通过GC-MS来分析这两个基因对脂肪酸代谢的影响,本文中敲除ppc基因对脂肪酸产量的影响是首次报道。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒: Escherichia coli MG1655由厦门大学馈赠;大肠杆菌DH5α由本实验室保存。质粒pTargetF和pCas由中国科学院上海生命科学研究院馈赠;基因突变株由本实验构建。详见表 1。| 菌株及质粒 Strains or plasmids |
特征 Characteristics |
来源 Source |
| Strains | ||
| E. coli DH5α | F-endA1 glnV44 thi-1 recA1 relA1 gyrA96deoRnupGΦ80dlacZΔM15Δ(lacZYA-argF)U169 hsdR17 (rK– mK-)λ– | Trans Gen |
| E. coli MG1655 | K-12; F–λ– rph-1 | |
| 1655ΔFadD 1655ΔPEPCa 1655ΔPEPCb 1655ΔPEPCa:ΔFadD 1655ΔPEPC b:ΔFadD |
MG1655ΔfadD MG1655Δppca MG1655Δppcb MG1655ΔppcaΔfadD MG1655ΔppcbΔfadD |
This study This study Thisstudy This study This study Donor |
| Plasmids | repA101(Ts) kan Pcas-cas9 ParaB-Red lacIq Ptrc-sgRNA-pMB | Donor |
| pCas pTargetF pTargetF-FadD pTargetF-PEPCa pTargetF-PEPC |
pMB1 aadA sgRNA pMB1 aadA sgRNA-fadD pMB1 aadA sgRNA-ppca pMB1 aadA sgRNA-ppcb |
This study This study This study |
| 注:a:敲除ppc基因的部分片段;b:敲除ppc基因. Note: a: Knock-out partial of ppc gene; b: Knock-out complete ppc gene. |
||
为了获得带有与基因组中目的片段20 bp碱基互补的sgRNA,分别以P1/P2和P3/P4为引物对,以质粒pTargetF为模板扩增出可以识别fadD和ppc基因的pTargetF, 分别命名为sgRNA-fadD和sgRNA-ppc,然后将扩增的PCR产物转化至E. coli DH5α,分别利用引物P13/P14和P15/P16进行PCR鉴定,将鉴定正确的阳性克隆分别命名为pTargetF-sgRNA-fadD和pTargetF-sgRNA-ppc。以E. coli MG1655为材料,用细菌基因组DNA提取试剂盒提取MG1655基因组。分别以引物P5/P6和P7/P8、P9/P10和P11/P12扩增用于同源重组的fadD和ppc基因的上下游同源臂片段fadD上/下和ppc上/下,然后通过SOEing PCR将fadD上/下和ppc上/下分别连接起来,将电泳条带正确的分别命名为fadD同和ppc同。引物见表 2。
| 引物 Primers |
序列 Sequence (5′→3′) |
大小 Size (bp) |
| P1 | CCCTGACCGTTATCAATCTCGTTTTAGAGCTAGAAATAGC | 40 |
| P2 | GAGATTGATAACGGTCAGGGACTAGTATTATACCTAGGAC | 40 |
| P3 | CAGTATGCTCGGCAAAGTGCGTTTTAGAGCTAGAAATAGC | 40 |
| P4 | GCACTTTGCCGAGCATACTGACTAGTATTATACCTAGGAC | 40 |
| P5 | CACGCCGCAACAAGATTTAG | 20 |
| P6 | AGTGGGCGTCAAAAAAAACGGCGAAGCGCGTCACTATTTA | 40 |
| P7 | TAAATAGTGACGCGCTTCGCCGTTTTTTTTGACGCCCACT | 40 |
| P8 | TAACAGTTGCAGACAGGCCA | 20 |
| P9 | CATCCGTATTCAGGGCCAGT | 20 |
| P10 | ATTGCCAGCGCAGCAAACGAATAAATAGCAGGA | 33 |
| P11 | TCGTTTGCTGCGCTGGCAATGATGCTAACC | 30 |
| P12 | TCAATACGGACCAGCGGTAC | 20 |
| P13 | CGACCTACACCGAACTGAGA | 20 |
| P14 | GAGATTGATAACGGTCAGGG | 20 |
| P15 | CGACCTACACCGAACTGAGA | 20 |
| P16 | GCACTTTGCCGAGCATACTG | 20 |
| P17 | TGTTACTTGGGGCGATTTTT | 20 |
| P18 | CAGCGATTCCACTGCTTTTT | 20 |
| P19 | AAGCGTCGTGAATTTAATGA | 20 |
| P20 | GCCGAATGTAACGACAATTC | 20 |
E. coli MG1655用于构建突变体菌株,感受态细胞的制备参见文献[16]。首先将pCas转化到E. coli MG1655中,然后挑取含有pCas的MG1655的单克隆,在加入50 mg/L卡那霉素的LB培养基中,30℃、220 r/min培养至OD600为0.2时,向摇瓶中加入终浓度为10 mmol/L的阿拉伯糖诱导pCas载体上λ-Red蛋白的表达,然后摇至OD600约为0.4−0.5时回收菌体制备电转感受态。电转化时,向40μL的感受态细胞加入80 ng的pTargetF质粒和400 ng的同源臂DNA片段,轻柔混合后,加入到预冷的1 mm电转杯中,在1.8 kv的条件下放入电转仪(Bio-rad)中电转,电转完后迅速加入1 mL的LB培养基(室温),30℃、180 r/min培养1 h进行复苏,然后接种菌体于50 mg/L卡那霉素和50 mg/L壮观霉素的LB双抗固体培养基上,30℃培养过夜。转化子用PCR进行检测。
1.4 总酯提取及脂肪酸甲酯化 1.4.1 总酯的提取: 挑各突变株及野生菌株单克隆,在37℃培养到OD600为1.4左右,取200 mL菌液,8 000 r/min离心10 min回收菌体,吸取上清后,冷冻干燥菌体,然后向干燥的菌粉中加入4 mL ddH2O,漩涡振荡,悬浮菌体,然后沸水浴15 min,分别向冷却的悬浮液中加入20 mL的氯仿/甲醇(2:1,体积比),漩涡振荡3 min后,过夜,次日,离心后取下相(氯仿相),氮气吹干,即为总酯。 1.4.2 脂肪酸甲酯化: 挑各突变体菌株及野生菌株单克隆,37℃、180 r/min培养到OD600为1.4左右,取50 mL菌液回收菌体,冷冻干燥菌体后,各取20 mg干菌粉,用1 mL的ddH2O漩涡振荡,悬浮菌体,然后沸水浴15 min,待悬浮冷却后,加入100μL 0.5 g/L的C19作为内标,后续操作采用曲璟秋等[17]的方法。 1.5 GC-MS分析甲酯化后的脂肪酸利用安捷伦7890/5975C-GC/MSD进行分析,检测条件为:进样器温度250℃,检测器温度280℃,毛细管色谱柱(HP,30 m×0.25 mm,Film thickness of 0.25 m),氦气为载气,线速度为1 ml/min,程序升温条件为75℃(2 min),以15℃/min速度升至250℃,最后250℃(2 min),离子源温度220℃,EI离子源70 eV。
1.6 重组菌中质粒的消除为了消除pCas和pTargetF质粒,将重组菌(含有pCas和pTargetF)培养在LB培养基中(补加50 mg/L的卡那霉素,并加入终浓度为0.5 mmol/L的IPTG诱导靶向pTargetF上的pMB1的sgRNA-pMB1的表达)培养过夜,并将其涂布于含50 mg/L卡那霉素的平板上用于消除pTargetF,待平板上长出单克隆,挑菌落在含50 mg/L壮观霉素的液体LB中培养过夜,不长即为pTargetF消除的工程菌,然后挑取该工程菌在37℃的环境中培养过夜,用以消除pCas。
2 结果 2.1 基因缺失菌株的构建结果 2.1.1 FadD和ppc基因同源片段的扩增: 以E. coli MG1655基因组DNA为模板,分别扩增用于敲除fadD和ppc基因的上/下同源臂片段,电泳结果显示产物大小与预期片段大小一致。 2.1.2 缺失突变菌的鉴定: 将消除了质粒pTargetF和pCas的菌株,挑其单克隆,30℃、180 r/min培养2 h,回收菌体,菌体沉淀用1 mL的ddH2O洗涤2次,取20μL菌液破菌,利用引物P17/P18和P19/P20 (分别在敲除位点的上下游设计)经菌液PCR鉴定,初步判定敲除了fadD和ppc基因,然后挑取单克隆,30℃、180 r/min培养过夜,用细菌DNA提取试剂盒分别对各突变株提取基因组DNA,以基因组DNA为模板,同样利用上述引物进行扩增,获得了敲除fadD和ppc基因成功的菌株,结果见图 1。
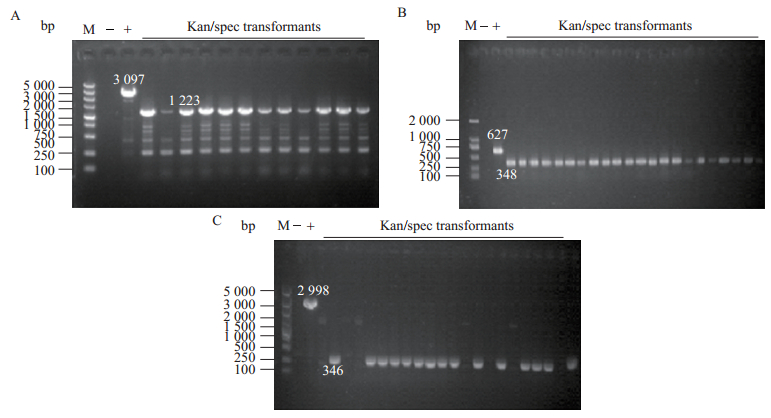
|
| 图 1 突变体菌株的鉴定 Figure 1 Identification of mutants by PCR. 注:M:DNA ladder marker;+:阳性克隆;−:阴性克隆. A和B分别为敲除fadD和ppc (部分敲除)基因的PCR鉴定,敲除效率为100%;C:敲除ppc基因的PCR鉴定,效率为68%±2%. Kan/spec transformants:在含有卡那霉素和壮观霉素双抗平板上挑取单克隆分别用引物P17/P18、P19/P20对其进行PCR鉴定. Note: M: DNA ladder marker; +: Positive control: −: Negative control. a and b: The strains, which successfully deleted fadD and ppc(partial), showed 100% mutation rate respectively. c: The ∆PEPC strain showed 68%±2% mutation rate. Kan/spec transformants: Identified the colonies by P17 and P18, P19 and P20 on the plate, which contains Kan and Spec resistance. |
|
|
从图 2中可以看出,无论是fadD和ppc基因分别敲除的菌株还是两个基因同时敲除的菌株,在生长情况上与野生型菌株相比,均没有很大的变化,说明在大肠杆菌体内,可能存在其他途径可以对这两个基因的作用进行补充,以弥补其缺失带来的生长缺陷,尤其是ppc基因,该基因的缺失会影响大肠杆菌蛋白质的合成,但并没有影响大肠杆菌的正常生长,这一发现需要后面作进一步探讨。
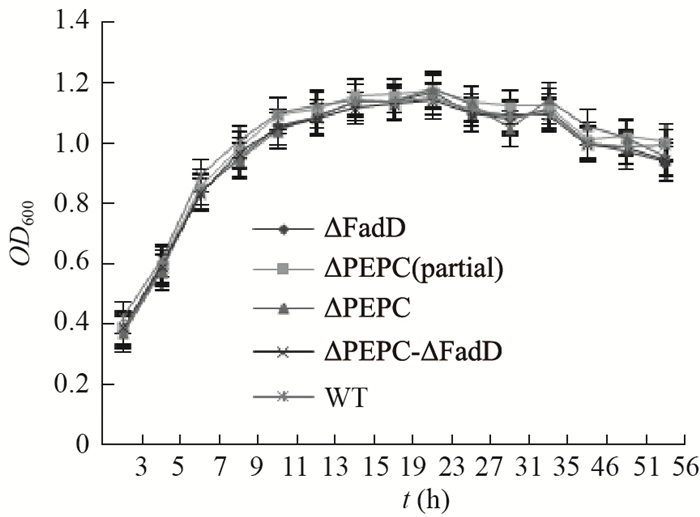
|
| 图 2 突变株及野生株的生长曲线 Figure 2 The growth curve of MG1655 and mutants 注:∆PEPC(partial)是指敲除部分ppc基因. Note: ∆PEPC(partial) represent knock-out partial ppc gene. |
|
|
提取重组菌株及野生菌株的总脂,发现重组菌株与野生型菌株相比,各重组菌株中脂肪酸的含量均有一定程度的提高,最高的增加了3.7% (表 3)。
| 菌株 Strains |
湿重 DCW (g/L) |
干重 DLW (mg/L) |
脂含量 Lipid content (%) |
| E. coil MG1655 | 1.45±0.09 | 103±4.2 | 7.1%±0.2% |
| 1655ΔFadD | 1.47±0.11 | 160±2.3 | 10.8%±0.3%* |
| 1655ΔPEPCa | 1.36±0.06 | 108±4.5 | 7.6%±0.1%* |
| 1655ΔPEPCb | 1.38±0.06 | 106±6.7 | 7.7%±0.2%* |
| 1655ΔPEPCa:ΔFadD | 1.46±0.09 | 153±11.0 | 10.5%±0.2%* |
| 1655ΔPEPC:ΔFadD | 1.37±0.06 | 115±15.2 | 10.7%±0.3%* |
| 注:a:敲除ppc基因的部分片段;b:敲除ppc基因;:对重组菌株脂肪酸含量与野生型菌株脂肪酸含量进行差异显著性分析表明差异显著(p=0.033 < 0.05). Note: a: Knock-out partial of ppc gene; b: Knock-out complete ppc gene; *: The fatty acid contents of recombinant strains and wild type strain indicates significant difference (p=0.033 < 0.05). |
|||
但是,敲除ppc基因菌株总脂含量比敲除fadD的低,同时部分敲除和全部敲除ppc基因的菌株在脂肪酸含量上几乎没有差别,说明只要敲除基因的开放阅读框的核心区,该基因就会失去作用。对于ppc和fadD基因双敲除的菌株,脂肪酸含量比单敲除ppc基因的含量要高3.1%。
为了进一步了解各菌株脂肪酸组成的成分变化,利用GC-MS对各菌株中的组分进行了定量分析。从图 3中可以看出,与敲除前相比,无论是ppc和fadD基因单敲除菌株还是ppc和fadD基因同时敲除的菌株,脂肪酸的组成都没有发生变化,都含有11:0、12:0、13:0、14:0、15:0、16:0、17:1、17:0和18:0,并且16:0、17:1和18:0占脂肪酸的主要组成部分。

|
| 图 3 脂肪酸组成变化 Figure 3 The change of fatty acid composition 注:∆PEPC(partial)是指敲除部分ppc基因. Note: ∆PEPC(partial) represent knock-out partial ppc gene. |
|
|
通过一个二元载体介导的CRISPR/Cas系统可以高效地构建大肠杆菌突变体菌株,相较于λ-Red重组系统,该系统也构建并表达了λ-Red重组酶,并且该系统只需要在PTargetF载体上构建一个与宿主目的基因NGG上游的20个碱基互补配对的寡核苷酸序列,然后与PCas共同发挥作用,就可以对宿主的基因进行编辑,从而快速而高效地获得突变体菌株,而且相较于Red重组系统会在菌株基因组上留下一段外源的碱基序列(包括FRT位点),CRISPR/Cas系统只会按照设计敲除或者插入目的基因,不会有其他基因被敲除或者插入,这样更有利于验证某些基因的作用,可以很好地控制实验结果。之前应用到大肠杆菌的CRISPR/Cas效率都很低[4],几乎无法获得阳性克隆。本系统的优势之处是利用了λ-Red重组酶与CRISPR/Cas系统结合,大大提高了重组效率,也很好地发挥了两者的优势。但是在多位点敲除并进行多位点的基因重组上还存在一定风险。
根据陈锦清[18]的底物竞争假说:在生物体的新陈代谢过程中, 葡萄糖酵解产物丙酮酸有两个去向:(1)在PEPC的催化下合成草酰乙酸,进而生成天冬氨酸进入蛋白质代谢;(2)在丙酮酸脱氢酶的作用下合成乙酰辅酶A,然后在乙酰辅酶A羧化酶(ACCase)的催化下生成丙二酸单酰辅酶A进入脂类代谢。如果抑制了PEPC的表达,将会有更多的丙酮酸用于合成脂类,蛋白质的合成即会减少。而本实验的结果却发现在敲除ppc基因时,菌株的脂肪酸含量相较于野生型并没有明显的变化,同时突变体菌株的生长状况与野生型相比也没有明显的变化,说明在大肠杆菌体内通过单独敲除控制蛋白质途径的基因并不能很好地促进前体物质流向脂肪酸合成的代谢途径。并且,脂肪酸和蛋白质存在一定的物质和能量的交流,蛋白质可以转化成脂肪,脂肪在特殊情况下也可以转化为蛋白质,所以控制这两种物质含量的比例或者控制这两个物质合成途径中的某些酶的相对活性可能才是脂肪酸合成的关键调控点[19]。
我们利用表达λ-Red重组酶的CRISPR/Cas系统,选取与脂肪酸合成途径相关和脂肪酸降解途径中的关键基因进行基因敲除,阐述这些基因与脂肪酸产量的关系。通过研究发现,阻断脂肪酸β-氧化途径的基因fadD可以明显地提高菌体脂肪酸的含量,说明敲除这一基因对脂肪酸的积累起到了一个积极的作用。阻断了控制丙酮酸流向蛋白质方向的ppc基因,使丙酮酸流向脂肪酸合成途径并没有引起菌体中脂肪酸的明显变化,表明生物体中合成的脂肪酸转化为了部分蛋白质,以补充蛋白质缺失对菌体的影响。
| [1] | Terns MP, Terns RM. CRISPR-based adaptive immune systems[J]. Current Opinion in Microbiology , 2011, 14 (3) : 321–327. DOI:10.1016/j.mib.2011.03.005 |
| [2] | Richter C, Chang JT, Fineran PC. Function and regulation of clustered regularly interspaced short palindromic repeats (CRISPR)/CRISPR associated (Cas) systems[J]. Viruses , 2012, 4 (10) : 2291–2311. |
| [3] | Wiedenheft B, Sternberg SH, Doudna JA. RNA-guided genetic silencing systems in bacteria and archaea[J]. Nature , 2012, 482 (7385) : 331–338. DOI:10.1038/nature10886 |
| [4] | Jiang WY, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nature Biotechnology , 2013, 31 (3) : 233–239. DOI:10.1038/nbt.2508 |
| [5] | Chang NN, Sun CH, Gao L, et al. Genome editing with RNA-guided Cas9 nuclease in Zebrafish embryos[J]. Cell Research , 2013, 23 (4) : 465–472. DOI:10.1038/cr.2013.45 |
| [6] | Feng ZY, Zhang BT, Ding WN, et al. Efficient genome editing in plants using a CRISPR/Cas system[J]. Cell Research , 2013, 23 (10) : 1229–1232. DOI:10.1038/cr.2013.114 |
| [7] | Si T. Genome engineering in Saccharomyces cerevisiae. Urbana-Champaign: Doctoral Dissertation of University of Illinois at Urbana-Champaign[M]. 2015 . |
| [8] | Court DL, Sawitzke JA, Thomason LC. Genetic engineering using homologous recombination 1[J]. Annual Review of Genetics , 2002, 36 (1) : 361–388. DOI:10.1146/annurev.genet.36.061102.093104 |
| [9] | Steen EJ, Kang YS, Bokinsky G, et al. Microbial production of fatty-acid-derived fuels and chemicals from plant biomass[J]. Nature , 2010, 463 (7280) : 559–562. DOI:10.1038/nature08721 |
| [10] | Lu XF, Vora H, Khosla C. Overproduction of free fatty acids in E. coli: implications for biodiesel production[J]. Metabolic Engineering , 2008, 10 (6) : 333–339. DOI:10.1016/j.ymben.2008.08.006 |
| [11] | Youngquist JT, Lennen RM, Ranatunga DR, et al. Kinetic modeling of free fatty acid production in Escherichia coli based on continuous cultivation of a plasmid free strain[J]. Biotechnology and Bioengineering , 2012, 109 (6) : 1518–1527. DOI:10.1002/bit.24420 |
| [12] | Jeon E, Lee S, Won J-I, et al. Development of Escherichia coli MG1655 strains to produce long chain fatty acids by engineering fatty acid synthesis (FAS) metabolism[J]. Enzyme and Microbial Technology , 2011, 49 (1) : 44–51. DOI:10.1016/j.enzmictec.2011.04.001 |
| [13] | Liu H, Yu C, Feng DX, et al. Production of extracellular fatty acid using engineered Escherichia coli[J]. Microbial Cell Factories , 2012, 11 (1) : 41–54. DOI:10.1186/1475-2859-11-41 |
| [14] | Li JQ, Zheng SX, Yu ZN, et al. Acetyl-coenzyme A Carboxylase: A key metabolic enzyme of fatty acid and progress of its gene clone[J]. Chinese Journal of Applied Environmental Biology , 2011, 17 (5) : 753–758. (in chinese) 李洁琼, 郑世学, 喻子牛, 等. 乙酰辅酶A羧化酶:脂肪酸代谢的关键酶及其基因克隆研究进展[J]. 应用与环境生物学报 , 2011, 17 (5) : 753–758. |
| [15] | Hou LJ, Shi DJ, Cai ZF, et al. Regulation of lipid synthesis in transgenic Escherichia coli by inserting Cyanobacterial sense and antisense pepcA gene[J]. Chinese Biotechnology , 2008, 28 (5) : 52–58. (in chinese) 侯李君, 施定基, 蔡泽富, 等. 蓝藻正反义pepcA基因导入对大肠杆菌中脂类合成的调控[J]. 中国生物工程杂志 , 2008, 28 (5) : 52–58. |
| [16] | Sharan SK, Thomason LC, Kuznetsov SG, et al. Recombineering: a homologous recombination-based method of genetic engineering[J]. Nature Protocols , 2009, 4 (2) : 206–223. DOI:10.1038/nprot.2008.227 |
| [17] | Qu JQ, Liu CH, Liu WF, et al. Construction of Escherichia coli gene konck-out mutants for engineering of fatty acid metabolism[J]. Acta Microbiologica Sinica , 2013, 53 (6) : 608–614. (in chinese) 曲璟秋, 刘翠花, 刘伟丰, 等. 应用基因敲除快速构建大肠杆菌突变体改造脂肪酸代谢途径[J]. 微生物学报 , 2013, 53 (6) : 608–614. |
| [18] | Chen JQ, Lang CX, Hu ZH, et al. Antisense PEP gene regulates to ration of protein and lipid content in Brassica napus seeds[J]. Journal of Agricultural Biotechnology , 1999, 7 (4) : 316–320. (in chinese) 陈锦清, 郎春秀, 胡张华, 等. 反义PEP基因调控油菜籽粒蛋白质/油脂含量比率的研究[J]. 农业生物技术学报 , 1999, 7 (4) : 316–320. |
| [19] | Kubis SE, Pike MJ, Everett CJ, et al. The import of phosphoenolpyruvate by plastids from developing embryos of oilseed rape, Brassica napus (L.), and its potential as a substrate for fatty acid synthesis[J]. Journal of Experimental Botany , 2004, 55 (402) : 1455–1462. DOI:10.1093/jxb/erh157 |
 2016, Vol. 48
2016, Vol. 48




