扩展功能
文章信息
- 黎志凤, 刘杨, 朱敬, SINGHRaghvendra Pratap, 李越中
- LI Zhi-Feng, LIU Yang, ZHU Jing, SINGH Raghvendra Pratap, LI Yue-Zhong
- 应用生物信息学的系统化综合性教学改革实践
- Teaching reforms the systematicness and comprehensiveness of Applied Bioinformatics
- 微生物学通报, 2016, 43(8): 1854-1862
- Microbiology China, 2016, 43(8): 1854-1862
- DOI: 10.13344/j.microbiol.china.160335
-
文章历史
- 收稿日期: 2016-04-26
- 接受日期: 2016-06-28
- 优先数字出版日期(www.cnki.net): 2016-06-30
2. 山东大学数学学院 山东 济南 250100
2. School of Mathematics, Shandong University, Jinan, Shandong 250100, China
20世纪90年代因特网诞生了,并伴随着信息高速公路计划的大力实施,这意味着信息时代的到来。与此同时,生命科学领域也经历着革命性的飞跃,科学家们开始了大规模的基因组研究。计算机因特网和基因组计划的历史性碰撞,不可阻挡地催生了新世纪新兴学科——生物信息学[1-5]。生物信息学出现还不到20年,但是已渗透到生命科学各领域的各个层次。毫无疑问,生物信息学应用技术将成为所有生物学工作者必须掌握的基本技能[4-5]。因此,为迎接这一历史潮流,应用生物信息学人才的培养成为新时期生命科学人才培养的重要环节。早在20世纪末,欧洲、美国、日本等国家便达成共识,即应高度优先满足对生物信息学技术人才培养的需求[3-4]。目前,发达国家已实现了对相关人才培养组织的大力资助[5-9]。随着生物信息学在生命科学领域的日益渗透,我国也开始在本科教育阶段普及这门新兴学科。当前,国内的北京大学、清华大学、浙江大学、武汉大学、华中科技大学、上海交通大学等各大高校如雨后春笋般地展开了本科生生物信息学教学。
早在1999年,山东大学在国内高校率先开设了研究生生物信息学课程,这在一定程度上推动了国内生物信息学教学的发展[10-11]。为进一步迎接新时期的机遇,2006年山东大学生命科学学院面向本科生开设了54学时的应用生物信息学课程,同时建设了生物信息学实践机房[12]。该课程适用于具备了生物化学、分子生物学、计算机基础等相关知识背景的学生学习。生物信息学借由计算机和网络而诞生,因此应用生物信息学的教学实践离不开计算机软件和网络数据库[10-11]。然而,实践中我们发现了一些问题,诸如:相关课程内容结构缺乏系统性,只见树木不见森林,内容设置偏重于就软件讲软件、就数据库讲数据库,未能将生物学问题贯穿于整个教学。由此,我们尝试了教学改革,并在2008年生命科学基础课程报告论坛上进行了交流[12]。如今教学改革已经实践了8年,本文将详尽深入地探讨如何提升课程内容的系统性和综合性。
1 应用生物信息学教学内容系统性布局 1.1 课程定位什么是生物信息学?这个问题看似简单却不易准确回答。生物信息学是一门高速发展的多学科交叉的新兴学科,其学科知识每天都发生着细微变化,而且不同领域的科学家对生物信息学的定义也有着不同的侧重点[7]。基于此,2000年美国国立卫生研究院(National Institutes of Health,NIH)规范了生物信息学的定义[13]——“生物信息学即研究、开发或者应用计算工具和方法来扩展对生物学、医学、行为科学和卫生学数据的利用,包括此类数据的采集、存储、整理、归档、分析与可视化”[3, 13]。在众多学科中有一门学科与生物信息学密切相关,即计算生物学。计算生物学的规范定义是“开发和应用数据分析、理论方法、数学模型和计算机仿真技术,用于生物学、行为学和社会群体系统的研究”[3, 13]。两者相比较,生物信息学侧重于应用计算机手段解决生物学问题,而计算生物学则侧重于开发数理方法应用于生物学研究。
考虑到生命科学背景的学生今后科研工作的切实需求,我们的应用生物信息学教学定位是以生物学问题作为灵魂贯穿于软件和数据库的应用中,指导学生利用生物学软件对实验产生的数据进行合理有效的处理,并启发学生利用数据库资源对数据背后的生物学问题进行有理有据的解析。课程宗旨是训练学生勤于实践、全方位解析信息以及系统思考问题的能力,同时激发学生对这门学科的兴趣和热爱。
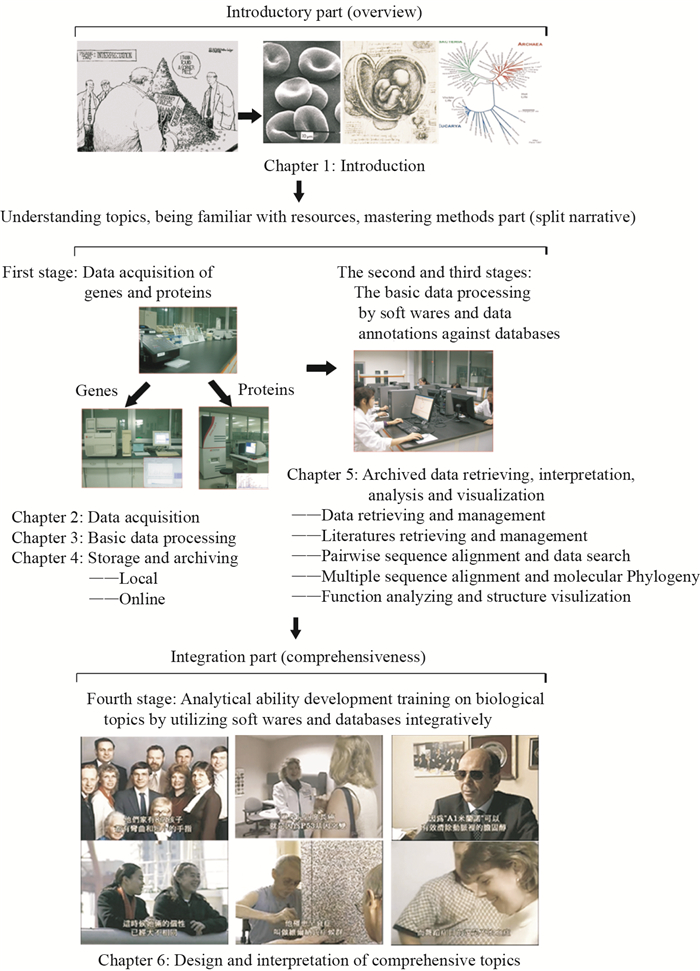
1.2 结构内容体系基于课程定位,为引导学生学好应用生物信息学课,我们强调了解问题、熟悉资源、掌握方法和融会贯通这4个方面[3-4]。具体的教学安排分为4个阶段(图 1)[12]:首先,采集基因与蛋白的数据(第一阶段);再使用软件对采集的数据进行基本处理(第二阶段);然后,应用各种一、二级数据库对处理后的数据进行注释与解析(第三阶段);最后,融会贯通地应用各种软件与数据库进行生物学问题的综合解析(第四阶段)。前三阶段的重点是了解问题、熟悉资源、掌握方法,第四阶段的重点是融会贯通,由此形成一套有层次的阶梯递进式教学体系。整个体系结构由合(绪论)到分(具体章节),再由分(具体章节)到合(综合问题设计解析)。具体内容由浅入深、环环相扣,包括:绪论→分子数据的序列采集→采集数据的基本处理→处理数据的存储、整理与归档(包括“本地处理”与“在线处理”两部分)→归档数据的获取、解读、解析与可视化分析(包括生物数据检索与管理,生物文献检索与管理,核酸蛋白的双序列比对与数据搜索,核酸蛋白的多序列比对与进化分析,以及核酸蛋白序列结构功能注释解析与可视化共五部分)→综合问题的设计与解析。基于多年的科研工作和学习实践,我们总结出来的这套内容布局有助于学生把握整体结构中各子环节的串并联关系,以及每个章节的来龙去脉、前因后果。
作为一门新兴交叉学科,应用生物信息学具有知识面覆盖广、更新快的特点。针对这些特点,我们在实践中应用了以下教学手段[12]:(1) 增设基因与蛋白数据采集源头。由于生物信息学是基于生物数据的累积而诞生,增设数据采集源头有利于两个方面:一方面,形成了一套由初始数据开始的完整体系;另一方面,能使学生重温当年生物信息学诞生的历史际遇,深刻感受到数据并不等于知识,体会到获得实验数据后更需要的是数据处理与解析,从而激发学生对后续章节的学习兴趣。(2) 把握时代气息,与时俱进。随着生命科学研究的深入,生物信息学资源广度和深度的变化可谓日新月异,相关软件工具处于不断的更新中,相关数据库也同样处于迅速的发展整合中。当前主流数据库资源信息入口包括:核酸研究期刊(Nucleic Acids Research)数据库专辑(Database issue)[14],欧洲生物信息研究所(European Bioinformatics Institute,EBI)[9],美国国家生物信息技术中心(National Center for Biotechnology Information,NCBI)[8]和日本京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)[15-16]。介绍原始资源链接能够使学生从静态链接入口把握动态更新的软件和数据库,与时俱进。由于授课内容和资源信息与国际同步,让学生们有了与国际接轨、站在时代前沿的深刻体会。(3) 利用英文视频提升专业英语水平。生物信息学是一门与时俱进的国际性学科,所以众多顶级软件公司和权威生物信息数据库维护中心制作了大量相关英文视频资源。积极地利用这些资源可帮助我们适时变换教学模式并强化学生专业英语听、说、读、写、解的能力。(4) 互动教学。利用网络设备和软件系统,构建互动课堂,邀请学生现场演示,达到师生间和学生间的实时互动与反馈。同时,设立班级公共邮箱,上传教学课件与拓展资源,设立优秀展示专区,择优选取某方面见长的优秀报告供大家揣摩学习,让学生们认识到自己当前的不足之处,进而取百家之长进行改进。(5) 考核评价。就实践性强且发展变化快的学科而言,传统闭卷考试显然不能对学生掌握情况和学习能力做出合理评价。由此,我们综合课下实践与课堂演练情况对学生进行考评。平时成绩占40%,由学生个人课下独立完成,主要考核学生应用具体单一软件或数据库的能力,执行中可寻求他人帮助但需要在致谢中声明。期末成绩占60%,考核学生就综合问题融会贯通地应用多种软件和数据库的解析能力。学生根据所学课下自主寻求优秀素材完成期末的综合实践性研究报告,允许学生组建小团队合作完成,但要求标明各自的分工与贡献。实践报告从排版完整规范度(15%)、思路流程清晰流畅度(20%)、概念正确结论完善度(20%)、态度认真及时度(20%)、思考发挥创造度(25%)五个角度评价,使学生各方面优点都能在成绩中得以体现。作为开卷考评,为规范道德,还需强调雷同报告罚分力度,直接引用必须标注文献出处等。
2 综合趣味性生物学问题解析实例第四阶段融会贯通的训练,取决于易理解的综合性生物学问题。通过积极地搜集分析,我们发现部分优秀科普视频将Science、Nature等顶级期刊的学术研究进行了科普化。我们若能善加利用,则可将视频内容设计转化为综合趣味性生物学问题进行实践演练。在此,以美国DISCOVERY和日本NHK公司合资制作的《DNA时代》科教视频中的“冰冻人(iceman)”片段剪辑(位于第五集4′30′′−12′00′′)[17]为例进行交流。
2.1 解读视频剪辑了解问题人体细胞内环状线粒体DNA远多于细胞核DNA,因此线粒体DNA很容易从古代骸骨收集。所以,通过分析线粒体DNA,可以发现古代人与现代人的关系,寻找人的根。
1991年,在阿尔卑斯山发现了冰冻的干尸。干尸旁边有箭、斧头、绳索等器物。这具干尸有5 300岁,被称为冰冻人。人们不禁提出以下生物学问题:当时这位冰冻人,身披毛皮背负猎具在山上行走,他是哪里人?他的后裔是谁?据此,牛津大学布莱安塞克斯教授通过样本的采集与信息分析,发现冰冻人与现代人差异较大的序列特征区,并找到了冰冻人的后裔(图 2)[17]。
2.2 建立视频剪辑细节与课程内容的关联我们对视频资料提供的生物学问题进行细节分析,并逐一将其与课程所学内容进行关联(表 1)。整个生物学问题具体实践下来,需要贯穿运用每个章节所学内容。
| 顺序 Order |
视频剪辑细节分析 Analyzing details in the video clips |
课程内容 Contents in the course |
| 采集 Acquisition |
首先,布莱安塞克斯教授对样本进行了数据采集 | 运用“分子数据的序列采集”章节所学 |
| 处理 Processing |
采集后,需对数据进行基本处理 | 运用“采集数据的基本处理”章节所学 |
| 存档 Archiving |
处理后,需对数据进行存储、整理、归档以便发表共享 | 运用“处理数据的在线存储、整理与归档”章节所学 |
| 文献 Literature |
研究成果已经报道,应该有文献依据可查 | 运用“生物文献检索与管理”章节所学,获取原文 |
| 记录 Record |
研究成果已经报道,相关序列应该已提交至数据库存档共享 | 运用“生物数据检索与管理”章节所学,获取序列 |
| 序列细节 Details of the sequence |
视频中提及布莱安塞克斯教授只是选取了一段序列进行分析,整个线粒体注释怎样?布莱安塞克斯教授选取了哪段序列? | 运用“核酸、蛋白序列结构功能注释解析与可视化”章节所学,可从相关二级或专有数据库找到答案 |
| 双序列比对 Pairwise sequence alignment |
在相对的世界里,对未知事物的认知,只能通过比较作出评判。同样,认知冰冻人的序列需进行比较研究 | 运用“生物数据搜索-核酸双序列比对与数据搜索”章节所学 |
| 多序列比对与进化 Multiple sequence alignment and molecular phylogeny |
通过多方面的比较才能对事物有相对完善的认知。就种群研究而言,构建进化树是个很好的直观展示途径 | 运用“核酸的多序列比对与进化分析”章节所学 |
理清思路后,引导学生融会贯通地运用课程所学内容进行具体解析求证,求证路线如图 3所示。具体步骤如下:

|
| 图 3 基于视频剪辑信息设计的实践论证路线 Figure 3 The practical comfirmation flow chart based on video clips information |
|
|
(1) 提炼视频剪辑[17]关键词信息。研究报道时间:1994年;考察人物:牛津大学布莱安塞克斯教授;考察对象:冰冻人(iceman or ice man)、新石器时代干尸(neolithic mummy)、线粒体(mitochondrion or mitochondrial)、D-loop区(D-loop region)等。
(2) 利用提炼的关键词对主题词(Medical Subject Headings,MeSh)数据库[18]进行检索,并获取原文。提交检索式(“Neolithic mummy”AND Mitiochondrial)、(“DNA, Mitochondrial”[Majr] AND“Mummies”[Majr] Limits: Publication Date from 1993/12/13 to 1995/1/1)、(“Neolithic mummy”AND“Pubmed nucleotide”[Filter])、(“DNA, Mitochondrial”[Majr:NoExp] AND“Neolithic mummy”)等执行Entrez检索[19],能殊途同归地获取原始文献。核对文献信息,明确该研究于1994年发表在Science期刊,研究题目为“Molecular genetic analyses of the Tyrolean Ice Man”[20],论文署名作者中的牛津大学布莱安塞克斯教授(Bryan Sykes,University of Oxford)正是视频中采访的科学家。
(3) 依据文献获取序列。Entrez是NCBI的集成数据库检索系统[19],其中pubmed[18]与二十几个数据库集成偶联。根据文献可轻易地通过“Secondary Source ID”或“Related information”获取关联的核酸序列。由此,顺藤摸瓜地获取研究论文已提交的序列。该序列GenBank登录号为S69989,定义为“Control region hypervariable segment: {Mummified DNA} [Human, Tyrolean Alps, Late Neolithic Ice Man, Mitochondrial, 354 nt]”。此处定义为Control region?与视频所提及的D-loop区是否同一区间?有待进一步了解详细注释信息后作答。
(4) 依二级或专有数据库全面了解线粒体注释信息。从NCBI基因组数据库查看线粒体完整信息。人的线粒体16.6 kb,包含37个基因(蛋白编码基因13个和RNA基因24个),编码基因比例68%。借助专有数据库MITOMAP (A human mitochondrial genome database)[21]得到详细注释信息:Control region (控制区)由1 122 bp组成,包含D环区(displacement-loop region,D-Loop)及其邻近的转录启动子区。作为线粒体DNA多态性分析的主选区域,Control region和D-loop区有时在文献中被当作同义词使用。
(5) 利用获得的序列号进行数据库比对与进化分析。登录NCBI数据库BLAST在线程序[22]界面提交序列号,利用限制性搜索(Entrez Query项中用布尔逻辑符“OR”限定各国人种[19]),执行数据库搜索比对与进化分析;也可利用Batch download tool、clustalX[23-24]、Bioedit[25]和MEGA[26]软件进行序列下载和本地化比对与进化分析。
2.4 视频剪辑实践结果与讨论基于冰冻人这一科普视频,我们从提炼关键词开始直至考证信息,最终得出结论:(1) 该视频剪辑内容[17]来源于1994年牛津大学Bryan Sykes教授发表在Science期刊的研究成果[20]。(2) 冰冻人序列已提交至数据库(序列登录号:S69989),且冰冻人序列中有两处不同于现代人(图 4A)。(3) 冰冻人与现代南欧人亲缘关系最近(图 4B),通过序列分析发现了遥遥相隔5 300年的冰冻人后裔(图 4C);(4) 实践解析结果与1994年Science文献报道[20]吻合。(5) 视频报道的[17]正是Bryan Sykes教授的研究[20],但视频中冰冻人特征序列信息描述有误,冰冻人区别于现代人的特征序列不是CCCC和TAGC,而是CCCT和TAGT;CCCC和TAGC是大多数现代人的序列,科普视频将大多数现代人的特征序列误报道为冰冻人的特征序列,对此我们予以纠正,以免以讹传讹(图 4A)。

|
| 图 4 冰冻人剪辑实践解析结果 Figure 4 The practice results of iceman video clips 注:A:多序列比对分析结果;B:进化分析结果:括号中的号码代表序列GenBank登录号,分支点上的数字代表计算1 000次聚类相关分支的支持概率,标尺代表 0.5%序列差异的单位长度;C:视频片段剪辑. Note: A: Multiple sequence alignment results; B: Evolutionary results: Numbers in parenthesis represent the sequence's accession number in GenBank, the number at each branch point indicates the percentage supported by bootstrap, bar means 0.5% sequence divergence; C: Video clips. |
|
|
因任何研究都不是一成不变的,研究论文发表后即会产生一定的影响并引发后续工作。所以,我们在实践最后增加了生物学问题研究进展。相关研究进展环节可启发学生以发展观而不是以僵化观认识问题。
经过相关检索,我们了解到如下进展:(1) 自1994年Bryan Sykes教授等在Science上发表了冰冻人研究以来,该报道已经被引用167次。(2) 为更好地理解欧洲史前人口种群情况,科学家们展开了相对广泛的古DNA研究,时间跨度从旧石器时代到史前时代[27]。(3) 伴随技术的进步,今天的科学家们已经通过测序获得了冰冻人完整线粒体和全基因组序列,并通过注释,对冰冻人可能的特征和潜在的患病基因等进行了分析预测,如冰冻人线粒体为现代罕见的单倍群、蓝眼睛、O型血、患乳糖不耐症、可能因血管钙化易患心胀病、携带莱姆疏螺旋(Lyme borreliosis)病原体等[28-29]。
3 教学改革效果与结论教学是教师的“教”和学生的“学”所组成的一种特有的人才培养活动。针对应用生物信息学这门课,我们在系统化综合性方面进行了改革实践,希望通过教学使学生由被动学习转化为主动学习、主动求教、主动探索,使学生能够将书本静态的死知识活学活用并解决实际生物学问题,从而促进学生多方面综合素质提升,进而提升教学效果。
就教学成效而言,学生的评价与感触是最具说服力的[30]。在此,选取了如下具代表性的学生感言:
“实践中遇到各种问题,但从未气馁,不断尝试,不断学习,克服了所有困难,取得令人欣慰的结果”,“虽然浪费了大量时间摸索,但通过实践明白机理后,兴奋异常”,“最喜欢的一门课”,“从一个只会被动接受知识的学生开始转变为主动寻找答案,主动去思考的学生”,“能利用Nature中的论文自设生物学问题解析实践”,“很庆幸选择生物信息学这门课”,“实践锻炼了勤于思考、敢于尝试创新的能力,得到很大帮助”,“对课程产生浓厚兴趣”,“感受到团队合作的重要性和亲自实践的成就感”,“第一次如此系统地接受如此专业的训练和学习,认识更加深刻”,“团队分工合作,亲身接触了生物信息学的研究方法,感受到它的魅力”。从这些反馈信息来看,学生们在实践中受益匪浅。我们的教学改革取得了很好的效果,可供广大同行参考交流,以期取长补短、共同进步。
| [1] | Jiang Y, Wang XX, Cao Y, et al. Basic Bioinformatics and Application[M]. Beijing: Tsinghua University Press, 2003: 255 . (in chinese) 蒋彦, 王小行, 曹毅, 等. 基础生物信息学及应用[M]. 北京: 清华大学出版社, 2003: 255 . |
| [2] | Luo JC. Teaching the ABCs of bioinformatics: a brief introduction to the Applied Bioinformatics Course[J]. Briefings in Bioinformatics , 2014, 15 (6) : 1004–1013. DOI:10.1093/bib/bbt065 |
| [3] | Pevsner J. Bioinformatics and Functional Genomics[M]. Sun ZR, Trans. Beijing: Chemical Industry Press, 2006: 706 (in Chinese) 乔纳森·佩夫斯纳.生物信息学与功能基因组学[M].孙之荣, 译.北京:化学工业出版社, 2006: 706 |
| [4] | Attwood TK, Parry-Smith DJ. Introduction to Bioinformatics[M]. Luo JC, Trans. Beijing: Peking University Press, 2002: 280 (in Chinese) Attwood TK, Parry-Smith DJ.生物信息学概论[M].罗静初, 译.北京:北京大学出版社, 2002: 280 |
| [5] | Neil SI. Editorial: Bioinformatics education in the 21st century[J]. Briefings in Bioinformatics , 2010, 11 (6) : 535–536. DOI:10.1093/bib/bbq071 |
| [6] | Revote J, Watson-Haigh NS, Quenette S, et al. Development of a cloud-based Bioinformatics Training Platform[J]. Briefings in Bioinformatics , 2016 . DOI:10.1093/bib/bbw032 |
| [7] | Cummings MP, Temple GG. Broader incorporation of bioinformatics in education: opportunities and challenges[J]. Briefings in Bioinformatics , 2010, 11 (6) : 537–543. DOI:10.1093/bib/bbq058 |
| [8] | Cooper PS, Lipshultz D, Matten WT, et al. Education resources of the National Center for Biotechnology Information[J]. Briefings in Bioinformatics , 2010, 11 (6) : 563–569. DOI:10.1093/bib/bbq022 |
| [9] | Wright VA, Vaughan BW, Laurent T, et al. Bioinformatics training: selecting an appropriate learning content management system-an example from the European Bioinformatics Institute[J]. Briefings in Bioinformatics , 2010, 11 (6) : 552–562. DOI:10.1093/bib/bbq023 |
| [10] | Wang LS, Gao PJ. Bioinformatics Application Technology[M]. Beijing: Chemical Industry Press, 2008: 253 . (in chinese) 王禄山, 高培基. 生物信息学应用技术[M]. 北京: 化学工业出版社, 2008: 253 . |
| [11] | Li YZ. Genomics and Bioinformatics[M]. Jinan: Shandong University Press, 2001: 311 . (in chinese) 李越中. 基因组研究与生物信息学[M]. 济南: 山东大学出版社, 2001: 311 . |
| [12] | Li ZF, Zhu J, Li YZ, et al. Applied Bioinformatics teaching method reform exploration and practice[A]//The forum corpus of the foundational life science course reports in colleges[C]. Xinxiang: Higher Education Press, 2008: 129-135 (in Chinese) 黎志凤, 朱敬, 李越中, 等.应用生物信息学教学方法改革探索与实践[A]//高校生命科学基础课程报告论坛文集[C].新乡:高等教育出版社, 2008: 129-135 |
| [13] | Huerta M, Haseltine F, Liu Y, et al. NIH working definition of bioinformatics and computational biology[S]. Biomedical Computation Review, BISTIC Definition Committee, 2000 |
| [14] | Rigden DJ, Fernández-Suárez XM, Galperin MY. The 2016 database issue of Nucleic Acids Research and an updated molecular biology database collection[J]. Nucleic Acids Research , 2016, 44 (D1) . |
| [15] | Ogata H, Goto S, Sato K, et al. KEGG: Kyoto Encyclopedia of genes and genomes[J]. Nucleic Acids Research , 1999, 27 (1) : 29–34. DOI:10.1093/nar/27.1.29 |
| [16] | Kanehisa M, Goto S, Sato Y, et al. KEGG for integration and interpretation of large-scale molecular data sets[J]. Nucleic Acids Research , 2012, 40 (D1) . |
| [17] | Ma Y (Trans). < DNA Age > (Human BodyⅢ. DNA Age)[CD]. USA: DISCOVERY and Japan: NHK, 2000 (in Chinese) 马英(译).《DNA时代》(Human BodyⅢ. DNA Age)[CD].美国: DISCOVERY和日本: NHK, 2000 |
| [18] | Giglia E. PubMed in progress: latest changes in MeSH and MyNCBI[J]. European Journal of Physical and Rehabilitation Medicine , 2011, 47 (3) : 525–528. |
| [19] | Baxevanis AD. Searching the NCBI databases using Entrez[A]//Current Protocols in Human Genetics[M]. New York: John Wiley & Sons, Inc., 2006 |
| [20] | Handt O, Richards M, Trommsdorff M, et al. Molecular genetic analyses of the Tyrolean Ice Man[J]. Science , 1994, 264 (5166) : 1775–1778. DOI:10.1126/science.8209259 |
| [21] | Brandon MC, Lott MT, Nguyen KC, et al. MITOMAP: a human mitochondrial genome database-2004 update[J]. Nucleic Acids Research , 2005, 33 (D1) . |
| [22] | Ladunga I. Finding homologs to nucleotide sequences using network BLAST searches[A]//Current Protocols in Bioinformatics[M]. New York: John Wiley & Sons, Inc., 2002 |
| [23] | Larkin MA, Blackshields G, Brown NP, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatic , 2007, 23 (21) : 2947–2948. DOI:10.1093/bioinformatics/btm404 |
| [24] | Sievers F, Higgins DG. Clustal Omega, accurate alignment of very large numbers of sequences[A]//Russell DJ. Multiple Sequence Alignment Methods: Methods in Molecular Biology[M]. New York: Humana Press, 2014, 1079: 105-116 |
| [25] | Hall TA. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series , 1999, 41 : 95–98. |
| [26] | Kumar S, Stecher G, Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution , 2016, 33 (7) : 1870–1874. DOI:10.1093/molbev/msw054 |
| [27] | Lacan M, Keyser C, Crubézy E, et al. Ancestry of modern Europeans: contributions of ancient DNA[J]. Cellular and Molecular Life Sciences , 2013, 70 (14) : 2473–2487. DOI:10.1007/s00018-012-1180-5 |
| [28] | Keller A, Graefen A, Ball M, et al. New insights into the Tyrolean Iceman's origin and phenotype as inferred by whole-genome sequencing[J]. Nature Communications , 2012, 3 : 698. DOI:10.1038/ncomms1701 |
| [29] | Coia V, Cipollini G, Anagnostou P, et al. Whole mitochondrial DNA sequencing in Alpine populations and the genetic history of the Neolithic Tyrolean Iceman[J]. Scientific Reports , 2016, 6 : 18932. DOI:10.1038/srep18932 |
| [30] | Zheng XJ, Meng ZN, Zhang ZG. Represent the research-oriented teaching life science classroom teaching quality evaluation[C]. Tianjin: Higher Education Press, 2007: 226-229 (in Chinese) 郑晓健, 孟振农, 张治国.体现研究型教学的生命科学课堂教学质量评价[C].天津:高等教育出版社, 2007: 226-229 |
 2016, Vol. 43
2016, Vol. 43