扩展功能
文章信息
- 张香美, 许冬倩, 闫洪波
- ZHANG Xiang-Mei, XU Dong-Qian, YAN Hong-Bo
- 细菌sRNA与群体感应系统相互作用研究进展
- Research progress on interaction between bacterial sRNA and quorum sensing system
- 微生物学通报, 2016, 43(8): 1822-1828
- Microbiology China, 2016, 43(8): 1822-1828
- DOI: 10.13344/j.microbiol.china.150725
-
文章历史
- 收稿日期: 2015-09-24
- 接受日期: 2015-12-14
- 优先数字出版日期(www.cnki.net): 2015-12-14
温度、pH、渗透压以及营养缺乏等各种环境压力往往会影响细菌的代谢过程,为适应变化的环境,细菌进化出多种响应环境刺激的调控系统或调控因子,如群体感应系统、sRNA、σ因子等以调节基因的表达。
1 细菌sRNA概述细菌sRNA即Small RNA或非编码RNA (noncoding RNA),是普遍存在于细菌中的一类长度在40−500 nt之间的RNA分子。目前发现的细菌sRNA主要位于基因间区[1],但也有位于编码基因5′和3′UTR区的报道[1]。
在20世纪就掀起了识别非编码sRNA的研究热潮[2-3]。早期的sRNA主要是通过总RNA放射标记法、免疫共沉淀法或者遗传分析方法发现的。随着生物信息学和分子生物学的发展,以及大量细菌基因组测序工作的完成,多种用于系统地发现和研究sRNA的方法被提出,主要包括:计算机预测、微阵列方法、鸟枪克隆方法以及SELEX (Systematic evolution of ligands by exponential enrichment)等方法。新近发展起来的高通量测序技术,如基因组Tiling分析、原核链特异性转录组测序等,为有效检测细菌sRNA并研究其特点提供了方便,大大加快了sRNA研究的进程。
目前,对sRNA的发掘多集中于致病菌,对有益菌sRNA的研究尚鲜有报道。
1.1 细菌sRNA的分类sRNA主要通过与靶基因5′UTR结合调控翻译或改变mRNA的稳定性,实现对靶标基因的表达调控。此外,sRNA还可以与蛋白质结合影响靶标蛋白质的生物活性。按照sRNA发挥生物学功能的形式,可以把细菌中的sRNA分为3种类型:(1)行使管家功能的sRNA,如4.5S RNA、tmRNA以及RNase P RNA;(2)与蛋白结合的调控sRNA,如6S RNA、CsrB、CsrC、RsmX、RsmY和RsmZ等;(3)与靶mRNA相互作用的调控sRNA,如SgrS、RyhB、MicA、OxyS等。
1.2 细菌sRNA的生理功能作为一种新型的环境压力应答调节元件,细菌sRNA可以随着生长和环境的改变而变化,每种环境胁迫因子至少和某一种sRNA相关,例如低温、营养缺乏或者应激等均可导致不同sRNA的表达[4]。与蛋白质调节因子相比,sRNA可以快速合成和降解,所以在细菌需要快速反应时,用sRNA进行调控比用蛋白质调控更为有利[5]。Geissmann等[6]对金黄色葡萄球菌(Staphylococcus aureus) sRNA的研究发现:大多数sRNA会随着氧气、温度、渗透压、pH值等生长环境的不同而发生改变。另据报道[7],sRNA的表达水平还会随着恶臭假单胞菌(Pseudomonas putida)生长温度的不同而发生变化。
细菌sRNA主要是通过感知生存条件的改变而对质粒复制、噬菌体发育、压力反应、群体感应、细菌毒性、碳代谢和铁的动态平衡调节等进行精细调控[8-9],它可将环境改变信号传导到细菌细胞内有关基因,通过上调或下调靶基因的表达来适应变化的环境。
在低温环境压力下,大肠杆菌(Escherichia coli)小调控RNA DsrA可以与rpoS mRNA的5′UTR通过碱基配对结合,激活RpoS蛋白的翻译。另一方面,DsrA还可保护rpoS mRNA,防止其被RNase E降解[10]。作为细菌的一个调控因子,RpoS启动一系列下游基因的表达,使细菌适应不利的环境,降低繁殖速度,进入稳定生长期。
E. coli MicA通过与群体感应系统PhoPQ的mRNA碱基配对,感应Mg2+浓度的变化,并与其他调控系统相互关联[11]。作为调控网络的一部分,E. coli 6S RNA调节一些关键的调控子,包括relA、crp和翻译装置,当营养缺乏时,放大全局响应[11]。
1.3 sRNA的应用作为一种重要的基因表达调控工具,sRNA在真核生物中得到了广泛应用。随着微生物中sRNA的不断发现及其调控机制的逐渐明确,新近开发的人工sRNA在微生物代谢工程方面也展现出了巨大优势。姚元锋等[12]通过对负调控大肠杆菌L-酪氨酸生物合成途径的碳贮藏调控因子csrA基因的sRNA进行人工设计和筛选,分析其对L-酪氨酸合成的影响。结果表明,所设计的sRNA能有效提高L-酪氨酸的合成。
VrrA是存在于霍乱弧菌(Vibrio cholerae)中的一种sRNA。Song等[13]研究发现,VrrA通过与rbmC转录物5′非翻译区结合负调控rbmC翻译,因此在V. cholerae中,可利用VrrA对生物被膜基质组分RbmC蛋白的合成进行调控。
鼠伤寒沙门菌(Salmonella typhimurium)的sRNA MicF[14],在Hfq蛋白协助下可调节脂多糖合成过程中起关键作用的类脂A修饰酶的合成,因此可以利用MicF来调节脂多糖的合成。
sRNA作为一种有效的代谢调节工具,在提高代谢产物产量以及微生物细胞工厂的构建上必将有着更广阔的应用前景。利用sRNA调控来避免环境条件对生物活性化合物合成的影响也不失为一个好的策略。
2 群体感应调控在细菌生长过程中,能够释放特定信号分子,使其感知菌群密度变化,当信号分子随细胞密度的增加积累到一定浓度阈值时,信号分子与受体蛋白结合,引起受体蛋白构象或基团的变化,进而启动一系列基因的表达,以调节菌体的群体行为,这种现象称为群体感应[15] (quorum sensing,QS),这类信号分子称为自诱导物(autoinducer,AI)。
QS系统是一个细菌与细菌在种内或者种间,通过信号分子彼此感知、交流、相互协调的系统,参与细菌生物发光、质粒转移、抗生素合成、细菌素合成[16]、生物被膜形成及毒力因子表达等许多重要生物学功能的调节。虽然不同物种之间的QS系统组分有很大差异,但所有已知的QS系统组分的功能相似。按QS系统内调节蛋白的组成不同可将QS系统分为LuxI/LuxR型、LuxS/AI-2型、LasI/LasR型、RhlI/RhlR型、GacS/GacA型、AgrC/AgrA型等多种类型[17-18];按QS信号分子的不同又可将其分为酰基高丝氨酸内酯介导型、AIP介导型、AI-2/LuxS型、AI-3/QseC型QS系统等。
群体感应调控是一个依据细胞密度来调节基因表达的过程,通常QS系统运用许多不同的基因调控机制来实现这一功能,如σ因子、sRNA调控等,使细菌能够适应环境并对环境变化快速做出响应[19]。
环境因子影响着AI分子的产生、释放及其稳定性[20],对群体感应的启动起着至关重要的作用。我们的研究发现,乳杆菌细菌素的合成受到QS调控[21-23],环境因子,如温度、pH、乙醇、NaCl等可以通过影响QS信号分子PlnA的诱导活性影响乳杆菌细菌素的合成[24],但具体机制尚不清楚。在许多QS调控中,AI分子对基因表达的启动是由多种sRNA决定的[25]。
细菌的QS系统存在许多反馈回路,然而这些反馈回路如何控制信号处理仍然还不十分清楚。Liu等[26]的研究结果表明,sRNA(Qrr)与AIs、LuxO、LuxU、LuxR相互交织组成调控网络,不同的反馈在调控过程中都起着类似开关的作用,对哈氏弧菌(V. harveyi)的信号传导进行精确控制。是否有sRNA参与了乳杆菌QS系统对细菌素合成的调控尚未见报道。
3 群体感应系统与sRNA相互作用调控基因表达通过对sRNA启动子区域的检测发现了感应调节子结合位点的存在[27-28],Reichenbach等则发现sRNA基因与QS系统编码基因共定位[29],Mandin等[30]通过筛选多拷贝sRNA启动子与lacZ报告基因融合载体差异表达基因文库也发现一些QS与sRNA相互关联,这都提示sRNA与QS系统的功能具有相关性。研究QS与sRNA的相互作用关系,揭示细菌代谢调控过程已成为广大科研人员的一个重点研究领域[19, 31]。
sRNA在QS调控过程中扮演着多种角色。越来越多的研究都表明sRNA可以和QS系统交互作用,形成广泛的调控网络,多方面多层次地调节细菌的多种生理功能。通常情况下,sRNA是QS系统和靶基因之间相互联系的桥梁和纽带,一些细菌的QS系统通过直接调控sRNA调控目的基因的表达,而另外一些细菌的QS系统则受到sRNA调控。
3.1 细菌QS系统通过调控sRNA调控靶基因表达sRNA是QS系统调控子的重要组成部分,越来越多的QS系统调控子中被证实存在sRNA[5]。E. coliK12已知的30个QS系统操纵子中就发现有6个含有sRNA。许多细菌的QS系统通过调控sRNA来调控靶基因的表达[19],如P. aeruginosa、V. harveyi、V. cholerae,这些QS系统的感应调节子通过与sRNA启动子的结合激活或抑制sRNA的表达,进而在转录后水平激活或抑制靶mRNA或蛋白质。
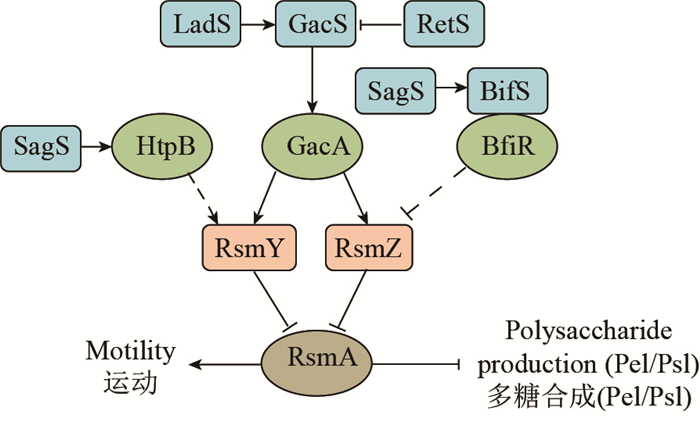
铜绿假单胞菌(P. aeruginosa)的QS系统(GacS/GacA)通过调控RsmY、RsmZ和RgsA三种sRNA的表达,调节RsmA蛋白所调控的胞外多糖基因Pel/Psl的表达[32-34]。GacS/GacA通过调控RsmY/RsmZ调控Pel/Psl表达见图 1。
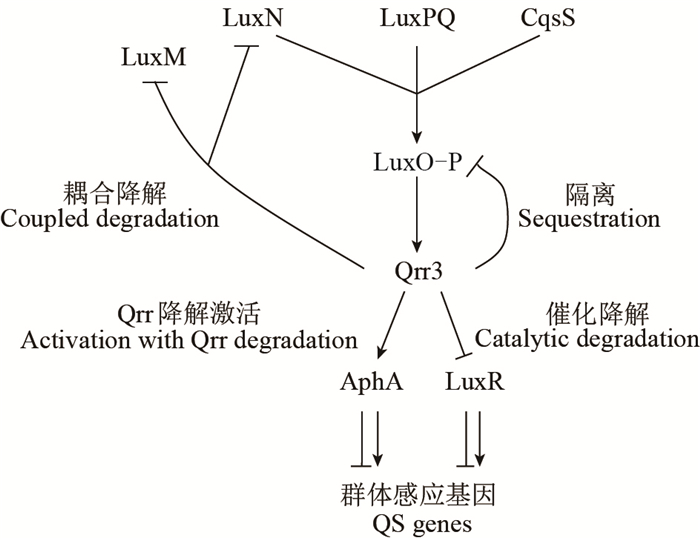
V. harveyi和V. cholerae的QS系统都包含一个sRNA(Qrr)通路,通过Qrr和蛋白分子伴侣Hfq来调节转录调控因子的表达。由于Qrr能够对细胞密度的变化快速做出响应,因此,弧菌利用Qrr抑制高细胞密度(HCD)相关基因的表达,激活与低细胞密度(LCD)相关基因的表达。对于V. harveyi,其磷酸化的QS组分LuxO可以通过激活Qrr间接激活与LCD相关主转录调节因子AphA的翻译,抑制与HCD相关的主转录调节因子LuxR的翻译[35] (图 2)。
|
| 图 2 V.harveyi QS系统通过激活Qrr调控靶基因 Figure 2 The V. harveyi quorum sensing system regulation of target genes through activation of Qrr |
|
|
维氏固氮菌(Azotobacter vinelandii)的QS系统GacS/GacA是合成PHB和胞外多糖藻酸盐的重要调控系统。GacA通过激活rsmZ1和rsmZ2来解除藻酸盐合成基因algD mRNA的翻译抑制[36]。
我们以Lactobacillus paraplantarum L-ZS9为研究对象,通过与Rfam基于结构性Infernal比对,找到255种候选sRNA;通过与sRNAMap、sRNATarBase以及SIPHT基于序列相似性的blast比对,获得7种候选sRNA,除sRNAPU0175外,其余6种均为新发现的sRNA。这些候选sRNA是否介导了乳杆菌细菌素合成的QS调控还有待于进一步验证。
研究sRNA介导的QS调控,对于阐明QS调控机理,进而对代谢过程进行精细调节具有十分重要的意义。
3.2 sRNA通过调控QS系统调控基因表达一些细菌的QS系统受到sRNA调控。在许多QS系统中,sRNA通过转录后调控机制直接或间接调节QS系统相关基因的表达。已有的研究[37]表明,QS系统双组分之一的感应调节蛋白及其同源蛋白多为细菌sRNA调控的目标基因。通过对一些Vibrio spp.的研究发现,V. harveyi和V. cholerae都包含5个sRNA:Qrr1、Qrr2、Qrr3、Qrr4和Qrr5,它们都独立转录,通过破坏QS组分的稳定性,在转录后水平抑制luxO[38]。
Rutherford等[39]的研究表明,在低细胞密度时,活化的sRNA促进V. cholera HapR (QS系统的感应调节蛋白组分)的降解;此外,sRNA还可通过激活毒力转录激活子AphA的表达来抑制HapR的表达,促进生物被膜的形成。
V. harveyi的sRNA (Qrr)可以结合到QS组分luxR的mRNA上,发挥调控作用,破坏其稳定性[40]。
CyaR(以前称作RyeE)则通过负调控nadE、yqaE、ompX和luxS等基因,使QS信号分子AI-2产量下降[38],进而调控基因表达。
我们采用IntaRNA软件(http://rna.informatik.uni-freiburg.de:8080/IntaRNA.jsp)预测分析发现,多个L. paraplantarum L-ZS9候选sRNA均以调控细菌素合成的QS组分HR_08945和HR_08955为靶基因(待发表),这些sRNA与其预测靶基因之间的对应关系以及它们之间的相互作用机制还有待进一步研究确认。
选取对QS系统起调控作用的sRNA进行研究,对于揭示环境条件造成的基因表达变化具有重要的意义。
3.3 sRNA通过对信息整合精确调控QS系统为了应对不断变化的生存环境,细菌进化出了一套复杂而有效的机制。QS系统是一个非常复杂的信号交流系统,该系统不仅整合来自信号分子的信息,而且也整合来自其他全局调控子的信息。sRNA作为全局调控的主要参与者[17, 41],通过转录后调节直接或间接调控基因的表达。越来越多的研究表明,QS系统对信息的整合是由非编码sRNA调控的。
Tu等[42]的研究发现V. harveyi的Qrr通过整合信息调控QS,使LuxR呈一定的梯度,进而实现对QS靶基因的差别调控。V. harveyi Qrr sRNAs参与4个负反馈调节通路,包括LuxR转录的自抑制、LuxR激活qrr (2−4)的表达、LuxO转录的自抑制以及Qrr sRNAs抑制luxO mRNA的翻译,从而使无论在低密度条件下还是在高密度条件下QS反应均保持高度忠实[40]。
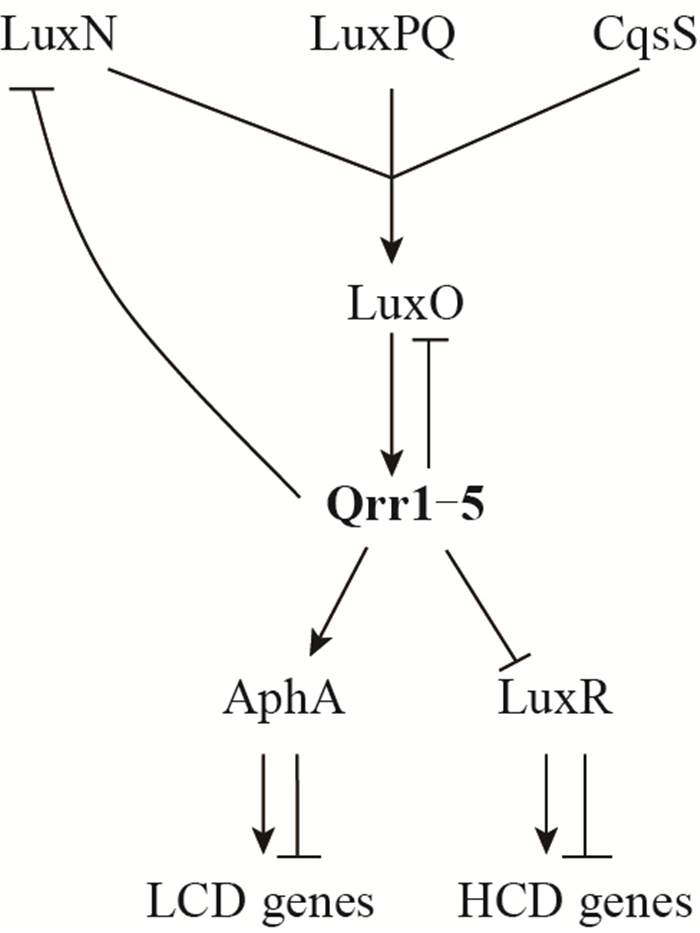
Feng等[43]的研究表明,V. harveyi Qrr3整合信息,运用4种不同的机制调控其靶标(图 3),确保QS调控处于最佳。
在已报道的大多数QS系统中,sRNA介导的调控通过多种功能性sRNA来完成,这些sRNA可能会响应不同的环境信号,因此,sRNA介导的调控有助于QS系统的弹性调控。sRNAs可以把来自环境的多种信息整合为一个单一的信号输出,即QS调控子调控[5],从而更有效地对靶基因表达进行调控。sRNAs可以同时作为多个靶基因的全局调控子,协调多个代谢过程。sRNAs对于细菌数量、细菌群体的种类、环境信号、代谢状况等信息的整合,在调控细菌群体行为时尤为重要[5]。
sRNA与QS系统相互作用的研究多集中在革兰氏阴性细菌,sRNA与革兰氏阳性菌QS系统相互作用方面的研究仅见于S. aureus[5]、李斯特菌、链球菌[44]等少数致病菌或有害菌,sRNA与乳杆菌等有益菌QS系统交互作用的研究则鲜见报道。产细菌素乳杆菌是一类天然食品防腐保鲜剂产生菌,其细菌素的合成受到温度、pH、盐浓度以及应激等多种环境条件的影响,鉴于sRNA多作为环境胁迫应答的调节元件,乳杆菌sRNA如何与QS组分组成调控网络对细菌素的合成进行精细调控是我们关注的重点。
识别响应环境条件变化并与QS调控相关的sRNA,研究其在基因表达中对环境信号的整合作用,必将为研究环境条件变化造成的基因表达波动提供大量有用的信息。
4 展望基于sRNA与QS系统相互作用研究现状及存在的问题,我们认为sRNA与QS系统相互作用的研究工作可从以下几个方面开展:(1)继续寻找与QS相关的新的细菌sRNA,尤其是与有益菌QS相关的sRNA。目前虽然已发现多种sRNA,但这些sRNA多与有害菌或致病菌相关,与有益菌相关的sRNA则鲜有报道,研究sRNA在有益菌代谢中的调控作用,对于调控代谢过程、提高生物活性代谢产物的产量具有重要意义。(2)由于sRNA的表达受环境影响较大,某些sRNA只在特定的条件下表达,因此,寻找在特定环境压力下表达的sRNA,研究其在代谢调控中的作用,对于揭示代谢产物的合成调控机理具有重要意义;另一方面,研究环境因子如何调节sRNA表达也一直是这一领域的研究热点。(3)通过生物信息学技术预测sRNA的靶标基因或靶结合位点,分析其可能的调控作用,继续研究sRNA如何调节基因表达,进一步通过构建sRNA突变株和互补株,分析野生株、突变株和互补株中靶标mRNA的表达变化,验证sRNA调控功能进而研究其具体作用机制。(4)研究sRNA与QS系统交互作用,揭示环境条件对代谢产物合成的影响机制。
相信随着分子生物学实验技术的发展和对sRNA分子特性研究的深入,越来越多的sRNA分子将会被发现和研究,必将有利于推进sRNA分子参与转录后加工及翻译调控等各个生命过程的研究。研究sRNA与QS系统的交互作用,揭示QS系统响应环境条件变化的基因表达调控机制,对于研究环境条件造成的代谢过程的波动,以便于更精准、更快速地调节代谢过程都具有极为重要的意义。
| [1] | Hershberg R, Altuvia S, Margalit H. A survey of small RNA-encoding genes in Escherichia coli[J]. Nucleic Acids Research , 2003, 31 (7) : 1813–1820. DOI:10.1093/nar/gkg297 |
| [2] | Massé E, Majdalani N, Gottesman S. Regulatory roles for small RNAs in bacteria[J]. Current Opinion in Microbiology , 2003, 6 (2) : 120–124. DOI:10.1016/S1369-5274(03)00027-4 |
| [3] | Wassarman KM. Small RNAs in bacteria: diverse regulators of gene expression in response to environmental changes[J]. Cell , 2002, 109 (2) : 141–144. DOI:10.1016/S0092-8674(02)00717-1 |
| [4] | Repoila F, Gottesman S. Temperature sensing by the dsrA promoter[J]. Bacteriology , 2003, 185 (22) : 6609–6614. DOI:10.1128/JB.185.22.6609-6614.2003 |
| [5] | Bejerano-Sagie M, Xavier KB. The role of small RNAs in quorum sensing[J]. Current Opinion in Microbiology , 2007, 10 (2) : 189–198. DOI:10.1016/j.mib.2007.03.009 |
| [6] | Geissmann T, Chevalier C, Cros MJ, et al. A search for small noncoding RNAs in Staphylococcus aureus reveals a conserved sequence motif for regulation[J]. Nucleic Acids Research , 2009, 37 (21) : 7239–7257. DOI:10.1093/nar/gkp668 |
| [7] | Fonseca P, Moreno R, Rojo F. Pseudomonas putida growing at low temperature shows increased levels of CrcZ and CrcY sRNAs, leading to reduced Crc-dependent catabolite repression[J]. Environmental Microbiology , 2013, 15 (1) : 24–35. DOI:10.1111/emi.2013.15.issue-1 |
| [8] | Romby P, Vandenesch F, Wagner EGH. The role of RNAs in the regulation of virulence-gene expression[J]. Current Opinion in Microbiology , 2006, 9 (2) : 229–236. DOI:10.1016/j.mib.2006.02.005 |
| [9] | Storz G, Altuvia S, Wassarman KM. An abundance of RNA regulators[J]. Annual Review of Biochemistry , 2005, 74 : 199–217. DOI:10.1146/annurev.biochem.74.082803.133136 |
| [10] | McCullen CA, Benhammou JN, Majdalani N, et al. Mechanism of positive regulation by DsrA and RprA small noncoding RNAs: pairing increases translation and protects rpoS mRNA from degradation[J]. Journal of Bacteriology , 2010, 192 (21) : 5559–5571. DOI:10.1128/JB.00464-10 |
| [11] | Storz G, Vogel J, Wassarman KM. Regulation by small RNAs in bacteria: expanding frontiers[J]. Molecular Cell , 2011, 43 (6) : 880–891. DOI:10.1016/j.molcel.2011.08.022 |
| [12] | Yao YF, Zhao Y, Zhao GR. Artificial sRNAs silencing csrA to optimize the production of L-tyrosine in Escherichia coli[J]. China Biotechnology , 2013, 33 (8) : 60–65. (in chinese) 姚元锋, 赵莹, 赵广荣. 人工sRNAs沉默csrA基因以优化大肠杆菌生产L-酪氨酸[J]. 中国生物工程杂志 , 2013, 33 (8) : 60–65. |
| [13] | Song TY, Sabharwal D, Wai SN. VrrA mediates Hfq-dependent regulation of OmpT synthesis in Vibrio cholerae[J]. Journal of Molecular Biology , 2010, 400 (4) : 682–688. DOI:10.1016/j.jmb.2010.05.061 |
| [14] | Waters LS, Storz G. Regulatory RNAs in bacteria[J]. Cell , 2009, 136 (4) : 615–628. DOI:10.1016/j.cell.2009.01.043 |
| [15] | Zhang XM, Li PL. Quorum sensing in class II bacteriocin-producing lactic acid bacteria and its application-a review[J]. Acta Microbiologica Sinica , 2011, 51 (9) : 1152–1157. (in chinese) 张香美, 李平兰. 产Ⅱ类细菌素乳酸菌群体感应及其应用[J]. 微生物学报 , 2011, 51 (9) : 1152–1157. |
| [16] | Rizzello CG, Filannino P, di Cagno R, et al. Quorum-sensing regulation of constitutive plantaricin by Lactobacillus plantarum strains under a model system for vegetables and fruits[J]. Applied and Environmental Microbiology , 2014, 80 (2) : 777–787. DOI:10.1128/AEM.03224-13 |
| [17] | Lalaouna D, Fochesato S, Sanchez L, et al. Phenotypic switching in Pseudomonas brassicacearum involves GacS-and GacA-dependent Rsm small RNAs[J]. Applied and Environmental Microbiology , 2012, 78 (6) : 1658–1665. DOI:10.1128/AEM.06769-11 |
| [18] | Rutherford ST, Bassler BL. Bacterial quorum sensing: its role in virulence and possibilities for its control[J]. Cold Spring Harbor Perspectives in Medicine , 2012, 2 (11) : a012427. |
| [19] | Hunter GAM. Mediating bacterial communication: the role of small RNA in quorum sensing[D]. Utah: Doctoral Dissertation of the University of Utah, 2013 |
| [20] | Horswill AR, Stoodley P, Stewart PS, et al. The effect of the chemical, biological, and physical environment on quorum sensing in structured microbial communities[J]. Analytical and Bioanalytical Chemistry , 2007, 387 (2) : 371–380. DOI:10.1007/s00216-006-0720-y |
| [21] | Zhang XM, Shang N, Zhang X, et al. Role of plnB gene in the regulation of bacteriocin production in Lactobacillus paraplantarum L-XM1[J]. Microbiological Research , 2013, 168 (5) : 305–310. DOI:10.1016/j.micres.2012.11.008 |
| [22] | Zhang XM, Liu GR, Zhao B, et al. New insight into the regulation of classⅡbacteriocin production by quorum sensing in Lactobacillus pentosus 31-1[J]. African Journal of Microbiology Research , 2012, 6 (15) : 3582–3588. |
| [23] | Zhang XM, Li PL. Bioinformatics analysis of the plnBCD gene from Lactobacillus paraplantarum L-XM1[J]. Journal of China Agricultural University , 2014, 19 (3) : 175–179. (in chinese) 张香美, 李平兰. 类植物乳杆菌L-XM1 plnBCD基因的生物信息学分析[J]. 中国农业大学学报 , 2014, 19 (3) : 175–179. |
| [24] | Zhang XM, Li PL. Effects of PlnA on bacteriocin production in Lactobacillus paraplantarum: influence of environmental factors[J]. Microbiology China , 2013, 40 (9) : 1624–1630. (in chinese) 张香美, 李平兰. 环境因素对PlnA诱导类植物乳杆菌产生细菌素效果的影响[J]. 微生物学通报 , 2013, 40 (9) : 1624–1630. |
| [25] | Lenz DH, Mok KC, Lilley BN, et al. The small RNA chaperone Hfq and multiple small RNAs control quorum sensing in Vibrio harveyi and Vibrio cholerae[J]. Cell , 2004, 118 (1) : 69–82. DOI:10.1016/j.cell.2004.06.009 |
| [26] | Liu X, Zhou PP, Wang RQ. Small RNA-mediated switch-like regulation in bacterial quorum sensing[J]. IET Systems Biology , 2013, 7 (5) : 182–187. DOI:10.1049/iet-syb.2012.0059 |
| [27] | Moon K, Gottesman S. A PhoQ/P-regulated small RNA regulates sensitivity of Escherichia coli to antimicrobial peptides[J]. Molecular Microbiology , 2009, 74 (6) : 1314–1330. DOI:10.1111/mmi.2009.74.issue-6 |
| [28] | G pel Y, Lüttmann D, Heroven AK, et al. Common and divergent features in transcriptional control of the homologous small RNAs GlmY and GlmZ in Enterobacteriaceae[J]. Nucleic Acids Research , 2011, 39 (4) : 1294–1309. DOI:10.1093/nar/gkq986 |
| [29] | Reichenbach B, GÖpel Y, GÖrke B. Dual control by perfectly overlappingσ54-andσ70-promoters adjusts small RNA GlmY expression to different environmental signals[J]. Molecular Microbiology , 2009, 74 (5) : 1054–1070. DOI:10.1111/mmi.2009.74.issue-5 |
| [30] | Mandin P, Gottesman S. Integrating anaerobic/aerobic sensing and the general stress response through the ArcZ small RNA[J]. The EMBO Journal , 2010, 29 (18) : 3094–3107. DOI:10.1038/emboj.2010.179 |
| [31] | Zhao XN. Non-coding small RNAs regulate multiple mRNA targets to control the Vibrio cholerae quorum sensing response[D]. Georgia: Doctoral Dissertation of Georgia Institute of Technology, 2013 |
| [32] | GÖpel Y, GÖrke B. Rewiring two-component signal transduction with small RNAs[J]. Current Opinion in Microbiology , 2012, 15 (2) : 132–139. DOI:10.1016/j.mib.2011.12.001 |
| [33] | Brencic A, McFarland KA, McManus HR, et al. The GacS/GacA signal transduction system of Pseudomonas aeruginosa acts exclusively through its control over the transcription of the RsmY and RsmZ regulatory small RNAs[J]. Molecular Microbiology , 2009, 73 (3) : 434–445. DOI:10.1111/mmi.2009.73.issue-3 |
| [34] | Chambers JR, Sauer K. Small RNAs and their role in biofilm formation[J]. Trends in Microbiology , 2013, 21 (1) : 39–49. DOI:10.1016/j.tim.2012.10.008 |
| [35] | Shao Y, Feng LH, Rutherford ST, et al. Functional determinants of the quorum-sensing non-coding RNAs and their roles in target regulation[J]. The EMBO Journal , 2013, 32 (15) : 2158–2171. DOI:10.1038/emboj.2013.155 |
| [36] | Hernandez-Eligio A, Moreno S, Castellanos M, et al. RsmA post-transcriptionally controls PhbR expression and polyhydroxybutyrate biosynthesis in Azotobacter vinelandii[J]. Microbiology , 2012, 158 (Pt 8) : 1953–1963. |
| [37] | Marx P, Nuhn M, Kovács M, et al. Identification of genes for small non-coding RNAs that belong to the regulon of the two-component regulatory system CiaRH in Streptococcus[J]. BMC Genomics , 2010, 11 (1) : 661. DOI:10.1186/1471-2164-11-661 |
| [38] | Carter KK, Valdes JJ, Bentley WE. Pathway engineering via quorum sensing and sRNA riboregulators—interconnected networks and controllers[J]. Metabolic Engineering , 2012, 14 (3) : 281–288. DOI:10.1016/j.ymben.2011.11.006 |
| [39] | Rutherford ST, van Kessel JC, Shao Y, et al. AphA and LuxR/HapR reciprocally control quorum sensing in vibrios[J]. Genes & Development , 2011, 25 (4) : 397–408. |
| [40] | Tu KC, Long T, Svenningsen SL, et al. Negative feedback loops involving small regulatory RNAs precisely control the Vibrio harveyi quorum-sensing response[J]. Molecular Cell , 2010, 37 (4) : 567–579. DOI:10.1016/j.molcel.2010.01.022 |
| [41] | Beisel CL, Storz G. Base pairing small RNAs and their roles in global regulatory networks[J]. FEMS Microbiology Reviews , 2010, 34 (5) : 866–882. DOI:10.1111/j.1574-6976.2010.00241.x |
| [42] | Tu KC, Bassler BL. Multiple small RNAs act additively to integrate sensory information and control quorum sensing in Vibrio harveyi[J]. Genes & Development , 2007, 21 (2) : 221–233. |
| [43] | Feng LH, Rutherford ST, Papenfort K, et al. A qrr noncoding RNA deploys four different regulatory mechanisms to optimize quorum-sensing dynamics[J]. Cell , 2015, 160 (1/2) : 228–240. |
| [44] | Xia L, Xia W, Li SH, et al. Identification and expression of small non-coding RNA, L10-Leader, in different growth phases of Streptococcus mutans[J]. Nucleic Acid Therapeutics , 2012, 22 (3) : 177–186. |
 2016, Vol. 43
2016, Vol. 43