扩展功能
文章信息
- 周会先, 王艳梅, 张宗啸, 周红伟, 赵一夫, 牛红星
- ZHOU Hui-Xian, WANG Yan-Mei, ZHANG Zong-Xiao, ZHOU Hong-Wei, ZHAO Yi-Fu, NIU Hong-Xing
- 普氏蹄蝠胃肠道菌群的多样性
- Microbial diversity in the gastrointestinal tract of Hipposideros pratti
- 微生物学通报, 2016, 43(8): 1739-1745
- Microbiology China, 2016, 43(8): 1739-1745
- DOI: 10.13344/j.microbiol.china.150654
-
文章历史
- 收稿日期: 2015-09-02
- 接受日期: 2015-12-07
- 优先数字出版日期(www.cnki.net): 2016-01-06
蝙蝠是翼手目动物的俗称,它们捕食农林业害虫、传播种子和花粉,对于维持生态系统的平衡起着重要作用[1-2]。蝙蝠与人类的关系非常密切,是人类生产与生活中必须重视的一大特殊类群。长期以来,人们利用蝙蝠作为中药,治疗哮喘、肾虚等疾病[3]。研究发现,虽然蝙蝠对生态系统和人类生活具有积极的作用,但它也是许多人兽共患病病原体的携带者,可引起多种人兽共患病[4-5]。迄今为止,已从蝙蝠体内分离出80多种病毒,其中一些病毒是重大人兽共患疾病的病原体[6]。除此之外,蝙蝠也是许多致病菌的自然宿主[4]。众所周知,动物的胃肠道内存在大量微生物,它们与动物的生理功能密切相关,是最密集的微生物生态系统[7]。因此,蝙蝠的胃肠道是研究蝙蝠体内细菌多样性的最佳部位。目前有关蝙蝠胃肠道细菌的研究较少,Graves (1988)[8]、Anand (2004)[9]、Mühldorfer (2010)[10]、Reyes (2011)[11]和Daniel (2013)[12]等从犬蝠(Cymopteruss phinx)、短耳犬蝠(Cymopteruss brachyotis)、印度狐蝠(Pteropus giganteus)、马来大狐蝠(Pteropus vampyrus)、大鼠耳蝠(Myotis myotis)、伏翼(Pipistrellus pipistrellus)等蝙蝠体内分离出腐败梭菌(Clostridium septicum)、金黄色葡萄球菌(Stapylococcus aureus)、奇异变形杆菌(Protues mirabilis)、弗氏柠檬酸杆菌(Citrobacter freundii)、肺炎克雷伯菌(Klebsiella pneumonia)、铜绿假单胞菌(Pseudomonas aeruginosa)、阴沟肠杆菌(Enterobacter cloacae)、假结核耶尔森氏菌(Yersinia pseudotuberculosis)和小肠结肠炎耶尔森氏菌(Y. enterocolitica)等20多种人类致病菌或条件致病菌,迄今为止,国内外关于蝙蝠胃肠道菌群的研究仅限于实验室可培养微生物种类,而系统剖析蝙蝠胃肠道菌群结构的研究尚未见报道。
实验室可培养微生物的种类非常有限[13],而高通量测序技术可使每个样品的测序量达到普通测序的几百倍,它极大地促进了对环境中不可培养微生物以及痕量菌的研究[14]。MiSeq高通量测序技术集中了Illumina HiSeq 2 500和Roche 454的优点,可对多个样品的多个可变区同时测序,而且在测序速度和测序通量上都有进一步提升,目前该方法已在微生物多样性研究中得到了广泛的运用[15-16]。为全面地剖析普氏蹄蝠胃肠道菌群的结构,采用MiSeq高通量测序技术对普氏蹄蝠胃肠道细菌的16S rRNA基因V1−V2区基因进行了测序和分析,为蝙蝠胃肠道微生物的研究以及预防人和蝙蝠共患疾病的发生提供基础资料。
普氏蹄蝠在国内分布于广西、四川、福建、陕西、江西、云南等11个省,为洞栖型蝙蝠[17-18]。近年来,随着旅游业的发展,许多普氏蹄蝠栖息的洞穴被开发成旅游景点,这既破坏了蝙蝠的生存环境,也可能加速蝙蝠体内一些病原体对人体的适应,从而传播给人类,这已引起了人们的高度关注[19]。因此,研究普氏蹄蝠胃肠道菌群的结构,尤其是致病菌的种类对预防人与蝙蝠共患疾病的发生意义重大。
1 材料与方法 1.1 材料2014年9月,在河南省南阳市南召县乔端镇仙人洞(N112°06',E33°34'),通过洞内架设雾网,采集到5只普氏蹄蝠,蝙蝠物种的鉴定依据《中国兽类野外手册》[14],将蝙蝠活体带回实验室备用。
1.2 方法 1.2.1 分离胃肠内容物: 将5只蝙蝠用过量氯仿麻醉处死,用70%的酒精浸泡进行体表消毒,无菌水漂洗后,在无菌条件下取出胃和肠(包括小肠、大肠)的内容物,分别放入无菌的5 mL离心管,将5只蝙蝠胃和肠的内容物分别混合均匀备用。 1.2.2 基因组DNA抽提: 基因组DNA提取使用Stool DNA Kit试剂盒(购自广州飞扬生物工程有限公司),提取步骤按说明书严格进行。 1.2.3 : 扩增及测序:以提取的总DNA为模板,以8F (5'-AGAGTTTGATYMTGGCTCAG-3')和338R (5'-TGCTGCCTCCCGTAGGAGT-3')为引物,扩增细菌16S rRNA基因V1−V2区。PCR扩增体系(50μL):2×Master Mix (购自康为世纪生物科技有限公司),25μL,引物各1μL (浓度为20μmol/L),模板2μL,灭菌双蒸水21μL。PCR扩增条件:98℃30 s;98℃10 s,50℃30 s,72℃30 s,20个循环;72℃7 min;4℃保存[20]。2%琼脂糖凝胶电泳检测,采用快速琼脂糖凝胶DNA回收试剂盒(购自康为世纪生物科技有限公司)对目的条带胶回收纯化。将纯化后的PCR产物送至江苏中宜金大分析检测有限公司进行测序,测序平台为Illumina MiSeq。 1.2.4 生物信息分析: 应用MG-RAST V3.3.6中的rRNA Detection pipeline进行数据分析,原始数据经过过滤处理,得到优化序列,对优化序列进行操作分类单元(Operational taxonomy unit,OTU)聚类分析和分类学分析。基于OTU聚类分析结果,对OTU进行多样性指数分析以及测序深度的检测;基于分类学信息,在门和属水平上进行群落结构的统计分析。计算在97%的相似水平上每个样本的OTU数量,分析内容包括:优化序列统计、多样性指数分析和高通量热图绘制。 2 结果与分析 2.1 胃肠道菌群样品测序产物丰度及多样性分析胃中菌群样品经测序共获得144 998条原始序列和48 332条有效序列,测序覆盖率为99.02%,说明胃内容物样品中99.02%的细菌已经被克隆,被测序的物种包含了胃中绝大多数细菌的种类,能够代表胃中物种的多样性,97%的相似性归并后得到894个OTUs。肠道菌群样品经测序共获得275 274条原始序列和91 758条有效序列,测序覆盖率为98.94%,说明该肠道内容物样品中98.94%的细菌已经被克隆,被测序的物种包含肠道中绝大多数细菌种类,能够代表肠道中物种的多样性,97%的相似性归并后得到756个OTUs (表 1)。
| 样品名称 Sample name |
优化序列 After trim |
测序深度 Sample depth |
操作分类单元 OTUs |
Chao指数 Chao index |
香农指数 Shannon index |
ACE指数 ACE index |
辛普森指数 Simpson index |
测序覆盖率 Coverage (%) |
| 肠Intestine | 91 758 | 48 332 | 756 | 2 051 | 2.407 | 3 556 | 0.151 | 98.94 |
| 胃Stomach | 48 332 | 48 332 | 894 | 1 490 | 2.405 | 2 221 | 0.168 | 99.02 |
胃中菌群丰度指数Chao指数(1 490)和ACE指数(2 221)分别低于肠道菌群的Chao指数(2 051)和ACE (3 556)指数;Shannon多样性指数(2.405)低于肠道(2.407),Simpson多样性指数(0.168)大于肠道(0.151),表明胃中菌群丰度和多样性均低于肠道菌群(表 1)。
2.2 高通量测序对胃肠道细菌群落结构的分析 2.2.1 门水平上胃肠道菌群结构: 胃肠中的细菌主要分布在6个门,分别是厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)、软膜菌门(Tenericutes)、放线菌门(Actinobacteria)和未分类细菌(Unclassified),这6个门在胃肠道中均有分布,但各门在胃肠道中所占比例不相同,其中占优势的主要是变形菌门(胃中占56.4%,肠中占46.0%)和厚壁菌门(胃中占40.7%,肠中占49.2%) (表 2)。胃肠道中95%以上的细菌属于变形菌门和厚壁菌门,但这两个优势菌门在胃肠道中所占的比例存在差异,其他类型的细菌在胃肠道中均只占较少的部分。| 门Phylum | 肠Intestine (%) | 胃Stomach (%) |
| 拟杆菌门 Bacteroidetes |
0.6 | 1.1 |
| 厚壁菌门 Firmicutes |
49.2 | 40.7 |
| 变形菌门 Proteobacteria |
46.0 | 56.4 |
| 软膜菌门 Tenericutes |
1.3 | - |
| 放线菌门 Actinobacteria |
1.4 | 0.3 |
| 未分类细菌 Unclassified |
1.1 | 0.5 |
| 其他Others | 0.4 | 1.0 |
| 注:其他:比例较少的细菌;−:含量很少. Note: others: rare genus. −: very low bacteria content. | ||
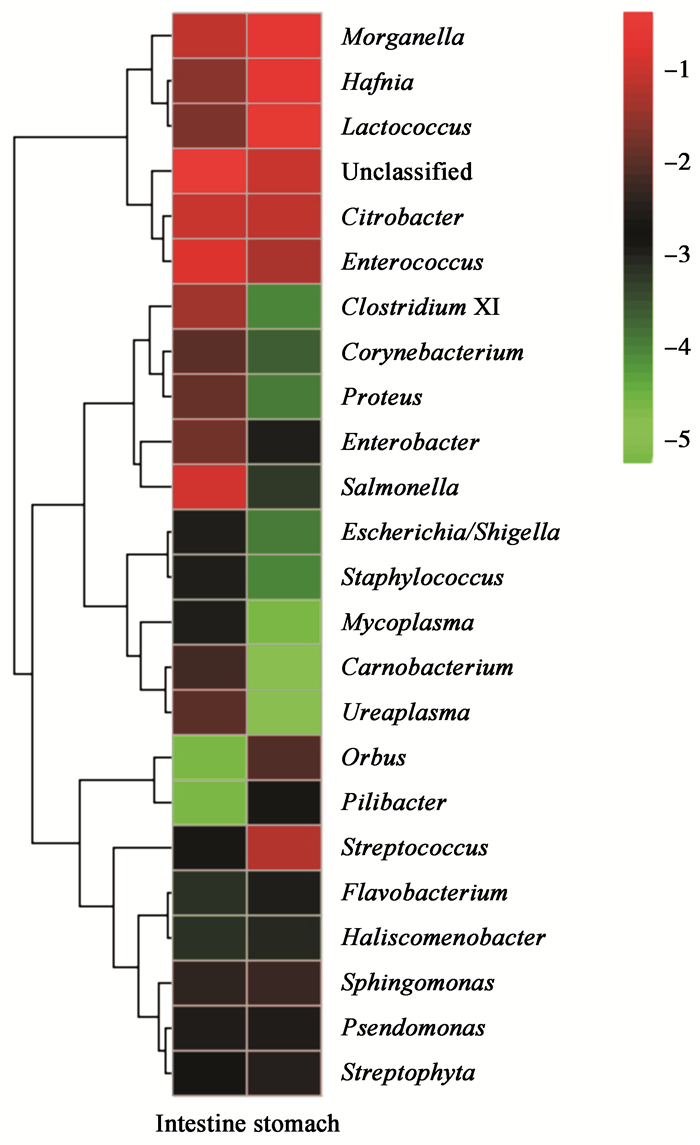
|
| 图 1 高通量分析属水平上的分类情况 Figure 1 The classification of the High-throughput sequencing analysis of genus level |
|
|
| 属Genus | 肠Intestine (%) | 胃Stomach (%) |
| 乳球菌属 Lactococcus |
2.0 | 26.1 |
| 哈夫尼菌属 Hafnia |
2.5 | 21.0 |
| 摩根菌属 Morganella |
7.3 | 20.1 |
| 柠檬酸杆菌属 Citrobacter |
9.9 | 6.9 |
| 肠球菌属 Enterococcus |
15.2 | 5.0 |
| 沙门氏菌属 Salmonella |
12.7 | 0.1 |
| 梭杆菌属 Clostridium XI |
4.2 | - |
| 巴氏杆菌属 Pasteurella |
0.0 | - |
| 支原体属 Mycoplasma |
0.1 | 0.0 |
| 其他Others | 46.1 | 20.8 |
| 注:其他:未能确定分类水平及比例较少的菌属;−:含量很少. Note: Others: all the unclassified and rare genus. −:very low bacteria content. | ||
在哺乳动物中,大多数的肠道微生物能把复杂的碳水化合物分解成简单的多糖,为生物体提供营养和能量[21]。蝙蝠食性不同肠道中的细菌种类也有所差异,如有学者从以植物的果实和花蜜等为食的印度狐蝠(Pteropus giganteus)肠道中分离出了能分解纤维素和木聚糖的细菌,而食虫蝙蝠大耳小蹄蝠(Hipposideros fulvus)肠道中不含有能分解纤维素和木聚糖的细菌[10]。Whitaker等(2004)[22]从印度9种食虫蝙蝠肠道中分离出能产生几丁质酶的细菌,普氏蹄蝠是典型的食虫蝙蝠,这些菌属在普氏蹄蝠肠道中也被发现。普氏蹄蝠胃肠道中主要有24个菌属,其中束缚杆菌属、鞘氨醇单胞菌属、Orbus、Pilibacter和Streptophyta的细菌首次在蝙蝠胃肠道中被发现,而曾有报道柠檬酸杆菌属、肠杆菌属、哈夫尼菌属、摩根菌属、假单胞菌属和链球菌属存在于其他食虫蝙蝠胃肠道中[23];Daniel等(2013)[12]从果蝠(Cynopterus brachyotis)的胃肠道中分离出了肠杆菌属、沙雷氏菌属、埃希氏菌属、肠球菌属和泛生菌属(pantoea)。其中除泛生菌属外,其余各菌属在普氏蹄蝠胃肠道中均有发现。由此可以看出,不同种类的蝙蝠,其胃肠道中菌群的组成不同,这可能与宿主自身的生理特性有关,也有可能是实验方法不同造成的差异。
普氏蹄蝠胃肠道中有些菌属为人类致病菌或条件致病菌,如:黄杆菌属细菌(图 1)可引起新生儿脑膜炎[24],该细菌在食血蝙蝠(Desmodus rotundus)胃中也曾被发现[25]。梭杆菌属细菌可引起出血性痢疾[26-27],巴氏杆菌属细菌可引起局部或全身感染[28-29],部分细菌可导致肺炎和皮肤脓肿[30],支原体可引起肺炎、泌尿生殖道感染、不育及自然流产,也可作为AIDS和肿瘤的协同致病因子[31]。胃中所占百分比较高的乳球菌属细菌可引起人类败血症、心内膜炎和骨髓炎等[32];哈夫尼亚菌属的细菌能引起人体腹泻、败血症等[33-34];摩根菌属细菌可致人体泌尿道感染和皮肤软组织及伤口感染[35],并可导致食物中毒或腹泻[36];肠道中所占百分比较高的肠球菌属细菌常栖居于人和动物肠道中,为医院感染的重要病原菌,并可引起心内膜炎、伤口感染、胆囊炎、尿道炎、膀脱炎、肾盂炎和脑膜炎等疾患[37];沙门氏菌作为一种致病菌频繁地被报道,常可引起人和动物肠胃炎[38-39];而柠檬酸杆菌属细菌常可引起人体呼吸道和泌尿系统感染[40]。普氏蹄蝠是典型的洞穴居住者,且携带有大量的人类致病菌,因此,有普氏蹄蝠居住的洞穴应禁止开发旅游,以防止人类和蝙蝠之间疾病的传播。
致谢: 感谢河南师范大学生命科学学院杨清香教授在论文写作过程中给予的指导与帮助;也感谢团队所有成员在标本采集及实验过程中给予的帮助。| [1] | Cox PA, Elmqvist T, Pierson ED, et al. Flying foxes as pollinators and seed dispersers in Pacific island ecosystems[C]//Wilson DE, Graham G. Pacific island flying foxes: proceedings of an international conservation conference. Washington, DC: U. S. Forest Service Biological Report, 1992: 18-23 |
| [2] | Fleming TH. Foraging strategies of plant-visiting bats[M]//Kunz TH. Foraging Strategies of Plant. New York: Plenum Press, 1982: 278-325 |
| [3] | Fujita MS, Tuttle MD. Flying foxes (Chiroptera: Pteropodidae): threatened animals of key ecological and economic importance[J]. Conservation Biology , 1991, 5 (4) : 455–463. DOI:10.1111/cbi.1991.5.issue-4 |
| [4] | Calisher CH, Childs JE, Field HE, et al. Bats: important reservoir hosts of emerging viruses[J]. Clinical Microbiology Reviews , 2006, 19 (3) : 531–545. DOI:10.1128/CMR.00017-06 |
| [5] | Wong S, Lau S, Woo P, et al. Bats as a continuing source of emerging infections in humans[J]. Reviews in Medical Virology , 2007, 17 (2) : 67–91. DOI:10.1002/rmv.v17:2 |
| [6] | Li WD, Liang GD, Liang B, et al. Progress of research on the viruses in bats[J]. Virologica Sinica , 2004, 19 (4) : 418–425. (in chinese) 李文东, 梁国栋, 梁冰, 等. 蝙蝠携带病毒的研究进展[J]. 中国病毒学 , 2004, 19 (4) : 418–425. |
| [7] | Xu B, Yang FY, Mu YL, et al. Molecular ecology applied to gastrointestinal tract microorganisms[J]. Microbiology China , 2014, 41 (1) : 136–145. |
| [8] | Graves SR, Kennelly-Merrit SA, Tidemann CR, et al. Antibiotic-resistance patterns of enteric bacteria of wild mammals on the Krakatau Islands and West Java, Indonesia[J]. Philosophical Transactions of the Royal Society of London. Series B: Biological Sciences , 2014, 41 (1) : 136–145. (in chinese) 许波, 杨富亚, 慕跃林, 等. 胃肠道微生物及其分子生态学技术研究进展[J]. 微生物学通报 , 2014, 41 (1) : 136–145. |
| [9] | Anand AAP, Sripathi K. Digestion of cellulose and xylan by symbiotic bacteria in the intestine of the Indian flying fox (Pteropus giganteus)[J]. Comparative Biochemistry and Physiology Part A: Molecular & Integrative Physiology , 2004, 139 (1) : 65–69. |
| [10] | Mühldorfer K, Wibbelt G, Haensel J, et al. Yersinia species isolated from bats, Germany[J]. Emerging Infectious Diseases , 2010, 16 (3) : 578–580. DOI:10.3201/eid1603.091035 |
| [11] | Reyes AWB, Rovira HG, Masangkay JS, et al. Polymerase chain reaction assay and conventional isolation of Salmonella spp. from Philippine bats[J]. Acta Scientiae Veterinariae , 2011, 39 (1) : 947. |
| [12] | Daniel DS, Ng YK, Chua EL, et al. Isolation and identification of gastrointestinal microbiota from the short-nosed fruit bat Cynopterus brachyotis brachyotis[J]. Microbiological Research , 2013, 168 (8) : 485–496. DOI:10.1016/j.micres.2013.04.001 |
| [13] | Li XR, Gong FM, Li J, et al. Active bacterial community diversity in traditional fermented soybean in Yunnan using mixed culture and barcoded pyrosequencing[J]. Food Science , 2014, 35 (7) : 90–94. (in chinese) 李晓然, 龚福明, 李洁, 等. 基于混合培养和高通量测序分析云南传统发酵豆豉中活性细菌群落[J]. 食品科学 , 2014, 35 (7) : 90–94. |
| [14] | Wang XC, Yang ZR, Wang M, et al. High-throughput sequencing technology and its application[J]. China Biotechnology , 2012, 32 (1) : 109–114. (in chinese) 王兴春, 杨致荣, 王敏, 等. 高通量测序技术及其应用[J]. 中国生物工程杂志 , 2012, 32 (1) : 109–114. |
| [15] | Jiao JK, Mo BH. Bacteria diversity of cheeses with high coverage by Illumina MiSeq Platform[J]. China Brewing , 2014, 33 (5) : 34–38. (in chinese) 焦晶凯, 莫蓓红. Illumina MiSeq平台高覆盖率测定干酪中的细菌微生物多样性[J]. 中国酿造 , 2014, 33 (5) : 34–38. |
| [16] | Zhou LW. MiSeq: A new personalized sequencing machine[J]. Biotech World , 2011 (5) : 14–16. (in chinese) 周林文. MiSeq:新一代个人化测序仪[J]. 生物技术世界 , 2011 (5) : 14–16. |
| [17] | Wang YX. A Complete Checklist of Mammal Species and Subspecies in China a Taxonomic and Geographic Reference[M]. Beijing: China Forestry Publishing House, 2003: 38 . (in chinese) 王应祥. 中国哺乳动物种和亚种分类名录与分布大全[M]. 北京: 中国林业出版社, 2003: 38 . |
| [18] | Smith AT, Xie Y. A Guide to the Mammals of China[M]. Translated by Chen TX. Changsha: Hunan Education Publishing House, 2009: 296-300(in Chinese) >Smith AT, 解焱.中国兽类野外手册[M].陈廷熹, 译.长沙:湖南教育出版社, 2009: 296-300 |
| [19] | Bu YZ, Wang YM, Zhang C, et al. Geographical distribution, roost selection, and conservation state of cave-dwelling bats in China[J]. Mammalia , 2014, 79 (4) : 409–417. DOI:10.1515/mammalia-2014-0008 |
| [20] | Chen N, Yang Y, Zhang L, et al. Changes of postnatal gut microbiota in preterm infants using Illumina high-throughput sequencing technology[J]. Chinese Journal of Evidence-Based Pediatrics , 2014, 9 (5) : 359–364. (in chinese) 陈娜, 杨毅, 张澜, 等. Illumina高通量测序技术分析早产儿出生后肠道菌群变化的初步研究[J]. 中国循证儿科杂志 , 2014, 9 (5) : 359–364. |
| [21] | Caroline J, Gross MS, Spillman DM. Fiber digestion in mammals[J]. Pakistan Journal of Biological Sciences , 2003, 6 (17) : 1564–1573. DOI:10.3923/pjbs.2003.1564.1573 |
| [22] | Whitaker Jr JO, Dannelly HK, Prentice DA. Chitinase in insectivorous bats[J]. Journal of Mammalogy , 2004, 85 (1) : 15–18. DOI:10.1644/1545-1542(2004)085<0015:CIIB>2.0.CO;2 |
| [23] | Di Bella C, Piraino C, Caracappa S, et al. Enteric microflora in Italian chiroptera[J]. Journal of Mountain Ecology , 2003, 7 (Suppl) : 221–224. |
| [24] | Zhang DZ, Wen JY, Zhou WC, et al. Hospital infection and multidrug resistance of Flavobacterium: characteristics and analysis[J]. Chinese Journal of Nosocomiology , 2009, 19 (15) : 2040–2043. (in chinese) 张德忠, 温建艳, 周文聪, 等. 黄杆菌属医院感染特性与多药耐药分析[J]. 中华医院感染学杂志 , 2009, 19 (15) : 2040–2043. |
| [25] | Chaverri G. Flora bacteriana aeróbica del tracto digestivo del vampiro común, Desmodus rotundus (Chiroptera: phyllostomidae)[J]. Revista de Biología Tropical , 2006, 54 (3) : 717–724. |
| [26] | Hajkova P, Pikula J. Veterinary treatment of evening bats (Vespertilionidae) in the Czech Republic[J]. The Veterinary Record , 2007, 161 (4) : 139–140. DOI:10.1136/vr.161.4.139 |
| [27] | Mühldorfer K, Speck S, Kurth A, et al. Diseases and causes of death in European bats: dynamics in disease susceptibility and infection rates[J]. PLoS One , 2011, 6 (12) : e29773. DOI:10.1371/journal.pone.0029773 |
| [28] | Simpson VR. Veterinary advances in the investigation of wildlife diseases in Britain[J]. Research in Veterinary Science , 2000, 69 (1) : 11–16. DOI:10.1053/rvsc.2000.0384 |
| [29] | Daffner B. Causes of morbidity and mortality in British bat species and prevalence of selected zoonotic pathogens[D]. London: Master's Thesis of University of London, 2001 |
| [30] | Helmick KE, Heard DJ, Richey L, et al. A Pasteurella-like bacterium associated with pneumonia in captive megachiropterans[J]. Journal of Zoo and Wildlife Medicine , 2004, 35 (1) : 88–93. DOI:10.1638/01-083 |
| [31] | Gorschlüter A, Mak LH, Sundermeier C, et al. Electro-magnetic base technology for extremly sensitive immunosensors and DNA-chips[J]. Biomedizinische Technik , 2002, 47 (s1a) : 213–216. DOI:10.1515/bmte.2002.47.s1a.213 |
| [32] | Li ZX, Zhang XH, Wang YX. Research advance in lactococci and its infection[J]. Section of Clinical Biochemistry & Laboratory Medicine Foreign Medical Sciences , 2005, 26 (12) : 928–931. (in chinese) 李仲兴, 张新华, 王永祥. 乳球菌及其感染的研究进展[J]. 国外医学临床生物化学与检验学分册 , 2005, 26 (12) : 928–931. |
| [33] | Li YQ, Yin SL, Li X. Report: Severe diarrhea patients were detected 84Hafnia alvei strains[J]. Shandong Medical Journal , 1999, 39 (16) : 63. (in chinese) 李悦庆, 殷淑兰, 李新. 重症腹泻患者检出峰房哈夫尼亚菌84株报告[J]. 山东医药 , 1999, 39 (16) : 63. |
| [34] | Jiang TJ. Hafnia alvei sepsis was misdiagnosed as upper respiratory tract infection[J]. Journal of Clinical Internal Medicine , 2002, 19 (6) : 465. (in chinese) 姜天俊. 蜂房哈夫尼亚菌败血症误诊为上呼吸道感染一例[J]. 临床内科杂志 , 2002, 19 (6) : 465. |
| [35] | Lin CZ, Xing WC, Ren ZQ, et al. Community acquired with Morganella morganii bacterium: a case report and review of literatures[J]. Chinese Journal of Clinicians (Electronic Edition) , 2013, 7 (11) : 4840–4843. (in chinese) 林存智, 邢维聪, 任兆强, 等. 社区获得性摩根摩根菌肺部感染一例并文献复习[J]. 中华临床医师杂志:电子版 , 2013, 7 (11) : 4840–4843. |
| [36] | Xu YF, Chu SR, Wang YN. A survey was conducted for food poisoning caused by Morganella morganii[J]. Prevetive Medicine Tribune , 2008, 14 (4) : 364. (in chinese) 许玉芬, 褚思瑞, 王亚楠. 1起由摩氏摩根氏菌引起的食物中毒调查[J]. 预防医学论坛 , 2008, 14 (4) : 364. |
| [37] | Zhao HY, SunYP, Zhang JK, et al. The analysis of pathogenicity's Enterococcus and resistance[J]. Chinese Medicine of Factory and Mine , 2009, 22 (2) : 198–199. (in chinese) 赵红艳, 孙彦平, 张静凯, 等. 肠球菌的致病性及耐药性分析[J]. 中国厂矿医学 , 2009, 22 (2) : 198–199. |
| [38] | Brenner FW, Villar RG, Angulo FJ, et al. Salmonella nomenclature[J]. Journal of Clinical Microbiology , 2000, 38 (7) : 2465–2467. |
| [39] | Foley SL, Lynne AM. Food animal-associated Salmonella challenges: pathogenicity and antimicrobial resistance[J]. Journal of Animal Science , 2008, 86 (14 Suppl) : E173–E187. |
| [40] | Yu XC, Zhu MY, Zhang DZ. Character of hospital infection and Multidrug Resistance of Citrobacter[J]. Chinese Journal of Nosocomiology , 2009, 19 (12) : 1603–1605. (in chinese) 俞锡灿, 朱美英, 张德忠. 柠檬酸杆菌属医院感染特性与多药耐药分析[J]. 中华医院感染学杂志 , 2009, 19 (12) : 1603–1605. |
 2016, Vol. 43
2016, Vol. 43




