扩展功能
文章信息
- 冯淑贞, 郑艺, 席晓霞, 张和平, 孙志宏
- FENG Shu-Zhen, ZHENG Yi, XI Xiao-Xia, ZHANG He-Ping, SUN Zhi-Hong
- 内蒙古锡林郭勒牧区发酵乳制品抗生素抗性基因多样性
- Study on the antibiotic resistance genes diversity of fermented products in Inner Mongolia Xilin Gol pasturing area
- 微生物学通报, 2016, 43(8): 1725-1731
- Microbiology China, 2016, 43(8): 1725-1731
- DOI: 10.13344/j.microbiol.china.150662
-
文章历史
- 收稿日期: 2015-08-25
- 接受日期: 2015-12-04
- 优先数字出版日期(www.cnki.net): 2015-12-31
自1928年英国细菌学家Fleming发现青霉素以来,抗生素便在人类医疗以及动物养殖方面发挥着重要的作用[1],然而随着其在全球范围内大量且无节制的使用,导致环境污染和生态毒害问题日益严重,更为严重的是在抗生素选择压力的影响下,携带抗性基因的微生物数量不断增加[2]。2006年Pruden等将抗生素抗性基因(ARGs)作为新型污染物提出[3],自此国内外关于抗性基因来源及传播机制的研究层出不穷,细菌携带的抗性基因包括内源抗性基因与外源抗性基因,其中外源抗性基因可通过转座子、接合质粒、整合子实现水平转移而具有更大的危害性[4]。目前研究表明土壤、地下水、地表水等众多生境均成为抗性基因的储存库[5-6],更令人担忧的是一些对人体健康有益的乳酸菌也可作为抗性基因的储存库,并且能够将抗生素抗性基因水平转移给肠道微生物或致病菌,造成抗生素抗性基因的面源污染[7]。因此在当今人们越来越关注食品安全的大环境下,关于乳酸菌及发酵乳制品中抗性基因的丰度研究也应该受到关注,但是国内关于此方面的研究鲜有开展。
内蒙古锡林郭勒牧区远离城市,是蒙古族的聚居地。相对于养殖场的大规模圈养方式,当地牧民仍保留着相对传统的畜牧方式,多数家畜以天然植被为食。此外,当地牧民仍保留着制作和饮用发酵乳的传统,其中发酵牛乳和马乳是当地最常见的乳制品。本研究应用q-PCR技术对采集自内蒙古锡林郭勒牧区的11份传统发酵乳制品中常见抗生素抗性基因丰度进行了绝对定量检测,旨在对当地抗生素的使用情况和抗性基因的污染程度作出客观评估,同时也为后续探索环境中抗性基因的来源、传播和扩散机制提供一定的数据基础。
1 材料与方法 1.1 材料 1.1.1 样品来源: 11份传统发酵乳制品均采集自内蒙古锡林郭勒镶黄旗牧区,其中包括6份传统发酵酸牛乳和5份酸马乳。样品采集后置于4℃便携式冰箱内,于当日到达实验室后保存于−80℃,48 h内提取DNA。 1.1.2 主要试剂和仪器: SYBR® Premix Ex Taq™Ⅱ,宝生物工程有限公司;SanPrep柱式DNA胶回收试剂盒,上海生工生物工程股份有限公司;PowerSoil DNA Isolation Kit,美国MOBIO公司。PCR仪,Applied Biosystems公司;Eppendorf 5810R型台式高速离心机、ND-1000型微量紫外分光光度计,基因有限公司;ML-30L型全自动高压蒸汽灭菌锅,Panasonic仪器公司;HH-SI-NI恒温水浴槽,金坛市正基仪器有限公司;CDS8000型UPV凝胶成像分析系统,Sagecreation公司;PL303/01电子天平,Mettler Toledo公司;DYY-12电泳仪,北京六一仪器厂。 1.2 方法 1.2.1 发酵乳中细菌宏基因组DNA的提取: 应用PowerSoil DNA Isolation Kit提取DNA,具体步骤参照试剂盒说明书。 1.2.2 抗性基因及其引物的筛选: 选取四环素类、磺胺类、红霉素类、利福平类、甲硝唑类、链霉素类、万古霉素类、氟喹诺酮类、氯霉素类和青霉素类常见抗生素抗性基因用于定量研究,经过查阅文献和与NCBI数据库比对以及前期预实验验证,最终确定了22种抗生素抗性基因引物用于后续定量研究,试验所用引物均由上海桑尼生物科技有限公司合成,引物序列见表 1。| ARGs | Forward primer (5′→3′) | Reverse primer (5′→3′) | Tm(℃) | Reference |
| tetO | AATGAAGATTCCGACAATTT | CTCATGCGTTGTAGTATTCCA | 50 | [8] |
| tetL | GTMGTTGCGCGCTATATTCC | GTGAAMGRWAGCCCACCTAA | 50 | [8] |
| rpp | GAYACNCCNGGNCAYRTNG | GCCCARWANGGRTTNGGNGG | 45 | [9] |
| int | GCGTGATTGTATCTCACT | GACGCTCCTGTTGCTTCT | 50 | [8] |
| sulA | CAGGCTCGTAAATTGATAGCAGAAG | CTTTCCTTGCGAATCGCTTT | 50 | [10] |
| mefA | CTGTATGGAGCTACCTGTCTGG | CCCAGCTTAGGTATACGTAC | 52 | [11] |
| ermB | CATTTAACGACGAAACTGGC | GGAACATCTGTGGTATGGCG | 52 | [12] |
| ermC | TTTGAAATCGGCTCAGGAAAA | ATGGTCTATTTCAATGGCAGTTACG | 50 | [10] |
| ermT | GTAAAATCCCTAGAGAATACTTTCATCCA | TGAGTGATATTTTTGAAGGGTGTCTT | 50 | [10] |
| rpoB | GGTCGGCATGTCGCGGATGG | GCACGTCGCGGACCTCCAGC | 64 | [13] |
| rdxA | GTTAGGGATTTTATTGTATG | ACGCCAAGCATTTGAGCAAA | 48 | [14] |
| frxA | TCTCAAGCGGAAAAATCCGG | AATTTTTGATGATTTGAGCG | 48 | [14] |
| strA | CCGGTGGCATTTGAGAAAAA | GTGGCTCAACCTGCGAAAAG | 55 | [10] |
| strB | GCTCGGTCGTGAGAACAATCT | CAATTTCGGTCGCCTGGTAGT | 56 | [10] |
| vanC | ACAGGGATTGGCTATGAACCAT | TGACTGGCGATGATTTGACTATG | 50 | [10] |
| gyrA | CAMCGKCGKATTCTTTACGGAATG | TTRTTGATATCRCGBAGCATTTC | 50 | [8] |
| floR | ATTGTCTTCACGGTGTCCGTT | CCGCGATGTCGTCGAACT | 50 | [10] |
| yidY | GCAGTTGCATATCGCCTTCT | CTTCCCGGCAAACAGCAT | 50 | [10] |
| cfr | GCAAAATTCAGAGCAAGTTACGAA | AAAATGACTCCCAACCTGCTTTAT | 50 | [10] |
| cmlA1 | TAGGAAGCATCGGAACGTTGAT | CAGACCGAGCACGACTGTTG | 50 | [10] |
| cat | TTAGGTTATTGGGATAAGTTA | GCATGRTAACCATCACAWAC | 48 | [9] |
| pbpla | AAACAAGGTCGGACTCAACC | AGGTGCTACAAATTGAGAGG | 50 | [15] |

|
应用绝对定量的方法获得样品中抗性基因的丰度,标准品的制备是至关重要的,当标准曲线的相关系数R2无限接近于1,定量结果可信度越高。试验绘制的标准曲线线性关系良好,R2均大于0.99。
2.2 发酵牛乳和马乳中红霉素类抗生素抗性基因丰度检测发酵牛乳和发酵马乳样品中红霉素类抗生素抗性基因(ermB、ermC、ermT和mefA)定量结果如图 1所示,在1 mL发酵牛乳中,红霉素类抗性基因ermB、ermC、ermT和mefA的丰度取对数后分别为:6.361±0.096、4.018±0.236、3.476±0.038、6.434±0.046。而在每mL发酵马乳中,红霉素类抗性基因ermB、ermC、ermT和mefA的丰度取对数后分别为:4.267±0.254、4.465±0.096、3.372±0.122、6.142±0.006。经过统计分析,ermB在发酵牛乳中的丰度显著高于发酵马乳(p < 0.05)。
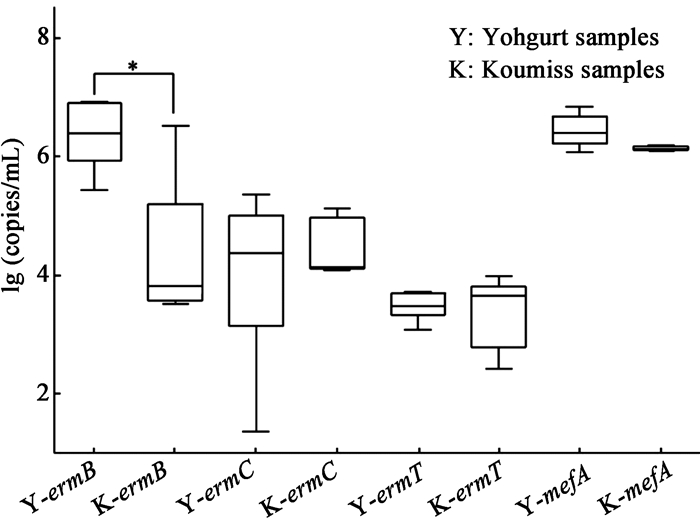
|
| 图 1 红霉素类抗生素抗性基因丰度 Figure 1 The abundance of erythrocin resistance genes in fermented dairy samples Note: Asterisks indicate statistical significance. *: P < 0.05. |
|
|
发酵牛乳和发酵马乳样品中甲硝唑、链霉素和万古霉素类抗生素抗性基因(rdxA、frxA、strA、strB、vanC和gyrA)定量结果如图 2所示,在1 mL发酵牛乳中,甲硝唑、链霉素和万古霉素类抗生素抗性基因rdxA、frxA、strA、strB、vanC和gyrA的丰度取对数后分别为:2.630±0.108、2.908±0.058、5.561±0.144、8.648±0.087、4.176±0.040、8.537±0.031。而在1 mL发酵马乳中甲硝唑、链霉素和万古霉素类抗生素抗性基因rdxA、frxA、strA、strB、vanC和gyrA的丰度取对数后分别为:2.754±0.127、2.709±0.077、3.251±0.378、7.009±0.207、3.807±0.040、7.453±0.275。经过统计分析,strA和strB在发酵牛乳中的丰度极显著高于发酵马乳(p < 0.001)。gyrA在发酵牛乳中的丰度显著高于发酵马乳(p < 0.05),而rdxA、frxA和vanC在发酵牛乳和发酵马乳中的丰度差异不显著。

|
| 图 2 甲硝唑、链霉素和万古霉素类抗生素抗性基因丰度 Figure 2 The abundance of metronidazole, streptomycin and vancomycin resistance genes in fermented dairy samples Note: Asterisks indicate statistical significance. ***: P < 0.001. |
|
|
发酵牛乳和发酵马乳样品中四环素类抗生素抗性基因(tetL、tetO、rpp和int)定量结果如图 3所示,在1 mL发酵牛乳中,四环素类抗生素抗性基因tetL、tetO、rpp和int的丰度取对数后分别为:3.529±0.051、4.020±0.082、3.529±0.051、5.220±0.109。而在1 mL发酵马乳中,四环素类抗生素抗性基因tetL、tetO、rpp和int的丰度取对数后分别为:3.187±0.084、1.028±0.338、3.187±0.084、4.720±0.317。经过统计分析,tetO在发酵牛乳中的丰度显著高于发酵马乳(p < 0.05),而tetL、rpp和int在发酵牛乳和发酵马乳中的丰度差异不显著。
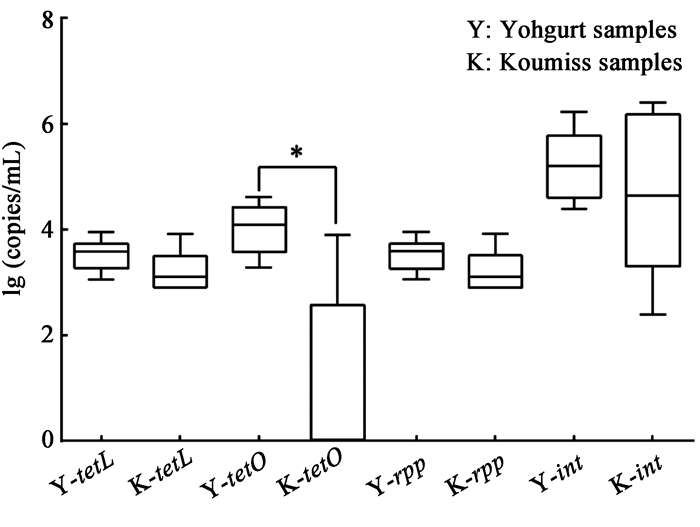
|
| 图 3 四环素类抗生素抗性基因丰度 Figure 3 The abundance of tetracyclines resistance genes in fermented dairy samples |
|
|
发酵牛乳和发酵马乳样品中氟喹诺酮类抗生素抗性基因(yidY、cfr、cmlA1和floR)定量结果如图 4所示,在1 mL发酵牛乳中,氟喹诺酮类抗生素抗性基因yidY、cfr、cmlA1和floR的丰度取对数后分别为:6.069±0.006、5.771±0.049、4.070±0.103、6.804±0.039。而在1 mL发酵马乳中,氟喹诺酮类抗生素抗性基因yidY、cfr、cmlA1和floR的丰度取对数后分别为:4.255±0.499、5.712±0.093、3.370±0.100、6.215±0.099。经过统计分析,yidY在发酵牛乳中的丰度显著高于发酵马乳(p < 0.05),cfr、cmlA1和floR的丰度差异不显著。
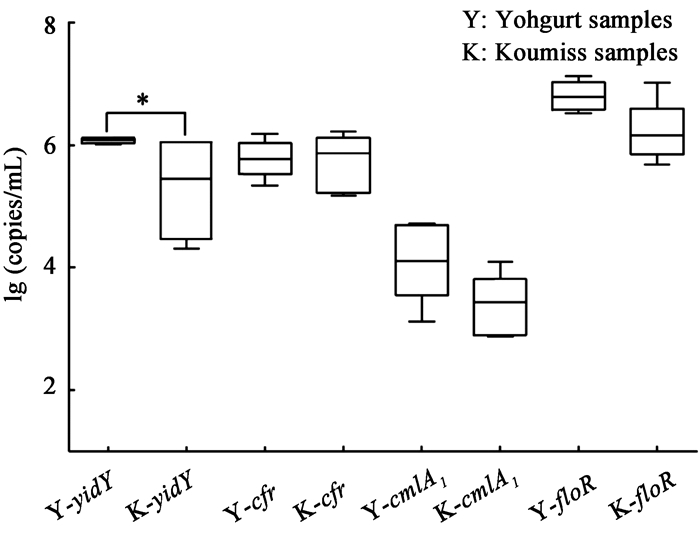
|
| 图 4 氟喹诺酮类抗生素抗性基因丰度 Figure 4 The abundance of fluoroquinolones resistance genes in fermented dairy samples |
|
|
发酵牛乳和发酵马乳样品中磺胺类药物、氯霉素、青霉素和利福平类抗生素抗性基因(sulA、cat、pbpla和rpoB)定量结果如图 5所示,在1 mL发酵牛乳中,磺胺类药物、氯霉素、青霉素和利福平类抗生素抗性基因sulA、cat、pbpla和rpoB的丰度取对数后分别为:3.937±0.096、6.950±0.120、4.256±0.034、7.310±0.092。而在1 mL发酵马乳中,磺胺类药物、氯霉素、青霉素和利福平类抗生素抗性基因sulA、cat、pbpla和rpoB的丰度取对数后分别为:3.836±0.160、6.084±0.040、3.932±0.070、6.720±0.149。经统计分析,cat在发酵牛乳中的丰度显著高于发酵马乳(p < 0.05),而sulA、pbpla和rpoB在发酵牛乳和发酵马乳中的丰度差异不显著。

|
| 图 5 磺胺类药物、氯霉素、青霉素和利福平类抗生素抗性基因丰度 Figure 5 The abundance of sulfonamides, chloramphenicol, penicillin and rifampicin resistance genes in fermented dairy samples |
|
|
本文利用q-PCR技术对内蒙古锡林郭勒牧区传统发酵乳制品中的抗生素抗性基因的多样性进行了分析。结果显示:22种抗性基因均在发酵酸牛乳以及酸马乳中被检测到,其中发酵酸牛乳中红霉素类(ermB)、链霉素类(strA、strB)、万古霉素类(gyrA)、四环素类(tetO)、氟喹诺酮类(yidy)、氯霉素类(cat)等7种抗性基因丰度显著高于发酵酸马乳(p < 0.05),其余15种常见抗性基因丰度在两组样品之间无显著差异。
抗性基因的直接载体是微生物,抗性基因的丰度与环境中微生物的种类之间存在一定的相关性[16-17],大量研究表明,乳酸菌携带有抗生素抗性基因[18-21],2005年乌日娜在内蒙古锡林郭勒盟牧区采集的传统酸马乳样品中共分离出49株乳杆菌,其中干酪乳杆菌和植物乳杆菌是优势菌,共占到分离菌株的53%[22]。2006年周丽霞在采集自内蒙古牧区的传统乳制品中共分离到53株乳杆菌,植物乳杆菌和干酪乳杆菌为优势菌群,共占到分离菌株66%[23],这可能是导致酸牛乳及酸马乳中均存在磺胺类、利福平类、甲硝唑类以及青霉素类抗性基因且无显著差异的原因。此外,抗性基因丰度与抗生素浓度具有一定的相关性[5-6]。本实验得出cat、ermB、tetO、strA和strB基因在酸牛乳中的丰度显著高于酸马乳(p < 0.05),这可能归结于原料牛乳中常使用氯霉素、红霉素、四环素、链霉素等抗生素用于预防治疗牛乳房炎[24]。此外链霉素可以与细菌核糖体小亚基结合使之变构,引起读码错误,并最终合成异常蛋白质而导致细菌死亡,因而草原地区大量使用链霉素来预防鼠疫,这也可能是导致链霉素抗性基因在样品中丰度较高的原因之一[25]。
本文对发酵乳制品中抗性基因丰度进行了绝对定量分析,结果表明这些样品可能是潜在的抗性基因储存库,其中发酵牛乳制品中strB和gyrA抗性基因的含量最高,分别达到(8.537±0.031)、(8.648±0.087) lg(copies/mL)。这一结果提醒我们有必要对上市的发酵乳制品从表型和基因水平进行抗生素抗性分析,并且需要在日后进一步深入研究不同来源的发酵乳制品中抗性基因的传播机制及控制措施,以防止抗性基因在食品链中的传播,否则进入人体的抗性基因将有可能传递给肠道微生物或致病菌,改变体内菌群抗药性,造成不可估量的后果。
| [1] | Bentley R. The development of penicillin: genesis of a famous antibiotic[J]. Perspectives in Biology & Medicine , 2005, 48 (3) : 444–452. |
| [2] | Luo Y, Mao D, Rysz M, et al. Trends in antibiotic resistance genes occurrence in the Haihe River, China[J]. Environmental Science and Technology , 2010, 44 (19) : 7220–7225. DOI:10.1021/es100233w |
| [3] | Pruden A, Pei R, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants: studies in Northern Colorado[J]. Environmental Science & Technology , 2006, 40 (23) : 7445–7450. |
| [4] | Zou W, Luo Y, Zhou QX. Pollution and environmental regulation of antibiotic resistance genes (ARGs) in Livestock Manure[J]. Journal of Agro-Environment Science , 2014, 33 (12) : 2281–2287. (in chinese) 邹威, 罗义, 周启星. 畜禽粪便中抗生素抗性基因(ARGs)污染问题及环境调控[J]. 农业环境科学学报 , 2014, 33 (12) : 2281–2287. |
| [5] | He LY, Liu YS, Su HC, et al. Dissemination of antibiotic resistance genes in representative broiler feedlots environments: identification of indicator ARGs and correlations with environmental variables[J]. Environmental Science & Technology , 2014, 48 (22) : 13120–13129. |
| [6] | Li JN, Cheng WX, Xu LK, et al. Antibiotic-resistant genes and antibiotic-resistant bacteria in the effluent of urban residential areas, hospitals, and a municipal wastewater treatment plant system[J]. Environmental Science and Pollution Research , 2015, 22 (6) : 4587–4596. DOI:10.1007/s11356-014-3665-2 |
| [7] | Hummel AS, Hertel C, Holzapfel WH, et al. Antibiotic resistances of starter and probiotic strains of lactic acid bacteria[J]. Applied and Environmental Microbiology , 2007, 73 (3) : 730–739. DOI:10.1128/AEM.02105-06 |
| [8] | Yang M. Antibiotic resistance and related genes of lactic acid bacteria isolated from traditional fermented dairy products[D]. Hohhot: Master's Thesis of Inner Mongolia Agricultural University, 2010 (in Chinese) 杨梅.传统发酵乳制品中乳酸菌的抗生素抗性及相关抗性基因的研究[D].呼和浩特:内蒙古农业大学硕士学位论文, 2010 http://cdmd.cnki.com.cn/Article/CDMD-10129-2010150337.htm |
| [9] | van Hoek AHAM, Mayrhofer S, Domig KJ, et al. Mosaic tetracycline resistance genes and their flanking regions in Bifidobacterium thermophilum and Lactobacillus johnsonii[J]. Antimicrobial Agents and Chemotherapy , 2008, 52 (1) : 248–252. DOI:10.1128/AAC.00714-07 |
| [10] | Zhu YG, Johnson TA, Su JQ, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America , 2013, 110 (9) : 3435–3440. DOI:10.1073/pnas.1222743110 |
| [11] | Cauwerts K, Pasmans F, Devriese LA, et al. Cloacal Lactobacillus isolates from broilers often display resistance toward tetracycline antibiotics[J]. Microbial Drug Resistance , 2006, 12 (4) : 284–288. DOI:10.1089/mdr.2006.12.284 |
| [12] | Ubukata K, Iwata S, Sunakawa K. In vitro activities of new ketolide, teithromycin, and eight other macrolide antibiotics against Streptococcus pneumonia having mefA and ermB genes that mediate macrolide resistance[J]. Journal of Infection and Chemotherapy , 2003, 9 (3) : 221–226. DOI:10.1007/s10156-003-0258-2 |
| [13] | Sharma M, Sethi S, Mishra B, et al. Rapid detection of mutations in rpoB gene of rifampicin resistant Mycobacterium tuberculosis strains by line probe assay[J]. Indian Journal of Medical Research , 2003, 117 : 76–80. |
| [14] | Aldana LP, Kondo M, Kondo T, et al. In vitro induction of resistance to metronidazole, and analysis of mutations in rdxA and frxA genes from helicobacter pylori isolates[J]. Journal of Infection and Chemotherapy , 2005, 11 (2) : 59–63. DOI:10.1007/s10156-004-0370-Y |
| [15] | Ubukata K, Muraki T, Igarashi T, et al. Identification of penicillin and other Beta-Lactam resistance in Streptococcus pneumoniae by polymerase chain reaction[J]. Journal of Infection and Chemotherapy , 1997, 3 (4) : 190–197. DOI:10.1007/BF02490033 |
| [16] | Cheng W, Chen H, Su C, et al. Abundance and persistence of antibiotic resistance genes in livestock farms: a comprehensive investigation in eastern China[J]. Environment International , 2013, 61 : 1–7. DOI:10.1016/j.envint.2013.08.023 |
| [17] | Zhang T, Zhang M, Zhang XX, et al. Tetracycline resistance genes and tetracycline resistant lactose-fermenting Enterobacteriaceae in activated sludge of sewage treatment plants[J]. Environmental Science & Technology , 2009, 43 (10) : 3455–3460. |
| [18] | Pan L, Hu XQ, Wang XY. Assessment of antibiotic resistance of lactic acid bacteria in Chinese fermented foods[J]. Food Control , 2011, 22 (8) : 1316–1321. DOI:10.1016/j.foodcont.2011.02.006 |
| [19] | Sharma P, Tomar SK, Goswami P, et al. Antibiotic resistance among commercially available probiotics[J]. Food Research International , 2014, 57 : 176–195. DOI:10.1016/j.foodres.2014.01.025 |
| [20] | Nawaz M, Wang J, Zhou AP, et al. Characterization and transfer of antibiotic resistance in lactic acid bacteria from fermented food products[J]. Current Microbiology , 2011, 62 (3) : 1081–1089. DOI:10.1007/s00284-010-9856-2 |
| [21] | atalolukO, GogebakanB. Presence of drug resistance in intestinal Lactobacilli of dairy and human origin in Turkey[J]. FEMS Microbiology Letters , 2004, 236 (1) : 7–12. |
| [22] | Wu RN. Study on identification and 16S rDNA homology analysis of Lactobacillus from koumiss in Inner Mongolia[D]. Hohhot: Master's Thesis of Inner Mongolia Agricultural University, 2005 (in Chinese) 乌日娜.内蒙古传统酸马奶中乳杆菌的分离鉴定及16S rDNA序列同源性分析[D].呼和浩特:内蒙古农业大学硕士毕业论文, 2005 |
| [23] | Zhou YX. Studies on biological properties and probiotic effects of Lactobacilli from traditional dairy products in Inner Mongolia[D]. Hohhot: Doctoral Dissertation of Inner Mongolia Agricultural University, 2006 (in Chinese) 周雨霞.内蒙古牧区传统乳制品中乳杆菌生物学特性及其益生作用的研究[D].呼和浩特:内蒙古农业大学博士毕业论文, 2006 |
| [24] | Zhu WJ, Deng GZ. Prevention and treatments of mastitis of dairy cow[J]. China Journal of Veterinary Medicine , 2008, 42 (3) : 40–45. (in chinese) 朱文娟, 邓干臻. 奶牛乳腺炎防治方法的研究进展[J]. 中国兽药杂志 , 2008, 42 (3) : 40–45. |
| [25] | Nizza S, Rando F, Fiorito F, et al. Fecal microbiota and antibiotic resistance in ferrets (Mustela putorius furo) from two captive breeding facilities in Italy[J]. Research in Veterinary Science , 2014, 96 (3) : 426–428. DOI:10.1016/j.rvsc.2014.03.015 |
 2016, Vol. 43
2016, Vol. 43




