扩展功能
文章信息
- 付瑞敏, 于烽, 常慧萍, 张红, 陈五岭
- FU Rui-Min, YU Feng, CHANG Hui-Ping, ZHANG Hong, CHEN Wu-Ling
- 一株扩展青霉拮抗菌的分离、鉴定及抑菌活性物质
- Isolation, identification and characterization of an antagonistic bacterium against Penicillium expansum
- 微生物学通报, 2016, 43(8): 1715-1724
- Microbiology China, 2016, 43(8): 1715-1724
- DOI: 10.13344/j.microbiol.china.150662
-
文章历史
- 收稿日期: 2015-09-04
- 接受日期: 2015-12-16
- 优先数字出版日期(www.cnki.net): 2015-12-16
2. 河南教育学院生命科学系 河南 郑州 450046
2. Population and Life Department, Henan Institute of Education, Zhengzhou, Henan 450046, China
随着人们环保意识的不断增强,生物农药越来越多应用于植物病害的防治。其中,芽孢杆菌以其对环境的高度耐受性和具有产生各种多肽类、脂肽类和细菌素等抑菌性物质的特点成为当前生物农药的研究热点。解淀粉芽孢杆菌次生代谢产物有抗菌性脂肽、蛋白或多肽等,不仅可抑制多种植物病原性真菌,而且可促进植物生长和诱导植物抗病性。特别是一些由非核糖体多肽合成酶途径合成的脂肽,主要包括IturinA、Fengycin和Surfactin[1]等,由于其两亲分子的结构特点和非特异性的作用模式使其能够广泛作用于多种病原菌,因此作为新一代抗生素受到关注[2-3]。如何将这些活性物质分离纯化并制成新型、高效的生物农药,是当前研究的工作重点。
近年来,随着基因组测序工作的全面推进,枯草芽孢杆菌、解淀粉芽孢杆菌等菌株的基因组已测序完成,为利用PCR技术快速检测同类菌株的抗生素合成基因创造了条件。由扩展青霉(Penicillium expansum)[4]感染苹果所致的青霉病是苹果采摘后导致果实腐烂的重要病害之一,针对该病原菌,本实验室从苹果表面分离出一株可有效抑制苹果青霉病的生防菌BA-16,结合该菌株的形态、生理生化和分子系统发育等特征对其进行鉴定。为确定该菌株所产的脂肽类型及含量,先利用NCBI上已知的脂肽抗生素(Surfactin inturinA fengycin)合成基因(sfp,fenB,ituD)设计引物,以菌株BA-16的基因组DNA为模板,采用PCR技术进行脂肽抗生素合成酶的相关基因检测,然后利用脂肽提取方法制备出粗提物,并采用HPLC和MALDI-TOF-MS对粗提物中的脂肽进行纯化和鉴定,以确定脂肽类型,本研究为认识解淀粉芽孢杆菌所产的脂肽类抗生素及将其应用于苹果采后的生物防治提供理论基础。
1 材料与方法 1.1 材料 1.1.1 菌种、供试苹果和培养基: 致病菌扩展青霉(Penicillium expansum)由陕西省微生物研究所惠赠;供试苹果为红富士(Red Fuji),由陕西洛川县渭北苹果种植园惠赠。YEB培养基和PDA培养基配方参见文献[5];摇瓶发酵培养基配方参见文献[6]。 1.1.2 主要试剂和仪器: Fengycin和Surfactin标准品购于sigma公司;乙腈、三氟乙酸、甲醇等皆为色谱纯,购自Sigma公司。Agilnt 1100series高效液相色谱系统购自美国安捷伦公司;液相色谱-电喷雾质谱仪(LC-ESI MS)由Waters Alliance2690高效液相色谱仪(美国Waters公司)和TSQ Quantum Discovery三级四极杆质谱仪(美国Thermo Fisher Scientific公司)组成。 1.2 细菌的分离、筛选从感染青霉病苹果的表面分离菌株。用无菌水清洗苹果健康部位,将所得液体经10−1−10−6的梯度稀释,将10−4、10−5、10−6梯度稀释液分别涂布在YEB平板上,37℃培养24 h。挑取各平板上的菌株分别进行纯化得到单克隆,将其分别编号以做进一步研究。
以扩展青霉为指示菌,用平板对峙法[7]对分离菌株进行拮抗能力的筛选。病原菌接种于PDA平板中央,待测菌株接种于离指示菌2.5 cm处,28℃培养48 h观察其生长状况,测量抑菌带宽度,实验均重复3次,挑取抑菌条带宽的菌株至YEB平板上,保存备用。
将初筛所得菌株接种于发酵培养基中,37℃、150 r/min振荡培养72 h。参照Pathak等的方法[8]制备菌株无细胞发酵液,取所制的发酵液200μL接入牛津杯中,采用牛津杯法[9]检测菌株无细胞发酵液拮抗扩展青霉的能力。28℃培养24 h后测量抑菌圈直径,挑选直径最大的菌株做进一步研究。整个实验以无菌水作为对照,实验均重复3次。
1.3 菌株的形态学与生理生化鉴定将复筛所选菌株经染色、光学显微镜观察菌体个体及群体形态,生理生化反应参照《微生物学实验指导》[10-11]。
1.4 16S rRNA基因序列分析使用上海生工有限公司的细菌基因组DNA快速抽提试剂盒提取所筛菌株的基因组DNA,以其为模板,参考文献[12]进行16S rRNA基因的PCR扩增,扩增引物见表 1。PCR反应体系(25μL):50 mg/L模板1μL,10μmol/L上下游引物各0.5μL,Pfu PCR MasterMix 12.5μL,双蒸水10.5μL。反应条件:94℃3 min;94℃1 min,55℃1 min,72℃2 min,30个循环;72℃10 min。目的片段经Sanprep柱式PCR试剂盒(上海生工生物工程有限公司)纯化后,提交至上海生工进行序列测定。用BLAST程序将所得序列结果与GenBank相关菌种的16S rRNA基因序列进行比对分析,利用MEGA 4.1构建系统发育树[13]。
| 基因 Gene |
引物序列 Primers sequence (5′→3′) |
长度 Size (bp) |
| 16S rRNA | AGAGTTTGATCATGGCTCAG CTACGGTTACCTTGTTACGAC |
1 468 |
| sfp | ATGAAGTTTACGGAATTTA TTATAAAAGCTCTTCGTACG |
675 |
| fenB | CTATAGTTTGTTGACGGCTC CAGCACTGGTTCTTGTCGCA |
1 400 |
| ItuD | ATGACAATCTTGCCTTTTTA TATTTTAAAATCCGCAATT |
1 203 |
将培养24 h的菌株发酵液于常温下8 000 r/min离心20 min,弃沉淀,将所得上清液置于无菌锥形瓶中,用浓度为7 mol/L的HCl将其pH调整至2.0,无菌条件下分装至10 mL的无菌离心管中(每管10 mL),4℃过夜,10 000 r/min离心20 min,取沉淀,在沉淀中加入0.5 mL的中性甲醇溶液,以此方法操作2次,将所得萃取液合并,并将其浓缩至5倍浓度,经0.2μm滤膜过滤后即为粗提液。
1.7 脂肽类抗生素的分离、纯化和鉴定 1.7.1 HPLC分离纯化脂肽类抗生素: 采用的HPLC分析系统为Agilent HPLC 1200series,色谱柱为ZORBAX Eclipse XDB-C18;检测波长为280 nm,柱温为30℃,进样量10μL,采用等梯度洗脱进行样品分析,洗脱条件如表 2所示。| 时间 Time (min) |
表面活性素 Surfactin |
丰原素 Fengycin |
|||||
| 乙腈(0.1%三氟乙酸) Acetonitrile (0.1% TFA) |
水(0.1%三氟乙酸) Water (0.1% TFA) |
流速 Flow rate (mL/min) |
乙腈(0.1%三氟乙酸) Acetonitrile (0.1% TFA) |
水(0.1%三氟乙酸) Water (0.1% TFA) |
流速 Flow rate (mL/min) |
||
| 0 | 20 | 80 | 0.8 | 60 | 40 | 1 | |
| 5 | 50 | 50 | 0.8 | 60 | 40 | 1 | |
| 15 | 65 | 35 | 0.8 | 90 | 10 | 1 | |
| 25 | 65 | 35 | 0.8 | 90 | 10 | 1 | |
经过初筛从苹果表面分离获得18株菌,其中酵母4株,细菌14株。通过平板对峙实验,筛选出可拮抗扩展青霉的6株。采用牛津杯法检测6株菌的无细胞发酵液抗扩展青霉活性,结果如表 3所示。结果表明:6株菌的菌株细胞和无细胞发酵液拮抗扩展青霉的效果表现出了显著的差异。其中,BA-16的拮抗效果最强,因此将其挑选出来做后续研究。
| 菌株 Strain |
抑菌带宽 Width of inhibition band (mm) |
抑菌圈直径 Diameter of inhibition zone (mm) |
| BA-3 | 4.196±0.025 | 3.338±0.026 |
| BA-6 | 4.980±0.035 | 4.013±0.028 |
| BA-8 | 5.980±0.016 | 4.580±0.023 |
| BA-12 | 6.650±0.033 | 5.380±0.024 |
| BA-17 | 6.880±0.003 | 5.560±0.002 |
| BA-16 | 7.050±0.016 | 5.880±0.036 |
菌株BA-16经革兰氏染色阳性,显微镜镜检为杆菌,有芽孢,有荚膜,具有运动性。该菌株在LB平板上形成乳白色菌落。菌落边缘不整齐,干燥不透明,中间有突起。生理生化结果见表 4。
| 项目 Items |
结果 Results |
| Anaerobic growth | + |
| Gelatin hydrolysis | + |
| Amylohydrolysis | + |
| Citrate utilization | − |
| Tyrosine reaction | − |
| V-P test | + |
| Indole test | + |
| 2% NaCl | + |
| 7% NaCl | − |
| 30℃ | + |
| 50℃ | − |
| D-glucose | + |
| Maltose | + |
| Mannitol | + |
| Note: +: positive; −: negative. | |
结合菌株BA-16的形态、染色及生理生化特征,将该菌株初步鉴定为Bacillus sp.。
2.3 16S rRNA基因序列分析提取菌株BA-16的总DNA进行PCR扩增,凝胶电泳回收扩增序列,得到一段1 468 bp长度的16S rRNA基因,所获序列提交至GenBank (登录号KR31430)。将其同GenBank中相近种属进行BLAST,对BLAST结果进行分析,通过MEGA 4.1软件构建系统发育树(图 1)。发现菌株BA-16与Bacillus amyloliquefaciens AY603658的相似性达到99%,综合形态学特征及生理生化鉴定结果,BA-16被鉴定为Bacillus amyloliquefaciens。
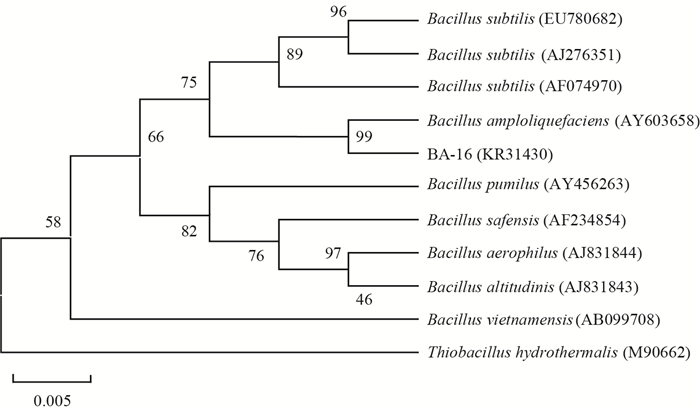
|
| 图 1 菌株BA-16 16SrRNA基因序列发育进化树 Figure 1 Phylogenetic tree of 16s rRNA gene sequences of isolated strain BA-16 注:参与比对的序列的GenBank登录号列于括号中,分支处标注有自展值,标尺所示长度为0.005核苷酸置换率. Note: The GenBank accession number of aligned sequences are shown in the brackets. the bootstrap value are shown at the node. bar 0.005 means the nucleotide substitution rate of 0.005. |
|
|
以所筛菌株BA-16的基因组DNA为模板进行PCR扩增,发现可得到两条大约为675 bp和1 400 bp的目的条带(图 2),初步确认为sfp基因和fenB基因,将PCR产物纯化测序后提交至NCBI进行相关序列的BLAST同源性分析,结果显示与GenBank公布的相关基因相似性高达99%,可确定菌株BA-16具有sfp和fenB基因,因此推测该菌株可产生Fengycin和Surfactin两种脂肽类抗生素。

|
| 图 2 脂肽类相关基因的PCR检测 Figure 2 PCR detection of lipopeptide related genes 注:M:DNA marker;1:ItuD基因;2:fenB基因;3:sfp基因. Note: M: DNA marker (Sangong) in base pairs of DL2000; 1: ItuD gene; 2: fenB gene; 3: sfp gene. |
|
|
粗提液经HPLC分离纯化后,所得片段如图 3、4所示,发现Surfactin标样在保留时间13.683、15.776、17.741和21.363 min处出现了特征峰(图 3A)。同一洗脱条件下,所测样品其Surfactin特征峰出现在13.690 min和17.781 min (图 3B),保留时间在标准峰附近,推测是Surfactin的同系物;Fengycin标样在保留时间20.717 min,33.665 min和41.260 min处出现了特征峰(图 4A);同一洗脱条件下,所测样品其Fengycin的特征峰出现在20.767 min和40.880 min (图 4B),这些峰也在标准峰附近,说明粗提液中也含有Fengycin。
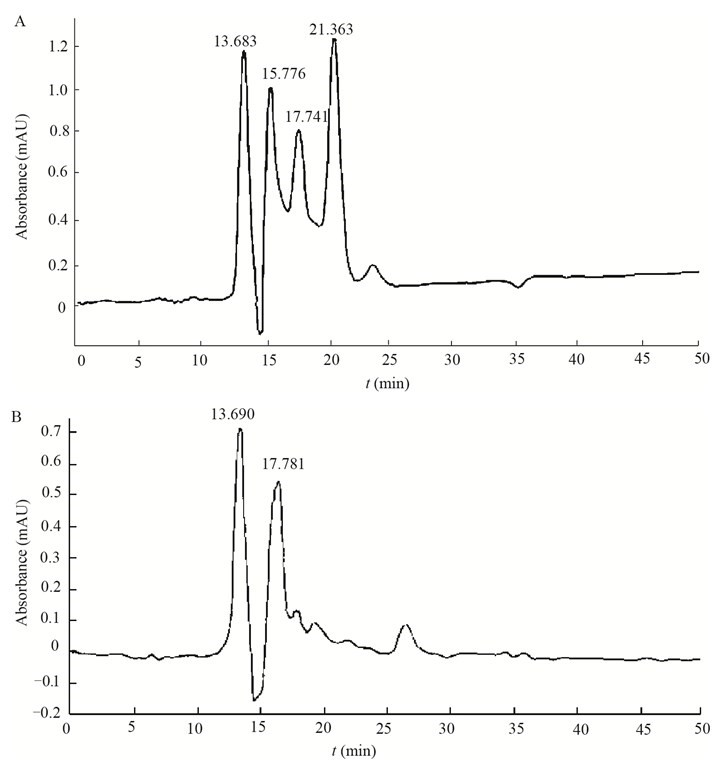
|
| 图 3 Surfactin标准品(A)和供试样本Surfactin (B)的HPLC色谱图温度-活力曲线图 Figure 3 HPLC spectra of standard surfactin (A) and test sample surfactin (B) |
|
|
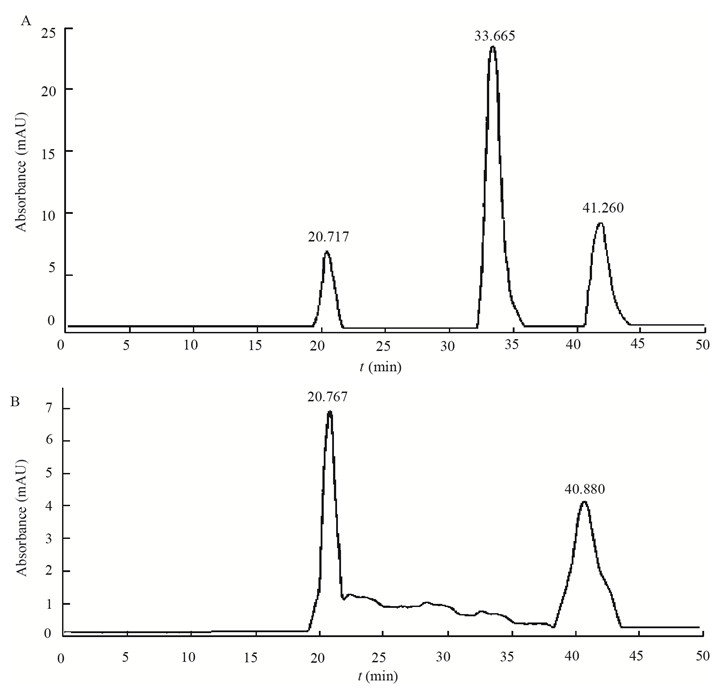
|
| 图 4 Fengycin标准品(A)和供试样本Fengycin (B)的HPLC色谱 Figure 4 HPLC spectra of standard Fengycin (A) and test sample Fengycin (B) |
|
|
将HPLC纯化得到的Surfactin和Fengycin分别采用牛津杯方法[9]检测其对扩展青霉的抑制效果,所得结果如表 5所示,Fengycin对扩展青霉展现了明显的抑菌效果,与之相反的是,Surfactin对扩展青霉几乎无抑制作用,推测在解淀粉芽孢杆菌抑制扩展青霉的过程中起主要作用的是脂肽类蛋白Fengycin。
| 组别 Group |
抑菌圈直径 Inhibition zone (mm) |
|
| 表面活性素Surfactin | 丰原素 Fengycin |
|
| Treatment group | 0.10±0.12 | 4.68±0.08 |
| Control group | 0 | 0 |
经飞行时间质谱检测分析,获得了粗提液中各脂肽产物同系物的分子量,所得结果如表 6所示。质谱图显示出2个系列的离子峰(图 5),图 5a显示了Surfactin家族同系物,其[M+H]+、[M+Na]+和[M+K]+离子如表 6所示,所得Surfactin分别为C13-Surfactin和C15-Surfactin,且第7位氨基酸为Leu;图 5b显示了Fengycin家族同系物,其[M+H]+、[M+Na]+和[M+K]+离子如表 6所示,所得Fengycin分别为C14-FengycinA、C15-FengycinA、C16-FengycinA和C17-FengycinA,这些Fengycin其第6位氨基酸为丙氨酸,C16-FengycinB和C17-FengycinB这些Fengycin其第6位氨基酸为缬氨酸。结合PCR检测结果、HPLC和质谱检测,所得发酵液抗菌性脂肽提取物中含有Fengycin和Surfactin。
| 片段 Fraction |
实测荷质比 Experimental charge-mass ratio (m/z) |
理论荷质比 Theoretical charge-mass ratio (m/z) |
强度 Intensity (%) |
结构判断 Structure assignment |
| Surfactin | 1 030.54 | 1 030.40 | 32 | C13-Surfactin, [M+Na]+ |
| 1 058.78 | 1 058.88 | 36 | C15-Surfactin, [M+Na]+ | |
| 1 435.61 | 1 435.58 | 28 | C14-FengycinA, [M+Na]+ | |
| 1 449.74 | 1 449.74 | 74 | C15-FengycinA, [M+Na]+ | |
| Fengycin | 1 463.75 | 1 463.78 | 50 | C16-FengycinA, [M+H]+ |
| 1 477.78 | 1 477.82 | 56 | C17-FengycinA, [M+H]+ | |
| 1 491.82 | 1 491.83 | 58 | C16-FengycinB, [M+H]+ | |
| 1 505.64 | 1 505.65 | 40 | C17-FengycinB, [M+H]+ |
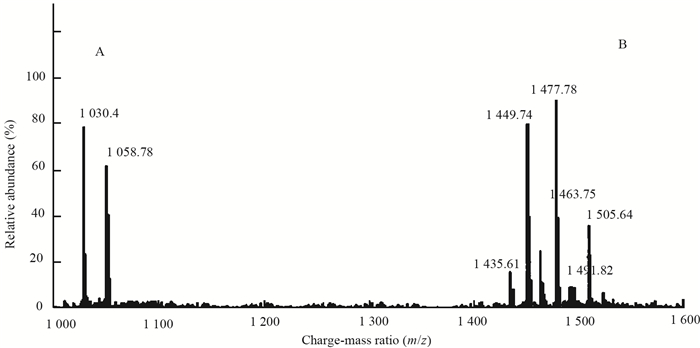
|
| 图 5 拮抗菌株BA-16发酵液中抗菌脂肽的质谱图 Figure 5 MS spectra of lipopeptides of BA-16 |
|
|
由于传统农药的广泛应用,不仅污染环境和损害人类身体健康,而且还容易使许多病原菌产生抗药性。抗菌脂肽作为生物农药,不仅没有二次污染,其非特异性的抗菌机制也可避免病菌抗药性的产生,在植物的病害防治中展现了巨大优势[14-16]。
本研究筛选出可有效拮抗扩展青霉的菌株BA-16,该菌株被鉴定为解淀粉芽孢杆菌。在此基础上进行了抗菌物质的分离、鉴定和抑菌机制的初步探讨。以分子生物学技术为辅助手段,首先通过基因检测推测拮抗菌代谢产物中可能存在的活性物质;然后利用HPLC技术对其进行分离、纯化和抑菌性能的初步鉴定;最后通过质谱检测获得了脂肽类产物的精确分子量。综合分析基因检测、HPLC和质谱鉴定的结果[17-18],发现扩展青霉拮抗菌BA-16可代谢产生Surfactin和Fengycin两类脂肽类抗生素,这与关于解淀粉芽孢杆菌可以产生脂肽类抗生素的多数报道相符[19]。
通过对比Surfactin和Fengycin两类脂肽类抗生素对扩展青霉的抑制效果,发现抑制扩展青霉活性的脂肽主要是Fengycin。Fengycin是由一条β-羟基脂肪酸链和一个10肽所形成的内酯环,它可作用于丝状真菌的磷脂双分子层,改变其细胞膜的通透性,从而显著抑制其活性[19-21]。虽然当前鲜有针对Fengycin的抗菌机制研究,但根据报道,IturinA与Fengycin同属于脂肽类抗生素,其抑菌活性主要源于其肽环氨基酸以及脂肪酸碳链的变化,例如通过结合到病菌细胞膜上,从而改变后者的通透性;或者通过肽环上的极性氨基酸嵌合到病菌核酸分子上从而影响后者的正常表达等[22-24]。由于Fengycin与IturinA在结构上具有很大的相似性,因此上述有关IturinA的抑菌机制为探讨Fengycin的抗菌原理提供了参考依据[24]。根据Fengycin 6号位置氨基酸的不同将其分为FengycinA和FengycinB两种类型,本研究中通过质谱检测分析发现菌株BA-16可代谢产生多种Fengycin同系物,其类型涵盖A、B两种类型。此外,HPLC纯化物质抑菌试验结果表明,Surfactin对扩展青霉的抑制效果不明显,Fengycin可能是菌株BA-16拮抗扩展青霉的主要物质,这有待于进一步的实验研究。
| [1] | Kim PI, Ryu J, Kim YH, et al. Production of biosurfactant lipopeptides Iturin A, fengycin and surfactin A from Bacillus subtilis CMB32 for control of Colletotrichum gloeosporioides[J]. Journal of Microbiology and Biotechnology , 2010, 20 (1) : 138–145. |
| [2] | Ongena M, Jourdan E, Adam A, et al. Surfactin and fengycin lipopeptides of Bacillus subtilis as elicitors of induced systemic resistance in plants[J]. Environmental microbiology , 2007, 9 (4) : 1084–1090. DOI:10.1111/emi.2007.9.issue-4 |
| [3] | Romero D, de Vicente A, Rakotoaly RH, et al. The iturin and fengycin families of lipopeptides are key factors in antagonism of Bacillus subtilis toward Podosphaera fusca[J]. Molecular Plant-Microbe Interactions , 2007, 20 (4) : 430–440. DOI:10.1094/MPMI-20-4-0430 |
| [4] | Waewthongrak W, Pisuchpen S, Leelasuphakul W. Effect of Bacillus subtilis and chitosan applications on green mold (Penicilium digitatum Sacc.) decay in citrus fruit[J]. Postharvest Biology and Technology , 2015, 99 : 44–49. DOI:10.1016/j.postharvbio.2014.07.016 |
| [5] | Afsharmanesh H, Ahmadzadeh M, Javan-Nikkhah M, et al. Improvement in biocontrol activity of Bacillus subtilis UTB1 against Aspergillus flavus using gamma-irradiation[J]. Crop Protection , 2014, 60 : 83–92. DOI:10.1016/j.cropro.2014.02.013 |
| [6] | Wang Y, Lu ZX, Bie XM, et al. Separation and extraction of antimicrobial lipopeptides produced by Bacillus amyloliquefaciens ES-2 with macroporous resin[J]. European Food Research and Technology , 2010, 231 (2) : 189–196. DOI:10.1007/s00217-010-1271-1 |
| [7] | Bonaterra A, Badosa E, Cabrefiga J, et al. Prospects and limitations of microbial pesticides for control of bacterial and fungal pomefruit tree diseases[J]. Trees , 2012, 26 (1) : 215–226. DOI:10.1007/s00468-011-0626-y |
| [8] | Pathak KV, Keharia H, Gupta K, et al. Lipopeptides from the banyan endophyte, Bacillus subtilis K1: mass spectrometric characterization of a library of fengycins[J]. Journal of the American Society for Mass Spectrometry , 2012, 23 (10) : 1716–1728. DOI:10.1007/s13361-012-0437-4 |
| [9] | Han XY, Chen ZH, Luo DQ, et al. Study on antimicrobial activity and stability of bioactive metabolite produced by tpb55 strain fermentation[J]. Chinese Agricultural Science Bulletin , 2012, 28 (27) : 260–264. (in chinese) 韩欣宇, 陈志厚, 罗定棋, 等. Tpb55菌株发酵液中活性代谢产物的抑菌作用及稳定性测定[J]. 中国农学通报 , 2012, 28 (27) : 260–264. |
| [10] | Wei W, Li SY, Wang MJ, et al. Probiotic lactic acid bacteria to fluoroquinolones sensitivity detection method[J]. Food Science and Technology , 2013, 38 (3) : 22–26. (in chinese) 韦婉, 李少英, 王梦姣, 等. 益生乳酸菌对氟喹诺酮类药物的敏感性检测方法的研究[J]. 食品科技 , 2013, 38 (3) : 22–26. |
| [11] | Huang WF, Zhang S. Microbiology Experiment Instruction[M]. Guangzhou: Ji'nan University Press, 2003: 122 -124. (in chinese) 黄文芳, 张松. 微生物学实验指导[M]. 广州: 暨南大学出版社, 2003: 122 -124. |
| [12] | Woo PCY, Lau SKP, Teng JLL, et al. Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories[J]. Clinical Microbiology and Infection , 2008, 14 (10) : 908–934. DOI:10.1111/j.1469-0691.2008.02070.x |
| [13] | Du ML, Fu RM, Gu YN, et al. Isolation, identification and breeding of petroleum-degrading bacteria in oilfield produced water by UV-plasma complex mutagenesis[J]. Microbiology China , 2015, 42 (6) : 1001–1009. (in chinese) 杜茂林, 付瑞敏, 谷亚楠, 等. 油田采出水石油烃降解菌分离、鉴定及高通量选育复合诱变菌株[J]. 微生物学通报 , 2015, 42 (6) : 1001–1009. |
| [14] | Caldeira AT, Arteiro JMS, Coelho AV, et al. Combined use of LC-ESI-MS and antifungal tests for rapid identification of bioactive lipopeptides produced by Bacillus amyloliquefaciens CCMI 1051[J]. Process Biochemistry , 2011, 46 (9) : 1738–1746. DOI:10.1016/j.procbio.2011.05.016 |
| [15] | Zhou XH, Lu ZX, Lü FX, et al. Antagonistic action of Bacillus subtilis strain fmbj on the postharvest pathogen Rhizopus stolonifer[J]. Journal of Food Science , 2011, 76 (5) . |
| [16] | Yánez-Mendizábal V, Usall J, Vi as I, et al. Potential of a new strain of Bacillus subtilis CPA-8 to control the major postharvest diseases of fruit[J]. Biocontrol Science and Technology , 2011, 21 (4) : 409–426. DOI:10.1080/09583157.2010.541554 |
| [17] | Chen H, Chen ZJ, Zhang YJ, et al. Identification of Antifungal Peptides from Bacillus subtilis BS-918[J]. Analytical Letters , 2014, 47 (18) : 2891–2899. DOI:10.1080/00032719.2014.928882 |
| [18] | Yu SM, Oh BT, Lee YH. Biocontrol of green and blue molds in postharvest satsuma mandarin using Bacillus amyloliquefaciens JBC36[J]. Biocontrol Science and Technology , 2012, 22 (10) : 1181–1197. DOI:10.1080/09583157.2012.719150 |
| [19] | Tanaka K, Ishihara A, Nakajima H. Isolation of anteiso-C17, iso-C17, iso-C16, and iso-C15 Bacillomycin D from Bacillus amyloliquefaciens SD-32 and Their Antifungal Activities against Plant Pathogens[J]. Journal of Agricultural and Food Chemistry , 2014, 62 (7) : 1469–1476. DOI:10.1021/jf404531t |
| [20] | Deng Y, Lu ZX, Lu FX, et al. Study on an antimicrobial protein produced by Paenibacillus polymyxa JSa-9 isolated from soil[J]. World Journal of Microbiology and Biotechnology , 2011, 27 (8) : 1803–1807. DOI:10.1007/s11274-010-0638-6 |
| [21] | Zamani-Zadeh M, Soleimanian-Zad S, Sheikh-Zeinoddin M, et al. Integration of Lactobacillus plantarum A7 with thyme and cumin essential oils as a potential biocontrol tool for gray mold rot on strawberry fruit[J]. Postharvest Biology and Technology , 2014, 92 : 149–156. DOI:10.1016/j.postharvbio.2014.01.019 |
| [22] | Pandya U, Saraf M. In vitro evaluation of PGPR strains for their biocontrol potential against fungal pathogens[M]//Kharwar RN, Upadhyay RS, Dubey NK, et al. Microbial Diversity and Biotechnology in Food Security. India: Springer, 2014, : 293-305 |
| [23] | Guilhabert-Goya M, Margolis JS. Synergistic combinations of polyene fungicides and non-ribosomal peptides and related methods of use: US, 8623813 [P]. 2014-07-01 |
| [24] | Lee GW, Ko JA, Oh BT, et al. Biological control of postharvest diseases of apples, peaches and nectarines by Bacillus subtilis S16 isolated from halophytes rhizosphere[J]. Biocontrol Science and Technology , 2012, 22 (3) : 351–361. DOI:10.1080/09583157.2012.658553 |
 2016, Vol. 43
2016, Vol. 43




