扩展功能
文章信息
- 费云燕, 盖钧镒, 赵团结
- FEI Yun-Yan, GAI Jun-Yi, ZHAO Tuan-Jie
- 突变肠杆菌株NRS-1中5个草甘膦逆境应答基因的克隆与功能研究
- Cloning and functional analysis of 5 glyphosate-response-genes in mutant Enterobacter NRS-1 strain
- 微生物学通报, 2016, 43(8): 1690-1698
- Microbiology China, 2016, 43(8): 1690-1698
- DOI: 10.13344/j.microbiol.china.150675
-
文章历史
- 收稿日期: 2015-09-10
- 接受日期: 2016-01-12
草甘膦为内吸传导型、非选择性的有机磷类除草剂,对许多一年生和多年生杂草有极强的控制能力[1],目前全球销量居首位,其广泛应用大大减少了杂草给农业带来的经济损失,抗草甘膦转基因育种是推动该类除草剂发展的动力。受aroA基因编码的5-烯醇式丙酮酸莽草酸-3-磷酸合成酶(EPSPS)是草甘膦的靶标酶,主要参与莽草酸途径,抗性aroA主要通过构象的改变使除草剂无法结合而阻止其对生物的伤害。
抗草甘膦生物中非靶基因的研究也受到一定的关注。目前发现调控草甘膦抗性的非靶基因包括C−N键氧化裂解基因,如草甘膦氧化还原酶基因GOX;参与裂解C−P键的C−P裂解酶;磷酸转移酶基因glpA、glpB和igrA,甘氨酸氧化酶基因GO;草甘膦N-乙酰转移酶基因GAT使草甘膦发生乙酰化,无法与EPSPS结合,将草甘膦转化为低毒的乙酰草甘膦;此外转运蛋白能够主动运输某些磷酸化合物,推测草甘膦也可能被转运蛋白运出细胞外[2-4]。
抗草甘膦基因被广泛应用于转基因育种研究,如Barry等将细菌中获得的GOX转入小麦获得了抗性植株[5];Pe aloza-Vázquez等也将磷酸转移酶基因转化烟草,提高了其抗性[6];谢龙旭等将抗性基因aroAM12转入棉花得到抗草甘膦的棉花[7]。在目前被挖掘的一系列抗性基因中,已被用于商业化转基因作物中的有aroA-cp4 (根癌土壤杆菌)、GOX (人苍白杆菌)、2mepsps (玉米的双突变EPSPS)、gat (地衣芽孢杆菌)、epsps (Ag) (球形节杆菌)、mepsps (玉米的经修饰的EPSPS)、epsps grg23ace5 [人工合成的类似于epsps (Ag)的基因]。
目前转基因抗草甘膦生物育种所用的基因主要是5-烯醇式丙酮酸莽草酸-3-磷酸合成酶,发掘和利用不同类型的抗草甘膦基因一直受到人们重视。一些研究也发现,微生物对草甘膦的抗性涉及大量调控不同层面抗性的相关基因,深入研究和利用这些基因具有重要的理论与应用价值[8]。本研究对抗草甘膦肠杆菌突变菌株NRS-1中5个草甘膦应答基因进行克隆研究,目前对这些基因与草甘膦的抗性研究较少,因此本实验期望通过相关实验初步了解这些基因在应对草甘膦逆境中的作用和应用潜力。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒: 肠杆菌NRS-1为草甘膦抗性菌株,农杆菌EHA105为植物表达菌株,本实验室保藏。大肠杆菌DH5α为质粒克隆宿主、大肠杆菌BL21为原核表达菌株,天根(北京)生化科技有限公司。PMDC83载体为本实验室保藏;PMD19载体、Pcold载体,日本TaKaRa公司。 1.1.2 培养基和抗生素: 研究中主要使用的培养基包括LB培养基[3]、YEB培养基和MS培养基[7]。主要抗生素包括氨苄青霉素50 mg/L、卡那霉素50 mg/L、潮霉素50 mg/L、利福平50 mg/L。 1.1.3 主要试剂: 限制性内切酶、T4 DNA连接酶、DNA凝胶回收试剂盒,生工生物(上海)工程有限公司;DNA聚合酶KOD-Plus,东洋纺(上海)生物科技有限公司;细菌双杂交载体试剂盒中所用菌株及质粒,重庆医科大学张雪梅、尹一兵教授惠赠;化学试剂过硫酸铵、甘氨酸、溴酚蓝、考马斯亮蓝、硫酸锌、氯化钙等均为国产分析纯或色谱纯,北京鼎国昌盛生物技术有限公司。 1.2 方法 1.2.1 应答高浓度草甘膦逆境重要差异表达基因的筛选与克隆: 根据肠杆菌NRS-1在60 g/L草甘膦处理下的基因芯片结果[8],按照预测的目标基因所在通路、作用特点进行筛选,结果从43个差异表达基因中筛选出5个基因用于研究,包括易位酶延伸因子基因fusA、丁二酸脱氢酶基因sdhA、胸苷磷酸化酶基因deoA、鸟氨酸氨甲酰转移酶基因argF、周质蛋白基因osmY。这些基因从不同层面参与抵御草甘膦逆境,osmY作用于细胞最表层,sdhA、deoA和argF属于代谢水平的保护,fusA则是与核糖体相关的保护。草甘膦的靶基因aroA[8]作为参照基因同样用于基因的功能验证。利用primer 3软件[9]进行引物的设计(表 1),DNA聚合酶KOD-Plus用于基因的PCR扩增,PCR反应体系及程序参照高保真聚合酶KOD-Plus简介说明书,PCR产物导入PMD19载体,详细步骤参照PMD19载体说明书,重组载体转化大肠杆菌DH5α,详细步骤参照大肠杆菌DH5α感受态说明书,获得的单克隆首先用琼脂糖胶检测,阳性克隆送上海英骏生物技术(上海)有限公司测序,获得序列在NCBI网站上进行BLAST比对,若为肠杆菌属的相应基因,相应单克隆提取重组质粒保留备用,同时获得的序列提交NCBI数据库。| 基因名 Symbol |
上游引物 Forward primer (5′→3′) |
下游引物 Reverse primer (5′→3′) |
| argF | ATTTTCACACACGGACGGG | ATCGGCATAATGCTGGCA |
| deoA | CACATTTCCCCCGATTCC | ATGTGGCCCATGGTATCAG |
| sdhA | TCGGTTTTTTCGCTACCAG | TTCATCCGCTTCCAGGG CGGCG |
| fusA | ATGACATATGGCTCGTACAACACCCATCG | ATTCTCGAGCTTACCACGGGCTTCGATAACG |
| osmY | ATGACTATGACAAGACTGAAGATTTCG | GCGTTAAAAATGATCTGAAAACTAAG |
| 基因名 Symbol |
上游引物 Forward primer (5′→3′) |
下游引物 Reverse primer (5′→3′) |
| fusA | CTGAAGTAACTGGCGTTAAG | GTGTATGATGCACGACCT |
| aroA | CGGAAATCGGCACCTATAAC | GCCGTACATTTCGGATCAAG |
| osmY | CGACGCTAAAGACACCTC | GGTTTCCACTTTCACCTTG |
| argF | GAAAGAGTTCGATCTGCACG | CACCATCACCGCCTTAAT |
| deoA | CGGCCCGATTGTGGATAAA | GATATCGAAGCCCGGAATAG |
| sdhA | CGTCTGGACGATACCTCA | TTCGGTACGGAAGTTTGC |
| actin2 | GTGGATTCCAGCAGCTTCCAT | GCTGAGAGATTCAGATGCCCA |
通过T-A克隆获得了fusA、sdhA、deoA、argF、osmY 5个目的基因及aroA基因(图 1)。在NRS-1中,其中fusA、osmY、deoA、sdhA、argF基因在高浓度草甘膦处理下为上调表达。其结构特点总结如下。
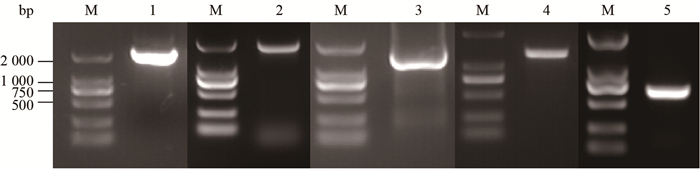
|
| 图 1 克隆基因的琼脂糖胶图 Figure 1 The agarose gel pictures of some cloned genes Note: M: Marker; 1: fusA; 2: sdhA; 3: deoA; 4: argF; 5: osmY. |
|
|
基因fusA (GenBank登录号KT366019)是易位酶延伸因子,开放阅读框为2 112 bp,编码704个氨基酸残基,等电点为4.90,帮助蛋白质翻译过程中tRNA和mRNA转移;在翻译结束时帮助tRNA的脱落;该基因还能够增强或抑制翻译过程[18]。除了参与蛋白质的合成,研究发现fusA能够作为分子伴侣[19],具有稳定蛋白质的作用。基因osmY (GenBank登录号KT366020)是功能未知的周质蛋白,开放阅读框为615 bp,编码205个氨基酸残基,等电点为5.16。它能够被高渗透压和生长速率改变表达水平,能够与磷脂相互作用,与质膜的空隙连接,防止胞质的皱缩[20]。基因deoA (GenBank登录号KT366022)为胸苷磷酸化酶基因,开放阅读框为1 332 bp;编码444个氨基酸残基;等电点为4.96。它可以将胸腺嘧啶转化成为胸苷,这些胸苷可以被用于合成氨基酸,这是含有胸腺嘧啶氨基酸的来源之一[21]。该基因也具有反向的功能,即将胸苷转化为胸腺嘧啶,这个过程帮助降解核苷[22],细胞在遇到逆境时,会发生一些错误,比如核苷合成发生错误,deoA可以帮助降解错误或多于的核苷。
基因sdhA (GenBank登录号KT366023)编码丁二酸脱氢酶,开放阅读框为1 764 bp,编码588个氨基酸残基,等电点为4.91。该酶位于质膜的内层,催化丁二酸和延胡索酸的互相转化[23];参与TCA循环和电子传递[24]。受细胞分裂和铁离子浓度的影响[25-26];此外高浓度的丁二酸能够降低超氧化物的浓度[27],因此sdhA下调或上调都有可能发挥作用。
基因argF (GenBank登录号KT366021)编码鸟氨酸氨甲酰转移酶,开放阅读框为1 002 bp;编码334个氨基酸残基;等电点为5.04。该转移酶利用鸟氨酸合成精氨酸,在各种生长环境中该基因都被反向调节,过多或过少的精氨酸或其他C源都会抑制argF的表达[28],该基因的下调一方面能够使鸟氨酸积累,具有抗渗透的作用,另一方面可以帮助合成其他氨基酸如脯氨酸、芳香族氨基酸等[29]。
基因aroA[8] (GenBank登录号KT366024)编码5-烯醇式丙酮酰莽草酸-3-磷酸合成酶,开放阅读框为1 281 bp,编码427个氨基酸残基,等电点为4.98。该基因参与莽草酸途径,与芳香族氨基酸(苯丙氨酸、酪氨酸、色氨酸)及其衍生物的合成相关,不仅影响细胞的代谢、蛋白质的合成,还参与细胞的抗逆。从已知的功能来看,克隆的6个基因都为逆境应答基因。
2.2 目的基因原核表达特点由于在原核表达实验中经常会出现蛋白质的包涵体而使基因失去功能,因此首先进行SDS-PAGE实验验证蛋白质的形成情况。从图 2可见,每个基因的第3个泳道与第2个对照泳道相比都有特异性的条带,说明基因在表达菌中全部能成功合成相应的蛋白质,fusA、sdhA、deoA、argF、osmY、aroA基因合成的蛋白大小分别是77.59、64.23、47.27、36.7、21.35、46.16 kD,与图 2中显示的特异性条带大小相似。所有基因在原核表达实验中都能够表达有功能的蛋白,可用于进一步的功能验证。

|
| 图 2 目的基因在E. coli中的表达情况 Figure 2 Genes expression in E. coli 注:M:Marker;1:诱导前对照;2:诱导后菌的总蛋白;3:总蛋白经超声波裂解后的沉淀;4:总蛋白经超声波裂解后的上清. Note: M: Marker; 1: the protein of E. coli before induction; 2: the protein of E. coli after induction; 3: precipitation of total protein after ultrasonic cracking; 4: supernatant of total protein after ultrasonic cracking. |
|
|
对正确含有重组质粒的大肠杆菌进行草甘膦的处理,24 h后测定OD600以观察其生长情况。结果表明含有6个基因的菌株OD600值都比对照大,对照的值约为0.14,而含有基因的菌株的最小值是sdhA为0.15,osmY最大,其OD600达0.4,因此可以推测体外表达这几个基因对微生物抵御草甘膦都有一定的作用,它们可以通过某种方式发挥作用,是否与NRS-1中的作用方式相同仍需进一步的验证。
2.3 转基因拟南芥的性状表现PCR及测序结果显示6个目的基因已被转到拟南芥基因组上(图 3A)。为验证基因对植物抗草甘膦是否起作用,对生长约14 d的转基因植株进行了草甘膦(终浓度0.5 mmol/L) 48 h处理。结果如图 3B所示,在草甘膦处理后,植株叶片出现失绿现象,但转不同基因拟南芥在草甘膦处理下的形态表现有所不同,其优劣顺序依次为:aroA、argF、deoA、sdhA、GFP (空载对照)、fusA、osmY。其中转aroA、argF、deoA基因的表型相差甚小,说明deoA、argF可能对拟南芥抗草甘膦有重要作用,转fusA、osmY基因的植株表型较对照组差,说明基因对拟南芥耐草甘膦无正向积极影响。

|
| 图 3 5个基因在植物中的表达 Figure 3 Genes expressed in plants 注:A:阳性拟南芥的分子鉴定;B:阳性拟南芥对草甘膦的耐性检测;C:阳性转基因拟南芥在草甘膦处理下目的基因的表达情况. M:Marker;1:fusA;2:sdhA;3:deoA;4:aroA;5:argF;6:osmY. Note: A: molecular identification of positive transgenic Arabidopsis thaliana; B: the phenotype of transgenic Arabidopsis thaliana in glyphosate stress; C: genes expression of transgenic Arabidopsis thaliana under glyphosate treatment. M: Marker; 1: fusA; 2: sdhA; 3: deoA; 4: aroA; 5: argF; 6: osmY. |
|
|
对处于草甘膦逆境下的转基因植株进行qPCR,检测相应基因的表达情况如图 3C所示。在草甘膦处理12 h时,过表达植物中6个基因aroA、argF、deoA、osmY、sdhA和fusA都表现为上调,将每一个基因的变化对应到基因的功能及其在草甘膦逆境下转基因植株的表型,含有基因fusA和osmY的转基因苗在草甘膦处理后,受到草甘膦的影响较大,fusA为微生物中的延伸因子,参与蛋白质转录翻译过程,osmY在微生物中与膜相关,两个基因可能对物种的特异性较强,在拟南芥中表达不能产生积极的抗逆效果,反而对细胞产生负担;基因aroA、argF和deoA在草甘膦处理0 h到12 h之间基因上调量的差异比较是aroA>argF≥deoA,这与草甘膦处理拟南芥后的表型有极大的相似性,说明aroA、argF和deoA基因对植物耐草甘膦具有一定的作用。
2.4 草甘膦应答基因与aroA靶基因的互作 2.4.1 基于KEGG分析的基因网络: 利用ClueGo程序进行KEGG分析,图 4显示了目的基因间的关系,不同的基因间通过参与不同的代谢途径连接形成一个小型的代谢网络。基因argF与aroA的相关性较大,有代谢的重叠,表明argF基因可能除了产物精氨酸对细胞抗草甘膦的作用外,还能够协助莽草酸途径抵御草甘膦;而aroA参与的过程与fusA、sdhA、deoA、osmY的关系较远,主要为间接的关系。推测本文获得的基因所参与的途径可能在不同程度上影响芳香族氨基酸及其衍生物的功能,由于NRS-1长期处于草甘膦逆境下,其耐性进化有着不可预测的一面,因此不排除基因改变其功能直接与莽草酸途径相关,从某种程度上说,这些基因可能帮助NRS-1细胞应答草甘膦存在的混合逆境,以及参与抵御草甘膦的毒性。
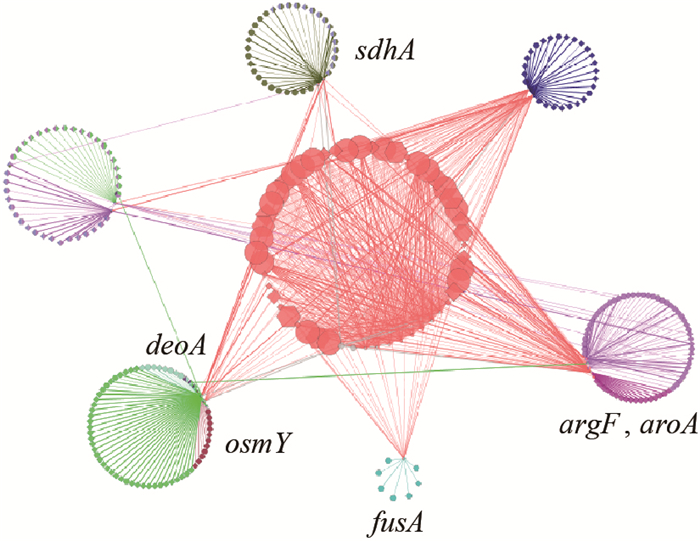
|
| 图 4 基因芯片中差异基因的关系 Figure 4 The relationships of different expressed genes in Genechip 注:相同颜色的线条表明代谢途径或生物过程相同. Note: Lines with same colour mean the identical metabolic pathway or bioprocess. |
|
|

|
| 图 5 细菌双杂实验中的Western blot图以验证基因能否在表达菌中表达 Figure 5 Western blot of bacterial two hybrid experiments to validate the gene expression in bacteria |
|
|
在基因芯片中上调表达的fusA和osmY在拟南芥中比对照组对草甘膦更敏感[8],尤其是osmY,可能是由植物细胞膜或翻译等过程与微生物的差异较大,另外细胞额外的表达功能不大的基因会浪费细胞资源,造成细胞的负担。基因argF和sdhA在NRS-1中表现为下调[8],但在转基因拟南芥中argF或sdhA对植物抵御草甘膦的过程效果较好,两个基因都为双功能的基因,不管为何种差异变化,对抵御逆境都有一定的作用。基因在植物中功能验证的结果说明草甘膦影响到转基因植株氨基酸的合成或利用,其中芳香族氨基酸对拟南芥抗草甘膦有较大的作用,其次是含有胸腺嘧啶的相关氨基酸,最后是精氨酸。此部分实验直接体现了拟南芥中目的基因对植物抗草甘膦的作用。
3.2 耐草甘膦非靶标基因的利用潜力抗草甘膦基因可分为靶基因和非靶标基因,基因资源的获得是抗草甘膦转基因育种的关键。aroA是草甘膦的靶基因,在细胞对草甘膦的耐性中起着重要作用,除获得一些抗性的靶基因外,非靶标基因的研究也有所必要。目前抗草甘膦的非靶标基因挖掘较少,主要的基因包括GAT、GOX、glpA、glpB、igrA、GO[4],其中只有GOX、gat被用于商业化转基因作物。我国在此方面基因的挖掘上有一些研究,但主要集中于获得高效的靶基因,为拓宽相关研究领域,加速抗草甘膦的育种,对生物抗草甘膦机制进一步的了解,非靶基因的研究也应该有一定的发展。
本研究中,fusA、sdhA、deoA、argF、osmY从功能上看都是逆境应答基因,参与酸、渗透、氧化胁迫等,同时作为草甘膦应答基因,是细胞进入新稳态时调用的逆境特异性的应答基因[30]。实验证明克隆获得的5个非靶标基因通过复杂的代谢网络,与草甘膦的靶基因aroA以间接的方式联系,通过莽草酸途径参与细胞抗草甘膦的过程。在拟南芥和大肠杆菌中对目的基因进行抗草甘膦的功能验证,表明这些基因在两种生物中均对细胞抗草甘膦有一定的作用,可能经过某种改造,仍能够具有高效抗草甘膦的作用,更深的调控机制研究仍需进一步的进行。
| [1] | Lorentz L, Beffa R, Kraehmer H. Recovery of plants and histological observations on advanced weed stages after glyphosate treatment[J]. Weed Research , 2011, 51 (4) : 333–343. DOI:10.1111/wre.2011.51.issue-4 |
| [2] | Zhu JW. Physiological and biochemical characteristics and glyphosate tolerance of alligator weed (alternanthera philoxeroides) under manganese stress[D]. Hangzhou: Doctoral Dissertation of Zhejiang University, 2008 (in Chinese) 朱金文.高锰胁迫下空心莲子草的生理生化特性和草甘膦耐性研究[D].杭州:浙江大学博士学位论文, 2008 http://cdmd.cnki.com.cn/article/cdmd-10335-2009156588.htm |
| [3] | Li L. Characterization of the functional domains among classⅡEPSP synthases and microarray analysis of the glyphosate stress response in Escherichia coli[D]. Beijing: Doctoral Dissertation of Chinese Academy of Agriculture Sciences, 2010 (in Chinese) 李亮.Ⅱ型EPSP合酶的功能域鉴定及大肠杆菌在草甘膦冲击下的基因表达谱分析[D].北京:中国农业科学院博士学位论文, 2010 |
| [4] | Pollegioni L, Schonbrunn E, Siehl D. Molecular basis of glyphosate resistance-different approaches through protein engineering[J]. FEBS Journal , 2011, 278 (16) : 2753–2766. DOI:10.1111/j.1742-4658.2011.08214.x |
| [5] | Barry G, kishore G, Padgette S, et al. Inhibitors of amino acid biosynthesis: strategies for imparting glyphosate tolerance to crop plants[J]. Current Topics in Plant Physiology (USA) , 1992 : 139–145. |
| [6] | Pe aloza-Vázquez A, Oropeza A, Mena GL, et al. Expression of the hygromycin B phosphotransferase gene confers tolerance to the herbicide glyphosate[J]. Plant Cell Reports , 1995, 14 (8) : 482–487. |
| [7] | Xie LX, Li YF, Xu PL. Glyphosate-resistant cotton (Gossypium hirsutum L.) transformed with aroAM12 gene via Agrobacterium tumefaciens[J]. Journal of Plant Physiology and Molecular Biology, 2004, 30(2): 173-178 (in Chinese) 谢龙旭, 李云锋, 徐培林.根癌农杆菌介导的转aroAM12基因棉花植株的草甘膦抗性[J].植物生理与分子生物学学报, 2004, 30(2): 173-178 http://www.cnki.com.cn/Article/CJFDTotal-ZWSI200402008.htm |
| [8] | Fei YY, Gai JY, Zhao TJ. Identification of regulated genes conferring resistance to high concentrations of glyphosate in a new strain of Enterobacter[J]. FEMS Microbiology Letters , 2013, 349 (2) : 135–143. DOI:10.1111/fml.2013.349.issue-2 |
| [9] | Untergasser A, Cutcutache I, Koressaar T, et al. Primer3-new capabilities and interfaces[J]. Nucleic Acids Research , 2012, 40 (15) : e115. DOI:10.1093/nar/gks596 |
| [10] | Sch gger H. Tricine-SDS-PAGE[J]. Nature Protocols , 2006, 1 (1) : 16–22. DOI:10.1038/nprot.2006.4 |
| [11] | Karimi M, InzéD, Depicker A. GATEWAY™ vectors for Agrobacterium-mediated plant transformation[J]. Trends in Plant Science , 2002, 7 (5) : 193–195. DOI:10.1016/S1360-1385(02)02251-3 |
| [12] | Martinez-Trujillo M, Limones-Briones V, Cabrera-Ponce JL, et al. Improving transformation efficiency of Arabidopsis thaliana by modifying the floral dip method[J]. Plant Molecular Biology Reporter , 2004, 22 (1) : 63–70. DOI:10.1007/BF02773350 |
| [13] | Pfaffl MW. A new mathematical model for relative quantification in real-time RT-PCR[J]. Nucleic Acids Research , 2001, 29 (9) : e45. DOI:10.1093/nar/29.9.e45 |
| [14] | Bindea G, Mlecnik B, Hackl H, et al. ClueGO: a Cytoscape plug-in to decipher functionally grouped gene ontology and pathway annotation networks[J]. Bioinformatics , 2009, 25 (8) : 1091–1093. DOI:10.1093/bioinformatics/btp101 |
| [15] | Joung JK, Ramm EI, Pabo CO. A bacterial two-hybrid selection system for studying protein-DNA and protein-protein interactions[J]. Proceedings of the National Academy of Sciences of the United States of America , 2000, 97 (13) : 7382–7387. DOI:10.1073/pnas.110149297 |
| [16] | Xu C, Ma X, Chen SW, et al. Bacterial cellulose membranes used as artificial substitutes for dural defection in rabbits[J]. International Journal of Molecular Sciences , 2014, 15 (6) : 10855–10867. DOI:10.3390/ijms150610855 |
| [17] | Cai YY. Screening and identification of DnaJ interacting proteins in Streptococcus pneumoniae[D]. Chongqing: Master's Thesis of Chongqing Medical University, 2013 (in Chinese) 蔡莺莺.肺炎链球菌热休克蛋白DnaJ相互作用蛋白的筛选及鉴定[D].重庆:重庆医科大学硕士学位论文, 2013 http://cdmd.cnki.com.cn/article/cdmd-10631-1014109327.htm |
| [18] | Długosz M, Huber GA, McCammon JA, et al. Brownian dynamics study of the association between the 70S ribosome and elongation factor G[J]. Biopolymers , 2011, 95 (6) : 616–627. |
| [19] | Len ACL, Harty DWS, Jacques NA. Stress-responsive proteins are upregulated in Streptococcus mutans during acid tolerance[J]. Microbiology , 2004, 150 (Pt 5) : 1339–1351. |
| [20] | Oh JT, Cajal Y, Skowronska EM, et al. Cationic peptide antimicrobials induce selective transcription of micF and osmY in Escherichia coli[J]. Biochimica et Biophysica Acta (BBA)-Biomembranes , 2000, 1463 (1) : 43–54. DOI:10.1016/S0005-2736(99)00177-7 |
| [21] | Wheeler PR. Biosynthesis and scavenging of pyrimidines by pathogenic mycobacteria[J]. Journal of General Microbiology , 1990, 136 : 189–201. DOI:10.1099/00221287-136-1-189 |
| [22] | Spinazzola A, Marti R, Nishino I, et al. Altered thymidine metabolism due to defects of thymidine phosphorylase[J]. The Journal of Biological Chemistry , 2002, 277 : 4128–4133. DOI:10.1074/jbc.M111028200 |
| [23] | Guest JR. Partial replacement of succinate dehydrogenase function by phage-and plasmid-specified fumarate reductase in Escherichia coli[J]. Journal of General Microbiology , 1981, 122 (2) : 171–179. |
| [24] | Johnson AS, van Horck S, Lewis PJ. Dynamic localization of membrane proteins in Bacillus subtilis[J]. Microbiology , 2004, 150 : 2815–2824. DOI:10.1099/mic.0.27223-0 |
| [25] | Righelato RC. The distrubution of iron in iron-deficient toxin-synthesizing and in excess-iron non-toxin-synthesizing Corynebacterium diphtheriae[J]. Journal of General Microbiology , 1969, 58 (3) : 411–419. DOI:10.1099/00221287-58-3-411 |
| [26] | Meredith DH, Plank M, Lewis PJ. Different patterns of integral membrane protein localization during cell division in Bacillus subtilis[J]. Microbiology , 2008, 154 : 64–71. DOI:10.1099/mic.0.2007/013268-0 |
| [27] | Franchini AG, Egli T. Global gene expression in Escherichia coli K-12 during short-term and long-term adaptation to glucose-limited continuous culture conditions[J]. Microbiology , 2006, 152 : 2111–2127. DOI:10.1099/mic.0.28939-0 |
| [28] | Voellmy R, Leisinger T. Regulation of enzyme synthesis in the arginine biosynthetic pathway of Pseudomonas aeruginosa[J]. Journal of General Microbiology , 1978, 109 (1) : 25–35. DOI:10.1099/00221287-109-1-25 |
| [29] | Weglenski P. The mechanism of action of proline suppressors in Aspergillus nidulans[J]. Journal of General Microbiology , 1967, 47 (1) : 77–85. DOI:10.1099/00221287-47-1-77 |
| [30] | Kreps JA, Wu Y, Chang HS, et al. Transcriptome changes for Arabidopsis in response to salt, osmotic, and cold stress[J]. Plant Physiology , 2002, 130 (4) : 2129–2141. DOI:10.1104/pp.008532 |
 2016, Vol. 43
2016, Vol. 43




