扩展功能
文章信息
- 李风娥, 马艳芳, 韩明渠, 毛志泉, 束怀瑞, 高峥, 周波
- LI Feng-E, MA Yan-Fang, HAN Ming-Qu, MAO Zhi-Quan, SHU Huai-Rui, GAO Zheng, ZHOU Bo
- 不同种植年限苹果根际土可培养根皮苷耐受菌的分布及多样性
- Distribution and diversity of culturable phlorizin-resistant bacteria from apple rhizosphere soil in different planting years
- 微生物学通报, 2016, 43(7): 1499-1512
- Microbiology China, 2016, 43(7): 1499-1512
- 10.13344/j.microbiol.china.150915
-
文章历史
- 收稿日期: 2015-11-13
- 接受日期: 2016-04-20
- 优先数字出版日期(www.cnki.net): 2016-05-10
2. 山东农业大学园艺科学与工程学院 山东 泰安 271018;
3. 山东农业大学 土肥资源高效利用国家工程实验室 山东 泰安 271018
2. College of Horticulture Science and Engineering,Shandong Agricultural University,Tai’an,Shandong 271018,China;
3. National Engineering Laboratory for Efficient Utilization of Soil and Fertilizer Resources,Shandong Agricultural University,Tai’an,Shandong 271018,China
连作障碍是指在同一块土壤先后种植同种或同科作物的过程中表现出明显的病态,如植株矮小、叶片发黄、生长不良、抗逆性差、成活率低甚至死亡的现象[1]。连作障碍形成及加重发生的原因包括土壤的传染性病害、土壤理化性状劣变、根系分泌物和残茬分解物等引起的自毒作用。其中,酚酸类自毒物质是造成植物连作障碍的重要因素[2, 3, 4, 5]。Börner首次提出苹果植株残体分解以及根系分泌到土壤中的根皮苷、根皮素、肉桂酸、对羟基安息香酸、间苯三酚等是苹果连作障碍发生的重要因子[6, 7]。根皮苷(Phlorizin,C21H24O10)是苹果属植物特有和主要的酚酸类自毒物质,多年生苹果果园根际土酚酸含量的动态变化与树体的生长具有一致性[8]。研究发现低浓度(0.001 mmol/L)根皮苷促进平邑甜茶幼苗生长,高浓度的根皮苷(1 mmol/L)通过降低光合速率和影响干物质积累,抑制平邑甜茶幼苗生长,且随着处理浓度增加,抑制作用增强[9]。Hofmann等通过对连作土壤和健康土壤中苹果幼苗根系分泌物的研究发现,根皮苷是苹果根系分泌物的主要酚酸物质;在连作障碍发生初期,连作土壤中苹果根系根皮苷分泌量高于健康土壤,且与根系干物质量显著相关[10]。Yim等也认为酚酸等物质的降解可能会促进植物的生长[11]。因而缓解高浓度根皮苷对苹果的自毒作用或可成为破解苹果连作障碍的一条有效途径。土壤微生物的种类繁多,数量庞大,以其强大的降解转化能力及多样的生物活性,在自然界物质循环中扮演着重要的角色,是从自毒物质降解角度进行连作障碍生物防治的重要潜在资源。因此从土壤微生物资源中筛选具有高效根皮苷降解功能的微生物具有重要意义。
尽管近几年人们在连作障碍中微生物群落组成方面进行了深入的研究[11, 12, 13],以期在一定程度上揭示连作障碍的机制,但目前对于根皮苷降解微生物的研究进展仍然较慢。黄园勇等分离获得630株放线菌,通过以根皮苷为唯一碳源初筛后,获得能降解转化根皮苷的放线菌29株,其中具有较高降解活性的菌株有4株,分别为链霉菌属、贪噬菌属、红球菌属、小单孢菌属[14]。张一等筛选到一株具有较高根皮苷降解效果的菌株,属于粘质沙雷菌属,对平邑甜茶的根系及地上部分无明显负作用,且对其生长发育有一定的促进作用[15]。因此,了解不同年限苹果根际土根皮苷耐受菌的群落分布及多样性,阐明根皮苷耐受菌群落与土壤理化因子的关系,对于获得高效根皮苷降解菌株,揭示和解决苹果连作障碍具有重要的指导意义。
黄土高原地区地处中国干旱和半干旱地区,气候干燥少雨,是我国四大苹果优势产区之一,其果园面积大,以乔化居多,如今这些果园已陆续进入更新改造期,而很多新果园是在老果园基础上进行换茬再植,则会发生连作障碍,成为苹果园更新的主要限制因素。本文以黄土高原苹果产区不同种植年限苹果树的根际土壤样品为研究对象,对根皮苷耐受菌进行筛选,通过ARDRA酶切聚类和16S rRNA基因序列分析确定分离的根皮苷耐受菌的分类地位,并通过生物信息学的分析方法对可培养根皮苷耐受菌的菌群分布及多样性进行研究,有助于理解根皮苷耐受菌与土壤理化因子的相关关系,为探索高效菌株的获得提供科学依据,以期为苹果连作障碍提供基础资料。
1 材料与方法 1.1 培养基和试剂10 mmol/L根皮苷液体培养基(g/L):根皮苷4.8,磷酸二氢铵1.0,氯化钾0.2,硫酸镁0.2,定容至1 L,pH 7.0。
根皮苷,Aladdin-上海阿拉丁生化科技股份有限公司;细菌基因组DNA提取试剂盒,天根生化科技(北京)有限公司;E.Z.N.A. Cycle-Pure Kit PCR产物纯化试剂盒,Omega Bio-Tek (美国);Msp Ⅰ、Hae Ⅲ,宝生物工程(大连)有限公司。
1.2 样品采集及理化性质测定黄土高原苹果(Malus Mill.)产区土壤样品于2012年7月16日−18日采集自陕西省延安市洛川果树实验站。该实验站占地面积约0.27 km2,栽植不同年限的同类果树,产区立地条件、管理模式相同,是研究苹果根际微生态结构和多样性的优质材料。在果树实验站,选择不同种植年限的苹果园,每个果园选择3棵苹果树,用抖落法取中根(0− 20 cm)的土壤,用力将根表面附着的土壤全部抖落下来,获得根际土壤,混合均匀,过2 mm孔径的筛子,装入样品袋中,4 °C保存,用于根皮苷耐受菌的分离及不同年限根皮苷耐受菌群落多样性的分析;取其根围土壤,混合均匀后装入样品袋中,用于土壤理化指标、酚酸含量测定。详细信息见表 1。
| 苹果树树龄 Age of the apple trees | 砧木类型 Type of rootstock | 品种 Cultivars | 地理位置 Geographical position |
| 3 | Vigorous rootstock of Malus baccata (Linn.) Borkh. | Fuji | 35°47′20.94′′N,109°21′45.07′′E |
| 8 | Vigorous rootstock of Malus baccata (Linn.) Borkh. | Fuji | 35°47′23.10′′N,109°21′43.41′′E |
| 15 | Dwarfing rootstock of Malus baccata (Linn.) Borkh. | Fuji | 35°47′10.42′′N,109°21′58.35′′E |
| 20 | Vigorous rootstock of Malus baccata (Linn.) Borkh. | Fuji | 35°47′03.01′′N,109°21′45.43′′E |
| 25 | Vigorous rootstock of Malus baccata (Linn.) Borkh. | Fuji | 35°47′01.42′′N,109°21′47.84′′E |
土壤样品自然风干,过筛处理后测定各项指标。全氮采用凯氏常量定氮法,有机质采用重铬酸钾容量法-外加热法测定,速效磷采用0.5 mol/L碳酸氢钠浸提-分光光度法测定,速效钾采用醋酸铵浸提-火焰光度法测定,有效铁、锰、铜、锌采用0.1 mol/L盐酸浸提-原子吸收法测定,水溶性钙、镁采用5:1水土比-EDTA滴定法测定,土壤与0.01 mol/L氯化钙按1:2.5比例浸提,采用酸度计测定土壤pH。土壤酚酸测定采用ASE-HPLC法[16]。
1.3 根皮苷耐受菌的筛选(1) 以根皮苷为唯一碳源的根皮苷耐受菌的富集。取不同年限苹果树根际土壤1 g,分别装入到99 mL 10 mmol/L根皮苷液体培养基中,30 °C、150 r/min培养5 d;取1 mL培养液加入到另一新的99 mL 10 mmol/L根皮苷液体培养基中,30 °C、150 r/min培养5 d,重复3次。
(2) 以根皮苷为唯一碳源的根皮苷耐受菌的分离及纯化。取富集后土壤样品的悬浮液,十倍稀释至10−7,共7个梯度,将稀释后的溶液各取100 μL均匀涂布于10 mmol/L根皮苷固体培养基中,3个平行,30 °C培养观察菌的生长情况。24−96 h挑取培养基中的单菌落进行划线纯化,4 °C保存备用。
1.4 基因组DNA的提取及细菌16S rRNA基因PCR扩增基因组DNA使用天根公司的细菌基因组DNA提取试剂盒(DP302),每个样品使用1−5 mL活化的菌液,依照试剂盒说明提取细菌基因组,获得的DNA通过琼脂糖凝胶电泳(1.0%)检测后−20 °C保存。
以提取的细菌基因组DNA为模板进行16S rRNA基因PCR扩增,引物为27F (5′-AGAGTTTGATCMTGGCTCAG-3′)和1492R (5′-GGYTACCTTGTTACGZCTT-3′)[17]。反应体系:10×PCR 缓冲液(Mg2+ plus) 5 μL,dNTPs mixture (2.5 mmol/L) 4 μL,rTaq酶(5 U/μL) 0.2 μL,引物(10 mmol/L)各1 μL,模板DNA 1 μL,灭菌双蒸水补足至50 μL。PCR反应条件:95 °C 5 min;94 °C 1 min,55 °C 1 min,72 °C 90 s,30个循环;72 °C 10 min。PCR产物使用E.Z.N.A. Cycle-Pure Kit进行纯化,纯化后使用1.0%琼脂糖电泳检测后于−20 °C保存备用。
1.5 ARDRA分析利用MspⅠ、Hae Ⅲ两种限制性内切酶对纯化后的PCR产物进行酶切。酶切反应体系(20 μL):MspⅠ 5 U,10×T Buffer 2 μL,0.1% BSA 2 μL,DNA≤1 μg;Hae Ⅲ 5 U,10×M Buffer 2 μL,DNA≤ 1 μg。酶切反应条件:37 °C酶切过夜,70 °C 15 min终止反应。酶切产物用2.5%的琼脂糖凝胶电泳检测。合并MspⅠ、Hae Ⅲ两种酶切图谱中≥100 bp的条带,利用美国Bio-Rad公司的Quanity one 4.62软件进行聚类分析,构建UPGMA树状图。
1.6 16S rRNA基因序列测序根据ARDRA图谱分析结果,选择代表菌株,将其PCR产物送至华大基因公司测序。将获得菌株的16S rRNA基因序列通过EzBio Cloud数据库(htttp://www.ezbiocloud.net/eztaxon)进行比对,并进行GenBank注册,注册号为KT452766−KT452783。利用MEGA 5.1软件[18]采用邻接法(Neighbor-Joining)构建系统发育树。
1.7 生物信息学分析韦恩图(Venn diagram)利用R软件(https://www.r-project.org/)中的VennDiagram程序包进行。主坐标分析(Principal coordinates analysis,PCoA)和多样性指数利用Paleontological Statistics (PAST)软件进行分析。皮尔森相关性分析(Pearson correlations analyses)使用Statistical Product and Solution Statistics (SPSS 19.0) for windows。冗余分析(Redundancy analysis,RDA)利用CANOCO for Windows软件(ver. 4.5)分析根皮苷耐受菌群落结构与不同年限苹果连作土的理化因子之间的关系。
2 结果与分析 2.1 不同种植年限苹果根际土的理化性质黄土高原苹果园土壤理化因子数据见表 2。土壤pH范围为8.40−8.53,表明黄土高原苹果园土壤呈弱碱性,与该地区降水较少,缺乏灌溉条件有关。种植年限为3年的苹果园土壤C/N最低,其他4个年份相差较小。种植年限为3年和8年的苹果树园土壤中有效磷含量高于20年和25年;相反,3年和8年生苹果树根围土壤中速效钾含量较低;微量元素中,Cu2+、Zn2+、Fe3+、Mn2+的含量以种植15年和25年的苹果树根围土壤中含量较多,种植3年和8年根围土壤样品中含量较低,推测与果园常年施用铜离子杀菌剂以及补充微量元素肥料等农艺措施,具有一定的相关性。
| Physicochemical parameters | 3 a | 8 a | 15 a | 20 a | 25 a |
| pH | 8.43 | 8.40 | 8.40 | 8.45 | 8.53 |
| C (g/kg) | 4.72 | 4.25 | 4.75 | 3.57 | 4.07 |
| N (g/kg) | 0.59 | 0.50 | 0.55 | 0.41 | 0.48 |
| P (mg/kg) | 8.35 | 5.70 | 12.23 | 4.21 | 4.66 |
| K (mg/kg) | 28.49 | 15.90 | 144.30 | 61.22 | 73.81 |
| Cu (mg/kg) | 1.16 | 1.50 | 2.31 | 1.08 | 2.59 |
| Zn (mg/kg) | 0.44 | 0.45 | 0.82 | 0.50 | 1.04 |
| Fe (mg/kg) | 7.80 | 8.71 | 9.37 | 7.82 | 10.02 |
| Mn (mg/kg) | 11.48 | 10.77 | 12.47 | 10.59 | 12.42 |
| Ca (mg/kg) | 32.15 | 30.43 | 13.93 | 42.11 | 30.53 |
| Mg (mg/kg) | 1.74 | 1.88 | 1.56 | 2.29 | 1.83 |
| C/N | 8.00 | 8.50 | 8.64 | 8.71 | 8.48 |
| Phloroglucinol (ng/100 g) | 180 425.37 | 85 100.92 | 135 686.71 | 0.00 | 97 048.34 |
| Catchin (ng/100 g) | 848.87 | 580.82 | 426.18 | 525.04 | 1 271.62 |
| Chlorogenic acid (ng/100 g) | 38.86 | 17.61 | 62.91 | 24.30 | 25.40 |
| Caffeic acid (ng/100 g) | 308.88 | 243.04 | 387.96 | 257.10 | 325.09 |
| P-hydroxybenzoic acid (PHBA) (ng/100 g) | 0.00 | 0.00 | 0.00 | 789.54 | 1 279.14 |
| Syringic acid (ng/100 g) | 0.00 | 0.00 | 0.00 | 0.00 | 45.72 |
| Ferulic acid (ng/100 g) | 26.04 | 28.41 | 24.10 | 0.00 | 0.00 |
| Salicylic acid (SA) (ng/100 g) | 137.25 | 233.93 | 1 919.99 | 218.88 | 0.00 |
| Phloridzin (ng/100 g) | 0.00 | 0.00 | 2 830.91 | 506.10 | 391.24 |
| Quercetin (ng/100 g) | 522.82 | 514.82 | 2 419.31 | 186.05 | 1 013.03 |
| Phloretin (ng/100 g) | 417.51 | 667.46 | 0.00 | 0.00 | 0.00 |
以根皮苷为唯一碳源,采用富集驯化培养和稀释涂布分离法,从不同生长年限苹果园根际土壤中筛选出103株根皮苷耐受菌。
对全部根皮苷耐受菌菌株的酶切图谱进行聚类分析,结果表明103株细菌菌株在65%[19]的相似性水平上可分为18个群(图 1)。分别从18个群中各选择一个作为代表性菌株,将所测菌株的16S rRNA基因序列进行EzBioCloud序列比对,并确定其分类地位(表 3)及构建根皮苷耐受菌系统发育树(图 2)。由表 3与图 2可知,18株代表菌株与模式菌株的相似性为97.58%−100%,其中Group 1、9、10、13分别与Rhizobium radiobacter ATCC 19358T、Bacillus aryabhattai B8W22T、Bacillus cereus ATCC 14579T、Acinetobacter calcoaceticus DSM 30006T的相似性为100%,而Group 3与Sphingobium aromaticiconvertens DSM 12677T的相似性较低,为97.58%。结合酶切图谱和表 3可知,筛选的根皮苷耐受菌属在数量上有一定差异,最多的是芽孢杆菌属,共24株(23.3%);最少的是奇异芽孢杆菌属、不动杆菌属和葡萄球菌属,为单菌株(0.97%)。

|
| 图 1 根皮苷耐受细菌的ARDRA聚类树状图 Fig. 1 Dendrogram shows the relationship of 103 phlorizin-resistant bacteria based on ARDRA fingerprints |

|
| 图 2 根据18株根皮苷耐受菌的16S rRNA基因序列构建的系统发育树 Fig. 2 Neighbor-joining (N-J) phylogenetic tree of the 18 group based on 16S rRNA gene sequences 注:括号内为GenBank登录号;分支上的数字为自展值百分比且大于50%;线段0.02为核苷酸替换率;以放线菌属为外围. Note: Numbers in parentheses represent accession numbers in GenBank; Bootstrap values of the Neighbor-Joining (NJ) are shown above the internodes and only values above 50% are given; Bar,0.02 substitutions per nucleotide position; The tree was rooted with Micrococcus luteus NCTC 2665T as an out group. |
| 门 Phylum | 属 Genus | 不同类群 Groups | 代表菌株 Representative strain | 最相似菌株 The most similar strain | 相似度 Similarity (%) | 菌株数目 Number of isolates in 5 years |
| Proteobacteria | Rhizobium | group 1 | 61 | Rhizobium radiobacter ATCC 19358T | 100 | 7 |
| Ancylobacter | Group 2 | 102 | Ancylobacter defluvii SK15T | 99.85 | 2 | |
| Sphingobium | Group 3 | 43 | Sphingobium aromaticiconvertens DSM 12677T | 97.58 | 8 | |
| Novosphingobium | Group 4 | 5 | Novosphingobium barchaimii LL02T | 99.25 | 8 | |
| Group 5 | 29 | Novosphingobium lindaniclasticum LE124T | 99.30 | 9 | ||
| Cupriavidus | Group 12 | 69 | Cupriavidus necator N-1T | 99.37 | 2 | |
| Acinetobacter | Group 13 | 64 | Acinetobacter calcoaceticus DSM 30006T | 100 | 1 | |
| Enterobacter | Group 14 | 24 | Enterobacter cancerogenus LMG 2693T | 99.47 | 10 | |
| Rhizobacter | Group 15 | 103 | Rhizobacter gummiphilus NS21T | 99.84 | 2 | |
| Variovorax | Group 16 | 16 | Variovorax boronicumulans BAM-48T | 99.85 | 8 | |
| Pseudomonas | Group 17 | 92 | Pseudomonas hunanensis LVT | 99.92 | 2 | |
| Group 18 | 11 | Pseudomonas umsongensis Ps 3-10T | 99.86 | 18 | ||
| Firmicutes | Fictibacillus | Group 6 | 84 | Fictibacillus barbaricus V2-BIII-A2T | 99.71 | 1 |
| Bacillus | Group 8 | 87 | Bacillus acidiceler CBD 119T | 99.55 | 1 | |
| Group 9 | 70 | Bacillus aryabhattai B8W22T | 100 | 9 | ||
| Group 10 | 40 | Bacillus cereus ATCC 14579T | 100 | 3 | ||
| Group 11 | 72 | Bacillus amyloliquefaciens subsp. plantarum FZB42T | 99.93 | 11 | ||
| Staphylococcus | Group 7 | 21 | Staphylococcus hominis subsp. novobiosepticus GTC 1228T | 99.86 | 1 |
研究发现,根皮苷耐受菌主要存在于变形杆菌门(74.8%)和厚壁菌门(25.2%),分属13个属,其中芽孢杆菌属数量最多,占菌株总数的23.3%,其次是假单胞菌属(19.4%)和新鞘脂菌属(16.5%),是耐受根皮苷的优势菌群(图 3a)。根皮苷耐受菌的优势菌群随年份不同而存在差异,种植3年苹果根际土分离的根皮苷耐受菌的优势菌种是假单胞菌属(36.7%),8年是肠杆菌属(50%),15年为新鞘脂菌属(20%),20年为芽孢杆菌属(52.4%),25年为鞘脂菌属(26.9%)和新鞘脂菌属(26.9%)(图 3b)。与其他3个年份相比,在属水平上种植年限为8年和20年苹果根际土分离的根皮苷耐受菌物种组成较简单。为了统计不同年份之间根皮苷耐受菌的组成情况,我们基于根皮苷耐受菌属水平绘制了韦恩图(图 4),结果表明,中间区域(F区)的2个属的菌种分布较广泛,分别为新鞘脂杆菌属和贪噬菌属,在5个不同的年限均有分布;种植3、8和25年的土壤中均分离到其特有的菌株。其中种植3年特有的根皮苷耐受菌属是贪铜菌属和不动杆菌属(A区),8年的是葡萄球菌属(B区),25年的是奇异芽孢杆菌(E区)。

|
| 图 3 根皮苷耐受菌群落分布 Fig. 3 The community distribution of phlorizin-resistant bacteria 注:a:根皮苷耐受菌不同属之间的分布;b:根皮苷耐受菌在各个年份属水平的变化. Note: a: The distribution of phlorizin-resistant bacteria from different genera; b: Changes of phlorizin-resistant bacteria at the genus level from different types of soils. |

|
| 图 4 不同年限根皮苷耐受菌属水平的分布 Fig. 4 Overlap of the genus level bacterial communities from five different years’ soils 注:圆圈内的数字代表不同年限分离到的根皮苷耐受菌属的数目. A:3年样品特有的菌属数目;B:8年样品特有的菌属数目;C:15年样品特有的菌属数目;D:20年样品特有的菌属数目;E:25年样品特有的菌属数目;F:5个样品共有的菌属数目. Note: The number in the circles represents the total number of genera from different years. A: The number of bacterial genera unique to sample of three year; B: The number of bacterial genera unique to sample of eight year; C: The number of bacterial genera peculiar to sample of fifteen year; D: The number of bacterial genera peculiar to sample of twenty year; E: The number of bacterial genera peculiar to sample of twenty-five year; F: The number of bacterial genera shared by five samples. |
在根皮苷耐受菌属水平上计算多样性指数(表 4)。结果显示,5个不同种植年限(3、8、15、20、25年)中筛选的根皮苷耐受菌的数目具有一定的差异。其中种植8年的苹果根际土壤,分离到根皮苷耐受菌的数目较少(14株),而种植25年筛选到的根皮苷耐受菌数目较多(26株),菌株数目随着年份无明显的变化规律。另外,种植3年根际土分离到的物种丰富度最高,种植20年最低,与香农指数的变化趋势相同。而主坐标分析(PCoA)研究了不同年限的根皮苷耐受菌的群落分布模式情况。根皮苷耐受菌群落PCoA排序图的第一轴(PC1)和第二轴(PC2)分别能解释59.07%和22.44%的物种变化信息,其中种植年限为15、20和25年在图 5中的距离相对较近,说明在种植初期,可培养根皮苷耐受菌群落变化较大;随着种植年限的增长,可培养根皮苷耐受菌菌群落趋于稳定,群落组成相似。

|
| 图 5 不同年限根皮苷耐受菌主坐标分析 Fig. 5 The PCoA analysis of the phlorizin-resistant bacteria communities from different years’ soils |
| Different years | Richness | No. of isolates | Shannon (H′) | Evenness (J′) | Chao-1 | No. of niche-specific genera |
| 3 | 9 | 22 | 1.93 | 0.77 | 9.6 | 2 |
| 8 | 6 | 14 | 1.43 | 0.70 | 12.0 | 1 |
| 15 | 8 | 20 | 1.92 | 0.85 | 9.5 | 0 |
| 20 | 5 | 21 | 1.27 | 0.71 | 5.0 | 0 |
| 25 | 8 | 26 | 1.82 | 0.77 | 9.5 | 1 |
皮尔森分析(Pearson correlations analysis)(表 5、6)研究了黄土高原苹果园土壤的理化性质与根皮苷耐受菌的群落组成的关系(基于属水平)。结果显示,根瘤菌属对环境因子中金属元素较敏感,与Cu2+ (P<0.01)、Zn2+ (P<0.05)、Fe3+ (P<0.05)、Mn2+ (P<0.05)呈显著正相关;新鞘脂杆菌属与奇异芽孢杆菌属次之,其中新鞘脂杆菌属与Cu2+ (P<0.05)、Zn2+ (P<0.05)、Mn2+ (P<0.01),奇异芽孢杆菌属与pH值(P<0.05)、儿茶素(P<0.05)、丁香酸(P<0.01)显著正相关;鞘脂杆菌属与2种酚酸呈显著正相关,分别为根皮苷(P<0.05)和水杨酸(SA)(P<0.05);而儿茶素与根瘤菌属(P<0.05)和贪噬菌属(P<0.01)显著正相关,根皮素与肠杆菌属显著正相关(P<0.05),阿魏酸与芽孢杆菌属呈显著负相关(P<0.05),并且C/N与贪铜菌属(P<0.05)、不动杆菌属(P<0.05)显著负相关,这表明C/N的比值越高,贪铜菌属和不动杆菌属的数量越少。这与土壤中理化性质的结果相符,由表 4可知,在种植3年的苹果根围土壤中,C/N含量最低,而贪铜菌属和不动杆菌属是种植3年根际土中筛选出的独特的2种菌。因此,贪铜菌属和不动杆菌属的数量可能与C/N的含量相关。Cu2+、Zn2+、Fe3+、Mn2+在种植15年和25年的土壤中含量较高,而根瘤杆菌属分别是种植15年和25年特有的菌株,可能与Cu2+、Zn2+、Fe3+、Mn2+的含量相关。同理,新鞘脂杆菌是种植15年和25年根际土中筛选的优势菌株,可能与Cu2+、Zn2+、Mn2+在土壤中的含量相关。
| Genus | pH | C | N | P | K | Cu | Zn | Fe | Mn | Ca | Mg | C/N |
| Ancylobacter | -0.118 | 0.185 | 0.049 | 0.094 | 0.114 | 0.600 | 0.361 | 0.711 | 0.267 | -0.526 | -0.415 | 0.321 |
| Rhizobium | 0.648 | 0.229 | 0.385 | -0.144 | -0.247 | 0.196 | 0.306 | 0.156 | 0.416 | 0.136 | -0.254 | -0.743 |
| Sphingobium | -0.634 | 0.314 | 0.149 | 0.752 | 0.741 | 0.057 | -0.018 | -0.055 | 0.175 | -0.588 | -0.269 | 0.447 |
| Novosphingobium | 0.662 | 0.091 | 0.012 | 0.152 | 0.659 | 0.914 | 0.988** | 0.851 | 0.890 | -0.441 | -0.395 | 0.200 |
| Bacillus | 0.593 | -0.759 | -0.728 | -0.495 | 0.180 | -0.090 | 0.210 | -0.132 | -0.121 | 0.575 | 0.709 | 0.402 |
| Staphylococcus | -0.438 | -0.025 | -0.049 | -0.224 | -0.543 | -0.186 | -0.417 | -0.020 | -0.490 | 0.033 | 0.041 | 0.068 |
| Cupriavidus | -0.125 | 0.510 | 0.683 | 0.222 | -0.403 | -0.464 | -0.438 | -0.544 | -0.042 | 0.128 | -0.249 | -0.938 |
| Acinetobacter | -0.125 | 0.510 | 0.683 | 0.222 | -0.403 | -0.464 | -0.438 | -0.544 | -0.042 | 0.128 | -0.249 | -0.938 |
| Enterobacter | -0.576 | 0.213 | 0.212 | -0.038 | -0.571 | -0.269 | -0.528 | -0.133 | -0.449 | -0.060 | -0.127 | -0.161 |
| Rhizobacter | 0.392 | 0.257 | 0.119 | 0.389 | 0.804 | 0.962** | 0.953 | 0.895 | 0.927 | -0.685 | -0.559 | 0.309 |
| Variovorax | 0.856 | 0.025 | 0.130 | -0.288 | -0.101 | 0.472 | 0.594 | 0.463 | 0.531 | 0.103 | -0.187 | -0.441 |
| Pseudomonas | 0.272 | 0.122 | 0.274 | 0.067 | -0.056 | -0.324 | -0.115 | -0.459 | 0.093 | 0.297 | 0.054 | -0.596 |
| Fictibacillus | 0.918 | -0.230 | -0.211 | -0.399 | 0.101 | 0.704 | 0.813 | 0.735 | 0.552 | 0.039 | -0.062 | 0.028 |
Note: : Correlation is significant at the 0.05 level; **: Correlation is significant at the 0.01 level.
| Genus | Phloroglucinol | Gallic acid | Catchin | Chlorogenic acid | Caffeic acid | P-hydroxybenzoic acid | Syringic acid | Ferulic acid | Salicylic acid | Phloridzin | Quercetin | Phloretin |
| Ancylobacter | 0.065 | -0.487 | 0.013 | -0.075 | 0.078 | -0.106 | 0.250 | 0.303 | 0.240 | 0.192 | 0.399 | 0.291 |
| Rhizobium | 0.532 | -0.068 | 0.884 | -0.085 | 0.198 | 0.348 | 0.612 | -0.170 | -0.496 | -0.423 | -0.169 | -0.024 |
| Sphingobium | 0.027 | 0.733 | -0.803 | 0.789 | 0.520 | -0.482 | -0.590 | 0.288 | 0.924 | 0.884 | 0.679 | -0.346 |
| Novosphingobium | 0.163 | -0.046 | 0.568 | 0.340 | 0.714 | 0.588 | 0.802 | -0.456 | 0.251 | 0.439 | 0.597 | -0.703 |
| Bacillus | -0.668 | -0.070 | 0.167 | -0.199 | -0.113 | 0.763 | 0.288 | -0.921 | -0.255 | -0.054 | -0.291 | -0.748 |
| Staphylococcus | -0.121 | -0.552 | -0.246 | -0.503 | -0.592 | -0.390 | -0.250 | 0.492 | -0.188 | -0.351 | -0.264 | 0.812 |
| Cupriavidus | 0.674 | 0.469 | 0.194 | 0.157 | 0.043 | -0.390 | -0.250 | 0.401 | -0.255 | -0.351 | -0.259 | 0.362 |
| Acinetobacter | 0.674 | 0.469 | 0.194 | 0.157 | 0.043 | -0.390 | -0.250 | 0.401 | -0.255 | -0.351 | -0.259 | 0.362 |
| Enterobacter | 0.122 | -0.349 | -0.281 | -0.354 | -0.499 | -0.599 | -0.383 | 0.701 | -0.128 | -0.334 | -0.218 | 0.923 |
| Rhizobacter | 0.228 | 0.080 | 0.317 | 0.524 | 0.821 | 0.348 | 0.612 | -0.232 | 0.524 | 0.665 | 0.812 | -0.639 |
| Variovorax | 0.315 | -0.317 | 0.985** | -0.183 | 0.221 | 0.621 | 0.875 | -0.409 | -0.479 | -0.342 | -0.077 | -0.211 |
| Pseudomonas | 0.314 | 0.524 | 0.295 | 0.221 | 0.196 | 0.114 | 0.000 | -0.199 | -0.224 | -0.168 | -0.214 | -0.277 |
| Fictibacillus | -0.022 | -0.552 | 0.888 | -0.261 | 0.199 | 0.817 | 1.000** | -0.609 | -0.352 | -0.167 | 0.052 | -0.391 |
Note: : Correlation is significant at the 0.05 level; **: Correlation is significant at the 0.01 level.
分别以根皮苷耐受菌菌株数目(基于属水平)和所检测的理化因子数据为依据,以5个不同年份为连接桥梁进行冗余分析(RDA),研究环境因子与根皮苷耐受菌群落结构之间的关系。通过数据预选(Forward selection)和蒙特卡洛-置换检验(Monte Carlo permutation tests,999次),选出解释度排名前三的环境因子展示在图 6的RDA排序图中。通过根皮苷耐受菌群落与环境因子的RDA排序图可以看出,排序图的第一轴和第二轴共可解释78.5%的物种-环境关系(其中第一轴Axis 1和第二轴Axis 2分别能解释59.9%和18.6%的物种-环境关系);RDA分析显示,在所检测的理化因子中,阿魏酸、对羟基苯甲酸、根皮素含量对可培养根皮苷耐受菌群落组成的解释度排在前三位,但统计学检验后,仅阿魏酸(P=0.017 0,F=2.512,999 Permutation tests)显著影响可培养根皮苷耐受菌群落的结构,这表明了阿魏酸在根皮苷耐受菌群落组成上发挥重要的作用。
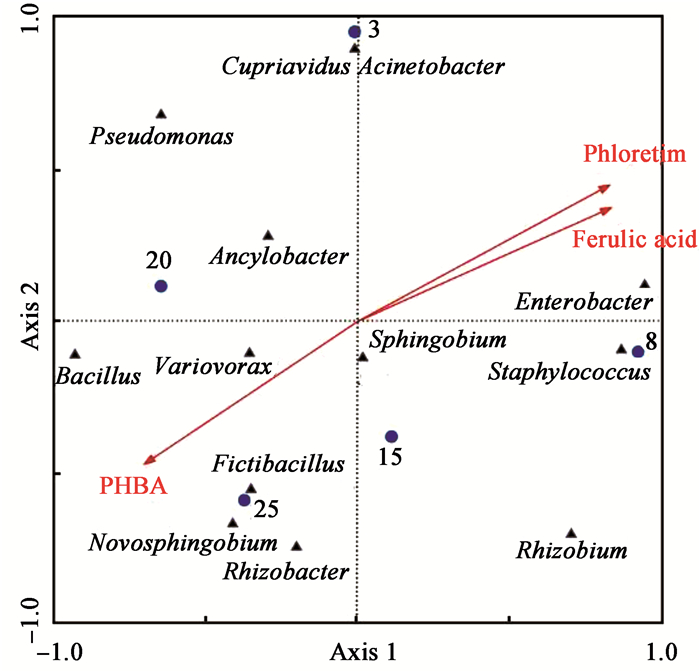
|
| 图 6 不同年限根皮苷耐受菌属与理化因子的冗余分析 Fig. 6 The RDA ordination plot for the relationship between the distribution of different genera and environment factors |
近几年我国苹果产业发展迅猛,趋于矮化密植栽培,生命周期缩短,品种更新加快,苗圃地、老果园重复利用时遇到的连作障碍问题越来越突出,严重制约果树生产的可持续发展。而根系与土壤直接接触,可以通过向土壤中分泌多种化学物质与其发生互作效应,其中的酚类物质是造成作物连作障碍的重要因素之一[20]。目前,对能够降解酚酸类物质的微生物研究较多,研究者已从外界环境中分离到多种能降解酚酸类物质的微生物,包括假单胞菌、醋酸钙不动杆菌、红球菌、根瘤菌、节杆菌、芽孢杆菌等[21],但对根皮苷降解菌研究较少。本研究以筛选得到的根皮苷耐受菌为基础,研究了根皮苷耐受菌的群落分布、多样性及其与理化因子的相关性,为根皮苷高效降解菌株的获得提供基础资料。从根皮苷耐受菌菌群分布来看,在5个不同种植年限的苹果根际土中,筛选得到103株根皮苷耐受菌,包括芽孢杆菌属、假单胞菌属、鞘脂杆菌属等13个属,显示了根皮苷耐受菌具有丰富的物种多样性。另外,由图 3B可知,与种植3年和8年分离得到的菌株相比,在种植年限为15、20和25年中分离得到的假单胞菌属、芽孢杆菌属、新鞘脂杆菌属和鞘脂杆菌属的数量显著增加,推测可能其具有解酚酸类的物质的能力。其中假单胞菌属可以降解土壤中酚酸类化感物质如β-香豆酸、阿魏酸、对羟基苯甲酸和对羟基苯甲醛等[22];芽孢杆菌可以降解苯酚[23];新鞘脂杆菌属可降解咖啡酸等酚酸类化合物[24];鞘脂杆菌属可以利用原儿茶酸和邻苯二酚等芳香族化合物[25]。另外,关于分离到的18个菌种的研究多见于石油烃及塑料的降解,部分芳香族和萜类化合物及农药的降解。例如鞘脂杆菌具有降解氯代二苯并呋喃化合物的能力[26];新鞘脂杆菌具有降解六氯环己烷的能力[27, 28];芽孢杆菌具有降解乙草胺除草剂的能力[29];贪铜菌能够降解芳香族和酚类化合物[30];醋酸钙不动杆菌能够降解苯酚[31];贪噬菌可以对新烟碱类杀虫剂噻虫啉进行生物转化[32],而根皮苷属于二氢查尔酮苷,具有多苯环结构,与芳香族及农药等部分物质的结构类似。因此,进一步推测分离到的菌株可能具有根皮苷降解能力。
主坐标分析可以观察群体间的差异。每一个点代表一个样本,两点之间距离越近,表明两者的群落构成差异越小。结果表明,从种植15、20和25年中分离到的根皮苷耐受菌菌群结构相似,推测可能与其对应的理化性质有关。由表 2可知,在不同种植年限的苹果土壤中,除C、N外,微量元素和速效钾、磷的含量较多,且以种植15、20、25年限中的含量较多。Cu2+、Zn2+、Mn2+的含量在15年和25年时最高,而种植15年和20年的优势菌种为新鞘脂杆菌,皮尔森分析表明新鞘脂杆菌与Cu2+、Zn2+、Mn2+正相关,可能具有相似的群落构成。
研究初步表明阿魏酸影响根皮苷耐受菌群落组成。结果表明,以15年为分水岭,随着种植年限的增长,阿魏酸的含量减少,厚壁杆菌门的数量上升(图 3B)。皮尔森分析表明阿魏酸与芽孢杆菌属具有一定的负相关性(P<0.05)。马瑞霞等发现3种浓度(5.15、2.58、0.26 mmol/L)的阿魏酸均表现出对枯草芽孢杆菌的生长具有抑制作用,并且随浓度的增大而增强[33]。前人研究也表明了阿魏酸等酚酸物质对土壤微生物的生长和活性有一定的影响。Sparling等认为人为添加酚酸可以改变土壤微生物量和活性,酚酸类物质与土壤微生物活性变化有着密切的关系,通过改变土壤的理化性质,促进一些微生物生长的同时也抑制其它一些相应土壤微生物的活性[21, 34]。研究发现,西瓜根系分泌物中的酚酸类物质可以促进尖孢镰刀菌孢子的形成和萌发,而水稻根系分泌物中的酚酸类物质能抑制尖孢镰刀菌孢子的形成和萌发[21, 35]。另外,冗余分析发现阿魏酸是影响根皮苷耐受菌群落组成的关键因子,根皮苷对于根皮苷耐受菌的群落结构没有显著的影响,可能是因为根皮苷在土壤中的部分降解。Jayasankar等研究表明土壤中的许多真菌可以将根皮苷分解为根皮素、对羟基苯甲酸、间苯三酚等[36]。
总之,文章研究了不同年限苹果根际土中根皮苷耐受菌的群落分布和多样性,丰富了对根皮苷耐受菌的了解,揭示了可培养的根皮苷耐受菌与环境因子可能性的潜在关系,结果对高效根皮苷降解菌的分离具有一定的指导意义,也对利用微生物缓解苹果连作障碍具有一定的借鉴意义。
| [1] | Mazzola M,Manici LM. Apple replant disease: role of microbial ecology in cause and control[J]. Annual Review of Phytopathology,2012,50(1): 45-65 |
| [2] | Inderjit. Plant phenolics in allelopathy[J]. The Botanical Review,1996,62(2): 186-202 |
| [3] | Wu FZ,Zhao FY,Ma FM. Phenolic acid substances and allelopathy mechanisms[J]. Journal of Northeast Agricultural University,2004,32(4): 402-407 (in Chinese)吴凤芝,赵凤艳,马凤鸣. 酚酸物质及其化感作用[J]. 东北农业大学学报,2004,32(4): 402-407 |
| [4] | Friebe A,Schulz M,Kück P,et al. Phytotoxins from shoot extracts and root exudates of Agropyron repens seedlings[J]. Phytochemistry,1995,38(5): 1157-1159 |
| [5] | Reigosa MJ,Sánchez-Moreiras A,González L. Ecophysiological approach in allelopathy[J]. Critical Reviews in Plant Sciences,1999,18(5): 577-608 |
| [6] | Börner H. The apple replant problem. I. The excretion of phlorizin from apple root residues[J]. Contributions of the Boyce Thompson Institute,1959,20: 39-56 |
| [7] | Börner H. Liberation of organic substances from higher plants and their role in the soil sickness problem[J]. The Botanical Review,1960,26(3): 393-424 |
| [8] | Zhang JH. Allelopathic effect of phenolics and its role on apple replant disease mechanism[D]. Tai’an: Doctoral Dissertation of Shandong Agricultural University,2005 (in Chinese)张江红. 酚类物质对苹果的化感作用及重茬障碍影响机理的研究[D]. 泰安: 山东农业大学博士学位论文,2005 |
| [9] | Zhang JH,Mao ZQ,Wang LQ,et al. Effect of phloridzin on physiological characteristics of Malus hupehensis Rehd. seedlings[J]. Scientia Agricultura Sinica,2007,40(3): 492-498 (in Chinese)张江红,毛志泉,王丽琴,等. 根皮苷对平邑甜茶幼苗生理特性的影响[J]. 中国农业科学,2007,40(3): 492-498 |
| [10] | Hofmann A,Wittenmayer L,Arnold G,et al. Root exudation of phloridzin by apple seedlings (Malus xdomestica Borkh.) with symptoms of apple replant disease[J]. Journal of Applied Botany and Food Quality,2012,82(2): 193-198 |
| [11] | Yim B,Winkelmann T,Ding GC,et al. Different bacterial communities in heat and gamma irradiation treated replant disease soils revealed by 16S rRNA gene analysis-contribution to improved aboveground apple plant growth?[J]. Frontiers in Microbiology,2015,6: 1224 |
| [12] | Sun J,Zhang Q,Zhou J,et al. Illumina amplicon sequencing of 16S rRNA tag reveals bacterial community development in the rhizosphere of apple nurseries at a replant disease site and a new planting site[J]. PLoS One,2014,9(10): e111744 |
| [13] | Yang J,Ruegger PM,McKenry MV,et al. Correlations between root-associated microorganisms and peach replant disease symptoms in a California soil[J]. PLoS One,2012,7(10): e46420 |
| [14] | Huang YY,Zhou GM,Yin GT,et al. Rhizosphere actinomycete isolation methods and phloridzin degradation activity analysis in plants[J]. Journal of Southern Agriculture,2013,44(1): 54-58 (in Chinese)黄园勇,周光明,尹国通,等. 植物根际放线菌分离方法初探及根皮苷降解活性分析[J]. 南方农业学报,2013,44(1): 54-58 |
| [15] | Zhang Y,Wang FZ,Zhang MS,et al. A strain of phlorhizin degrading bacterium screened from apple orchard soil[J]. Shandong Agricultural Sciences,2011(12): 59-63 (in Chinese)张一,王凤忠,张铭铄,等. 苹果园土壤中一株根皮苷降解菌的筛选[J]. 山东农业科学,2011(12): 59-63 |
| [16] | Yin CM,Wang GS,Li YY,et al. A new method for analysis of phenolic acids in the soil-soil from replanted apple orchards was investigated[J]. Scientia Agricultura Sinica,2013,46(21): 4612-4619 (in Chinese)尹承苗,王功帅,李园园,等. 一种分析土壤中酚酸类物质含量的新方法——以连作苹果园土壤为试材[J]. 中国农业科学,2013,46(21): 4612-4619 |
| [17] | Lane DJ. 16S/23S rRNA sequencing[A]//Stackebrandt E,Goodfellow M. Nucleic Acid Techniques in Bacterial Systematics[M]. Chichester,UK: John Wiley and Sons,1991: 115-175 |
| [18] | Tamura K,Peterson D,Peterson N,et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J]. Molecular Biology and Evolution,2011,28(10): 2731-2739 |
| [19] | Shi Y. Study on the polymorphism biodiversity of Bacillus and classification of the silicate bacterium[D]. Harbin: Master’s Thesis of Northeast Agricultural University,2008 (in Chinese)石禹. 芽孢杆菌的多态性研究及硅酸盐细菌的分类鉴定[D]. 哈尔滨: 东北农业大学硕士学位论文,2008 |
| [20] | Zhao X. A preliminary study on the relationship between phenolics in root exudate of different apple rootstocks and resistance to replant disease[D]. Yangling: Master’s Thesis of Northwest A&F University,2009 (in Chinese)赵新. 苹果砧木根系分泌物中酚类物质与连作障碍抗性关系的初步研究[D]. 杨凌: 西北农林科技大学硕士学位论文,2009 |
| [21] | Xie XG,Chen Y,Bu YQ,et al. A review of allelopathic researches on phenolic acids[J]. Acta Ecologica Sinica,2014,34(22): 6417-6428 (in Chinese)谢星光,陈晏,卜元卿,等. 酚酸类物质的化感作用研究进展[J]. 生态学报,2014,34(22): 6417-6428 |
| [22] | Zhang ZY,Pan LP,Li HH. Isolation,identification and characterization of soil microbes which degrade phenolic allelochemicals[J]. Journal of Applied Microbiology,2010,108(5): 1839-1849 |
| [23] | Yuan LJ,Jiang LC,Peng ZS,et al. Breeding of the high phenol-degraded bacterium JY01 and study on phenolic biodegradation[J]. Microbiology China,2009,36(4): 587-592 (in Chinese)袁利娟,姜立春,彭正松,等. 一株高效苯酚降解菌的选育及降酚性能研究[J]. 微生物学通报,2009,36(4): 587-592 |
| [24] | Bermek H,Catal T,Akan SS,et al. Olive mill wastewater treatment in single-chamber air-cathode microbial fuel cells[J]. World Journal of Microbiology and Biotechnology,2014,30(4): 1177-1185 |
| [25] | Inoue D,Hara S,Kashihara M,et al. Degradation of bis (4-hydroxyphenyl) methane (bisphenol F) by Sphingobium yanoikuyae strain FM-2 isolated from river water[J]. Applied and Environmental Microbiology,2008,74(2): 352-358 |
| [26] | Wittich RM,Busse HJ,Kämpfer P,et al. Sphingobium aromaticiconvertens sp. nov.,a xenobiotic-compound-degrading bacterium from polluted river sediment[J]. International Journal of Systematic and Evolutionary Microbiology,2007,57(2): 306-310 |
| [27] | Saxena A,Anand S,Dua A,et al. Novosphingobium lindaniclasticum sp. nov.,a hexachlorocyclohexane (HCH)-degrading bacterium isolated from an HCH dumpsite[J]. International Journal of Systematic and Evolutionary Microbiology,2013,63(Pt 6): 2160-2167 |
| [28] | Niharika N,Moskalikova H,Kaur J,et al. Novosphingobium barchaimii sp. nov.,isolated from hexachlorocyclohexane-contaminated soil[J]. International Journal of Systematic and Evolutionary Microbiology,2013,63(Pt 2): 667-672 |
| [29] | Xu J,Yang M,Dai JY,et al. Degradation of acetochlor by four microbial communities[J]. Bioresource Technology,2008,99(16): 7797-7802 |
| [30] | Belchik SM,Xun LY. Functions of flavin reductase and quinone reductase in 2,4,6-trichlorophenol degradation by Cupriavidus necator JMP134[J]. Journal of Bacteriology,2008,190(5): 1615-1619 |
| [31] | Cordova-Rosa SM,Dams RI,Cordova-Rosa EV,et al. Remediation of phenol-contaminated soil by a bacterial consortium and Acinetobacter calcoaceticus isolated from an industrial wastewater treatment plant[J]. Journal of Hazardous Materials,2009,164(1): 61-66 |
| [32] | Zhang HJ,Zhou QW,Zhou GC,et al. Biotransformation of the neonicotinoid insecticide thiacloprid by the bacterium Variovorax boronicumulans strain J1 and mediation of the major metabolic pathway by nitrile hydratase[J]. Journal of Agricultural and Food Chemistry,2011,60(1): 153-159 |
| [33] | Ma RX,Feng Y,Li X. Effects of allelochemicals on growth of Bacillus subtilis and its denitrification under anaerobic condition[J]. Acta Ecologica Sinica,2000,20(3): 452-457 (in Chinese)马瑞霞,冯怡,李萱. 化感物质对枯草芽孢杆菌(Bacillus subtilis)在厌氧条件下和生长[J]. 生态学报,2000,20(3): 452-457 |
| [34] | Sparling GP,Ord BG,Vaughan D. Changes in microbial biomass and activity in soils amended with phenolic acids[J]. Soil Biology and Biochemistry,1981,13(6): 455-460 |
| [35] | Hao WY,Ren LX,Ran W,et al. Allelopathic effects of root exudates from watermelon and rice plants on Fusarium oxysporum f. sp. niveum[J]. Plant and Soil,2010,336(1/2): 485-497 |
| [36] | Jayasankar NP,Bandoni RJ,Towers GHN. Fungal degradation of phloridzin[J]. Phytochemistry,1969,8(2): 379-383 |
 2016, Vol. 43
2016, Vol. 43




