扩展功能
文章信息
- 邹洁, 张继文, 尤逊, 杨江科
- ZOU Jie, ZHANG Ji-Wen, YOU Xun, YANG Jiang-Ke
- 马克斯克鲁维酵母菌菊粉酶的异源表达及低聚果糖制备工艺优化
- Heterelogous expression of Kluyveromyces marxianus inulinase gene and optimization of reaction parameters for oligofructose preparation from inulin
- 微生物学通报, 2016, 43(7): 1429-1437
- Microbiology China, 2016, 43(7): 1429-1437
- 10.13344/j.microbiol.china.150562
-
文章历史
- 收稿日期: 2015-07-23
- 接受日期: 2015-11-20
- 优先数字出版日期(www.cnki.net): 2016-01-05
2. 武汉轻工大学生物与制药工程学院 湖北 武汉 430023
2. School of Biology and Pharmaceutical Engineering, Wuhan Polytechnic University, Wuhan, Hubei 430023, China
菊粉是植物中的一种储备性多糖,以最早从菊科植物中分离出来而得名。菊粉广泛存在于各类植物中,其中以菊芋(Helianthus tuberosus L.)和菊苣(Cichorium intybus L.)含量最高。菊粉是由呋喃D-果糖经β-2,l糖苷键脱水聚合而成,且通常在果糖残基末端连有一个葡萄糖残基。菊粉是制备果糖和低聚果糖的理想原料。在稀酸或菊粉酶水解下,依照水解程度的不同可生成果糖或低聚果糖[1]。低聚果糖是由数个果糖基通过β-2,l糖苷键与蔗糖中的果糖基结合生成的蔗果三糖、四糖和五糖等的混合物。低聚果糖的甜度比蔗糖稍低,但其甜味纯正且更加清爽。低聚果糖是一种天然活性物质。它具有调节肠道菌群、调节血脂、提高免疫机能以及抗龋齿等保健功能,被誉为最具潜力的新一代“益生原”。目前已在食品、乳制品等行业应用[1,2]。
在低聚果糖的制备工艺中,以菊粉为原料采用菊粉酶水解生产低聚果糖的酶法工艺是先进的绿色制造。获得高效的菊粉酶是低聚果糖酶法制备工艺的前提。菊粉酶在植物和微生物中分布广泛。但由于植物组织中菊粉酶含量低且提取工艺复杂,微生物是工业用菊粉酶的主要来源。常见的产菊粉酶的微生物包括芽孢杆菌、曲霉、酵母等[3,4]。由于从自然菌种中分离制备菊粉酶纯度低、工艺复杂且成本高,因此限制了菊粉酶的酶法制备技术的推广和应用。利用基因工程手段实现菊粉酶的高效表达,不仅可获得优质、高纯度的菊粉酶,而且可为菊粉酶在的工业领域的广泛应用奠定良好的基础。
马克斯克鲁维酵母菌(Kluyveromyces marxianus)是具有良好生物安全性的菊粉酶生产菌[5,6]。本研究克隆了K.marxianus CICC1726的菊粉酶基因,并实现了该基因在毕赤酶母(Pichia pastoris)中的分泌表达;以毕赤酵母表达的菊粉酶为催化剂,系统研究了以菊粉为原料制备低聚果糖的工艺条件。本研究为菊粉酶的大量生产及低聚果糖的制备提供了参考。
1 材料与方法 1.1 材料马克斯克鲁维酵母菌K. marxianus CICC1726购自中国工业微生物菌种保藏中心;大肠杆菌DH5α、Pichia pastoris GS115、质粒pPIC9K为本实验室保存。
1.2 培养基低钠LB培养基(g/L):酵母粉5.0,蛋白胨10.0,氯化钠5.0,加蒸馏水至1 L。
YPD培养基(g/L):酵母粉10.0,蛋白胨20.0,葡萄糖20.0,加蒸馏水至1 L。
BMGY培养基(g/L):酵母粉10,蛋白胨20,葡萄糖20,酵母氮碱0.5,生物素0.004,加蒸馏水至1 L。
1.3 主要试剂和仪器限制性内切酶、T4 DNA连接酶、Pfu聚合酶等购自宝生物工程(大连)有限公司;质粒提取试剂盒、PCR产物纯化试剂盒、胶回收试剂盒等购自美国Omega公司。
小型台式Bioflo450型发酵罐购自德国Eppendorf公司;高效液相色谱仪(Waters2695型)购自美国Waters公司;PCR仪购自美国Bio-Rad公司;恒温摇床购自上海智诚实验设备有限公司;5424R型高速冷冻离心机购自德国Eppendorf公司;UV-2102型紫外可见分光光度计购自尤尼柯(上海)仪器有限公司。
1.4 方法 1.4.1菊粉酶基因的克隆及在P. pastoris中的表达: K. marxianus菊粉酶基因KMI的PCR扩增以菌株K. marxianus CICC1726总DNA为模板,以菊粉酶基因5′端序列为上游引物KMIF(5′-GAATTCATGAAGTTAGCATACTCCCTC-3′,EcoR I),以菊粉酶基因3′端序列为下游引物YLR1(5′-GCGGCCGCTCAAACGTTAAATTGGGTAAC-3′,Not I)。100 μL PCR反应体系:200 μmol/L dNTPs 10 μL、0.1 μmol/L上下游引物各2 μL、2 U Pfu、DNA模板20 ng。PCR反应条件:94 ℃ 2 min;94 ℃ 30 s,52 ℃ 30 s,72 ℃ 1 min,共28个循环;72 ℃ 5 min;4 ℃保温。PCR扩增产物经序列分析确认为菊粉酶基因。菊粉酶基因KMI的PCR产物经EcoR I/Not Ⅰ酶切位点克隆至P. pastoris甲醇诱导型表达载体pPICZa,获得重组子pPICZ-KMI(图 1)。重组子经Sac I线性化后采用电转化法导入P. pastoris GS115。电转化体系为:GS115细胞90 μL、线性化质粒5 μg。电转化条件为:电压2 500 V、电阻200 Ω。转化后的酵母细胞在含Zeocin的YPD平板筛选后获得阳性转化子。详细操作参见文献[7]。

|
|
图 1
K. marxianus菊粉酶基因KMI的PCR扩增产物与表达载体酶切产物的电泳检测
Figure 1
PCR amplification of K. marxianus inulinase gene KMI and enzymatic digestion of the recombinant plasmid pPICZ-KMI
注:M:Marker. A:菊粉酶KMI基因的PCR产物电泳图;B:重组质粒pPICZ-KMI双酶切产物电泳图. Note: M: Marker. A: The PCR product of inulinase KMI gene; B: Recombinant pPICZ-KMI was digested by EcoR I/Not I. The arrows indicate the bands of the vector pPICZa and KMI gene. |
(2)菊粉酶在不同温度下的酶活测定。试验采用的方法与1.4.4所述基本一致。不同之处在于反应温度分别设定为30、35、40、45、50、55和60 ℃。
(3)菊粉酶在含不同金属离子缓冲液中的酶活测定。分别以0.l mol/L、pH 5.0的醋酸缓冲液配制含2 mmol/L不同种类的二价金属盐离子CaCl2、MgCl2、FeCl2、ZnCl2、CuCl2和离子螯合剂EDTA的反应液。测定菊粉酶在上述反应液中的酶活,获得反映菊粉酶在不同金属离子和EDTA条件下的酶活变化情况。测定方法同于1.4.4所述。
(4)最适底物浓度测定。试验采用的方法与1.4.4所述基本一致。不同之处在于反应体系中的菊粉底物浓度分别设定为2%、4%、6%、8%、10%、15%和20%。
1.4.6菊粉水解产物的HPLC检测: 菊粉水解溶液的配制。精密量取2.0 mL菊粉水解溶液,加3.0 mL三蒸水,用乙腈定容至10.0 mL容量瓶中。高效液相色谱仪为Waters2695型并配置ELSD检测器和Amide 4.6 mm×250 mm规格色谱柱。进样量为15 μL,流动相为乙腈与水的混合液。初始条件下乙腈与水体积比为75:25,在0−35 min之间,体积比等度梯度变化为50:50。参照混标溶液出峰时间(依次为果糖、葡萄糖、蔗糖、蔗果三糖、蔗果四糖、蔗果五糖),按照出峰数目类推,统计低聚果糖各组分和峰面积和相对含量。 2 结果与分析 2.1 K. marxianus菊粉酶基因以K.marxianus CICC1726菌株总DNA为模板扩增了菊粉酶基因KMI(图 2)。所扩增的菊粉酶基因开放式阅读框全长1 671 bp,编码556个氨基酸,其中前23个氨基酸为信号肽。该酶属于糖基水解酶家族GH32成员,包括2个主要的结构域:果糖苷水解酶和糖基水解酶C32。果糖苷水解酶活性中心氨基酸分别为:Asp53、Asp182和Glu238(或者为D53、D182和E238)。

|
|
图 2
K. marxianus菊粉酶基因序列和主要功能区段
Figure 2
The sequence of K. marxianus inulinase gene and its functional domain
注:蓝色背景为信号肽区,“*”所指为菊粉酶活性位点氨基酸. Note: Blue background indicates the signal peptide region, and * indicates the active site of enzyme. |
在摇瓶条件下检测了P.pastoris基因工程菌诱导表达菊粉酶的情况。由图 3A、B可见,在前24 h的营养生长阶段,未检测到菊粉酶酶活;开始甲醇诱导后,随着诱导时间的延长,菊粉酶活及蛋白质含量稳步增加。当到达96 h时,发酵液中菊粉酶活为286 U/mL,蛋白质含量为0.4 g/L。表明菊粉酶基因KMI在毕赤酵母中实现了分泌表达。

|
|
图 3
毕赤酵母基因工程菌在摇瓶条件和发酵罐条件下菊粉酶的产酶变化趋势
Figure 3
Expression of K. marxianus inulinase gene in P. pastoris
注:A:揺瓶条件下酶活变化;B:摇瓶发酵液电泳检测;C:10 L发酵罐条件下酶活变化;D:10 L发酵罐中发酵液电泳检测. Note: A: Under the condition of the flasks; B: The protein profiles in the supernatant of the fermentation broth in flasks; C: Under the condition of the 10 L fermenter; D: The protein profiles in the supernatant of the fermentation broth in fermenter. |
进一步测定了工程菌株在10 L发酵罐水平下的产酶能力(图 3C、D)。主要工艺参数为:(1)在营养生长阶段(前24 h,菌体生长温度为28 ℃,pH 6.0),与摇瓶条件相一致,未检测到菊粉酶活;(2)在甲醇诱导表达阶段,甲醇流加速度控制在0.5−2.5 mL/(L·h)、温度为25 ℃、pH 5.5。在此条件下,发酵时间达96 h时,发酵液中菊粉酶活性达1 570 U/mL、蛋白质含量为2.75 g/L发酵液。
2.3 菊粉酶在不同条件下的酶活变化趋势分别测定了菊粉酶在不同pH、温度、金属离子以及底物浓度下的酶活变化趋势,以期为菊粉酶水解菊粉制备低聚果糖提供合适的反应参数。
(1)菊粉酶在不同pH条件下酶活变化趋势。采用不同的缓冲体系NaAc-Hac(pH 4.0−6.0)、Tris-HCl(pH 7.0−8.0)和Gly-NaOH(pH 9.0−10.0)测定了菊粉酶在pH 4.0−10.0之间酶活的变化趋势。由图 4A可见,菊粉酶的最适pH为5.0。当pH在5.0−7.0之间变化时,表观酶活稳定在最适酶活的80%以上。当pH>7.0后,随着pH值的增大,酶活迅速降低。表明研究所获得的菊粉酶适合于弱酸性的环境。
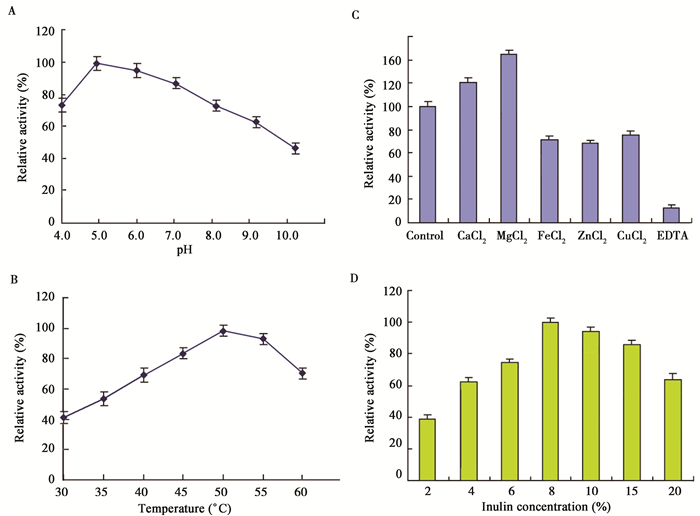
|
|
图 4
不同单因子条件下菊粉酶水解菊粉的酶活变化趋势
Figure 4
The activity of inulinase under different conditions
注:A:在不同pH条件下的酶活;B:在不同温度下的酶活;C:在不同金属离子及鳌合剂下的酶活;D:不同菊粉浓度下的酶活. Note: A: Effects of pH on inulinase activity; B: Effects of temperature on inulinase activity; C: Effect of cations and EDTA on inulinase activity; D: Effects of substrate concentration on inulinase activity. |
(2)菊粉酶的最适温度测定。在30−60 ℃下分别测定了菊粉酶的表观酶活力,由图 4B可见,菊粉酶的最适温度为50 ℃。当温度在40−55 ℃之间变化时,菊粉酶的酶活均能保持在最适酶活的80%以上。即使当温度达60 ℃时,酶活仍为最适酶活的72%。
(3)金属离子对菊粉酶活性的影响。分别检测了Ca2+、Mg2+、Fe2+、Zn2+、Cu2+以及离子螯合剂EDTA对菊粉酶的影响。试验在反应体系中分别加入2 mmol/L不同种类的二价金属盐离子和离子螯合剂EDTA,并测定其菊粉酶的活性。菊粉酶在不同金属离子中的酶活见图 4C。由图 4C可见,Ca2+和Mg2+能提高显著提高菊粉酶的活力,与出发酶液相比,酶活分别提高20%和75%;Cu2+、Fe2+和Zn2+对此菊粉酶则有明显的抑制作用,残余酶活力分别为出发酶液的75%、72%和70%;金属离子螯合剂EDTA对菊粉酶活力的影响极为显著,残余酶活仅为12.8%。
(4)最适底物浓度测定。分别配制2%、4%、6%、8%、10%、15%和20%的菊粉溶液,检测菊粉酶在不同底物浓度下的酶活。由图 4D可见,随着底物浓度从2%−10%逐渐增加,菊粉酶液的表观酶活逐渐增大。当菊粉底物浓度为8%时,酶活达到最大值;此后,随着底物浓度的增加,菊粉酶表观酶活逐渐下降。
2.4 不同条件下水解菊粉产物的HPLC分析基于2.3的结果,在pH 5.0、反应温度50 ℃、含0.2 mmol/L Mg2+以及菊粉底物浓度为8%的条件下,系统测定了不同酶量的水解产物的组分及其含量(表 1,图 5)。由表 1和图 5结果可见,随着酶量的增加,菊粉被逐渐水解,长链产物含量逐渐降低。在菊粉酶量为10 U/L的情况下,菊粉被完全水解。水解产物中单糖(葡萄糖)二糖(蔗糖)含量为9.25%,而低聚果糖(C3−C8)含量为90.75%,而其中,C3−C5低聚果糖含量高达72.92%。
| Component | Amount of inulinase in reaction mixture (U/L) | |||||
| 2 | 4 | 6 | 8 | 10 | 12 | |
| C1+C2 | 9.36 | 9.79 | 9.46 | 9.76 | 9.25 | 9.25 |
| C3 | 12.51 | 18.72 | 20.29 | 22.00 | 25.71 | 29.47 |
| C4 | 15.05 | 22.32 | 25.10 | 25.67 | 28.08 | 31.55 |
| C5 | 13.07 | 16.56 | 17.84 | 19.13 | 19.13 | 17.23 |
| C6 | 12.34 | 13.51 | 13.23 | 14.08 | 12.56 | 11.83 |
| C7 | 11.06 | 10.83 | 10.42 | 7.70 | 5.12 | 0.67 |
| C8 | 7.17 | 3.53 | 2.03 | 1.02 | 0.15 | − |
| C9 | 3.83 | 1.24 | 0.60 | 0.27 | − | − |
| C10 | 2.70 | 0.79 | 0.34 | 0.19 | − | − |
| C11 | 2.18 | 0.66 | 0.24 | 0.17 | − | − |
| C12 | 1.93 | 0.51 | 0.23 | − | − | − |
| C13 | 1.55 | 0.51 | 0.10 | − | − | − |
| C14 | 1.38 | 0.43 | 0.10 | − | − | − |
| C15 | 0.89 | 0.22 | − | − | − | − |
| C16 | 0.81 | 0.15 | − | − | − | − |
| C17 | 0.75 | 0.11 | − | − | − | − |
| C18 | 0.74 | 0.10 | − | − | − | − |
| C19 | 0.59 | − | − | − | − | − |
| C20 | 0.66 | − | − | − | − | − |
| C21 | 0.47 | − | − | − | − | − |
| C22 | 0.33 | − | − | − | − | − |

|
|
图 5
菊粉酶水解菊粉产物的HPLC图谱
Figure 5
The HPLC chromatograms of the inulin products digested with inulinase KMI
注:A:2 U菊粉酶水解菊粉的产物检测;B:10 U菊粉酶水解菊粉的产物检测. Note: A: The profile of the products digested the inulin with 2 U enzyme; B: The profile of the products digested the inulin with 10 U enzyme. |
低聚果糖被广泛应用于食品、乳制品等领域,具有广阔的市场需求。以菊粉为原料,采用菊粉酶水解生产低聚果糖的菊法工艺是先进的绿色制造工艺,也是今后的发展方向。实现菊粉酶的大量生产是低聚果糖酶法制造的前提。早期制备低聚果糖均直接以曲霉等菌体为催化剂(菌体中含有少量的菊粉酶)制备低聚果糖。但由于菌体酶系成分复杂、菊粉酶含量低等因素造成产物不纯,该方法已被淘汰。另一方面,从天然微生物中获取高纯度的菊粉酶需要经过一系列复杂的纯化技术,如(NH4)2SO4的分级沉淀、离子交换层析及凝胶过滤色谱等。工艺复杂、成本高昂、限制了菊粉酶的工业化应用[3,4]。因此,利用基因工程手段,实现菊粉酶的高效表达,不仅可获得优质、高纯度的酶,而且可为菊粉酶的工业化应用奠定良好的基础。
本研究选用的P. pastoris表达系统是目前广泛使用的真核生物表达体系。它具有生物安全性好、遗传元件稳定、高密度发酵工艺成熟以及目的蛋白分离纯化简便等优点,是异源蛋白质工业化生产的理想宿主[8]。从本研究结果可知,毕赤酵母菊粉酶基因工程菌在10 L发酵罐中酶活达到了1 570 U/mL发酵液。该酶活与He等对A. niger菊粉酶在Pichia中异源表达的酶活相当(1 349 U/mL)[10],但高于Cryptococcus aureus菊粉酶基因INU1在Pichia中异源表达的酶活(16.3±0.24 U/mL)[11]和Candida membranifaciens菊粉酶基因INU1在Saccharomyces中表达的酶活(16.8 U/mL)[12],更远高于直接从环境中筛选出的野生型Penicillium菌株的产酶能力(2 U/mL)[13]。
提高菊粉酶酶解效率既需要提供合适的反应条件,又需要提高酶活和增加酶对底物的敏感性。不同酶种间底物专一性严格程度不同。通常,菊粉酶不仅可以作用于菊粉,也可以作用于蔗糖及棉子糖。根据对多聚果糖底物作用方式的不同,菊粉酶被分为内切型菊粉酶(EC3.2.1.7)和外切型菊粉酶(EC3.2.1.80)两种类型。但多数情况下菊粉酶内切和外切活性兼而有之。对糖苷水解酶类作用方式可用i/s值的大小来区分内切型和外切型的区别。i是以多聚果糖(菊粉)作底物时的酶活,s是以蔗糖作底物时的酶活。通常,外切菊粉酶的i/s值比内切菊粉酶的i/s值低[3]。由实验结果可知(图 5),本研究中所获得的K. marxianus菊粉酶的主要水解产物为低聚果糖,其含量远高于一糖和二糖,因此该酶应为内切型菊粉酶。
本研究克隆了K. marxianus菊粉酶基因,并实现了其在P. pastoris中的异源高效表达,完成了10 L规模发酵罐中的发酵工艺优化;通过对该菊粉酶反应参数和反应产物的测定,获得了制备低聚果糖的最佳工艺条件。本研究为菊粉酶的生产及低聚果糖酶法制备提供了参考。
| [1] | Coussement PA. Inulin and oligofructose: safe intakes and legal status[J]. The Journal of Nutrition, 1999, 129(7): 1412S-1417S |
| [2] | Roberfroid MB. Inulin-type fructans: functional food ingredients[J]. The Journal of Nutrition, 2007, 137(11): 2493S-2502S |
| [3] | Vijayaraghavan K, Yamini D, Ambika V, et al. Trends in inulinase production-a review[J]. Critical Reviews in Biotechnology, 2009, 29(1): 67-77 |
| [4] | Housseiny MM. Production of an endoinulinase from Aspergillus niger AUMC 9375, by solid state fermentation of agricultural wastes, with purification and characterization of the free and immobilized enzyme[J]. Journal of Microbiology, 2014, 52(5): 389-398 |
| [5] | Rouwenhorst RJ, Ritmeester WS, Scheffers WA, et al. Localization of inulinase and invertase in Kluyveromyces species[J]. Applied and Environmental Microbiology, 1990, 56(11): 3329-3336 |
| [6] | Hong SJ, Kim HJ, Kim JW, et al. Optimizing promoters and secretory signal sequences for producing ethanol from inulin by recombinant Saccharomyces cerevisiae carrying Kluyveromyces marxianus inulinase[J]. Bioprocess and Biosystems Engineering, 2015, 38(2): 263-272 |
| [7] | Yang JK, Liu LY, Dai JH, et al. De novo design and synthesis of Candida antarctica lipase B gene and a-factor leads to high-level expression in Pichia pastoris[J]. PLoS One, 2013, 8(1): e53939 |
| [8] | Zhou WJ, Yang JK, Mao L, et al. Codon optimization, promoter and expression system selection that achieved high-level production of Yarrowia lipolytica lipase in Pichia pastoris[J]. Enzyme and Microbial Technology, 2015, 71: 66-72 |
| [9] | Sengupta S, Jana ML, Sengupta D, et al. A note on the estimation of microbial glycosidase activities by dinitrosalicylic acid reagent[J]. Applied Microbiology and Biotechnology, 2000, 53(6): 732-735 |
| [10] | He M, Wu D, Wu J, et al. Enhanced expression of endoinulinase from Aspergillus niger by codon optimization in Pichia pastoris and its application in inulooligosaccharide production[J]. Journal of Industrial Microbiology & Biotechnology, 2014, 41(1): 105-114 |
| [11] | Cao TS, Wang GY, Chi Z, et al. Cloning, characterization and heterelogous expression of the INU1 gene from Cryptococcus aureus HYA[J]. Gene, 2013, 516(2): 255-262 |
| [12] | Zhang LL, Tan MJ, Liu GL, et al. Cloning and characterization of an inulinase gene from the marine yeast Candida membranifaciens subsp. flavinogenie W14-3 and its expression in Saccharomyces sp. Wo for ethanol production[J]. Molecular Biotechnology, 2015, 57(4): 337-347 |
| [13] | Rawat HK, Ganaie MA, Kango N. Production of inulinase, fructosyltransferase and sucrase from fungi on low-value inulin-rich substrates and their use in generation of fructose and fructo-oligosaccharides[J]. Antonie Van Leeuwenhoek, 2015, 107(3): 799-811 |
 2016, Vol. 43
2016, Vol. 43




