扩展功能
文章信息
- 梁园梅, 刘瑛, Maurycy Daroch, 成家杨
- LIANG Yuan-Mei, LIU Ying, Maurycy Daroch, Jay J. CHENG
- 破囊壶菌脂肪酶基因的克隆、双温控自诱导表达及酶学性质研究
- Cloning, auto-induced expression by dual temperature controlling and characterization of lipase from Aurantiochytrium sp. PKU#SW7
- 微生物学通报, 2016, 43(6): 1190-1198
- Microbiology China, 2016, 43(6): 1190-1198
- DOI: 10.13344/j.microbiol.china.150553
-
文章历史
- 收稿日期: 2015-07-21
- 接受日期: 2015-09-09
- 优先数字出版日期(www.cnki.net): 2015-09-28
脂肪酶(Lipase,EC3.1.1.3)属于羧基酯水解酶类,α/β折叠水解酶超家族。脂肪酶广泛存在于人、动植物、微生物体中。由于微生物脂肪酶具有种类繁多、底物专一性类型多、产量可控性强、易分离纯化以及便于工业化生产等优点,成为国内外的研究热点。Hasan等[1]研究发现,微生物界可产脂肪酶的种类大约有65个属,其中细菌有28个属、酵母菌10个属、放线菌4个属、其他真菌23个属,但实际环境中微生物脂肪酶分布广度会更加宽泛。近几年,脂肪酶作为一种重要的工业酶,被大量运用到食品、医疗、皮革、日用化工、废渣处理等领域[2]。根据脂肪酶在有机相中高效催化脂肪酸和醇类进行转酯化的反应特性,脂肪酶可作为生产生物柴油的直接催化剂,提高生物柴油产率[3]。
破囊壶菌是一类分布在海洋海水、底泥、红树林根部的类真菌微生物[4],在生长过程中可以分解环境中动植物残体为自身提供营养,意味着破囊壶菌在生长过程中会分泌一系列胞外酶降解物质。在过去几十年间对破囊壶菌的研究主要集中在生物多样性以及化学成分多样性[5];破囊壶菌在生长过程中会积累大量棕榈酸和二十二碳六烯酸(Docosahexaenoic acid,DHA),商业价值巨大。目前对破囊壶菌DHA的开发利用已有相关报道[6, 7],但对于如何利用开发棕榈酸却鲜有研究。破囊壶菌在某种特定环境条件下是否可自身催化棕榈酸转酯合成生物柴油,这是一个非常值得探索的问题。基于这个问题,需要先对生物自身脂肪酶酶学性质进行深入了解,判断是否符合生物柴油生物催化剂的要求。然而,目前对于破囊壶菌胞外酶活性检测和分泌酶基因挖掘的研究却罕见报道。
Aurantiochytrium是破囊壶菌科中一个属,具有破囊壶菌科在环境中的普遍特性。在实验室前期工作中,分离得到一株命名为Aurantiochytrium sp. PKU#SW7的菌株,对其研究表明该菌株脂肪酸含量高,并且脂肪酸组成成分简单(主要为棕榈酸和DHA),是生产DHA保健品和生物柴油的良好菌株[8]。此外,在分析Aurantiochytrium sp. PKU#SW7转录组数据(待发表)研究脂肪酸合成途径过程中,我们发现有36条标记为脂肪酶或者酯酶的基因片段,说明该菌株可能存在脂肪酶活性,具有自身转化合成生物柴油的潜力。本文主要以Aurantiochytrium sp. PKU#SW7基因组DNA为模板,利用PCR技术扩增获取脂肪酶lip基因,应用表达载体pET30a(+)构建重组质粒, 转化至表达菌株大肠杆菌Rosetta(DE3),组建表达脂肪酶基因工程菌,利用自诱导双温控制方式大量表达蛋白,并初步研究重组酶的酶学性质,为综合开发利用破囊壶菌生产生物柴油奠定基础。
1 材料与方法 1.1 实验材料1.1.1 菌株和质粒: 宿主Escherichia coli DH5α、Rosetta(DE3)、载体pMD18-T和pET30a(+),购于thermo公司;破囊壶菌Aurantiochytrium sp. PKU#SW7由北京大学环境与能源学院分离自深圳大鹏湾沿岸海水(22°31′32.632″N,114°28′40.185″E),保藏于中国微生物菌种保藏管理委员会普通微生物中心,保藏编号为CGMCC 8575。
1.1.2 主要试剂: 限制性内切酶EcoR I、Xho I,T4 DNA连接酶,Taq DNA聚合酶,PageRuler™ Prestained Protein Ladder,购于Fermentas公司;pMD18-T Vector Cloning Kit,购于宝生物工程(大连)有限公司;PCR相关试剂、基因组DNA提取试剂盒、DNA凝胶回收试剂盒及质粒小量提取试剂盒,购于上海捷瑞生物工程有限公司;DNA Marker、Millipore Amicon Ultrafiltration Centrifuge Tubes、piece#BCA Protein Assay Kit,购于Thermo Fisher Scientific公司;6×His-Tagged Protein Purification Kit,购于北京康为世纪生物科技有限公司。
1.1.3 培养基:液体M4培养基和自诱导培养基,分别按照参考文献[9]和[10]配制。
脂肪酶筛选培养基(g/L):无水葡萄糖20.0,酵母浸出粉1.0,蛋白胨1.5,0.01%苯酚红以及1%的橄榄油,琼脂12.0,天然海水过滤后配制,1×105 Pa灭菌30 min,摇匀倒平板。
1.2 实验方法1.2.1 破囊壶菌Aurantiochytrium sp. PKU#SW7 脂肪酶活性研究:在含有1%橄榄油和0.01%苯酚红的筛选平板上打小孔(直径约1 cm),将培养2-3 d的破囊壶菌菌液于4 000 r/min离心10 min,实验组取培养液上清100 μL接种到小孔中,对照组添加100 μL M4培养基,把接种后的筛选平板放置于28 °C培养,24 h后观察筛选平板颜色变化,定性判断菌株是否存在脂肪酶活性。
1.2.2 目的基因lip 克隆:破囊壶菌的脂肪酶基因表达序列从转录组测序数据库中获得。将转录组测序数据库中36条标记为脂肪酶或者酯酶的基因片段进行分析,获得一个有完整开放阅读框的基因片段,具备转录翻译表达成熟脂肪酶的能力。分析该开放阅读框,设计引物如下:F:5′-CCGGAATT CGGGTCTGAAGAATATACCTTGGATG-3′;R:5′-CCGCTCGAGCTATAACTGAAGGGCAAATCTTTCAGC-3′。其中,下划线处分别为EcoR I和Xho I酶切位点。
以破囊壶菌Aurantiochytrium sp. PKU#SW7基因组序列为模板,通过PCR扩增lip基因全长序列。PCR反应体系(20 μL):基因组DNA 2.0 μL,2×UltraTaq Master Mix 10.0 μL,引物F和引物R (10 μmol/L)各1.0 μL,ddH2O 6.0 μL。PCR扩增条件为:94 °C 3 min;94 °C 30 s,60 °C 30 s,72 °C 1 min,28个循环;72 °C 10 min。将完整lip基因克隆至pMD18-T中,即为pMD18-lip质粒。PCR产物和pMD18-lip质粒测序由华大基因完成。
1.2.3 表达载体构建:将pMD18-lip质粒用EcoR I和Xho I双酶切,回收目的基因lip片段;与经过相同内切酶双酶切的pET30a(+)质粒连接,构建重组表达质粒pET30a-lip,转化至感受态细胞Rosetta(DE3),挑选阳性转化子,提取质粒通过华大基因测序分析。
用Expasy中ProtParam (http://web.expasy.org/cgi-bin/protparam/protparam)预测脂肪酶基因lip的蛋白质分子量以及等电点等;SOPMA预测脂肪酶的二级结构等;采用SignalP 4.1 (http://www.cbs.dtu.dk/services/SignalP/)预测蛋白质信号肽以及剪切位点,为目的蛋白高效表达提供一定基础条件。
1.2.5 重组质粒pET30a-lip 双温控自诱导表达:将重组菌Rosetta(DE3)/pET30-lip接种至含有卡那霉素(50 g/L)和氯霉素(50 g/L)的自诱导培养基中,37 °C、200 r/min培养4 h后降温到25 °C培养过夜。4 °C、4 000 r/min离心15 min收集诱导菌体。
1.2.6 脂肪酶蛋白纯化:收集双温控自诱导菌体,用预冷的结合缓冲液(20 mmol/L Tris-HCl,3 mol/L NaCl,50 mmol/L咪唑,pH 8.0)洗涤2次菌体后均匀悬浮菌体,冰浴中进行超声波破碎处理(20 min,每个循环运行5 s后暂停8 s)。4 °C、4 000 r/min离心20 min后获得含有His-tag-lip融合蛋白上清液。在自然条件下,采用Ni-Agarose His标签蛋白纯化试剂盒纯化,将获得的纯化酶液用脱盐柱(Millipore Amicon Ultrafiltration Centrifuge Tubes)进行脱盐处理后,用于酶学性质研究。纯化酶液通过SDS-PAGE电泳检测纯化效果。
1.2.7 对硝基苯酚法测LIP 水解活力:脂肪酶活力测定以对硝基苯酚丁酸酯(p-NPB)为底物,按照Gupta等[11]方法测定。反应体系总体积4 mL,反应液包含3.6 mL底物反应液(溶液A:176 μL p-NPB溶于10 mL乙腈,-20 °C保存;溶液B:50 mmol/L Tris-HCl缓冲液,pH 8.0;底物反应液:溶液A:溶液B=1:9)和400 μL粗酶液,最适温度下反应10 min后,加入等体积无水乙醇终止反应,在405 nm处测定吸光值。脂肪酶酶活单位的定义:每分钟释放1 μmol对硝基苯酚所需的酶量定义为一个酶活单位U。
1.2.8 考马斯亮蓝法测定酶蛋白浓度[12] :使用Pierce#BCA Protein Assay Kit测定蛋白浓度,以BSA (牛血清蛋白)为标准蛋白,测定不同浓度BSA在595 nm处的吸光值,以OD595为横坐标,蛋白浓度为纵坐标,绘制蛋白浓度标准曲线,据此计算目的蛋白的表达量。
1.2.9 重组脂肪酶酶学性质研究:(1) 酶最适反应温度以及稳定性研究。根据测酶活法测定纯化酶液在20-60 °C下酶活力,确定脂肪酶最适反应温度。设定酶活最高点的相对酶活力为100%,以相对酶活绘制温度-酶活曲线。
(2) 酶最适pH研究。以不同pH缓冲液配制底物反应液,pH 4.0-6.0采用50 mmol/L CH3COOH-CH3COONa缓冲液,pH 5.0-8.0采用50 mmol/L NaH2PO4-Na2HPO4缓冲液,pH 7.0-9.0采用50 mmol/L Tris-HCl缓冲液,pH 9.0-11.0采用50 mmol/L Gly-NaOH缓冲液。最适温度下测定酶活,确定最适pH。设定酶活最高点的相对酶活力为100%,以相对酶活绘制pH-酶活曲线。
(3) 金属离子及其他试剂对酶的影响。在5 mL酶液中分别加入不同金属离子(Ca2+、Mg2+、Mn2+、Na+、Li+、K+、Co2+)、PMSF (蛋白酶抑制剂)、SDS (十二烷基磺酸钠)、DMSO (二甲基亚砜)、EDTA (乙二胺四乙酸),最适温度下孵育30 min后按照1.2.7中酶活测定方法在最适条件下测定脂肪酶活性,设定不添加任何试剂的粗酶液酶活为100%。
(4) 醇类物质对酶的影响。在5 mL酶液中分别加入不同醇类物质(甲醇、乙醇、丙三醇、异丙醇),添加量分别为5%和10% (体积比),最适温度下孵育30 min后按照上述酶活测定方法在最适条件下测定脂肪酶活性,设定不添加醇类物质的粗酶液酶活为100%。
(5) 脂肪酶底物作用特性。在最适pH条件下,考察不同温度下脂肪酶对短链底物对硝基苯酚丁酸酯(p-NPB)和长链底物对硝基苯酚十六酸酯(p-NPP)的水解活性,设定p-NPB酶活最高点的相对酶活力为100%,绘制脂肪酶对不同底物的催化曲线。
2 结果与分析 2.1 Aurantiochytrium sp. PKU#SW7 脂肪酶活性鉴定通过分析Aurantiochytrium sp. PKU#SW7转录组数据,发现该菌株存在脂肪酶基因。利用筛选平板对菌株脂肪酶活性进行定性检测,结果如图1所示。筛选平板中的酸碱指示剂苯酚红在pH 7.3-7.4时为红色,当pH下降后转变为黄色。筛选平板内包含脂肪酶水解底物橄榄油,当有细胞分泌脂肪酶时,橄榄油会被分解为小分子脂肪酸,导致pH下降,筛选平板随之转变为黄色[13]。在筛选平板上打孔加入菌液上清后,28 °C培养2 d,发现小孔周围培养基变黄,说明该菌株存在显著的脂肪酶活性。该方法可以广泛用于其他微生物的脂肪酶活性定性筛选鉴定[14]。
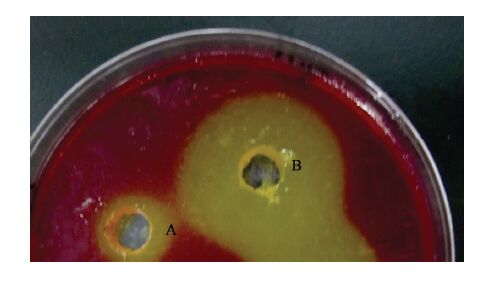
|
| 图 1 苯酚-橄榄油平板检测脂肪酶活性 Figure 1 lipase activity detected by the phenol-olive oil plate 注:A:对照组;B:实验组. 空气中CO2和培养基原液导致小孔A周围培养基pH下降,颜色变黄. Note: A: control group; B: experimental group. CO2 and M4 medium caused a decrease in pH,then slight yellow halo appeared around hole A. |
|
|
利用Expasy中的ProtParam预测该蛋白的氨基酸组成和理化性质,发现其可以编码290个氨基酸,分子式为C1467H2318N402O416S15,分子量约为32.7 kD,理论等电点为7.63。带负电的氨基酸Asp和Glu共有30个,而带正电的氨基酸Arg和Lys有37个,由此推断此蛋白为一个碱性蛋白,目前催化合成生物柴油的脂肪酶多为碱性蛋白[15, 16];含有6个Cys,说明蛋白中可能形成二硫键。通过SOPMA对LIP二级结构进行预测发现,LIP中α-螺旋占51.38%,无规则卷曲占24.48%,延伸链占16.9%,β-折叠占7.24%。SingnalP预测结构表明蛋白N端含有28个氨基酸的N端信号肽序列,最优剪切位点位于第28和第29个氨基酸之间,未剪切信号肽脂肪酶主要以无活性的包涵体形式存在,切除信号肽后,克隆表达到具有活性的成熟脂肪酶。通过NCBI中blastp对预测蛋白序列分析,该酶属于Esterase-lipase超家族;但是该蛋白序列与已知报道的微生物脂肪酶氨基酸序列相似度低,可能为微生物脂肪酶的一种新酶。根据脂肪酶保守基因序列设计引物,去除编码信号肽序列,PCR扩增后获得873 bp lip基因片段,该基因序列已提交至GenBank,登录号为KT305964。
2.3 重组LIP 的表达与纯化经过双温自诱导表达系统,重组Rosetta(pET30-lip)大量表达分泌脂肪酶蛋白LIP。使用超声破碎菌液,低温离心上样SDS-PAGE验证如图2所示,得到分子量大约为35 kD的重组酶,经过Ni柱纯化后可获得单一条带。通过考马斯亮蓝法测定粗酶液蛋白浓度和经Ni柱纯化后目的蛋白浓度,测得粗酶液蛋白量约为2.1 g/L,脂肪酶蛋白表达量约为0.8 g/L,占细胞总蛋白38%左右,说明重组质粒在宿主中可以高效表达。利用Millipore Amicon Ultrafiltration Centrifuge Tubes对经Ni柱纯化的重组LIP和His-Tag组成的融合蛋白洗脱液进行盐处理,在最适条件下测定脂肪酶活性,酶活高达102.5 U/mg。

|
| 图 2 镍柱纯化后LIP 蛋白的SDS-PAGE 分析 Figure 2 Analysis of Ni-Agarose purified LIP by SDS-PAGE 注:M:蛋白标准;A:诱导Rosetta(pET30-lip);B-E:纯化蛋白;F-G:未诱导Rosetta(DE3);H:未诱导Rosetta(pET30-lip),渗漏表达;I:诱导Rosetta(pET30-lip). Note:M: marker; A: Induced Rosetta(pET30-lip); B-E: purified; F-G: Uninduced Rosetta(DE3); H: Uninduced Rosetta(pET30-lip),leaky expressed; I: Induced Rosetta(pET30-lip). |
|
|
2.4.1 脂肪酶最适反应条件: 通过对粗酶液在20-70 °C之间进行酶活检测,设定最高酶活为100%,以相对酶活为纵坐标,做出温度与相对酶活曲线关系,如图3所示。从图3A中可以看出,该菌株脂肪酶活性最适温度在40 °C左右,但在破囊壶菌的最适生长温度30 °C左右,该酶有将近60%的相对酶活。在高温条件下(60 °C左右),该酶存在有50%酶活,但在70 °C左右该酶接近失活状态。
以不同pH缓冲液作为底物反应液,按照Gupta等[11]方法测定。在pH 5.0-6.0下,采用50 mmol/L CH3COOH-CH3COONa缓冲液、50 mmol/L NaH2PO4-Na2HPO4缓冲液分别测定活性,酶活值分别是41.2±3.4 U/mg和88.2±4.1 U/mg、42.3±4.6 U/mg和90.9±3.5 U/mg;而pH 7.0-8.0采用50 mmol/L NaH2PO4-Na2HPO4缓冲液、50 mmol/L Tris-HCl缓冲液分别测定活性,酶活值分别是95.3±2.2 U/mg和101.5±1.7 U/mg、97.4±5.4 U/mg和103.5±2.8 U/mg;pH 9.0采用50 mmol/L Tris-HCl缓冲液、50 mmol/L Gly-NaOH缓冲液分别测定活性,酶活值分别是78.9±4.2 U/mg、81.5±2.9 U/mg,说明在相同pH下更换缓冲液对该脂肪酶活性影响不大,以两种缓冲液测定酶活的平均值为该pH下脂肪酶活性。不同缓冲液主要测定pH 4.0-11.0之间的酶活变化,取最高酶活设定为100%,绘制pH-相对酶活曲线,如图3B所示。从图3中发现,该脂肪酶反应最适pH值在8.0左右,pH过酸或过碱都对酶活有极大的影响。在pH 6.0-8.0左右酶活变化幅度小,说明该酶在这一区间都具有较好作用能力。
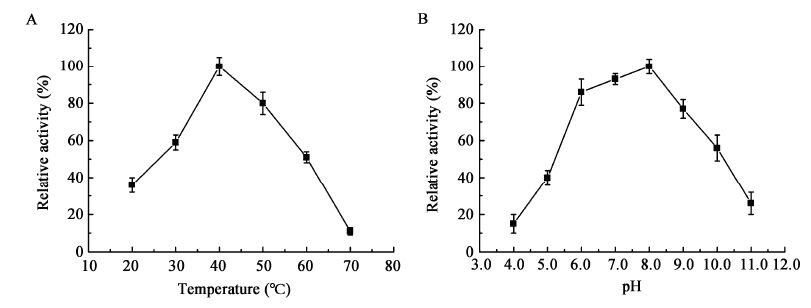
|
| 图 3 温度(A) 和pH (B) 对重组酶LIP 活性的影响 Figure 3 Effects of temperature (A) and pH (B) on the recombination LIP activity |
|
|
2.4.2 金属离子以及其他试剂对酶活影响:表1描述了7种金属离子和4种试剂对脂肪酶活性的影响情况。在金属离子中,可以发现Ca2+和Co2+在两个不同处理浓度下都对脂肪酶有激活作用,脂肪酶活性提高将近30%;相对地,Li+在两个不同浓度对脂肪酶活性都有显著抑制作用;其他的金属离子对酶活性没有表现出显著作用。除EDTA外,其他3种试剂对酶活都有显著抑制作用,由于PMSF属于细胞蛋白酶抑制剂,可以显著性地抑制细胞中各种酶的活性,因此PMSF对脂肪酶活性抑制作用最为显著。
| 添加剂 Additives | 浓度 Concentration | 相对酶活 Relative activity (%,x±s) |
| 空白对照 blank | - | 100 |
| Ca2+ | 10 mmol/L | 122±2* |
| 20 mmol/L | 136±6* | |
| Mg2+ | 10 mmol/L | 106±5 |
| 20 mmol/L | 98±3 | |
| Mn2+ | 10 mmol/L | 98±3 |
| 20 mmol/L | 95±5 | |
| Na+ | 10 mmol/L | 107±6 |
| 20 mmol/L | 110±4 | |
| Li+ | 10 mmol/L | 83±5* |
| 20 mmol/L | 70±6* | |
| K+ | 10 mmol/L | 109±3 |
| 20 mmol/L | 106±2 | |
| Co2+ | 10 mmol/L | 130±2* |
| 20 mmol/L | 141±6* | |
| PMSF | 1 mmol/L | 20±5** |
| 2 mmol/L | 0±5** | |
| EDTA | 15 mmol/L | 95±1 |
| 30 mmol/L | 93±5 | |
| SDS | 5% (V/V) | 82±4* |
| 10% (V/V) | 63±1* | |
| DMSO | 5% (V/V) | 76±5* |
| 10% (V/V) | 60±4* | |
| 注:采用Duncan’s multiple range test方法分析. *:该值与其他数值的显著性差异(p<0.05,n=3);**:该值与其他数值的显著性差异(p<0.01,n=3). | ||
| Note: Analyzed with Duncan’s multiple range test. *: the data is significantly different from others (p<0.05,n=3); **: the data is significantly different from others (p<0.01,n=3). | ||
2.4.3 醇类物质对脂肪酶酶活的影响:表2显示了4种不同醇类对脂肪酶酶活的影响。甲醇对脂肪酶抑制效果不显著;在10% (体积比)添加量情况下,其他3种醇类(乙醇、异丙醇、丙三醇)分别抑制了脂肪酶约40%、22%、17%酶活。在20% (体积比)添加量情况下,其他3种醇类(乙醇、异丙醇、丙三醇)分别抑制了脂肪酶约58%、40%、25%的酶活。
| 添加剂 Additives | 浓度 Concentration (%,V/V) | 相对酶活 Relative activity (%,x±s) |
| 空白对照 blank | - | 100 |
| 甲醇 Methanol | 10 | 95±2 |
| 20 | 90±4 | |
| 乙醇 Ethanol | 10 | 60±5* |
| 20 | 42±3** | |
| 丙三醇 Glycerol | 10 | 83±3* |
| 20 | 75±5* | |
| 异丙醇 Isopropanol | 10 | 78±6* |
| 20 | 60±4* | |
| 注:采用Duncan’s multiple range test方法分析. *:该值与其他数值的显著性差异(p<0.05,n=3);**:该值与其他数值的显著性差异(p<0.01,n=3). | ||
| Note: Analyzed with Duncan’s multiple range test. *: the data is significantly different from others (p<0.05,n=3); **: the data is significantly different from others (p<0.01,n=3). | ||
2.4.4 脂肪酶底物作用特性: 在最适pH 8.0条件下,考察了重组LIP对短链底物p-NPB和长链底物p-NPP的酶解活力,结果如图4所示,该脂肪酶对p-NPB的最适作用条件为pH 8.0、40 °C,对p-NPP的最适作用条件为pH 8.0、50 °C;对p-NPB的酶解活力高于p-NPP。根据每分钟释放1 μmol对硝基苯酚所需的酶量定义为一个酶活单位,lip脂肪酶在不同最适温度下酶解短链底物p-NPB和p-NPP的活力分别为102.5±2.6 U/mg和70±3.1 U/mg。
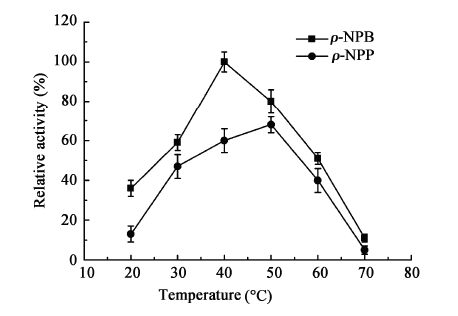
|
| 图 4 脂肪酶对p -NPB 和p- NPP 的水解活力 Figure 4 Enzyme activity of hydrolysis of p-NPB and p-NPP |
|
|
传统IPTG方法诱导重组质粒pET30a-lip只能微量表达,获得目的蛋白量少。然而采用双温度控制自诱导培养技术表达脂肪酶蛋白,经Ni柱纯化后目的蛋白占粗蛋白的38%,对底物p-NPB脂肪酶酶活约为102.5±2.6 U/mg,因此双温控自诱导培养可以实现异源系统高效表达。杨鑫等[17]利用自诱导双温控制方式在大肠杆菌细胞内表达海藻糖合成酶,与传统IPTG恒温培养方式相比酶活提高了55.25%,海藻糖得率达71%。在酶的水解反应过程中,脂肪酶活性通常会受到重金属离子的影响[18]。在本研究中发现Ca2+和Co2+对该酶活性有显著促进作用,可将酶活提高1.3倍左右;金属离子对酶活的促进作用可能是由于金属离子与酶结合,使底物更容易与酶的活性中心部位结合,酶的最佳活力构象更趋于稳定[19]。4种不同生化试剂均对该酶具有显著的抑制作用,丝氨酸脂肪酶抑制剂PMSF可完全抑制该酶酶活,由此可以推测出该酶具有以丝氨酸为活性中心的催化三元组。优良的生物柴油 催化剂需要具备温和反应温度和醇类物质耐受性等特征[20],在醇类对酶活影响研究中,发现甲醇对重组LIP抑制效果不显著;甲醇作为生物柴油炼制另一原材料,减少甲醇对生物催化剂的毒害作用是提高生产效率的关键[21],重组LIP对甲醇存在耐受性,可以在20% (体积比)甲醇浓度条件下保持90%以上酶活。丙三醇作为生物柴油炼制的副产品,对生物催化剂也有毒害作用。重组LIP在20% (体积比)丙三醇浓度条件下保持75%左右酶活,说明该酶对丙三醇存在一定耐受性。
目前关于海洋微生物脂肪酶克隆的表达报道[22, 23, 24]产物多为低温碱性脂肪酶,但在生物柴油生产反应中最适温度要求在35-60 °C[20],本研究发现海洋微生物破囊壶菌Aurantiochytrium sp. PKU#SW7脂肪酶在30-60 °C对中长链脂肪酸底物都存在酶解活力,可以满足生物柴油转化过程中的温度要求。Zhang等[23]、Park等[25]发现的海洋微生物脂肪酶作用底物范围在C4-C10之间,而该酶主要作用底物范围在C10-C16的中长链脂肪酸,属于生物柴油原材料范畴。LIP在pH 6.0-10.0存在60%以上酶活,pH 8.0时达到最高酶活,已知报道的用于催化合成生物柴油的脂肪酶也多为碱性脂肪酶[15, 16],LIP与之相符也属于碱性脂肪酶。目前工业上利用酶促反应催化生产生物柴 油成本太高,往往取决于酶的固定化[3]。破囊壶菌Aurantiochytrium sp. PKU#SW7本身具有脂肪酶活力和高含量的脂肪酸(如棕榈酸),从微生物全细胞脂肪酶催化合成生物柴油的角度出发,将破囊壶菌细胞作为直接催化剂对脂肪酸(如棕榈酸)进行转酯化,可以极大地降低生产成本,实现绿色经济生物炼制。
基于本研究结果,优化脂肪酶LIP表达条件,提高脂肪酶产量和比酶活,摸索破囊壶菌脂肪酶转酯化反应条件等可作为未来研究探索方向,从而为利用该菌株自身脂肪酶生物转化棕榈酸合成生物柴油奠定基础。
| [1] | Hasan F, Shah AA, Hameed A. Industrial applications of microbial lipases[J]. Enzyme & Microbial Technology, 2006, 39 (2): 235–251. |
| [2] | Tan ZF, Wang YP, Chen LH, et al. Application and research advances of microbial lipases in the industry[J]. Science and Technology of Food Industry, 2006, 27 (7): 193–195. (in chinese) 谈重芳, 王雁萍, 陈林海, 等. 微生物脂肪酶在工业中的应用及研究进展[J]. 食品工业科技, 2006, 27 (7):193–195. |
| [3] | Xue Y. The research of microbial lipases and catalytic synthesis of biodiesel[D]. Wuhan: Master’s Thesis of Huazhong University of Science and Technology, 2004 (in Chinese) 薛勇. 微生物脂肪酶及其催化合成生物柴油的研究[D]. 武汉: 华中科技大学硕士学位论文, 2004 |
| [4] | Liu Y, Singh P, Sun Y, et al. Culturable diversity and biochemical features of thraustochytrids from coastal waters of Southern China[J]. Applied Microbiology & Biotechnology, 2013, 98 (7): 3241–3255. |
| [5] | Calbet A, Landry MR. Phytoplankton growth, microzooplankton grazing, and carbon cycling in marine systems[J]. Limnology & Oceanography, 2004, 49 (1): 51–57. |
| [6] | Huang HQ, Bao SX. Studies on production of docosahexaenoic acid by Thraustochytrium aureum[J]. Acta Microbiologica Sinica, 2002, 42 (4): 9–11. (in chinese) 黄惠琴, 鲍时翔. 金黄色破囊壶菌发酵生产DHA的研究[J]. 微生物学报, 2002, 42 (4):9–11. |
| [7] | Du B. Optimization and metabolic control of DHA fermentation[D]. Guangzhou: Doctoral Dissertation of South China University of Technology, 2001 (in Chinese) 杜冰. DHA的发酵生产及代谢调控研究[D]. 广州: 华南理工大学博士学位论文, 2001 |
| [8] | Liu Y, Tang J, Li JJ, et al. Efficient production of triacylglycerols rich in docosahexaenoic acid (DHA) by osmo-heterotrophic marine protists[J]. Applied Microbiology & Biotechnology, 2014, 98 (23): 9643–9652. |
| [9] | Damare S, Raghukumar C, Raghukumar S. Fungi in deep-sea sediments of the Central Indian Basin[J]. Deep Sea Research Part I: Oceanographic Research Papers, 2006, 53 (1): 14–27. |
| [10] | Studier FW. Protein production by auto-induction in high-density shaking cultures[J]. Protein Expression and Purification, 2005, 41 (1): 207–234. |
| [11] | Gupta N, Rathi P, Gupta R. Simplified para-nitrophenyl palmitate assay for lipases and esterases[J]. Analytical Biochemistry, 2003, 311 (1): 98–99. |
| [12] | Yang JX. Biochemistry and Molecular Biology Technology Tutorials. Beijing: Science Press[M]. 2009: 35 -36. (in chinese) 杨建雄. 生物化学与分子生物学实验技术教程. 北京: 科学出版社[M]. 2009: 35 -36. |
| [13] | Singh R, Gupta N, Goswami VK, et al. A simple activity staining protocol for lipases and esterases[J]. Applied Microbiology & Biotechnology, 2006, 70 (6): 679–682. |
| [14] | Wang H, He LP, Zhou HJ, et al. Determination methods for lipase activity and its application in the screening of microbial lipase[J]. Biotechnology Bulletin, 2013 (1): 203–208. (in chinese) 王欢, 何腊平, 周换景, 等. 脂肪酶活力测定方法及其在筛选产脂肪酶微生物中的应用[J]. 生物技术通报, 2013 (1):203–208. |
| [15] | Yoo HY, Simkhada JR, Cho SS, et al. A novel alkaline lipase from Ralstonia with potential application in biodiesel production[J]. Bioresource Technology, 2011, 102 (10): 6104–6111. |
| [16] | Cai XH, Ma J, Wei DZ, et al. Functional expression of a novel alkaline-adapted lipase of Bacillus amyloliquefaciens from stinky tofu brine and development of immobilized enzyme for biodiesel production[J]. Antonie van Leeuwenhoek, 2014, 106 (5): 1049–1060. |
| [17] | 杨鑫, 吴茜, 崔怀言, 等. 灰产色链霉菌海藻糖合成酶的自诱导表达及两阶段温度控制发酵的优化[J]. 食品与发酵工业, 2015, 41 (5):14–18. |
| [18] | Wang JZ, Feng X, Wang LJ. Study on the influence of metal ions to activity of rice bran lipase[J]. Food Science & Technology, 2007, 32 (6): 46–49. (in chinese) 王吉中, 冯昕, 王丽娟. 金属离子对米糠解脂酶活性影响的研究[J]. 食品科技, 2007, 32 (6):46–49. |
| [19] | Invernizzi G, Papaleo E, Grandori R, et al. Relevance of metal ions for lipase stability: structural rearrangements induced in the Burkholderia glumae lipase by calcium depletion[J]. Journal of Structural Biology, 2009, 168 (3): 562–570. |
| [20] | Antczak MS, Kubiak A, Antczak T, et al. Enzymatic biodiesel synthesis-key factors affecting efficiency of the process[J]. Renewable Energy, 2009, 34 (5): 1185–1194. |
| [21] | Kaieda M, Samukawa T, Kondo A, et al. Effect of methanol and water contents on production of biodiesel fuel from plant oil catalyzed by various lipases in a solvent-free system[J]. Journal of Bioscience and Bioengineering, 2001, 91 (1): 12–15. |
| [22] | Jeon JH, Kim JT, Kim YJ, et al. Cloning and characterization of a new cold-active lipase from a deep-sea sediment metagenome[J]. Applied Microbiology and Biotechnology, 2009, 81 (5): 865–874. |
| [23] | Zhang JW, Lin S, Zeng RY. Cloning, expression, and characterization of a cold-adapted lipase gene from an Antarctic deep-sea psychrotrophic bacterium, Psychrobacter sp[J]. Journal of Microbiology and Biotechnology, 2007, 17 (4): 604–610. |
| [24] | Florczak T, Daroch M, Wilkinson MC, et al. Purification, characterisation and expression in Saccharomyces cerevisiae of LipG7 an enantioselective, cold-adapted lipase from the Antarctic filamentous fungus Geomyces sp[J]. Enzyme and Microbial Technology, 2013, 53 (1): 18–24. |
| [25] | Park HJ, Jeon JH, Kang SG, et al. Functional expression and refolding of new alkaline esterase, EM2L8 from deep-sea sediment metagenome[J]. Protein Expression and Purification, 2007, 52 (2): 340–347. |
 2016, Vol. 43
2016, Vol. 43




