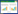扩展功能
文章信息
- 崔恒林
- CUI Heng-Lin
- 嗜盐古菌分类学研究进展
- Taxonomy of halophilic archaea: a review
- 微生物学通报, 2016, 43(5): 1113-1122
- Microbiology China, 2016, 43(5): 1113-1122
- DOI: 10.13344/j.microbiol.china.150920
-
文章历史
- 收稿日期: 2015-11-15
- 接受日期: 2016-01-07
- 优先数字出版日期(www.cnki.net): 2016-02-01
地球上的盐环境按起源可分为海洋型(Thalassohaline)和非海洋型(Athalassohaline)两大类。海洋型盐环境是由海水蒸发而成,盐离子组成比例与浓缩海水相同或相近的高盐体系,主要包括人工的海水盐田(Marine solar saltern)和天然的海洋泻湖(Marine lagoon);非海洋型盐环境是由环境中的矿物质溶解积累而成、盐离子组成比例与海水不同的高盐体系,主要包括内陆盐湖(Salt lake)、盐矿(Salt mine)和盐井(Salt well)[1]。基于16S rRNA基因文库的免培养研究结果表明,两大类起源不同的盐环境均蕴藏着丰富多样的嗜盐古菌类群[2-3]。作为高盐环境的特有类群,嗜盐古菌驱动着高盐生态系统的生物地球化学循环,成为盐环境保护和利用的重要指示微生物。
到目前为止,已发现的嗜盐古菌都属于古菌域(Archaea)的广古菌门(Euryarchaeota),主要包括:嗜盐甲烷古菌类群(Halophilic methanogenic archaea)、嗜盐古菌纲(Halobacteria)的全部成员以及尚不能培养的纳米嗜盐古菌类群(Nanohaloarchaea)[4]。嗜盐甲烷古菌类群主要包括甲烷八叠球菌科(Methanosarcinaceae)和甲烷卵石形菌科(Methanocalculaceae)的部分物种,这些物种除Methanohalobium evestigatum为极端嗜盐菌(生长盐浓度范围15%−30%,最适生长盐浓度25%)外,其余的都属于中度嗜盐菌或耐盐菌[4-5]。由于这些嗜盐甲烷古菌类群生长缓慢、对氧极其敏感导致实验操作困难,关于此类群的研究较少。这些嗜盐甲烷古菌类群所处的属、科也存在非嗜盐类群,因此嗜盐甲烷古菌类群并非单系的分类群,因而不属于传统意义上的嗜盐古菌。传统意义上的嗜盐古菌是指属于古菌域、广古菌门的嗜盐古菌纲的全部物种[4]。纳米嗜盐古菌类群是近年通过“宏基因组”组装技术揭示的“微生物暗物质”类群,可能代表广古菌门的一个未知的纲,目前尚不能培养[6-7]。
我国东、南部沿海建设有上百家海水晒盐场,中西部地区分布着近千个内陆盐湖、盐矿和盐井,这些不同起源的盐环境蕴藏极为丰富的嗜盐古菌,形成了独特的种群分布与群落结构,是嗜盐古菌分类研究的珍贵资源。在过去的30年中,我国在嗜盐古菌分类方面取得了较多的研究成果,但相对于数量巨大、类型丰富的盐环境资源而言,我国嗜盐古菌分类研究仍需拓展。本文根据近年来的研究,综合前人研究成果,聚焦传统意义上的嗜盐古菌,从嗜盐古菌分类学地位的变迁、分类学方法、分类学研究现状等方面,总结我国嗜盐古菌分类学研究领域取得的成绩,探讨未来嗜盐古菌分类学研究的重点与方向。
1 嗜盐古菌分类学地位的变迁 1.1 古代先民对“红池”现象的记述我国有着悠久的制盐历史[8],古代先民在制盐生产中对卤水变红的现象早有记述。我国北宋沈括(1031−1095) 所著《梦溪笔谈》(卷三,辨证一)就有“卤色正赤”的描述。在气温较高的春末至早秋季节,水分的蒸发导致盐池卤水盐浓度升高,在较适宜的温度及盐浓度下,盐池卤水中的嗜盐古菌大量生长繁殖使得卤水呈现红色,即“红池”现象。“红池”也是盐田产盐丰收的先兆,相关地方志或盐志类古代文献可能不乏此类记载。从可查阅的相关古代文献中并未见我国古代先民对“红池”现象生物学本质的认识和记载,只是古代先民不自觉地将“红池”现象与“盐田丰收”联系在一起。在圣经和古埃及的相关文献中也有对死海(Dead Sea)、尼罗河(Nile)和瓦迪纳特伦湖(Wadi Natrun lake)卤水变红的记载[1]。
1.2 早年关于嗜盐菌的研究腌肉、咸鱼以及盐渍毛皮的腐败变红常常影响着人们的生产和生活,对这一现象的关注和研究最终导致对嗜盐菌生物学本质的认识。
1771年,Monro记述了用产自利比亚的黎波里(Tripoli)的海盐腌制的肉会变红[1]。1880年,Farlow指出腌制鳕鱼在夏季变红的主要原因——红色嗜盐球菌的生长[9]。1914年,Becker首次报道嗜盐菌导致腌制皮张变红[10]。Pierce在1914年首次确切地描述了卤水中的红色细菌(现归入古菌域)[1]。Klebahn在1919年首次描述了盐杆菌(Halobacterium),这些分离自变红的腌鱼的红色杆状嗜盐菌被命名为Bacillus halobius ruber[1]。1922年Harrison和Kennedy在加拿大也分离到与Bacillus halobius类似的微生物并描述为Pseudomonas salinaria,后来又改名为Serratia salinaria,并收录在第一版的伯杰氏细菌鉴定手册中[1]。1934年Lochhead描述了分离自加拿大西部地区腌制牛皮的Serratia cutirubra[11]。1943年,Lochhead认识到Serratia salinaria、Serratia cutirubra与Serratia属的模式种Serratia marcescens在多个特征上存在差异,建议将前两个物种重新划归Pseudomonas属,分别命名为Pseudomonas salinaria和Pseudomonas cutirubra[12]。
1.3 《伯杰氏手册》中的嗜盐古菌类群早年的研究者对红色嗜盐菌的研究主要包括菌株分离及生物学特性描述。由于研究者之间缺乏密切交流和对相关物种的比较研究,往往出现同一物种的多次分离及重复描述,由此导致同一物种分类学地位的混乱。不同版《伯杰氏手册》对嗜盐古菌分类学地位的不同界定反映了早期研究中嗜盐古菌分类学地位的变迁。
《伯杰氏细菌鉴定手册》第六版[13]将嗜盐菌划入Pseudomonas (Pseudomonas salinaria、Pseudomonas cutirubra)、Micrococcus (Micrococcus littoralis、Micrococcus morrhuae)、Sarcina (Sarcina littoralis)和Flavobacterium (Flavobacterium halobium、Flavobacterium marismortui、Flavobacterium trapanicum)四个属。
在《伯杰氏细菌鉴定手册》第七版[14]中,设立盐杆菌属(Halobacterium),包括Halobacterium salinarium、Halobacterium cutirubrum、Halobacterium halobium、Halobacterium marismortui和Halobacterium trapanicum。而球状嗜盐菌则仍归入Micrococcus (Micrococcus morrhuae)和Sarcina (Sarcina littoralis)两个属。
值得关注的是,在1974年出版的《伯杰氏细菌鉴定手册》第八版[15]中建立了嗜盐菌科(Halobacteriaceae)下设盐杆菌属(Halobacterium)和盐球菌属(Halococcus)。盐杆菌属(Halobacterium)包含Halobacterium salinarium、Halobacterium halobium两个种和三个未确定的种:Halobacterium marismortui、Halobacterium trapanicum和Amoebobacter morrhuae。盐球菌属(Halococcus)只有Halococcus morrhuae一个种。至此,红色嗜盐菌作为同一个类群划归同一科。
具有里程碑意义的是,在“三域学说”的影响下,1990年出版的《伯杰氏系统细菌学手册》第一版第三卷[16]中,设立古细菌类群(Archaeobacteria),其下包含极端嗜盐古细菌群,并依次设立嗜盐菌目(Halobacteriales)、嗜盐菌科(Halobacteriaceae),其下包括盐杆菌属(Halobacterium)、盐球菌属(Halococcus) 、盐小盒菌属(Haloarcula)、盐富饶菌属(Haloferax)、盐碱杆菌属(Natronobacterium)和盐碱球菌属(Natronococcus)六个属。
2001年出版的《伯杰氏系统细菌学手册》第二版第一卷[17]设立古菌域(Archaea)、广古菌门(Euryarchaeota)、嗜盐菌纲(Halobacteria)、嗜盐菌目(Halobacteriales)及嗜盐菌科(Halobacteriaceae),其下包括14个属:盐杆菌属(Halobacterium)、盐小盒菌属(Haloarcula)、盐棒菌属(Halobaculum)、盐球菌属(Halococcus)、盐富饶菌属(Haloferax)、盐几何型菌属(Halogeometricum)、盐红菌属(Halorubrum)、盐土菌属(Haloterrigena)、钠白菌属(Natrialba)、钠线菌属(Natrinema)、盐碱杆菌属(Natronobacterium)、盐碱球菌属(Natronococcus)、盐碱单胞菌属(Natronomonas)和盐碱红菌属(Natronorubrum)。至此,嗜盐古菌的纲级分类单元确立。
2 嗜盐古菌分类学方法嗜盐古菌分类学是基于20世纪70年代Colwell提出的多相分类学[18-19],即综合考虑微生物的表型、基因型和系统发育等多种信息的分类。1997年,Oren等[20]提出了“描述嗜盐古菌分类单元的最低标准”(表 1),其中规定了描述嗜盐古菌分类单元的最低标准,从而规范了嗜盐古菌的分类和鉴定。
| 重要性 Importance | 指标 Minimal standards |
| 必须做 Required | 菌落形态、细胞形态 Colonial morphology,cell morphology |
| 运动性 Motility | |
| 色素产生 Pigmentation | |
| 革兰氏染色 Gram stain | |
| 细胞保持完整形态所需的盐浓度 Salt concentrations required to prevent cell lysis | |
| 生长所需的最适NaCl和MgCl2浓度 Optimum NaCl and MgCl2 concentrations for growth | |
| 生长盐浓度范围 Range of salt concentrations enabling growth | |
| 生长温度和pH范围 Temperature and pH ranges for growth | |
| 利用硝酸盐厌氧生长 Anaerobic growth in the presence of nitrate | |
| 硝酸盐还原、硝酸盐产气 Reduction of nitrate to nitrite,formation of gas from nitrate | |
| 利用精氨酸厌氧生长 Anaerobic growth in the presence of arginine | |
| 碳源利用 Ability to grow on a range of single carbon sources | |
| 利用碳源产酸 Production of acids from a range of carbohydrates | |
| 过氧化氢酶和氧化酶活性 Catalase and oxidase activities | |
| 吲哚产生 Formation of indole | |
| 淀粉水解、明胶水解 Starch hydrolysis,gelatin hydrolysis | |
| 酪素水解、吐温80水解 Casein hydrolysis,Tween 80 hydrolysis | |
| 抗生素敏感性 Sensitivity to different antimicrobials | |
| 极性脂组分 Characterization of polar lipids | |
| 糖脂类型 Types of glycolipids present | |
| 是否存在磷脂酰甘油硫酸(PGS) Presence or absence of phosphatidylglycerosulfate (PGS) | |
| DNA G+C mol%含量 G+C content of DNA | |
| 16S rRNA核苷酸序列信息 16S rRNA nucleotide sequence information | |
| 与相近种的DNA-DNA杂交(仅用于新种的描述) DNA-DNA hybridization with related species (for descriptions of new species only) | |
| 建议做 Recommended | 电镜照片 Electron microscopy |
| 利用DMSO或TMAO厌氧生长 Anaerobic growth in the presence of DMSO or TMAO | |
| 磷酸酶活性、尿素酶活性 Phosphatase activity,urease activity | |
| β-半乳糖苷酶活性β-Galactosidase activity | |
| 赖氨酸脱羧酶活性 Lysine decarboxylase activity | |
| 鸟氨酸脱羧酶活性 Ornithine decarboxylase activity | |
| 细胞外被是否存在糖蛋白 Presence of glycoprotein in the cell envelope | |
| 是否产生PHA、是否存在质粒 Presence of PHA,presence of plasmids | |
| 全细胞蛋白电泳 Electrophoresis of cellular proteins |
在嗜盐古菌的分类鉴定中,经典或传统表型特征是非常重要的特征指标,包括形态及培养特征、生理生化特性和对抗生素的敏感性等[20]。表型特征主要运用于纲、目、科、属和种各级分类单元的描述。如何界定各级分类单元的特征?不同的研究者可能会给出不同的描述。只有建立在一定样本数基础之上的特征描述才是正确的、可信的,即在描述种级特征时,获得多株且来自不同地域菌株的表型特征,在平行比较的基础之上,提出这些菌株的共有特征作为种级特征,高级分类单元的表型特征描述依赖于足够多的低一级分类单元特征。
细胞化学组分主要包括细胞壁氨基酸组成、细胞膜脂类型、全细胞水解液单糖组分、全细胞脂肪酸组成以及醌类型。对于大多数细菌来说,上述指标有着重要的分类学价值。对嗜盐古菌而言,细胞膜脂类型即细胞中的极性脂组分在属级分类单元上具有较好的分辨力。极性脂由极性的头部和非极性的尾部构成。所有嗜盐古菌极性脂的尾部都含有二植烷(C20C20)甘油醚核心脂,但一些菌株另外还含有植烷半萜(C20C25)甘油醚核心脂,某些嗜盐碱古菌的菌株含二半萜(C25C25)甘油醚脂。嗜盐古菌极性脂的头部都含磷脂酰甘油(Phosphatidyl glycerol,PG)、磷脂酰甘油磷酸甲基酯(Phosphatidyl glycerol phosphate methyl ester,PGP-Me),有些属的嗜盐古菌还含磷脂酰甘油硫酸(Phosphatidyl glycerol sulphate,PGS)。在中性嗜盐古菌细胞中含有一种或多种糖脂,这些糖脂包括:二糖基二醚(Diglycosyl diether,DGD)、三糖基二醚(Triglycosyl diether,TGD)、硫酸二糖基二醚(Sulfated diglycosyl diether,S-DGD)、硫酸三糖基二醚(Sulfated triglycosyl diether,S-TGD)、硫酸四糖基二醚(Sulfated tetraglycosyl diether,S-TeGD)和二硫酸四糖基二醚(Disulfated diglycosyl diether,S2-DGD)[20-21]。
基因组DNA G+C mol%含量是一个经典的基因型特征指标。一般认为,同种内的不同菌株的DNA G+C mol%含量的差别应小于3%,同属内的不同种的G+C mol%含量的差别应小于5%。嗜盐古菌属于高G+C mol%含量的类群,染色体DNA G+ C mol%含量介于46.9%−71.2%之间[21-22]。不同的测定方法测定的G+C mol%含量可能存在差异,随着基因组测序技术的普及,G+C mol%含量应该会更精确。
16S rRNA基因序列分析及系统进化关系推断在嗜盐古菌种级以上分类单元的界定中具有较强的分辨力[23]。RNA聚合酶B亚基基因(rpoB′)[24-25]在嗜盐古菌分类单元的界定中也具有重要应用价值。多位点序列分析(Multilocus sequence analysis)在菌株水平上具较高的分辨力[26]。
DNA-DNA同源性分析是原核微生物新种描述中非常重要的“黄金标准”,一般认为,DNA-DNA杂交同源性大于等于70%的菌株应归入同一个 种[27-28]。由于运用不同杂交方法获得的DNA-DNA同源性数据不一致,且不同研究者获得的相关菌株间的DNA-DNA杂交同源性数据不具可比性,因此,这一费时费力的方法被研究者排斥。根据已有数据,研究者统计出70%的DNA-DNA同源性与98.2%−99.0%的16S rRNA基因相似性对应[29]。而基于全基因组数据的“平均核苷酸一致性”(Average nucleotide identity,ANI)可成为替代DNA-DNA同源性的重要指标[30]。Kim等分析了属于22个门的 6 787个原核微生物的基因组,认为95%−96%的ANI对应于98.65%的16S rRNA基因相似性[31]。基于全基因组数据的系统基因组学分析(Phylogenomic analyses)可能更准确的界定嗜盐古菌的各级分类单元[32]。
3 嗜盐古菌分类学研究现状近年来,随着研究者对可培养研究技术的探索,来自不同盐环境的多种嗜盐古菌新类群获得纯培养[22, 33]。一个典型的例子就是Walsby四方形嗜盐古菌的分离与培养。此类微生物1980年首次被发现[34],普遍存在于盐池卤水或盐田卤水中,且数量极大,但这种微生物一直难以获得纯培养。直至2004年,两个研究组获得此类微生物的纯培养[35, 36],并于2007年描述了Haloquadratum walsbyi[22],从而了却二十多年一直萦绕在许多嗜盐古菌分类学研究者心头的缺憾。由此可知,设计新的实验方案,运用不同于以往的分离技术肯定能更加全面的认识嗜盐古菌物种资源的多样性。截至2015年12月底,嗜盐古菌纲包含52个属、213个种(图 1)。

|
| 图 1 基于16S rRNA基因的嗜盐古菌纲各模式种的系统发育树 Figure 1 Phylogenetic tree for type species of genera of the class Halobacteria based on 16S rRNA gene sequences 括号内序号为该物种序列在GenBank中的登录号,分支点上的数字表示该分支的自举检验值,标尺为每核苷酸位点上预期的替换率. Numbers in brackets are GenBank accession numbers. Numbers on branch points are bootstrap values (%). Bar represents expected substitutions per nucleotide position. |
|
|
随着大量新类群的描述,嗜盐古菌纲的分类学存在以下问题:
(1) 基于极性脂组分的化学分类标准在属级分类界定上的不一致性。中性嗜盐古菌一般既含磷脂又含糖脂,而嗜碱类群一般只含磷脂。因此,在全部由中性嗜盐古菌或全部由嗜碱类群组成的属中,属级特征极性脂组成就是其模式种的极性脂组分。而那些包含中性类群和嗜碱类群的属(Halalkalicoccus、Halobiforma、Halorubrum、Halostagnicola、Natrialba、Natronococcus、Natronomonas、Natronorubrum)的特征性极性脂则与模式种的极性脂组成不一致[21]。加上不含糖脂的中性类群的发现[37],撼动了基于极性脂组分的化学分类标准在嗜盐古菌属级分类中的重要地位。
(2) 对16S rRNA基因异源性认识不足导致嗜盐古菌属级界定不清晰。
嗜盐古菌纲有15个属存在种内的rRNA基因多态性,嗜盐古菌同基因组内不同拷贝的16S rRNA基因序列差异最高达9%[38],16S rRNA基因异源性必然会影响嗜盐古菌属级界定。Haloterrigena和Natrinema两个属的分类地位之争由来已久[39],而Natrinema ejinorense和Natronorubrum sediminis两物种混乱的归类,使得上述问题更加复杂[40-41];加上Halosarcina和Halogeometricum两个属成员的错误归类[42-43],导致基于16S rRNA基因的分子分类体系呈现混乱状态,甚至已经影响到嗜盐古菌分子生态学研究[3, 44],势必会高估环境中微生物的多样性[45]。
(3) 基于rpoB'基因的系统发育格局与基于16S rRNA基因系统发育格局的矛盾。与16S rRNA基因系统发育格局不同,rpoB'基因的系统发育分析结果将Natronolimnobius属的Natronolimnobius innermongolicus划入Haloterrigena-Natrinema类群,而将Natronorubrum tibetense组建一个新属[25]。
这些分类学问题的出现,归根结底是对嗜盐古菌多样性类群认识不够,缺乏对多样性类群的表型特征、化学组分和多基因系统发育关系的全面系统的比较研究。笔者认为,可以借助现代质谱分析技术,从确切的化学组分分析入手,比较嗜盐古菌种属水平的极性脂组成;依托基因组测序和分析技术,基于多基因分析,推断嗜盐古菌系统演化关系,进而解决目前嗜盐古菌分类学存在的问题。
在嗜盐古菌纲的目和科级分类单元的分类上,Gupta等运用系统基因组学和特征性分子指征分析技术考察了来自30个属的100多个嗜盐古菌的基因组,发现近三分之二的分类单元构成两个主要的分支群(Clade A和Clade B),Gupta等根据这一结果建立两个新目:盐富饶菌目(Haloferacales)和钠白菌目(Natrialbales)以及对应的两个新科:盐富饶菌科(Haloferacaceae)和钠白菌科(Natrialbaceae)[32]。剩余的嗜盐古菌仍留在原来的盐杆菌目(Halobacteriales)下的盐杆菌科(Halobacteriaceae)。值得注意的是,这些剩余的嗜盐古菌类群之间的系统发育关系属于并系类群而非单系类群,可能代表不同的目级分类单元,因此,Gupta等建议的分类格局仍有待更多基因组数据的支撑。
4 我国的嗜盐古菌分类学研究我国的嗜盐古菌分类学研究开始于20世纪 80年代初,中国科学院微生物研究所的王大珍、周培瑾、田新玉和复旦大学的张纪忠、南京师范大学的秦怀兰等前辈,对我国西藏、新疆、内蒙、青海地区的盐(碱)湖以及东部沿海的盐田开展了大规模的嗜盐微生物资源调查,分离了一大批具有区域特色的嗜盐古菌菌株[46-52]。先后描述了“Halobacterium dachaidanensis”、“Halobacterium tangguensis”、“Halobacterium zhabeiensis”、“Halobacterium haloalcaligenum”、“Haloarcula aidinensis”、“Natronobacterium innermongoliae”、“Natrilba wudunaoensis”等新类群。虽然这些嗜盐古菌种名未获“有效发表”(valid publication),但这些开创性的工作为我国嗜盐古菌分类学后续研究奠定了坚实的基础。
1999年中国科学院微生物研究所的徐毅等[53]研究了分离自西藏班戈湖的嗜盐古菌菌株A33和GA33,建立Natronorubrum属,描述两个嗜盐碱嗜盐古菌Natronorubrum bangense和Natronorubrum tibetense。此研究结果发表于“International journal of systematic bacteriology”上,这是我国研究者首次在国际微生物分类学权威期刊上发表嗜盐古菌新分类单元。
进入21世纪,中国科学院微生物研究所微生物资源前期开发国家重点实验室的研究者开展了嗜盐古菌分类学的国际合作研究[54]。先后合作建立了Halopiger、Halostagnicola、Halovivax、Natronolimnobius四个新属,描述了12个新种。相关国外的合作者近期仍在发表分离自我国的新类群[55],足见国际合作研究对我国嗜盐古菌分类学研究的影响。截止2015年11月底,我国研究者描述或参与描述的分离自我国的嗜盐古菌新类群共 18个属、102个种。值得一提的是,笔者以第一作者或通讯作者先后描述了其中的11个属、55个种。
在嗜盐古菌分类学方面,Qiu等[43]理清了Halogeometricum和Halosarcina两个属的分类关系,将Halosarcina属现有的种归入Halogeometricum属。
目前,在我国的嗜盐古菌分类学前辈们的带领、指导下,国内不少高校和研究机构的研究人员开展了嗜盐古菌的分类学及相关研究。他们是浙江大学的吴敏课题组、国家海洋局第二海洋研究所的许学伟课题组、中山大学(云南大学)的李文均课题组、塔里木大学的张利莉课题组以及江苏大学的崔恒林课题组等。这些课题组聚焦于我国不同地域的高盐环境,从单纯的描述新种发展为开展不同盐环境嗜盐古菌多样性的比较研究,并利用免培养和可培养技术揭示不同盐域的嗜盐古菌多样性,获得了多样性的嗜盐古菌类群。可以预见,这些课题组的研究成果将会极大推动我国嗜盐古菌分类学的研究。
5 展望为了嗜盐古菌分类学的发展,针对目前存在的分类学问题,必须建立新的分类标准。新分类标准的建立必须在多相分类学理论的指导下,依赖于基因组测序和分析技术、现代质谱检测和分析技术从基因组层面、细胞化学组分层面考察与总结嗜盐古菌种、属、科、目、纲等分类单元的特征,建立种、属、科、目、纲等分类单元的新分类标准。同时,新的分类标准还必须建立在针对不同盐环境多样性类群研究的基础上。大量的免培养研究结果表明,高盐环境中蕴藏着大量尚不能培养的嗜盐古菌类群[56]。由于免培养研究难以揭示这些类群细胞水平的生物学特征,因此,其分类学价值也无从谈起。只有重视可培养技术研究,以基于高通量测序技术的宏基因组学研究结果为指导,开展不同盐环境嗜盐古菌可培养研究,培养出先前发现的未培养类群,获得多样性的嗜盐古菌,才能真正认识曾经出现在进化树上的未知类群。
随着研究者对高盐环境的关注,对嗜盐古菌的分类学研究可能会在以下方面重点展开:
(1) 难培养嗜盐古菌的分离和纯培养。在利用培养和免培养技术研究高盐环境嗜盐古菌多样性时,免培养技术揭示的物种多样性远高于可培养物种多样性。设计针对性的培养基及添加特定的诱导因子可能获得更多的从未获分离的嗜盐古菌新类群。
(2) 特殊生理、生态型嗜盐古菌的基因组学和蛋白质组学的比较分类学研究。随着基因组测序技术的发展,越来越多的特殊生理、生态型嗜盐古菌的基因组被测定,在基因水平和蛋白质水平比较研究这些特殊类群的特殊机能,不仅能够揭示这些特殊生命的适应机制,而且能够建立嗜盐古菌比较分类学体系。
(3) 立足于应用的嗜盐古菌分类学研究。嗜盐古菌产生的独特嗜盐与耐盐酶在含盐体系的生物转化或生物催化中具有重要的应用价值,是常规酶类所不能替代的。
过去几十年的研究大大增加了我们对嗜盐古菌分类学、生理学、生态学以及生物信息学的知识,这一特殊类群潜在的应用价值也促进了研究者进一步对它们的研究和了解。高盐环境的嗜盐古菌承传着不同于真核生物和细菌系统进化世系的“第三种生命形式”,激发了人们对这一类生命现象探索的兴趣。作为特殊的生命形式,嗜盐古菌为生命体对高盐的适应性和生物系统进化等理论研究提供极好的素材;作为高盐环境的重要居住者,嗜盐古菌物种多样性反映了高盐生态系统多样性,对盐环境资源的保护与利用具有重要意义;作为特殊基因及其产物的拥有者和生产者,对嗜盐古菌的研究将为工农业及人类健康的进一步发展提供新的机遇,为生物技术带来革命性进步。
| [1] | Oren A. Halophilic microorganisms and their environments[M]. New York: Kluwer Academic Publishers, 2002 . |
| [2] | Oh D, Porter K, Russ B, et al. Diversity of Haloquadratum and other haloarchaea in three, geographically distant, Australian saltern crystallizer ponds[J]. Extremophiles, 2010, 14 (2): 161–169. |
| [3] | Youssef NH, Ashlock-Savage KN, Elshahed MS. Phylogenetic diversities and community structure of members of the extremely halophilic archaea (Order Halobacteriales) in multiple saline sediment habitats[J]. Applied and Environmental Microbiology, 2012, 78 (5): 1332–1344. |
| [4] | Oren A. Taxonomy of halophilic archaea: current status and future challenges[J]. Extremophiles, 2014, 18 (5): 825–834. |
| [5] | Sorokin DY, Abbas B, Merkel AY, et al. Methanosalsum natronophilum sp. nov., and Methanocalculus alkaliphilus sp. nov., haloalkaliphilic methanogens from hypersaline soda lakes[J]. International Journal of Systematic and Evolutionary Microbiology, 2015, 65 (10): 3739–3745. |
| [6] | Narasingarao P, Podell S, Ugalde JA, et al. De novo assembly reveals abundant novel major lineage of Archaea in hypersaline microbial communities[J]. The ISME Journal, 2012, 6 (1): 81–93. |
| [7] | Martínez-García M, Santos F, Moreno-Paz M, et al. Unveiling viral-host interactions within the ‘microbial dark matter’[J]. Nature Communications, 2014, 5 : 4542. |
| [8] | Baas-Becking LGM. Historical notes on salt and salt-manufacture[J]. Scientific Monthly, 1931, 32 (5): 434–446. |
| [9] | Kocur M, Hodgkiss W. Taxonomic status of the genus Halococcus Schoop[J]. International Journal of Systematic Bacteriology, 1973, 23 (2): 151–156. |
| [10] | Flannery WL. Current status of knowledge of halophilic bacteria[J]. Bacteriology Reviews, 1956, 20 (2): 49–66. |
| [11] | Lochhead AG. Agricultural and industrial bacteriology A14 bacteriological studies of the red discoloration of salted hides[J]. Journal of Bacteriology, 1934, 27 (1): 51–74. |
| [12] | Lochhead AG. Note on the taxonomic position of the red chromogenic halophilic bacteria[J]. Journal of Bacteriology, 1943, 45 (6): 574–575. |
| [13] | Breed RS, Murry EGD, Hithchens AP. Bergey’s Manual of Determinative Bacteriology[M].6th Edition. Baltimore: Williams and Wilkins, 1948 . |
| [14] | Breed RS, Murry EGD, Smith NR. Bergey’s Manual of Determinative Bacteriology[M].7th Edition. Baltimore: Williams and Wilkins, 1957 . |
| [15] | Buchanan RE, Gibbons NE. Bergey’s Manual of Determinative Bacteriology[M].8th Edition. Baltimore: Williams and Wilkins, 1974 . |
| [16] | Staley JT, Bryant MP, Pfennig N, et al. Bergey’s Manual of Systematic Bacteriology[M]. Baltimore: Williams and Wilkins, 1990 . |
| [17] | Boone DR, Castenholz RW. Bergey’s Manual of Systematic Bacteriology[M].2nd Edition. New York: Springer, 2001 . |
| [18] | Citarella RV, Colwell RR. Polyphasic taxonomy of the genus Vibrio: polynucleotide sequence relationships among selected Vibrio Species[J]. Journal of Bacteriology, 1970, 104 (1): 434–442. |
| [19] | Colwell RR. Polyphasic taxonomy of the genus Vibrio: numerical taxonomy of Vibrio cholerae and Vibrio parahaemolyticus, and related Vibrio species[J]. Journal of Bacteriology, 1970, 104 (1): 410–433. |
| [20] | Oren A, Ventosa A, Grant WD. Proposed minimal standards for description of new taxa in the order Halobacteriales[J]. International Journal of Systematic Bacteriology, 1997, 47 (1): 233–238. |
| [21] | Oren A, Arahal DR, Ventosa A. Emended descriptions of genera of the family Halobacteriaceae[J]. International Journal of Systematic and Evolutionary Microbiology, 2009, 59 (3): 637–642. |
| [22] | Burns DG, Janssen PH, Itoh T, et al. Haloquadratum walsbyi gen. nov., sp. nov., the square haloarchaeon of Walsby, isolated from saltern crystallizers in Australia and Spain[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57 (2): 387–392. |
| [23] | Wright ADG. Phylogenetic relationships within the orderHalobacterialesinferred from 16S rRNA gene sequences[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 56 (6): 1223–1227. |
| [24] | Enache M, Itoh T, Fukushima T, et al. Phylogenetic relationships within the familyHalobacteriaceaeinferred from rpoB′ gene and protein sequences[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57 (10): 2289–2295. |
| [25] | Minegishi H, Kamekura M, Itoh T, et al. Further refinement of the phylogeny of the Halobacteriaceae based on the full-length RNA polymerase subunit B′ (rpoB′) gene[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60 (10): 2398–2408. |
| [26] | Papke RT, White E, Reddy P, et al. A multilocus sequence analysis approach to the phylogeny and taxonomy of the Halobacteriales[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61 (12): 2984–2995. |
| [27] | Wayne LG, Brenner DJ, Colwell RR, et al. Report of the ad hoc committee on reconciliation of approaches to bacterial systematics[J]. International Journal of Systematic Bacteriology, 1987, 37 (4): 463–464. |
| [28] | Stackebrandt E, Goebel BM. Taxonomic note: a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J]. International Journal of Systematic Bacteriology, 1994, 44 (4): 846–849. |
| [29] | Meier-Kolthoff JP, Göker M, Spröer C, et al. When should a DDH experiment be mandatory in microbial taxonomy?[J]. Archives of Microbiology, 2013, 195 (6): 413–418. |
| [30] | Goris J, Konstantinidis KT, Klappenbach JA, et al. DNA-DNA hybridization values and their relationship to whole-genome sequence similarities[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57 (1): 81–91. |
| [31] | Kim M, Oh HS, Park SC, et al. Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64 (2): 346–351. |
| [32] | Gupta RS, Naushad S, Baker S. Phylogenomic analyses and molecular signatures for the class Halobacteria and its two major clades: a proposal for division of the class Halobacteria into an emended order Halobacterialesand two new orders, Haloferacales ord. nov. and Natrialbales ord. nov, containing the novel families Haloferacaceae fam. nov. and Natrialbaceae fam. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 2015, 65 (3): 1050–1069. |
| [33] | Cui HL, Zhang WJ. Salinigranum rubrum gen. nov., sp. nov., a new member of the family Halobacteriaceae isolated from a marine solar saltern[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64 (6): 2029–2033. |
| [34] | Walsby AE. A square bacterium[J]. Nature, 1980, 283 (5742): 69–71. |
| [35] | Bolhuis H, te Poele EM, Rodriguez-Valera F. Isolation and cultivation of Walsby’s square archaeon[J]. Environmental Microbiology, 2004, 6 (12): 1287–1291. |
| [36] | Burns DG, Camakaris HM, Janssen PH, et al. Cultivation of Walsby’s square haloarchaeon[J]. FEMS Microbiology Letters, 2004, 238 (2): 469–473. |
| [37] | Cui HL, Yang X, Mou YZ. Salinarchaeum laminariae gen. nov., sp. nov.: a new member of the family Halobacteriaceae isolated from salted brown alga Laminaria[J]. Extremophiles, 2011, 15 (6): 625–631. |
| [38] | Cui HL, Zhou PJ, Oren A, et al. Intraspecific polymorphism of 16S rRNA genes in two halophilic archaeal genera, Haloarcula and Halomicrobium[J]. Extremophiles, 2009, 13 (1): 31–37. |
| [39] | Tindall BJ. Taxonomic problems arising in the genera Haloterrigena and Natrinema[J]. International Journal of Systematic and Evolutionary Microbiology, 2003, 53 (5): 1697–1698. |
| [40] | Castillo AM, Gutiérrez MC, Kamekura M, et al. Natrinema ejinorense sp. nov., isolated from a saline lake in Inner Mongolia, China[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 56 (11): 2683–2687. |
| [41] | Gutiérrez MC, Castillo AM, Corral P, et al. Natronorubrum sediminis sp. nov., an archaeon isolated from a saline lake[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60 (8): 1802–1806. |
| [42] | Savage KN, Krumholz LR, Oren A, et al. Halosarcina pallida gen. nov., sp. nov., a halophilic archaeon from a low-salt, sulfide-rich spring[J]. International Journal of Systematic and Evolutionary Microbiology, 2008, 58 (4): 856–860. |
| [43] | Qiu XX, Zhao ML, Han D, et al. Taxonomic study of the genera Halogeometricum and Halosarcina: transfer of Halosarcina limi and Halosarcina pallida to the genus Halogeometricumas Halogeometricum limi comb. nov. and Halogeometricum pallidumcomb. nov., respectively[J]. International Journal of Systematic and Evolutionary Microbiology, 2013, 63 (10): 3915–3919. |
| [44] | Pagaling E, Wang HZ, Venables M, et al. Microbial biogeography of six salt lakes in Inner Mongolia, China, and a salt lake in Argentina[J]. Applied and Environmental Microbiology, 2009, 75 (18): 5750–5760. |
| [45] | Sun DL, Jiang X, Wu QL, et al. Intragenomic heterogeneity of 16S rRNA genes causes overestimation of prokaryotic diversity[J]. Applied and Environmental Microbiology, 2013, 79 (19): 5962–5969. |
| [46] | Wang DZ, Zhou PJ, Tian XY, et al. Identification of new species of extreme halophilic bacteria[J]. Acta Microbiologica Sinica, 1984, 24 (4): 304–309. (in chinese) 王大珍, 周培瑾, 田新玉, 等. 极端嗜盐菌新种的鉴定[J]. 微生物学报, 1984, 24 (4):304–309. |
| [47] | Zhang JZ, Huang JJ, Xu DQ, et al. A new species of Halobacterium[J]. Acta Microbiologica Sinica, 1990, 30 (2): 87–93. (in chinese) 张纪忠, 黄静娟, 徐德强, 等. 嗜盐杆菌属一新种[J]. 微生物学报, 1990, 30 (2):87–93. |
| [48] | Zhou PJ, Tian XY, Ma YH, et al. An extreme haloalkaligenous bacterium[J]. Acta Microbiologica Sinica, 1990, 30 (1): 1–6. (in chinese) 周培瑾, 田新玉, 马延和, 等. 一株产碱极端嗜盐杆菌[J]. 微生物学报, 1990, 30 (1):1–6. |
| [49] | Zhou PJ, Xu Y, Xiao CS, et al. New species of Haloarcula[J]. Acta Microbiologica Sinica, 1994, 34 (2): 89–95. (in chinese) 周培瑾, 徐毅, 肖昌松, 等. 嗜盐小盒菌属新种的鉴定[J]. 微生物学报, 1994, 34 (2):89–95. |
| [50] | Tian XY, Xu Y, Liu HC, et al. New species of Natronobacterium[J]. Acta Microbiologica Sinica, 1997, 37 (1): 1–6. (in chinese) 田新玉, 徐毅, 刘洪灿, 等. 嗜盐嗜碱杆菌属的一个新种[J]. 微生物学报, 1997, 37 (1):1–6. |
| [51] | Wang ZX, Xu Y, Zhou PJ. Taxonomy of a new species of haloalkalophilic archaeon[J]. Acta Microbiologica Sinica, 2000, 40 (2): 115–120. (in chinese) 王振雄, 徐毅, 周培瑾. 嗜盐碱古生菌新种的系统分类学研究[J]. 微生物学报, 2000, 40 (2):115–120. |
| [52] | Cui HL, Dai CH, Dong Y, et al. Identification of a bacterioruberin-producing microorganism and analysis of its pigment[J]. Marine Sciences, 2005, 29 (2): 43–48. (in chinese) 崔恒林, 代春华, 董英, 等. 一株菌红素产生菌的鉴定及色素组分分析[J]. 海洋科学, 2005, 29 (2):43–48. |
| [53] | Xu Y, Zhou PJ, Tian XY. Characterization of two novel haloalkaliphilic archaea Natronorubrum bangense gen. nov., sp. nov. and Natronorubrum tibetense gen. nov., sp. nov.[J]. International Journal of Systematic Bacteriology, 1999, 49 (1): 261–266. |
| [54] | Xu Y, Wang ZX, Xue Y, et al. Natrialba hulunbeirensis sp. nov. and Natrialba chahannaoensis sp. nov., novel haloalkaliphilic archaea from soda lakes in Inner Mongolia Autonomous Region, China[J]. International Journal of Systematic and Evolutionary Microbiology, 2001, 51 (5): 1693–1698. |
| [55] | Corral P, Corcelli A, Ventosa A. Halostagnicola bangensis sp. nov., an alkaliphilic haloarchaeon from a soda lake[J]. International Journal of Systematic and Evolutionary Microbiology, 2015, 65 (3): 754–759. |
| [56] | Sorokin DY, Kublanov IV, Gavrilov SN, et al. Elemental sulfur and acetate can support life of a novel strictly anaerobic haloarchaeon[J]. The ISME Journal, 2015, 10 (1): 240–252. |
 2016, Vol. 43
2016, Vol. 43